Method Article
Genoma Ingegneria mouse usando Nucleases Designer
In questo articolo
Riepilogo
Nucleasi di stilisti come nucleasi dito di zinco (ZFNs) e attivatore di trascrizione come nucleases effettrici (Talens) possono essere utilizzati per modificare il genoma di embrioni preimpianto di topo attivando sia la fine nonhomologous giunzione (NHEJ) e la ricombinazione omologa (HR) percorsi. Questi progressi consentono la rapida generazione di mouse con precise modificazioni genetiche.
Abstract
Topi transgenici portatori modificazioni sito-specifiche genoma (knockout, knock-in) sono di importanza vitale per sezionare sistemi biologici complessi nonché per la modellazione malattie umane e testare strategie terapeutiche. I recenti progressi nell'uso di nucleasi design come nucleasi dito di zinco (ZFNs), trascrizione attivatore-come nucleases effettrici (Talens), ed i cluster brevi ripetizioni palindromiche regolarmente intervallati (CRISPR) / CRISPR-associato (CAS) Sistema 9 per il sito- specifico ingegneria genomica aperta la possibilità di effettuare una rapida modificazione genoma mirata in virtualmente qualsiasi specie di laboratorio senza la necessità di fare affidamento su staminali embrionali (ES) tecnologia cellulare. Un esperimento editing genoma inizia tipicamente con l'identificazione di design nucleasi siti bersaglio all'interno di un gene di interesse seguita dalla costruzione di domini leganti il DNA personalizzati per dirigere l'attività nucleasi al locus genomico investigatore definito. Plasmidi Designer nucleasi sono in vitro </ Em> trascritto per produrre mRNA per la microiniezione degli ovociti di topo fecondati. Qui, forniamo un protocollo per realizzare la modifica del genoma di mira da iniezione diretta di TALEN mRNA in oociti di topo fecondati.
Introduzione
I topi sono di gran lunga la piattaforma più popolare per la generazione di modelli animali transgenici. Il toolbox versatile per l'ingegneria genetica dell'embrione topo 1-3 è stato recentemente prorogato di approcci di modifica del genoma basato su nucleases stilisti come nucleasi dito di zinco (ZFN) 4-6, trascrizione nucleases effettrici attivatore-like (Talen) 7,8, ed i cluster brevi ripetizioni palindromiche regolarmente intervallati (CRISPR) / Sistema CRISPR-associato (CAS) 9 9. ZFN e la funzione TALEN come coppie di due domini progettati su misura a base di proteine che legano il DNA (array di proteine dita di zinco e ripetere variabile di-residui (RVDS), rispettivamente) che sono ciascuno accoppiato al endonucleasi FokI 10-12. Viceversa, la specificità di taglio del DNA Cas9-mediata è fornito da transattivazione RNA CRISPR (crRNA e tracrRNA, che possono anche essere combinati in una singola molecola di RNA chimerico guida definito RNA) 11 che agiscono in un complesso con l'Proteine CRISPR.
Talens con una sequenza definita di RVDS può essere rapidamente costruito dagli sperimentatori individuali con una moltitudine di strategie di assemblaggio scegliere 13-17. CRISPR/Cas9 promette ancor meno generazione ad alta intensità di manodopera di nucleasi di design, tuttavia la specificità della guida RNA-DNA-binding non è ancora completamente risolto 18,19. Generazione di ZFNs misura è stata finora limitata a laboratori accademici specializzati e fornitori commerciali come Sangamo Biosciences e il servizio Sigma CompoZr.
In generale, genome editing con nucleasi design mira a introdurre doppie rotture del filamento (DSB) a loci genomici definito, che poi attirano fine nonhomologous giunzione (NHEJ) o ricombinazione omologa (HR) di riparazione del DNA macchinari 10,12. Riparazione NHEJ-mediata di un DSB provoca spesso l'introduzione di inserzioni e delezioni in prossimità del sito di riparazione. Così NHEJ riparazione cun essere sfruttati per buttare giù la funzione di un gene bersaglio introducendo una mutazione frame-shift ai geni codificanti proteine sequenza 4,7,9. In alternativa, aggiunta o sostituzione di informazioni genetiche definite possono essere raggiunti realizzando un donatore DNA insieme alle nucleasi progettista. Un donatore di DNA comprende ricercatore-progettato sequenze di DNA fiancheggiati da regioni di omologia con il locus bersaglio, quindi servire come modello per la riparazione DSB da HR. Entrambi i plasmidi 5,6,20 e singolo filamento oligonucleotidi 8,9,21 sono stati utilizzati con successo come donatori. Né-NHEJ né HR-mediata editing genoma richiedono l'introduzione di un marcatore selezionabile nel genoma dell'embrione mouse, che rende queste strategie particolarmente adatto per la creazione di piccole alterazioni nella sequenza nucleotidica senza disturbare l'architettura genetica complessiva.
In questo protocollo si descrivono tutte le procedure essenziali per l'editing genoma nellaembrione di topo usando Talens. Questi includono 1) individuazione di un sito di destinazione TALEN 22, 2) la costruzione di Talens dal Golden Gate clonazione 13, 3) la sintesi in vitro di TALEN mRNA, 4) la microiniezione di mRNA TALEN in ovociti di topo fecondati, 5) le procedure chirurgiche per il trasferimento di embrioni e 6) analisi di mutagenesi TALEN indotta in animali fondatori. Ci concentriamo su TALEN mRNA microiniezione e lo screening dei fondatori per NHEJ-indotte inserzioni / delezioni. A questo scopo abbiamo generato bifunzionali costrutti Talen che permettono sia l'espressione in cellule di mammifero, quando trasfettate come plasmidi e nella sintesi in vitro di TALEN mRNA per microiniezione in embrioni di topo. Questi costrutti comprendono una spina dorsale RACCONTO troncato 23 fusi per eterodimerica FokI domini 24,25 per ottimizzare la modifica del genoma nelle cellule di mammifero. Questo protocollo può anche essere adottata per microiniezione di altre nucleasi di design o per le iniezioni combinate di nucleasi di designe costrutti donatori (disegno di donatori di DNA è stata descritta in eccellenti pubblicazioni tecniche Wefers et al. 26,27).
Dichiarazione etica
Tutti gli esperimenti sugli animali sono stati eseguiti in conformità con le linee guida e regolamenti del veterinario cantonale del Canton Zurigo.
Protocollo
1. Identificazione dei Talen siti bersaglio
- Visitare il sito Web 2.0 TAL Effettuatore Nucleotide targeter (http://tale-nt.cac.cornell.edu) e scegliere "TALEN targeter".
- Inserire la sequenza del gene bersaglio. Se l'espressione pCAG-T7-TALEN costruisce viene utilizzato (Figura 1C) scegliere "Miller et al., 2011" sotto "Usa un architettura Preset" al fine di prevedere siti di destinazione che possono essere avviati con la lunghezza ottimale distanziale (15-20 bp) e il numero di RVDS Tale (15-20) per questa architettura TALEN.
- Scegliere "NN" sotto "G Substitute" (NH RVDS specifico per guanosina sono inoltre disponibili ma non ancora ampiamente testati per il montaggio TALEN).
- Facoltativo: per altre impostazioni e opzioni seguire le istruzioni sul sito e link forniti qui.
- Un file di testo con i potenziali siti bersaglio viene generato, che può essereimportato in un foglio di calcolo per l'osservazione più conveniente. Selezionare la coppia TALEN (s) per Golden Gate montaggio.
- Facoltativo: Sequenza della regione genomica di interesse nel ceppo di topi che verrà utilizzato per la microiniezione per rilevare eventuali polimorfismi a singolo nucleotide (SNPs) che potrebbero non essere contabilizzati in banche dati pubbliche e potrebbe potenzialmente impedire nucleasi vincolante.
- Opzionale: Fare riferimento alla Tabella 3 per design aggiuntive nucleasi-relative risorse online.
- La maggior parte dei plasmidi necessari per ZFN e TALEN costruzione possono essere ottenuti da Addgene:
http://www.addgene.org/special-collections/
Addgene ID per il kit Golden Gate TALEN e mammiferi costrutti di espressione Talen sono riportati nella Tabella 2.
In alternativa, moduli particolari funzionali come i domini zinc finger possono essere ordinati da un fornitore di servizi di sintesi del gene.
2. Golden Gate TALEN Assembly
Questa sezione descrive l'assemblaggio di Talens utilizzando un protocollo pubblicato da Cermak et al. Talens 13 lunghi sono costruiti utilizzando due successive fasi di clonazione Golden Gate (Figura 1). Questo approccio consente l'incorporazione di un numero qualsiasi di RVDS tra 12 e 31 nei costrutti di espressione finale. Il protocollo assemblea è stato adattato a vettori di destinazione progettato per la generazione di mRNA (Figura 1C) che esprimono Talens altamente attivi seguenti ovocita microiniezione. Si prega di fare riferimento anche al protocollo on line del kit Golden Gate TALEN per stabilire e mantenere la biblioteca plasmide ( http://www.addgene.org/TALeffector/goldengateV2/ ).
- Giorno 1 - Golden Gate Reaction # 1 - Assemblea dei RVD Array in pFUS Vettori
- Opzionale: Per ogni coppia TALEN progettato nel Protocol 1, immettere la sequenza del DNA dei due siti bersaglio (compresi iniziale T, 5 'a 3') nel foglio TALEN_Voytas_Pipetting per generare uno schema di pipettamento per ciascuna reazione Golden Gate (inserire anche concentrazioni di tutti plasmidi utilizzati per gli assembly ). Il foglio di calcolo fornisce anche la sequenza di DNA atteso di tutti RVDS che possono essere utilizzati per l'allineamento dei risultati sequenziamento al punto 2.5.2.
- Per ogni unica TALEN progettato nel protocollo 1:
Se la lunghezza TALEN è 12-21 (standard), selezionare Ripetizione variabile di-residui (RVDS) 1-10 e la destinazione vettore pFUS_A. Selezionare restanti RVDS e una destinazione vettore pFUS_B, ad esempio per un TALEN con 15 RVDS (tra cui l'ultima ripetizione, che verrà aggiunta al montaggio finale al punto 2.3.3, selezionare RVDS 11-14 e pFUS_B4 destinazione vettore (Figura 1A).
Se la lunghezza TALEN è 22-31, uso RVDS 1-10 e destinazione vector pFUS_A30A. Scegli RVDS 11-20 e la destinazione vettore pFUS_A30B. Selezionare restanti RVDS e un vettore pFUS_Bdestination, ad esempio per TALEN con 24 RVDS raccogliere RVDS 21-23 e la destinazione vettore pFUS_B3. - Impostare Golden Gate reazione # 1 per ogni pFUS + combinazione RVD separatamente, vale a dire 1-10 + pFUS_A e restanti RVDS + rispettivo vettore pFUS_B o per TALEN più di 21 RVDS, 1-10 + pFUS_A30A, 11-20 pFUS_A30B e le restanti RVDS + rispettivo vettore pFUS_B. Utilizzare 150 ng di ciascun vettore RVD, 150 ng di pFUS vettore, 1 ml BsaI, 1 ml T4 DNA ligasi, 2 microlitri di buffer 10x T4 DNA ligasi, e H 2 O a 20 microlitri di volume totale di reazione. (Utilizzo di aliquote freschi di T4 ligasi tampone per ogni turno di assembly Golden Gate è raccomandato in quanto ripetuto scongelamento / congelamento del tampone di ligasi T4 può ridurre l'efficienza delle reazioni.)
- Posizionare le reazioni Golden Gate in un cycler termico.
Programma:
37 ° C 5 min
16 ° C 10 min
10 cicli
50 ° C 5 min
80 ° C 5 min - Aggiungere 1 ml ATP 10 mM e 1 ml Plasmid-Safe nucleasi per ciascuna miscela e incubare a 37 ° C per 1 ora. Questo trattamento rimuove prodotti di ligazione incompleti lineari che possono essere clonati in vettori di destinazione in vivo ricombinazione omologa in batteri trasformati.
- Trasforma E. coli con reazioni individuali ligazione (elettrocompetenti o chimicamente competente E. coli che facilitano α-complementazione come XL1-Blue o DH5a può essere usato qui e nelle trasformazioni successive).
- Batteri piastra su spectinomicina (50 mg / ml) piastre con X-Gal e IPTG (40 mcg / ml) per il blu / selezione colonia bianca.
- Giorno 2 - Conferma del corretto montaggio pFUS-RVDS
- Utilizzando colonia PCR con primer pCR8_F1 e pCR8_R1 (Vedi Tabella 1 per sequenze primer) schermo 1-3 whitecolonie di ogni piatto. Correggere pFUS-RVDS assemblaggi in genere mostrano una banda corrispondente alla lunghezza combinata di tutti RVDS clonati (ad esempio circa 1,1 kb per 10 RVDS) e una "scala" dei piccoli gruppi meno prominenti (Figura 1B).
Programma PCR:
95 ° C 3 min
95 ° C 30 sec
55 ° C 30 sec
72 ° C 1 min 45 sec
30-35 cicli
72 ° C 10 min - Utilizzare confermato cloni per iniziare cultura durante la notte (2-5 ml LB con 50 mg / ml spectinomicina).
- Utilizzando colonia PCR con primer pCR8_F1 e pCR8_R1 (Vedi Tabella 1 per sequenze primer) schermo 1-3 whitecolonie di ogni piatto. Correggere pFUS-RVDS assemblaggi in genere mostrano una banda corrispondente alla lunghezza combinata di tutti RVDS clonati (ad esempio circa 1,1 kb per 10 RVDS) e una "scala" dei piccoli gruppi meno prominenti (Figura 1B).
- Giorno 3 - Golden Gate Reazione # 2 - Array RVD in Talen vettori di espressione
- Eseguire "minipreps" per isolare i gruppi pFUS-RVD (a seconda del numero di RVDS sia pFUS_A e pFUS_B o pFUS_A30A, pFUS_A30B e pFUS_B).
- Optional: Sequenza singoli vettori pFUS utilizzando primer pCR8_F1, pCR8_R1 (vedi Tabella 1 per le sequenze di primer). Sequencing può anche essere effettuata sul finale TALEN costruisce (sezione 2.5.2); tuttavia, per più Talens completa legge di tutti RVDS potrebbe non essere possibile utilizzando il sequenziamento Sanger.
- Impostare Golden Gate reazione # 2 per ogni singolo TALEN. 150 ng di ciascun vettore pFUS, 150 ng di PLR-HD, PLR-NG, PLR-NI, PLR-NN (ultimo "mezzo-repeat"), secondo il disegno della sequenza RVD e per l eft TALEN utilizzare 75 ng di pCAG-T7-TALEN-ELD-destinazione e per il diritto TALEN utilizzare 75 ng pCAG-T7-TALEN-KKR-destinazione (o viceversa). Aggiungere 1 ml Esp3I, 1 ml T4 DNA ligasi, 2 microlitri di buffer 10x T4 DNA ligasi, H 2 O a 20 microlitri volume totale di reazione.
- Posizionare le reazioni Golden Gate in un cycler termico.
Programma:
37 ° C 5 min
16 ° C 10 min
10 cicli
37 ° C 15 min
80 ° C 5 min - Utilizzare reazioni sezione 2.3.4 per trasformare E. coli.
- Giorno 4 - Conferma del corretto montaggio TALEN
- Schermo 1-3 colonie bianche da ogni piastra da colonia PCR con primer TAL_F1 e TAL_R2 (cfr. tabella 1 per le sequenze di primer). Talens correttamente assemblati mostrano un prodotto di PCR con una lunghezza corrispondente al numero totale di RVDS Incorporated (Figura 1D, questa banda è talvolta difficile da rilevare, mentre l'effetto "scaletta" rappresenta un indicatore robusto di montaggio successo).
Programma PCR:
95 ° C 3 min
95 ° C 30 sec
55 ° C 30 sec
72 ° C 3 min
30-35 cicli
72 ° C 10 min - Usa confermato cloni corretti per avviare colture batteriche durante la notte (2-5 ml LB con 100 mg / ml ampicillina).
- Schermo 1-3 colonie bianche da ogni piastra da colonia PCR con primer TAL_F1 e TAL_R2 (cfr. tabella 1 per le sequenze di primer). Talens correttamente assemblati mostrano un prodotto di PCR con una lunghezza corrispondente al numero totale di RVDS Incorporated (Figura 1D, questa banda è talvolta difficile da rilevare, mentre l'effetto "scaletta" rappresenta un indicatore robusto di montaggio successo).
- Giorno 5 - Conferma del corretto montaggio TALEN
- Eseguire "minipreps" per isolare i plasmidi pCAG-T7-ta.
- Se sequenziamento non è stata eseguita in séction 2.3.2, utilizzare primer TAL_Seq_5-1 e TAL_R2 (vedi Tabella 1 per le sequenze dei primer) per determinare il montaggio RVD corretto in full-length TALEN.
3. Nuclease mRNA sintesi
- Generazione di modello di DNA
- Preparare midi o maxipreps di plasmidi pCAG-T7-ta per la sintesi di mRNA di alta qualità.
- Linearizzare 10 pg del TALEN o ZFN mRNA sintesi plasmide con un enzima di restrizione che preferenzialmente fende valle e in prossimità del codone di STOP nucleasi (per vettori pCAG-T7-ta utilizzano SacI). plasmidi sintesi di mRNA tipicamente includono un promotore T7 o SP6 fago monte della sequenza codificante nucleasi.
- Eseguire 200-500 ng di plasmide digerito su un gel di agarosio 0,7-1% per verificare la completa digestione.
- Rimuovere sali dal plasmide digerire facendo precipitare il DNA con acetato di sodio 0,1 volumi e 3 volumi di etanolo per 1 ora a camera temperature. Sedimentare il DNA mediante centrifugazione a 14.000 xg per 10 o più minuti, lavare il pellet con 200 ml di etanolo 70%, centrifugare per altri 5 minuti, rimuovere l'etanolo, asciugare il pellet e risospendere in un volume adeguato di acqua priva di RNasi. La purificazione del plasmide linearizzato è possibile anche utilizzando un sistema basato su colonne, ad esempio QIAquick PCR Purification Kit.
- Determinare la concentrazione del modello lineare e utilizzando 1 mg per impostare trascrizione in vitro.
- mRNA Sintesi e poliadenilazione
- Per la trascrizione in vitro di pCAG-T7-TALEN plasmidi usa il mMESSAGEmMACHINE T7 Ultra Kit. Impostare la reazione di trascrizione per ogni TALEN sul ghiaccio: a 20 ml con acqua priva di nucleasi, 10 microlitri T7 2x NTP / ARCA, 2 ml 10x T7 Reaction Buffer, 1 mg di DNA stampo, 2 microlitri T7 mix di enzimi. Mescolare la reazione e incubare per 1-2 ore a 37 ° C.
- Utilizzare la completa trascrizione 20 microlitrizione miscela di reazione per impostare la reazione di poliadenilazione su ghiaccio: 36 ml di acqua priva di nucleasi, 20 microlitri di buffer 5x EPAP, 10 microlitri 25 MnCl2 mM, ATP 10 microlitri 10 mm. Mescolare e togliere 2,5 microlitri della miscela di reazione come campioni di controllo L1 ed R1 (per nucleasi sinistra e destra, rispettivamente). Aggiungere 4 ml di E-PAP enzima e incubare la reazione per 45-60 minuti a 37 ° C. Rimuovere altri 2,5 ml di miscela di reazione come campioni di controllo L2 e R2.
- mRNA Purificazione
- Utilizzare NucAway Colonne Spin per la purificazione di mRNA (scambio di buffer e la rimozione di nucleotidi prive di personalità giuridica). Toccare le colonne di stabilirsi gel secco in fondo della colonna. Colonna Idratare con 650 ml di buffer di RNase-free microiniezione (1 mM Tris-Cl, 0.1 EDTA, pH 7.5). Cap, vortice, toccare le bolle d'aria, e idratare per 5-15 minuti a temperatura ambiente.
- Posizionare la colonna in un tubo di raccolta e centrifugaa 750 xg, a 4 ° C per 2 minuti per rimuovere fluido interstiziale in eccesso. Eliminare colonna in tubo di raccolta e posto in una provetta 1,5 ml di eluizione.
- Applicare la miscela di reazione completo dal punto 3.2.2 alla colonna e centrifugare a 750 xg, 4 ° C per 2 min. Nucleasi mRNA verrà ora disciolto in tampone microiniezione. Rimuovere 2,5 ml di campioni purificati L3 e R3.
- Conservare mRNA a -80 ° C fino aliquote microiniezione sono preparati.
- mRNA elettroforesi su gel
- Pulire una camera di gel per rimuovere le contaminazioni RNasi con la soluzione di SDS 10% o RNaseZAP.
- Preparare un gel di agarosio 1% in 1xTBE tampone di corsa.
- Mescolare ogni L1/R1 campione di RNA, L2/R2, L3/R3 con 3 volumi di NorthernMax formaldeide Load Dye e incubare per 15 minuti a 65 ° C.
- Caricare i campioni e una scala di dimensioni RNA (formato marcatore es RNA Millennium) sul geled eseguire gel a 10 V / cm in TBE 1x fino loading dye raggiunge la fine del gel.
- Macchiare il gel usando soluzione verde SYBR (Invitrogen) per 30-60 minuti con agitazione a temperatura ambiente.
- Immagine il gel e confrontare le dimensioni di L1/R1 con L2/R2 e L3/R3. I campioni L2/R2 e L3/R3 dovrebbero mostrare nastri di dimensione maggiore rispetto al L1/R1 indicando poliadenilazione successo (Figura 2).
- Determinare la concentrazione di mRNA utilizzando uno spettrofotometro.
- Preparare aliquote mRNA per microiniezioni mescolando nuclease sinistra e mRNA nucleasi destra in un rapporto di 1:1. Si consiglia di preparare aliquote con una concentrazione totale di 200 ng / mL (100 ng / ml di ogni nucleasi) diluendo con tampone microiniezione. Conservare mRNA aliquote a -80 ° C.
4. Isolamento dell'embrione e microiniezione
- Isolamento embrione. Questo protocollo è stato utilizzato con successo con C57BL/6J e la tensione BDF1s e può probabilmente essere adattato ad altri ceppi comunemente usati in esperimenti di microiniezione come FVB o CBF1. I topi sono alloggiati sotto una ciclo luce-buio 12 hr-12 ore in un impianto temperatura e umidità controllata con cibo e acqua ad libitum.
- Donatrici Superovulate per aumentare il rendimento degli embrioni. 16 femmine (4 settimane) sono superovulate da intraperitoneale (ip) iniezione di gonadotropina siero del 5 IU cavalla gravida (PMSG), seguito da iniezione ip su 5 UI di gonadotropina corionica umana (hCG), 48 ore più tardi.
- Mate i 16 femmine superovulate a 16 anni di allevamento (2-8 mesi) maschi subito dopo l'iniezione di hCG.
- Sacrifica le femmine utilizzando un protocollo eutanasia riconosciuto come CO 2 inalazione.
- Recuperare gli ovociti fecondati. Ovociti vengono raccolti da ovidotti la mattina successiva e poi liberati da tutte le cellule del cumulo rimanenti da un trattamento di 3-5 min a 0,1% ialuronidasi bovina disciolto in M2 medio. A seconda della capacità di accoppiamento e il ceppo utilizzato il rendimento embrione può variare. 16 femmine C57BL/6J solito producono 150-250, mentre BDF1 cede 300-400 ovociti fecondati, rispettivamente.
- Embrione microiniezione
- Iniettare il nucleasi mRNA. Ovociti stadio pronucleari sono tipicamente iniettati nel primo pomeriggio. Citoplasmatica mRNA microiniezione viene effettuata in M2 liquido sotto olio minerale usando un microscopio invertito dotato Nomarski DIC usando obiettivo 20X e con micromanipolatori embrioni nonché un gruppo di iniezione. Si consiglia di iniziare le iniezioni di TALEN mRNA ad una concentrazione di 10 ng / ml (20 ng / ml in totale).
- Aspirare il ovocita con un capillare detenzione e iniettare il mRNA nucleasi nel citoplasma dell'embrione evitando il contatto con il pronuclei. L'iniezione deve essere superficiale, vicino alla membrana plasmatica. Il volume di iniezione deve essere mantenuto basso e l'ago microiniezione dovrebbe essere withdrawn al primo segno di distensione citoplasmatica. Una serie microiniezione formata di norma da 100-300 ovociti. Tipicamente, almeno il 80-90% degli embrioni dovrebbe sopravvivere l'iniezione senza lisi immediato.
- A seguito di microiniezione, posizionare gli embrioni per 1 ora a M16 (Sigma) medium a 37 ° C e 5% di CO 2 e trasferire gli embrioni sopravvissuti in pseudopregnant madri adottive.
5. Surgical Embryo Transfer
- Preparare i destinatari embrione pseudopregnant (madri affidatarie) un giorno prima mRNA microiniezione. Femmine mature (3-6 mesi) di un ceppo outbred robusto, come CD-1, è adatto per il ruolo. Indurre pseudogravidanza con l'accoppiamento le femmine in un giorno precedente con i maschi chirurgicamente o geneticamente vasectomized 28. Solo 0,5 femmine DPC con una spina copula chiaro sono utilizzati come embrioni destinatari. Nelle femmine non utilizzate la pseudogravidanza scompare dopo circa 3 weeks permettono il loro uso ripetuto.
- Anestetizzare il 0,5 DPC affidatari femmine madre mediante iniezione ip di ketamina, xylazina (120 mg / kg e 16 mg / kg, rispettivamente). Questa formulazione garantisce ~ 30 min tempo di tolleranza chirurgica è più che sufficiente per il tempo di funzionamento previsto di 5-10 min.
- Posizionare l'animale anestetizzato sull'addome su una superficie calda e disinfettare la zona di incisione con un disinfettante adeguato, come il 70% di etanolo e clorexidina o miscela alcol.
- Incidere la pelle sul dorso dell'animale e aprire la cavità peritoneale con forbici sterili.
- Visualizzare e esternare il corno uterino tirando il cuscinetto adiposo attaccato al ovaio. Tenere gli organi umido con soluzione calda 0,9% NaCl. Tenete a mente che il sito chirurgico può essere drappeggiato con garza sterile o rasato al fine di conformarsi alle linee guida veterinari locali.
- Immobilizzare gli organi riproduttivi dal ritaglio del cuscinetto adiposo ovario con una fascetta bulldog.
- Afferrare nell'ovidotto appena a monte del ampolla con una pinza Orologiai e usare un ago ipodermico di 30 G per creare un piccolo buco nel muro ovidotto.
- Ritirare l'ago e reinserire un capillare di vetro sottile che contiene gli embrioni nel foro che permette il posizionamento degli embrioni nella ampolla soffiando delicatamente nel boccaglio del supporto capillare. Aspirare il capillare volta gli embrioni vengono depositati nel ampolla e sostituire gli organi riproduttivi indietro nella cavità del corpo.
- Chiudere la cavità peritoneale con una serie di suture sterili (Prolene 6-0).
- Chiudere la pelle con 1-2 clip ferita (Autoclip 9 mm) a seconda delle dimensioni dell'apertura.
- A seguito dell'operazione tornare gli animali per la loro gabbia casa e supervisionare loro fino a quando l'effetto dell'anestesia svanire.
- Al fine di minimizzare lo stress, ospitare i topi in gruppi sociali stabili (2-4 animali / tipo III gabbia), quando possibile.
- Applicare analgesico post-operatorios in forma di Dafalgan aggiunti all'acqua potabile (paracetamolo 200 mg / kg di peso corporeo) per 3 giorni dopo l'intervento.
6. Analisi dei Fondatori mediante PCR e T7 Endonuclease o enzimi di restrizione
- Primer design per amplificare una regione tra 200-700 bp intorno al sito di legame nucleasi. Le distanze avanti regione spacer primer nucleasi e nucleasi spacer regione-reverse primer dovrà essere sufficientemente differente per permettere il rilevamento di due bande separate per Prodotti digeriti su un gel di agarosio (vedi figure 3 e 4 per gli esempi).
- Eseguire PCR con condizioni ottimizzate. Controllare dimensione amplicone PCR su un gel di agarosio.
- Optional: prodotti Purify PCR, ad esempio con QIAquick PCR Purification Kit per rimuovere i sali e nucleotidi dal mix PCR. Molti enzimi di restrizione sono attivi in campioni PCR integrate con il buffer di enzima di restrizione e purificazione del caso non è necessaria. Abbiamo anche successfuusato lly endonucleasi T7 in tampone QiagenTaq PCR completata con NEBuffer 2.
- T7 endonuclease dosaggio. Mescolare 17 ml di prodotto di PCR con 2 ml di NEBuffer 2 ed eseguire la formazione eteroduplici (Figura 3b) programma in un termociclatore:
95 ° C 2 min
95 ° C a 85 ° C (-2 ° C / sec)
85 ° C a 25 ° C (-0,1 ° C / sec)
4 ° C attesa.
Aggiungere 1 ml di T7 endonucleasi a ciascun campione e incubare a 37 ° C per 20 min. (Il saggio geometra (Transgenomic), che si basa su CEL 1 nucleasi, può essere utilizzato anche qui. Prega di fare riferimento alle istruzioni del produttore.) - Limitazione digest del prodotto della PCR. Mescolare 17 ml di prodotto di PCR con 2 ml di tampone 10x di restrizione enzimatica e 1 ml di enzima di restrizione. Digerire a temperatura appropriata per 1 ora o più.
- T7 endonuclease dosaggio. Mescolare 17 ml di prodotto di PCR con 2 ml di NEBuffer 2 ed eseguire la formazione eteroduplici (Figura 3b) programma in un termociclatore:
- Aggiungere DNA loading dye per campioni e correre su un gel di agarosio al 2% per rilevare digestione distintamodelli per animali wild-type e fondatori portano alleli mutati. Vedere le figure 3 e 4 per i risultati attesi.
- Clone prodotti di PCR dei fondatori positivi per gli alleli mutati di sequenziamento Sanger (ad esempio mediante TA-clonazione in pGEM-T Easyor sequenza direttamente da miscele di prodotti PCR utilizzando sequenziamento di prossima generazione.
Risultati
Abbiamo costruito plasmidi destinazione compatibili con l'assieme dorato GateTALEN pubblicato da Cermak et al. 13 che permettono l'espressione di Talens in cellule di mammifero e in vitro sintesi di mRNA dal promotore T7 fago (Figura 1C). Questi plasmidi carry domains Foki eterodimeriche (mutazioni eld o KKR) oggi dimostrati ridurre gli effetti fuori bersaglio relativamente per omodimeri Foki ea potenziare l'attività scissione rispetto alla prima generazione FokI eterodimeri 25,29. Le reazioni di montaggio Golden gate # 1 e # 2 sono di solito molto efficiente e ogni colonia bianca, quando analizzato da colonia PCR, mostra il modello previsto per il particolare numero di RVDS clonati (Figure 1B e 1D).
Gel di analisi in vitro sintetizzato mRNA (Figura 2) dovrebbe rivelare una singola banda distinguibile con poca o nessuna sbavatura per ogni campione analizzato. Ci dovrebbe essere un cambiamento chiara dimensione tra i campioni L1/R1 e L2/R2, L3/R3, che indica poliadenilazione successo.
Animali fondatori possono essere sottoposti a screening per alleli mutati NHEJ indotte mediante genotipizzazione PCR seguito da digestione T7 endonucleasi (figura 3) o una digestione di restrizione usando un enzima che scinde la sequenza wild type all'interno della regione distanziatore della coppia nucleasi (Figura 4). Il saggio T7 endonucleasi è applicabile a qualsiasi tipo di mutazione indipendentemente dalla sequenza genomica specifica all'interno della regione distanziatore della coppia nucleasi iniettato; tuttavia, si rileva solo i disallineamenti tra i filamenti di DNA in prodotti di PCR eteroduplici. Così, nel raro caso in cui uno dei fondatori porta due alleli mutati in modo identico, i prodotti di PCR potrebbero non mostrare alcun modello T7 digestione. Questa distinzione è tuttavia sempre possibile quando un sito di restrizione specifico si trova all'interno della regione spacer nucleasi che verrà eliminato NHEJIndotta inserzioni / delezioni (Figura 5). Qui, le bande non digerito indicano la presenza di mutazioni, e l'assenza di qualsiasi prodotto digeriti suggerisce fortemente fondatore trasportano mutazioni in entrambi gli alleli del gene targeting (contrassegnati con l'asterisco sono obbligatori in Figura 4b).
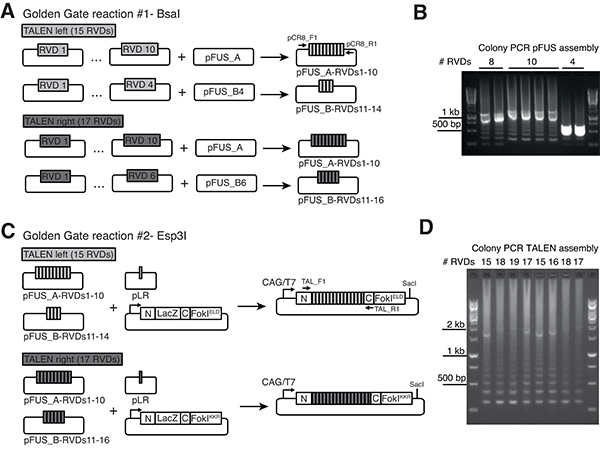
Figura 1. Golden Gate clonazione di Talen RVDS in eterodimeriche vettori pCAG-T7-Destination. A) Assemblea dei RVD matrici in vettori pFUS. Ecco un esempio viene mostrato per una coppia di TALEN con singoli array TALE composto da 15 RVDS e 17 RVDS, rispettivamente (per array TALE più di 21 RVDS, tre assemblee pFUS-RVDS sono necessari, non mostrato). Le frecce indicano primer per b) i prodotti pFUS specifico reazioni colonia PCR PCR.amplificato dalle assemblee pFUS corrette in genere mostrano una banda corrispondente alla durata totale di tutte RVDS clonati (ad esempio circa 1,1 kb per 10 RVDS) e C "scala" dei piccoli gruppi meno importanti a causa della natura ripetitiva di array RVD.) assemblaggio finale di array pFUS-RVD e un plasmide contenente l'ultima ripetizione (PLR) in eterodimeriche vettori di espressione Talen con FokIELD e FokIKKR varianti, rispettivamente. La spina dorsale TALEN (annotato come N e C) ricorda l'architettura pubblicato da Miller et al. 23 Il CAG (CMV elemento presto enhancer / pollo beta-actina) promotore assicura alti livelli di espressione in cellule di mammifero trasfettate, mentre il promotore T7 fago permette in sintesi vitro mRNA (utilizzare SacI per la linearizzazione del vettore a valle del codone di stop nucleasi). D) Colony PCR utilizzando primer indicati dalle frecce C) permette di identificare correttamente montato Talens. Full-lungezzah prodotti di PCR sono spesso meno evidenti, mentre l '"effetto scala" rappresenta un indicatore affidabile di montaggio successo. Clicca qui per vedere l'immagine ingrandita .
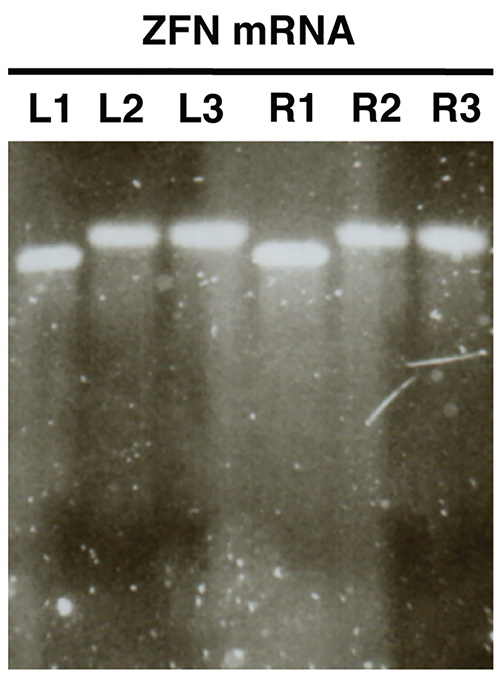
Figura 2. . Controllo di qualità dei nuclease mRNA in sintesi vitro mediante elettroforesi su gel di agarosio mRNA ZFN sono mostrati come esempio (L, sinistra ZFN, R, destra ZFN). I campioni L1/R1 mostrano mRNA prima di poliadenilazione, i campioni L2/R2 spettacolo poliadenilato mRNA e L3/R3 mostra purificato mRNA poliadenilato. Clicca qui per vedere l'immagine ingrandita .
alt = "Figura 3" fo: content-width = "6in" fo: src = "/ files/ftp_upload/50930/50930fig3highres.jpg" src = "/ files/ftp_upload/50930/50930fig3.jpg" width = "600" />
Figura 3. Esempio di endonucleasi saggio T7 utilizzato per l'identificazione di animali fondatori portatori di mutazioni nucleasi indotta del locus bersaglio. A) Una coppia TALEN è stato progettato per fendere all'interno della regione codificante del mouse gene della proteina prionica (PRNP, sequenza bersaglio TALEN può essere fornito su richiesta). Un prodotto di PCR viene generato utilizzando un primer forward (F) situato 110 bp a monte e un primer reverse (R) 250 bp a valle del sito di clivaggio TALEN. B) Il prodotto di PCR viene successivamente sottoposto a heteroduplex formazione e T7 digestione con endonucleasi. C) TALEN mutagenesi indotta all'interno della regione genomica mirata di singoli fondatori è rivelato dalla presenza di un full-length prodotto PCR con prodotti di digestione di 250 e 110 bps.50930/50930fig3highres.jpg "target =" _blank "> Clicca qui per vedere l'immagine ingrandita.

Figura 4. Esempio di una restrizione digest di prodotti di PCR utilizzati per l'identificazione degli animali fondatori portatori di mutazioni nucleasi-indotte del luogo di destinazione. ZFN specifici per il mouse Rosa26 locus 29 bersaglio di un sito di restrizione XbaI all'interno introni 1. A) Fondatori sono stati proiettati per la genotipizzazione PCR utilizzando un primer forward (F) situato a 500 bp a monte e un primer reverse (R) 250 bp a valle del sito di taglio. B) Digestione dei prodotti di PCR con XbaI svela i modelli di digestione che indicano topi con mutazioni bi-alleliche (contrassegnati con asterisco), mono alleliche mutazioni (bande digerito e digerito, ad esempio animali 2)e topi WT (digestione completa, ad esempio animali 21). C) sequenziamento di topi con potenziali modifiche bi-allelica mostra fino a 3 (24 animali) distinti inserzioni / delezioni. Clicca qui per vedere l'immagine ingrandita .
| Nome del Primer | Sequenza 5 'a 3' |
| pCR8_F1 | ttgatgcctggcagttccct |
| pCR8_R1 | cgaaccgaacaggcttatgt |
| TAL_F1 | ttggcgtcggcaaacagtgg |
| TAL_R2 | ggcgacgaggtggtcgttgg |
| TAL_Seq_5-1 | catcgcgcaatgcactgac |
Tabella 1. Sequenze di primer utilizzati per la PCR e sequenziamento delle colonie all'interno del gruppo di Golden Gate TALENprotocollo.
| Plasmide / Collezione | Collaboratore | Addgene ID | Commenti |
| Golden Gate TALEN e TAL Effector Kit 2.0 | Voytas lab | 1000000024 | Contiene tutti i plasmidi necessari per l'assemblaggio Golden Gate TALEN |
| vettori di destinazione pCAG-T7-TALEN -KKR/ELD | Pelczar lab | 40131, 40132 | Add-on plasmidi per l'espressione TALEN in cellule di mammifero e in vitro sintesi di mRNA |
Tabella 2. Plasmidi e collezioni plasmidi necessari per il montaggio Golden Gate TALEN possono essere ottenuti da Addgene ( www.addgene.org ).
| OnlineRisorsa | Commenti |
| http://tale-nt.cac.cornell.edu | Progettazione di TALEN; TALEN previsione off-target |
| http://zifit.partners.org/ZiFiT/ | Progettazione di TALEN, APERTO ZFN, CoDA ZFN, CRISPR/Cas9 |
| http://www.genome-engineering.org | Progettazione di TALEN, CRISPR/Cas9; CRISPR/Cas9 previsione off-target |
| http://baolab.bme.gatech.edu/Research/BioinformaticTools/assembleTALSequences.html | Assemblea dei TALEN sequenze per la conferma dei risultati di sequenziamento |
| http://pgfe.umassmed.edu/ZFPmo dularsearchV2.html | Progettazione di assemblaggio modulare ZFN |
| www.genomecenter.ucdavis.edu/s egallab / segallabsoftware | Progettazione di assemblaggio modulare ZFN |
Tabella 3. Risorse online per la progettazione ZFN, TALEN, e CRISPR/Cas9.
Discussione
Genoma approcci editing nucleasi-driven Designer hanno notevolmente ampliato la gamma di specie suscettibili di modifiche mirate dei rispettivi genomi 10,12. Nei topi,-gene targeting nelle cellule ES è una tecnica standard per oltre due decenni; tuttavia, si è rivelato difficile per adattarsi alle cellule staminali provenienti da specie diverse del mouse, anche se c'è stata qualche recente successo in cellule ES ratto. Anche con la disponibilità di "off-the-shelf" cloni di cellule ES topo gene targeting fornite da consorzi come EUCOMM, KOMP, o NorCOMM 3 modifica del genoma da ZFN e TALEN fornisce una maggiore precisione e flessibilità per quanto riguarda la gamma di modifiche che possono essere introdotto nel genoma del mouse. Fondatore animali portatori di mutazioni nucleasi-mediata sembrano essere altamente linea germinale competente 4-6,20,21, che non è sempre il caso di chimere provenienti da blastocisti iniezioni di cellule staminali. Pertanto, in alcuni casi microiniezione di desnucleasi igner possono provocare generazione significativamente più veloce delle linee di nuovi mouse con modificazioni del genoma mirati.
Il successo generazione di topi knockout per iniezione di ZFN e TALEN dipende in larga misura l'attività della coppia nucleasi iniettato. Talens hanno dimostrato di avere un alto tasso di successo in mira una vasta gamma di geni in un numero di organismi; Tuttavia, studi recenti suggeriscono che TALEN legame è sensibile alla citosina metilazione 30,31. Così, coppie nucleasi appena generati, per esempio Talens clonati in vettori pCAG-T7, possono essere trasfettate transitoriamente in una linea cellulare di topo come NIH-3T3 o Neuro -2a, che imitano lo stato della cromatina embrione di topo in una certa misura. Qui, nucleasica può essere stimata utilizzando la T7 endonucleasi test o una restrizione digest del prodotto di PCR come descritto nel capitolo 5 prima sintesi di mRNA e microiniezione. Si consiglia di sequenziamento della regione genomica di interesse nel rispettoive linea cellulare e il ceppo del mouse utilizzato per esperimenti di microiniezione.
In zigoti del mouse, diverse coppie Talen o ZFN lavoreranno in maniera ottimale a diverse concentrazioni di mRNA e quindi la concentrazione di lavoro ottimale della microiniezione nucleasi mRNA possono essere determinati sperimentalmente. A seconda della coppia di nucleasi, una concentrazione troppo bassa comporterà alcuna scissione, mentre troppo alta può causare letalità embrionale. A seconda della coppia nucleasi, abbiamo avuto successo usando concentrazioni di mRNA totale compresi fra 2 ng / ml e alto come 200 mcg / ul. Questi effetti sono difficili da prevedere da esperimenti in coltura cellulare e la concentrazione ottimale nucleasi sia per la sopravvivenza degli embrioni e la velocità di modifica del locus bersaglio deve essere determinato empiricamente.
Altamente attivo ZFN o TALEN possono fendere la loro sequenza bersaglio oltre la fase unicellulare dell'embrione microiniezione e quindi causare complessi schemi di mutagenesi und mosaicismo in fondatori. Noi e altri 4 abbiamo osservato tre o più alleli mutati distinte in un unico fondatore (Figura 5C). Così, quando si stabilisce una nuova linea di topi da questi fondatori, prole devono essere attentamente monitorati mediante sequenziamento per la presenza della mutazione favorevole da saggi di digestione forniscono la prova che solo una mutazione indefinito è presente.
Una delle critiche spesso hanno espresso contro i sistemi ZFN e Talen è la possibilità che questi nucleases sono anche in grado di scindere sequenze presenti altrove nel genoma che sono simili ai siti di destinazione. Tali effetti off-target sono stati osservati con i reagenti di prima generazione utilizzando il dominio omodimerica FokI e costrutti eterodimero stati progettati per alleviare gli effetti 25 off-target. Potenziali siti off-bersaglio possono essere previsti in una certa misura in silico 32,33 e screening mediante PCR e sequenziamento. Un evidente vantaggio of usando ZFNs e Talens per la generazione di topi piuttosto che linee cellulari è la possibilità di rimuovere off mutazioni di destinazione non collegate alla modificazione del genoma desiderato eseguendo diversi reincroci ad un ceppo selvatico di scelta. Per l'analisi di un gran numero di topi fondatori, prossima generazione sequenziamento dei prodotti di PCR generati dal locus nucleasi targeting e in silico previsti off-target loci potrebbe offrire una lettura qualitativa e quantitativa alternativa ai saggi di digestione dei prodotti di PCR.
Le tecniche di riproduzione assistita descritte in questo protocollo sono ottimizzati per i ceppi mouse standard utilizzati per esperimenti di microiniezione come C57BL/6J o B6D2F1. I topi di diversa origine, quali ceppi dell'esoincrocio, in linea di principio essere utilizzati per approcci modifica del genoma e potrebbero fornire un background genetico più adatto per domande di ricerca specifiche. Le prestazioni di tecniche di riproduzione assistita come la superovulazione può essere predicatoCTED per un numero di ceppi 34-36 ma potrebbe richiedere ulteriore ottimizzazione per i ceppi non standard al fine di ottenere un numero sufficiente di embrioni per nucleasi microiniezione.
Oltre ZFN e TALEN, nuove nucleasi di design come il sistema CRISPR/Cas9 RNA-guidata 9,37,38 ora sono stati introdotti per applicazioni di editing genoma. Tutti i metodi per la microiniezione e l'analisi di animali fondatori qui descritti sono applicabili a CRISPR/Cas9 e le modalità future di modifica del genoma anche.
Divulgazioni
Gli autori dichiarano nessun conflitto di interessi.
Riconoscimenti
Vorremmo ringraziare Monika Tarnowska, Cornelia Albrecht, e Ewa Skoczylas per l'eccellente assistenza tecnica. Questo studio è stato finanziato dal FNS Sinergia concessione CRSI33-125.073 a PP.
Materiali
| Name | Company | Catalog Number | Comments |
| BsaI | NEB | R0535S or L | |
| Esp3I | Thermo Scientific | ER0451 | |
| T4 Ligase | NEB | M0202S or L | |
| Spectinomycin | Sigma | S0692-1ML | |
| Ampicillin | Sigma | A0166 | |
| X-Gal | Sigma | B4252 | |
| IPTG | Sigma | I6758 | |
| Plasmid-Safe nuclease | Epicentre | E3101K | |
| QIAprep Spin Miniprep Kit | Qiagen | 27106 | |
| QIAGEN Plasmid Midi Kit | Qiagen | 12143 | |
| QIAquick PCR Purification Kit | Qiagen | 28106 | |
| mMESSAGE mMACHINE T7 Ultra Kit | Invitrogen | AM1345 | |
| NucAway Spin Columns | Invitrogen | AM10070 | |
| RNaseZAP | Sigma | R2020-250ML | |
| NorthernMax Formaldyde Load Dye | Invitrogen | AM8552 | |
| RNA Millennium Markers | Invitrogen | AM7150 | |
| 10x TBE buffer | Thermo Scientific | B52 | |
| T7 endonuclease | NEB | M0302S or L | |
| pGEM-T EasyVector System I | Promega | A3600 | |
| SYBR Green I Nucleic Acid Gel Stain | Invitrogen | S-7563 | |
| pregnant mare's serum gonadotrophin (PMSG) | Sigma | G4877 | |
| human chorionic gonadotropin (hCG) | Sigma | CG5 | |
| M2 embryo culture medium | Sigma | M7167 | |
| M16 embryo culture medium | Sigma | M7292 | |
| Mineral oil, embryo tested | Sigma | M8410 | |
| Ketamine | CentraVet | Ket 201 | |
| Xylazine | Sigma Aldrich | 46995 | |
| Equipment/Tools | |||
| Inverted microscope with Nomarski DIC optics (for example Nikon Eclipse TE200) | Nikon | ||
| Micromanipulator units (for example Narishige, NT88NF) | Narishige | ||
| Embryo holding capillaries | Sutter Instruments | B100-75-10 | |
| Embryo injection capillaries | Narishige | GD-1 | |
| Capillary puller (for example Sutter P97) | Sutter Instruments | ||
| Microforge (for example Narishige MF-900) | Narishige | ||
| Walton skin scissors | FST | 14077-10 | |
| Surgical scissors | FST | 14041-10 | |
| Surgical probe | FST | 10140-03 | |
| Reflex wound clip system (9 mm) | FST | 12031-09 | |
| Reflex wound clips (9 mm) | FST | 12032-09 | |
| Dumont fine forceps 5 | FST | 11254-20 | |
| Moria curved forceps | FST | 11370-31 | |
| Moria fine forceps | FST | 11399-80 | |
| Dietrich bulldog clamp | FST | 18038-45 | |
| C57BL/6J mice | Jackson Labs | strain code 000664 | |
| CD-1 mice | Charles River | strain code 000664 | |
Riferimenti
- Capecchi, M. R. Gene targeting in mice: functional analysis of the mammalian genome for the twenty-first century. Nat. Rev. Genetics. 6, 507-512 (2005).
- Johansson, T., et al. Building a zoo of mice for genetic analyses: a comprehensive protocol for the rapid generation of BAC transgenic mice. Genesis. 48, 264-280 (2010).
- Skarnes, W. C., et al. A conditional knockout resource for the genome-wide study of mouse gene function. Nature. 474, 337-342 (2011).
- Carbery, I. D., et al. Targeted genome modification in mice using zinc-finger nucleases. Genetics. 186, 451-459 (2010).
- Meyer, M., de Angelis, M. H., Wurst, W., Kuhn, R. Gene targeting by homologous recombination in mouse zygotes mediated by zinc-finger nucleases. Proc. Natl. Acad. Sci. U.S.A. 107, 15022-15026 (2010).
- Cui, X., et al. Targeted integration in rat and mouse embryos with zinc-finger nucleases. Nat. Biotechnol. 29, 64-67 (2011).
- Sung, Y. H., et al. Knockout mice created by TALEN-mediated gene targeting. Nat. Biotechnol. 31, 23-24 (2013).
- Wefers, B., et al. Direct production of mouse disease models by embryo microinjection of TALENs and oligodeoxynucleotides. Proc. Natl. Acad. Sci. U.S.A. 110, 3782-3787 (2013).
- Wang, H., et al. One-Step generation of mice carrying mutations in multiple genes by CRISPR/Cas-mediated genome engineering. Cell. 153, 910-918 (2013).
- Urnov, F. D., Rebar, E. J., Holmes, M. C., Zhang, H. S., Gregory, P. D. Genome editing with engineered zinc finger nucleases. Nat. Rev. Genetics. 11, 636-646 (2010).
- ZFN, T. A. L. E. N. CRISPR/Cas-based methods for genome engineering. Trends Biotechnol. 31, 397-405 (2013).
- Joung, J. K., Sander, J. D. TALENs: a widely applicable technology for targeted genome editing. Nat. Rev. Mol. Cell Biol. 14, 49-55 (2013).
- Cermak, T., et al. Efficient design and assembly of custom TALEN and other TAL effector-based constructs for DNA targeting. Nucleic Acids Res. 39, (2011).
- Reyon, D., et al. FLASH assembly of TALENs for high-throughput genome editing. Nat. Biotechnol. 30, 460-465 (2012).
- Sanjana, N. E., et al. A transcription activator-like effector toolbox for genome engineering. Nat. Protoc. 7, 171-192 (2012).
- Kim, Y., et al. A library of TAL effector nucleases spanning the human genome. Nat. Biotechnol. 31, 251-258 (2013).
- Schmid-Burgk, J. L., Schmidt, T., Kaiser, V., Honing, K., Hornung, V. A ligation-independent cloning technique for high-throughput assembly of transcription activator-like effector genes. Nat. Biotechnol. 31, 76-81 (2013).
- Hsu, P. D., et al. DNA targeting specificity of RNA-guided Cas9 nucleases. Nat. Biotechnol. 31, 827-832 (2013).
- Fu, Y., et al. High-frequency off-target mutagenesis induced by CRISPR-Cas nucleases in human cells. Nat. Biotechnol. 31, 822-826 (2013).
- Hermann, M., et al. Evaluation of OPEN zinc finger nucleases for direct gene targeting of the ROSA26 locus in mouse embryos. PLoS ONE. 7, (2012).
- Meyer, M., Ortiz, O., Hrabe de Angelis, M., Wurst, W., Kuhn, R. Modeling disease mutations by gene targeting in one-cell mouse embryos. Proc. Natl. Acad. Sci. U.S.A. 109, 9354-9359 (2012).
- Doyle, E. L., et al. TAL Effector-Nucleotide Targeter (TALE-NT) 2.0: tools for TAL effector design and target prediction. Nucleic Acids Res. 40, 117-122 (2012).
- Miller, J. C., et al. A TALE nuclease architecture for efficient genome editing. Nat. Biotechnol. 29, 143-148 (2011).
- Guo, J., Gaj, T., Barbas, C. F. Directed evolution of an enhanced and highly efficient FokI cleavage domain for zinc finger nucleases. J. Mol. Biol. 400, 96-107 (2010).
- Doyon, Y., et al. Enhancing zinc-finger-nuclease activity with improved obligate heterodimeric architectures. Nat. Methods. 8, 74-79 (2011).
- Wefers, B., et al. . Current Protocols in Mouse Biology. , (2011).
- Wefers, B., Wurst, W., Kühn, R. . Current Protocols in Mouse Biology. , (2011).
- Haueter, S., et al. Genetic vasectomy-overexpression of Prm1-EGFP fusion protein in elongating spermatids causes dominant male sterility in mice. Genesis. 48, 151-160 (2010).
- Perez-Pinera, P., Ousterout, D. G., Brown, M. T., Gersbach, C. A. Gene targeting to the ROSA26 locus directed by engineered zinc finger nucleases. Nucleic Acids Res. 40, 3741-3752 (2012).
- Bultmann, S., et al. Targeted transcriptional activation of silent oct4 pluripotency gene by combining designer TALEs and inhibition of epigenetic modifiers. Nucleic Acids Res. 40, 5368-5377 (2012).
- Valton, J., et al. Overcoming TALE DNA binding domain sensitivity to cytosine methylation. J. Biol. Chem. 287, 38427-38432 (2012).
- Cradick, T. J., Ambrosini, G., Iseli, C., Bucher, P., McCaffrey, A. P. ZFN-Site searches genomes for zinc finger nuclease target sites and off-target sites. BMC Bioinform. 12, (2011).
- Doyle, E. L., et al. TAL Effector-Nucleotide Targeter (TALE-NT) 2.0: tools for TAL effector design and target prediction. Nucleic Acids Res. 40, 117-122 (2012).
- Byers, S. L., Payson, S. J., Taft, R. A. Performance of ten inbred mouse strains following assisted reproductive technologies (ARTs). Theriogenology. 65, 1716-1726 (2006).
- Luo, C., et al. Superovulation strategies for 6 commonly used mouse strains. J. Am. Assoc. Lab. Animal Sci. 50, 471-478 (2011).
- Pease, S., Saunders, T. L. . International Society for Transgenic Technologies. Advanced protocols for animal transgenesis : an ISTT manual. , (2011).
- Cong, L., et al. Multiplex genome engineering using CRISPR/Cas systems. Science. 339, 819-823 (2013).
- Mali, P., et al. RNA-Guided human genome engineering via Cas9. Science. 339, 823-826 (2013).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon