È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Identificazione di acidi grassi a
In questo articolo
Riepilogo
We propose a protocol to identify fatty acids without the need to purify them. It combines information on the retention times with the mass spectra of three types of fatty acid derivatives: fatty acid methyl esters (FAMEs), 4,4-dimethyl oxazoline derivatives (DMOX), and 3-pyridylcarbinyl esters (picolinyl).
Abstract
Le specie Bacillus contengono ramificata catena e acidi grassi insaturi (FAS) con diverse posizioni del ramo di metile (ISO o anteiso) e del doppio legame. Cambiamenti nella composizione FA giocano un ruolo cruciale nell'adattamento dei batteri al loro ambiente. Queste modifiche comportano una variazione del rapporto di iso rispetto anteiso ramificato AF, e nella proporzione di insaturo FAs rispetto al satura AF, con doppi legami creati in posizioni specifiche. Identificazione precisa del profilo di FA è necessario comprendere i meccanismi di adattamento di specie Bacillus.
Molte delle federazioni da Bacillus non sono disponibili in commercio. La strategia proposta nel presente documento identifica FAs combinando le informazioni sul tempo di ritenzione (calcolo della lunghezza della catena equivalente (ECL)), con gli spettri di massa di tre tipi di derivati FA: gli esteri metilici degli acidi grassi (FAME), 4,4-dimetil ossazolinici derivati (DMOX), e3-pyridylcarbinyl estere (picolinyl). Questo metodo può identificare FAS senza la necessità di purificare l'ignoto AF.
Confrontando i profili cromatografici di FAME preparato da Bacillus cereus con una miscela commerciale di standard consente l'identificazione di catena lineare saturo AF, il calcolo della ECL, e ipotesi sull'identità dell'altro AF. FAMEs di ramificata saturi Federcalcio, iso o anteiso, visualizzare un cambiamento negativo costante nella ECL, rispetto alla FAS lineari saturi con lo stesso numero di atomi di carbonio. FAMEs di insaturo AF possono essere rilevati dalla massa dei loro ioni molecolari, e determinano uno spostamento positivo nella ECL rispetto ai corrispondenti federazioni saturi.
La posizione ramificazione del FAS e la posizione del doppio legame insaturi FAs possono essere identificati dagli spettri di massa a ionizzazione di elettroni picolinyl e DMOX derivati, rispettivamente. Questo approccio identifica tutto il ramo saturo sconosciutaEd FAs, insaturi a catena lineare FAs e AF ramificati insaturi dall'estratto cereus B..
Introduzione
Acidi grassi gascromatografia estere metilico (FAME) (GC) è un metodo essenziale per la caratterizzazione dei lipidi. Si separa rapidamente e quantifica i vari acidi grassi (FAS) di un campione dopo una breve fase di estrazione. Derivati di esteri metilici sono altamente volatili, stabile ed inerte verso la colonna cromatografica, evitando così i picchi di decantazione. La loro identificazione è piuttosto semplice quando il campione è costituito da FAs ben noti in quanto i profili cromatografici o sono pubblicati o rispetto agli standard. Inoltre, l'iniezione ripetuta di standard di calibrazione per la quantificazione di vari FAs non è necessario, data la loro pressoché costante risposta al rilevamento a ionizzazione di fiamma (FID) 1.
Oltre a FID, spettrometria di massa (MS) di rilevamento fornisce una serie complementare di informazioni per confermare FAMEs. Tuttavia, quando FAMEs pagano usando ionizzazione elettronica (EI), gli spettri risultanti non sempre consentono °e l'identificazione di FA struttura fine. Per esempio, la posizione ramificazione (cioè, un gruppo metile ramificato) è difficile da prevedere perché gli ioni diagnostici sono difficili da rilevare 1 e il cambiamento caratteristico in abbondanza ione bersaglio dipende dalla macchina, evitando l'uso di librerie di spettri di massa 2. Un'altra sfida consiste nell'individuare la posizione di doppio legame, perché EI provoca la migrazione doppio legame. Così, isomeri FA con diverse posizioni doppio legame non possono essere differenziati per la loro spettri di massa. Fortunatamente, altri strumenti sono stati sviluppati per l'identificazione FA. Per esempio, la presenza e la posizione di ramificazione o di doppi legami in FAs ipotizzabile calcolando la lunghezza della catena equivalente (ECL) 3.
Altri metodi di derivatizzazione comportano diversi spettri di massa, dipende dalla posizione di un doppio legame o un gruppo metile ramificato. 4,4-dimetil derivati ossazolinici (DMOX) 4 permettono di easIdentificazione y della posizione di doppi legami di acidi grassi monoinsaturi. 3-pyridylcarbinyl estere (picolinyl estere) i derivati permettono l'identificazione univoca della posizione di metile ramificata FAs 5. Combinando ritenzione cromatografica (ECL) e spettrometria di massa (DMOX e picolinyl) informazioni consente l'identificazione della maggior parte delle federazioni senza la necessità di utilizzare complessi metodi di purificazione, come richiesto per la risonanza magnetica nucleare (NMR), il metodo incontestabile per la caratterizzazione strutturale 1 .
I batteri del genere Bacillus, che include alcuni agenti patogeni umani ed animali, sono in grado di colonizzare estremamente diverse nicchie e sono quindi ampiamente distribuiti nell'ambiente 6. Tra genere Bacillus, composizione FA è influenzata dalla nicchia ecologica della specie con modulazioni nei modelli FA di adattarsi ad un'ampia gamma di cambiamenti ambientali (ad esempio, il terreno di coltura, la temperatura,pH, ecc) 7-9. A causa della relativa omogeneità del modello FA attraverso specie del genere Bacillus durante la crescita in condizioni standardizzate, determinazione della composizione FA è uno dei criteri essenziali usati per definire le specie Bacillus. Un attributo unico del genere Bacillus è l'abbondanza del FAS a catena ramificata contenenti 12-17 atomi di carbonio 10-12 con il rapporto tra ISO e anteiso isomeri di essere un fattore determinante di adattamento alle condizioni ambientali. Specie Bacillus adattano anche alle fluttuazioni ambientali alterando la percentuale di acidi grassi insaturi. In alcune specie, come il Bacillus cereus, due desaturasi di acidi grassi creano doppi legami in diverse posizioni della catena alchilica 13 con diversi ruoli in adattamento 9. L'esempio del genere Bacillus illustra l'importanza di individuare con precisione la posizione doppio legame e FA ramificazione. Collezionarevamente, l'identificazione di modelli di Bacillus FA ha diverse applicazioni utili. Qui, proponiamo un nuovo approccio GC-MS per l'identificazione modello Bacillus FA che supera i limiti intrinseci di una classica analisi GC-MS.
Questo approccio innovativo può essere utilizzato direttamente sul materiale biologico grezzo, e consiste in una combinazione di tecniche esistenti: informazioni sui tempi di ritenzione (ECL) e spettri di massa dei diversi derivati AF (FAME, DMOX e picolinyl-estere).
Usiamo la seguente nomenclatura FA. i, a, e n indicano iso, anteiso metil ramificata, e acidi grassi a catena lineare, rispettivamente. FAs insaturi sono stati nominati da C: d dove C è il numero di atomi di carbonio dell'acido grasso e d è il numero di doppi legami. Δ x indica la posizione del doppio legame, in cui il doppio legame è situato il legame carbonio-carbonio xth, contando dal terminale acido carbossilico.
Protocollo
1. colture batteriche
- Preparare un prato di batteri (Bacillus cereus ceppo ATCC 14579) diffondendo 100 ml di una coltura durante la notte del ceppo incubato a 30 ° C in LB (Luria-Bertani medio), sulla superficie di una piastra di terreno LB agar. Incubare la piastra per una notte a 30 ° C.
2. ECL: lunghezza della catena Equivalente
- Calcolare ECL come segue:
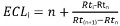 con:
con:
i, il soluto di interesse;
n, il numero di carbonio del rettilineo catena di acido grasso saturo estere metilico eluendo prima soluto I;
n + 1, il numero di carbonio del rettilineo estere metilico catena satura di acido grasso eluendo dopo soluto I;
Rti, Rtn, Rt (n + 1) i tempi di ritenzione dei picchi FAME sopra descritti.
NOTA: ottenere i tempi di ritenzione di esteri acidi grassi saturi a catena metilici rettilinei mediante iniezione di una miscela di standard (BAME).
3. FAME preparazione e l'analisi
- Per ottenere acidi grassi lipidi, cellule batteriche raccolta raschiando colonie dalla piastra di agar e trasferire 40 mg (peso fresco, equivalente a 10 9 cellule vitali) di batteri in una provetta di vetro da 10 ml con tappo a vite e guarnizioni PTFE.
- Eseguire transesterificazione tramite il metodo di collegamento estere 14,15 descritto di seguito.
- Aggiungere 5 ml di 0,2 M KOH in metanolo alle cellule batteriche fresche e incubare a 37 ° C per 1 ora. Questa reazione è costituito metanolisi alcalina, interrompendo il collegamento di estere nel lipidico e produrre esteri metilici degli acidi grassi.
- Aggiungere 1 ml di 1 M di acido acetico per abbassare il pH a 7,0. Controllare il pH con le strisce reattive pH.
- Aggiungere 3 ml di esano per estrarre FAMEs.
- Trasferire il surnatante (fase organica) in provette pulite e concentrato mediante evaporazione a temperatura ambiente sotto un flusso continuo di azoto per ottenere circa 200 microlitridi estratto. Trasferire il campione in una fiala GC con inserto.
- Iniettare estratti in una spettrometria di massa cromatografia gas sistema (GC-MS).
4. GC / MS Condizioni
- Iniettare campioni FAME in uno strumento GC-MS equipaggiato con una colonna capillare ZB-WAX (lunghezza 30 m, diametro, 0,25 mm, spessore del film, 0,25 micron).
- Impostare la porta di iniezione (in modalità splitless) temperatura a 250 ° C. Utilizzare l'elio come gas di trasporto, con una velocità lineare di 37 cm / sec. Mantenere la temperatura del forno a 50 ° C per 1 min, aumentare a 190 ° C ad una velocità di 20 ° C / min, e aumentare ulteriormente ad una temperatura finale di 230 ° C ad una velocità di 2 ° C / min.
- Ai MS, registrare gli spettri di massa per ionizzazione elettronica (EI) a 70 eV, e impostare l'acquisizione della corrente ionica totale tra 50 e 400 unità atomiche di massa (amu) (2 scans / sec).
- Quando necessario, iniettare DMOX e picolinyl derivati sotto la stessa condizione eccet forno programma di temperatura come segue:
DMOX: 50 ° C (1 minuto), 20 ° C / min fino 210 ° C e 2 ° C / min fino a 240 ° C (5 min);
Picolinyl: 6 ° C (1 minuto), 20 ° C / min fino a 220 ° C e 2 ° C / min fino a 250 ° C (20 min).
5. Picolinyl Ester Preparazione a partire da 16 FAME
- Evaporare l'estratto FAME dalla Sezione 3 con un flusso di azoto (almeno 10 mg materiale secco) e sciogliere in 1 ml di diclorometano anidro.
- Preparare una soluzione di ter di potassio terz.butossido in tetraidrofurano 1.0 M.
- Aggiungere l'estratto FAME e 0,2 ml di 3-pyridinemethanol a 0,1 ml di soluzione fatte nella fase 5.2.
- Riscaldare la soluzione a 40 ° C per 30 min in una fiala chiusa.
- Dopo raffreddamento a temperatura ambiente, aggiungere acqua deionizzata purificata (2 ml, vedi Materiali Tavolo) ed esano (4 ml). Mescolare con un vortice, permettono di fase per separare e raccogliere la fase organica.
- Dry aggiungendo solfato di sodio anidro fino alla fase organica è perfettamente chiaro. Trasferire in una provetta pulita. Poi evaporare a 200 l. Trasferire il campione in una fiala GC con inserto.
6. DMOX Preparazione a partire da 17 FAME
- Evaporare l'estratto FAME dalla Sezione 3 con un flusso di azoto (materiale secco, di almeno 10 mg).
- Per l'estratto secco FAME, aggiungere 250 mg di 2-ammino-2-metil-1-propanolo. Lavare il recipiente con azoto, aggiungere un tappo, e metterlo in un blocco di riscaldamento durante la notte a 190 ° C.
- Dopo raffreddamento a temperatura ambiente, aggiungere 3 ml diclorometano nel tubo, e 5 ml di acqua deionizzata purificata (vedi Materiali Tabella).
- Agitare per separazione di fase e quindi rimuovere la fase acquosa.
- Lavare la fase organica con acqua 5 ml. Agitare per separazione di fase e quindi rimuovere la fase acquosa.
- Lavaggio aggiungendo solfato di sodio anidro fino alla fase organica è perfettamente limpida etrasferirlo in una provetta pulita. Evaporare sotto corrente di azoto fino a raggiungere un volume di 200 microlitri. Trasferire il campione in una fiala GC con inserto.
Risultati
La strategia di identificazione FA da cellule batteriche è presentato in Figura 1. Ogni passo fornisce informazioni spettrali complementari o informazioni su di ritenzione cromatografica. Fase 1 consiste preliminare identificazione FA utilizzando una soluzione standard. Step 2 permette l'interpretazione di FAME EI spettri e la loro ECL, al fine di tentare di identificare i prodotti. Fase 3 identifica la posizione esatta ramificazione nella catena ramificata-FAS. Inf...
Discussione
I profili cromatogramma AF indicati nella tabella 1 corrispondono a B. cereus ATCC 14579 cresciuta su una superficie della piastra di agar. Profili simili sono stati ottenuti quando il batterio è stato coltivato in terreni liquidi aerato alla stessa temperatura 8. Nel caso dei batteri coltivati in terreno liquido, la biomassa batterica viene raccolto per centrifugazione del mezzo di crescita e può essere lavato secondo protocolli precedentemente descritti a seconda delle condizioni...
Divulgazioni
The authors declare that they have no competing financial interests.
Riconoscimenti
Gli autori sono grati a Thomas Mison per il suo supporto tecnico, e di Rachel Kopec per la revisione del manoscritto.
Materiali
| Name | Company | Catalog Number | Comments |
| GC/MS | Shimadzu | QP2010 | |
| capillary column ZB WAX | Phenomenex | 7HG-G007-11 | 30 m x 0.25 mm x 0.25 µm |
| Methanol Lichrosolv | VWR | 1.06018.2500 | |
| potassium hydroxide | Aldrich | P1767 | |
| THF | Hipersolv Chromanorm | 28559.320 | |
| Dichloromethane | Hipersolv Chromanorm | 23373.320 | |
| Hexane | Hipersolv Chromanorm | 24575.320 | |
| 3-pyridinemethanol | Aldrich | P6-680-7 | |
| potassium tertiobutoxide | Aldrich | 156671 | |
| 2-amino-2-methyl-1-propanol | A-9879 | ||
| MilliQ Academic | Millipore | ZMQS50001 | |
| Bacterial Acid Methyl Ester (BAME) Mix | Sigma-Aldrich | 47080-U Supelco |
Riferimenti
- Christie, W. W., Han, X. . Lipid Analysis 4th Edition. , (2010).
- HÜbschmann, H. -. J. . Handbook of GC-MS: fundamental and application. Third edition. , (2015).
- Sasser, M. Identification of Bacteria by Gas Chromatography of Cellular Fatty Acids. MIDI Technical note. 101, 1-6 (1990).
- Spitzer, V. Structure analysis of fatty acids by gas chromatography - Low resolution electron impact mass spectrometry of their 4,4-dimethyloxazoline derivatives - A review. Prog Lipid Res. 35 (4), 387-408 (1996).
- Harvey, D. J., Christie, W. W. . Advances in lipid methodology. Volume 1. , 19-80 (1992).
- Diomande, S. E., Nguyen-The, C., Guinebretière, M. -. H., Broussolle, V., Brillard, J. Role of fatty acids in Bacillus environmental adaptation. Front Microbiol. 6, (2015).
- Brillard, J., et al. Identification of Bacillus cereus Genes Specifically Expressed during Growth at Low Temperatures. Appl Environ Microbiol. 76 (8), 2562-2573 (2010).
- de Sarrau, B., et al. Influence of Anaerobiosis and Low Temperature on Bacillus cereus Growth, Metabolism, and Membrane Properties. Appl Environ Microbiol. 78 (6), 1715-1723 (2012).
- Diomandé, S. E., et al. Involvement of the CasK/R two-component system in optimal unsaturation of the Bacillus cereus fatty acids during low-temperature growth. Int J Food Microbiol. 213, 110-117 (2015).
- Berkeley, R. C. W., Heyndrickx, M., Logan, N., De Vos, P., Berkeley, R. C. W. . Applications and Systematics of Bacillus and Relatives. , 1-7 (2002).
- Kämpfer, P. Limits and Possibilities of Total Fatty Acid Analysis for Classification and Identification of Bacillus Species. System. Appl. Microbiol. 17 (1), 86-98 (1994).
- Kaneda, T. Fatty-acids of genus bacillus - example of branched-chain preference. Bacteriol Rev. 41 (2), 391-418 (1977).
- Chazarreta Cifre, L., Alemany, M., de Mendoza, D., Altabe, S. Exploring the Biosynthesis of Unsaturated Fatty Acids in Bacillus cereus ATCC 14579 and Functional Characterization of Novel Acyl-Lipid Desaturases. Appl Environ Microbiol. 79 (20), 6271-6279 (2013).
- Sasser, M., et al. Identification of Bacillus anthracis from culture using gas chromatographic analysis of fatty acid methyl esters. J AOAC Int. 88 (1), 178-181 (2005).
- Schutter, M. E., Dick, R. P. Comparison of fatty acid methyl ester (FAME) methods for characterizing microbial communities. Soil Sci Soc Am J. 64 (5), 1659-1668 (2000).
- Destaillats, F., Angers, P. One-step methodology for the synthesis of FA picolinyl esters from intact lipids. J Am Oil Chem Soc. 79 (3), 253-256 (2002).
- Fay, L., Richli, U. Location of double-bonds in polyunsaturated fatty-acids by gas-chromatography mass-spectrometry after 4,4-dimethyloxazoline derivatization. J Chromatogr. 541 (1-2), 89-98 (1991).
- Zhang, J. Y., Yu, Q. T., Liu, B. N., Huang, Z. H. Chemical modification in mass spectrometry IV-2-alkenyl-4,4-dimethyloxazolines as derivatives for the double bond location of long-chain olefinic acids. Biol Mass Spect. 15 (1), 33-44 (1988).
- de Sarrau, B., et al. Unsaturated fatty acids from food and in the growth medium improve growth of Bacillus cereus under cold and anaerobic conditions. Food Microbiol. 36 (2), 113-122 (2013).
- Miwa, T. K., Mikolajczak, K. L., Earle, F. R., Wolff, I. A. Gas chromatographic characterization of fatty acids.Identification constants for mono- and dicarboxylic methyl esters. Anal Chem. 32 (13), 1739-1742 (1960).
- van Den Dool, H., Kratz, P. A generalization of the retention index system including linear temperature programmed gas-liquid partition chromatography. J Chromatogr A. 11, 463-471 (1963).
- Stransky, K., Jursik, T., Vitek, A. Standard equivalent chain length values of monoenic and polyenic (methylene interrupted) fatty acids. J High Res Chromatogr. 20 (3), 143-158 (1997).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon