È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Studi struttura-funzione nel topo cellule staminali embrionali Uso ricombinasi mediata Cassette Scambio
In questo articolo
Riepilogo
Le proteine contengono spesso più domini che possono esercitare diverse funzioni cellulari. Gene knock-out (KO) non considerano questa diversità funzionale. Qui riportiamo un cambio cassette ricombinazione mediata (RMCE) approccio basato lo struttura-funzione in cellule staminali embrionali KO che consente la dissezione molecolare dei vari domini funzionali o varianti di una proteina.
Abstract
ingegneria gene in embrioni di topo o cellule staminali embrionali (mESCs) permette per lo studio della funzione di una data proteina. Le proteine sono i cavalli della cellula e spesso costituiti da più domini funzionali, che possono essere influenzate da modificazioni post-traduzionali. La riduzione dello intera proteina in condizionale o costitutiva knock-out (KO) topi non tiene conto di questa diversità funzionale e regolazione. Una linea Mesc e un modello di topo derivato, in cui è stato inserito un sito di attracco per FLPE scambio cassette ricombinazione mediata (RMCE) all'interno del locus ROSA26 (R26), è stato riportato in precedenza. Qui riportiamo un approccio struttura-funzione che permette di dissezione molecolare delle diverse funzionalità di una proteina multidominio. A tal fine, i topi RMCE-compatibili devono essere incrociati con topi KO e mESCs KO quindi compatibili con RMCE devono essere isolati. Successivamente, un gruppo di costrutti di soccorso putativi può essere introdotto nel locus R26 via RMCE targeting. I cDNA di soccorso candidati possono essere facilmente inseriti tra i siti RMCE del vettore targeting utilizzando la ricombinazione clonazione. Successivamente, mESCs KO sono transfettate con il vettore targeting in combinazione con un'espressione ricombinasi plasmide FLPE. RMCE riattiva il gene neomicina-resistenza promotore-meno negli attracco siti ROSA26 e consente la selezione della manifestazione di targeting corretto. In questo modo, l'efficienza elevata di targeting vicino al 100% si ottengono, consentendo l'inserimento di diversi costrutti salvataggio putativi in modo semi-alta produttività. Infine, un gran numero di costrutti di soccorso R26-driven possono essere testati per la loro capacità di salvare il fenotipo che è stato osservato in parentali mESCs KO. Presentiamo uno studio struttura-funzione a prova di principio in p120 catenina (p120ctn) mESCs KO utilizzando differenziazione endoderma in corpi embrionali (EBS) come la lettura fenotipica. Questo approccio consente l'identificazione di domini importanti vie a valle putativi, e punto malattie rilevantimutazioni che sono alla base fenotipi KO per una data proteina.
Introduzione
Si stima che i genomi dei mammiferi contengono circa 20.000 geni codificanti proteine. Lo splicing alternativo e modificazioni post-aumentare ulteriormente il repertorio di proteine. Proteine hanno una struttura modulare 1 e spesso contengono più domini di interazione, che permettono la loro assunzione in diversi complessi proteici e la loro partecipazione molteplici processi cellulari 2. Un esempio è la proteina multi-funzionale chiamato p120ctn. p120ctn è codificata dal gene Ctnnd1 e consiste in una grande dominio centrale armadillo ripetizione affiancato da un N-terminale ed una regione C-terminale. Il dominio di armadillo p120ctn lega ad un dominio juxtamembrana altamente conservata caderine classiche, che sono coinvolti nella adesione cellula-cellula, ma si lega anche al repressore trascrizionale Kaiso. Il dominio N-terminale di p120ctn interagisce con diverse chinasi, fosfatasi, piccoli RhoGTPases e associata ai microtubuli proteins 3. È interessante notare che, a seguito di splicing alternativo, isoforme p120ctn possono essere generati da quattro codoni di inizio alternativi 4. p120ctn isoforma 1A è la più lunga, come viene tradotto da più-5' codone di inizio e contiene il segmento N-terminale full-length. In p120ctn isoforme 3 e 4, questo segmento N-terminale è soppresso parzialmente e completamente, rispettivamente. Comprendere il ruolo preciso delle proteine (o isoforme della proteina) ed i loro domini in diverse funzioni cellulari rimane una sfida.
Gene targeting in mESCs consente lo studio della funzione di una proteina attraverso la delezione genetica del gene corrispondente ed ha ampiamente contribuito alla identificazione di evolutivamente importanti e malattie rilevanti geni e vie. Questo importante passo avanti nel campo della genetica inversa è il risultato di progressi nel campo di isolamento Mesc e gene targeting grazie alla ricombinazione omologa 5 . ricombinazione omologa è un processo in cui vengono scambiati frammenti di DNA tra due porzioni nucleici simili o identici dopo doppio stranded (ds) DNA si rompe. Normalmente, HR è inefficiente perché pause dsDNA sono frequenti. Recentemente, l'efficienza del gene omologia-diretto mira potrebbe essere aumentato utilizzando nucleasi site-specific 6, 7, ma purtroppo, questi sono inclini a effetti fuori bersaglio 8. Una tecnica più affidabile per abilitare gene targeting è RMCE, che si basa su sistemi di ricombinazione sito-specifica, come Cre / loxP o FLPE / Frt. LoxP e sequenza Frt si trovano in P1 batteriofago e Saccharomyces cerevisiae, rispettivamente, e consistono di 34 bp, compresa una sequenza bp asimmetrica 8 che determina l'orientamento del sito. D'altra parte, l'orientamento, ad esempio, due siti loxP all'interno di un tratto di DNA a stabilire se il DNA floxed viene escisso o inversed upon ricombinazione mediata da Cre 9. Inoltre, Cre può anche indurre una traslocazione se due siti si trovano su cromosomi diversi. RMCE sfrutta siti di ricombinazione heterospecific che non cross-reagiscono e che sono incorporati in un locus genomico. In presenza di un plasmide donatore che contiene un frammento di DNA fiancheggiata da siti stessi heterospecific, la ricombinasi inserirà questo frammento di DNA nel locus genomico RMCE compatibile causa della traslocazione doppio simultaneo (Figura 1). Qui, cloni correttamente solo RMCE targeting possono rendere farmacoresistenza grazie ad un promotore sul vettore entrante che restituisce una "trappola" promotore-minore resistenza neomicina gene (Neo R) presenti nel genoma R26 delle cellule di aggancio (figura 1) 10, 11. Questo si traduce in un rendimento di targeting molto alta, spesso vicino al 100% 11, </ sup> 12. In conclusione, il targeting RMCE-based è molto efficace e può essere utilizzato per studi struttura-funzioni; tuttavia, richiede un locus genomico pre-ingegnerizzato.
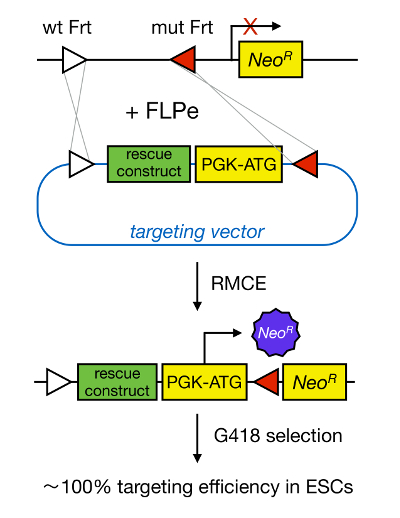
Figura 1. Schema del Targeting RMCE-mediata. RMCE permette lo scambio di segmenti di DNA da un vettore targeting in ingresso a un locus genomico definito se entrambi nutrono due siti FRT heterospecific (rappresentate da triangoli bianchi e rossi). Inoltre, il locus genomico ingegnerizzato contiene un promoterless e gene troncato resistenza alla neomicina (Neo R). Fornendo un promotore e codone di inizio nel frammento di DNA in entrata, eventi di ricombinazione solo corretti restauro resistenza neomicina, con conseguente elevate efficienze di targeting. Si prega di cliccare qui per vedere una versione più grande di tla sua figura.
ingegneria del genoma in mESCs consente la generazione di topi RMCE-compatibili. Nel 1981, due gruppi sono riusciti a catturare cellule pluripotenti dalla massa cellulare interna (ICM) di blastocisti e mantenendoli in coltura 13, 14. mESCs sono in grado di auto-rinnovamento e la differenziazione in tutti i tipi di cellule embrionali e adulte, tra cui il lignaggio cellule germinali. Pertanto, gene targeting in mESCs consente studi reverse-genetici attraverso lo sviluppo di topi costitutivi o condizionali (utilizzando il sistema / LoxP Cre) KO. Tuttavia, il modo classico per isolare cellule di topo ES è molto inefficiente. Diversi importanti miglioramenti hanno notevolmente aumentato il tasso di successo per derivare linee Mesc, compreso l'uso di un siero sostituzione definito (SR) medium 15, alternando mezzo Mesc contenente siero bovino fetale e SR (FBS) 16, e l'uso di farmacocomposti logici quali pluripotin o 2i 17. Pluripotin, una piccola molecola sintetica, consente la propagazione di mESCs in uno stato indifferenziato in assenza del fattore inibitorio della leucemia (LIF) e fibroblasti embrionali di topo (MEF) 18. Infine, è stato dimostrato che mESCs possono essere isolati con un'efficienza molto elevata (vicina al 100%) quando un / FBS protocollo mezzo alternanza SR è combinato con LIF e pluripotin 19, 20. Questi protocolli consentono l'isolamento efficace di mESCs KO RMCE compatibili che possono successivamente essere utilizzati per studi struttura-funzione.
Questo documento descrive un metodo che permette di identificare i settori chiave o residui all'interno di una proteina che sono responsabili per i processi cellulari specifici. A tal fine, una pipeline di tecnologie avanzate che permettono l'isolamento efficiente Mesc, mira assemblaggio vettoriale, e Mesc il targeting è stato creared. Come tali, i grandi pannelli con isoforme della proteina, i mutanti di dominio e effettori a valle possono essere introdotti in mESCs KO e possono essere valutati per la loro capacità di salvare la vitro KO fenotipo in.
Protocollo
Tutti gli esperimenti sui topi sono stati condotti secondo le norme animali istituzionali, nazionali, ed europei.
1. Isolamento di mESCs KO-compatibili RMCE
- Razza topi eterozigoti KO con i topi RMCE-compatibile, come ad esempio i topi ROSALUC 10 o topi ROSA26-IPSC 21. Entrambi i topi RMCE-compatibili sono stati mantenuti su un 129 / C57BL6 / sfondo misto svizzero.
NOTA: L'incrocio con topi KO eterozigoti si consiglia di superare letalità embrionale nei topi KO omozigoti. - Utilizzare PCR per selezionare topi KO eterozigoti che contengono una cassetta RMCE nel locus R26 12.
- Razza, topi KO eterozigoti RMCE-compatibile con i topi KO eterozigoti e isolare RMCE-compatibile, omozigoti blastocisti KO.
- Impostare accoppiamenti tempo la sera e verificare la presenza di copulazione si inserisce la mattina successiva.
NOTA: Le spine sono fatte di secrezioni coagulate dalle coagulazione e vescicolari ghiandole del maschio.Queste spine riempiono la vagina della femmina e persistono per 8 - 24 ore dopo l'allevamento. femmine Plugged si considerano trasportare 0,5 DPC (giorni dopo coitum) embrioni.- Per verificare la presenza di spine, sollevare la femmina dalla base della coda e da esaminare la sua apertura vaginale per una massa biancastra. Stendere le labbra della vulva po 'con una sonda angolata quando la spina è difficile da vedere. femmine separati collegati da loro maschile.
- Raccogliere blastocisti al 3,5 DPC.
- Eutanasia femmine gravide dal metodo approvato (ad esempio, dislocazione cervicale). Effettuare un'incisione midventral e sezionare l'utero ed ovidotto (ancora attaccato gli uni agli altri) utilizzando forbice fine e pinze.
- Piegare un ago 26-gauge in un angolo di 45 °. Attaccare una siringa da 1 ml riempito con mezzo M2 per questo ago piegato e utilizzarla per irrigare le blastocisti dall'utero nel coperchio di un piatto 10 cm.
- Inserire l'ago nell'estremità dell'utero che più si avvicina aovidotto. Tenere l'ago in posizione con una pinza sottile mentre si spinge il pistone; gonfiore dell'utero indica un risciacquo di successo.
- Utilizzare una pipetta bocca (con un diametro di 100-200 micron) per raccogliere tutti gli embrioni e lavarli due volte in una goccia di mezzo di M2 fresca. Subito dopo il lavaggio, trasferire le blastocisti verso le piastre di coltura (vedi sotto).
NOTA: La dissezione e la manipolazione di blastocisti di dovrebbe essere fatto nel flusso d'aria laminare.
- Impostare accoppiamenti tempo la sera e verificare la presenza di copulazione si inserisce la mattina successiva.
- Isolare mESCs RMCE compatibile KO
- Preparare una 12-pozzetti con mitomicina-C-trattati DR4 MEF (vedere la tabella dei materiali) un giorno prima l'isolamento blastocisti.
NOTA: Questi MEF sono stati isolati da topi Tg (DR4) 1Jae / J contenenti quattro geni droga selezionabili e conferiscono resistenza a neomicina, puromicina, igromicina, e 6-tioguanina 22.- Rivestire tutte piastre di coltura con 0,1% di gelatina. Aggiungere 0,1% di gelatinaalle piastre di coltura, incubare per 5 min a 37 ° C in 5% CO 2, ed aspirare la soluzione di gelatina. Seme un quarto di una fiala di P2 MEF in un 12-pozzetti e crescere in 2 mL di terreno MEF (vedi Tabella 1, la tabella dei materiali) ad un monostrato confluente 19.
- Li inattivare con mitomicina-C (10 ug / mL) per 3 ore e lavare due volte con soluzione salina tamponata con fosfato (PBS) 19.
- Usando una pipetta bocca, piastra blastocisti Onto gelatinizzazione piastre da 12 pozzetti (1 pozzetto / embrione), con i MEF mitomicina-C-trattati in terreno cella SR-ES (2 mL / pozzetto) integrato sia con 2 mM pluripotin o con 2i (1 uM inibitore Erk PD0325901 e 3 pM GSK3 inibitore CHIR99021). Incubare a 37 ° C in 5% CO 2.
- Aggiornare la media delle cellule SR-ES (integrato con pluripotin o 2i) ogni 2 - 3 giorni.
- Esaminare ogni blastocisti sotto uno stereomicroportata a 4.0X ingrandimenti e controllare cova e l'attaccamento allo strato MEF.
NOTA: quando blastocisti portellone, perdono la zona pellucida che li racchiude. Wells con blastocisti senza legami devono essere aggiornati utilizzando il pipettaggio bocca. - Scegli i singoli escrescenze ICM (utilizzando uno stereomicroscopio) dopo 10 - 12 giorni di coltura con una pipetta P10 con puntali monouso. Trasferire la conseguenza in circa 10 ml di mezzo di una piastra a 96 pozzetti a V contenente 30 pl / pozzetto di PBS (a temperatura ambiente).
- Aggiungere 50 ml di 0,25% tripsina ad ogni pozzetto usando una pipetta multicanale e incubare per 3 minuti a 37 ° C in 5% CO 2.
- Aggiungere 100 ml di FBS contenente medio Mesc; dissociare le escrescenze ICM in singole cellule pipettando 10-15 volte; e trasferire le cellule dissociate per mitomicina-C-trattata, 96-pozzetti MEF che sono stati preparati un giorno prima colonie ICM vengono raccolte.
- Da questa fase in poi, omi t pluripotin o 2i dal mezzo Mesc. Il giorno successivo, cambiare il supporto da FBS- medio Mesc SR-contenenti (100 pl / pozzetto).
- Ampliare le linee Mesc stabilite da 96- a 24 pozzetti formato 19.
- Lavare le cellule con 200 microlitri di PBS, aggiungere 50 ml di tripsina, e incubare per 5 min a 37 ° C in 5% CO Aggiungere 100 pl di mezzo Mesc FBS-based.; dissociarsi pipettando 10 - 15 volte con una pipetta multicanale; e trasferire le cellule dissociate a mitomicina-C-trattati, 24-pozzetti MEF.
- Passare alla media SR-base il giorno successivo. Ampliare le mESCs in modo simile da 24- formato 6 pozzetti. Fare 3 - 4 gelate da un confluenti 6 pozzetti 19.
- Identificare RMCE-compatibile, mESCs omozigoti KO utilizzando primer PCR per il locus R26 23 e KO allele di scelta (in questo caso, p120ctn; PCR per p120ctn nullo e alleli floxed stati descritti primalass = "xref"> 12).
- Preparare una 12-pozzetti con mitomicina-C-trattati DR4 MEF (vedere la tabella dei materiali) un giorno prima l'isolamento blastocisti.

Tabella 1. Culture Media. Tutti i mezzi sono stati conservati a 4 ° C e riscaldati a 37 ° C 30 minuti prima dell'uso.
2. Generazione di un vettore targeting RMCE-compatibile tramite ricombinazione Clonazione
- Clone salvato costruisce in vettori di ricombinazione compatibile con enzima di restrizione (RE) a base di o PCR-basato 24 tecniche di clonazione. Assicurarsi che i cDNA contengono un codone di stop.
- Progettazione primer 24 ATTB-tagged. Assicurarsi che il primer forward contiene i seguenti elementi: un tratto GGGG, un sito AttB1, un linker, una sequenza di Kozak, e circa 25 nucleotidi di cDNA soccorso (iniziano con il suo ATG). Assicurarsi che il primer inverso ha una composizione simile: un tratto GGGG, un sito attB2, un linker, e circa 25 NUCleotides di cDNA salvataggio (complemento inverso).
- Amplificare il cDNA di soccorso tramite PCR per ottenere il cDNA ATTB-fiancheggiati.
- Eseguire una reazione di 10 microlitri BP con 100 ng di ATTB-fiancheggiato cDNA e 150 ng del vettore donatore ricombinazione-compatibile, che contiene un gene di kanamicina-resistenza.
- Trasformare 5 microlitri delle miscele BP in heat-shock-competenti MC1061 Escherichia coli (E. coli) batteri (simili a quelle descritte nel passo 2.3).
- Identificare colonie contenenti le corrette soccorso vettori di cDNA contenente (simili a quelli descritti nello stadio 2.4)
- Eseguire una reazione LR 10 microlitri con 100 ng di salvataggio cDNA contenente vettoriale; 150 ng di pRMCE-DV1 Cre-escissa vettoriale 11 (LMBP 8195); e 2 microlitri di miscela ricombinasi, che contiene un'integrasi fago-codificato e excisionase e fattore ospitante integrazione batterica. Incubare per 2 ore a 25 ° C.
NOTA: Una reazione LR è una ricombinazione reagireionico in cui un clone di voce contenente siti Attl e un vettore di destinazione contenente siti attr vengono ricombinati dal mix clonase enzima LR. Ciò si traduce in un clone di espressione contenente siti attB che fiancheggiano il gene di interesse. - Trasformare 5 ml di miscele LR in MC1061 batteri E. coli heat-shock-competente.
- Aggiungere 5 ml di LR miscele a coste, costeggiato, tubo 2 mL tappo a vite con 40 ml di heat-shock-competenti di E. coli e incubare per 20 min in ghiaccio. Incubare per 5 min a 37 ° C.
- Aggiungere 1 ml di Luria Broth (LB) media e incubare per 1 ora a 37 ° C. Piastra 50 microlitri di ampicillina (Amp; 100 ug / mL) -contenente piastre di agar e crescere durante la notte a 37 ° C.
- Identificare le colonie con la corretta vettore targeting.
- Scegliere 5 colonie casualmente usando una punta p200. Trasferire la punta di una provetta di vetro contenente 2 - 5 ml di terreno LB e crescere durante la notte a 37 ° C.
- ExTract il DNA plasmidico da colture batteriche utilizzando kit disponibili in commercio.
- Convalida utilizzando digerisce RE e sequenziamento. Tagliare, 0,5 - 2 pg di plasmide utilizzando 0,2 ml di RE (20 U / ml) e 1 ml di tampone 10x corrispondente in una reazione di 10 microlitri. Incubare per almeno 1 ora a 37 ° C e separato su un gel di agarosio all'1%. Selezionare per le colonie con il modello previsto di frammenti di DNA.
- Analizzare i vettori confermati (50 ng / ml) con (GGG GCG GAA TTC GAT ATC AAG) primer (5 pmol / mL) Tlox F (ATC ATG TCT GGA TCC CCA TC) e IRES R utilizzando sequenziamento Sanger.
3. Mesc RMCE mediata Targeting di Rescue Costruisce al R26 Locus
- Avviare una cultura di mESCs e il passaggio li KO RMCE-compatibile almeno due volte il MEF a medio Mesc FBS-based. Dividere i mESCs su un 6-pozzetti gelatinizzato.
- Il giorno successivo, aggiornare le cellule, a circa il 50% di confluenza, con 1.5mL di terreno Mesc FBS-based e trasfezione delle mESCs con Cre-escisso pRMCE-DV1 di targeting vettore contenente cDNA di soccorso.
- Fare un mix di DNA. Aggiungere 1 mg di colpire vettoriale e 1 mg di FLPE-plasmide di espressione 25-250 ml di terreno DMEM puro.
- Fare un mix lipofezione. Aggiungere 7 ml di reagente trasfezione lipofezione-based (ad esempio, Lipofectamine 2000) a 250 ml di terreno DMEM pura e incubare per 5 minuti a temperatura ambiente (RT).
NOTA: RMCE simile mira efficienze sono stati ottenuti utilizzando altri reagenti lipofezione-based (ad esempio, Lipofectamine LTX e Effectene). - Mescolare il mix di DNA con il mix lipofezione e incubare per 20 minuti a temperatura ambiente. Pipettare la miscela di trasfezione sulle mESCs rinfrescati. Agitare delicatamente e lasciare per una notte.
- Un giorno dopo la trasfezione, raggruppati tutti mESCs dal 6 pozzetti di una piastra di coltura da 10 cm con uno strato confluenti di MEF DR4 e m 10 ml di FBS-basedmedia ESC.
- Due giorni dopo la trasfezione, selezionare cloni Mesc con il corretto scambio cassette FLPE mediata aggiungendo 0,2 mg / ml di G418 (100x) al mezzo.
NOTA: Fare una curva uccidere per ogni lotto di G418 per identificare la più bassa concentrazione di G418 che uccide tutti i mESCs. - Aggiornare i mESCs quotidiano con il mezzo Mesc G418 contenente. Le colonie dovrebbero apparire dopo 7 - 10 giorni, in modo da raccogliere ed espandere questi come per step 1.4.
- Confermare i cloni corretti tramite PCR 11.
4. Differenziazione di mESCs in corpi embrionali (EBS)
- Avviare una cultura di mESCs KO con costrutti di soccorso R26-driven e il passaggio di loro almeno due volte il MEF a medio Mesc FBS-based. Passage le mESCs una volta su gelatinizzati 6 pozzetti per sbarazzarsi dei MEF.
- Lavare con PBS e trypsinize culture Mesc quasi confluenti. Piastra dissociato mESCs in diverse diluizioni (1/20 e 1/40) sulla non aderente, pet batterica gradori-piatti in terreno di differenziamento.
- Lasciare EBS per formare in questi piatti per 30 giorni. Aggiornare la media ogni 2 - 3 giorni con la seguente procedura: trasferire la sospensione EB ad un tubo da 50 ml, lasciate che i EBs depositano per gravità, rimuovere il surnatante, aggiungere mezzo fresco, e trasferire la sospensione EB di nuovo ad un batterica-grade piatto.
- Analizzare i mESCs mirati e EBs mediante immunofluorescenza e microscopia elettronica a trasmissione utilizzando protocolli che sono stati descritti in precedenza 12.
Risultati
La procedura di isolare linee KO Mesc RMCE-compatibile è illustrato nella figura 2. Due allevamenti consecutivi devono ottenere RMCE compatibile blastocisti KO. In primo luogo, i topi KO eterozigoti sono incrociate con i topi RMCE-compatibile, per ottenere topi KO eterozigoti RMCE-compatibili. Questi topi sono poi utilizzati per accoppiamenti a tempo con altri topi KO eterozigoti per ottenere 3,5-DPC, compatibile con RMCE, omozigoti blastocisti KO. La possibilità di ot...
Discussione
Il nostro metodo di isolamento Mesc è user-friendly e non richiede competenze o attrezzature avanzate, come la microchirurgia della blastocisti. Così, questa tecnologia è accessibile a una larga parte della comunità scientifica. Chiunque abbia esperienza di base coltura cellulare può propagare escrescenze ICM e stabilire linee mESCs. Tuttavia, il lavaggio e la manipolazione di blastocisti richiede una certa pratica. Una pipetta bocca è usato per trasferire blastocisti e consiste di una micropipetta, un supporto mi...
Divulgazioni
Gli autori non hanno nulla da rivelare.
Riconoscimenti
Ringraziamo Jinke D'Hont, Frederique Van Rockeghem, Natalie Farla, Kelly Lemeire, e Riet De Rycke per il loro eccellente supporto tecnico. Ringraziamo anche Eef Parthoens, Evelien Van Hamme e Amanda Goncalves dal Bioimmagini Nucleo strumento della infiammazione Centro di ricerca per la loro assistenza di esperti. Riconosciamo i membri del nostro gruppo di ricerca per le discussioni di valore. Questo lavoro è stato sostenuto dalla politica belga Science (Belspo Interuniversitario di attrazione Poli - Premio IAP VII-07 DevRepair; https://devrepair.be), dalla Fondazione Regina Elisabetta Medical, Belgio (GSKE 2008-2010; http: // www .fmre-gske.be), e dalle azioni concertate di ricerca (GOA) 01G01908 di Università di Gand, Belgio (http://www.ugent.be/en/ghentuniv). SG è un borsista postdottorato della Ricerca Fondi Fiandre (FWO-V).
Materiali
| Name | Company | Catalog Number | Comments |
| ROSALUC Mice | made in house | frozen sperm available upon request | |
| R26-iPSC mice | made in house | frozen sperm available upon request | |
| Vessel probe | Fine Science Tools | 10160-13 | to check for copulation plugs |
| M2 medium | Sigma-Aldrich | M7167 | make aliquots and store at -20 °C |
| Fine forceps (Dumont #5 Standard tip Student forceps) | Fine Science Tools | 11251-10 | spray with 70% EtOH before use (do not autoclave) |
| 23 G needles | Fine-ject | 8697 | |
| 1-mL syringes | Soft-ject | 6680 | |
| 60-mm bacterial grade plates (for flushing) | Gosselin | BB60-01 | |
| Mouth pipette | made in house | see discussion | |
| Mouse embryonic fibroblasts (MEFs, TgN (DR4)1 Jae strain) | ATTC | SCRC-1045 | |
| TgN (DR4)1 Jae mice | The Jackson Laboratory | 3208 | |
| Mitomycin C | Sigma-Aldrich | M0503 | |
| Phosphate buffered saline (PBS) without calcium or magnesium | Gibco | 14190-094 | |
| Tg(DR4)1Jae/J mice | JAX | 3208 | mice that contain four drug-selectable genes and DR4 MEFS confers resistance to neomycin, puromycin, hygromycin and 6-thioguanine |
| 0.1% Gelatin | Sigma-Aldrich | G1393 | Dissolve 0.5 g in 500 mL distilled water, autoclave and store at 4 °C. |
| Trypsin (0.25%) | Gibco | 25200-056 | |
| 2 μM pluripotin | Cayman Chemical | 10009557 | |
| pRMCE-DV1 | BCCM/LMBP collection | LMBP 08870 | public available from the BCCM/LMBP collection (http://bccm.belspo.be) |
| cre-excised pRMCE-DV1 | BCCM/LMBP collection | LMBP 08195 | public available from the BCCM/LMBP collection (http://bccm.belspo.be) |
| pCAG-FlpE-IRES-Puro-pA | Addgene | 20733 | |
| heat-shock competent DH5α bacteria | made in house | ||
| Gateway pDONR221 vector | Thermo Fisher | 12536-017 | contains kanamycin-resistance gene |
| BP clonase II mix | Thermo Fisher | 11789-020 | |
| LR clonase II mix | Thermo Fisher | 11791-020 | |
| Luria Broth (LB) | |||
| Ampicillin | |||
| Applied Biosystems 3730XL DNA Analyzer | Thermo Fisher | 3730XL | |
| G418 | Thermo Fisher | 11811-023 | |
| Lipofectamine 2000 transfection reagent | Thermo Fisher | 11668027 | |
| Lipofectamine LTX transfection reagent | Thermo Fisher | 15338100 | |
| Effectene transfection reagent | Qiagen | 301425 | |
| GATEWAY pENTR 1A vector | Thermo Fisher | A10462 | recombination-compatible vector |
| mouse monoclonal anti-p120ctn antibody | BD Transduction Laboratories | 610134 | |
| mouse monoclonal anti-Ecadherin antibody | BD Transduction Laboratories | 610181 | |
| General equipment | |||
| Sterile dissection tools | fine scissors and forceps for dissecting the uterus | ||
| Sterile pipettes: 5 mL, 10 mL and 25 mL | |||
| 15-mL and 50-mL conical centrifuge tubes | |||
| 96-well culture plates V-shaped bottom and flat bottom) | |||
| Culture dishes: 24 wells, 12 wells and 6 wells | |||
| Multichannel pipettes (to pipette 30, 50, 100 and 200 μL) | |||
| Sterile multichannel reservoirs | |||
| Access to a laminar air flow | |||
| Access to an incubator at 37 °C with 5% CO2 | |||
| Access to an inverted microscope | |||
| Access to a bench-top centrifuge | |||
| Access to a stereo microscope with transmitted-light | |||
| Culture media | |||
| MEF Medium | stored at 4 °C; warm 30 min at 37 °C before use | ||
| Dulbecco’s modified Eagle’s medium (DMEM) | Gibco | 41965-062 | |
| 10% fetal bovine serum (FBS) | Sigma-Aldrich | F-7524 | |
| L-glutamine (2 mM) | Gibco | 25030-024 | |
| Sodium pyruvate (0.4 mM) | Gibco | 11360-039 | |
| penicillin (100 U/mL) | Gibco | 15140-122 | |
| streptomycin (100 µg/mL) | Gibco | 15140-122 | |
| SR-based mESC medium | stored at 4 °C; warm 30 min at 37 °C before use | ||
| DMEM/F12 | Gibco | 31330-038 | mixed in a 1:1 ratio |
| 15% knock-out serum replacement (SR) | Gibco | 10828–028 | |
| L-glutamine (2 mM) | Gibco | 25030-024 | |
| 0.1 mM non-essential amino acids | Gibco | 11140-050 | |
| penicillin (100 U/mL) | Gibco | 15140-122 | |
| streptomycin (100 µg/mL) | Gibco | 15140-122 | |
| β-mercaptoethanol (0.1 mM) | Sigma-Aldrich | M 3148 | |
| 2,000 U/mL recombinant mouse LIF | (IRC/VIB Protein Service facility) | ||
| FBS-based mESC medium (similar to SR-based mESC medium) | stored at 4°C; warm 30 min at 37°C before use | ||
| Knockout DMEM | Gibco | 10829-018 | |
| 15% FBS | Hyclone | SH30070.03E | |
| Differention Medium | stored at 4 °C; warm 30 min at 37 °C before use | ||
| Iscove's Modified Dulbecco's Medium (IMDM) | Gibco | 21980-032 | |
| 15% FBS | Hyclone | SH30070.03E | |
| 5% CD Hybridoma Medium(1x) liquid | Gibco | 11279-023 | |
| 2 mM L-glutamine | Gibco | 25030-024 | |
| 0.4 mM 1-thioglycerol | Sigma-Aldrich | M-6145 | |
| 50 μg/mL ascorbic acid | Sigma-Aldrich | A-4544 | |
| penicillin (100 U/mL) | Gibco | 15140-122 | |
| streptomycin (100 µg/mL) | Gibco | 15140-122 | |
| 2i | |||
| 1 μM Erk inhibitor PD0325901 | Axon Medchem | Axon 1408 | |
| 3 μM Gsk3 inhibitor CHIR99021 | Axon Medchem | Axon 1386 |
Riferimenti
- Gul, I. S., Hulpiau, P., Saeys, Y., van Roy, F. Metazoan evolution of the armadillo repeat superfamily. Cell Mol Life Sci. , (2016).
- Valenta, T., Hausmann, G., Basler, K. The many faces and functions of beta-catenin. EMBO J. 31, 2714-2736 (2012).
- Pieters, T., van Hengel, J., van Roy, F. Functions of p120ctn in development and disease. Front Biosci. 17, 760-783 (2012).
- Keirsebilck, A., et al. Molecular cloning of the human p120ctn catenin gene (CTNND1): Expression of multiple alternatively spliced isoforms. Genomics. 50, 129-146 (1998).
- Capecchi, M. R. Gene targeting in mice: functional analysis of the mammalian genome for the twenty-first century. Nat Rev Genet. 6, 507-512 (2005).
- Wang, H., et al. One-step generation of mice carrying mutations in multiple genes by CRISPR/Cas-mediated genome engineering. Cell. 153, 910-918 (2013).
- Yang, H., et al. One-step generation of mice carrying reporter and conditional alleles by CRISPR/Cas-mediated genome engineering. Cell. 154, 1370-1379 (2013).
- Fu, Y., et al. High-frequency off-target mutagenesis induced by CRISPR-Cas nucleases in human cells. Nat Biotechnol. 31, 822-826 (2013).
- Branda, C. S., Dymecki, S. M. Talking about a revolution: The impact of site-specific recombinases on genetic analyses in mice. Dev Cell. 6, 7-28 (2004).
- Sandhu, U., et al. Strict control of transgene expression in a mouse model for sensitive biological applications based on RMCE compatible ES cells. Nucleic Acids Res. 39, 1 (2010).
- Haenebalcke, L., et al. Efficient ROSA26-based conditional and/or inducible transgenesis using RMCE-compatible F1 hybrid mouse embryonic stem cells. Stem Cell Rev. 9, 774-785 (2013).
- Pieters, T., et al. p120 Catenin-Mediated Stabilization of E-Cadherin Is Essential for Primitive Endoderm Specification. PLoS Genet. 12, e1006243 (2016).
- Evans, M. J., Kaufman, M. H. Establishment in culture of pluripotential cells from mouse embryos. Nature. 292, 154-156 (1981).
- Martin, G. R. Isolation of a pluripotent cell line from early mouse embryos cultured in medium conditioned by teratocarcinoma stem cells. Proc Natl Acad Sci U S A. 78, 7634-7638 (1981).
- Cheng, J., Dutra, A., Takesono, A., Garrett-Beal, L., Schwartzberg, P. L. Improved generation of C57BL/6J mouse embryonic stem cells in a defined serum-free media. Genesis. 39, 100-104 (2004).
- Bryja, V., et al. An efficient method for the derivation of mouse embryonic stem cells. Stem Cells. 24, 844-849 (2006).
- Ying, Q. L., et al. The ground state of embryonic stem cell self-renewal. Nature. 453, 519-523 (2008).
- Chen, S., et al. Self-renewal of embryonic stem cells by a small molecule. Proc Natl Acad Sci U S A. 103, 17266-17271 (2006).
- Pieters, T., et al. Efficient and User-Friendly Pluripotin-based Derivation of Mouse Embryonic Stem Cells. Stem Cell Rev. 8, (2012).
- Yang, W., et al. Pluripotin combined with leukemia inhibitory factor greatly promotes the derivation of embryonic stem cell lines from refractory strains. Stem Cells. 27, 383-389 (2009).
- Haenebalcke, L., et al. The ROSA26-iPSC Mouse: A Conditional, Inducible, and Exchangeable Resource for Studying Cellular (De)Differentiation. Cell Rep. 3, (2013).
- Tucker, K. L., Wang, Y., Dausman, J., Jaenisch, R. A transgenic mouse strain expressing four drug-selectable marker genes. Nucleic Acids Res. 25, 3745-3746 (1997).
- Nyabi, O., et al. Efficient mouse transgenesis using Gateway-compatible ROSA26 locus targeting vectors and F1 hybrid ES cells. Nucleic Acids Res. 37, 55 (2009).
- Katzen, F. Gateway((R)) recombinational cloning: a biological operating system. Expert Opin Drug Discov. 2, 571-589 (2007).
- Schaft, J., Ashery-Padan, R., vander Hoeven, F., Gruss, P., Stewart, A. F. Efficient FLP recombination in mouse ES cells and oocytes. Genesis. 31, 6-10 (2001).
- Spencer, H. L., et al. E-cadherin inhibits cell surface localization of the pro-migratory 5T4 oncofetal antigen in mouse embryonic stem cells. Mol Biol Cell. 18, 2838-2851 (2007).
- Stryjewska, A., et al. Zeb2 Regulates Cell Fate at the Exit from Epiblast State in Mouse Embryonic Stem Cells. Stem Cells. , (2016).
- Nagy, A., Gertsenstein, M., Vintersten, K., Behringer, R. . Manipulating the mouse embryo: A laboratory manual. , (2003).
- van den Berghe, V., et al. Directed migration of cortical interneurons depends on the cell-autonomous action of Sip1. Neuron. 77, 70-82 (2013).
- Li, J., et al. The EMT transcription factor Zeb2 controls adult murine hematopoietic differentiation by regulating cytokine signaling. Blood. , (2016).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon