Method Article
Una spettroscopia Raman integrata e una piattaforma di spettrometria di massa per studiare l'assorbimento di farmaci a singola cellula, il metabolismo e gli effetti
In questo articolo
Riepilogo
Questo protocollo presenta una piattaforma integrata di spettroscopia-massa spectromo (MS) in grado di raggiungere la risoluzione a cella singola. La spettroscopia raman può essere utilizzata per studiare la risposta cellulare ai farmaci, mentre la SM può essere utilizzata per l'analisi mirata e quantitativa dell'assorbimento e del metabolismo dei farmaci.
Abstract
Le cellule sono noti per essere intrinsecamente eterogenee nelle loro risposte ai farmaci. Pertanto, è essenziale che l'eterogeneità a una cellula sia spiegata negli studi sulla scoperta di farmaci. Ciò può essere ottenuto misurando con precisione la pletora di interazioni cellulari tra una cellula e un farmaco a livello di singola cellula (ad esempio, assunzione di farmaci, metabolismo ed effetto). Questo documento descrive una piattaforma di spettroscopia Raman e spettrometria di massa (MS) a cella singola per monitorare i cambiamenti metabolici delle cellule in risposta ai farmaci. Utilizzando questa piattaforma, i cambiamenti metabolici in risposta al farmaco possono essere misurati dalla spettroscopia Raman, mentre il farmaco e il suo metabolita possono essere quantificati utilizzando la spettrometria di massa nella stessa cellula. I risultati suggeriscono che è possibile accedere alle informazioni sull'assorbimento di droga, metabolismo, e la risposta a livello di singola cellula.
Introduzione
Le cellule rispondono in modo diverso ai cambiamenti nel loro microambiente a livello di singola cellula, un fenomeno chiamato eterogeneità cellulare1. Nonostante questo, gli attuali studi di scoperta di farmaci si basano su misurazioni medie delle popolazioni di cellule, che offuscano le informazioni sulle potenziali sottopopolazioni e le variazioni monocellulari2. Queste informazioni mancanti possono spiegare perché alcune cellule sono più suscettibili ai farmaci, mentre altri sono resistenti. È interessante notare che la mancanza di informazioni unicellulari sulla risposta ai farmaci è una possibile ragione per il fallimento degli studi clinici di fase II dei farmaci3. Pertanto, per affrontare questo problema, le interazioni cellulari con il farmaco (cioè l'assorbimento, il metabolismo e la risposta) devono essere misurate a livello di singola cellula.
Per raggiungere questo obiettivo, abbiamo progettato un sistema unico in cui le singole cellule viventi vengono sottoposte a screening utilizzando la spettroscopia Raman senza etichette, quindi ulteriormente caratterizzata utilizzando la spettrometria di massa4. La spettroscopia Raman fornisce un'impronta molecolare dello stato cellulare, uno spettro complesso derivante dai contributi di molte molecole all'interno della cellula. Nonostante questa complessità, si può considerare che le impronte digitali Raman riflettono la struttura e il metabolismo di un'intera cellula5,6. La spettroscopia Raman eccelle nella misurazione degli stati cellulari in modo non invasivo e relativamente elevato, il che lo rende utile per lo screening e la valutazione della risposta ai farmaci a livello di singola cellula.
Al contrario, la SM fornisce la sensibilità e la selettività necessarie per misurare l'assorbimento di droga a livello di singola cellula. Poiché la SM è distruttiva (il campione [cellula] viene in genere consumato durante l'analisi), integrarlo con spettroscopia Raman non distruttiva e priva di etichette può fornire un'elevata velocità effettiva e un sistema sensibile. Questa piattaforma combinata è in grado di fornire ulteriori informazioni sull'assorbimento di droga, metabolismo, ed effetti a livello di singola cellula.
Questo manoscritto chiarisce un protocollo utilizzato per studiare le interazioni cellulari con i farmaci a livello di singola cellula utilizzando colture in vitro utilizzando una piattaforma Raman-MS integrata. Per fare ciò, le cellule carcinoma epatocellulare (HepG2) e il tamoxifene vengono utilizzate come modello. Cellule di HepG2 sono state scelte perché prendono tamoxifene e metabolizzare il farmaco, e sono contemporaneamente colpiti a causa dei suoi effetti epatotossici. Due stati sono utilizzati in questo manoscritto: cellule trattate con farmaci contro cellule non trattate (controllo).
Protocollo
1. Cultura cellulare
- cellule di coltura di interesse in un supporto di coltura appropriato. La penicillina-streptomicina può essere aggiunta per evitare la contaminazione. Nel caso delle cellule HepG2, le cellule di coltura in un supporto di coltura contenente il mezzo di Aquila modificato di Dulbecco (DMEM) sono state integrate con 10% siero bovino fetale (FBS) e 0,1% penicillina-streptomicina. Per facilare le misurazioni della spettroscopia Raman, le cellule possono essere coltivate su un piatto in vetro rivestito di gelatina dello 0,1% o vetrini al quarzo.
- Incubare le cellule per 2 giorni a 37 e 5% DI CO2 in un'incubatrice umidificata.
- Sincronizza le colture cellulari per raggiungere la confluenza del 70%.
- Le cellule di sottocoltura in una piastra di griglia inferiore in vetro da 35 mm o vetrini al quarzo utilizzando lo stesso mezzo ad una densità di semina di 0,7 x 106, quindi incubano a 37 gradi centigradi per 24 h.
NOTA: I piatti di coltura o i vetrini possono essere pre-rivestiti con una soluzione di rivestimento di collagene o gelatina con un rapporto di superficie di coltura di 5 ug/cm2 per consentire loro di fissare, garantendo la loro sopravvivenza durante la misurazione.
2. Trattamento della droga
- Rimuovere le colture cellulari dall'incubatrice e lavare 2x con tampone PBS preriscaldato (37 gradi centigradi).
NOTA: È ottimale rimuovere le cellule per il trattamento farmacologico a una confluenza del 50%-60%. - Dividere le cellule in sottogruppi trattati con farmaci e non trattati in piatti di coltura 35 mm.
- Mescolare la droga di scelta con i media di coltura. Ad esempio, sciogliere il tamoxifene nel solforosi dimetilo (DMSO) e mescolare con i mezzi di coltura per ottenere un volume finale di 2 mL e tamoxifene di 10 mL. Questo sarà il gruppo trattato con la droga.
- Mescolare un volume corrispondente di solvente (DMSO) nel mezzo come controllo per studiare gli effetti del DMSO. Questo sarà il gruppo di controllo.
- Incubare entrambi i gruppi in 2 mL del supporto a spillo preparato nei passi 2.3-2.4 per 24 h. La confluenza prevista dopo l'incubazione dovrebbe essere del 70%-80%.
3. Imaging spettrale Raman ed elaborazione spettrale
NOTA: Sebbene i sistemi di spettroscopia Raman siano disponibili in commercio, il sistema di spettroscopia Raman utilizzato qui è un microscopio confocale a scansione lineare costruito in casa descritto in precedenza7,8. In breve, questo sistema è dotato di un laser a stato solido pompato da 532 nm. La luce laser viene modellata in un piano utilizzando una lente cilindrica, che consente la misurazione di 400 spettri in un'unica esposizione. Gli spettri Raman sono stati registrati utilizzando una telecamera CCD raffreddata montata su un policromatore che utilizza una griglia 1.200 scanalature/mm per massimizzare la risoluzione spettrale della regione delle impronte digitali (da 500-1.800 cm-1). Questa area spettrale contiene un'alta densità di frequenze specifiche per le molecole che genera la dispersione Raman. Viene utilizzata anche una lente per l'acqua-immersione (NA - 0,95). La risoluzione spaziale di questo sistema è di 300 nm e la risoluzione spettrale è 1 cm-1. Per garantire la sopravvivenza delle cellule durante l'esperimento, viene utilizzata una microcamera fissata su uno stadio motorizzato al microscopio.
- Prima delle misurazioni spettrali, verificare l'allineamento dell'ottica. Per verificare che il foro stenopeico e la posizione laser corrispondano esattamente. Inserire lo spettrofotometro fessura quando si restringe il più possibile.
- Utilizzare l'etanolo per calibrare lo spettrofotometro prima di ogni esperimento. Per farlo, posiziona EtOH in un piatto con fondo di vetro, misura lo spettro a una data intensità laser (misurata al campione) per 1 s e associa il picco alle lunghezze d'onda note7.
- Ridurre l'intensità del laser al campione a 2,4 mW/m2 in modo che le cellule sopravvivano all'esposizione al laser.
- Impostare la microcamera al 5% di CO2 e 37 gradi centigradi.
- Una volta che il sistema di microscopio è pronto, rimuovere le cellule dall'incubatrice e risciacquare immediatamente le cellule 2x con tampone PBS (37 gradi centigradi) riscaldato, quindi aggiungere 2 mL di PBS riscaldato (37 o C) o DMEM per risospendere le cellule.
NOTE: Sia i supporti basati su PBS che FluoroBrite DMEM sono opzioni sufficienti per le misurazioni della spettroscopia Raman perché producono un segnale di fondo minimo. - Aggiungere 10 l'acqua sulla lente dell'obiettivo di immersione dell'acqua e posizionare delicatamente la teglia a cellule del fondo di vetro sullo stadio del microscopio.
- Misurare ogni cella concentrando la linea laser. Un tempo di esposizione di 15 s per cella è sufficiente qui per ottenere una sezione trasversale di una cella con un segnale Raman chiaro. Uno specchio galvano consente la scansione di una cella o di un gruppo di celle in diverse decine di minuti.
NOTE: Una maggiore risoluzione dell'imaging spettrale completo delle cellule richiede più tempo e può causare fotodanni. Qui, le cellule sono state misurate utilizzando un'esposizione a riga singola per ottenere una sezione trasversale di ogni cellula. Questo approccio è un buon compromesso per aumentare la produttività e ottenere informazioni sufficienti per discriminare le cellule, garantendo al contempo la vitalità delle cellule limitando i danni fotografici.
4. Pre-elaborazione di dati spettrali e analisi multivariate
NOTA: la pre-elaborazione è un passaggio necessario prima dell'analisi aggiuntiva per rimuovere le variazioni tecniche indesiderate all'interno dei dati spettrali. A causa della diversità dei metodi e del software, non è possibile fornire un elenco esaustivo e ci sono molte recensioni utili trovate nella letteratura7,8. In questa sezione viene brevemente descritto l'approccio utilizzato per analizzare e interpretare i dati spettrali Raman ottenuti da singole cellule viventi.
- Estrarre e pre-elaborare le immagini Raman per rimuovere possibili interferenze cosmiche dei raggi.
NOTA: L'asse spettrale degli spettri ottenuti durante diversi giorni/settimane/mesi può includere alcune variazioni dovute a piccole variazioni tecniche durante la calibrazione utilizzando l'etanolo. Ciò avrà un forte impatto sulle successive analisi multivariate e sui confronti statistici. Nel caso in cui gli esperimenti vengano eseguiti durante settimane/mesi diversi, si prevedono piccole variazioni ottiche. In questo caso, i dati devono essere interpolati per correggere eventuali spostamenti spettrali dei dati tra gli esperimenti. Qui viene utilizzata l'interpolazione con spline cubica. Dopo questo passaggio, tutti gli assi degli spettri devono essere allineati. Un intervallo di 500-1.800 cm-1 è considerato per l'analisi successiva. - Estrarre i dati spettrali delle celle e dello sfondo (assenza di celle) da ogni immagine utilizzando un algoritmo fatto in casa. Sottrarre il segnale di sfondo dal segnale della cella. Quindi, calcolare la media degli spettri dei pixel rimanenti, che devono corrispondere a una singola cella. I passaggi seguenti vengono eseguiti utilizzando gli spettri 2D delle celle.
- Eseguire una correzione della linea di base utilizzando il ModPoly9 o qualsiasi altro algoritmo che ha stimato di adattarsi sufficientemente. Tagliare la gamma spettrale a 600-1.700 cm-1 per selezionare la regione di impronte digitali e assicurarsi che non vi siano effetti indesiderati del bordo sugli spettri a causa di un cattivo raccordo polinomiale.
- Eseguire un passaggio di normalizzazione come la normalizzazione vettoriale (l'intensità a ogni numero d'onda è divisa per la norma l2 globale o il valore massimo singolare di uno spettro) per normalizzare l'intensità spettrale10, anche se altre normalizzazioni possono essere considerate.
- Preparare un set di dati con l'etichetta appropriata per ogni classe/condizione.
NOTA: È possibile analizzare comparativamente le analisi spettrali per esplorare la natura delle possibili differenze tra la classe/condizioni della cellula (ad esempio, sottraendo lo spettro medio del gruppo di controllo ad altri gruppi per identificare le regioni di interesse [come i potenziali biomarcatori]). I calcoli dei punteggi ANOVA e Fisher possono anche essere eseguiti10. - Per identificare le cellule trattate e non trattate in base alle caratteristiche spettrali, è possibile applicare analisi multivariate. I dati spettrali normalizzati devono essere usati come set di dati di training e un set di dati sconosciuto (senza etichetta) di un esperimento di replica deve essere usato come dati di test, se possibile.
NOTA: l'analisi discriminante eseguita su un vettore di un'analisi dei componenti principali (PCA-DA10),la proiezione sui punteggi latenti seguita dall'analisi discriminante (PLS-DA) e le macchine vettoriali di supporto (SVM)11 sono modelli spesso utilizzati sul campo e ciascuno presenta diverse considerazioni statistiche. Di conseguenza, è necessario eseguire la pre-elaborazione dei dati. - Usare l'apprendimento automatico che si adatta agli obiettivi sperimentali. Qui, una proiezione sul modello di struttura latente (PLS) viene costruita utilizzando la regione di impronte spettrali degli spettri Raman (600-1,710 cm-1)11,12. Media-centra i dati in base alle esigenze. Per la convalida incrociata del modello, è possibile applicare diverse tecniche.
NOTA: Qui viene applicata una cross-validation cieca veneziana con 10 divisioni. La complessità del modello (numero di componenti o variabile latente) deve essere testata in modo che il modello migliore riduca al minimo il valore di errore quadrato medio radice (RMSE). Si è scoperto che tre vettori latenti (LV) hanno fornito la migliore discriminazione con il nostro set di dati. - Identificare quali picchi spettrali Raman contribuiscono alla discriminazione delle cellule (ad esempio, tracciando il punteggio di importanza variabile nella proiezione [VIP] per ogni numero d'onda Raman o la grandezza del coefficiente di regressione).
NOTE: Il punteggio VIP di una variabile viene calcolato come somma ponderata delle correlazioni al quadrato tra i componenti PLS-DA e la variabile originale. Dettagli per quanto riguarda PLS e VIP punteggi algoritmo possono essere trovati nella letteratura11,12.
5. Impostazione e procedure di campionamento a cella singola
- Fissare il sistema di campionamento cellulare sul microscopio di Raman, come illustrato nella Figura 1. Collegare il micromanipolatore 3D al supporto capillare di vetro collegato a una siringa vuota per succhiare il campione applicando una pressione negativa (Figura 1).
- Impostare il microscopio su un campo di ingrandimento elevato (40x) per osservare la punta del vetro capillare e assicurarsi che non sia rotto. Controllare la posizione del vetro capillare utilizzando il micromanipolatore (x-, y-, z-axes). Assicurarsi che la punta capillare sia centrata nel campo visivo, quindi spostare il capillare sull'asse z per dare spazio al piatto di coltura in un secondo momento.
NOTA: Il microcampionamento delle cellule viene eseguito dal capillare del vetro per le cellule con diametri compresi tra 10-15 m. Si raccomanda un capillare di dimensioni del foro di 5 m. Se la dimensione del foro è troppo piccola, la punta capillare sarà tappata dalla cellula, e se è troppo grande, la sensibilità delle successive misurazioni della SM potrebbe essere compromessa. - Posizionare la piastra/piatto campione sullo stage del microscopio, regolare l'ingrandimento e mettere a fuoco, selezionare la cella di destinazione sulla piastra della griglia e spostarla al centro della vista. Quindi, abbassare con attenzione il vetro capillare utilizzando micromanipolatore (asse z) fino a quando la punta entra a fuoco.
NOTA: Assicurarsi di non spostare il capillare negli assi x e y fino a quando il capillare non è a fuoco. - Sotto osservazione microscopica, toccare la singola cellula bersaglio con la punta capillare, quindi iniziare ad applicare una pressione negativa utilizzando la siringa per intrappolare la cellula all'interno della punta capillare. Registrare questa procedura scattando una foto o un video per controllare con precisione la tempistica e la posizione della cella, se necessario.
- Spostare il capillare verso l'alto sull'asse z. Quindi, staccare il capillare dal supporto capillare utilizzando pinze in preparazione per l'analisi MS.
6. Misurazioni della spettrometria di massa
- Calibrare la precisione di massa dello strumento MS secondo le raccomandazioni del produttore. Dopo la calibrazione, assicurarsi che l'errore di massa non sia maggiore di 3 ppm.
- Ottimizzare lo strumento MS a parametri più adatti per l'analita di interesse.
NOTA: Nel caso di tamoxifene e 4-OHT analisi, i parametri dello strumento sono impostati sul seguente: ingresso capillare temperatura: 400 C, tensione a spruzzo: 1500 V, obiettivo di controllo automatico del guadagno (AGC): 5.00E-06, livello RF S-lens: 90%, gamma SIM: 347-397 m/z, Tempo massimo di iniezione SIM: 200 ms, risoluzione SIM: 140.000 FWHM, gamma MS/MS: 50-400 m/z, MS/MS AGC target: 2.00E-05, MS/MS tempo massimo di iniezione: 100 ms, risoluzione MS/MS: 17,500 FWHM, MS/MS finestra di isolamento: 1 m/z, MS/MS(MS). - Impostare un metodo di acquisizione automatica con una durata di 5 min per la modalità SIM per ottenere la quantificazione relativa, e un altro metodo MS / MS per l'identificazione positiva del farmaco e il suo metabolita. I parametri del metodo di acquisizione devono essere impostati sui valori ottimizzati indicati nel passaggio 6.2.
- Preparare il solvente per la ionizzazione sotto un cofano di fumi. La composizione del solvente dipende dall'analita di interesse. Qui, il solvente organico utilizzato è costituito da 80% MeOH, 10% DMSO, e 0.1% acido formico.
- Mescolare uno standard interno appropriato con il solvente organico prima delle misurazioni. In questo esperimento, 5,31 nM di d5-tamoxifene viene utilizzato come standard interno.
- Per evitare falsi positivi, aspirare i supporti che circondano le cellule trattate con il farmaco utilizzando un capillare di dimensioni 1 m con costante osservazione microscopica per evitare il campionamento di parti cellulari.
- Aggiungere 2 -L del solvente per ionizzazione all'estremità larga del capillare contenente il supporto utilizzando una pipetta attaccata alle punte del caricatore. Quindi, analizzare il supporto campionato dalla SM, verificare la presenza dell'analita di interesse (normalmente, non dovrebbe essere rilevabile).
- Aggiungere 2 - L il solvente di ionizzazione al capillare contenente la cellula, fissare il capillare a un adattatore nanoelettrospray (nano-ESI) collegato ad un adeguato spettrometro di massa e avviare il metodo di acquisizione automatica.
7. Elaborazione e analisi dei dati di spettrometria di massa
NOTA: Qualsiasi software adatto può essere utilizzato per eseguire l'analisi dei dati. Tuttavia, se i ricercatori desiderano eseguire l'analisi dei dati utilizzando un software che non è fornito dal fornitore MS, quindi i dati grezzi devono essere convertiti dal formato fornitore proprietario in un formato aperto o come un file di testo prima (che è stato fatto qui).
- Normalizzare i dati dividendo l'area di picco dell'analita per quella dello standard interno dalla stessa scansione MS. Quindi, log trasformare i rapporti di picco per ridurre l'asimmetria.
- Tracciare l'intensità normalizzata del farmaco o del suo metabolita come boxplot o curva di densità per visualizzare la distribuzione tra singole cellule. Qui, viene utilizzato il software statistico R, insieme al pacchetto ggplot2.
- Calcolare il farmaco metabolizzato al rapporto farmacologico non metabolizzato dividendo l'abbondanza del metabolita del farmaco per quella della molecola genitore non metabolizzata in ogni cellula (cioè, 4-OHT e tamoxifene, rispettivamente.
NOTA: La correlazione tra variazioni di specifici picchi di interesse Raman e le variazioni nei picchi di SM del farmaco o dei suoi metaboliti può essere studiata. Questa è un'aggiunta alla possibile correlazione tra il farmaco stesso e il suo metabolita in singole cellule. Questo può essere fatto calcolando il coefficiente di correlazione di Pearson utilizzando un test a due code. Dovrebbero essere presi in considerazione anche approcci integrativi più avanzati.
Risultati
L'analisi a singola cellula delle interazioni farmacologiche (assorbimento, metabolismo ed effetti) è essenziale per scoprire qualsiasi sottopopolazione nascosta o resistente ai farmaci e comprendere gli effetti dell'eterogeneità cellulare. In questo protocollo, sono state utilizzate due tecniche complementari per misurare le interazioni di cui sopra nelle singole cellule: la spettroscopia Raman e la spettrometria MS Raman identifica rapidamente le cellule colpite da farmaci a base di biomarcatori spettrali della risposta al farmaco. La SM viene utilizzata per monitorare l'assorbimento e il metabolismo del farmaco in modo selettivo e semi-quantitativo. Le cellule sono state prima vagliate dalla spettroscopia Raman e poi campionate individualmente per l'analisi dalla SM.
Un'analisi comparativa dello spettro medio di ogni condizione (con e senza trattamento farmacologico) è illustrata nella figura 2. Lo spettro medio delle due condizioni differisce chiaramente a vari picchi, che sono stati precedentemente identificati e assegnati ai composti molecolari2. In particolare, i picchi a 1000 cm- (assegnati a composti aromatici come fenilalanina e tirosina) mostrano forti differenze. L'importanza della differenza statistica dovrebbe essere valutata mediante ulteriori analisi multivariate.
Il set di dati è stato poi utilizzato per addestrare un modello PLS (passaggi 4.5-4.8) volto a distinguere i due trattamenti cellulari (con la droga: n - 290, senza droga: n - 115). La capacità predittiva di classificare le cellule coltivate in presenza di tamoxifene ha raggiunto il 100% di sensibilità e il 72% di specificità nei dati di test (sconosciuta dal modello addestrato convalidate incrociate). La sensibilità è una misura dei veri positivi che sono correttamente identificati dal modello, mentre la specificità è una misura dei negativi effettivi identificati dal modello. Modelli alternativi come SVM, LDA e reti neurali possono fornire risultati simili o migliori, anche se un confronto completo non è stato eseguito in questo studio.
Sulla base del modello PLS, sono stati calcolati i punteggi VIP, che rappresentano l'importanza delle lunghezze d'onda (spostamenti Raman) nella discriminazione delle condizioni sperimentali (Figura 3). È importante sottolineare che le vette più alte dei profili VIP corrispondevano alle vette Raman per le quali si sono osservate forti differenze tra i due trattamenti. Ciò ha confermato le differenze molecolari specifiche tra cellule trattate e non trattate. Di conseguenza, i ricercatori possono identificare possibili biomarcatori spettrali che riflettono la risposta delle singole cellule al trattamento farmacologico. Questi biomarcatori possono essere testati ulteriormente per verificarne la rilevanza biologica e la generalizzazione attraverso varie condizioni e linee cellulari.
Un sistema di spettrometria di massa unicellulare dal vivo (LSC-MS) è stato in grado di rilevare sia il farmaco che i suoi metaboliti in singole cellule HepG2 trattate con farmaci precedentemente misurate dalla spettroscopia Raman. Inoltre, la SM in tandem può essere utilizzata per confermare la struttura di entrambe le molecole. Dopo l'identificazione positiva, l'abbondanza relativa del farmaco e dei suoi metaboliti sono stati misurati in ogni cellula e confrontati con picchi di fondo nelle cellule non trattate. Forte variazione è stata osservata nell'abbondanza di tamoxifene, e questo fenomeno è stato ancora più pronunciato nel caso del suo metabolita, 4-OHT (Figura 4). È stata studiata anche la relazione tra l'abbondanza di tamoxifene e i suoi metaboliti, in cui è stata osservata una correlazione positiva significativa tra i due (r - 0,54, p - 0,0001, n - 31).

Figura 1: Sistema di raccolta delle celle montato in fase di microscopio. Fare clic qui per visualizzare una versione più grande di questa figura.
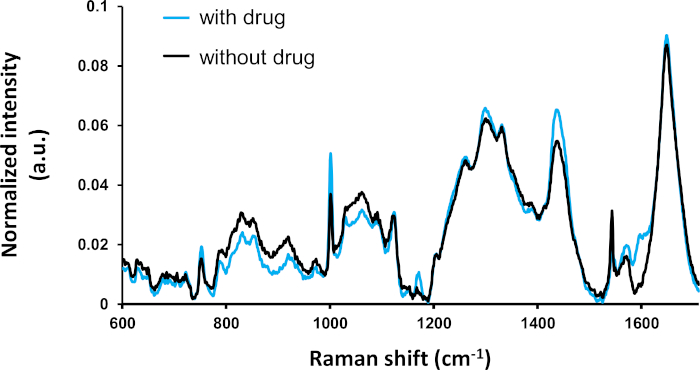
Figura 2: Spettro medio delle cellule trattate con farmaci (con tamoxifene: n - 295) e cellule non trattate (senza tamoxifene: n Le cime di Raman possono essere identificate dalla letteratura. La maggior parte delle forti differenze spettrali sono statisticamente significative (ANOVA, p - 0,5) come descritto in precedenza4. Questa cifra è stata modificata da una precedente pubblicazione4. Fare clic qui per visualizzare una versione più grande di questa figura.
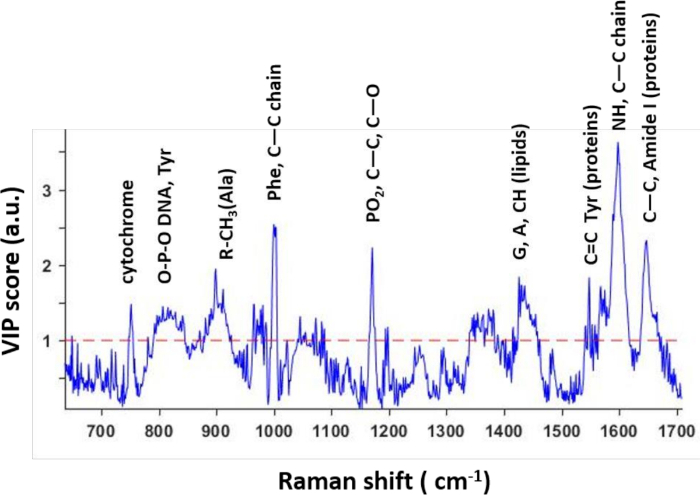
Figura 3: punteggi VIP estratti dal modello PLS predittivo. I punteggi VIP riflettono le lunghezze d'onda che contribuiscono a distinguere tra le due classi nel modello. La maggior parte dei picchi corrispondono a molecole specifiche che vengono osservate come biomarcatori spettrali degli effetti dei farmaci sulle cellule trattate con farmaci. Questa cifra è stata modificata da una precedente pubblicazione4. Fare clic qui per visualizzare una versione più grande di questa figura.
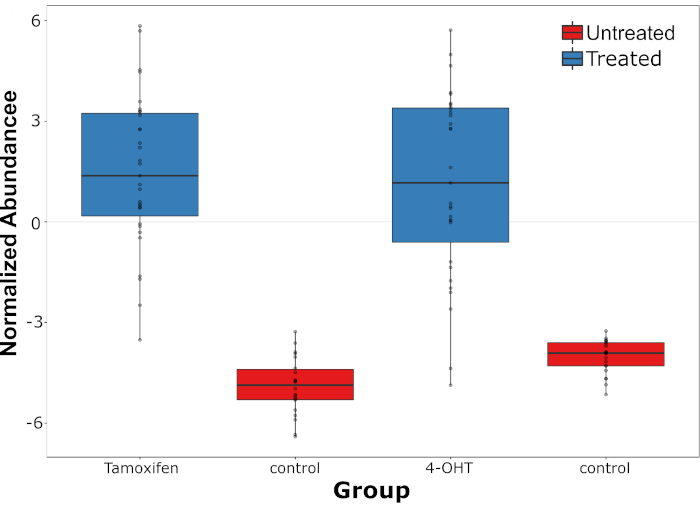
Figura 4: Distribuzione dell'abbondanza di tamoxifene e del suo metabolita. Distribuzione dell'abbondanza di tamoxifene e del suo metabolita, 4-OHT (misurato a livello di singola cellula) rispetto ai picchi endogeni nelle cellule non trattate (controllo). Questa cifra è stata modificata da una precedente pubblicazione4. Fare clic qui per visualizzare una versione più grande di questa figura.
Discussione
In questo manoscritto, è stato scelto un semplice caso in cui le cellule HepG2 sono state esposte (o meno) a tamoxifene. La capacità di un sistema di spettroscopia Raman e spettrometria di massa è dimostrata per monitorare gli effetti del tamoxifene sulle cellule. La spettroscopia Raman ha permesso l'identificazione di potenziali biomarcatori che riflettevano una risposta generale delle singole cellule all'esposizione ai farmaci. È stata osservata una certa eterogeneità tra singole cellule, suggerendo che alcune cellule non hanno risposto all'esposizione ai farmaci. D'altra parte, LSC-MS era in grado di eseguire un'analisi mirata del farmaco e del suo metabolita a livello di singola cellula, in cui è stato osservato un alto grado di eterogeneità nel farmaco e la sua abbondanza di metaboliti. Questa eterogeneità aiuta a spiegare perché alcune cellule sono colpite dal farmaco, mentre altre non sono apparentemente, nonostante le cellule provenienti da una popolazione apparentemente uniforme12.
Tra gli aspetti particolari di questa tecnica che richiedono attenzione, è importante valutare la qualità della configurazione del microscopio e l'elaborazione del segnale per garantire la riproducibilità dei dati. Se la pre-elaborazione degli spettri viene eseguita con attenzione, le variazioni del segnale devono essere massimizzate al massimo locale di ogni picco. Al contrario, la linea di base e il bordo degli spettri dovrebbero sovrapporsi tra le condizioni delle celle testate. Un altro aspetto importante è il modello multivariato utilizzato per studiare le differenze tra i trattamenti. È necessario valutare attentamente i modelli e i parametri del modello per garantire un'analisi precisa e accurata. Un vantaggio del modello PLS, a differenza delle reti neurali, è che consente l'accesso ai pesi associati a ogni lunghezza d'onda (spostamenti Raman) che meglio distinguono le condizioni testate dal modello.
Nonostante la spettroscopia Raman abbia discriminato con successo la risposta al farmaco, va sottolineato che questa tecnica è limitata nel suo uso per fornire l'interpretazione biologica. Ciò è dovuto principalmente alla complessità del segnale spettrale, che comprende una miscela di migliaia di molecole. Pertanto, sono necessarie ulteriori indagini per valutare le variazioni sistematiche tra le intensità spettrali Raman e le variazioni nelle concentrazioni di droga. Inoltre, studi simili di altre linee cellulari sono necessari per valutare la generalizzazione dei biomarcatori spettrali associati al tamoxifene.
Inoltre, può essere di interesse eseguire misurazioni dei tessuti viventi per valutare la farmacodinamica e studiare come i farmaci penetrano e scorrono all'interno di ogni cellula. Inoltre, va notato che la fase di campionamento in LSC-MS dipende fortemente dalle competenze dell'operatore. Parametri come la risoluzione spaziale, la posizione della cella all'interno del capillare dopo il campionamento e la resistenza alla velocità effettiva dipendono dall'operatore, il che limita l'adozione su larga scala di LSC-MS. Anche se, sistemi di campionamento automatizzato possono alleviare questo problema. Inoltre, mentre LSC-MS eccelle nel campionamento di cellule aderenti o galleggianti nei loro stati nativi, si comporta in modo più scarso nel campionamento delle cellule incorporate nelle sezioni di tessuto. Ciò è dovuto alla tendenza della punta capillare di campionamento a rompersi se la densità del campione è alta. Pertanto, un altro approccio come il single-probe può essere più adatto in questi casi14,15.
Poiché le cellule qui utilizzate sono campionate in condizioni ambientali con una preparazione minima del campione, LSC-MS può essere facilmente integrato con altre tecnologie, come dimostra la sua integrazione con Raman in questo protocollo. Un'altra integrazione simile con l'olografia 3D ha permesso di ottenere la quantificazione assoluta dei metaboliti cellulari al livello subcellulare16. Inoltre, l'integrazione con la citometria di flusso ha permesso di scoprire i biomarcatori metabolici in singole cellule tumorali circolanti di pazienti affetti da cancro al neuroblastoma17,18.
In futuro, a causa del recente crescente interesse a combinare set di dati da modalità di imaging19, potrebbe anche essere interessante studiare le variazioni sistematiche tra i segnali Raman e i risultati della spettrometria di massa (così come altri metodi omici) utilizzando approcci computazionali integrativi. È interessante notare che abbiamo già trovato diverse correlazioni lineari deboli ma significative tra le intensità dei picchi Raman identificati dai punteggi VIP e l'abbondanza di tamoxifene o il suo metabolita a livello di singola cellula identificato da MS4. Questi dati possono suggerire una relazione metabolica tra i profili MS e gli spettri Raman e la possibilità di prevedere questi valori.
Divulgazioni
Gli autori non dichiarano conflitti di interesse.
Riconoscimenti
Gli autori ringraziano Toshio Yanagida per il suo sostegno e i fondi collaborativi interni RIKEN attribuiti al Dr. Arno Germond.
Materiali
| Name | Company | Catalog Number | Comments |
| 0.1% penicillin-streptomycin | Nacalai Tesque | 09367-34 | |
| 35mm glass bottom grid dish | Matsunami | ||
| 4-Hydroxy Tamoxifen standard | Sigma-Aldrich | 94873 | |
| 532 nm diode pumped solid-state laser | Ventus, Laser Quantum | ||
| BIOS-L101T-S motorized microscope stage | OptoSigma | ||
| CT-2 cellomics coated sampling capillaries | HUMANIX | ||
| d5-Tamoxifen standard | Cambridge Isotope Laboratories | ||
| Dimethyl sulfoxide LC-MS grade | Nacalai Tesque | D8418 | |
| Dulbecco's Modified Eagle's medium | Sigma-Aldrich | D5796 | |
| Eppendorf GELoader tips | Eppendorf | ||
| fetal bovine serum | Hyclone laboratories | SH3006603 | |
| FluoroBrite DMEM | Thermo Fisher Scientific | ||
| Formic acid LC-MS grade | Sigma-Aldrich | 33015 | |
| HepG2 cell line (RCB1886) | RIKEN cell bank center | RCB1886 | |
| MC0-19A1C Incubator | Sanyo Electric Co. | MC0-19A1C | |
| Methanol LC-MS grade | Sigma-Aldrich | 1060352500 | |
| MMO-203 3-D Micromanipulator | Narshige | MMO-203 | |
| NA:0.95, UPL40 water-immersion Olympus objective lens | Olympus | ||
| Nanoflex nano-ESI adaptor | Thermo Fisher Scientific | ES071 | |
| On-stage incubator | ibidi | ||
| Pierce LTQ Velos ESI calibration solution | Thermo Fisher Scientific | 88323 | |
| PIXIS BR400 cooled CCD camera | Princeton Instruments | ||
| Q-Exactive Orbitrap | Thermo Fisher Scientific | ||
| Rat-tail collagen coating solution | Cell Applications Inc. | ||
| Tamoxifen standard | Sigma-Aldrich | 85256 |
Riferimenti
- Altschuler, S. J., et al. Cellular heterogeneity: do differences make a difference?. Cell. 141 (4), 559-563 (2010).
- Ali, A., et al. Single-cell metabolomics by mass spectrometry: Advances, challenges, and future applications. TrAC Trends in Analytical Chemistry. , (2019).
- Bunnage, M., et al. Target validation using chemical probes. Nature Chemical Biology. 9 (4), 195-199 (2013).
- Ali, A., et al. Single-Cell Screening of Tamoxifen Abundance and Effect Using Mass Spectrometry and Raman-Spectroscopy. Analytical Chemistry. 91 (4), 2710-2718 (2019).
- Wu, H., et al. In vivo lipidomics using single-cell Raman spectroscopy. Proceedings of the National Academy of Sciences of the United States of America. 108 (9), 3809-3814 (2011).
- Okada, M., et al. Label-free Raman observation of cytochrome c dynamics during apoptosis. Proceedings of the National Academy of Sciences of the United States of America. 109 (1), 28-32 (2012).
- Palonpon, A. F., et al. Raman and SERS microscopy for molecular imaging of live cells. Nature Protocols. 8 (4), 677-692 (2013).
- Butler, H. J., et al. Using Raman spectroscopy to characterize biological materials. Nature Protocols. 11 (4), 664-687 (2016).
- Mark, H., Workman, J. . Chemomtrics in Spectroscopy. , (2018).
- Lieber, C. A., Mahadevan-Jansen, A. Automated method for subtraction of fluorescence from biological Raman spectra. Applied Spectroscopy. 57, 1363-1367 (2003).
- Germond, A., et al. Raman spectral signature reflects transcriptomic features of antibiotic resistance in Escherichia coli. Communications Biology. 1, 85 (2018).
- Wold, S., et al. Partial Least Squares Projections to Latent Structures (PLS) in Chemistry. Encyclopedia of Computational Chemistry. , (2002).
- Chong, I. G., Jun, C. H. Performance of some variable selection methods when multicollinearity is present. Chemometrics and Intelligent Laboratory Systems. 78, (2005).
- Inde, Z., Dixon, S. J. The impact of non-genetic heterogeneity on cancer cell death. Critical Reviews in Biochemistry and Molecular Biology. 53 (1), 99-114 (2018).
- Pan, N., et al. The single-probe: a miniaturized multifunctional device for single cell mass spectrometry analysis. Analytical chemistry. 86 (19), 9376-9380 (2014).
- Rao, W., et al. Applications of the Single-probe: Mass Spectrometry Imaging and Single Cell Analysis under Ambient Conditions. Journal of Visualized Experiments. (112), e53911 (2016).
- Ali, A., et al. Quantitative Live Single-cell Mass Spectrometry with Spatial Evaluation by Three-Dimensional Holographic and Tomographic Laser Microscopy. Analytical Sciences: the International Journal of the Japan Society for Analytical Chemistry. 32 (2), 125-127 (2016).
- Abouleila, Y., et al. Live single cell mass spectrometry reveals cancer-specific metabolic profiles of circulating tumor cells. Cancer Science. 110, 697-706 (2018).
- Hiyama, E., et al. Direct Lipido-Metabolomics of Single Floating Cells for Analysis of Circulating Tumor Cells by Live Single-cell Mass Spectrometry. Analytical Sciences: the International Journal of the Japan Society for Analytical Chemistry. 31 (12), 1215-1217 (2015).
- Ryabchykov, O., et al. Fusion of MALDI Spectrometric Imaging and Raman Spectroscopic Data for the Analysis of Biological Samples. Frontiers in Chemistry. 6, 257 (2018).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon