Method Article
Caratterizzare l'efficienza di salto di esone nei campioni di pazienti DMD negli studi clinici sugli oligonucleotidi antisenso
* Questi autori hanno contribuito in egual misura
In questo articolo
Riepilogo
Qui, presentiamo la caratterizzazione molecolare dell'espressione distrofina 38 usando il sequenziamento Sanger, RT-PCR e il gonfiore occidentale nello studio clinico.
Abstract
La distrofia muscolare di Duchenne (DMD) è una malattia muscolare degenerativa che causa una progressiva perdita di massa muscolare, che porta alla morte prematura. Le mutazioni spesso causano un telaio di lettura distorto e codoni prematuri, con conseguente quasi totale mancanza di proteine della distrofina. Il telaio di lettura può essere corretto utilizzando oligonucleotidi antisenso (AON) che inducono il salto esone. Il morpholino AON viltolarsen (nome in codice: NS-065/NCNP-01) ha dimostrato di indurre exon 53 saltando, ripristinando il telaio di lettura per i pazienti con esone 52 delezioni. Recentemente abbiamo somministrato NS-065/NCNP-01 per via endovenosa a pazienti affetti da DMD in uno studio esplorativo iniziato da uno sperimentatore di NS-065/NCNP-01. In questo articolo di metodi, presentiamo la caratterizzazione molecolare dell'espressione della distrofina usando il sequenziamento di Sanger, RT-PCR e il gonfiore occidentale nello studio clinico. La caratterizzazione dell'espressione della distrofina è stata fondamentale nello studio per mostrare l'efficacia poiché non sono stati eseguiti test di esito funzionale.
Introduzione
La distrofia muscolare di Duchenne (DMD) è una malattia muscolare degenerativa che causa una progressiva perdita di massa muscolare, insufficienza respiratoria e cardiomiopatia, e porta alla morte prematura1. La malattia è causata dalla mancanza della grande proteina muscolare strutturale distrofina2. Le mutazioni nel gene DMD sul cromosoma X sono recessive e la malattia colpisce 1 su 3500-5000 maschiappena nati 3,4,5. Le mutazioni sono spesso grandi delezioni in una regione hotspot tra gli esoni 44 e 55 che portano ad un quadro di lettura distorto e a codoni di arresto prematuro, causando decadimento mediato da sciocchezze e una quasi totale mancanza di proteina distrofina6,7,8. Il telaio di lettura può essere corretto utilizzando oligonucleotidi antisenso (AON) che inducono l'esone saltando e ripristinando il telaio di lettura, ripristinando parzialmente l'espressione della distrofina e ritardando la progressione della malattia9,10,11. Il morpholino AON eteplirsen, che è stato recentemente approvato dalla Federal Drug Agency (FDA), induce saltando l'esone 51 e può ripristinare il telaio di lettura in pazienti con esone 52delezioni 12,13. Tuttavia, exon 53 saltando ripristina il telaio di lettura per i pazienti con esone 52 delezioni, e può potenzialmente trattare circa il 10% dei pazienti dmD14. Il farmaco morpholino AON NS-065/NCNP-01 ha dimostrato di indurre l'esone 53 saltando nelle cellule umane, e abbiamo recentemente somministrato NS-065/NCNP-01 a pazienti affetti da DMD in uno studio clinico di escalation della dose open-label di fase 1 (qui di seguito, indicato come "lo studio") (registrato come UMIN: 000010964 e ClinicalTrials.gov: NCT02081625)14. Lo studio ha mostrato un aumento dipendente dalla dose di esone 53 saltando sulla base di RT-PCR e livelli di proteine distrofina sulla base di gonfiore occidentale, e non sono stati osservati gravi eventi di farmaci avversio abbandoni 14.
In tutti gli studi clinici, l'analisi dei risultati è di fondamentale importanza. Per gli studi clinici DMD, è ancora in corso un dibattito sul metodo migliore per mostrare un beneficio terapeutico. I test clinici come il test di 6 minuti a piedi hanno alcuni inconvenienti. La caratterizzazione molecolare dell'espressione della distrofina può essere eseguita utilizzando diversi metodi, come RT-PCR, qPCR, digital-PCR, blotting occidentale e immunohistochimica. Tuttavia, l'entità del ripristino dell'espressione proteica necessaria per impartire un beneficio clinico rimane poco chiara. In questo articolo di metodi, descriviamo in dettaglio i metodi RT-PCR e western blotting utilizzati per determinare i livelli di salto e proteina esone, rispettivamente, nella fase 1 prova del farmaco AON saltando il farmaco NS-065/NCNP-0114.
Protocollo
La procedura operativa per la sperimentazione avviata dall'investigatore è stata approvata dal comitato etico NCNP (ID approvazione: A2013-019).
1. Preparazione dei campioni muscolari
NOTA: I muscoli dei bicipiti brachii o quadricipiti sono spesso selezionati come siti di biopsia. Tuttavia, il tibialis anteriore è stato utilizzato nello studio clinico.
-
Biopsia muscolare da pazienti
- Contrassegnare il sito di incisione prima della preparazione della pelle.
- Preparare una garza salina per il campione di biopsia. Spremere bene la garza.
- Iniettare l'anestesia locale nella pelle e nel tessuto sottocutaneo, ma non nel muscolo. Assicurarsi della profondità dell'anestesia dall'assenza di dolore cutaneo.
NOTA: L'anestesia generale è generalmente utilizzata in pazienti pediatrici sotto i 15 anni di età. - Fare un'incisione dalla pelle al tessuto sottocutaneo. Utilizzare piccoli retrattini per aprire l'incisione e separare il grasso sottocutaneo.
NOTA: In questa fase, una parte della pelle può essere tagliata per la coltura fibroblasta. - Fare un'altra piccola incisione nella fascia e tagliare ulteriormente la fascia. Bloccare i bordi utilizzando le forcepi di zanzara per esporre il muscolo.
- Utilizzare una sutura per tirare su la porzione selezionata del muscolo. Fare un piccolo tunnel utilizzando le forbici Iris e inserire le forcerse di zanzara nel tunnel.
- Separare il fascio muscolare con le force e tagliare entrambe le estremità della porzione bersaglio.
NOTA: I campioni di biopsia muscolare devono essere intorno alle dimensioni di una matita per i pazienti adulti (lunghezza 1,0-1,5 cm, diametro 0,8-1,0 cm). Il campione deve avere una lunghezza di circa 1 cm e un diametro di 5 mm nei pazienti pediatrici. - Avvolgere il campione in garza salata e trasportare immediatamente il muscolo fresco a temperatura ambiente (RT) in un impianto dove il campione può essere preparato per ulteriori analisi.
NOTA: I campioni devono essere trasportati a 4 gradi centigradi se la durata del trasporto supera 1 ora.
2. Preparazione del campione muscolare
- Mescolare volumi uguali di gomma e acqua tragacanth fino a quando la gomma diventa morbida e appiccicosa. Caricare il composto in siringhe da 25 mL. Le siringhe possono essere conservate in frigorifero.
- Posizionare circa 0,5 a 1 cm di gomma da tragacanth su dischi di sughero. Etichettare i dischi sul lato opposto.
- Mettere un contenitore di isopentane in azoto liquido fino a quando parte del liquido si congela.
- Collocare l'esemplare nella gomma tragacanth. L'asse longitudinale di ogni muscolo dovrebbe essere perpendicolare al sughero. Posizionare la gomma intorno al fondo di ogni muscolo per aiutare il corretto posizionamento.
- Utilizzando una pinzetta, posizionare il campione di muscolo/sughero in isopentane freddo preparato al punto 2.3 per congelare. Spostare il campione costantemente per 1 min o fino a quando completamente congelato, e posizionare temporaneamente sul ghiaccio secco.
- Collocare l'esemplare in fiale di vetro e conservate a -80 gradi centigradi.
3. Sezionazione muscolare
- Impostare il criostato per la sezionazione con una temperatura di lavoro di -25 gradi centigradi.
- Montare il blocco di sughero/muscolo e assetre fino a ottenere sezioni piatte.
- Utilizzare uno spessore di sezione di 10 m per RT-PCR e gonfiore occidentale. Utilizzare uno spessore di 6-8 m e 10-12 m per l'immunohistochimica o la colorazione ematossilina ed eosin, rispettivamente. Mettere le sezioni affettate in tubi da 2,0 mL e conservare a -80 gradi centigradi per RT-PCR e gonfiore occidentale.
4. Estrazione dell'RNA e reazione a catena di polimerasi di trascrizione inversa (RT-PCR)
- Estrarre circa 10 fette con uno spessore di 10 m dal muscolo congelato da un criostato. Raccogliere l'RNA totale purificato utilizzando il kit di purificazione dell'RNA totale secondo le istruzioni del produttore.
- Misurare la concentrazione di RNA utilizzando uno spettrofotometro.
- Combinare i reagenti necessari per una reazione RT-PCR nei tubi PCR secondo la tabella 1. Vedere la tabella 2 per le sequenze di primer.
NOTA: Il primer in avanti era 44F per il paziente NS-03 e 46F per il paziente NS-07. Il primer inverso era 54/55R come impostazione predefinita. Se i prodotti non specifici erano evidenti, 54R è stato utilizzato come alternativa. - Posizionare i tubi PCR contenenti la miscela in un termo-ciclo. Eseguire il termo-ciclo secondo la tabella 3.
- Conservare il prodotto PCR a 4 gradi centigradi per lo storage a breve termine o a -20 gradi centigradi per l'archiviazione a lungo termine.
5. Microchip elettroforesi ed esone saltando il calcolo
NOTA: Un sistema di elettroforesi a microchip (MCE) è spesso selezionato per analizzare l'efficienza del salto esodi. In questo protocollo, descriviamo i passaggi necessari per analizzare l'efficienza di salto esone utilizzando un sistema MCE dal produttore A e dal produttore B, in seguito chiamato sistema A e B. Durante lo studio clinico, l'efficienza di salto esone è stata analizzata sul sistema A. Tuttavia, il sistema A non è più in vendita e consigliamo al sistema B di analizzare l'efficienza di salto esodi. Sono stati inclusi protocolli per entrambi i sistemi (vedere la sezione 5.1 per il sistema A e 5.2 per il sistema B). Per informazioni sul sistema A e B, vedere Tabella dei materiali. Inoltre, questo passaggio può essere eseguito anche con il normale elettroforesi gel agarose, ma la sensibilità diminuisce notevolmente.
- Elettroforesi a microchip con sistema A
NOTA: il sistema A dispone di due tipi di chip. Qui, descriviamo i passaggi per il chip del DNA.- Reagenti kit equilibrate (buffer di stain, buffer di caricamento, ladder e buffer gel) a RT, vortice e spin verso il basso.
- Per preparare il buffer gel-stain (GS), aggiungere 12,5 L di buffer di macchia a 250 L di buffer gel e vortice per 10 s. Spostare la soluzione in un tubo di filtro di spin e centrifugare a 2.400 x g per 15 min. Eliminare il filtro.
NOTA: Il GS filtrato può essere conservato a 4 gradi centigradi per 1 mese. - Se i campioni sono altamente concentrati, diluire a circa 50 ng/L con tampone TE o acqua senza Dnase.
NOTA: Se i campioni sono in una concentrazione di sale : 200 mM KCl (o NaCl) e/o 15 mM MgCl2, scambiare il buffer. - Aggiungere 12 L di soluzione GS al pozzo di innesco del gel (evidenziato ed etichettato GS) del chip del DNA e posizionare il chip nella stazione di innesco.
NOTA: Per evitare bolle d'aria, inserire la punta della pipetta verticalmente e sul fondo del pozzo durante l'erogazione. Erogare lentamente fino alla prima fermata sulla pipetta e non espellere l'aria alla fine del passo di pipettatura. - Selezionare la modalità C3 e premere il pulsante Start. Al termine dell'innesco, rimuovere il chip.
- Ispezionare visivamente i microca canali alla ricerca di bolle d'aria intrappolate o innesco incompleto.
- Caricare i campioni preparati e la scala sul chip come di seguito.
- Pipette 9 di soluzione GS negli altri 3 pozzi GS.
- Pipette 5 -L del buffer di carico nel pozzo L e ogni pozzo campione (1-11).
- Pipette 1 L di scala nel pozzo L.
- Pipetta 1 L del campione di DNA in ciascuno degli 11 pozzi campione.
- Pipette 1 L di acqua senza TE o DNase in tutti i pozzi inutilizzati. La guida rapida del kit mostra una figura del layout del chip.
NOTA: Ispezionare tutti i pozzetti alla ricerca di bolle d'aria tenendo il chip sopra uno sfondo leggermente colorato. Spostare eventuali bolle d'aria intrappolate sul fondo di un pozzo con una punta di pipetta pulita o rimuovendo e ricaricando la soluzione.
- Posizionare il chip nella stazione del vortice e premere Mix. La stazione di vortice si arresta automaticamente dopo 1 min.
- Eseguire il chip nella stazione di elettroforesi entro 5 minuti dal caricamento.
- Selezionare Nuova esecuzione nella barra degli strumenti del software. Nella schermata Nuova esecuzione selezionare DNA e DNA 1K dall'elenco a discesa Assay.
- Selezionare una cartella di progetto per l'esecuzione dall'elenco a discesa Progetto oppure creare una nuova cartella di progetto immettendo un nome nel campo Progetto.
- Immettere un nome per l'esecuzione nel campo Prefisso esecuzione e fare clic su Avvia esecuzione.
- Lo strumento espe a causa del numero di esoffi al termine dell'analisi e si apre una finestra che indica la fine dell'esecuzione. Selezionare OK e rimuovere il chip dalla piattaforma chip.
- Selezionare File Esporta dati per esportare i dati. Selezionate le opzioni desiderate nella finestra di dialogo Esporta.
- Per pulire gli elettrodi, riempire un chip di pulizia con 800 L di acqua deionizzata e posizionarlo sulla piattaforma del chip, chiudere il coperchio e lasciarlo chiuso per 1 min. Dopo 1 min, aprire il coperchio, rimuovere il chip di pulizia e lasciare asciugare gli elettrodi per 1 min.
- Elettroforesi a microchip con sistema B
- Aprire il software operativo e immettere le informazioni di esempio. Dopo aver inserito le informazioni, il software calcola automaticamente la quantità di soluzioni buffer e marker di separazione.
- Preparare la quantità necessaria di buffer di separazione calcolato dal software. Preparare innanzitutto la soluzione di macchia di gel acido nucleico 100x diluendo la soluzione 10.000x con la quantità appropriata di buffer TE. La soluzione 100x può essere conservata a -20 gradi centigradi.
- Mescolare una quantità adeguata di 100x macchia di gel acido nucleico con tampone di sicurezza nel tubo specifico fornito dall'azienda per preparare una soluzione 1x. Vortice la soluzione.
- Preparare la quantità richiesta di soluzione marcatore nel tubo.
- Avviare il programma di lavaggio della sonda.
- Al termine, avviare il programma di lavaggio dei microchip.
- Contemporaneamente, pipetta i campioni a una multiplate PCR (96-pozzo, chiaro) e diluire 4 volte con acqua deionizzata. Il volume minimo che la macchina è in grado di gestire è di 6 L, mentre il massimo è di 30 L. Suggeriamo un volume totale di 10 L (2,5 L del campione e 7,5 L di acqua/TE-buffer).
- Coprire le piastre con fogli di carta di tenuta PCR adesivi e far girare i campioni.
- Impostare la piastra, la soluzione del marcatore e il buffer di separazione 1x nella macchina in base al posizionamento mostrato dalla macchina. Premere il pulsante Start.
- Al termine dell'esecuzione, esportare i dati dei risultati come file .csv dal software e calcolare l'efficienza di salto esone utilizzando la concentrazione molare come segue:
Efficienza di salto esone (%) - (Banda saltata / (Banda saltata - Banda non saltata) X 100
6. Sequenziamento del DNA complementare (cDNA)
-
Preparazione del gel Agarose
- Misurare 1,5 g di polvere di agarose e mescolare la polvere con 100 mL di tampone 1x TAE in una fiaschetta microwavable (con conseguente gel di agarose dell'1,5%).
- Microonde per 1-3 minuti fino a quando l'agarose è completamente sciolto.
NOTA: Non sovrabostare la soluzione, poiché parte del buffer evapora e altera la percentuale finale di agarose del gel. - Lasciare raffreddare la soluzione di agarose a circa 50 gradi centigradi.
- Aggiungere 10 coloranti per acido nucleico fluorescente da 10.000X alla soluzione di agarose.
- Versare l'agarose in un vassoio di gel con il pettine del pozzo in posizione. Lasciare a RT per 20-30 min fino a quando non si è completamente solidificato.
-
Sequenziamento
- Mescolare 5 L del prodotto RT-PCR (dal punto 4.4) e 1 L di buffer di carico 6x. Caricarli nei pozzetti del gel di agarose 1,5%.
- Eseguire il gel a 135 V per 5 min e 120 V per 20 min.
- Visualizzare le bande utilizzando un transilluminatore secondo il protocollo del produttore.
- Bande di accise di interesse dal gel e utilizzare un gel e kit di pulizia PCR per recuperare i prodotti RT-PCR.
- Misurare la concentrazione utilizzando uno spettrofotometro.
NOTA: La banda purificata può essere inviata per il sequenziamento presso un'azienda o un impianto di sequenziamento se non è disponibile alcuna apparecchiatura di sequenziamento Sanger in-house. - Preparare i reagenti necessari per un kit di sequenziamento del ciclo e la pipetta in una piastra PCR multiplata secondo la tabella 4. Vedere la tabella 2 per le sequenze di primer.
- Sigillare la piastra con pellicola adesivo trasparente.
- Vortice la piastra per 2 a 3 s. Centrifugare brevemente in una centrifuga oscillante in modo che il contenuto si stabilizzi sul fondo dei pozzi (5 a 10 s) a 1.000 x g.
NOTA: Le bolle d'aria possono essere presenti nei pozzi, ma non influiscono negativamente sulla reazione. - Mettere il composto in un termo-cycler ed eseguire secondo la tabella 5.
- Purificare le reazioni di sequenziamento con piastre secondo le istruzioni del produttore.
- Utilizzare un analizzatore di DNA per determinare le sequenze in base al protocollo del produttore.
- Confrontare la sequenza ottenuta con la sequenza prevista del paziente.
7. Gonfiore occidentale
- Preparazione del campione
- Preparare il buffer campione SDS (4% SDS, 4 M urea, 10% 2-mercaptoethanol, 10% glicerolo, 70 mM Tris-HCl pH 6.4, 0.001% bromophenol blu e inibitore della proteasi).
- Collocare circa 100 fette delle 10 sezioni muscolari di 10 m raccolte dal crio-sezione in 150 L di buffer SDS.
- Omogeneizzare brevemente i campioni di proteine sul ghiaccio per 30 s utilizzando un pratico micro omogeneizzatore.
- Centrifugare a 16.500 x g per 15 min e trasferire il supernante in un tubo fresco. Procedere con l'analisi o conservare a -80 gradi centigradi.
- SDS-PAGE per la misurazione della concentrazione proteica
NOTA: Per determinare la concentrazione proteica è stata utilizzata una macchia di gel fluorescente.- Determinare il normale peso del campione di controllo sano e la concentrazione utilizzando il kit di analisi della proteina BCA secondo le istruzioni del produttore.
- Preparare i campioni per l'elettroforesi gel. Pipette 10 g di proteine totali del controllo sano e 5 L dei campioni del paziente, 5 L di buffer campione 4x, 2 L di 10x agente di riduzione del campione. Una volta che questi sono stati mescolati, aggiungere l'acqua deionizzata ad un volume finale di 20 L in ogni campione.
- Campioni di calore a 70 gradi centigradi per 10 min.
- Preparare 2.000 mL di buffer in esecuzione 1x SDS utilizzando 50 mL di buffer in esecuzione 40x SDS. Diluire con 1.950 mL di acqua deionizzata.
NOTA: a questo punto, 1.000 mL di buffer in esecuzione è sufficiente. Il buffer rimanente di 1.000 mL viene utilizzato al punto 7.3.1. - Mescolare accuratamente e mettere da parte 800 mL del buffer 1x SDS per l'uso nella camera tampone inferiore (esterna) della scatola gel.
- Immediatamente prima dell'elettroforesi, aggiungere 500 L di buffer antiossidante a 200 mL di 1x SDS buffer in esecuzione da utilizzare nella camera tampone superiore (interna) della scatola gel.
- Preparare l'elettroforesi gel impostando un gel Tris-acetato del 3-8% secondo il protocollo del produttore. Caricare 20 L di ogni campione sul gel.
- Eseguire l'elettroforesi a 150 V per 75 min.
- Dopo l'elettroforesi, posizionare il gel direttamente in un vassoio pulito contenente 50 mL di soluzione di macchia di gel fluorescente.
- Coprire il vassoio, posizionare su uno shaker e agitare senza spruzzare liquido o danneggiare il gel per 90 min.
- Trasferire il gel in acqua prima dell'imaging in un sistema di fluorescenza a 312 nm di illuminazione con filtro di emissione di bromuro di etidio per rilevare la fluorescenza.
- Misurare le concentrazioni dei campioni di pazienti sulla base di una curva analitica costruita utilizzando il normale llysate di controllo con concentrazione nota.
- SDS-PAGE per il gonfiore occidentale
- Preparare la scatola gel secondo i punti 7.2.4-7.2.6.
- Sulla base della misura di concentrazione ottenuta al punto 7.2.11, caricare 100 g per corsia di lizia di controllo sano e 300 g per corsia del campione del paziente. Elettroforese utilizzando le stesse impostazioni del punto 7.2.7.
- Trasferimento di proteine
- Preparare il buffer di trasferimento. Immergere una membrana PVDF per 20 s in metanolo. Spostarlo nel buffer B fino all'uso.
- Immergere una carta gonfiante extra-spessa nel buffer A, B e C per almeno 30 min per buffer.
- Dopo l'elettroforesi, immergere il gel nel tampone B per 5 min.
- Posizionare le carte gonfiabili, la membrana PVDF e il gel sulla macchina di trasferimento semi-asciutta secondo il disegno della Figura 1. Stendere bolle d'aria tra il gel e la membrana con un rullo.
- Trasferimento a 2 mA/cm2 membrana per 1 h a RT.
NOTA: Dopo il trasferimento, si consiglia di eseguire una macchia proteica totale simile alla colorazione Ponceau per confermare il trasferimento riuscito delle grandi proteine di peso molecolare.
- Blocco e colorazione anticorpo
- Risciacquare la membrana due volte con PBST (1x PBS con 0,1% Tween 20).
- Posizionare la membrana in agente di blocco 1% e roccia delicatamente per 1 h a RT per bloccare.
- Incubare la membrana con anticorpo anti-distrofina in soluzione di blocco (1:125) e anticorpo anti-spectrin (1:25,000) per 1 h a RT o durante la notte a 4 gradi centigradi.
- Lavare la membrana tre volte per 10 min ciascuno con PBST a RT.
- Incubare la membrana con perossidasi di rafano (HRP)-coniugato anticorpo secondario anti-topo/coniglio in PBST (1:100) per 40 min a RT.
- Lavare di nuovo la membrana tre volte per 10 min con PBST a RT.
- Rilevamento
- Soluzioni di rilevamento mix A e B in un rapporto di 1:1. Il volume finale del reagente di rilevamento richiesto è di 0,1 mL/cm2 membrana.
- Rimuovere il PBST in eccesso dalla membrana lavata e posizionarlo con il lato proteico su un foglio di involucro di plastica o altre superfici pulite adatte. Aggiungere il reagente di rilevamento misto sulla membrana e incubare per 5 minuti a RT.
- Rimuovere il reagente di rilevamento in eccesso tenendo la membrana delicatamente in fopando e toccando il bordo contro un tessuto.
- Posizionare il lato della proteina macchia su un vassoio campione. Utilizzare il sistema fluorescente in base alla documentazione dell'utente. Impostare la macchina come segue. Tipo di esposizione: Incremento, Tempo intervallo: 10 s, Sensibilità/Risoluzione: alta.
NOTA: Le aree misurate sono state boxate con un rettangolo blu (Figura 4a-b): BG: sfondo; D: distrofia 427 kDa, SL: spectrin beta long isoform (274 kDa); SS: breve isoforme (253 kDa). Il rapporto segnale distrofina/spectrin è stato calcolato come (D-BG)/[(SL-BG) - (SS-BG)]. Il rapporto tra il controllo normale è stato impostato al 100%.
Risultati
Per utilizzare RT-PCR per rilevare il salto exon, i primer su entrambi i lati dell'esone che verrà saltato sono stati progettati e valutati per produrre solo bande specifiche (vedere la Figura 2 per un diagramma schematico di salto e posizione primer). I primer dovrebbero generare prodotti che possono essere facilmente distinti per dimensioni su un sistema MCE o elettroforesi gel agarose normale se MCE non è disponibile. Figura 3a mostraun sistema MCE Immagini gel di reazioni RT-PCR e i risultati del sequenziamento della banda saltata dai pazienti NS-03 e NS-07 prima e dopo il trattamento con NS-065/NCNP-01 nello studio di fase 1 di dose-escalation. NS-03 e NS-07 porto eliminazioni che si estendono exons 45-52 e 48-52, rispettivamente. NS-03 ha ricevuto 5 mg/kg e NS-07 20 mg/kg di NS-065/NCNP-01 settimanale per 12 settimane. Come previsto prima del trattamento, entrambi i pazienti non hanno mostrato alcun salto, e solo una banda non saltata potrebbe essere visualizzata. Dopo 12 settimane di trattamento, una banda inferiore saltata è stata vista per NS-07. Tuttavia, era ancora difficile rilevare qualsiasi prodotto saltato per il paziente NS-03. I risultati del sequenziamento hanno mostrato una concatenazione di esoni 47 e 54 per NS-07, così come exons 44 e 54 per NS-03. Secondo la sequenza, questi potrebbero teoricamente produrre un isoforme distrofina funzionale ma accorciato. Per calcolare la percentuale di salto mostrata nella figura 3b, la concentrazione molare per la banda saltata è stata divisa per la banda saltata e non saltata. Per il paziente NS-07, la percentuale dopo il trattamento era del 72%, ed era del 3,4% per NS-03. I dati di western blot (in triplicate) dei pazienti NS-03, NS-07 e un controllo sano sono illustrati nella Figura 4a. Previsto, non è stata rilevata alcuna banda distrofina prima del trattamento. Dopo il trattamento è stata rilevata una banda del paziente NS-07 con un peso molecolare inferiore rispetto al controllo sano (la distrofina di tipo selvaggio ha un peso molecolare di 427 kDa e la distrofina NS-07 ha un peso molecolare di 389 kDa). A causa della cancellazione esone e saltare, l'isoforme distrofina dal paziente NS-07 mancava di diversi esoni, e ci si aspettava di avere un peso molecolare inferiore. Il paziente NS-03 non ha mostrato livelli rilevabili di distrofina dopo il trattamento. Nella Figura 4b, viene mostrata la quantità di distrofina rispetto a un controllo sano per il paziente NS-07. La posizione in cui sono state misurate le intensità del segnale per il calcolo del ripristino della distrofina è indicata con quadrati blu. Il rapporto di spettro della distrofina è stato utilizzato per calcolare la quantità di distrofina rispetto a quella del controllo sano. A questo scopo, il segnale dalla distrofina meno sfondo è stato diviso per la somma dei due isoformi spectrin (lungo e corto) meno sfondo (vedi passo 8.6.5). Il rapporto per il controllo sano è stato impostato al 100%. La quantità di distrofina rispetto al controllo sano nel paziente NS-07 dopo il trattamento era del 9,1%. Tuttavia, la percentuale non poteva essere calcolata per il paziente NS-03 a causa di una banda distrofina debole, che simile a quelle ottenute per i campioni di trattamento precedenti per entrambi i pazienti.

Figura 1: diagramma schematico dello stack di trasferimento per l'analisi delle macchie occidentali. Assemblaggio della pila di trasferimento della macchia occidentale con carta assorbente imbevuta di tampone A nella parte inferiore seguita da carta assorbente imbevuta di tampone B, membrana PVDF, il 3-8% Tris-Acetate Gel e sulla parte superiore 2 carte blotting imbevute di tampone blotting C è mostrato. Si prega di fare clic qui per visualizzare una versione più grande di questa figura.
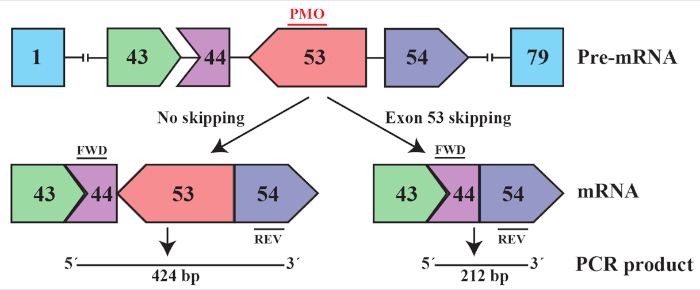
Figura 2: Disegno schematico di exon saltando l'esone 53 in un paziente con esone 45-52 delezione nel gene DMD. Per esempio, paziente NS-03 nello studio clinico dose-escalation ha avuto questa delezione. Se l'esone 53 viene mantenuto, l'mRNA sarà fuori dal telaio poiché la giunzione exon-exon tra exon 44 e 53 interrompe il telaio di lettura, causando un codon stop in exon 53. Il telaio di lettura viene ripristinato quando l'esone 53 viene saltato e viene prodotta un isoforma più corta di distrofina. Per rilevare l'esone 53-skipping da RT-PCR, vengono utilizzati primer in exon 44 e 54 in modo che il prodotto PCR tra mRNA saltato e non saltato può essere facilmente rilevato. FWD: primer in avanti, REV: primer inverso, PMO: fosforodiamidate morpholino oligomer. Si prega di fare clic qui per visualizzare una versione più grande di questa figura.
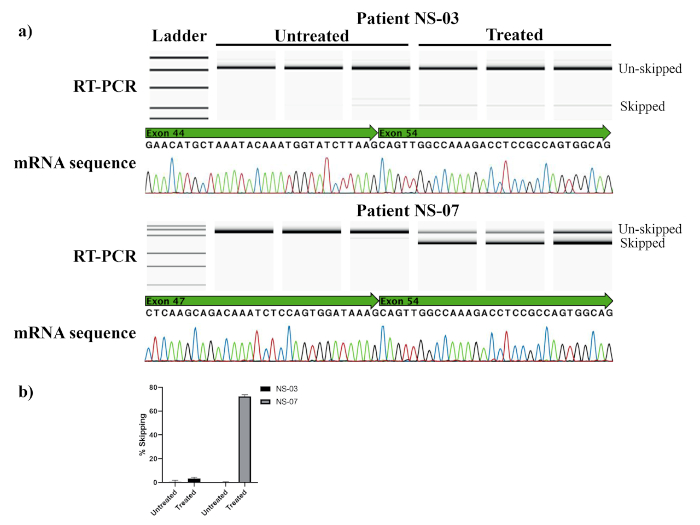
Figura 3: Saltare l'efficienza per i pazienti NS-03 e NS-07 da campioni di biopsia muscolare anteriore tibialis. a) Immagine Gel generata dal sistema di elettroforesi A di campioni RT-PCR di campioni non trattati e trattati da pazienti NS-03 e NS-07. Il pannello superiore mostra NS-03 e il pannello inferiore NS-07 in triplicete. Per NS-03, la banda non saltata è 422 bp e la banda saltata è 212 bp. Per NS-07, le bande sono rispettivamente 836 bp e 624 bp. L'analisi della sequenza ha mostrato una concatenazione di exon 44 ed exon 54 per NS-03 ed exon 47 a exon 54 per NS-07. b) L'efficienza di salto prima e dopo il trattamento è indicata per i pazienti NS-03 e NS-07, calcolata in base alla concentrazione molare delle due bande fornite dal sistema A come banda saltata/(banda saltata e banda non saltata) x 100. L'efficienza di salto per NS-03 era molto bassa dopo il trattamento; per NS-07, l'efficienza di salto è stata di oltre il 70%. Si prega di fare clic qui per visualizzare una versione più grande di questa figura.
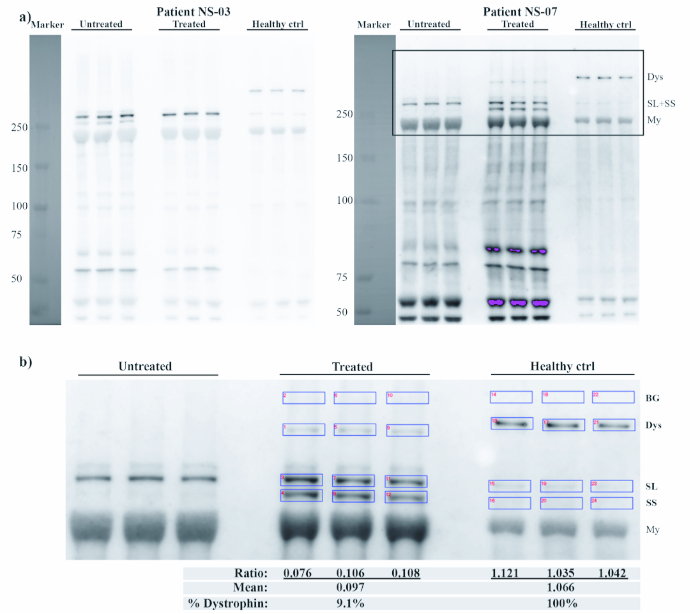
Figura 4: Quantificazione delle proteine delle isoformi della distrofina prima e dopo la terapia di salto eso. a) Quantificazione proteica delle isoformi della distrofina prima e dopo la terapia di salto dell'esone. Macchia occidentale di biopsia muscolare da tibialis anteriore da pazienti NS-03 e NS-07 prima e dopo il trattamento e dal controllo sano in triplicato. Dystrophin può essere visto a 427 kDa per il controllo sano e a 389 kDa per NS-07 dopo il trattamento. Nessun distrofina potrebbe essere rilevata per entrambi i pazienti prima del trattamento, o nessuno dei due potrebbe essere rilevato dopo il trattamento per il paziente NS-03. L'area nella casella nera è illustrata nella figura 4b. A sinistra in ogni macchia viene mostrato il marcatore. b) La quantità di distrofina ripristinata dopo il trattamento per il paziente NS-07. Il restauro della distrofina è stato calcolato in base all'intensità delle bande per la distrofina, lo sfondo e l'isoforma lunga e corta di spectrin secondo la formula: (distrofina - sfondo)/(spectrin L -background) - (spectrin S - background) dove il rapporto per il controllo sano è impostato al 100%. L'intensità di ogni banda è stata misurata all'interno delle caselle blu mostrate nella figura. Il ripristino della distrofina per NS-07 è stato del 9,1% dopo il trattamento. Il segnale della distrofina era troppo debole per essere misurata nei campioni non trattati. Le dimensioni dei marcatori sono mostrate in kilodaltons. Dismi: Distrofina, BG: sfondo, SL: Spectrin isoform lungo, SS: Spectrin breve isoform, Mio: Myosin tipo 1. Le dimensioni dei marcatori sono mostrate in kilodaltons. Si prega di fare clic qui per visualizzare una versione più grande di questa figura.
| Soluzione | Volume/Reazione (l) | Concentrazione finale |
| Acqua senza RNase (fornita) | Variabile | - |
| 5x Buffer QIAGEN OneStep RT-PCR | 5.0 | 1x |
| dNTP Mix (contenente 10 mM per ogni dNTP) | 1.0 | 400 M di ogni dNTP |
| Primer in avanti (10 M) | 1.5 | 0,6 M |
| Primer inverso (10 M) | 1.5 | 0,6 M |
| QIAGEN OneStep RT-PCR Enzyme Mix | 1.0 | - |
| Inibitore RNase (opzionale) | Variabile | 5-10 unità/reazione |
| Modello RNA | 50 ng | |
| Totale | 25 |
Tabella 1: reagenti RT-PCR. I composti necessari per una reazione del RT-PCR.
| Primer | Sequenza |
| 44F | 5'-CCTGAGAATTGGGAACATGC-3' |
| 46F | 5'-AACCTGGAAAAGAGCAGCAA-3' |
| 48F | 5'-CCAAGAAGGACCATTTGACG'3' |
| 54/55R | 5'-TCTCCCTCACTCACCCTTTT-3' |
| 54R | 5'-GTGGACTTTTTTTTTATCAT-3' |
Tabella 2: Elenco primer. Sequenze per i primer utilizzati in questo studio. F: Avanti, R: Inverti.
| 1 ciclo | Trascrizione inversa | 30 min | 50 gradi centigradi |
| 1 ciclo | Fase di attivazione PCR iniziale | 15 min | 95 gradi centigradi |
| 35 cicli | Denaturazione | 1 min | 94 gradi centigradi |
| Ricottura | 1 min | 60 gradi centigradi | |
| Estensione | 1 min | 72 gradi centigradi | |
| 1 ciclo | Estensione finale | 7 min | 72 gradi centigradi |
| Tenere | ∞ | 4 gradi centigradi |
Tabella 3: PCR condizionato. Mostra le condizioni PCR per la reazione RT-PCR.
| Soluzione | Volume/Reazione (l) |
| Acqua senza RNase | Variabile |
| BigDye Terminator 3.1 Pronto Reazione Mix | 3.5 |
| Primer in avanti/invertitore (3,2 M) | 2.0 |
| Modello RNA | 20 ng |
| Totale | 20 |
Tabella 4: Reagenti necessari per la reazione di sequenziamento. Utilizzare Forward o Reverse primer nella configurazione, non entrambi contemporaneamente.
| 1 ciclo | 1 min | 96 gradi centigradi |
| 25 cicli | 10 s | 96 gradi centigradi |
| 5 s | 50 gradi centigradi | |
| 4 min | 60 gradi centigradi | |
| Tenere | ∞ | 4 gradi centigradi |
Tabella 5: condizioni PCR per il sequenziamento Diger.
Discussione
Le sperimentazioni cliniche di DMD hanno prodotto successi e fallimenti negli ultimi anni. Sia RT-PCR che western blotting sono tecniche comuni per valutare l'efficienza di salto generata dai composti che saltano l'esodi somministrati ai pazienti. Tuttavia, RT-PCR è stato segnalato per sopravvalutare l'efficienza saltando rispetto al digitale PCR15. Anche se questo è dovuto a una serie di motivi, è principalmente causato dall'amplificazione più efficiente dei frammenti saltati più piccoli durante i cicli PCR. Sembra che la RT-PCR utilizzata in questo studio clinico ha generato anche efficienze di salto più elevate rispetto all'espressione proteica stimata dal gonfiore occidentale. Secondo la FDA, questo è un modo più affidabile per quantificare il ripristino della distrofina12. Pertanto, occorre prestare attenzione nell'interpretare i risultati di salto RT-PCR; tuttavia i campioni possono ancora essere confrontati. I campioni che mostrano efficienze di salto più elevate basate sui risultati di RT-PCR comunemente esano livelli di espressione proteica più elevati nelle analisi delle macchie occidentali.
Poiché tutti i pazienti negli studi clinici DMD non hanno lo stesso modello di deleghe, può essere difficile progettare primer e sonde che siano adeguatamente specifiche per eseguire operazioni digitali o qPCR su tutti i campioni. Quindi, RT-PCR è ancora una buona alternativa per una prima valutazione dell'efficienza di salto. Prima dell'inizio della sperimentazione clinica di NS-065/NCNP-01, era noioso valutare l'efficienza di salto per ogni paziente in vitro poiché una biopsia muscolare o cutanea era obbligatoria per generare mioblasti specifici per il paziente. Tuttavia, abbiamo recentemente pubblicato una nuova tecnica per generare cellule specifiche di MYOD1(UDC) convertite in origine dal paziente come un nuovo modello di cellule muscolari DMD16. Pertanto, solo l'urina raccolta dal paziente è necessaria per generare i mioblasti, e non è necessaria alcuna procedura invasiva. Crediamo che questo metodo possa essere utilizzato per lo screening di diversi AON in cellule specifiche del paziente. Inoltre, diversi primer e sonde possono essere testati prima che il paziente inizi qualsiasi studio clinico. Questo può facilitare l'uso di qPCR o PCR digitale per eso ignorare la misurazione negli studi clinici DMD in futuro.
Per l'esecuzione dell'analisi delle macchie occidentali in questo studio clinico, è stato utilizzato un singolo anticorpo contro la distrofina, e solo un controllo sano è stato utilizzato come campione di riferimento. Pertanto, la specificità dell'anticorpo non è stata convalidata in modo appropriato. Questo, insieme al fatto che l'anticorpo riconosce solo il dominio C-terminale della distrofina, è una limitazione del protocollo. In futuro sono consigliati controlli e anticorpi più sani diretti contro diversi domini della molecola distrofina.
Qui, abbiamo riassunto i protocolli che sono stati utilizzati nel recente processo esplorativo iniziato da investigatori, primo in-umano di NS-065/NCNP-01. NS-065/NCNP-01 è potenzialmente applicabile al 10,1% dei pazienti con DMD.
Divulgazioni
Nessuno.
Riconoscimenti
Siamo grati al dottor Takashi Saito, al dottor Tetsuya Nagata, al dottor Satoshi Masuda e al dottor Eri Takeshita per la discussione scientifica.
Materiali
| Name | Company | Catalog Number | Comments |
| 100 bp DNA Ladder | Takara | 3407A | marker solution for MultiNA |
| 1mol/l-Tris-HCl Buffer Solution(pH 7.6) | Nacalai | 35436-01 | |
| 2-mercaptoethanol | Nacalai | 21418-42 | |
| 2-Methylbutane (Isopentane) | Sigma-Aldrich | 15-2220 | |
| 5 mL Round Bottom Polystyrene Test Tube | Falcon | 352235 | |
| 6× Loading Buffer | Takara | 9156 | |
| Agarose ME | Iwai chemical | 50013R | |
| Applied Biosystems 3730 DNA Analyzer | Applied Biosystems | A41046 | |
| BD Matrigel Matrix | BD Biosciences | 356234 | |
| BigDye Terminator v3.1 Cycle Sequencing Kit | Applied Biosystems | 4337455 | |
| Bovine Serum Albumin solution | Sigma-Aldrich | A8412 | |
| Bromophenol blue | Nacalai | 05808-61 | |
| Centri-Sep 96-Well Plates | Applied Biosystems | 4367819 | |
| cOmplete Protease Inhibitor Cocktail | Roche | 4693116001 | |
| DMEM/F-12 | Gibco | 11320033 | |
| ECL Prime Blocking Reagent | GE healthcare | RPN418 | |
| ECL Prime Western Blotting Detection Reagent | GE healthcare | RPN2232 | |
| Endo-Porter | GeneTools | 2922498000 | |
| Experion Automated Electrophoresis Station | Bio-Rad | 7007010 | Microchip electrophoresis system A |
| Experion DNA 1K Analysis Kit for 10 Chips | Bio-Rad | 7007107JA | |
| Experion Priming Station | Bio-Rad | 7007030 | |
| Experion Vortex Station II | Bio-Rad | 7007043 | |
| Extra Thick Blot Filter Paper | Bio-Rad | 1703965 | |
| EzBlot | Atto | AE-1460 | |
| GelRed Nucleic Acid Gel Stain | Biotium | 41003 | |
| glass vial | Iwaki | 1880 SV20 | |
| Glycerol | Nacalai | 17045-65 | |
| Histofine Simple Stain MAX PO | Nichirei | 424151 | |
| Horse Serum | Gibco | 16050122 | |
| Immobilon-P Transfer Membrane | Millipore | IPVH304F0 | |
| ITS Liquid Media Supplement | Sigma-Aldrich | I3146 | |
| Methanol | Sigma-Aldrich | 34860 | |
| MicroAmp Clear Adhesive Film | Applied Biosystems | 4306311 | |
| MultiNA | SHIMADZU | MCE-202 | Microchip electrophoresis system B |
| Multiplate 96-Well PCR Plates | Bio-Rad | mlp9601 | |
| NanoDrop One Microvolume UV-Vis Spectrophotometer with Wi-Fi | Thermo Scientific | ND-ONE-W | |
| NovocastraTM Lyophilized Mouse Monoclonal Antibody Dystrophin (C-terminus) | Leica biosystems | NCL-DYS2 | |
| NovocastraTM Lyophilized Mouse Monoclonal Antibody Spectrin | Leica biosystems | NCL-SPEC1 | |
| NuPAGE 3-8% Tris-Acetate Protein Gels | Invitrogen | EA03785BOX | |
| NuPAGE Antioxidant | Invitrogen | NP0005 | |
| NuPAGE LDS Sample Buffer | Invitrogen | NP0007 | |
| NuPAGE Sample Reducing Agent | Invitrogen | NP0009 | |
| NuPAGE Tris-Acetate SDS Running Buffer | Invitrogen | LA0041 | |
| Oriole Fluorescent Gel Stain | Bio-Rad | 1610496 | |
| PBS | Gibco | 10010023 | |
| Penicillin-Streptomycin Solution Stabilized | Sigma-Aldrich | P4458 | |
| Physcotron Handy micro-homogenizer | MICROTEC | NS-310E3 | |
| Pierce BCA Protein Assay Kit | Thermo Scientific | 23227 | |
| Polybrene Infection / Transfection Reagent | Sigma-Aldrich | TR-1003 | |
| Propidium Iodide Staining Solution | BD Pharmingen | 556463 | |
| QIAGEN OneStep RT-PCR Kit | Qiagen | 210212 | |
| RNeasy Fibrous Tissue Mini Kit | Qiagen | 74704 | |
| SDS | Nacalai | 31606-75 | |
| Semi-dry transfer machine | Bio Craft | 41-1293 | |
| SYBR Gold Nucleic Acid Gel Stain | Invitrogen | S11494 | for MultiNA |
| TAE Buffer | Nacalai | 35430-61 | |
| Tragacanth Gum | Wako | 200-02245 | |
| Tris-EDTA Buffer | Nippon Gene | 316-90025 | for MultiNA |
| Trypsin-EDTA (0.05%) | Gibco | 25300054 | |
| TWEEN 20 | Sigma-Aldrich | P9416 | |
| Urea | Nacalai | 35907-15 | |
| Wizard SV Gel and PCR Clean-Up System | Promega | A9281 | |
| XCell SureLock Mini-Cell | Invitrogen | EI0001 |
Riferimenti
- Bushby, K., et al. Diagnosis and management of Duchenne muscular dystrophy, part 2: implementation of multidisciplinary care. Lancet Neurology. 9 (2), 177-189 (2010).
- Hoffman, E. P., Brown, R. H. Jr, Kunkel, L. M. Dystrophin: the protein product of the Duchenne muscular dystrophy locus. Cell. 51 (6), 919-928 (1987).
- Mendell, J. R., et al. Evidence-based path to newborn screening for Duchenne muscular dystrophy. Annals of Neurology. 71 (3), 304-313 (2012).
- Moat, S. J., Bradley, D. M., Salmon, R., Clarke, A., Hartley, L. Newborn bloodspot screening for Duchenne muscular dystrophy: 21 years experience in Wales (UK). European Journal of Human Genetics. 21 (10), 1049-1053 (2013).
- Greenberg, C. R., et al. Gene studies in newborn males with Duchenne muscular dystrophy detected by neonatal screening. Lancet. 2 (8608), 425-427 (1988).
- Forrest, S. M., et al. Further studies of gene deletions that cause Duchenne and Becker muscular dystrophies. Genomics. 2 (2), 109-114 (1988).
- Monaco, A. P., Bertelson, C. J., Liechti-Gallati, S., Moser, H., Kunkel, L. M. An explanation for the phenotypic differences between patients bearing partial deletions of the DMD locus. Genomics. 2 (1), 90-95 (1988).
- Flanigan, K. M., et al. Mutational spectrum of DMD mutations in dystrophinopathy patients: application of modern diagnostic techniques to a large cohort. Human Mutation. 30 (12), 1657-1666 (2009).
- Aoki, Y., et al. In-frame dystrophin following exon 51-skipping improves muscle pathology and function in the exon 52-deficient mdx mouse. Molecular Therapy. 18 (11), 1995-2005 (2010).
- Lu, Q. L., et al. Functional amounts of dystrophin produced by skipping the mutated exon in the mdx dystrophic mouse. Nature Medicine. 9 (8), 1009-1014 (2003).
- Yokota, T., et al. Efficacy of systemic morpholino exon-skipping in Duchenne dystrophy dogs. Annals of Neurology. 65 (6), 667-676 (2009).
- Kesselheim, A. S., Avorn, J. Approving a Problematic Muscular Dystrophy Drug: Implications for FDA Policy. Journal of the American Medical Association. 316 (22), 2357-2358 (2016).
- Mendell, J. R., et al. Eteplirsen for the treatment of Duchenne muscular dystrophy. Annals of Neurology. 74 (5), 637-647 (2013).
- Komaki, H., et al. Systemic administration of the antisense oligonucleotide NS-065/NCNP-01 for skipping of exon 53 in patients with Duchenne muscular dystrophy. Science Translational Medicine. 10 (437), (2018).
- Verheul, R. C., van Deutekom, J. C., Datson, N. A. Digital Droplet PCR for the Absolute Quantification of Exon Skipping Induced by Antisense Oligonucleotides in (Pre-)Clinical Development for Duchenne Muscular Dystrophy. PLoS One. 11 (9), e0162467(2016).
- Takizawa, H., et al. Modelling Duchenne muscular dystrophy in MYOD1-converted urine-derived cells treated with 3-deazaneplanocin A hydrochloride. Scientific Reports. 9 (1), 3807(2019).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon