Method Article
Un test di citometria a flusso basato su DNA / Ki67 per l'analisi del ciclo cellulare di cellule T CD8 antigene-specifiche in topi vaccinati
* Questi autori hanno contribuito in egual misura
In questo articolo
Erratum Notice
Riepilogo
L'espansione clonale è una caratteristica chiave della risposta delle cellule T antigene-specifiche. Tuttavia, il ciclo cellulare delle cellule T che rispondono all'antigene è stato scarsamente studiato, in parte a causa di limitazioni tecniche. Descriviamo un metodo citometrico a flusso per analizzare le cellule T CD8 antigene-specifiche in espansione clonale nella milza e nei linfonodi di topi vaccinati.
Abstract
Il ciclo cellulare delle cellule T antigene-specifiche in vivo è stato esaminato utilizzando alcuni metodi, ognuno dei quali presenta alcune limitazioni. La bromodeossiuridina (BrdU) contrassegna le cellule che sono in fase S o recentemente completate e l'estere carbossifluoresceina succinimidyl (CFSE) rileva le cellule figlie dopo la divisione. Tuttavia, questi coloranti non consentono l'identificazione della fase del ciclo cellulare al momento dell'analisi. Un approccio alternativo è quello di sfruttare Ki67, un marcatore che è altamente espresso dalle cellule in tutte le fasi del ciclo cellulare tranne la fase quiescente G0. Sfortunatamente, Ki67 non consente un'ulteriore differenziazione in quanto non separa le cellule in fase S che sono impegnate nella mitosi da quelle in G1 che possono rimanere in questa fase, procedere nel ciclo o passare a G0.
Qui, descriviamo un metodo citometrico a flusso per catturare una "istantanea" delle cellule T in diverse fasi del ciclo cellulare negli organi linfoidi secondari del topo. Il metodo combina Ki67 e la colorazione del DNA con la colorazione del complesso di istocompatibilità maggiore (MHC)-peptide-multimero e un'innovativa strategia di gating, permettendoci di distinguere con successo tra cellule T CD8 antigene-specifiche in G0, in G1 e nelle fasi S-G2/ M del ciclo cellulare nella milza e linfonodi drenanti dei topi dopo la vaccinazione con vettori virali portatori del gag dell'antigene modello del virus dell'immunodeficienza umana (HIV)-1.
Le fasi critiche del metodo sono state la scelta del colorante DNA e la strategia di gating per aumentare la sensibilità del test e per includere cellule T antigene-specifiche altamente attivate / proliferanti che sarebbero state perse dagli attuali criteri di analisi. Il colorante a DNA, Hoechst 33342, ci ha permesso di ottenere una discriminazione di alta qualità dei picchi di DNA G0/G1 e G2/M, preservando la colorazione di membrana e intracellulare. Il metodo ha un grande potenziale per aumentare le conoscenze sulla risposta delle cellule T in vivo e per migliorare l'analisi di immunomonitoraggio.
Introduzione
Le cellule T naïve subiscono espansione clonale e differenziazione dopo l'innesco dell'antigene. Le cellule T differenziate mostrano funzioni effettrici essenziali per la clearance dell'antigene e per il mantenimento della memoria antigene-specifica, che è fondamentale per una protezione duratura. Durante le prime fasi della risposta primaria, l'interazione naïve delle cellule T con cellule presentanti l'antigene (APC) all'interno di nicchie specializzate negli organi linfoidi è fondamentale per indurre l'enorme proliferazione delle cellule T che caratterizza la fase di espansione clonale1,2,3. L'interazione cellula T-APC è finemente regolata dalla concentrazione e dalla persistenza dell'antigene, dei segnali co-stimolatori e dei fattori solubili (citochine e chemochine) che influenzano la quantità e la qualità della progenie clonale delle cellule T4,5,6,7.
Nonostante gli intensi studi sull'espansione clonale delle cellule T, non è ancora noto se le cellule T innescate dall'antigene completino il loro intero ciclo cellulare nel sito di riconoscimento dell'antigene o se migrino verso altri organi durante la progressione del ciclo cellulare. Questa mancanza di conoscenza è dovuta alle proprietà degli strumenti disponibili per l'analisi del ciclo cellulare. Questi includono anticorpi monoclonali (mAbs) specifici per il marcatore nucleare, Ki67, e coloranti cellulari che identificano le cellule che hanno subito la fase S del ciclo cellulare (ad esempio, Bromodeoxyuridina (BrdU)) o discriminano tra le cellule figlie e i loro antenati (ad esempio, carbossifluoresceina succinimidyl estere (CFSE)).
Tuttavia, i coloranti per l'etichettatura cellulare, come CFSE e BrdU, non consentono di determinare se le cellule presenti in un particolare organo proliferassero localmente o piuttosto migrassero in questo sito dopo la divisione8,9. Inoltre, la proteina intranucleare, Ki67, è in grado di distinguere solo le cellule in G0 (cellule Ki67-negative) da quelle in qualsiasi altra fase del ciclo cellulare (cellule Ki67-positive). Pertanto, l'analisi Ki67 non distingue le cellule in proliferazione attiva(cioèin S, G 2 o M) da quelle in G1, che possono progredire rapidamente alla divisione o rimanere per lunghi periodi in G1 o tornare alla quiescenza10,11.
Qui, descriviamo un nuovo metodo citometrico a flusso per l'analisi del ciclo cellulare delle cellule T CD8 antigene-specifiche12 dalla milza e dai linfonodi (LNs) di topi vaccinati (Figura 1). Il metodo sfrutta una combinazione di Ki67 e colorazione del DNA che è stata precedentemente utilizzata per analizzare il ciclo cellulare delle cellule ematopoietiche del midollo osseo di topo (BM)13,14. Qui, abbiamo applicato con successo Ki67 plus DNA staining, insieme all'innovativa strategia di gating12recentemente pubblicata, all'analisi dell'espansione clonale delle cellule T CD8. Siamo stati in grado di discriminare chiaramente tra cellule T CD8 antigene-specifiche in G0,in G1e nelle fasi S-G2/M nella milza e nei LN drenanti dei topi vaccinati.
Protocollo
I topi sono stati ospitati presso la Plaisant Animal Facility e il lavoro è stato eseguito con il numero di autorizzazione del Ministero della Salute italiano 1065/2015-PR. Il protocollo ha seguito le linee guida per la cura degli animali secondo le leggi e le politiche nazionali e internazionali (Direttiva UE 2010/63/UE; D.Lgs. 26/2014).
1. Preparazione del mezzo e della soluzione colorante
- Preparare il mezzo completo: roswell Park Memorial Institute (RPMI) medio con 2 mM di glutammina, 100 U / mL di penicillina / streptomicina, 50 μM di beta-mercaptoetanolo e 10% di volume / volume (v / v) di siero bovino fetale (FBS)
- Preparare il tampone colorante: soluzione salina tamponata con fosfato senza Ca2+/ Mg2 + (PBS) con 1% peso / volume (p / v) albumina sierica bovina (BSA) e 2 mM di sale disodico dell'acido etilendidatetraacetico (EDTA)
2. Trattamento con topo
- Topi Balb/c femmina di 7-8 settimane mediante iniezione intramuscolare (i.m.) nei quadricipiti del virus dell'immunodeficienza umana (HIV)-1-gag-expressing-scimpanzé vettore adenovirale (ChAd3-gag) con una dose di 107 particelle virali.
- A 1-4 mesi dopo l'innesco, aumentare una volta i topi con i.m. iniezione di VIRUS Ankara vaccinia modificato che esprime HIV-1-gag (MVA-gag) con una dose di 106 unità di formazione della placca.
- Al giorno 3 post-boost, sacrifica i topi potenziati dalla lussazione cervicale e analizzali in parallelo con i topi non trattati.
- Raccogli i LNs drenando i quadricipiti (iliaci, poplitei e inguinali) e la milza da topi potenziati e non trattati. Inoltre, raccogliere il BM dalle due zampe posteriori da topi non trattati e utilizzare questo BM per le impostazioni del citometro a flusso e come controllo positivo per l'analisi del ciclo cellulare (Figura 2).
NOTA: genera vettori ChAd3-gag e MVA-gag come descritto in precedenza12,15,16,17.
3. Isolamento delle cellule LN, milza e BM drenanti
- Isolamento delle cellule della milza e LN
- Posizionare 5 mL di mezzo completo in ciascuno dei due tubi da 15 mL e tenerli sul ghiaccio, pronti per la raccolta degli organi.
- Sacrificare un topo adulto per lussazione cervicale.
- Posizionare il mouse sul dorso e sterilizzare la superficie della pelle con etanolo al 70% v/v.
- Per raccogliere gli LN inguinali, fare un'incisione longitudinale di ~ 1 cm sull'addome con le forbici e allungare l'incisione con la pinza.
- Visualizza gli LN inguinali sulla superficie interna della pelle e raccoglili con la pinza. Posizionare gli LN inguinali in uno dei due tubi da 15 mL preparati al punto 3.1.1.
- Per raccogliere la milza, fare un'incisione peritoneale con le forbici e rimuovere la milza. Dopo aver tagliato il tessuto connettivo circostante, posizionare la milza nel secondo tubo da 15 ml preparato al punto 3.1.1.
- Per raccogliere gli LN iliaci, sposta le viscere da parte e visualizza i LN iliaci vicino alla vena cava inferiore, quindi raccoglili usando la pinza. Posizionare gli LN iliaci nello stesso tubo contenente gli LN inguinali.
NOTA: Per ottenere un numero sufficiente di cellule LN per la colorazione (vedere paragrafo 4), è spesso necessario raggruppare IN poplitea, inguinali e iliaci da un topo. Questi LN drenano tutti i quadricipiti (il sito di i.m. vaccinazione). Questo protocollo utilizza un solo tubo da 15 ml di LN in pool. - Per raccogliere LN poplitei, afferrare la pelle delle zampe posteriori e tirarla delicatamente verso il basso per scoprire i muscoli. Quindi, inserire la pinza tra i muscoli sotto l'articolazione del ginocchio e raccogliere i LN poplitei. Posizionare i LN poplitei nello stesso tubo contenente LN inguinali e iliaci.
NOTA: vedere la nota dopo il 3.1.7. - Posizionare la milza in un filtro cellulare da 70 μm all'interno di una capsula di coltura da 60 mm riempita con 5 mL di terreno completo. Utilizzando uno stantuffo a siringa da 5 ml, schiacciare delicatamente l'organo fino alla sua completa disaggregazione.
- Rimuovere il filtro e trasferire la sospensione cellulare in un tubo pulito da 15 ml.
- Aggiungere 5 ml di terreno completo al piatto di coltura e lavare accuratamente il piatto e il colino per assicurarsi che tutte le cellule siano state recuperate. Piscina con il resto della sospensione della cellula della milza nel tubo da 15 ml.
- Per i LN inguinali, iliaci e popliteali raggruppati, preparare una sospensione a singola cellula seguendo una procedura simile a quella utilizzata nei passaggi da 3.1.9 a 3.1.11 per la milza.
- Centrifugare celle a 400 × g per 10 min a 4 °C. Scartare il surnatante e risospesciare i pellet cellulari in PBS.
- Contare le cellule con una camera Neubauer utilizzando il tampone di lisi dei globuli rossi e lo 0,04% v/v di tripano blu in PBS.
- Isolamento delle cellule BM
- Posizionare 5 mL di mezzo completo in un tubo da 15 ml e tenerlo sul ghiaccio, pronto per la raccolta delle zampe posteriori.
- Sacrificare un topo adulto per lussazione cervicale.
- Sterilizzare la superficie della pelle con etanolo al 70% v/v.
- Fai un'incisione trasversale di ~ 1 cm sulla pelle ventrale con le forbici, afferra saldamente la pelle su entrambi i lati del taglio e tira delicatamente verso il basso per scoprire i muscoli delle zampe posteriori.
- Per eliminare la pelle dalla parte posteriore delle zampe posteriori, mantenendo il mouse in posizione supina, posizionare il morsetto sotto il ginocchio e tirare verso l'alto per esporre i muscoli.
- Tagliare le ossa alle due estremità di una zampa posteriore: l'articolazione pelvica / anca e la caviglia.
- Trasferire entrambe le zampe posteriori sul tubo da 15 mL preparato al punto 3.2.1. Tenere il tubo sul ghiaccio.
- Prendi le zampe posteriori dal tubo da 15 ml e trasferiscile su carta velina. Tagliare le zampe posteriori appena sotto l'articolazione del ginocchio per rimuovere la tibia. Sezionare il femore e la tibia dai muscoli circostanti, rimuovere il tessuto in eccesso usando le forbici e bagnare la carta velina.
- Tagliare le estremità dell'osso con le forbici per esporre l'albero del midollo interno. Inserire la tibia e il femore nel tubo di estrazione BM (vedere preparazione in 3.2.9.1-3.2.9.218), con l'estremità più larga nella parte inferiore.
- Tagliare una punta della pipetta da 200 μL sulla linea appena sopra l'estremità della punta e sulla linea da 100 μL.
- Posizionare la parte centrale nella sezione superiore e più grande della punta e posizionarla in un tubo di microfuga da 1,5 ml.
- Ruotare il tubo di estrazione BM a 800 × g per 1 min.
- Scartare l'osso e risospescire vigorosamente il pellet in 1 mL di mezzo completo per rimuovere eventuali grappoli. Filtrare la sospensione cellulare attraverso un filtro da 70 μm posto sulla parte superiore di un tubo da 15 ml.
- Lavare il tubo di estrazione BM due volte con 1 mL di mezzo completo ogni volta. Filtrare attraverso un filtro da 70 μm e raggruppare il volume con il resto della sospensione cellulare ottenuta nel passaggio 3.2.11.
NOTA: un singolo tubo da 15 ml conterrà cellule da entrambe le zampe posteriori di un topo. - Centrifugare celle a 400 × g per 10 min a 4 °C. Scartare il surnatante e risospescere il pellet cellulare in PBS.
- Contare le cellule con una camera Neubauer utilizzando il tampone di lisi dei globuli rossi e lo 0,04% v/v di tripano blu in PBS.
4. Colorazione delle cellule della milza, LN e BM
- Dividere i campioni cellulari da colorare in 3 sottogruppi: campioni cellulari per la compensazione,comprese le cellule BM di topi non trattati da colorare solo con Hoechst 33342 (d'ora in poi denominato Hoechst) e le cellule della milza di topi non trattati da utilizzare per preparare una miscela di cellule morte/vive per la compensazione del colorante a cellule morte; controllo positivo per l'analisidel ciclo cellulare, costituito da un campione di BM di topi non trattati; e campioni sperimentali contenenti campioni di milza e LN di topi non trattati e vaccinati.
NOTA: Assicurarsi che vi siano abbastanza cellule della milza e LN per l'analisi di un numero sufficiente di cellule T CD8 specifiche del gag. Spesso è necessario utilizzare cellule della milza raggruppate e cellule LN raggruppate da 3 topi vaccinati e macchiare due o più campioni identici di cellule raggruppate, ciascuna contenente 3 × 106 cellule. Unire campioni identici nella fase di colorazione hoechst. Allo stesso modo, le cellule della milza e le cellule LN di 3 topi non trattati hanno colorato e fondono campioni identici alla fine. Mettere da parte un campione non macchiato di cellule della milza da un topo non trattato da utilizzare per la configurazione dello strumento e della compensazione. - Preparare la miscela di cellule morte / vive per la compensazione del colorante a cellule morte (questa miscela di cellule sarà macchiata solo con il colorante a cellule morte).
- Riscaldare un bagno d'acqua a 65 °C.
- Prendi un'aliquota delle cellule della milza (~3 × 106).
- Trasferire la sospensione cellulare in un tubo di microfuga, metterla a bagnomaria a 65 °C per 5 minuti, quindi metterla immediatamente sul ghiaccio per 10 minuti.
- Mescolare le cellule uccise dal calore con cellule della milza vive (~ 3 × 106) in un rapporto di 1: 1 e trasferire metà della miscela in una piastra inferiore ben rotonda 96 (~ 3 × 106 cellule / pozzetto per il controllo della colorazione delle cellule morte).
- Colorazione di cellule morte di campioni sperimentali, controllo positivo per l'analisi del ciclo cellulare e miscela di cellule morte/vive
- Trasferire le cellule milza, LN, BM (3 × 106 cellule/pozzetto) e la miscela cellulare morta/viva (sezione 4.2) in una piastra a fondo tondo a 96 pozzetti, secondo lo schema di colorazione (fase 4.1), e centrifugare a 400 × g per 3 minuti a 4 °C.
- Risospescire ogni pellet cellulare in 50 μL di colorante a cellule morte diluito in PBS e risospesso mediante pipettaggio su e giù 3 volte immediatamente.
- Incubare per 30 minuti a 4 °C, al riparo dalla luce.
- Lavare le celle 2 volte con tampone colorante; la prima volta con 200 μL e la seconda volta con 250 μL. Per ogni lavaggio centrifugare la piastra a 400 × g per 3 min a 4 °C.
- Scartare il surnatante e risospesciare il pellet cellulare in 20 μL di PBS.
- Colorazione delle cellule di membrana con multimeri e mAbs e mAbs del complesso maggiore di istocompatibilità (MHC).
- Tenendo conto dei volumi necessari secondo lo schema di colorazione (impostazioni del citometro a flusso, Tabella 1), preparare i seguenti reagenti:
- Diluire mAb 2.4G2 nel tampone di colorazione secondo l'appropriata diluizione (vedi Tabella dei materiali); per ogni campione da colorare, utilizzare 10 μL di questa diluizione.
NOTA: 2.4G2 mAb blocca il legame non antigene-specifico delle immunoglobuline ai recettori FcγIII e FcγII. - Diluire il tetramero marcato con alloficocianina (TEtr-gag) H-2k(d) AMQMLKETI (APC) nel tampone di colorazione per ottenere la diluizione appropriata (vedere Tabella dei materiali); per ogni campione da colorare, utilizzare 20 μL di questa diluizione.
- Preparare la miscela di anticorpi diluendo mAbs nel tampone colorante secondo l'appropriata diluizione (vedi Tabella dei materiali)che è stata precedentemente determinata negli esperimenti di titolazione; per ogni campione da colorare, utilizzare 20 μL di questa miscela di anticorpi.
NOTA: Qui sono stati utilizzati la proteina clorofilla anti-CD3e peridina (PerCP-Cy5.5) (clone 145-2C11), l'ultravioletto brillante anti-CD8a (BUV805) (clone 53-6.7) e la ficoeritrina cyanine7 anti-CD62L (PECy7) (clone MEL-14).
- Diluire mAb 2.4G2 nel tampone di colorazione secondo l'appropriata diluizione (vedi Tabella dei materiali); per ogni campione da colorare, utilizzare 10 μL di questa diluizione.
- Aggiungere 10 μL del mAb 2.4G2 precedentemente diluito (passo 4.4.1.1) e incubare per 10 minuti a 4 °C, al riparo dalla luce.
- Aggiungere 20 μL di Tetr-gag APC precedentemente diluito (punto 4.4.1.2) e 10 μL di H-2k(d) AMQMLKETI phycoerythrin (PE) pentamer (pent-gag). Incubare per 15 minuti a 4 °C, al riparo dalla luce.
- Aggiungere 20 μL della miscela di anticorpi precedentemente preparata (fase 4.4.1.3) e incubare 15 minuti a 4 °C, al riparo dalla luce.
NOTA: Quindi, il volume finale è di 80 μL per pozzetto (passo 4.3.5, passaggi da 4.4.2 a 4.4.4). - Lavare le celle con 200 μL di tampone colorante. Centrifugare a 400 × g per 5 min a 4 °C.
- Risospendare il pellet cellulare in 250 μL di tampone colorante e trasferire la sospensione cellulare in tubi da 5 mL. Aggiungere 1 mL di tampone colorante al tubo e centrifugare a 400 × g per 5 minuti a 4 °C.
- Prelevare l'aliquota delle cellule BM (3 × 106 cellule) (vedere elenco dei campioni cellulari, sezione 4.1) da utilizzare per compensare il canale di Hoechst (Hoechst 33342 è eccitato da un laser ultravioletto (impostazioni del citometro a flusso (Tabella 2)) e trasferire la sospensione cellulare in un tubo da 5 ml. Aggiungere 1 mL di tampone colorante al tubo e centrifugare 400 × g per 5 minuti a 4 °C.
- Tenendo conto dei volumi necessari secondo lo schema di colorazione (impostazioni del citometro a flusso, Tabella 1), preparare i seguenti reagenti:
5. Fissazione/permeabilizzazione
- Preparare un tampone di fissazione / permeabilizzazione fresco diluendo 1 parte di concentrato di fissazione / permeabilizzazione con 3 parti di diluente di fissazione / permeabilizzazione, secondo le istruzioni del produttore.
- Scartare il surnatante e pulsare vortex i campioni per disaggregare completamente il pellet.
- Aggiungere 1 mL del tampone di fissazione/permeabilizzazione appena preparato a ciascun tubo, compreso un tubo con cellule della milza non macchiate (3 x 106,vedere elenco dei campioni cellulari, sezione 4.1) e vortice.
- Incubare per 16 ore a 4 °C.
NOTA: il protocollo può essere messo in pausa qui.
6. Colorazione intracellulare
- Colorazione Ki67
- Preparare il tampone di permeabilizzazione fresco 1x diluendo il tampone di permeabilizzazione 10x con acqua distillata, secondo le istruzioni del produttore. Prima dell'uso, il tampone di permeabilizzazione 1x deve essere filtrato attraverso un filtro da 0,45 μm per eliminare gli aggregati.
- Diluire mAb Ki67 fluoresceina isotiocianato (FITC) (clone SolA15) nel tampone di permeabilizzazione 1x (vedere Tabella dei materiali),come determinato in precedenza negli esperimenti di titolazione (volume finale di 100 μL per campione).
- Aggiungere 3 mL di tampone di permeabilizzazione 1x a ciascun tubo e centrifugare a 400 × g per 5 minuti a temperatura ambiente (RT).
- Scartare il surnatante e ripetere il passaggio 6.1.3.
- Scartare il surnatante e risospesciare il pellet cellulare in 100 μL di mAb Ki67 FITC precedentemente diluito (fase 6.1.2).
- Incubare per 30 minuti a RT, al riparo dalla luce.
- Lavare le celle 2 volte con 4 mL di tampone di permeabilizzazione 1x. Per ogni centrifuga di lavaggio a 400 × g per 5 min a RT.
- Risospesso il pellet cellulare in PBS considerando i seguenti volumi: 350 μL di PBS per i campioni da acquisire direttamente al citometro a flusso; 250 μL di PBS per i campioni da incubare con Hoechst poco prima della citometria a flusso (paragrafo 6.2).
- Colorazione del DNA
- Aggiungere 250 μL di 4 μg/mL di Hoechst in PBS a ciascun campione (la concentrazione finale di Hoechst è di 2 μg/mL).
NOTA: Nel caso in cui siano stati preparati due o più campioni identici di 250 μL in PBS, unirli in questa fase e aggiungere un volume uguale di 4 μg/mL di soluzione di Hoechst in PBS (la concentrazione finale di Hoechst è di 2 μg/mL). Il numero di cellule influenza notevolmente la fase di colorazione del DNA. Utilizzare lo stesso numero di cella in ogni campione. Essere consapevoli del fatto che anche un numero di cellule leggermente ridotto (ad esempio, a causa della perdita di cellule nelle precedenti fasi di lavaggio) si traduce in un maggiore legame di Hoechst al DNA e in una maggiore intensità di Hoechst. - Incubare per 15 minuti a RT, al riparo dalla luce.
- Centrifugare i campioni a 400 × g per 5 minuti a RT.
- Risospesciare il pellet di cella in 350 μL di PBS.
- Aggiungere 250 μL di 4 μg/mL di Hoechst in PBS a ciascun campione (la concentrazione finale di Hoechst è di 2 μg/mL).
7. Preparazione di campioni di perline di compensazione
- Preparare 5 μL dell'anticorpo diluendo mAb nel tampone colorante in modo appropriato.
NOTA: Per ogni mAb coniugato al fluorocromo utilizzato nell'esperimento, preparare il campione di perline di compensazione corrispondente. - Vortex Negative Control e Anti-Rat/Hamster Ig,κ Comp Beads prima dell'uso.
- Per ogni campione, introdurre una goccia (~20 μL) di CompBeads a controllo negativo e una goccia di Anti-Rat/Hamster Ig,k CompBeads.
- Aggiungere 5 μL dell'anticorpo prediluito (fase 7.1) al tubo e pipettare su e giù.
- Incubare per 15 minuti a 4 °C, al riparo dalla luce.
- Lavare i campioni con 2 ml di tampone colorante. Centrifugare a 400 × g per 5 min a 4 °C.
- Scartare il surnatante e risospesciare il pellet aggiungendo 500 μL di PBS a ciascun tubo e vortice.
8. Strumentazione e configurazione della compensazione e acquisizione sperimentale del campione al citometro a flusso
NOTA: fare riferimento alle impostazioni del citometro a flusso (Tabella 2) per la configurazione del citometro.
- Configurazione generale dello strumento e della compensazione
- Aprire il software per l'acquisizione di campioni (vedere Tabella dei materiali)e creare un nuovo esperimento facendo clic su Nuovo esperimento nella sezione della barra multifunzione dell'area di lavoro e selezionando Nuovo esperimento vuoto.
- Fare doppio clic sull'esperimento creato per aprirlo.
- Nella finestra Impostazioni citometro, fare clic su Parametri e selezionare tutti i canali (ad esempio, PE, APC, ecc.) utilizzati nel pannello di colorazione, inclusi i parametri Forward Scatter (FSC) e Side Scatter (SSC).
- Selezionare la scala lineare come parametro Hoechst deselezionando la scala di log e controllare la larghezza (W) dell'impulso di tensione per FCS, SSC e Hoechst.
NOTA: tutti i parametri sono visualizzati per impostazione predefinita in scala logaritmica (log), ad eccezione di FSC e SSC che sono in scala lineare. Tutti i parametri vengono analizzati dall'Area (A) e dall'Altezza (H) dell'impulso di tensione. - Nel foglio di lavoro globalecreare un dot plot con FSC-A sull'asse x e SSC-A sull'asse y.
- Eseguire l'esempio di milza non macchiata facendo clic su Acquisisci dati nel dashboard di acquisizione.
- Impostare le impostazioni FSC e SSC appropriate per visualizzare le celle modificando i valori di tensione nella sezione Parametri e creare un gate per selezionare tutte le celle visualizzate nel dot plot FSC-A/SSC-A facendo clic su Polygon Gate sulla barra degli strumenti dell'area di lavoro del foglio di lavoroglobale .
- Visualizzare le cellule gated in istogrammi con ogni parametro di fluorescenza sull'asse x.
- Eseguire campioni di milza non macchiati e completamente macchiati per regolare il rilevatore di fluorescenza (PMT) in modo da avere una chiara separazione tra segnali negativi e positivi delle cellule colorate per ciascun parametro di fluorescenza.
- Per eseguire l'impostazione della compensazione, fare clic su Esperimento nella barra multifunzione dell'area di lavoro e nella sezione Impostazione compensazione selezionare Crea controlli di compensazione. Deselezionate Includi tubo di controllo non macchiato/pozzo (Include Unstained Control Tube/Well) e fate clic su OK.
NOTA: questa operazione comporterà la creazione di un campione denominato Compensation Controls e di un foglio di lavoro normale contenente diversi fogli corrispondenti a ciascun parametro selezionato. - Eseguire un campione di perline di compensazione (vedere paragrafo 7); impostare le impostazioni FSC e SSC appropriate per visualizzare le perline modificando i valori di tensione e la soglia di acquisizione di 5.000 sui parametri FSC nella finestra Del citometro.
- Regolare il gate P1 sulla popolazione di perline e verificare che i picchi positivi e negativi siano entrambi visibili sull'asse x. Ripetere questa operazione per ogni campione di perline di compensazione e infine registrare ogni file di esempio facendo clic su Registra dati nel dashboard di acquisizione (registrare almeno 5.000 eventi per ogni campione).
- Per ogni campione di perline registrato, impostare le porte P2 e P3 rispettivamente sui picchi positivi e negativi.
- Eseguire i campioni di cellule per la compensazione (vedere i passaggi 4.2 e 4.4.7 e i paragrafi 5 e 6). Modificare le tensioni FSC e SSC e il valore di soglia per visualizzare le celle, regolare il gate P1 e infine registrare ogni file di esempio (registrare almeno 10.000 eventi). Impostare le porte P2 e P3 rispettivamente sui picchi positivi e negativi.
NOTA: per la compensazione del canale di Hoechst, utilizzare G0/G1 come picco negativo (P3) e G2/M come positivo (P2). - Fare clic su Esperimento nella sezione della barra multifunzione dell'area di lavoro e nella sezione Impostazione compensazione selezionare Calcola compensazione.
- Assegna un nome all'impostazione di compensazione creata, al collegamento e salvala nell'esperimento corrente.
- Acquisizione sperimentale di campioni
- Aprire un campione facendo clic su Nuovo campione sulla barra degli strumenti del browser e creare la strategia di gating nel foglio di lavoro globale.
NOTA: la strategia di gating di acquisizione del campione è simile a quella dell'analisi del campione, descritta nella Figura 3 e nella sezione 9. - Visualizzare la popolazione di tutti gli eventi in un istogramma con CD3-A sull'asse x. Creare un Interval Gate per selezionare solo le celleCD3 +.
- Nel dashboard di acquisizioneselezionare il gate di archiviazione come Tutti gli eventi per i campioni LN e Tutti gli eventi o Le celle CD3+ per i campioni di milza.
- Esegui i campioni sperimentali a bassa velocità e infine registra tutti i file assicurandoti di raccogliere almeno 100-200 cellule T CD8 antigene-specifiche per ogni campione dai topi vaccinati.
NOTA: la dimensione del file dei campioni sperimentali è solitamente grande (30-120 MB), specialmente quando la frequenza delle cellule T CD8 antigene-specifiche è bassa. Quindi, è necessario raccogliere un numero elevato di eventi (> 1 ×10 6) per registrare almeno 100-200 cellule T CD8 antigene-specifiche. I file di grandi dimensioni potrebbero rallentare il successivo processo di analisi dei dati. L'acquisizione di sole cellule CD3+ in campioni di milza (vedere il passaggio 8.2.2 sopra) è utile per mantenere le dimensioni del file più piccole. - Eseguire e registrare il controllo positivo per l'analisi del ciclo cellulare, cioè il campione BM di topi non trattati.
- Aprire un campione facendo clic su Nuovo campione sulla barra degli strumenti del browser e creare la strategia di gating nel foglio di lavoro globale.
9. Analisi dei dati
- Aprire il software (vedere Tabella dei materiali)e creare diversi gruppi corrispondenti ai diversi organi da analizzare facendo clic su Crea gruppo nella sezione della barra multifunzione dell'area di lavoro (ad esempio, creare un gruppo "a-LNs"; "b-milza"; "c-BM").
NOTA: i gruppi appena creati appariranno nell'elenco dei gruppi, mentre il gruppo "Compensazione" verrà generato automaticamente dal software. - Aprire la finestra Modifica gruppo facendo doppio clic sul nome del gruppo e verificare che i gruppi appena creati siano sincronizzati. In caso contrario, inserire un segno di spunta sulla funzione Sincronizzato.
- Trascinare ogni file .fcs nel gruppo corrispondente.
- Crea la strategia di gating iniziando con il gruppo "a-LNs".
- Fare doppio clic sul campione completamente macchiato nel gruppo per aprire la finestra del grafico; gli assi x e y sono etichettati come nei file fcs (vedere impostazioni del citometro a flusso, Tabella 2).
- Visualizzare gli eventi totali acquisiti per questo campione in un dot plot con DNA-A sull'asse x e DNA-W sull'asse y.
- Selezionare solo la popolazione di celle singole facendo clic su Rettangolo nella sezione dello strumento gating della finestra del grafico.
NOTA: le singole cellule hanno valori di DNA-A come segue: 2N (basso): tra 2N e 4N (intermedio), o uguale a 4N (alto), mentre i valori di DNA-W sono identici per tutti loro (fase 1 della Figura 3). - Fare doppio clic al centro del cancello rettangolare per visualizzare celle singole in un dot plot con parametro FSC-A sull'asse x e colorante a cellule morte sull'asse y.
- Selezionare solo la popolazione di celle vive facendo clic su Poligono nella sezione dello strumento gating della finestra del grafico. Le cellule vive sono negative per il colorante a cellule morte (fase 2 della Figura 3).
- Fate doppio clic al centro del gate poligonale per visualizzare le celle in un dot plot con il parametro FSC-A sull'asse x e il parametro SSC-A sull'asse y.
- Fare clic su Rettangoloe creare un cancello "rilassato" per includere tutte le singole celle vive in quelgrafico 12 (passaggio 3 della Figura 3).
- Fare doppio clic al centro del cancello "rilassato" per visualizzare le celle in un dot plot con CD3 sull'asse x e CD8 sull'asse y.
- Selezionare le celle CD3+CD8+ facendo clic su Poligono (passaggio 4 della Figura 3).
- Fare doppio clic al centro del gate CD3+CD8+ per visualizzare le celle in un dot plot con Tetr-gag sull'asse x e Pent-gag sull'asse y.
- Selezionare le cellule T CD8 antigene-specifiche (positive sia per Tetr-gag che per Pent-gag) cliccando su Polygon (step 5 of Figure 3).
- Fare doppio clic al centro del gate specifico del gag per visualizzare le celle in un dot plot con DNA-A sull'asse x e Ki67 sull'asse y (Figura 4).
- Selezionare le celle nelle diverse fasi del ciclo cellulare facendo clic su Quad nella sezione dello strumento gating della finestra del grafico.
NOTA: Le cellule in fase G0 sono cellule basse Ki67neg-DNA (quadrante in basso a sinistra); le cellule in G1 sono Ki67pos-DNA basso (quadrante in alto a sinistra); le cellule in S-G2/M sono Ki67pos-DNA intermedio/alto (quadrante in alto a destra) (Figura 4). - Copiare la strategia di gating creata in un campione nel gruppo corrispondente per applicare i gate a tutti i campioni del gruppo.
- Ripetere i passaggi da 9,5 a 9,18 per il "gruppo a-LN".
- Verificare che tutte le porte siano appropriate per ciascun campione del gruppo "b-milza". Per analizzare il ciclo cellulare tra le cellule BM (controllo positivo), fare clic al centro del cancello "rilassato" per visualizzare le cellule in un dot plot con DNA-A sull'asse x e Ki67 sull'asse y.
- Verificare che tutte le porte siano appropriate per ogni campione dei 3 gruppi (ad esempio, per le cellule di milza, LN e BM).
NOTA: Single cell population gate (step 9.7) e Quad gate for cell cycle (step 9.17) possono avere coordinate di gate diverse in campioni diversi, principalmente a causa delle possibili lievi differenze dell'intensità del colorante Hoechst tra i campioni (sezione 6.2). Per questo motivo, potrebbe essere necessario modificare il single cell population gate e il Quad gates per il ciclo cellulare in ciascun campione. Questo verrà fatto come segue: fare doppio clic sul nome del gruppo e rimuovere la sincronizzazione dalle proprietà del gruppo. Questa operazione permette la modifica dei cancelli in un campione senza modificare gli stessi cancelli in tutti gli altri campioni del gruppo. Dopo la rimozione della sincronizzazione, modificare i cancelli se necessario. - Per visualizzare i risultati ottenuti da questa analisi, fare clic su Editor layout nella sezione della barra multifunzione dell'area di lavoro per aprirla. Trascinate ogni gate della strategia di gating nel riquadro di esempio nell'editor di layout e posizionate i grafici in base alla sequenza della strategia di gating. Se necessario, modificare il tipo di grafico facendo doppio clic sul plottaggio corrispondente nel layout e selezionando il tipo appropriato nella finestra Definizione grafico.
- Fare clic sul gruppo e scorrere per funzioni sulla barra multifunzione del layout per visualizzare i risultati ottenuti in ciascun organo e confrontare diversi campioni.
Risultati
Le fasi del ciclo cellulare delle cellule di milza, LNs e BM di topi Balb/ c sono state analizzate utilizzando il colorante fluorescente del DNA, Hoechst, e un mAb anti-Ki67, secondo il protocollo riassunto in Figura 1. Questa colorazione ha permesso la differenziazione delle cellule nelle seguenti fasi del ciclo cellulare: G0 (Ki67neg, con 2N di DNA definito come DNAlow), G1 (Ki67pos, DNAlow), e S-G2/M(Ki67pos, con un contenuto di DNA compreso tra 2N e 4N, o pari a 4N di DNA definito come DNAintermediato/alto).
Abbiamo prima eseguito l'analisi del ciclo cellulare delle cellule BM per riprodurre i risultati precedentemente pubblicati13,14 e poi analizzato le cellule di interesse, cioè le cellule T CD8. La Figura 2 mostra un tipico esempio di analisi del ciclo cellulare delle cellule BM (Figura 2A). Il protocollo ha prodotto un basso coefficiente di variazione (CV) dei picchi di DNA G0/G1 e G2/M, indicando l'ottima qualità della colorazione del DNA(Figura 2B,mostrando un esempio con CV < 2.5; CV era sempre < 5 in tutti gli esperimenti).
Abbiamo quindi applicato lo stesso protocollo alle cellule T CD8 antigene-specifiche di topi vaccinati. I topi BALB / c sono stati vaccinati contro il gag dell'antigene dell'HIV-1 usando Chad3-gag per l'innesco e MVA-gag per il potenziamento, entrambi progettati per trasportare il gag hiv-1. Al giorno (d) 3 post-boost, abbiamo analizzato la frequenza delle cellule T CD8 specifiche del gag dalla milza e dai LN drenanti. Abbiamo approfittato della strategia di gating appena definita per le cellule T nella fase iniziale della risposta immunitaria, che in contrasto con la strategia convenzionale, è appropriata per rilevare le cellule T CD8 che rispondono all'antigene altamente attivate12. Abbiamo eseguito la nuova strategia in cinque fasi successive. Nella fase 1, abbiamo escluso doppietti o aggregati per porta DNA-A / -W e nella fase 2, abbiamo identificato le cellule vive mediante esclusione di marcatori di cellule morte. Nella fase 3, abbiamo identificato la popolazione di interesse utilizzando un gate FSC-A / SSC-A "rilassato" non convenzionale(Figura 3A)invece del gate linfocitario stretto canonico12. Dopo aver gating su celle CD3+CD8+ (fase 4 della Figura 3A), abbiamo identificato le cellule T CD8 specifiche del gag utilizzando due diversi multimeri MHC, cioè Pent-gag e Tetr-gag (passo 5 della Figura 3A). Abbiamo usato due multimeri invece di uno per migliorare la sensibilità del rilevamento delle cellule T CD8 specifiche del gag nei topi vaccinati, senza aumentare lo sfondo di colorazione nei topi non trattati(Figura 3B e C,fase 5). Pertanto, abbiamo distinto con successo topi non trattati (0,00% e 0,00% di cellule T CD8 antigene-specifiche in LNs e milza, rispettivamente) da topi vaccinati (0,46% e 0,29% di cellule T CD8 antigene-specifiche in LNs e milza, rispettivamente, Figura 3B e C).
In particolare, il protocollo ci ha permesso di avere uno sfondo estremamente basso nella porta delle cellule T CD8 antigene-specifiche dei LN e della milza di topi non trattati (di solito 0,00% e al massimo 0,02%). Il confronto tra grafici FSC-A / SSC-A specifici del gag e non specifici del gag ha mostrato che le cellule specifiche del gag avevano alti SSC-A e FSC-A(Figura 3D),confermando la necessità di utilizzare un gate FSC-A / SSC-A "rilassato" per catturare queste cellule. Abbiamo quindi valutato le percentuali di cellule T CD8 specifiche del gag in diverse fasi del ciclo cellulare (Figura 4A). Abbiamo scoperto che le cellule T CD8 specifiche del gag nella milza e ancora di più nei LN drenanti contenevano un'alta percentuale di cellule nelle fasi S-G2/ M al giorno 3 post-boost (18,60% e 33,52%, rispettivamente).
Inoltre, abbiamo scoperto che le cellule T CD8 specifiche del gag nelle fasi S-G2/ M avevano un alto FSC-A e SSC-A, quando sovrapposte alle cellule T CD8 totali dello stesso organo (Figura 4B). L'espressione di CD62L da parte di cellule T CD8 specifiche del gag era bassa, come previsto per le cellule T attivate, ad eccezione di alcune cellule in G0 negli LNs (Figura 4C). Complessivamente, questi risultati hanno confermato che il gate "rilassato" (fase 3 della Figura 3A, B e C)era necessario per includere tutte le cellule T CD8 antigene-specifiche proliferanti12. Il protocollo è stato estremamente prezioso per una valutazione "istantanea" delle fasi del ciclo cellulare delle cellule T CD8 antigene-specifiche al momento dell'analisi e dell'espressione di CD62L da parte delle cellule in diverse fasi del ciclo cellulare.

Figura 1: Schema del protocollo per l'analisi del ciclo cellulare di cellule T CD8 antigene-specifiche. Fare clic qui per visualizzare una versione più grande di questa figura.
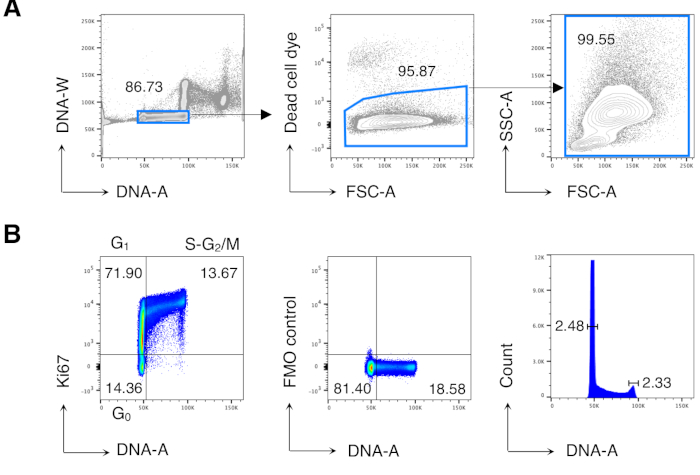
Figura 2: Analisi del ciclo cellulare delle cellule BM. Le cellule BM di topi Balb/c non trattati sono state colorate e analizzate mediante citometria a flusso. (A) Esempio di strategia di gating. Abbiamo recintato su singole cellule nel diagramma DNA-A /-W (a sinistra) e successivamente su cellule vive per esclusione di coloranti a cellule morte (al centro). Quindi, un cancello FSC-A / SSC-A "rilassato" è stato utilizzato per tutte le celle BM (a destra). (B) Esempio di analisi del ciclo cellulare di cellule BM (a sinistra). Abbiamo usato una combinazione di Ki67 e colorazione del DNA per identificare le cellule nelle seguenti fasi del ciclo cellulare: G0 (quadrante in basso a sinistra, cellule Ki67neg-DNAlow), G1 (quadrante in alto a sinistra, Ki67pos-DNAlow), S-G2/ M (quadrante in alto a destra, Ki67pos-DNAintermediato / alto). Vengono mostrati il controllo della fluorescenza Meno Uno (FMO) di Ki67 mAb (al centro) e l'istogramma del DNA (a destra). Nel grafico dell'istogramma del DNA, le porte sinistra e destra corrispondono rispettivamente al picco del DNA G0/G1 e G2/M e i numeri rappresentano i coefficienti di variazione (CV) di ciascun picco. In tutti gli altri grafici, i numeri rappresentano le percentuali di cella nelle porte indicate. La figura mostra 1 esperimento rappresentativo su 5. In ogni esperimento, abbiamo analizzato le cellule BM raggruppate da 3 topi. Fare clic qui per visualizzare una versione più grande di questa figura.
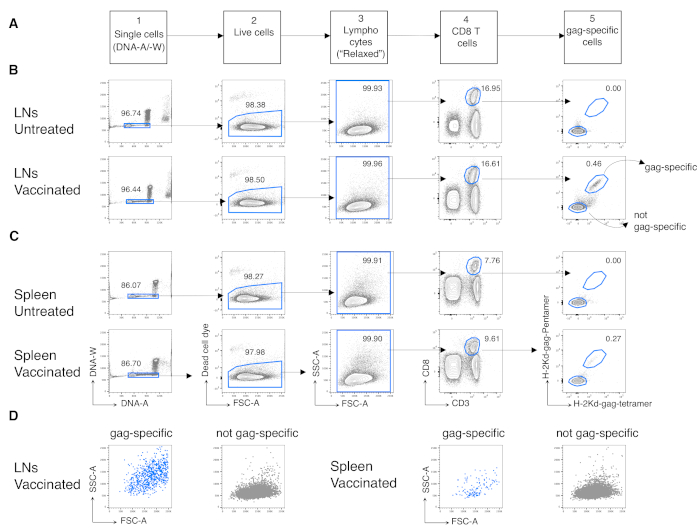
Figura 3: Analisi di cellule T CD8 antigene-specifiche da LNs e milza. I topi Balb/c sono stati innescati per via intramuscolare (i.m.) con Chad3-gag e potenziati i.m. con MVA-gag. Al giorno 3 post-boost, le cellule LN e della milza drenanti da topi di controllo vaccinati e non trattati sono state colorate e analizzate mediante citometria a flusso. (A) Schema della strategia di gating in cinque fasi per identificare le singole cellule (fase 1); cellule vive (Fase 2); linfociti (Fase 3); Cellule T CD8 (Fase 4); e celle specifiche del gag (Passaggio 5). (B-C) Esempio di grafici: analisi di cellule da (B) LNs e (C) milza di topi non trattati (in alto) e vaccinati (in basso). Abbiamo identificato singole cellule sul grafico DNA-A / -W nella fase 1. Quindi, nella fase 2, abbiamo selezionato le cellule vive per esclusione del colorante a cellule morte. Nella fase 3, abbiamo usato un cancello "rilassato" non canonico per i linfociti. Nella fase 4, abbiamo identificato le cellule T CD8 dalla loro doppia espressione di CD3 e CD8. Abbiamo quindi identificato cellule specifiche del gag e non specifiche del gag nella fase 5, in base alla loro capacità di legare rispettivamente H-2kd-gag-pentamero marcato con fluorocromo (Pent-gag) e H-2kd-gag-tetramero (Tetr-gag), o meno. (D) Profili FSC-A/SSC-A di cellule specifiche del gag (blu) e non specifiche del gag (grigie) dopo la gating come descritto sopra. I numeri rappresentano le percentuali di cella nelle porte indicate. La figura mostra 1 esperimento rappresentativo su 5. In ogni esperimento, abbiamo analizzato la milza aggregata e le cellule LN raggruppate da 3 topi vaccinati e 3 topi non trattati. Fare clic qui per visualizzare una versione più grande di questa figura.
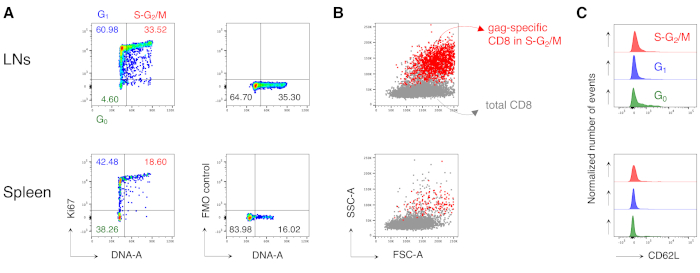
Figura 4: Analisi del ciclo cellulare di cellule T CD8 antigene-specifiche. I topi sono stati vaccinati come nella Figura 3 e l'analisi del ciclo cellulare delle cellule specifiche del vomito è stata eseguita al giorno 3 post-boost, dopo il gating in 5 fasi come nella Figura 3. (A)Esempio di analisi del ciclo cellulare di cellule T CD8 gag-specifiche da LNs (in alto) e milza (in basso) di topi vaccinati. Le fasi del ciclo cellulare sono state identificate come nella Figura 2B. I pannelli rappresentano celle in G0, in G1, e in S-G2/ M (a sinistra) e Fluorescenza Minus One (FMO) controllo di Ki67 mAb (a destra). I numeri rappresentano le percentuali di cella nelle porte indicate. (B) Grafici a punti FSC-A/SSC-A che mostrano cellule T CD8 specifiche del gag nelle fasi S-G2/M (in rosso) sovrapposte alle cellule T CD3+CD8+ totali (in grigio) da LNs (in alto) e milza (in basso) di topi vaccinati. (C) Istogrammi offset che mostrano l'espressione di CD62L da parte di cellule T CD8 specifiche del gag in G0 (verde), in G1 (blu) e in S-G2/ M (rosso) da LNs (in alto) e milza (in basso) di topi vaccinati. Gli assi y indicano il numero normalizzato di eventi. La figura mostra 1 esempio rappresentativo su 5 esperimenti indipendenti con un totale di 15 topi. Fare clic qui per visualizzare una versione più grande di questa figura.
Materiale supplementare: Impostazioni del citometro a flusso. Fare clic qui per scaricare questo file.
Discussione
Sebbene l'espansione clonale delle cellule T sia stata intensamente studiata, alcuni aspetti rimangono sconosciuti, soprattutto perché gli strumenti disponibili per indagarla sono pochi e hanno i loro svantaggi. Da questo punto di vista, abbiamo impostato un metodo citometrico a flusso altamente sensibile per analizzare il ciclo cellulare delle cellule T CD8 antigene-specifiche nei primi momenti dopo la vaccinazione in un modello murino. Il protocollo si basa su una combinazione di Ki67 e colorazione del DNA, che è stata precedentemente utilizzata per analizzare il ciclo cellulare delle cellule ematopoietiche BM nei topi13,14. Per adattare il protocollo alle cellule T CD8 antigene-specifiche, abbiamo dovuto considerare alcune criticità, tra cui la scelta del colorante DNA, le condizioni appropriate per ottenere una colorazione del DNA comparabile su diversi campioni e la strategia di gating per l'analisi dei dati.
Molti coloranti sono disponibili per la colorazione del DNA, tra cui ioduro di propidio e 7-aminoactinomicina D; abbiamo scelto Hoechst perché era compatibile con la colorazione a membrana e il protocollo di fissazione / permeabilizzazione delicata richiesto per la colorazione Ki67. Allo stesso tempo, la colorazione con Hoechst ci ha permesso di ottenere un istogramma del DNA di ottima qualità, cioè i picchi di DNA G0/ G1 e G2/ M avevano un coefficiente di variazione (CV) molto più basso rispetto ai picchi di DNA solitamente ottenuti con altri coloranti del DNA, ad esempio DRAQ519. Infatti, Hoechst può macchiare il DNA anche nelle cellule vive20.
Alcune strategie sono state utilizzate per evitare la fluttuazione dell'intensità di Hoechst in diversi campioni dello stesso esperimento. La colorazione di Hoechst è stata eseguita poco prima dell'acquisizione del campione al citometro a flusso per ridurre al minimo il declino dell'intensità del colorante nel tempo. Per coloro che sono interessati a riprodurre il protocollo in grandi esperimenti con numerosi campioni, si consiglia di eseguire la colorazione Hoechst su pochi campioni alla volta. Un altro inconveniente è che l'intensità di Hoechst può essere fortemente influenzata dal numero di cellule durante l'incubazione con il colorante. Per questo motivo, consigliamo vivamente di utilizzare sempre lo stesso numero di cellule e lo stesso volume per campione per la colorazione del DNA. Se è necessario un numero elevato di cellule per l'acquisizione al citometro a flusso, si consiglia di preparare due o più campioni identici e quindi di unirli appena prima della fase di colorazione di Hoechst.
Un punto chiave del protocollo è la strategia di gating per l'analisi dei dati. Abbiamo recentemente pubblicato una nuova strategia per l'analisi delle cellule T nei primi momenti della risposta immunitaria, che ci ha permesso di aumentare la sensibilità di rilevamento delle cellule T antigene-specifiche12. Abbiamo applicato questa strategia ai dati mostrati qui come segue. In primo luogo, abbiamo escluso gli aggregati cellulari nel grafico DNA-A/W. In secondo luogo, dopo aver eliminato le cellule morte, abbiamo usato un cancello linfocitario abbastanza grande nella trama FSC / SSC ("cancello rilassato"). Con questa strategia, siamo stati in grado di includere cellule T CD8 antigene-specifiche altamente attivate in S-G2/ M che di solito sono mancate dalle attuali strategie di gating, poiché queste cellule hanno un alto FSC-A e SSC-A. In sintesi, l'analisi dei dati rappresenta una parte critica del metodo, essenziale per ottenere una rilevazione sensibile di cellule T antigene-specifiche attivate/proliferanti.
Il metodo previene la possibilità di perdere dati critici sulle cellule T nelle prime fasi della risposta immunitaria e apre nuove prospettive per l'immuno-monitoraggio delle cellule T. Un miglioramento futuro potrebbe essere quello di includere la colorazione per il fosfo-istone 3 che consentirebbe la differenziazione tra G2 e M21. Una limitazione attuale è che le cellule devono essere fissate e permeabilizzate per colorare il marcatore nucleare, Ki67. Pertanto, le celle non possono essere utilizzate per altri tipi di analisi come l'ordinamento e la successiva analisi funzionale. Inoltre, i coloranti del DNA, incluso Hoechst, di solito interferiscono con l'analisi genomica del DNA e non sono adatti a questo tipo di valutazione. L'identificazione di marcatori di membrana che si correlano con diverse fasi del ciclo cellulare e che possono essere colorati su cellule vive potrebbe superare questa limitazione. In conclusione, il metodo ha un grande potenziale per la valutazione delle cellule T attivate/proliferanti in diversi contesti come la vaccinazione, l'infezione, le malattie immuno-mediate e l'immunoterapia.
Divulgazioni
A. Folgori e S. Capone sono dipendenti di Reithera Srl. A. Nicosia è nominata inventore sulla domanda di brevetto WO 2005071093 (A3) "Chimpanzee adenovirus vaccine carriers". Gli altri autori non hanno nulla da rivelare.
Riconoscimenti
Questo lavoro è stato sostenuto da Reithera, dal progetto MIUR 2017K55HLC_006 e dal contributo di 5 × 1000 dell'Associazione Italiana Ricerca sul Cancro (AIRC). Il seguente tetramero è stato ottenuto attraverso la NIH Tetramer Facility: H-2K (d) HIV gag 197-205 AMQMLKETI coniugato apc..
Materiali
| Name | Company | Catalog Number | Comments |
| 1-200 μL universal fit bulk packed pipet tips | Corning | CLS4866-1000EA | |
| 2.4G2 anti-FcγR mAb | BD | 553141 | 10 μg/ml final concentration |
| 5 ml syringe plunger | BD Emerald | 307733 | |
| 15 ml conical tubes | MercK Millipore | SBHA025SB | |
| 60 mm TC-treated Cell Culture Dish | Falcon | 353002 | |
| 70 μm cell strainer | Falcon | 352097 | |
| 96-well Clear Round Bottom TC-treated Culture Microplate | Falcon | 353077 | |
| Anti-Rat/Hamster Ig,k/Negative Control Compensation Particles | BD- Bioscience | 552845 | |
| Beta-mercaptoethanol | Sigma | M3148 | |
| Bovine Serum Albumin | Sigma | A07030 | |
| BUV805 Rat Anti-Mouse CD8a | BD- Bioscience | 564920 | 4 μg/ml final concentration |
| Dulbecco's Phosphate Buffer Saline w/o Calcium w/o Magnesium | Euroclone | ECB4004L | |
| Eppendorf Safe-Lock Tubes, 1.5 mL | Eppendorf | 30120159 | |
| Ethanol | Sigma | 34852-1L-M | |
| Ethylenediaminetetraacetic Acid Disodium Salt solution (EDTA) | Sigma | E7889 | |
| Fetal Bovine Serum | Corning | 35-079-CV | |
| Filcon, Sterile, Syringe-Type 70 μm | Falcon | 352350 | |
| Fixable Viability Dye eFluor 780 | eBioscience | 65-0865-14 | 1:1000 final concentration |
| Foxp3 / Transcription Factor Staining Buffer Set | eBioscience | 00-5523-00 | This Set contains fixation/permeabilization concentrate and diluent, and permeabilization buffer 10x |
| H-2k(d) AMQMLKETI allophycocyanin (APC)-labelled tetramer | provided by NIH Tetramer Core Facility | 6 μg/ml final concentration | |
| H-2k(d) AMQMLKETI phycoerythrine (PE) labelled pentamer | Proimmune | F176-2A-E - 176 | 10 μL / sample |
| Hoechst 33342, Trihydrochloride, Trihydrate - 10 mg/mL Solution in Water | ThermoFisher | H3570 | |
| Ki-67 Monoclonal Antibody (SolA15), FITC | eBioscience | 11-5698-82 | 5 μg/ml final concentration |
| L-Glutamine 100X (200 mM) | Euroclone | ECB3000D | |
| Millex-HA Filters 0,45 µm | BD | 340606 | |
| Penicillin/Streptomycin 100X | Euroclone | ECB3001D | |
| PE/Cyanine7 anti-mouse CD62L Antibody | Biolegend | 104418 | 0.2 μg/ml final concentration |
| PerCP-Cy™5.5 Hamster Anti-Mouse CD3e | BD- Bioscience | 551163 | 4.4 μg/ml final concentration |
| Red Blood Cell Lysis Buffer | Sigma | R7757 | |
| Round-Bottom Polystyrene Tubes, 5 mL | Falcon | 352058 | |
| RPMI 1640 Medium without L-Glutamine with Phenol Red | Euroclone | ECB9006L | |
| Software package for analyzing flow cytometry data | FlowJo | v.10 | |
| Software for acquisition of samples at flowcytometer | BD FACSDiva | v 6.2 | |
| Trypan Blue Solution | Euroclone | ECM0990D |
Riferimenti
- Castellino, F., et al. Chemokines enhance immunity by guiding naive CD8+ T cells to sites of CD4+ T cell-dendritic cell interaction. Nature. 440 (7086), 890-895 (2006).
- Zhang, N., Bevan, M. J. CD8(+) T cells: foot soldiers of the immune system. Immunity. 35 (2), 161-168 (2011).
- Bajénoff, M., et al. Highways, byways and breadcrumbs: directing lymphocyte traffic in the lymph node. Trends Immunology. 28 (8), 346-352 (2007).
- Bevan, M. J., Fink, P. J. The CD8 response on autopilot. Nature Immunology. 2 (5), 381-382 (2001).
- Van Stipdonk, M. J., Lemmens, E. E., Schoenberger, S. P. Naïve CTLs require a single brief period of antigenic stimulation for clonal expansion and differentiation. Nature Immunology. 2 (5), 423-429 (2001).
- Kaech, S. M., Wherry, E. J., Ahmed, R. Effector and memory T-cell differentiation: implications for vaccine development. Nature Review Immunology. 2 (4), 251-262 (2002).
- Beverley, P. C. Primer: making sense of T-cell memory. Nature Clinical Practice Rheumatology. 4 (1), 43-49 (2008).
- Parretta, E., et al. CD8 cell division maintaining cytotoxic memory occurs predominantly in the bone marrow. Journal of Immunology. 174 (12), 7654-7664 (2005).
- Di Rosa, F. Maintenance of memory T cells in the bone marrow: survival or homeostatic proliferation. Nature Review Immunology. 16 (4), 271 (2016).
- Di Rosa, F. Two niches in the bone marrow: a hypothesis on life-long T cell memory. Trends in Immunology. 37 (8), 503-512 (2016).
- Di Rosa, F. Commentary: Memory CD8(+) T cells colocalize with IL-7(+) stromal cells in bone marrow and rest in terms of proliferation and transcription. Frontiers in Immunology. 7, 102 (2016).
- Simonetti, S., et al. Antigen-specific CD8 T cells in cell cycle circulate in the blood after vaccination. Scandinavian Journal of Immunology. 89 (2), 12735 (2019).
- Wilson, A., et al. c-Myc controls the balance between hematopoietic stem cell self-renewal and differentiation. Genes & Development. 18 (22), 2747-2763 (2004).
- Hirche, C., et al. Systemic virus infections differentially modulate cell cycle state and functionality of long-term hematopoietic stem cells in vivo. Cell Report. 19 (11), 2345-2356 (2017).
- Colloca, S., et al. Vaccine vectors derived from a large collection of simian adenoviruses induce potent cellular immunity across multiple species. Science Translational Medicine. 4 (115), (2012).
- Di Lullo, G., et al. Marker gene swapping facilitates recombinant Modified Vaccinia Virus Ankara production by host-range selection. Journal of Virological Methods. 156 (1-2), 37-43 (2009).
- Di Lullo, G., et al. The combination of marker gene swapping and fluorescence-activated cell sorting improves the efficiency of recombinant modified vaccinia virus Ankara vaccine production for human use. Journal of Virological Methods. 163 (2), 195-204 (2010).
- . Mouse phenotype Available from: https://www.mousephenotype.org/data/secondaryproject/3i (2020)
- Yoon, H., Kim, T. S., Braciale, T. J. The cell cycle time of CD8+ T cells responding in vivo is controlled by the type of antigenic stimulus. PLoS One. 5 (11), 15423 (2010).
- Pauklin, S., Vallier, L. The cell-cycle state of stem cells determines cell fate propensity. Cell. 155 (1), 135-147 (2013).
- Vignon, C., et al. Flow cytometric quantification of all phases of the cell cycle and apoptosis in a two-color fluorescence plot. PLoS One. 8 (7), 68425 (2013).
Erratum
Formal Correction: Erratum: A DNA/Ki67-Based Flow Cytometry Assay for Cell Cycle Analysis of Antigen-Specific CD8 T Cells in Vaccinated Mice
Posted by JoVE Editors on 11/03/2021. Citeable Link.
An erratum was issued for: A DNA/Ki67-Based Flow Cytometry Assay for Cell Cycle Analysis of Antigen-Specific CD8 T Cells in Vaccinated Mice. The Authors section and a figure were updated.
The authors section was updated from:
Sonia Simonetti*1,2, Ambra Natalini*1,2, Giovanna Peruzzi3, Alfredo Nicosia4, Antonella Folgori5, Stefania Capone5, Angela Santoni2, Francesca Di Rosa1
1Institute of Molecular Biology and Pathology, National Research Council of Italy (CNR),
2Department of Molecular Medicine, University of Rome “Sapienza”,
3Center for Life Nano Science, Istituto Italiano di Tecnologia,
4Department of Molecular Medicine and Medical Biotechnology, University of Naples Federico II,
5Reithera Srl
* These authors contributed equally
To:
Sonia Simonetti*1,2, Ambra Natalini*1,2, Giovanna Peruzzi3, Alfredo Nicosia4, Antonella Folgori5, Stefania Capone5, Angela Santoni2,6, Francesca Di Rosa1
1Institute of Molecular Biology and Pathology, National Research Council of Italy (CNR),
2Department of Molecular Medicine, University of Rome “Sapienza”,
3Center for Life Nano Science, Istituto Italiano di Tecnologia,
4Department of Molecular Medicine and Medical Biotechnology, University of Naples Federico II,
5Reithera Srl
6IRCCS, Neuromed
* These authors contributed equally
Figure 1 was updated from:
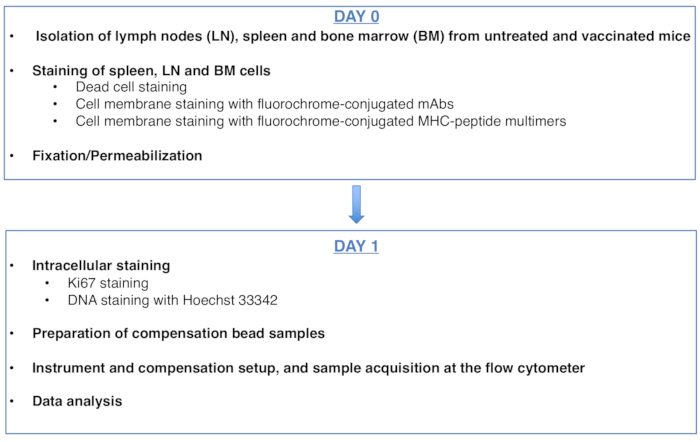
Figure 1: Scheme of the protocol for cell cycle analysis of antigen-specific CD8 T cells. Please click here to view a larger version of this figure.
To:

Figure 1: Scheme of the protocol for cell cycle analysis of antigen-specific CD8 T cells. Please click here to view a larger version of this figure.
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon