È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Caratterizzazione viscoelastica di fantocci di gelatina che imitano i tessuti molli mediante indentazione ed elastografia a risonanza magnetica
In questo articolo
Riepilogo
Questo articolo presenta una dimostrazione e un riassunto dei protocolli di produzione di fantasmi di gelatina che imitano i tessuti molli e la corrispondente caratterizzazione viscoelastica utilizzando l'indentazione e l'elastografia a risonanza magnetica.
Abstract
La caratterizzazione delle proprietà biomeccaniche dei tessuti biologici molli è importante per comprendere la meccanica dei tessuti ed esplorare i meccanismi biomeccanici correlati alla malattia, alle lesioni e allo sviluppo. Il metodo di prova meccanica è il modo più semplice per la caratterizzazione dei tessuti ed è considerato come verifica per la misurazione in vivo . Tra le molte tecniche di test meccanici ex vivo , il test di indentazione fornisce un modo affidabile, specialmente per campioni piccoli, difficili da fissare e viscoelastici come il tessuto cerebrale. L'elastografia a risonanza magnetica (MRE) è un metodo clinicamente utilizzato per misurare le proprietà biomeccaniche dei tessuti molli. Sulla base della propagazione dell'onda di taglio nei tessuti molli registrata utilizzando la risonanza magnetica, le proprietà viscoelastiche dei tessuti molli possono essere stimate in vivo sulla base dell'equazione delle onde. Qui, le proprietà viscoelastiche dei fantocci di gelatina con due diverse concentrazioni sono state misurate mediante MRE e indentazione. Sono stati presentati i protocolli di fabbricazione fantasma, test e stima del modulo.
Introduzione
La maggior parte dei tessuti biologici molli sembrano avere proprietà viscoelastiche che sono importanti per comprendere la loro lesione e sviluppo 1,2. Inoltre, le proprietà viscoelastiche sono importanti biomarcatori nella diagnosi di una varietà di malattie come la fibrosi e il cancro 3,4,5,6. Pertanto, la caratterizzazione delle proprietà viscoelastiche dei tessuti molli è cruciale. Tra le molte tecniche di caratterizzazione utilizzate, i test meccanici ex vivo di campioni di tessuto e l'elastografia in vivo mediante imaging biomedico sono i due metodi ampiamente utilizzati.
Sebbene siano state utilizzate varie tecniche di prova meccanica per la caratterizzazione dei tessuti molli, i requisiti relativi alle dimensioni del campione e alle condizioni di prova non sono facili da soddisfare. Ad esempio, la prova di taglio deve avere campioni fissati saldamente tra le piastre di taglio7. Il test biassiale è più adatto per il tessuto della membrana e ha requisiti specifici di serraggio 8,9. Un test di compressione è comunemente usato per il test dei tessuti, ma non può caratterizzare posizioni specifiche all'interno di un campione10. Il test di indentazione non ha requisiti aggiuntivi per fissare il campione di tessuto e può essere utilizzato per misurare molti campioni di tessuto biologico come il cervello e il fegato. Inoltre, con una piccola testa di penetratore, è possibile testare le proprietà regionali all'interno di un campione. Pertanto, sono stati adottati test di indentazione per testare una varietà di tessuti molli 1,3,11.
La caratterizzazione delle proprietà biomeccaniche dei tessuti molli in vivo è importante per gli studi traslazionali e le applicazioni cliniche della biomeccanica. Le modalità di imaging biomedico come l'ecografia (US) e la risonanza magnetica (RM) sono le tecniche più utilizzate. Sebbene l'imaging statunitense sia relativamente economico e facile da eseguire, soffre di basso contrasto ed è difficile misurare organi come il cervello. In grado di visualizzare strutture profonde, l'elastografia MR (MRE) potrebbe misurare una varietà di tessuti molli6,12, in particolare il cervello13,14. Con la vibrazione esterna applicata, la MRE potrebbe misurare le proprietà viscoelastiche dei tessuti molli a una frequenza specifica.
Gli studi hanno dimostrato che a 50-60 Hz, il modulo di taglio del cervello normale è ~1,5-2,5kPa 5,6,13,14,15 e ~2-2,5 kPa per il fegato normale 16. Pertanto, i fantocci di gelatina che hanno proprietà biomeccaniche simili sono stati ampiamente utilizzati per imitare i tessuti molli per test e convalida17,18,19. In questo protocollo, sono stati preparati e testati fantocci di gelatina con due diverse concentrazioni. Le proprietà viscoelastiche dei fantocci di gelatina sono state caratterizzate utilizzando un dispositivo MRE elettromagnetico personalizzato14 e un dispositivo di indentazione 1,3. I protocolli di test potrebbero essere utilizzati per testare molti tessuti molli come il cervello o il fegato.
Protocollo
1. Preparazione fantasma di gelatina
- Pesare gelatina, glicerolo e acqua secondo la Tabella 1. Mescolare la polvere di gelatina con acqua per ottenere la soluzione di gelatina.
NOTA: Le concentrazioni dei singoli componenti per la preparazione dei due fantocci sono indicate nella tabella 1. Maggiore è la concentrazione di gelatina, più rigido è il fantasma. - Riscaldare la soluzione di gelatina a 60 °C a bagnomaria. Aggiungere glicerolo alla soluzione di gelatina mantenendo la temperatura.
NOTA: Il glicerolo stabilizza le miscele di gelatina aumentando la loro temperatura di fusione e il modulo di taglio17. - Mescolare la soluzione e riscaldarla nuovamente a 60 °C. Versare la soluzione miscelata in un contenitore che verrà utilizzato per i test MRE e di indentazione. Raffreddare la soluzione a temperatura ambiente e attendere che la soluzione si solidifichi.
2. Test MRE
- Mettere la piastra vibrante sopra il fantasma di gelatina. Assicurarsi che il contatto tra il fantasma e la piastra vibrante sia saldo (Figura 1A).
NOTA: La piastra vibrante è realizzata in poliammide con una dimensione di 50 x 50 x 5 mm3. - Posizionare il fantasma di gelatina nella bobina della testa. Metti spugne e sacchetti di sabbia intorno al fantasma di gelatina per assicurarti che il fantasma sia saldamente posizionato. Utilizzare un attuatore elettromagnetico costruito su misura con una barra di trasmissione14,18. Montare un attuatore elettromagnetico sulla bobina della testa. Collegare la barra di trasmissione alla piastra vibrante (Figura 1B).
- Collegare le linee di alimentazione dell'attuatore con l'amplificatore. Collegare le linee di controllo con il controller (Figura 1C).
- Impostazioni dei parametri di scansione dell'attuatore e della risonanza magnetica
- Impostare la forma d'onda, la frequenza di vibrazione e l'ampiezza nel generatore di funzioni. Impostare l'ampiezza di vibrazione desiderata regolando l'amplificatore di potenza.
NOTA: Qui, la forma d'onda è impostata su sinusoidale nel generatore di funzioni; la frequenza di vibrazione è impostata su 40 Hz o 50 Hz e l'ampiezza è impostata su 1,5 Vpp. Nell'amplificatore di potenza, il rapporto di amplificazione è impostato al 40%. - Impostare il generatore di funzioni in modo che funzioni in modalità trigger. Collegare la linea di trigger alla porta di trigger esterna della macchina MRI.
- Impostare la frequenza di scansione MRE (attuatore) uguale a quella del generatore di funzioni, in modo che il gradiente di codifica del movimento sia sincronizzato con il movimento della piastra vibrante.
- Impostare la forma d'onda, la frequenza di vibrazione e l'ampiezza nel generatore di funzioni. Impostare l'ampiezza di vibrazione desiderata regolando l'amplificatore di potenza.
- Misurazione e analisi dei dati
- Seguire le procedure di posizionamento delle immagini di routine. Utilizzare una sequenza MRE basata su gradiente 2D (GRE) per l'imaging del fantasma di gelatina20. Impostare i parametri di imaging GRE-MRE come segue: Angolo di rotazione = 30°; TR/TE = 50/31 ms; Campo visivo = 300 mm; Spessore fetta = 5 mm; Dimensioni voxel = 2,34 x 2,34 mm2.
- Misurare le immagini di fase in quattro punti temporali in un ciclo sinusoidale. Applicate gradienti di codifica del movimento sia positivi che negativi in ogni punto temporale.
- In base all'immagine di fase acquisita, rimuovere la fase di sfondo sottraendo le immagini di fase codificate positive e negative. Scartare la fase con un algoritmo basato sull'ordinamento dell'affidabilità21.
- Estraete la componente principale del movimento applicando la trasformata di Fourier veloce alle immagini di fase senza wrapping. Filtrare l'immagine di fase con un filtro passa banda digitale. Stimare il modulo di taglio con un algoritmo di inversione diretta (DI) 2D per ottenere il modulo di archiviazione G' e il modulo di perdita G''13,14.
NOTA: la frequenza di taglio del filtro passa banda è [0,04 0,08]. La dimensione della finestra di raccordo dell'algoritmo DI è 11 x 11.
3. Prova di indentazione
- Utilizzare un punzone circolare o una lama chirurgica per tagliare il fantasma di gelatina in un campione cilindrico o cuboide, rispettivamente. Assicurarsi che lo spessore del campione sia compreso tra 3 e 10 mm e che il diametro del campione cilindrico o del lato lungo del cuboide sia maggiore di 4 mm. Utilizzare una lama affilata per tagliare la superficie del campione in modo da renderlo il più liscio possibile per la rientranza.
- Accendere il tester di rientro. Eseguire le seguenti operazioni utilizzando il programma di controllo del penetratore progettato per automatizzare la procedura di contatto dell'indentatore (programma personalizzato; vedere Tabella dei materiali).
- Fare clic sul pulsante Back off nella GUI per inizializzare il processo di calibrazione (Figura 2B). Leggere il valore dal sensore laser e digitare il valore nella casella BaseLine .
NOTA: durante il processo di calibrazione, la distanza tra il sensore laser e la piastra deflettore viene regolata su un valore predefinito specifico. - Posizionare un vetrino sulla piastra del deflettore e registrare il valore mostrato dal sensore laser. Quindi, mettere il campione sul vetrino e posizionarli insieme sulla piastra del deflettore. Leggere il valore dal sensore laser e digitarlo nella casella Campione+Diapositiva .
NOTA: Il sensore laser viene utilizzato per registrare lo spostamento della rientranza, ma viene anche utilizzato per misurare lo spessore del campione prima del test. - Prendiamo la differenza tra i due valori ottenuti nel punto 3.2.2 come spessore del campione nella regione di interesse (ROI).
- Posizionare con attenzione il campione insieme al vetrino sottostante proprio sotto il penetratore, quindi fare clic sul pulsante Contatto per avviare il contatto automatico tra il penetratore e la superficie del campione.
NOTA: se il contatto automatico non è soddisfacente, cioè se il penetratore preme in profondità nel campione o non ha un contatto, regolare la posizione del penetratore digitando un valore compreso tra 0,05 e 0,1 mm nella casella Offset e ripetere i passaggi 1.2.1-1.2.4. - Sulla base dello spessore del campione misurato (fase 3.2.3), stimare lo spostamento di indentazione (cioè la profondità totale di indentazione) moltiplicando lo spessore con la deformazione di prova rientrata (qui, è impostato su ≤8% per mantenere l'impronta all'interno dell'ipotesi di piccola deformazione).
- Digitare i valori di spostamento (passo 3.2.5) nella casella Spostamento (mm). Impostare il tempo di rilassamento su 180 s nella casella Tempo di permanenza . Fare clic sul pulsante Rientro . Lo spostamento e la forza reattiva durante la procedura di ramp-hold verranno automaticamente registrati e salvati in un file nel percorso del file specificato.
NOTA: il percorso del file può essere predefinito come percorso per il salvataggio dei dati di test.
- Fare clic sul pulsante Back off nella GUI per inizializzare il processo di calibrazione (Figura 2B). Leggere il valore dal sensore laser e digitare il valore nella casella BaseLine .
- Esportare i dati di rientro in un foglio di calcolo. Utilizzare una serie
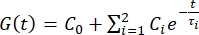 di Prony a due termini per adattarsi alla curva di rilassamento della forza 1,3,11:
di Prony a due termini per adattarsi alla curva di rilassamento della forza 1,3,11: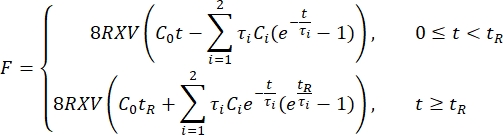
- Stimare il modulo di taglio istantaneo (G 0) e il modulo di taglio a lungo termine (G∞) in base ai parametri adattati:
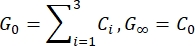
NOTA: Nelle equazioni precedenti, C 0, C i e τi sono parametri del modello della serie di Prony, F è la forza di indentazione, R è il raggio del penetratore, X è il fattore di compensazione per l'ipotesi del mezzo spazio infinito, V è la velocità di indentazione, t è la variabile temporale e t R è il tempo di rampa.
Risultati
Seguendo il protocollo MRE, è stata osservata una chiara propagazione dell'onda di taglio nei fantocci di gelatina a 40 e 50 Hz (Figura 3). Le proprietà viscoelastiche misurate dalla MRE e i test di indentazione sono mostrati nella Figura 4. I valori stimati di G' e G" ad ogni prova per ciascun fantasma sono riassunti nella Tabella 2. Seguendo il protocollo di indentazione, le proprietà viscoelastiche di ciascun fantoccio in ciascun punto di ...
Discussione
I fantocci di gelatina sono comunemente usati come materiali che imitano i tessuti per testare e convalidare algoritmi e dispositivi 17,19,22,23,24,25,26,27. Uno degli studi pionieristici che utilizzano il fantasma di gelatina per confrontare MRE e test di ...
Divulgazioni
Gli autori non dichiarano conflitti di interesse.
Riconoscimenti
È riconosciuto il sostegno finanziario della National Natural Science Foundation of China (grant 31870941), Natural Science Foundation of Shanghai (grant 22ZR1429600) e della Science and Technology Commission of Shanghai Municipality (grant 19441907700).
Materiali
| Name | Company | Catalog Number | Comments |
| 24-channel head & Neck coil | United Imaging Healthcare | 100120 | Equipment |
| 3T MR Scanner | United Imaging Healthcare | uMR 790 | Equipment |
| Acquisition board | Advantech Co | PCI-1706U | Equipment |
| Computer-Windows | HP | 790-07 | Equipment |
| Electromagnetic actuator | Shanghai Jiao Tong University | Equipment | |
| Function generator | RIGOL | DG1022Z | Equipment |
| Gelatin | CARTE D’OR | Reagent | |
| Glycerol | Vance Bioenergy Sdn.Bhd | Reagent | |
| Indenter control program | custom-designed | Software; accessed via: https://github.com/aaronfeng369/FengLab_indentation_code. | |
| Laser sensor | Panasonic | HG-C1050 | Equipment |
| Load cell | Transducer Technique | GSO-10 | Equipment |
| MATLAB | Mathworks | Software | |
| Power amplifier | Yamaha | A-S201 | Equipment |
| Voice coil electric motor | SMAC Corporation | DB2583 | Equipment |
Riferimenti
- Qiu, S., et al. Viscoelastic characterization of injured brain tissue after controlled cortical impact (CCI) using a mouse model. Journal of Neuroscience Methods. 330, 108463 (2020).
- Garcia, K. E., et al. Dynamic patterns of cortical expansion during folding of the preterm human brain. Proceedings of the National Academy of Sciences of the United States of America. 115 (12), 3156-3161 (2018).
- Qiu, S., et al. Characterizing viscoelastic properties of breast cancer tissue in a mouse model using indentation. Journal of Biomechanics. 69, 81-89 (2018).
- Yin, Z., et al. A new method for quantification and 3D visualization of brain tumor adhesion using slip interface imaging in patients with meningiomas. European Radiology. 31 (8), 5554-5564 (2021).
- Streitberger, K. -. J., et al. How tissue fluidity influences brain tumor progression. Proceedings of the National Academy of Sciences of the United States of America. 117 (1), 128 (2020).
- Bunevicius, A., Schregel, K., Sinkus, R., Golby, A., Patz, S. REVIEW: MR elastography of brain tumors. NeuroImage: Clinical. 25, 102109 (2020).
- Namani, R., et al. Elastic characterization of transversely isotropic soft materials by dynamic shear and asymmetric indentation. Journal of Biomechanical Engineering. 134 (6), 061004 (2012).
- Potter, S., et al. A novel small-specimen planar biaxial testing system with full in-plane deformation control. Journal of Biomechanical Engineering. 140 (5), 0510011 (2018).
- Zhang, W., Feng, Y., Lee, C. -. H., Billiar, K. L., Sacks, M. S. A generalized method for the analysis of planar biaxial mechanical data using tethered testing configurations. Journal of Biomechanical Engineering. 137 (6), 064501 (2015).
- Delaine-Smith, R. M., Burney, S., Balkwill, F. R., Knight, M. M. Experimental validation of a flat punch indentation methodology calibrated against unconfined compression tests for determination of soft tissue biomechanics. Journal of the Mechanical Behavior of Biomedical Materials. 60, 401-415 (2016).
- Chen, Y., et al. Comparative analysis of indentation and magnetic resonance elastography for measuring viscoelastic properties. Acta Mechanica Sinica. 37 (3), 527-536 (2021).
- Garteiser, P., Doblas, S., Van Beers, B. E. Magnetic resonance elastography of liver and spleen: Methods and applications. NMR in Biomedicine. 31 (10), 3891 (2018).
- Arani, A., Manduca, A., Ehman, R. L., Huston Iii, J. Harnessing brain waves: a review of brain magnetic resonance elastography for clinicians and scientists entering the field. British Journal of Radiolology. 94 (1119), 20200265 (2021).
- Qiu, S., et al. An electromagnetic actuator for brain magnetic resonance elastography with high frequency accuracy. NMR in Biomedicine. 34 (12), 4592 (2021).
- Hiscox, L. V., et al. Standard-space atlas of the viscoelastic properties of the human brain. Human Brain Mapping. 41 (18), 5282-5300 (2020).
- Seyedpour, S. M., et al. Application of magnetic resonance imaging in liver biomechanics: A systematic review. Frontiers in Physiology. 12, 733393 (2021).
- Okamoto, R. J., Clayton, E. H., Bayly, P. V. Viscoelastic properties of soft gels: comparison of magnetic resonance elastography and dynamic shear testing in the shear wave regime. Physics in Medicine and Biology. 56 (19), 6379-6400 (2011).
- Feng, Y., et al. A multi-purpose electromagnetic actuator for magnetic resonance elastography. Magnetic Resonance Imaging. 51, 29-34 (2018).
- Zeng, W., et al. Nonlinear inversion MR elastography with low-frequency actuation. IEEE Transactions on Medical Imaging. 39 (5), 1775-1784 (2020).
- Wang, R., et al. Fast magnetic resonance elastography with multiphase radial encoding and harmonic motion sparsity based reconstruction. Physics in Medicine and Biology. 67 (2), (2022).
- Herraez, M. A., Burton, D. R., Lalor, M. J., Gdeisat, M. A. Fast two-dimensional phase-unwrapping algorithm based on sorting by reliability following a noncontinuous path. Applied Optics. 41 (35), 7437-7444 (2002).
- Gordon-Wylie, S. W., et al. MR elastography at 1 of gelatin phantoms using 3D or 4D acquisition. Journal of Magnetic Resonance. 296, 112-120 (2018).
- McGarry, M., et al. Uniqueness of poroelastic and viscoelastic nonlinear inversion MR elastography at low frequencies. Physics in Medicine and Biology. 64 (7), 075006 (2019).
- Zampini, M. A., Guidetti, M., Royston, T. J., Klatt, D. Measuring viscoelastic parameters in Magnetic Resonance Elastography: a comparison at high and low magnetic field intensity. Journal of the Mechanical Behavior of Biomedical Materials. 120, 104587 (2021).
- Ozkaya, E., et al. Brain-mimicking phantom for biomechanical validation of motion sensitive MR imaging techniques. Journal of the Mechanical Behavior of Biomedical Materials. 122, 104680 (2021).
- Guidetti, M., et al. Axially- and torsionally-polarized radially converging shear wave MRE in an anisotropic phantom made via Embedded Direct Ink Writing. Journal of the Mechanical Behavior of Biomedical Materials. 119, 104483 (2021).
- Badachhape, A. A., et al. The relationship of three-dimensional human skull motion to brain tissue deformation in magnetic resonance elastography studies. Journal of Biomechanical Engineering. 139 (5), 0510021 (2017).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon