Method Article
Ricostituzione e caratterizzazione di compositi actina-microtubuli con dinamica e meccanica azionata da motori sintonizzabili
In questo articolo
Erratum Notice
Riepilogo
Questo articolo presenta protocolli per l'ingegnerizzazione e la caratterizzazione di reti composite tridimensionali sintonizzabili di filamenti di actina co-entangled e microtubuli. I compositi subiscono una ristrutturazione attiva e un movimento balistico, guidati dai motori della miosina II e della chinesina, e sono sintonizzati dalle concentrazioni relative di actina, microtubuli, proteine motorie e reticolanti passivi.
Abstract
Il citoscheletro composito, che comprende reti interagenti di filamenti di actina semiflessibili e microtubuli rigidi, ristruttura e genera forze utilizzando proteine motorie come la miosina II e la kinesina per guidare processi chiave come migrazione, citochinesi, adesione e meccanorilevamento. Mentre le interazioni actina-microtubuli sono fondamentali per la versatilità e l'adattabilità del citoscheletro, la comprensione della loro interazione con l'attività della miosina e della chinesina è ancora nascente. Questo lavoro descrive come progettare reti composite tridimensionali sintonizzabili di filamenti di actina co-entangled e microtubuli che subiscono una ristrutturazione attiva e un movimento balistico, guidati da motori di miosina II e kinesina, e sono sintonizzati dalle concentrazioni relative di actina, microtubuli, proteine motorie e reticolanti passivi. Vengono inoltre dettagliati i protocolli per l'etichettatura a fluorescenza dei microtubuli e dei filamenti di actina per visualizzare in modo più efficace la ristrutturazione e il movimento composito utilizzando l'imaging confocale multispettrale. Infine, vengono presentati i risultati dei metodi di analisi dei dati che possono essere utilizzati per caratterizzare quantitativamente la struttura, la dinamica e la meccanica del non equilibrio. Ricreare e studiare questa piattaforma biomimetica sintonizzabile fornisce preziose informazioni su come l'attività motoria accoppiata, la meccanica composita e la dinamica dei filamenti possano portare a una miriade di processi cellulari dalla mitosi alla polarizzazione alla meccano-sensazione.
Introduzione
Il citoscheletro è una rete composita dinamica di biopolimeri interagenti che fornisce supporto strutturale e meccanico alle cellule. I motori molecolari associati e le proteine leganti ristrutturano e adattano il citoscheletro per consentire alle cellule di crescere, cambiare forma, irrigidirsi, muoversi e persino auto-guarirsi, consentendo una miriade di processi cellulari che vanno dalla migrazione e divisione al meccanorilevamento 1,2. Oltre al suo significato nella biofisica cellulare, il citoscheletro è anche un esempio per eccellenza di materia attiva con potenziali applicazioni di materiali che vanno dalla guarigione delle ferite e dalla somministrazione di farmaci alla filtrazione e alla robotica morbida 1,3,4,5,6,7,8,9.
Le due caratteristiche chiave che conferiscono al citoscheletro la sua diversità strutturale e meccanica unica e la sua multifunzionalità sono: 1) la sua natura composita, comprendente più filamenti proteici interagenti, come filamenti di actina semiflessibili e microtubuli rigidi, nonché le loro proteine di legame e reticolazione associate 3,5,10; e 2) la sua capacità di ristrutturare, spostare, grossolano ed eseguire continuamente il lavoro attraverso motori che consumano energia, come miosine e chinesine, spingendo e tirando le proteine filamentose 1,7,11,12,13. Mentre questa elegante complessità consente al citoscheletro di mediare processi diversi come la motilità cellulare, la citochinesi e la guarigione delle ferite 3,6,7,11, ostacola la capacità dei ricercatori di riprodurre le caratteristiche della firma in vivo del citoscheletro in sistemi in vitro ricostituiti.
Gli attuali sforzi di ricostituzione delle frontiere si concentrano su compositi di filamenti di actina e microtubuli entangled e cross-linked 3,10,14,15,16,17, reti di actomiosina che generano forza2,8,18,19,20,21 e nematica attiva guidata da kinesin-microtubuli interazioni 22,23,24,25,26. È stato dimostrato che i compositi actina-microtubuli allo stato stazionario mostrano proprietà meccaniche emergenti15,16,27, come una maggiore mobilità del filamento e una maggiore rigidità rispetto ai sistemi monocomponenti 27. Studi su sistemi di actomiosina in vitro hanno riportato una vasta gamma di proprietà strutturali e dinamiche che dipendono dalle concentrazioni di actina, miosina e reticolanti 28,29,30,31. Ad esempio, con sufficiente reticolazione, le reti di actomiosina subiscono una contrazione e una rottura su larga scala 2,28,30,32,33,34,35,36, mentre senza reticolanti, le reti mostrano un flusso rapido e destabilizzante e si rompono 19,29 . È stato riportato che la nematica attiva ricostituita basata su microtubuli che utilizza gruppi di motori kinesina per reticolare e tirare su fasci di microtubuli mostra flussi turbolenti di lunga durata, estensione, instabilità, fratturazione e guarigione 12,22,23,24,25,37,38,39,40,41, 42,43,44,45,46,47.
Più recentemente, i compositi actina-microtubuli guidati da mini-filamenti di miosina II hanno dimostrato di portare a una contrazione più ordinata e all'integrità della rete rispetto al flusso disordinato e alla rottura della rete che le reti di actomiosina senza reticolanti mostrano 17,26,48. Inoltre, la combinazione di robustezza composita e generazione di forza è ottimizzata quando actina e microtubuli sono presenti a concentrazioni comparabili. Le principali caratteristiche emergenti in questa regione dello spazio di formulazione includono una maggiore resistenza meccanica 26, il movimento coordinato di actina e microtubuli26, una contrazione costante e sostenuta e una ristrutturazione a mesoscala17.
Qui, vengono descritti protocolli per progettare e sintonizzare compositi co-entangled e cross-linked di microtubuli e filamenti di actina che vengono spinti fuori equilibrio da mini-filamenti di miosina II e cluster di chinesina che agiscono rispettivamente su filamenti di actina e microtubuli (Figura 1). La dinamica, la struttura e la meccanica di questa classe di compositi possono essere regolate dalle concentrazioni relative dei filamenti, dei motori e dei reticolanti per mostrare un ricco spazio di fasi di flusso avvettivo e turbolento, contrazione isotropa, accelerazione, decelerazione, demiscelazione, irrigidimento, rilassamento e rottura. L'obiettivo di questo lavoro è la preparazione e la messa a punto di questa classe di compositi citoscheletrici attivi. Tuttavia, per aiutare i ricercatori a confrontare e caratterizzare i compositi attivi descritti, vengono dettagliati anche metodi di imaging efficaci che utilizzano la microscopia confocale multispettrale. Infine, vengono presentati i risultati dei principali metodi di analisi computazionale che possono essere utilizzati per misurare la dinamica, la struttura e la meccanica dei compositi. I ricercatori sono incoraggiati ad adottare questi metodi - che includono la microscopia dinamica differenziale (DDM), l'autocorrelazione dell'immagine spaziale (SIA) e la velocimetria dell'immagine delle particelle (PIV) - poiché sono stati ottimizzati per caratterizzare la dinamica complessa e la diversità strutturale dei compositi 17,26,49.
I passaggi descritti di seguito si concentrano sulla preparazione dei compositi e sull'imaging mediante microscopia confocale. I protocolli che descrivono l'analisi dei dati post-acquisizione e le misurazioni delle pinzette ottiche possono essere trovati nei precedenti lavori 17,26,48,50 e forniti su richiesta. Tutti i materiali sono elencati nella tabella dei materiali fornita.
Protocollo
1. Preparare vetrini di copertura silanizzati e vetrini per microscopio per prevenire l'adsorbimento di proteine sulle superfici della camera
NOTA: questo è un processo di 2 giorni. I vetrini silanizzati possono essere preparati fino a 1 mese prima dell'uso.
- Posizionare n. 1 coprivetrini (24 mm x 24 mm) e vetrini per microscopio (1 in x 3 in) in un rack designato che si adatti al pulitore al plasma. Posizionare il rack nel pulitore al plasma e correre per 20 minuti.
- Trasferire i vetrini e le guide in un nuovo rack designato solo per l'uso con silano e posizionare il rack in un contenitore di vetro per pulire i bicchieri come descritto di seguito.
- Immergere coprivetrini e vetrini in acetone al 100% per 1 ora. Immergere coprivetrini e vetrini in etanolo al 100% per 10 minuti.
- Immergere i coperchi e gli scivoli in acqua deionizzata (DI) per 5 min. Ripeti i passaggi di pulizia altre due volte.
- Immergere le copertine e le guide in 0,1 M KOH appena preparati per 15 minuti. Immergere le copertine e le diapositive in DI fresco per 5 minuti. Ripetere questo passaggio altre due volte.
- Coprivetrini e scivoli asciugati all'aria per 10 min. Trattare i vetrini e i vetrini puliti con silano per produrre superfici idrofobiche come descritto di seguito.
NOTA: completare i seguenti passaggi in una cappa aspirante.- Immergere i vetrini e i vetrini essiccati in silano al 2% (sciolto in toluene) per 5 minuti. Utilizzare un imbuto per versare il silano nella sua bottiglia designata per riutilizzare fino a cinque volte.
- Immergere le copertine e le guide in etanolo al 100% per 5 minuti. Sostituire l'etanolo con etanolo fresco. Immergere copertine e scivoli per 5 min.
- Immergere le copertine e le diapositive in DI fresco per 5 minuti. Ripetere il passaggio di lavaggio dell'etanolo e del DI altre due volte utilizzando etanolo fresco e DI ogni volta. Coprivetrini e scivoli asciugati all'aria per 10 min.
2. Preparazione di un composito attivo actina-microtubulo guidato da mini-filamenti di miosina
- Rimuovere la miosina inattiva tramite il legame del filamento di actina ed eseguire il pull-down tramite ultracentrifugazione come descritto di seguito.
- Polimerizzare l'actina in filamenti. Utilizzando una micropipetta di precisione e punte di pipette sterili, combinare in una provetta da microcentrifuga: 1,87 μL di DI, 1,3 μL di 10x G-buffer, 1,3 μL di 10x F-buffer, 1,63 μL di 4 M KCl, 4,53 μL di actina (47,6 μM) e 1,08 μL di 100 μM di falloidina.
NOTA: Per garantire una polimerizzazione sufficiente, la concentrazione di actina e il rapporto molare actina:falloidina devono essere rispettivamente di 18,4 μM e 2:1. - Pipe delicatamente la soluzione su e giù per mescolare e poi mettere in ghiaccio al buio per ≥1 ora. Ultracentrifuga fredda a 4 °C. Togliere l'aliquota di miosina da -80 °C e metterla in ghiaccio.
NOTA: completare il passaggio 2.2 a questo punto mentre l'actina è polimerizzante. - Dopo ≥1 h di polimerizzazione dell'actina, aggiungere 1,3 μL di 10 mM di ATP e 2 μL di 19 μM di miosina polimerizzata.
NOTA: Il rapporto molare actina/miosina deve essere >5 per garantire una rimozione sufficiente dei motori della miosina inattivi (cioè teste morte). - Pipe delicatamente la soluzione su e giù per mescolare. Trasferimento in una provetta di qualità ultracentrifuga.
Centrifugare a 4 °C e 121,968 x g per 30 min.
- Polimerizzare l'actina in filamenti. Utilizzando una micropipetta di precisione e punte di pipette sterili, combinare in una provetta da microcentrifuga: 1,87 μL di DI, 1,3 μL di 10x G-buffer, 1,3 μL di 10x F-buffer, 1,63 μL di 4 M KCl, 4,53 μL di actina (47,6 μM) e 1,08 μL di 100 μM di falloidina.
- Preparare una rete composita co-entangled di filamenti di actina e microtubuli come descritto di seguito.
NOTA: Iniziare 30 minuti prima dello spin-down della miosina (punto 2.1.4).- Impostare un blocco termico a 37 °C. Utilizzare una micropipetta di precisione e punte di pipette sterili per aggiungere quanto segue a una provetta per microcentrifuga: 13,9 μL di PEM, 3 μL di 1% Tween20, 1,55 μL di 47,6 μM di actina, 0,36 μL di 34,8 μM di R-actina, 0,3 μL di 250 mM ATP, 0,87 μL di 100 μM di falloidina, 1,91 μL di 5-488-tubulina, 0,3 μL di 100 mM GTP, e 0,75 μL di Taxolo da 200 μM, per un volume totale di 23 μL.
NOTA: Le concentrazioni di actina e tubulina elencate sono per un composito con 2,9 μM di actina e 2,9 μM di tubulina. La concentrazione proteica totale è c = c A + c T = 5,8 μM e la frazione molare di actina è c A/(c A + cT) = Φ A = 0,5. Vedere il passaggio 2.5 per regolare questi valori. - Pipe delicatamente la soluzione su e giù per miscelare e posizionare su un blocco termico a 37 °C al riparo dalla luce per 1 ora.
- Impostare un blocco termico a 37 °C. Utilizzare una micropipetta di precisione e punte di pipette sterili per aggiungere quanto segue a una provetta per microcentrifuga: 13,9 μL di PEM, 3 μL di 1% Tween20, 1,55 μL di 47,6 μM di actina, 0,36 μL di 34,8 μM di R-actina, 0,3 μL di 250 mM ATP, 0,87 μL di 100 μM di falloidina, 1,91 μL di 5-488-tubulina, 0,3 μL di 100 mM GTP, e 0,75 μL di Taxolo da 200 μM, per un volume totale di 23 μL.
- Preparare le camere campione per gli esperimenti di imaging confocale come descritto di seguito.
NOTA: completare i passaggi 2.1.4 e 2.2.2 durante i periodi di attesa.- Posizionare due vetrini silanizzati affiancati su una piastra riscaldante (spenta), posare due strisce di pellicola sigillante termoplastica attraverso i vetrini a ~ 3 mm di distanza e posizionare due vetrini di copertura silanizzati sul film sigillante termoplastico per formare una camera di campionamento.
- Ruotare la piastra riscaldante sull'impostazione bassa fino a quando i coperchi non si legano saldamente ai vetrini con pellicola sigillante termoplastica fusa (~1-2 min). Premere verso il basso con una pressione uniforme per garantire l'incollaggio mantenendo una spaziatura di ~ 100 μm tra le due superfici.
- Rimuovere le camere e spegnere la piastra calda. Camere di etichettatura con (+) e (-). La camera (+) sarà per il campione attivo (con miosina) e la camera (-) sarà il controllo (senza miosina). Assicurarsi che ogni camera possa contenere ≤10 μL di fluido.
- Preparare i campioni per l'immagine come descritto di seguito.
Nota : è importante completare questo passaggio immediatamente dopo il completamento dei passaggi 2.1 e 2.2.- Rimuovere con cautela il campione di miosina-actina dall'ultracentrifuga (fase 2.1.4) e convogliare immediatamente i 7,5 μL superiori del surnatante e trasferirli in una nuova provetta da microcentrifuga.
- Rimuovere il campione di microtubuli di actina dal blocco termico e mescolare delicatamente 1,5 μL di 10x D-glucosio, 1,5 μL di 10x GOC e 1,5 μL di 1 mM di blebbistatina. Dividere la soluzione in due aliquote da 13,7 μL ed etichettare come (+) e (-).
- Miscelare 1,28 μL di surnatante dalla fase 2.4.1 all'aliquota (+). Mescolare 1,28 μL di DI all'aliquota (-). Far fluire lentamente ogni soluzione nella camera corrispondente (fase 2.3) tramite azione capillare. Fare attenzione a non introdurre bolle d'aria nel canale.
- Sigillare le due estremità aperte di ciascun canale con colla epossidica o UV ad asciugatura rapida. Assicurarsi che l'adesivo sia completamente asciutto prima di posizionarlo sul microscopio. Immagine immediatamente come descritto al punto 3.
NOTA: la colla UV è vantaggiosa perché polimerizza quasi istantaneamente dopo l'esposizione ai raggi UV. Tuttavia, poiché la blebbistatina è sensibile ai raggi UV, è importante illuminare solo localmente la colla (ai bordi della camera del campione) utilizzando una piccola bacchetta UV per evitare di disattivare la blebbistatina.
- Opzionale: variare le concentrazioni proteiche per sintonizzare la dinamica e la struttura dei compositi.
NOTA: I seguenti passaggi sono suggeriti modifiche ai passaggi precedenti per variare le concentrazioni di actina, microtubuli e miosina, se lo si desidera.- Seguire i passaggi descritti sopra, ad eccezione delle seguenti modifiche nei punti 2.2.1 e 2.4.3.
- Per variare le concentrazioni di actina e microtubuli, regolando così c e ΦA, aumentare o diminuire il volume di actina, R-actina e 5-488-tubulina utilizzati nella fase 2.2.1, come desiderato26. Quando si varia la concentrazione di actina, regolare proporzionalmente le concentrazioni molari di R-actina e falloidina per mantenere gli stessi rapporti molari con l'actina. Regolare il volume di PEM in modo tale che il volume finale della miscela rimanga di 23 μL. Tutti gli altri volumi e concentrazioni dei componenti rimangono gli stessi.
- Per variare la concentrazione di miosina, regolare il volume di miosina aggiunto all'aliquota (+) al punto 2.4.3 come desiderato. Regolare di conseguenza il volume DI aggiunto all'aliquota (-). Regolare il volume PEM al punto 2.2.1 per tenere conto dell'aumento o della diminuzione del volume di miosina (+) e DI (-), assicurandosi che il volume finale di ciascun campione ((+) e (-)) sia di 14,98 μL.
3. Imaging e caratterizzazione di compositi attivi mediante microscopia confocale
- Per visualizzare i compositi actomiosina-microtubuli preparati nella fase 2, utilizzare un microscopio confocale a scansione laser (LSCM) o un microscopio simile, con un obiettivo di immersione in olio 60x 1,4 NA. Per visualizzare simultaneamente filamenti di actina e microtubuli in canali di fluorescenza separati, utilizzare un laser da 561 nm con filtri di eccitazione/emissione da 565/591 nm e un laser da 488 nm con filtri di eccitazione/emissione da 488/525 nm.
- Posizionare la camera del campione sul microscopio in modo che il canale di controllo sia posizionato direttamente sopra l'obiettivo. Assicurarsi che ci sia un'interfaccia olio tra l'obiettivo e il coprifoglio.
- Utilizzate i controlli di scena per mettere a fuoco il composito di controllo, quindi trovate entrambe le superfici della camera di campionamento. Spostare la posizione z al centro della camera del campione. Verificare la presenza di reti filamentose chiare come mostrato nella Figura 2.
- Sempre visualizzando la camera di controllo, regolare l'intensità di ciascun laser per consentire la visualizzazione simultanea di filamenti di actina e microtubuli. Mantenere la più bassa intensità laser possibile per prevenire il fotosbiancamento (più prevalente nel canale dell'actina) e il sanguinamento (tipicamente dai microtubuli nel canale dell'actina).
- Per caratterizzare il campione di controllo inattivo, raccogliere tre serie temporali (video) di 256 x 256 pixel quadrati (213 μm x 213 μm) immagini a 2,65 fps per un totale di ≥1000 fotogrammi. Raccogliere ogni serie temporale in una regione diversa della camera del campione separata da ≥500 μm. Assicurarsi che vi sia un movimento rilevabile minimo e nessun flusso o ristrutturazione.
- Spegnere il laser a 488 nm e utilizzare i controlli del palcoscenico per passare alla camera (+).
- Utilizzando il laser a 568 nm, visualizzare i microtubuli nel canale (+) per garantire la corretta formazione della rete (Figura 2) e identificare il centro assiale della camera del campione (che può essere diverso dalla posizione z centrale della camera di controllo).
- Accendere il laser a 488 nm e ripetere il passaggio 3.5 precedente con le seguenti modifiche. Raccogli le serie temporali per un massimo di 45 minuti, interrompendo l'acquisizione quando il campione si sposta fuori dal campo visivo, si rompe o si sbianca. Registrare 5-10 serie temporali e tenere traccia dell'ora in cui ogni serie temporale inizia rispetto all'inizio della prima serie temporale.
- Analizzare i dati utilizzando DDM, SIA e PIV come descritto nella Figura 3, nella Figura 4, nella Figura 5 e in precedenza 17,48,50,51.
NOTA: Il laser a 488 nm attiva localmente l'attività della miosina ATPasi disattivando la blebbistatina, quindi dovrebbe essere attivato solo all'inizio dell'acquisizione dei dati in modo tale che t = 0 sia all'inizio della serie temporale. Questi parametri di acquisizione sono ottimizzati per l'analisi della microscopia dinamica differenziale (DDM) come fatto in precedenza26.
4. Preparazione di compositi attivi actina-microtubuli azionati da motori kinesin
NOTA: I seguenti passaggi creano compositi actina-microtubuli che vengono spinti fuori equilibrio dai motori della chinesina o da una combinazione di chinesina e miosina50.
- Preparare i motori kinesina e miosina come descritto di seguito.
- Se si incorpora miosina, seguire il passaggio 2.1.
- Per formare gruppi motori di kinesina che legano ed esercitano forze tra coppie di microtubuli, utilizzare una micropipetta e punte di pipette sterili per aggiungere quanto segue a una provetta sterile da 1,5 mL di microcentrifuga: 1,16 μL PEM, 2,74 μL 8,87 μM dimeri di kinesina, 7,29 μL 83,3 μM NeutrAvidin, 0,81 μL 2mM DTT. Mescolare delicatamente pipettando la soluzione su e giù e incubare al riparo dalla luce (utilizzare una provetta da microcentrifuga nera o avvolgere in un foglio) per 30 minuti a 4 °C.
NOTA: Il rapporto molare tra dimeri di kinesina e NA è 1:25.
- Seguire il passaggio 2.3 per preparare le camere campione e creare tre camere invece di due. Eseguire questa fase durante l'incubazione della kinesina (fase 4.1.2) e l'ultracentrifugazione della miosina (fase 4.1.1).
- Preparare una rete composita co-intrecciata di filamenti di actina e microtubuli.
- Impostare il blocco termico a 37 °C. Utilizzare una micropipetta e punte di pipette sterili per aggiungere quanto segue a una provetta sterile da 1,5 mL di microcentrifuga: 3,21 μL PEM, 4,5 μL di 1% Tween20, 2,18 μL di 47,6 μM di actina, 3,46 μL di 5-R-tubulina, 4,5 μL di 100 mM ATP, 4,5 μL di 10 mM GTP, 1,13 μL di 200 μM Taxol e 1,57 μL di 20 μM 488-falloidina. Assicurarsi che il volume totale sia di 25 μL.
- Versare delicatamente la soluzione su e giù per miscelare e posizionarla sul blocco termico a 37 °C al riparo dalla luce per 1 ora. Rimuovere il tubo dal blocco termico e utilizzare una micropipetta per miscelare delicatamente 0,84 μL di falloidina da 100 μM. Incubare per 5-10 minuti a temperatura ambiente, al riparo dalla luce.
NOTA: L'aggiunta di falloidina in questa fase, piuttosto che nel punto 4.3.1, migliora l'etichettatura a fluorescenza dei filamenti di actina, poiché la 488-falloidina non deve competere con la falloidina non marcata per i siti di legame dell'actina.
- Preparare compositi attivi per l'imaging confocale.
- Aggiungere 1,13 μL di 200 μM di blebbistatina, 1,35 μL di 10x Glu e 1,35 μL di 10x GOC alla soluzione dal punto 4.3.2 e mescolare delicatamente pipettando su e giù. Dividere la soluzione in tre aliquote da 10 μL ed etichettarla come (K), (K+M) e (-).
- Mescolare 2,54 μL di miosina dal punto 2.1.4 all'aliquota (K+M). Mescolare in 2,54 μL PEM in (K) e (-) aliquote.
- Utilizzare una micropipetta e punte di pipette sterili per aggiungere 2,5 μL di cluster di kinesina dal punto 4.1.2 alle aliquote (K) e (K+M). Pipet su e giù per mescolare. Miscelare in 2,5 μL PEM a (-) usando la stessa tecnica.
NOTA: Le concentrazioni di actina e tubulina elencate sono per un composito con 2,32 μM di actina e 3,48 μM di tubulina. La concentrazione totale di proteine è c = c A + c T = 5,8 μM e la frazione molare di actina è c A/(c A + cT) = Φ A = 0,4. Le concentrazioni di kinesina e miosina sono rispettivamente 0,35 μM e 0,47 μM. Vedere il punto 2.5 per le linee guida generali per regolare c A, cT, c e ΦA. - Utilizzando una micropipetta, far fluire lentamente ciascuna soluzione nel canale corrispondente delle camere di campionamento preparate (fase 4.2) tramite azione capillare. Spingere verso il basso molto lentamente e delicatamente sul pipet in modo da non introdurre bolle d'aria nel canale.
- Sigillare le due estremità aperte di ciascun canale con colla epossidica o polimerizzabile UV ad asciugatura rapida. Assicurarsi che l'adesivo sia completamente asciutto prima di posizionarlo sul microscopio.
NOTA: è importante che questo passaggio venga eseguito rapidamente per ridurre al minimo il tempo in cui la kinesina agisce senza essere monitorata. Per questo motivo, si consiglia la resina epossidica che polimerizza in 1 minuto (anziché 5 o 10 minuti). La colla polimerizzabile UV è vantaggiosa in questo senso perché polimerizza quasi istantaneamente dopo l'esposizione ai raggi UV.
- Immagine preparata campioni immediatamente, dopo il passaggio 3, ad eccezione delle seguenti importanti modifiche. Poiché la kinesina non è controllata dall'attivazione della luce, inizia a funzionare immediatamente dopo il punto 4.4.3, quindi segna questa volta come t = 0. Per visualizzare il composito il più vicino possibile allo stato di inattività iniziale (t = 0), visualizzare prima i canali (K) e (K+M) e notare il tempo trascorso tra il passaggio 4.4.3 e l'inizio dell'acquisizione dei dati (passaggio 3.8). In pratica, questo tempo trascorso è ~ 5 min.
5. Incorporare reticolanti passivi in compositi attivi
NOTA: Questi passaggi descrivono come utilizzare le subunità biotinilate di actina e tubulina e NeutrAvidin (NA) per reticolare passivamente l'actina all'actina (A-A) o i microtubuli ai microtubuli (M-M) nei compositi attivi descritti al punto 4.
- Preparare complessi reticolanti A-A o M-M con proteine biotinilate (biotina-actina o biotina-tubulina), NA e biotina in un rapporto di 2:2:1 biotina-actina / tubulina: biotina: NA. Avviare questo processo prima del passaggio 4 .
- Per i reticolanti A-A, utilizzare una micropipetta e punte di pipette sterili per aggiungere 2 μL di 11,6 μM di biotina-actina, 1,39 μL di 8,33 μM NA, 2,27 μL di 1,02 μM di biotina e 4,34 μL di PEM a un tubo di microcentrifuga. Mescolare delicatamente pipettando su e giù.
- Per i reticolanti M-M, utilizzare una micropipetta e punte di pipette sterili per aggiungere 1,86 μL di 4,55 μM di biotina-tubulina, 1,11 μL di 8,33 μM NA, 1,82 μL di 1,02 μM di biotina e 5,21 μL di PEM a una provetta per microcentrifuga. Mescolare delicatamente pipettando su e giù.
- Avvolgere il/i tubo/i dei punti 5.1.1 e/o 5.1.2 in pellicola sigillante termoplastica per creare una tenuta stagna. Collocare in una zattera di galleggiamento in un bagno sonicatore a temperatura controllata impostato a 4 °C.
- Sonicare per 90 minuti a 4 °C. In pratica è meglio mettere il sonicatore in una cella frigorifera e aggiungere impacchi di ghiaccio al bagno di sonicazione per mantenere bassa la temperatura.
- Per incorporare complessi reticolanti nei campioni per l'imaging, seguire il passaggio 4.3, modificando il passaggio 4.3.1 come descritto di seguito per la reticolazione A-A (fase 5.2.1) o la reticolazione M-M (fase 5.2.2).
- Per la reticolazione A-A, combinare quanto segue in una provetta per microcentrifuga: 1,94 μL di PEM, 4,50 μL di 1% Tween20, 2,18 μL di 47,6 μM di actina, 3,46 μL di 45,5 μM 5-R-tubulina, 1,13 μL di reticolanti A-A (fase 5.1.1), 4,50 μL di 100 mM ATP, 4,50 μL di 10 mM GTP, 1,13 μL di 200 μM Taxol, e 1,57 μL di 20 μM 488-falloidina. Assicurarsi che il volume totale sia di 25 μL.
- Per la reticolazione M-M, combinare in una provetta per microcentrifuga: 1,97 μL di PEM, 4,50 μL di Tween20 all'1%, 2,18 μL di actina da 47,6 μM, 3,76 μL di 5-R-tubulina da 45,5 μM, 1,13 μL di diluizione 1:4 di reticolanti M-M (fase 5.1.2), 4,50 μL di 100 mM ATP, 4,50 μL di 10 mM GTP, 1,13 μL di 200 μM Taxol, e 1,57 μL di 20 μM 488-falloidina. Assicurarsi che il volume totale sia di 25 μL.
- Seguire i punti 4.3.2-4.5 con le concentrazioni specifiche per un rapporto molare reticolante:actina di RA = 0,02 e un rapporto molare reticolante:tubulina di RT = 0,005. Questi valori di R A e R T danno luogo a lunghezze simili tra i reticolanti lungo i filamenti di actina e i microtubuli (d A 60 nm e d MT 67 nm), stimati utilizzando d A = I monomero/2R A
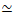 , dove I monomero è la lunghezza di unmonomero di actina, e dMT
, dove I monomero è la lunghezza di unmonomero di actina, e dMT 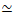 = anello I/26RT,dove I anello è la lunghezza di unanello di 13 tubuline 15, 17.
= anello I/26RT,dove I anello è la lunghezza di unanello di 13 tubuline 15, 17.
Risultati
Per determinare la corretta preparazione di compositi attivi (Figura 1) e per caratterizzarne la dinamica e la struttura, viene utilizzato un microscopio a fluorescenza a scansione laser con almeno due canali di fluorescenza per visualizzare contemporaneamente i filamenti di actina e i microtubuli (Figura 2 e Figura 6). Tutti i filamenti di actina e i microtubuli nei compositi sono scarsamente etichettati, piuttosto che drogati in filamenti brillanti traccianti, come spesso viene fatto negli studi in vitro. Questo metodo garantisce che la dinamica e la struttura misurate siano rappresentative del composito stesso piuttosto che dei traccianti che si formano in condizioni diverse rispetto ai compositi. Per questo motivo, i singoli filamenti di actina e i microtubuli non possono tipicamente essere risolti, piuttosto le immagini ritraggono la struttura della rete a mesoscala (Figura 2 e Figura 6).
Questo approccio di marcatura è stato ottimizzato per l'autocorrelazione delle immagini spaziali (SIA) e la microscopia dinamica differenziale (DDM) che esaminano la dinamica e la struttura nello spazio di Fourier reciproco (Figura 4, Figura 5 e Figura 8)52,53,54,55. La velocimetria particella-immagine (PIV) può anche essere utilizzata per rappresentare e caratterizzare dinamiche e campi di flusso (Figura 3 e Figura 7), ma richiede pixel-binning (risoluzione spaziale inferiore) e incrementi di tempo di ritardo maggiori (risoluzione temporale inferiore) rispetto a SIA e DDM per eliminare vettori errati che derivano dal rumore nelle immagini dense e a basso segnale. Tuttavia, PIV è raccomandato per l'esame qualitativo dei campi di flusso e la conferma dei risultati DDM (Figura 4 e Figura 8)26,50.
La caratterizzazione del campione delle reti descritte utilizzando queste analisi (ad esempio, DDM, SIA, PIV) viene fornita per aiutare i ricercatori ad adottare analisi simili per confrontare e caratterizzare i loro campioni. Tuttavia, le descrizioni dettagliate di queste tecniche esulano dallo scopo di questo lavoro. Per descrizioni dettagliate di come eseguire DDM su questi e altri sistemi simili, incluso il codice Python user-friendly, fare riferimento ai lavori precedenti 17,26,49,50 e ai riferimenti in esso contenuti. Per i dettagli su come eseguire SIA e PIV sui sistemi qui descritti, il lettore è indirizzato ai lavori precedenti17,50.
Diversi controlli, descritti di seguito, dovrebbero essere eseguiti per garantire che i compositi funzionino come previsto. Un composito senza miosina o chinesina dovrebbe apparire essenzialmente statico con fluttuazioni termiche minime o deriva. I filamenti di actina e i microtubuli dovrebbero apparire co-intrecciati e distribuiti in modo omogeneo, con un minimo raggruppamento, aggregazione o separazione di fase di actina e microtubuli in un campo visivo di ~200 μm x 200 μm (Figura 2, estrema sinistra)17. Ci si dovrebbe aspettare un risultato simile per i compositi che contengono miosina ma non sono esposti alla luce di 488 nm (per disattivare la blebbistatina).
Dopo l'incorporazione della miosina e l'esposizione alla luce a 488 nm, i compositi subiscono una contrazione che è in gran parte isotropa e simile per l'actina e i microtubuli, come si vede nelle immagini al microscopio prese prima e dopo l'attività della miosina (Figura 2), così come i corrispondenti campi di flusso PIV per tempi variabili durante l'attività (Figura 3). Per determinare se il moto è balistico, diffusivo, subdiffusivo, ecc., il tempo di decorrelazione caratteristica τ(q) determinato da DDM è valutato in funzione del vettore d'onda (cioè spazio reciproco). Vedere come descritto in dettaglio in precedenza 17,26,49. Nella Figura 4 viene inoltre illustrato come utilizzare DDM per caratterizzare questi materiali compositi. La scala della legge di potenza τ(q)~1/vq β, con β = 1, indica il moto balistico con velocità v. Per riferimento, β = 2 rappresenta la dinamica diffusiva con v come coefficiente di diffusione. Tutti i compositi attivi mostrano una scala balistica (Figura 4A) con velocità che sono regolate dalle concentrazioni di actina e miosina (Figura 4B) e che possono variare nel tempo durante l'attività, accelerando o decelerando (Figura 4C, D).
La ristrutturazione e il clustering della rete, visibili nella Figura 2 e più evidenti per concentrazioni più elevate di actina e miosina, possono essere caratterizzati utilizzando SIA, come illustrato nella Figura 5 e descritto in precedenza 17,48,50. In breve, una lunghezza di correlazione ξ, che è una misura della dimensione caratteristica delle caratteristiche in un'immagine, può essere determinata adattando ogni curva di autocorrelazione dell'intensità spaziale g(r) a una funzione esponenziale della distanza r tra i pixel. Picchi g(r) più grandi che persistono per distanze maggiori indicano caratteristiche strutturali più grandi (ad esempio, raggruppamento, raggruppamento dei singoli filamenti). Come mostrato nella Figura 5, per frazioni di actina più elevate e concentrazioni di miosina, una significativa ristrutturazione e aggregazione si riflette nell'aumento di ξ nel tempo.
Le proprietà viscoelastiche e la risposta meccanica non lineare dei compositi attivi possono anche essere misurate utilizzando la microreologia delle pinzette ottiche (OTM). Tuttavia, i protocolli e i risultati rappresentativi per questi esperimenti sono al di fuori dello scopo di questo lavoro. I lettori interessati sono rimandati ai precedenti lavori48,56 che descrivono dettagliatamente come eseguire le misurazioni OTM e i risultati attesi.
Utilizzando lo stesso programma di strumenti sperimentali e di analisi descritti sopra, la sezione seguente descrive come la dinamica e la struttura cambiano quando i motori kinesina e i reticolanti biotina-NA sono incorporati nei compositi (Figura 6, Figura 7 e Figura 8). La Figura 6 mostra immagini confocali rappresentative di compositi guidati da sola chinesina (K) o kinesina e miosina (K+M), con e senza reticolazione passiva (XL) di filamenti di actina o microtubuli.
L'incorporazione della kinesina nei compositi inizialmente si traduce in dinamiche e ristrutturazioni simili a quelle dei compositi guidati dalla miosina, come si vede nella riga superiore della Figura 7 (Classe 1). Tuttavia, la dinamica passa tipicamente al flusso anisotropo su larga scala (Figura 7 riga centrale, Classe 2), accelerazione e decelerazione (Figura 7 riga inferiore, Classe 3). Queste caratteristiche si accoppiano con il clustering e l'aggregazione su mesoscala dopo 5-30 minuti (Figura 6 e Figura 8B). I campi di flusso generati da PIV e le mappe temporali a colori mostrati nella Figura 7 rappresentano esempi di ristrutturazione isotropica (Classe 1, pannello superiore), flusso diretto (Classe 2, pannelli centrali) e accelerazione bidirezionale (Classe 3, pannelli inferiori).
Le velocità dell'actina e dei microtubuli in diversi punti temporali durante l'attività, determinate tramite adattamenti alle curve τ(q), illustrano l'accelerazione seguita dalla decelerazione (Figura 8), che dipende dalla reticolazione. Come mostrato anche nella Figura 8, quando entrambe le proteine motorie sono incorporate, la dinamica è in realtà più lenta dei compositi solo chinesina, e c'è un inizio ritardato del flusso a mesoscala. La miosina supporta anche una compenetrazione più omogenea delle reti di actina e microtubuli per tutta la durata dell'attività, nonché una minore aggregazione e ristrutturazione. Questi effetti possono essere visti nelle immagini in Figura 6 e sono quantificati dalle lunghezze di correlazione variabili nel tempo calcolate tramite SIA, che sono generalmente più piccole in presenza di miosina (Figura 8B).
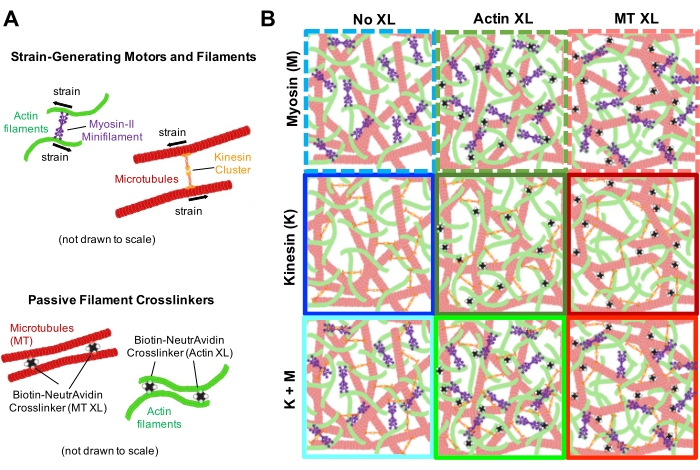
Figura 1. Progettazione e caratterizzazione di compositi attivi actina-microtubuli con motori multipli generatori di forza e reticolanti passivi. (A) I monomeri di actina e i dimeri di tubulina sono copolimerizzati a concentrazioni molari c A e c T di 0,73-11,6 μM e frazioni molari di actina Φ A = c A/ (c A + c T) = 0, 0,25, 0,5, 0,75 e 1, per formare reti co-intrecciate di filamenti di actina (verde) e microtubuli (rosso). La reticolazione passiva si ottiene utilizzando NA per collegare filamenti di actina biotinilati (Actin XL) o microtubuli (MT XL) a rapporti molari crosslinker:proteina di R A = 0,01-0,08 e RMT = 0,001-0,01 per actina e microtubuli, rispettivamente. Mini-filamenti di miosina-II (viola) e cluster di chinesina (arancione), a concentrazioni di c M = 0,12 - 0,48 μM e cK = 0,2 - 0,7 μM, spingono e tirano sui filamenti per spingere i compositi fuori dallo stato stazionario. (B) Schema dello spazio di formulazione. I mini-filamenti di miosina II (M), i cluster di chinesina (K) o entrambi i motori (K + M) sono incorporati in compositi senza reticolanti passivi (No XL), reticoli actina-actina (Actin XL) e reticoli microtubulo-microtubuli (MT XL). Tutti i cartoni animati non sono disegnati in scala. Fare clic qui per visualizzare una versione ingrandita di questa figura.
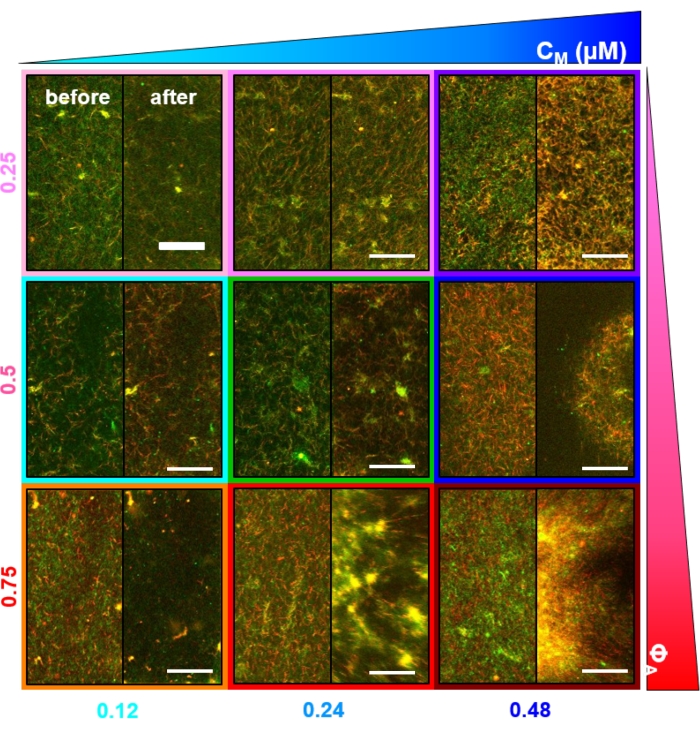
Figura 2. Imaging confocale a due colori di compositi citoscheletri guidati dalla miosina con concentrazioni variabili di miosina cM e frazioni molari di actina ΦA. (A) Le immagini al microscopio confocale a due colori da 256 x 128 pixel quadrati (212 x 106 μm2) mostrano come i compositi di filamenti di actina (verde) e microtubuli (rosso) vengono riorganizzati tramite l'attività motoria della miosina. Non sono presenti motori chinesinici o reticolanti passivi. In ogni pannello vengono mostrate le immagini scattate all'inizio (a sinistra, prima) e alla fine (a destra, dopo) dell'attivazione della miosina di 45 minuti (tramite illuminazione con luce a 488 nm per disattivare la blebbistatina). I pannelli sono ordinati aumentando la concentrazione molare di miosina (cM), andando da sinistra a destra, e aumentando la frazione molare di actina (ΦA), andando dall'alto verso il basso. I colori che delineano ciascun pannello corrispondono alla codifica dei colori utilizzata nella Figura 4 e nella Figura 5. Le barre della scala sono 50 μM. Per catturare al meglio le dinamiche e la struttura per l'analisi, utilizziamo frame rate di 1-5 fps, ROI con lati 50-250 μm e durate di serie temporali di 5-45 minuti, a seconda della velocità di contrazione e riarrangiamento. I pannelli in cui le immagini prima e dopo sembrano simili indicano una ristrutturazione minima, come si vede nei pannelli rosa, magenta e ciano. Il clustering su piccola scala, evidenziato da una maggiore eterogeneità e dalla presenza di caratteristiche punteggiate luminose, può essere visto nei pannelli arancione, verde e rosso. La contrazione su larga scala, vista come una rete uniformemente ridotta, è evidente nei pannelli blu e viola. Questa cifra è stata modificata rispetto al riferimento17. Fare clic qui per visualizzare una versione ingrandita di questa figura.
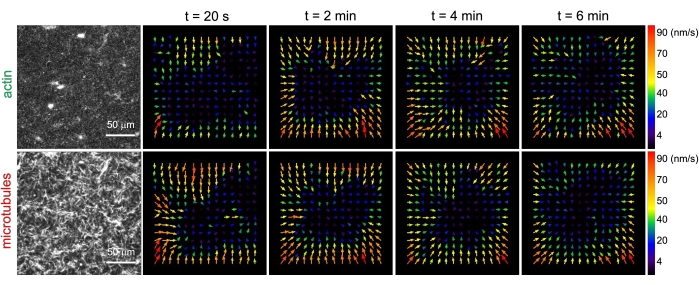
Figura 3. La velocimetria ad immagine di particelle (PIV) mostra che l'attività dell'actomiosina innesca dinamiche contrattili coordinate di actina e microtubuli in compositi co-entangled. Campi di flusso PIV per actina (riga superiore) e microtubuli (riga inferiore) in un composito guidato dalla miosina con (ΦA, cM) = (0,5, 0,24) in tempi crescenti durante una serie temporale di 6 minuti. I campi di flusso sono stati generati utilizzando il plugin Fiji/ImageJ PIV con un lag-time di 20 s e 2 pixel x 2 pixel binning. Sia l'actina che i microtubuli mostrano un movimento costante diretto verso la regione centrale del campo visivo per tutta la durata del film. Le barre della scala in tutte le immagini sono 50 μm. Diversi colori delle frecce corrispondono a velocità diverse, come indicato nella scala dei colori a destra dei campi vettoriali. Questa cifra è stata modificata rispetto al riferimento26. Fare clic qui per visualizzare una versione ingrandita di questa figura.
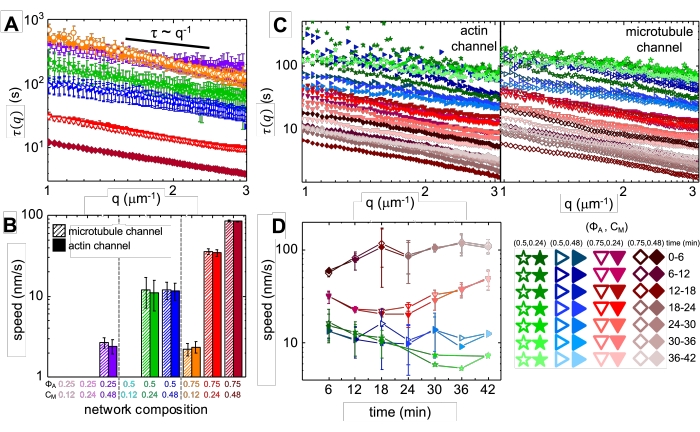
Figura 4. La microscopia dinamica differenziale risolta nel tempo (DDM) misura la velocità e il tipo di movimento dell'actina e dei microtubuli nei compositi attivi. (A) Il DDM viene eseguito sui canali microtubuli (simboli in alto, aperti) e actina (in basso, simboli pieni) di serie temporali per determinare i tempi di decadimento caratteristici τ rispetto al numero d'onda q sia per l'actina (simboli riempiti) che per i microtubuli (simboli aperti) come descritto in precedenza17,26. Tutte le curve seguono la scala τ ~ q-1, che indica il moto balistico, con velocità v che sono determinate tramite adattamenti a τ(q) = (vq)-1. Velocità più elevate corrispondono a valori τ(q) più piccoli per ogni dato q. I colori e le forme dei simboli corrispondono alle combinazioni (Φ A, cM) mostrate in B. (B) Le velocità di contrazione v sono determinate tramite adattamenti alle curve τ(q) mostrate in A, che sono mediate su tutti i tempi di ritardo per la durata di ogni serie storica di 45 minuti. (C) Il DDM risolto nel tempo (trDDM) quantifica come la dinamica varia nel tempo valutando τ(q) per actina (simboli riempiti, a sinistra) e microtubuli (simboli aperti, a destra) per intervalli consecutivi di 6 minuti (indicati da diverse tonalità dello stesso colore) durante il tempo di attivazione di 45 minuti. trDDM viene eseguito per ogni combinazione (ΦA, cM) (indicata da diversi simboli e colori) come descritto nella legenda in basso a destra. Le curve τ(q) mostrate in C seguono scale e tendenze simili a quelle in A, ma mostrano anche una dipendenza dal tempo per alcune composizioni (ΦA, cM), in particolare per ΦA= 0,75. (D) Le velocità di contrazione dei filamenti di actina (simboli chiusi) e dei microtubuli (simboli aperti) sono determinate dalle corrispondenti curve τ(q). Le barre di errore in tutti i grafici rappresentano l'errore standard dei valori tra tre e cinque repliche. Questa cifra è stata modificata rispetto al riferimento17. Fare clic qui per visualizzare una versione ingrandita di questa figura.
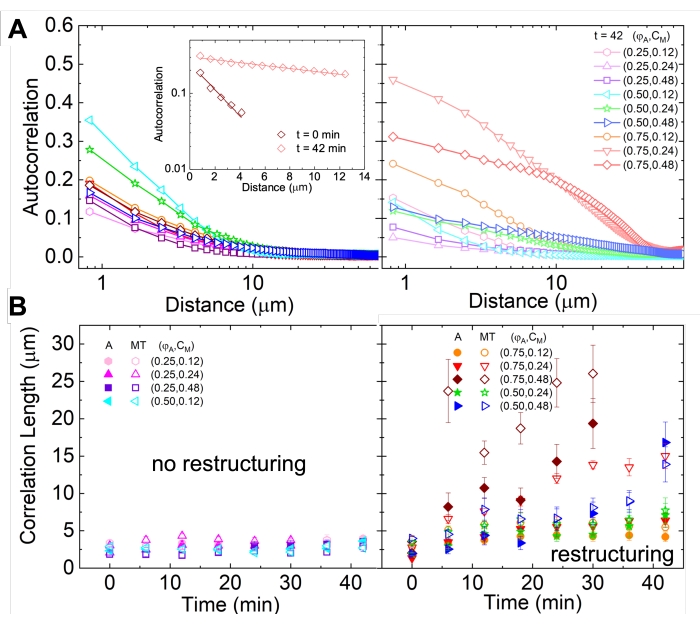
Figura 5. L'analisi dell'autocorrelazione delle immagini spaziali (SIA) quantifica la ristrutturazione motoria dei compositi citoscheletrici attivi. (A) Autocorrelazione g(r) per i microtubuli all'inizio (sinistra, t = 0 min, tonalità scure) e alla fine (destra, t = 42 min, tonalità chiare) dell'esperimento per le formulazioni (ΦA, cM) elencate nella legenda. Inserimento: esempio di adattamento dei dati all'ora 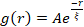 iniziale e finale per (ΦA, cM) = (0,75, 0,12). (B) Lunghezze medie di correlazione ξ per actina (simboli chiusi) e microtubuli (simboli aperti) per ciascuno (Φ A, cM) determinate tramite adattamenti esponenziali di ciascuna curva g(r), come mostrato nell'inserto in A. I dati sono divisi in quelli che mostrano una ristrutturazione minima (a sinistra) rispetto a quella sostanziale (a destra). Le barre di errore in A e B rappresentano l'errore standard tra tre e cinque repliche. Questa cifra è stata modificata rispetto al riferimento17. Fare clic qui per visualizzare una versione ingrandita di questa figura.
iniziale e finale per (ΦA, cM) = (0,75, 0,12). (B) Lunghezze medie di correlazione ξ per actina (simboli chiusi) e microtubuli (simboli aperti) per ciascuno (Φ A, cM) determinate tramite adattamenti esponenziali di ciascuna curva g(r), come mostrato nell'inserto in A. I dati sono divisi in quelli che mostrano una ristrutturazione minima (a sinistra) rispetto a quella sostanziale (a destra). Le barre di errore in A e B rappresentano l'errore standard tra tre e cinque repliche. Questa cifra è stata modificata rispetto al riferimento17. Fare clic qui per visualizzare una versione ingrandita di questa figura.
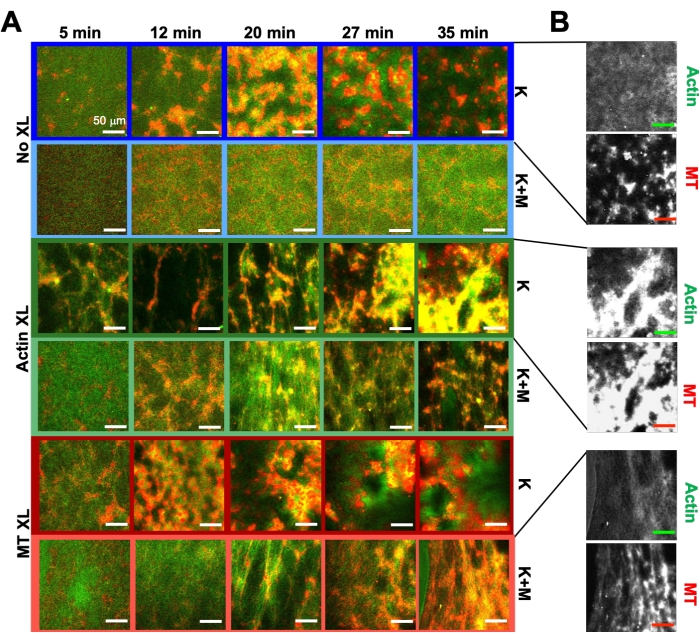
Figura 6. Incorporare motori kinesin e reticolanti passivi in compositi attivi per aumentare la programmabilità ed espandere lo spazio delle fasi della dinamica e della struttura. (A) Le immagini confocali a due colori di actina (verde) e microtubuli (rosso) nei compositi attivi mostrano una complessa ristrutturazione dipendente dalla formulazione nel tempo (elencata in min). Le cinque immagini in ogni riga corrispondono a cinque fotogrammi di una serie temporale di 2000 fotogrammi acquisita per un composito guidato da kinesina (K, righe 1, 3, 5) o kinesina e miosina (K + M, righe 2, 4, 6), e che include nessun reticolante passivo (No XL, righe 1, 2), reticoli actina-actina (Actin XL, righe 3, 4) o reticoli microtubulo-microtubuli (MT XL, righe 5, 6). Le barre della scala sono tutte da 50 μm. I colori dei contorni corrispondono alla combinazione di colori nella Figura 8. (B) Canali separati di fluorescenza di actina e microtubuli per i compositi solo chinesina mostrano strutture varie con co-localizzazione actina-MT e separazione micro-fase. Le immagini mostrate sono per compositi con c A = 2,32 μM, c T = 3,48 μM, c K = 0,35 μM, c M = 0,47 μM (righe 2, 4, 6), R A = 0,02 (righe 3, 4) e RMT = 0,005 (righe 5, 6). Tutti i compositi iniziano con reti interpenetranti uniformemente distribuite di actina e microtubuli (colonna 1). I compositi guidati dalla kinesina senza reticolanti (riga 1) formano cluster amorfi liberamente connessi che sono ricchi di MT. L'actina co-localizza inizialmente nei centri di questi aggregati, ma poi viene spremuta dalle regioni ricche di MT che continuano a contrarsi e disconnettersi l'una dall'altra. La reticolazione actina-actina (riga 3) ostacola questa separazione actina-MT su microscala, e invece gli aggregati ricchi di MT sono collegati tramite lunghi filamenti di actina. Il crosslinking dell'actina consente anche un lento assorbimento di actina nelle regioni ricche di MT, in modo tale che il composito diventi una rete connessa di cluster di actina e MT co-localizzati. La reticolazione dei microtubuli (riga 5) porta a un raggruppamento amorfo di MT che si fondono nel tempo, con conseguente separazione di fase su scala più ampia di actina e MT. L'aggiunta di miosina (righe 2, 4, 6) riduce la demiscelazione e la ristrutturazione guidate dalla chinesina. Senza reticolanti (riga 2), i compositi mostrano pochi riarrangiamenti nel corso delle ore. La reticolazione aumenta la ristrutturazione e la co-localizzazione di actina e microtubuli (righe 4, 6). In particolare, quando i microtubuli sono reticolati (riga 6), vi è una significativa compenetrazione e riorganizzazione in reti di fibre simili a ragnatele. Questa cifra è stata modificata rispetto alla referenza50. Fare clic qui per visualizzare una versione ingrandita di questa figura.
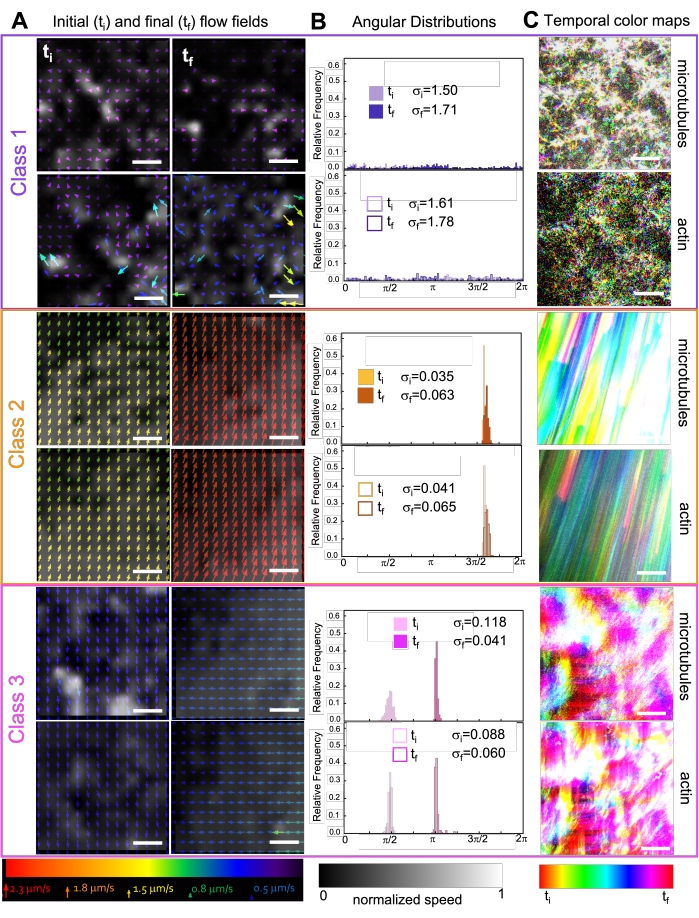
Figura 7. PIV mostra che i compositi attivi presentano tre classi di campi di flusso spaziotemporalmente distinti. (A) Campi di flusso PIV per il primo (t i) e l'ultimo (tf) frame di tre serie temporali rappresentative, che mostrano le diverse classi dinamiche mostrate dai compositi mostrati nella Figura 6. Video di esempio dei campi di flusso PIV per microtubuli (in alto) e actina (in basso) per i video di esempio di classe 1 (in alto, viola), classe 2 (al centro, arancione) e classe 3 (in basso, magenta), con i colori delle frecce corrispondenti alla scala di velocità universale in basso e la mappa dei colori in scala di grigi che mostra la distribuzione della velocità spaziale, normalizzata separatamente per ciascun campo di flusso in base alla scala mostrata in basso. Le barre della scala sono tutte da 50 μM. (B) Distribuzioni angolari dei vettori di velocità da A (in unità di radianti) con deviazioni standard iniziali e finali elencate σ i e σf. (C) Le mappe temporali a colori per i video analizzati in A e B mostrano la posizione da fotogramma a fotogramma di ciascun pixel rispetto al suo punto iniziale. Le mappe di classe 1 mostrano movimenti casuali su piccola scala; Le mappe di classe 2 rappresentano un movimento unidirezionale veloce con variazioni spaziali o temporali minime; Le mappe di Classe 3 presentano caratteristiche sia di Classe 1 che di Classe 2. Questa cifra è stata modificata rispetto alla referenza50. Fare clic qui per visualizzare una versione ingrandita di questa figura.
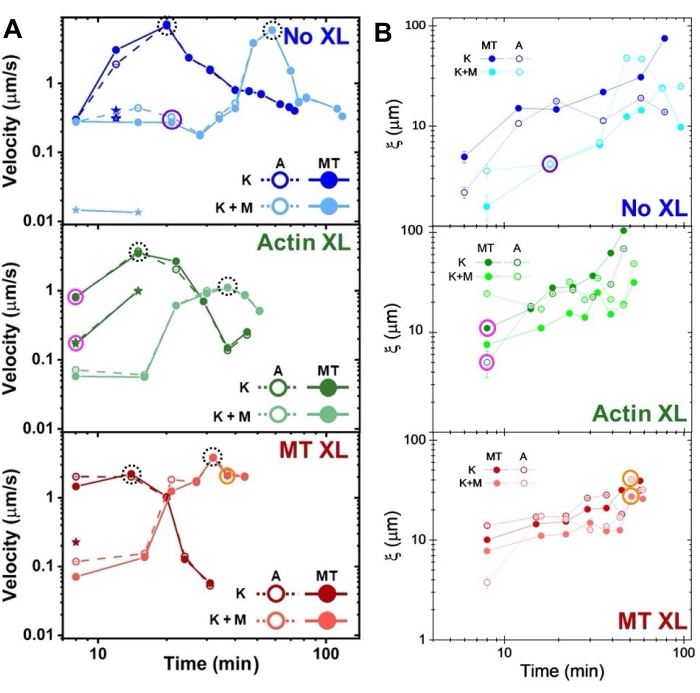
Figura 8. DDM e SIA misurano la dinamica e la struttura variabili nel tempo dei compositi actina-microtubuli a due motori. (A) Le velocità per i compositi descritte nelle figure 6 e 7, misurate tramite DDM, mostrano l'accelerazione e la decelerazione dei compositi, programmate mediante reticolazione e attività della miosina. Le velocità dei microtubuli (MT, cerchi chiusi) e dell'actina (A, cerchi aperti) sono tracciate in funzione del tempo di attività in compositi senza reticolazione (superiore, blu), reticolazione di actina (centrale, verde), reticolazione di microtubuli (in basso, rosso), senza miosina (K, tonalità più scure) e con miosina (K + M, tonalità più chiare). Per i casi di classe 3, che hanno due velocità, la velocità più bassa è indicata da una stella. I punti dati racchiusi da cerchi neri tratteggiati corrispondono alla velocità massima vmaxper ogni formulazione. Le barre di errore (la maggior parte troppo piccole per essere viste) sono l'errore standard sulla legge di potenza del corrispondente τ(q). (B) Lunghezze di correlazione strutturale ξ, determinate tramite SIA, rispetto al tempo di attività, per lo stesso insieme di serie temporali valutate in A. Ogni punto dati è una media delle lunghezze di correlazione determinate per il primo e l'ultimo fotogramma della serie temporale corrispondente. In generale, ξ aumenta nel tempo sia per l'actina che per i microtubuli in tutti i sistemi compositi, e i compositi guidati esclusivamente dalla kinesina hanno lunghezze di correlazione maggiori rispetto a quelli in cui è presente anche la miosina. I punti dati in A e B che corrispondono alle tre serie storiche analizzate nella Figura 7 sono cerchiati nel colore della classe corrispondente (1 = viola, 2 = arancione, 3 = magenta). Questa cifra è stata modificata rispetto alla referenza50. Fare clic qui per visualizzare una versione ingrandita di questa figura.
Discussione
Un progresso chiave del sistema ricostituito sopra descritto è la sua modularità e sintonizzazione, quindi gli utenti sono incoraggiati a modificare le concentrazioni di proteine, motori, reticolanti, ecc. per soddisfare i risultati desiderati, sia che si tratti di emulare un particolare processo cellulare o di ingegnerizzare un materiale con funzionalità specifiche o proprietà meccaniche. Le limitazioni sull'intervallo di concentrazione di actina e tubulina sono fissate al limite inferiore dalla concentrazione critica necessaria per polimerizzare l'actina (~0,2 μM)57,58,59 e la tubulina (~3 - 4 μM)60, e al limite superiore dalla transizione all'allineamento nematico dei filamenti di actina (~90 μM)61,62 o dei microtubuli (~35 μM)63 . I monomeri di actina e i dimeri di tubulina dovrebbero essere polimerizzati insieme in filamenti, piuttosto che mescolati insieme dopo la polimerizzazione, per garantire che formino reti percolate omogeneamente interpenetranti che si supportano sinergicamente a vicenda. Le nuove dinamiche che i compositi esibiscono si basano su questa interazione. Mentre è generalmente importante seguire tutti i passaggi come indicato nel protocollo per riprodurre correttamente i risultati mostrati, alcuni passaggi sono più impegnativi, mentre altri hanno spazio per modificare e regolare per soddisfare esigenze specifiche e risorse disponibili.
Ad esempio, un passo importante per garantire risultati riproducibili è preparare e conservare correttamente i reagenti seguendo le linee guida fornite nella tabella dei materiali. Le proteine citoscheletriche (actina, tubulina, miosina, chinesina) sono labili e devono essere aliquotate, congelate con azoto liquido e conservate a -80 °C in aliquote monouso. Una volta rimosse da -80 °C, le aliquote devono essere conservate in ghiaccio. Le proteine citoscheletriche non mantengono in modo affidabile la funzione dopo ulteriori cicli di congelamento-scongelamento.
I microtubuli sono più sensibili alla depolimerizzazione e alla denaturazione rispetto all'actina. Una volta rimossa da -80 °C, la tubulina deve essere conservata su ghiaccio prima della polimerizzazione e utilizzata entro 12 ore. Una volta polimerizzati, i microtubuli devono essere mantenuti a temperatura ambiente. È anche fondamentale stabilizzare i microtubuli con taxolo per prevenire la depolimerizzazione. La stabilizzazione della falloidina dei filamenti di actina è anche importante per sopprimere il tapis roulant di actina che consuma ATP che compete con l'attività della miosina e della chinesina.
L'ultracentrifugazione dei motori della miosina è un altro passaggio critico, in quanto rimuove le teste morte della miosina inattive. Non rimuovere i monomeri enzimaticamente inattivi provoca la reticolazione passiva della rete di actina e la perdita di attività. Per prolungare l'attività dell'ATPasi dei motori, è possibile incorporare un sistema di rigenerazione dell'ATP come la creatina fosfato e la creatinfosfochinasi64 .
Infine, il mantenimento dell'attività composita richiede l'inibizione dell'adsorbimento di filamenti e motori alle pareti della camera del campione, che può essere ottenuto mediante passivazione dei vetrini e dei vetrini del microscopio. Le proteine motorie sono particolarmente inclini all'adsorbimento, il che si traduce in un composito che viene tirato sulla superficie della camera del campione, si sposta fuori dal campo visivo, collassa in 2D e non subisce più attività. Silinizzare i vetrini e i vetrini è un modo efficace per passivare le superfici e prevenire l'adsorbimento (vedi fase 1). Un metodo alternativo di passivazione utilizzato efficacemente negli esperimenti di citoscheletro in vitro è il rivestimento della superficie con un doppio strato lipidico, simile alla membrana cellulare18. Questo metodo è vantaggioso se si desidera legare le proteine alla superficie o introdurre altre interazioni specifiche proteina-superficie, perché il doppio strato può essere funzionalizzato. Per gli esperimenti con pinzette ottiche, anche la passivazione delle microsfere è critica, e può essere ottenuta rivestendo microsfere carbossilate con BSA o PEG tramite chimica reticolante carbodiimmide48.
Ci sono alcuni aspetti dei protocolli presentati che i ricercatori possono considerare di modificare per soddisfare le loro esigenze. In primo luogo, i ricercatori possono scegliere di sostituire i crosslinker biotina-NA non nativi con reticolanti biologici, come l'alfa-actina o MAP65 che relazionano actina e microtubuli, rispettivamente 28,65,66. L'uso di reticolanti non nativi nei compositi qui descritti è motivato dalla loro maggiore riproducibilità, stabilità e sintonizzazione rispetto ai reticolanti nativi. A causa del forte legame biotina-NA, si può presumere che i reticolanti siano permanenti, piuttosto che la maggior parte dei reticolanti nativi che si legano transitoriamente con tassi di turnover ad ampio raggio. La dinamica della reticolazione transitoria complica l'analisi dei contributi dei reticolanti e dei motori alla dinamica. Inoltre, i linker biotina-NA possono essere utilizzati in modo versatile per reticolare sia l'actina che i microtubuli, nonché l'actina reticolata ai microtubuli. In questo modo, è possibile effettuare un confronto univoco tra i motivi di reticolazione, mantenendo fisse tutte le altre variabili (ad esempio, dimensione del reticolante, affinità di legame, stechiometria, ecc.). Infine, i reagenti necessari per incorporare i linker biotina-NA sono ampiamente disponibili in commercio, ben caratterizzati e comunemente usati in molti laboratori di biofisica. Tuttavia, uno dei punti di forza della piattaforma in vitro qui descritta è la sua modularità, quindi i ricercatori dovrebbero essere in grado di sostituire senza problemi i linker biotina-NA con linker nativi se lo desiderano.
In secondo luogo, nel protocollo attuale, i monomeri di actina e i dimeri di tubulina vengono polimerizzati in filamenti insieme in una provetta da centrifuga prima di essere aggiunti alla camera del campione. Il flusso della soluzione di proteine filamentose entangled nella camera del campione può causare l'allineamento del flusso, in particolare dei microtubuli, che rompe l'isotropia desiderata e l'omogeneità dei compositi. In effetti, un importante progresso nel lavoro precedente sui compositi actina-microtubuli allo stato stazionario è stata la capacità di copolimerizzare actina e microtubuli in situ (nella camera del campione) per garantire la formazione di reti isotrope interpenetranti di actina e microtubuli15,16,27. Tuttavia, estendere questo approccio ai compositi attivi richiederebbe l'aggiunta dei motori al campione prima della polimerizzazione di actina e tubulina e l'incubazione dell'intero campione a 37 °C prima degli esperimenti. I test di questa variazione del protocollo hanno portato a una ridotta polimerizzazione dell'actina e all'assenza di attività motoria distinguibile, probabilmente a causa dell'attività concorrente dell'ATPasi e della prolungata incubazione a 37 °C dei motori. Fortunatamente, non vi è alcun allineamento del flusso percepibile dei compositi quando si seguono i protocolli attuali, come si può vedere nella Figura 2, nella Figura 3 e nella Figura 6. Tuttavia, i ricercatori sono incoraggiati a progettare protocolli che consentano la formazione in situ di compositi attivi.
Un altro punto da considerare è lo schema di etichettatura a fluorescenza, che comporta l'etichettatura scarsamente di tutti i filamenti di actina e dei microtubuli nella rete. Questo approccio di etichettatura è stato ottimizzato per visualizzare direttamente la struttura della rete piuttosto che dedurre struttura e dinamica tramite filamenti traccianti o microsfere. Tuttavia, il compromesso è che i singoli filamenti non sono etichettati e risolvibili. Un approccio che i ricercatori potrebbero adottare sia per risolvere singoli filamenti che per visualizzare la struttura della rete è quello di drogare in filamenti preformati etichettati con un altro fluoroforo, in modo che sia la rete circostante che i singoli filamenti possano essere visualizzati contemporaneamente. Tuttavia, quando si utilizzano più di due fluorofori e canali di eccitazione / emissione, il bleed-through tra i canali è spesso difficile da eliminare, quindi è necessario prestare attenzione nella scelta dei fluorofori, dei filtri e delle intensità laser.
Una limitazione correlata è l'incapacità di visualizzare i motori della miosina o della chinesina nei compositi. I monomeri di actina marcati con fluorescenza e i dimeri di tubulina utilizzati sono disponibili in commercio, mentre la visualizzazione della miosina o della chinesina nei compositi richiede un'etichettatura interna. I ricercatori sono incoraggiati a fare il passo successivo per etichettare i motori, come fatto in precedenza18,67, per essere in grado di collegare inequivocabilmente l'attività motoria e il legame con le dinamiche e le strutture che i nostri compositi esibiscono.
Infine, è importante notare che, nell'attuale protocollo, l'insorgenza e la durata dell'attività della kinesina non sono controllate. Poiché l'attività della miosina è controllata usando la foto-disattivazione della blebbistatina, come descritto sopra, per costruire un'attivazione luminosa simile della chinesina, si può incorporare ATP attivato dalla luce.
Per costruire la complessità dei progetti qui descritti, per imitare meglio le condizioni cellulari e ampliare lo spazio dei parametri dinamica-struttura-funzione, il lavoro futuro si concentrerà sull'incorporazione di filamenti intermedi, come vimentin68,69, così come altri motori come dynein13,70. La gelsolina sarà anche incorporata a diverse concentrazioni per controllare la lunghezza dell'actina14, così come la proteina tau per controllare la rigidità dei microtubuli.
In sintesi, i protocolli presentati descrivono come progettare, creare e caratterizzare la dinamica, la struttura e la meccanica dei sistemi di sostanza attiva ispirati al citoscheletro, che contengono due componenti separati che generano forza attiva che agiscono su substrati diversi in un unico sistema. Questa piattaforma sintonizzabile e modulare porta gli sforzi di ricostituzione un importante passo avanti verso l'imitazione del citoscheletro cellulare e offre la capacità unica di programmare le sue proprietà attraverso un ampio spazio delle fasi incorporando, rimuovendo e sintonizzando in modo indipendente i diversi componenti. Inoltre, tutti i componenti di questo versatile sistema sono disponibili in commercio (vedi Tabella dei materiali), ad eccezione dei dimeri di kinesina che sono purificati nel Ross Lab, come descritto in precedenza50, e disponibili su richiesta. Infine, tutto il codice di analisi è liberamente disponibile tramite GitHub49 ed è basato su linguaggi di programmazione e software gratuiti (Python e Fiji). Si spera che la diffusione trasparente dei protocolli per progettare questi sistemi renderà questa piattaforma più accessibile a un gruppo diversificato di utenti con competenze, background, affiliazioni istituzionali e obiettivi di ricerca diversi.
Divulgazioni
Gli autori non hanno nulla da rivelare
Riconoscimenti
Ringraziamo Maya Hendija e il Dr. Jonathan Michel per l'assistenza con l'analisi dei dati, e il Dr. Janet Sheung, il Dr. Moumita Das e il Dr. Michael Rust per le utili discussioni e indicazioni. Questa ricerca è stata supportata da un William M. Keck Foundation Research Grant e NSF DMREF Award (DMR 2119663) assegnato a RMRA e JLR e National Institutes of Health R15 Grants (R15GM123420, 2R15GM123420-02) assegnato a RMR-A e RJM.
Materiali
| Name | Company | Catalog Number | Comments |
| (-)-Blebbistatin Abbreviation used in paper: blebbistatin | Sigma Aldrich | B0560 | Stock Concentration: 200 μM in DMSO Storage: dessicated, in DMSO, -20ºC Stock and Experiment Recipes: dissolve 1 mg of powder to 200 μM in DMSO Storage, Handling, Troubleshooting Notes: limited shelf-life, typically stops functioning reliably after 3-4 months. purchase and prepare new solution every 3 months. |
| 1:20 488-tubulin:tubulin mixture Abbreviation used in paper: 5-488-tubulin | NA | NA | Stock Concentration: 5 mg/ml in PEM Storage: single use aliquots, -80ºC, avoid freeze-thaw cycles Stock and Experiment Recipes: mix tubulin and 488-tubulin at a 20:1 ratio, flash freeze with LN2 Storage, Handling, Troubleshooting Notes: each aliquot can be used for up to 12 hrs stored on ice at 4ºC, protect from light |
| 1:20 R-tubulin:tubulin mixture Abbreviation used in paper: 5-R-tubulin | NA | NA | Stock Concentration: 5 mg/ml in PEM Storage: single use aliquots, -80ºC, avoid freeze-thaw cycles Stock and Experiment Recipes: mix tubulin and rhodamine tubulin at a 20:1 ratio, flash freeze with LN2 Storage, Handling, Troubleshooting Notes: each aliquot can be used for up to 12 hrs stored on ice at 4ºC, protect from light |
| actin (biotin): skeletal muscle Abbreviation used in paper: biotin-actin | Cytoskeleton | AB07 | Stock Concentration: 1 mg/ml in G-buffer Storage: single use aliquots, -80ºC, avoid freeze-thaw cycles Stock and Experiment Recipes: reconstitute to 1 mg/ml in G-buffer, flash freeze with LN2 Storage, Handling, Troubleshooting Notes: (1) immediately prior to use dilute to 0.5 mg/ml in PEM, (2) once removed from -80ºC, store aliquot on ice at 4ºC for up to 1 week |
| actin (rhodamine): rabbit skeletal muscle Abbreviation used in paper: R-actin | Cytoskeleton | AR05 | Stock Concentration: 1.5 mg/ml in G-buffer Storage: single use aliquots, -80ºC, avoid freeze-thaw cycles Stock and Experiment Recipes: reconstitute to 1.5 mg/ml in G-buffer, flash freeze with LN2 Storage, Handling, Troubleshooting Notes: once removed from -80ºC, store aliquot on ice at 4ºC, can be used for up to 1 week |
| adenosine triphosphate Abbreviation used in paper: ATP | Thermo Fisher Scientific | A1048 | Stock Concentration: 100 mM Storage: in solution (pH 7), -20ºC Stock and Experiment Recipes: reconsitute in DI H20, bring pH to 7 with NaOH Storage, Handling, Troubleshooting Notes: routinely check pH and adjust as needed, hydrolyzes over time, replace every ~6-12 months |
| AlexaFluor488 Phalloidin Abbreviation used in paper: 488-phalloidin | Thermo Fisher Scientific | A12379 | Stock Concentration: 100 μM DMSO Storage: protected from light, dessicated, -20ºC Stock and Experiment Recipes: reconstitute to 100 μM with DMSO Storage, Handling, Troubleshooting Notes: immediately prior to use dilute to 20 μM in PEM (1 μL in 4 μL PEM) |
| AlexaFluor488–labeled actin Abbreviation used in paper: 488-actin | Thermo Fisher Scientific | A12373 | Stock Concentration: 1.5 mg/ml in G-buffer Storage: single use aliquots, -80ºC, avoid freeze-thaw cycles Stock and Experiment Recipes: reconstitute to 1.5 mg/ml in G-buffer, flash freeze with LN2 Storage, Handling, Troubleshooting Notes: this item has been discontinued |
| Basic Plasma Cleaner Abbreviation used in paper: plasma cleaner | Harrick Plasma | PDC-32G | |
| Bemis Parafilm M Laboratory Wrapping Film Abbreviation used in paper: transparent film | Thermo Fisher Scientific | 13-374-5 | |
| D-(+)-Glucose Abbreviation used in paper: | Thermo Fisher Scientific | A1682836 | Stock Concentration: 100x Storage: store at stock concentration (100x) or 10x concentration, dessicated, at -20ºC Stock and Experiment Recipes: reconstitute powder to 4.5 mg/ml in DI H20 Storage, Handling, Troubleshooting Notes: final concentration in solution should 45 μg/mL |
| D-Biotin Abbreviation used in paper: biotin | Fisher Scientific | BP232-1 | Stock Concentration: 1.02 mM in PEM Storage: dessicated, 4ºC |
| deionized nanopure water Abbreviation used in paper: DI | |||
| Dimethyldichlorosilane Abbreviation used in paper: silane | Thermo Fisher Scientific | D/3820/PB05 | Stock Concentration: 2% dissolved in Toulene |
| Dithiothreitol Abbreviation used in paper: DTT | Thermo Fisher Scientific | R0861 | Stock Concentration: 1 M in DMSO Storage: dessicated, -20ºC Stock and Experiment Recipes: dilute to 2 mM in PEM immediately before each experiment |
| DMSO Anhydrous Abbreviation used in paper: DMSO | Thermo Fisher Scientific | D12345 | |
| F-Buffer Abbreviation used in paper: F-buffer | NA | NA | Stock Concentration: 10x Storage: dessicated, -20ºC Stock and Experiment Recipes: 10 mM Imidazole (pH 7.0), 50 mM KCl, 1 mM MgCl2, 1 mM EGTA, 0.2 mM ATP |
| G-Buffer Abbreviation used in paper: G-buffer | NA | NA | Stock Concentration: 10x Storage: dessicated, -20ºC Stock and Experiment Recipes: 2.0 mM Tris (pH 8), 0.2 mM ATP, 0.5 mM DTT, 0.1 mM CaCl2. Store at -20°C. |
| glass microscope slide Abbreviation used in paper: slide | Thermo Fisher Scientific | 22-310397 | |
| Glucose oxidase + catalase + β-mercaptoethanol Abbreviation used in paper: GOC | Sigma Aldrich | G2133-250KU, C1345, 63689 | Stock Concentration: 100x Storage: store at stock concentration (100x) or 10x concentration, dessicated, at -20ºC Stock and Experiment Recipes: For 100x: 4.3 mg/ml glucose oxidase, 0.7 mg/ml catalase, 0.5% v/v β-mercaptoethanol in DI H20 Storage, Handling, Troubleshooting Notes: final concentration in solution should be: 0.005% β-mercaptoethanol, 43 μg/mL glucose oxidase, 7 μg/mL catalase |
| glu-GOC oxygen scavenging system Abbreviation used in paper: glu-GOC | NA | NA | Stock Concentration: 100x Storage: prepare fresh each time Stock and Experiment Recipes: mix equal parts Glu and GOC and add at 1/100 final sample volume immediately before imaging Storage, Handling, Troubleshooting Notes: prepare from Glu and GOC immediately before imaging |
| Guanosine triphosphate Abbreviation used in paper: GTP | Thermo Fisher Scientific | R0461 | Stock Concentration: 100 mM Storage: 100 μL aliquots at -20ºC |
| Instant Mix 1-minute epoxy Abbreviation used in paper: epoxy | Loctite | 1366072 | |
| Kinesin-1 401 BIO 6x HIS Abbreviation used in paper: kinesin | Prepared in JL Ross Lab at Syracuse University | NA | Stock Concentration: 8.87 μM in PEM Storage: single use aliquots, -80ºC, avoid freeze-thaw cycles Storage, Handling, Troubleshooting Notes: biotinylated dimers form kinesin clusters, each aliquot can be used for up to 12 hrs stored on ice at 4ºC |
| NeutrAvidin Abbreviation used in paper: NA | Thermo Fisher Scientific | 31000 | Stock Concentration: 5 mg/ml in PEM Storage: dessicated, -20ºC Stock and Experiment Recipes: reconstitute powder to 5 mg/ml in PEM |
| No 1. glass coverslips (24 mm x 24 mm) Abbreviation used in paper: coverslip | Thermo Fisher Scientific | 12-548-CP | |
| Paclitaxel Abbreviation used in paper: Taxol | Thermo Fisher Scientific | P3456 | Stock Concentration: 2 mM in DMSO Storage: protected from light, dessicated, -20ºC Stock and Experiment Recipes: reconstitute to 2 mM with DMSO Storage, Handling, Troubleshooting Notes: immediately prior to use dilute to 200 μM in DMSO (0.4 μL in 3.6 μL DMSO) |
| PEM-100 Abbreviation used in paper: PEM | NA | NA | Stock Concentration: 1x Storage: room temperature (RT) Stock and Experiment Recipes: 100 mM K-PIPES (pH 6.8), 2 mM EGTA, 2 mM MgCl2 Storage, Handling, Troubleshooting Notes: use KOH to adjust pH to 6.8, recheck pH often and adjust accordingly |
| phalloidin Abbreviation used in paper: phalloidin | Thermo Fisher Scientific | P3457 | Stock Concentration: 100 μM in DMSO Storage: protected from light, dessicated, -20ºC, adhere closely to storage/handling conditions Stock and Experiment Recipes: reconstitute to 100 μM with DMSO Storage, Handling, Troubleshooting Notes: susceptible to impurities in its preparation and denaturing, identifiable as large amorphous aggregates of actin in samples |
| porcine brain tubulin Abbreviation used in paper: tubulin | Cytoskeleton | T240 | Stock Concentration: 5 mg/ml in PEM Storage: single use aliquots, -80ºC, avoid freeze-thaw cycles Stock and Experiment Recipes: reconstitute powder to 5 mg/ml in PEM, flash freeze with LN2 Storage, Handling, Troubleshooting Notes: each aliquot can be used for up to 12 hrs stored on ice at 4ºC |
| Potassium Chloride Abbreviation used in paper: KCl | Thermo Fisher Scientific | AM9640G | Stock Concentration: 4 M Storage: RT |
| Rabbit skeletal actin Abbreviation used in paper: actin | Cytoskeleton | AKL99 | Stock Concentration: 2 mg/ml in G-buffer Storage: single use aliquots, -80ºC, avoid freeze-thaw cycles Stock and Experiment Recipes: reconstitute to 2 mg/ml in G-buffer, flash freeze with LN2 Storage, Handling, Troubleshooting Notes: once removed from -80ºC, store aliquot on ice at 4ºC, can be used for up to 1 week |
| Rabbit skeletal myosin II Abbreviation used in paper: myosin | Cytoskeleton | MY02 | Stock Concentration: 10 mg/ml in PEM Storage: single use aliquots, -80ºC, avoid freeze-thaw cycles Stock and Experiment Recipes: reconstitute powder to 10 mg/ml in PEM, flash freeze with LN2 Storage, Handling, Troubleshooting Notes: monomers form minifilaments at low KCl, each aliquot can be used for up to 12 hrs stored on ice at 4ºC |
| Tubulin (biotin): porcine brain Abbreviation used in paper: biotin-tubulin | Cytoskeleton | T333P | Stock Concentration: 5 mg/ml in PEM Storage: single use aliquots, -80ºC, avoid freeze-thaw cycles Stock and Experiment Recipes: reconstitute powder to 5 mg/ml in PEM, flash freeze with LN2 Storage, Handling, Troubleshooting Notes: immediately prior to use dilute to 0.5 mg/ml in PEM |
| Tubulin (fluorescent HiLyte 488): porcine brain Abbreviation used in paper: 488-tubulin | Cytoskeleton | TL488M | Stock Concentration: 5 mg/ml in PEM Storage: single use aliquots, -80ºC, avoid freeze-thaw cycles Stock and Experiment Recipes: reconstitute powder to 5 mg/ml in PEM, flash freeze with LN2 Storage, Handling, Troubleshooting Notes: each aliquot can be used for up to 12 hrs stored on ice at 4ºC, protect from light |
| tubulin (rhodamine): porcine brain Abbreviation used in paper: R-tubulin | Cytoskeleton | TL590M | Stock Concentration: 5 mg/ml in PEM Storage: single use aliquots, -80ºC, avoid freeze-thaw cycles Stock and Experiment Recipes: reconstitute powder to 5 mg/ml in PEM, flash freeze with LN2 Storage, Handling, Troubleshooting Notes: each aliquot can be used for up to 12 hrs stored on ice at 4ºC, protect from light |
| Tween 20 Abbreviation used in paper: Tween20 | Thermo Fisher Scientific | J20605.AP | Stock Concentration: 1% v/v in DI H20 Storage: RT |
| ultracentrifuge grade microtubes Abbreviation used in paper: Beckman-Coulter Optima Max XP | Beckman Coultier | 343776 | Storage, Handling, Troubleshooting Notes: 8x34 mm PC |
| UV light curing glue Abbreviation used in paper: UV glue | Pharda | SKG-2869 |
Riferimenti
- Fletcher, D. A., Mullins, R. D. Cell mechanics and the cytoskeleton. Nature. 463 (7280), 485-492 (2010).
- Koenderink, G. H., Paluch, E. K. Architecture shapes contractility in actomyosin networks. Current Opinion in Cell Biology. 50, 79-85 (2018).
- Dogterom, M., Koenderink, G. H. Actin-microtubule crosstalk in cell biology. Nature Reviews Molecular Cell Biology. 20 (1), 38-54 (2019).
- Burla, F., Mulla, Y., Vos, B. E., Aufderhorst-Roberts, A., Koenderink, G. H. From mechanical resilience to active material properties in biopolymer networks. Nature Reviews Physics. 1 (4), 249-263 (2019).
- Wen, Q., Janmey, P. A. Polymer physics of the cytoskeleton. Current Opinion in Solid State and Materials Science. 15 (5), 177-182 (2011).
- Xiao, Q., Hu, X., Wei, Z., Tam, K. Y. Cytoskeleton molecular motors: structures and their functions in neuron. International Journal of Biological Sciences. 12 (9), 1083-1092 (2016).
- Ajeti, V. et al. Wound healing coordinates actin architectures to regulate mechanical work. Nature Physics. 15 (7), 696-705 (2019).
- Jung, W. et al. Dynamic motions of molecular motors in the actin cytoskeleton. Cytoskeleton. 76 (11-12), 517-531 (2019).
- Pollard, T. D., O'Shaughnessy, B. Molecular mechanism of cytokinesis. Annual Review of Biochemistry. 88 (1), 661-689 (2019).
- Huber, F., Boire, A., López, M. P., Koenderink, G. H. Cytoskeletal crosstalk: when three different personalities team up. Current Opinion in Cell Biology. 32, 39-47 (2015).
- Rivero, F. et al. The role of the cortical cytoskeleton: F-actin crosslinking proteins protect against osmotic stress, ensure cell size, cell shape and motility, and contribute to phagocytosis and development. Journal of Cell Science. 109 (11), 2679-2691 (1996).
- Duclos, G. et al. Topological structure and dynamics of three-dimensional active nematics. Science. 367 (6482), 1120-1124 (2020).
- Baclayon, M. et al. Optical tweezers-based measurements of forces and dynamics at microtubule ends. Optical Tweezers. 1486, 411-435 (2017).
- Gurmessa, B., Fitzpatrick, R., Falzone, T. T., Robertson-Anderson, R. M. Entanglement density tunes microscale nonlinear response of entangled actin. Macromolecules. 49 (10), 3948-3955 (2016).
- Francis, M. L. et al. Non-monotonic dependence of stiffness on actin crosslinking in cytoskeleton composites. Soft Matter. 15 (44), 9056-9065 (2019).
- Ricketts, S. N. et al. Varying crosslinking motifs drive the mesoscale mechanics of actin-microtubule composites. Scientific Reports. 9 (1), 12831 (2019).
- Lee, G. et al. Active cytoskeletal composites display emergent tunable contractility and restructuring. Soft Matter. 17 (47), 10765-10776 (2021).
- Murrell, M. P., Gardel, M. L. F-actin buckling coordinates contractility and severing in a biomimetic actomyosin cortex. Proceedings of the National Academy of Sciences. 109 (51), 20820-20825 (2012).
- Soares e Silva, M. et al. Active multistage coarsening of actin networks driven by myosin motors. Proceedings of the National Academy of Sciences. 108 (23), 9408-9413 (2011).
- Sonn-Segev, A., Bernheim-Groswasser, A., Roichman, Y. Dynamics in steady state in vitro acto-myosin networks. Journal of Physics: Condensed Matter. 29 (16), 163002 (2017).
- Ideses, Y., Sonn-Segev, A., Roichman, Y., Bernheim-Groswasser, A. Myosin II does it all: assembly, remodeling, and disassembly of actin networks are governed by myosin II activity. Soft Matter. 9 (29), 7127 (2013).
- Fürthauer, S. et al. Self-straining of actively crosslinked microtubule networks. Nature Physics. 15 (12), 1295-1300 (2019).
- Lemma, L. M. et al. Multiscale microtubule dynamics in active nematics. Physical Review Letters. 127 (14), 148001 (2021).
- Fan, Y., Wu, K.-T., Aghvami, S. A., Fraden, S., Breuer, K. S. Effects of confinement on the dynamics and correlation scales in kinesin-microtubule active fluids. Physical Review E. 104 (3), 034601 (2021).
- Triclin, S. et al. Self-repair protects microtubules from destruction by molecular motors. Nature Materials. 20 (6), 883-891 (2021).
- Lee, G. et al. Myosin-driven actin-microtubule networks exhibit self-organized contractile dynamics. Science Advances. 7 (6), eabe4334 (2021).
- Ricketts, S. N., Ross, J. L., Robertson-Anderson, R. M. Co-entangled actin-microtubule composites exhibit tunable stiffness and power-law stress relaxation. Biophysical Journal. 115 (6), 1055-1067 (2018).
- Bendix, P. M. et al. A quantitative analysis of contractility in active cytoskeletal protein networks. Biophysical Journal. 94 (8), 3126-3136 (2008).
- Linsmeier, I. et al. Disordered actomyosin networks are sufficient to produce cooperative and telescopic contractility. Nature Communications. 7 (1), 12615 (2016).
- Stam, S. et al. Filament rigidity and connectivity tune the deformation modes of active biopolymer networks. Proceedings of the National Academy of Sciences. 114 (47), E10037-E10045 (2017).
- Yadav, V. et al. Filament nucleation tunes mechanical memory in active polymer networks. Advanced Functional Materials. 29 (49), 1905243 (2019).
- Ennomani, H. et al. Architecture and connectivity govern actin network contractility. Current Biology. 26 (5), 616-626 (2016).
- Alvarado, J., Sheinman, M., Sharma, A., MacKintosh, F. C., Koenderink, G. H. Molecular motors robustly drive active gels to a critically connected state. Nature Physics. 9 (9), 591-597 (2013).
- Alvarado, J., Cipelletti, L., Koenderink, G. H. Uncovering the dynamic precursors to motor-driven contraction of active gels. Soft Matter. 15 (42), 8552-8565 (2019).
- Jung, W., Murrell, M. P., Kim, T. F-actin cross-linking enhances the stability of force generation in disordered actomyosin networks. Computational Particle Mechanics. 2 (4), 317-327 (2015).
- Lenz, M., Thoresen, T., Gardel, M. L., Dinner, A. R. Contractile units in disordered actomyosin bundles arise from f-actin buckling. Physical Review Letters. 108 (23), 238107 (2012).
- Memarian, F.L. et al. Active nematic order and dynamic lane formation of microtubules driven by membrane-bound diffusing motors. Proceedings of the National Academy of Sciences. 118 (52), e2117107118 (2021).
- Needleman, D., Dogic, Z. Active matter at the interface between materials science and cell biology. Nature Reviews Materials. 2 (9), 17048 (2017).
- Foster, P. J., Fürthauer, S., Shelley, M. J., Needleman, D. J. Active contraction of microtubule networks. eLife. 4, e10837 (2015).
- Thijssen, K. et al. Submersed micropatterned structures control active nematic flow, topology, and concentration. Proceedings of the National Academy of Sciences. 118 (38), e2106038118 (2021).
- Sanchez, T., Chen, D. T. N., DeCamp, S. J., Heymann, M., Dogic, Z. Spontaneous motion in hierarchically assembled active matter. Nature. 491 (7424), 431-434 (2012).
- Colen, J. et al. Machine learning active-nematic hydrodynamics. Proceedings of the National Academy of Sciences. 118 (10), e2016708118 (2021).
- Mitchell, K. A., Tan, A. J., Arteaga, J., Hirst, L. S. Fractal generation in a two-dimensional active-nematic fluid. Chaos: An Interdisciplinary Journal of Nonlinear Science. 31 (7), 073125 (2021).
- Pandolfi, R. J., Edwards, L., Johnston, D., Becich, P., Hirst, L. S. Designing highly tunable semiflexible filament networks. Physical Review E. 89 (6), 062602 (2014).
- Tan, A. J. et al. Topological chaos in active nematics. Nature Physics. 15 (10), 1033-1039 (2019).
- Roostalu, J., Rickman, J., Thomas, C., Nédélec, F., Surrey, T. Determinants of polar versus nematic organization in networks of dynamic microtubules and mitotic motors. Cell. 175 (3), 796-808.e14 (2018).
- Ndlec, F. J., Surrey, T., Maggs, A. C., Leibler, S. Self-organization of microtubules and motors. Nature. 389 (6648), 305-308 (1997).
- Sheung, J. Y. et al. Motor-driven restructuring of cytoskeleton composites leads to tunable time-varying elasticity. ACS Macro Letters. 10 (9), 1151-1158 (2021).
- McGorty, R. PyDDM v0.2.0. Zenodo. (2022).
- Achiriloaie, D. H. et al. Kinesin and myosin motors compete to drive rich multi-phase dynamics in programmable cytoskeletal composites. arXiv. (2021).
- Wulstein, D. M., Regan, K. E., Garamella, J., McGorty, R. J., Robertson-Anderson, R. M. Topology-dependent anomalous dynamics of ring and linear DNA are sensitive to cytoskeleton crosslinking. Science Advances. 5 (12), eaay5912 (2019).
- McGorty, R. Image-Correlation. at <https://github.com/rmcgorty/Image-Correlation>. (2020).
- Robertson, C. Theory and practical recommendations for autocorrelation-based image correlation spectroscopy. Journal of Biomedical Optics. 17 (8), 080801 (2012).
- McGorty, R. Differential Dynamic Microscopy - Python. at <https://github.com/rmcgorty/Differential-Dynamic-Microscopy---Python>. (2021).
- Cerbino, R., Trappe, V. Differential dynamic microscopy: probing wave vector dependent dynamics with a microscope. Physical Review Letters. 100 (18), 188102 (2008).
- Robertson-Anderson, R. M. Optical tweezers microrheology: from the basics to advanced techniques and applications. ACS Macro Letters. 7 (8), 968-975 (2018).
- Pollard, T. D. Polymerization of ADP-actin. Journal of Cell Biology. 99 (3), 769-777 (1984).
- Coué, M., Brenner, S. L., Spector, I., Korn, E. D. Inhibition of actin polymerization by latrunculin A. FEBS Letters. 213 (2), 316-318 (1987).
- Pollard, T. D. Actin and actin-binding proteins. Cold Spring Harbor Perspectives in Biology. 8 (8), a018226 (2016).
- Kumar, N. Taxol-induced polymerization of purified tubulin. Mechanism of action. Journal of Biological Chemistry. 256 (20), 10435-10441 (1981).
- Käs, J. et al. F-actin, a model polymer for semiflexible chains in dilute, semidilute, and liquid crystalline solutions. Biophysical Journal. 70 (2), 609-625 (1996).
- Viamontes, J., Narayanan, S., Sandy, A. R., Tang, J. X. Orientational order parameter of the nematic liquid crystalline phase of F -actin. Physical Review E. 73 (6), 061901 (2006).
- Hitt, A. L., Cross, A. R., Williams, R. C. Microtubule solutions display nematic liquid crystalline structure. Journal of Biological Chemistry. 265 (3), 1639-1647 (1990).
- Andexer, J. N., Richter, M. Emerging enzymes for ATP regeneration in biocatalytic processes. ChemBioChem. 16 (3), 380-386 (2015).
- Farhadi, L. et al. Actin and microtubule crosslinkers tune mobility and control co-localization in a composite cytoskeletal network. Soft Matter. 16 (31), 7191-7201 (2020).
- Falzone, T. T., Lenz, M., Kovar, D. R., Gardel, M. L. Assembly kinetics determine the architecture of α-actinin crosslinked F-actin networks. Nature Communications. 3 (1), 861 (2012).
- Thoresen, T., Lenz, M., Gardel, M. L. Reconstitution of contractile actomyosin bundles. Biophysical Journal. 100 (11), 2698-2705 (2011).
- Sanghvi-Shah, R., Weber, G. F. Intermediate filaments at the junction of mechanotransduction, migration, and development. Frontiers in Cell and Developmental Biology. 5, 81 (2017).
- Shen, Y. et al. Effects of vimentin intermediate filaments on the structure and dynamics of in vitro multicomponent interpenetrating cytoskeletal networks. Physical Review Letters. 127 (10), 108101 (2021).
- Laan, L., Roth, S., Dogterom, M. End-on microtubule-dynein interactions and pulling-based positioning of microtubule organizing centers. Cell Cycle. 11 (20), 3750-3757 (2012).
Erratum
Formal Correction: Erratum: Reconstituting and Characterizing Actin-Microtubule Composites with Tunable Motor-Driven Dynamics and Mechanics
Posted by JoVE Editors on 10/11/2022. Citeable Link.
An erratum was issued for: Reconstituting and Characterizing Actin-Microtubule Composites with Tunable Motor-Driven Dynamics and Mechanics. The Authors section was updated.
Mehrzad Sasanpour1
Daisy H. Achiriloaie1,2
Gloria Lee1
Gregor Leech1
Christopher Currie1
K. Alice Lindsay3
Jennifer L. Ross3
Ryan J. McGorty1
Rae M. Robertson-Anderson1
1Department of Physics and Biophysics, University of San Diego
2W. M. Keck Science Department, Scripps College, Pitzer College, and Claremont McKenna College
3Department of Physics, Syracuse University
to:
Mehrzad Sasanpour1
Daisy H. Achiriloaie1,2
Gloria Lee1
Gregor Leech1
Maya Hendija1
K. Alice Lindsay3
Jennifer L. Ross3
Ryan J. McGorty1
Rae M. Robertson-Anderson1
1Department of Physics and Biophysics, University of San Diego
2W. M. Keck Science Department, Scripps College, Pitzer College, and Claremont McKenna College
3Department of Physics, Syracuse University
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon