Method Article
組み合わせるQD - FRETとマイクロ流体はリアルタイムでDNAのNanocomplex自己組織化を監視する
要約
我々は将来の核酸ベースの治療のために均一とカスタマイズ可能なpolyplexesのよりよい制御および合成を提供することが期待される高分子電解質polyplexes、の形成を調査するナノフォトニクス(QD - FRET)とマイクロフルイディクスの小説や、強力な統合を提示する。
要約
ゲノミクスの進歩により、細胞および分子レベルでの病態をターゲットにできる治療薬の開発に燃料を供給し続けます。細胞内で一般的に機能的、核酸ベースの治療法は、効率的な細胞内送達システムが必要です。 One広く採用されているアプローチは、劣化に対してそれを保護しながら、DNAの細胞取り込みを促進する、自己組織化電経由nanocomplexesを形成するために遺伝子キャリアーとの複合体のDNAにすることです。時期尚早の解離または過度に安定な結合が細胞への取り込みや治療効果に有害となるので、課題は、効率的な遺伝子キャリアの合理的な設計にあります。混合バルクで合成さNanocomplexesはnanocomplexesの大きさと安定性の異質性に起因した、開梱、細胞内及び人身売買の行動の多様な範囲を示した。そのような異質性が自己組織化の動態の正確な評価を妨げられ、トランスフェクション効率や生理活性にその物理的性質を相関させるのが困難に追加されます。我々は、層流の下nanocomplexの自己組織化のリアルタイム動態を特徴づけるために、新規ナノフォトニクスの収束(すなわち、QD - FRET)とマイクロフルイディクスを提示する。マイクロフルイディクスは、空間的に高時間分解能(〜ミリ秒)を使用してプロセスを分析するために十分に制御された微小環境を提供するのに対し、QD - FRETは、合成プロセス全体を通して、分子間相互作用との定量的測定値の発症の高感度指標を提供する。高分子nanocomplexesのモデルシステムの場合は、自己組織化プロセスの2つの異なるステージがこの分析プラットフォームによって捕獲された。マイクロスケールで得られた自己組織化プロセスの速度論的側面は、多くのマイクロおよびナノスケールのアプリケーションに関連するマイクロリアクターベースの反応は特に有用であろう。さらに、nanocomplexesは、マイクロ流体デバイスの適切な設計を使用してカスタマイズすることができ、結果として得られる高分子DNAのnanocomplexesが容易な構造と機能の関係を確立するために適用される可能性QD - FRET。
プロトコル
DNAのA.ビオチン化
プラスミドDNAを共有結合製造業者によって記載されたようグアニン特異的ビオチンラベルしたビオチン(Mirusバイオ、マディソン、ウィスコンシン州)が、DNAあたり1〜2ビオチンラベルを持つようにスケールされた。プラスミドDNA(pEGFPを- C1、4.9キロバイト、クロンテック、マウンテンビュー、カリフォルニア州)は、このプロトコルで標識した。
- 1μg/μLDNA溶液を作るためにDNaseフリー、RNaseフリーのTEバッファー(分子生物学グレードの品質)水にPDNAの所望量を溶解する。
- 以下の反応混合物を用いて標識反応を行う。 ITは、試薬最後のラベルを追加します。
100μgのDNA反応のための:
| DNaseフリー、RNaseフリーの水 | 75μL |
| 10X標識バッファー | 20μL |
| 1μg/μLDNA | 100μL |
| IT試薬ラベルを付けます。 | 5μL |
| 合計ボリューム | 200μL |
- 37反応℃で1時間インキュベートする。
- 標準的なプロトコルは、次のエタノールまたはイソプロパノール沈殿により標識された試料を精製する。
注:ゲルろ過ベースの列には、DNAの定量化や蛍光特性に影響を与える可能性が、高いUV吸光度または蛍光バックグラウンドにつながる可能性があります。
注:ビオチン化のレベルはHABAベースのテストによって決定されることがあります。
Cy5標識カチオン性ポリマーのB.標識
キトサンは(390 kDaの、脱アセチル化83.5%、バンソン、レッドモンド、ワシントン州)、本研究におけるモデルカチオン性ポリマーとして使用されました。キトサンポリマー骨格の空き第一級アミンは、Cy5標識NHS(アマシャムバイオサイエンス社、ピスカタウェイ、NJ)で標識した。
- Cy5の色素の完全な接合を容易にするために、Cy5標識NHSの必要量を計算するCy5のモル比というような:200:第一級アミンは1です。
- NaOHの添加により〜6.5にキトサン溶液(25mMの酢酸緩衝液中)のpHを調整します。 NHSの反応は、塩基性pHでより効率的ですが、ここキトサンの溶解度が動作pH範囲を制限することに注意してください。
- 撹拌しながら、ゆっくりとドロップによるドロップの方法でキトサン溶液にCy5標識NHSの計算量(1 mg / mlのDMSO)を追加します。
- 一晩室温で暗所で混合物をかき混ぜる。
- 精製するために、暗所で室温で1%の酢酸緩衝液に対して2時間10K MWCOスライド- A - Lyzer(Pierce)で透析。
- バッファーを交換し、暗所で室温で別の2時間を透析。
- バッファーを交換し、4℃で一晩透析·ダークでCを。
- ストア精製は、-20℃でポリマーを標識し
注:本研究では、標準曲線は670 nmでCy5標識NHSエステルの発光強度を測定することによって構成されている。標準曲線にCy5標識キトサンから670 nmで得られた発光を測定することにより、ラベルの密度を特徴づける。吸光度は、標識効率を決定するために使用されることがありますが、ここで実行されませんでした。
QD -標識DNAとCy5 -ポリマーのC.の準備
PDNAへの量子ドットの完全な接合を確保するためにQDにPDNAのモル比が過剰(2::QD≈1 PDNA)に保たれた。各PDNAに標識された量子ドットの数は、TEM像や他の同等の機能を介して推定することができる。我々の研究では、PDNAあたりのドット数は、TEMおよび単一分子分光法による〜1月3日と推定される。調製時1使用ミリポアのMilli - Q勾配の水(> 18.0 MW、0.2umのフィルタリング)。
- 所望のN / P比によると10μgのPDNA、DNA溶液に負のリン酸にキトサン溶液中のプロトン化アミンの理論上の比率のためのキトサンの必要量を計算します。
- ビオチン化PDNA液にストレプトアビジン官能605QDs(Qdot ®二605 ITK、Invitrogen社、カールスバッド、カリフォルニア州)を追加します。
- 15分間暗所に室温で溶液をインキュベートする。
- 最終的な音量200μLを作るために、50mMの硫酸ナトリウム溶液にQD -標識DNAを追加。
- 最終的な音量200μLを作るためにMilli - Q水で、所望のN / P比によると、Cy5標識キトサンを希釈する。
注意:可能な光退色を防ぐために、暗所で反応しておいてください。
重要:このカタログの量子ドットは、FRETの目的のために設計されているとしてQdot ®二605 ITK™ストレプトアビジンコンジュゲート(ITKシリーズ)を使用するように注意が必要です。正規のQdot ®二シリーズは、特に携帯電話ラベリングのために、非特異的結合を防ぐためにPEG層との共役複素数である。しかし、この追加のコーティングはrで、その結果、ドナー - アクセプターの距離を拡大エネルギー伝達効率をeduced。
標準的なフォトリソグラフィーを用いたSU - 8マスターズのD.作製
- Siウェーハはピラニア℃で5分間洗浄し、200で焼成です。
- 25μmのの指定されたマスターの厚さ、スピンコート用2000rpmでシリコンウェハ上にネガ型フォトレジスト(SU - 8 2025、マイクロケム、ニュートン、マサチューセッツ州)30秒。
- ソフトベーク65のランプとホットプレート上にウェーハ℃〜95℃/時間℃の
- マイクロチャネルの設計を含むマスク膜(CAD /アートサービス、バンドン、OR)を介して250mJ/cm 2のUV光(波長365nm)にさらす。
- 暴露後は、65のランプをホットプレート上にウエハを焼く° C / hrで95℃を
- SU - 8フォトレジストの現像剤を用いてウェハを開発する。
- パターン付きウェーハは、ハード65のランプをホットプレートで焼いている° C / hrで200℃室温へのダウンウェハその後徐々に涼しく、少なくとも5時間、200℃でウェハを保持する。
重要:SU - 8マスターベーキング処理が必要な時に徐々にランプが、そうでなければSU - 8構造体は、SU - 8構造が応力緩和により誘導されることがありますでシリコンウェハやクラックから剥離することがあります。
マスターとボンディングからカバーガラスにPDMSのE.レプリカ成形
- SU - 8のマスターは、秤量ボートに配置されます。
- 10のミックスシリコーンエラストマーと硬化剤(ポリ(ジメチルシロキサン)、PDMS、Sylgard 184、ダウコーニング、ミッドランド、MI):1の割合。
- SU - 8マスターにPDMSの混合物を注ぎ、気泡を除去するため真空デシケーター中で秤量ボートを残す。
- 1〜2時間、65℃でPDMSを治す。
- のSiマスターモールドからPDMSストリップピール。
- パンチのチャネル入口と流体デバイスのアウトレット。
- PDMSのストリップを洗浄し、エタノールとして空気乾燥してガラスをカバーする。
- 酸素プラズマによるクリーニングPDMSストリップとカバーガラスを(1分間20W)と同様に取り扱う。
- カバーガラスとすぐに結合PDMSストリップ。
- 95℃で一晩オーブンに結合マイクロ流体チップを残す。
重要:プラズマ処理と一晩ベーキングは、強化された接着強さには不可欠です。
F.モニターマイクロ流体デバイス中のDNA Nanocomplexesの形成
- 実験時のスムーズな流れを確保するために試薬をロードする前に、水とマイクロ流体チャネルを(マイクロ流体チャネル内に気泡がないことを確認するために)記入してください。
- ビデオで説明されているチューブを介して、二つの個々のガラスシリンジにQD -標識DNAとCy5標識キトサンソリューションを読み込みます。
- マイクロ流体デバイスには2つの入口とチューブを接続します。プロセス中に空気を導入しないように注意が必要です。層流条件下で、20nL/min(PHD - 2000シリンジポンプ、ホリストン、MA)で流量を設定します。
- 顕微鏡下でマイクロチャネルを確認してください。
- 流れは、(〜15〜20分)安定した量子ドットを介したフレットの場合はチャネルの中心に観察する必要があります。
- チャネルに沿って異なる位置で蛍光画像を(冷却CCD、Qimaging、BC、カナダ)を取る。
- ImageJとOriginLabのと蛍光画像を解析する。
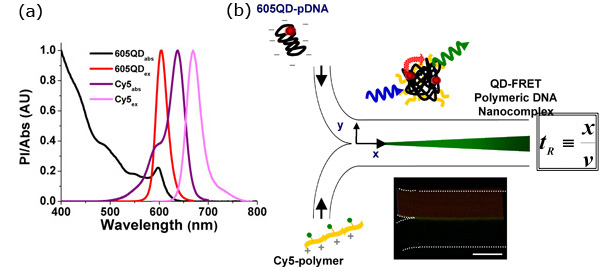
図1。 QD - FRETは、DNAのNanocomplexes自己組織化の発症の敏感な指標を提供
- 量子ドットを介した蛍光共鳴エネルギー移動(QD - FRET)は、DNAや遺伝子のキャリア間の相互作用の発症の明確な検出を可能にする、余分なまたは細胞内の環境のいずれかでpolyplex安定性の定量的かつ高感度指標を提供することができます。 FRETのペア、605QDとCy5は、ドナーとアクセプター間のスペクトルの重なりを最大化し、潜在的なクロストークを最小限に基づいて選択されました。このペアの場合は、フェルスター距離は69.4Åです3。
- QD - FRETのDNA nanocomplexesの自己集合。アニオンプラスミドDNA(PDNA)とカチオン性遺伝子キャリアは、それぞれ、QD(エネルギー供与体)とCy5(エネルギーアクセプター)で標識した。 QD - FRET nanocomplexesは、静電複合コアセルベーションを経て形成された。 488nmで励起されると、QD - FRETを介したCy5の発光は、コンパクトでそのままnanocomplexの形成を示した。滞留時間は、(T R)2つのストリームが調査対象の反応の位置、及び平均流の速度(v)に合致した場所から測定する距離(X)により算出することができる。混合、層流の性質上は、t Rの関数としての質量輸送の正確な計算を可能にする、インターフェース(各画像の中央)で行われます。時間分解能はapplieを変えることによって調整することができます。dフローのレート。 (挿入図)FRETの介在シグナルは、適用される流量に応じて数ミリ秒以内に起こる、その結合が急速であったことを示す、2つのストリームが満たさインターフェースですぐに観察された。スケールバー:100μmの。
ディスカッション
- 我々の仕事の意義:
- これは、単純なマイクロ流体チップ内にQD - FRET応答を通じてリアルタイム(ミリ秒の分解能)で高分子DNAのnanocomplex自己組織化の動態を監視する最初の試みです。
- マイクロフルイディクスは、空間的にDNAのnanocomplexの合成時にプロセスを分析するために十分に制御された微小環境を提供するのに対し、QD -媒介FRETは、分子間相互作用の発症のと自己組織化プロセス全体で高感度かつ定量的な指標を提供する。
- マイクロフルイディクスとナノフォトニクスの統合は、錯体形成反応の任意のタイプを調査するために新しいし、面白いアプローチを示唆している。
- 結果として得られる高分子DNAのnanocomplexesが容易な構造と機能の関係を確立するために適用される可能性QD - FRET。1,2
開示事項
The authors have nothing to disclose.
謝辞
NIHの助成HL89764、NSFの助成金0546012、0730503と0725528で提供される資金調達のサポート。
資料
| Name | Company | Catalog Number | Comments |
| SU-8 | MicroChem Corp. | SU-8 2025 | |
| PDMS | Dow Corning | Sylgard 184 | |
| Plasmid DNA | Clontech Laboratories | pEGFP-C1 | 4.9 kb, MW = 3.3 x 106 |
| LabelIT Biotin Labeling Kit | Mirus Bio LLC | MIR 3400 | Standard protocol yields labeling efficiency of approximately one label every 20-60 bp of double-stranded DNA, The density of labeling was adjusted in this work. |
| Streptavidin 605QD | Invitrogen | Qdot® 605 ITK® Streptavidin Conjugate | |
| Cy5-NHS Ester | Amersham | PA15101 | |
| Chitosan | Vanson | 390 kDa | 83.5% deacetylated |
| Cover Glass | Fisher Scientific | 12-545C | No. 1; Size: 40 x 22mm |
| Gastight Glass Syringe | Hamilton Co | TLL series | 50μL to 500μL depending on sample volume |
| Tygon Tubing | Small Parts, Inc. | 0.02 ID, 100ft | Tygon Tubes Microbore, 0.02 ID, 100ft |
| Connector | Small Parts, Inc. | HTX-23R | Customized in length of 0.750" |
| Syringe Pump | Harvard Apparatus | PHD-2000 | |
| CCD | QImaging | Intensified Retiga Cooled | |
| Microscope | Olympus Corporation | BX-51 | 100W mercury arc lamp |
| ImageJ | National Institutes of Health | v1.36b | http://rsb.info.nih.gov/ij |
| Origin Pro8 | OriginLab | Student Version | |
| Microscope Filter sets | Omega Optical | 475AF40 | Excitation filter in both channels |
| Microscope Filter sets | Omega Optical | 595AF60 | Emission filter in 605QD channel |
| Microscope Filter sets | Omega Optical | 670DF40 | Emission filter in QD-FRET channel |
| Microscope Filter sets | Omega Optical | 500 DRLP | Long pass dichroic in 605QD channel |
| Microscope Filter sets | Omega Optical | 595DRLP | Long pass dichroic in QD-FRET channel |
参考文献
- Ho, Y. P., Chen, H. H., Leong, K. W., Wang, T. H. Evaluating the intracellular stability and unpacking of DNA nanocomplexes by quantum dots-FRET. J Control Release. 116, 83-89 (2006).
- Chen, H. H. Quantitative comparison of intracellular unpacking kinetics of polyplexes by a model constructed from quantum dot-FRET. Mol. Ther. 16, 324-332 (2008).
- Zhang, C. Y., Yeh, H. C., Kuroki, M., Wang, T. H. Single-Quantum-Dot-Based DNA Nanosensor. Nat Mat. 4, 826-831 (2005).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved