Method Article
Combinando QD-FRET e microfluidica per il monitoraggio Nanocomplex DNA di auto-assemblaggio in tempo reale
In questo articolo
Riepilogo
Vi presentiamo un nuovo e potente integrazione di nanofotonica (QD-FRET) e microfluidica per studiare la formazione di polyplexes polielettrolita, che si prevede di fornire un migliore controllo e la sintesi di polyplexes uniforme e personalizzabili per i futuri nucleici terapie a base di acido.
Abstract
I progressi nella genomica continuano ad alimentare lo sviluppo di terapie che possono bersaglio patogenesi a livello cellulare e molecolare. Tipicamente funzionale all'interno della cellula, acidi nucleici terapie a base richiedono un efficiente sistema di distribuzione intracellulare. Un metodo ampiamente adottato è quello del DNA complesso con un vettore gene per formare nanocomplexes tramite auto-assemblaggio elettrostatico, facilitando l'assorbimento cellulare di DNA, mentre la protezione contro la degradazione. La sfida consiste nella progettazione razionale dei vettori gene efficiente, dal momento prematuro dissociazione o eccessivamente vincolante stabile sarebbe dannoso per la captazione cellulare e l'efficacia terapeutica. Nanocomplexes sintetizzato mescolando di massa ha mostrato una vasta gamma di comportamenti intracellulari disimballaggio e il traffico, che è stata attribuita alla eterogeneità di dimensioni e la stabilità del nanocomplexes. Tale eterogeneità ostacola la corretta valutazione del self-assembly cinetica e aggiunge alla difficoltà di correlare le loro proprietà fisiche di efficienza di trasfezione o bioactivities. Vi presentiamo una convergenza romanzo di nanofotonica (cioè QD-FRET) e microfluidica per caratterizzare in tempo reale cinetica del nanocomplex self-assembly sotto flusso laminare. QD-FRET fornisce un'indicazione molto sensibile della insorgenza di interazioni molecolari e misura quantitativa per tutto il processo di sintesi, mentre microfluidica offre una ben controllata microambiente per analizzare spazialmente il processo con alta risoluzione temporale (~ millisecondi). Per il sistema modello di nanocomplexes polimerici, due fasi distinte nel processo di auto-assemblaggio sono stati catturati da questa piattaforma analitica. L'aspetto cinetico del processo di auto-assemblaggio presso la microscala sarebbe particolarmente utile per microreattore basato reazioni che sono rilevanti per molte applicazioni micro-e nano-scala. Inoltre, nanocomplexes può essere personalizzato attraverso la corretta progettazione di dispositivi microfludic, e la conseguente QD-FRET nanocomplexes DNA polimerici può essere facilmente applicato per stabilire relazioni struttura-funzione.
Protocollo
A. biotinilazione del DNA
DNA plasmidico sono state covalentemente biotinilato con guanina-specifiche etichette biotina, come descritto dal produttore (Mirus Bio, Madison, WI), ma in scala di avere ~ 1-2 etichette biotina al DNA. DNA plasmidico (pEGFP-C1, 4,9 kb, Clontech, Mountain View, CA) è stato etichettato in questo protocollo.
- Sciogliere quantità desiderata di PDNA in TE buffer di DNasi e senza RNasi-free (biologia molecolare-grado di qualità) di acqua per ottenere una soluzione 1μg/μL DNA.
- Condurre la reazione di marcatura usando le miscele di reazione seguente. Aggiungere l'etichetta IT reagente scorso.
Per reazione il DNA 100μg:
| DNasi RNasi-free e senza acqua | 75 microlitri |
| Etichettatura Buffer 10X A | 20 l |
| 1μg/μL DNA | 100 ul |
| Etichetta IT reagente | 5 microlitri |
| Volume totale | 200 l |
- Incubare la reazione a 37 ° C per 1 ora.
- Purificare il campione etichettato da etanolo o isopropanolo precipitazione seguenti protocolli standard.
Nota: le colonne di filtrazione gel a base può portare ad assorbimento UV ad alta o fluorescenza di fondo, che possono influenzare la quantificazione del DNA o la caratterizzazione fluorescenza.
Nota: Il livello di biotinilazione può essere determinata da HABA a base di test.
B. Etichettatura dei Cy5-polimeri cationici
Chitosano (390 kDa, 83,5% deacetilata, Vanson, Redmond, WA) è stato utilizzato come modello di un polimero cationico in questo studio. Le ammine primarie libero sulla dorsale chitosano polimero sono stati etichettati con Cy5-NHS (Amersham Biosciences, Piscataway, NJ).
- Per facilitare la coniugazione completa di Cy5 colorante, calcolare in modo tale che il rapporto molare tra Cy5 la quantità richiesta di Cy5-SSN: ammine primarie è di 1: 200.
- Regolare il pH della soluzione di chitosano (in tampone acetato 25mM) per ~ 6,5 con l'aggiunta di NaOH. Si noti che la reazione NHS è più efficiente a pH basico, ma la solubilità del chitosano qui limita l'intervallo di pH di lavoro.
- Sotto agitazione, aggiungere lentamente la quantità calcolata di Cy5-NHS (1 mg / ml DMSO) alla soluzione chitosano in una goccia a goccia modo.
- Agitare la miscela al buio a temperatura ambiente per una notte.
- Per purificare, Dializzare con 10k MWCO Slide-a-Lyzer (Pierce) per 2 ore contro tampone acetato 1% a temperatura ambiente al buio.
- Sostituire il tampone e Dializzare altre 2 ore a temperatura ambiente al buio.
- Sostituire il tampone e Dializzare notte a 4 ° C al buio.
- Conservare purificato etichettati polimero a -20 ° C.
Nota: In questo studio, una curva standard è costruito misurando l'intensità di emissione di Cy5-NHS estere a 670 nm. Caratterizzano la densità etichettatura misurando l'emissione ottenuti a 670 nm da Cy5 marcato chitosano nella curva standard. Assorbanza può anche essere usato per determinare l'efficienza etichettatura, ma non è stato eseguito qui.
Preparazione C. QD marcato DNA e Cy5-Polymer
Il rapporto molare di PDNA di QD è stato mantenuto in eccesso (PDNA: QD ≈ 1: 2) per garantire la coniugazione completa di QD di PDNA. Il numero di QD etichetta su ogni PDNA può essere stimata attraverso immagini TEM o altre strutture equivalenti. Nel nostro studio, il numero di QD per PDNA è stimato in ~ 1-3 da TEM e spettroscopia di singola molecola. 1 Utilizzare Millipore Milli-Q acqua pendenza (> 18,0 MW, 0.2um filtrata) durante la preparazione.
- Calcolare la quantità di chitosano per 10μg PDNA secondo desiderato N / P, il rapporto teorico di ammine protonate nella soluzione di chitosano per i fosfati negativo nella soluzione del DNA.
- Aggiungi streptavidina-funzionalizzati 605QDs (Qdot 605 ITK, Invitrogen, Carlsbad, CA) nella soluzione biotinilato PDNA.
- Incubare la soluzione a temperatura ambiente al buio per 15 min.
- Aggiungere il QD marcato DNA in 50 mM soluzione di solfato di sodio per rendere il volume finale 200μL.
- Diluire Cy5-chitosano, secondo desiderato N / P, con acqua Milli-Q per rendere il volume finale 200μL.
Nota: Tenere la reazione di scuro per impedire alla photobleaching possibile.
Importante: Siate cauti di utilizzare il Qdot 605 ITK ™ streptavidina coniugato (la serie ITK), come Punti quantici in questo catalogo sono progettati per lo scopo di FRET. La serie Qdot regolari sono coniugati con uno strato PEG per evitare legame non specifico, in particolare per l'etichettatura cellulare. Tuttavia, questa ulteriore rivestimento allarga il donatore-accettore distanza, con conseguente reduced trasferimento di efficienza energetica.
D. Realizzazione della SU-8 Masters Utilizzando Fotolitografia standard
- Wafer di silicio è piranha pulito e cotto a 200 ° C per 5 min.
- Per lo spessore di 25 micron padrone designato, cappotto girare il photoresist negativo (SU-8 2025, MicroChem, Newton, MA) su wafer di silicio a 2000 rpm per 30 sec.
- Morbido cuocere la cialda su una piastra con una rampa di 65 ° C / hr a 95 ° C.
- Esporre ai raggi UV (365nm) per 250mJ/cm 2 a un film maschera (CAD / Servizi Arte, Bandon, OR) contenente la progettazione di microcanali.
- Post-esposizione cuocere la cialda su una piastra con una rampa di 65 ° C / hr a 95 ° C.
- Sviluppare il wafer utilizzando SU-8 sviluppatore photoresist.
- Il wafer fantasia è duro cotta su una piastra con una rampa di 65 ° C / hr a 200 ° C. Mantenere il wafer a 200 ° C per almeno 5 ore, poi gradualmente raffreddare la cialda a temperatura ambiente.
Importante: rampa graduale durante il SU-8 processo di cottura master è necessario, altrimenti il SU-8, la struttura può staccato dal wafer di silicio o crepe sulla struttura di SU-8 può essere indotta da stress-release.
E. Stampaggio Replica del PDMS dai maestri e l'adesione al vetro di copertura
- Il SU-8 master è posto in una barca di pesatura.
- Mix di silicone elastomero e catalizzatore (Poly (dimetilsiloxano), PDMS, Sylgard 184, Dow Corning, Midland, MI) in un 10: 1 rapporto.
- Versare il composto PDMS sul SU-8 master e lasciare la barca pesa in un essiccatore a vuoto per rimuovere le bolle.
- Curare il PDMS a 65 ° C per 1-2 ore.
- Sbucciare la striscia PDMS dallo stampo maestro Si.
- Punch insenature canale e uscite del dispositivo fluidica.
- Pulire la striscia di PDMS e vetro di copertura con etanolo e poi asciugare all'aria.
- Trattare la pulizia PDMS striscia di vetro e coprire con plasma di ossigeno (20 W per 1 minuto).
- Immediatamente la striscia PDMS legame con il vetro di copertura.
- Lascia il chip incollato microfluidica in forno a 95 ° C per una notte.
Importante: il trattamento al plasma e da forno durante la notte sono essenziali per la forza di legame maggiore.
F. Monitorare la formazione di DNA Nanocomplexes Nel dispositivo a microfluidi
- Riempire il canale microfluidica con acqua (per assicurarsi che ci sia assenza di bolle all'interno del canale microfluidica), prima di caricare i reagenti per assicurare un flusso regolare durante l'esperimento.
- Caricare il DNA QD-etichettati e marcati con Cy5 soluzioni di chitosano in due siringhe di vetro singolo, attraverso il tubo descritto nel video.
- Collegare il tubo con le due insenature di dispositivi microfluidici. Essere cauti a non introdurre aria durante il processo. Impostare la velocità di flusso a 20nL/min (PHD-2000 pompa a siringa, Holliston, MA), in condizioni di flusso laminare.
- Controllare la microcanali sotto il microscopio.
- Quando il flusso è stabile (~ 15 a 20 minuti), QD-mediata FRET devono essere osservate al centro del canale.
- Scatta foto di fluorescenza (CCD raffreddata, Qimaging, BC, Canada) in luoghi diversi lungo il canale.
- Analizzare le immagini a fluorescenza con ImageJ e OriginLab.
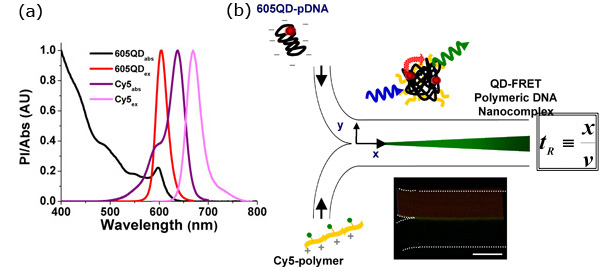
Figura 1. QD-FRET fornisce un'indicazione sensibile l'insorgenza di Nanocomplexes DNA auto-assemblaggio
- Quantum dot-mediato trasferimento di energia di risonanza di fluorescenza (QD-FRET) in grado di fornire una indicazione quantitativa e altamente sensibile di stabilità Polyplex sia in ambienti extra o intra-cellulari, permettendo per la rilevazione inequivocabile l'inizio delle interazioni tra il DNA e il vettore genico. Il FRET coppia, 605QD e Cy5, è stato scelto sulla base di massimizzare sovrapposizione spettrale tra il donatore e accettore e riducendo al minimo il potenziale cross-talk. Per questa coppia, la distanza di Förster è 69.4Å 3.
- Auto-assemblaggio del QD-FRET nanocomplexes DNA. Anionici DNA plasmidico (PDNA) e il vettore gene cationici sono stati etichettati con QD (donatore di energia) e Cy5 (energia accettore), rispettivamente. QD-FRET nanocomplexes si sono formati attraverso elettrostatiche coacervazione complessi. Al momento di eccitazione a 488 nm, QD-FRET-mediata Cy5 emissione indicati formazione di un nanocomplex compatta e intatta. Il tempo di permanenza (t R) può essere calcolato in base alla distanza (x) che misura, da dove i due flussi si incontrano per la posizione di reazione sotto inchiesta, e la velocità di flusso medio (v). A causa della natura del flusso laminare, la miscelazione avviene esclusivamente a livello di interfaccia (centro di ogni immagine), che consente il calcolo preciso di trasporto di massa in funzione di t R. Risoluzione temporale può essere regolata variando la applied portate. (Interno) FRET-mediata segnale è stato osservato immediatamente a livello di interfaccia, quando i due corsi d'acqua ha incontrato, a indicare che il legame è stato rapido, che si verificano in pochi millisecondi in base ai tassi di flusso applicati. Scala Bar: 100μm.
Discussione
- Significato del nostro lavoro:
- Questo è il primo tentativo di monitorare polimerici DNA nanocomplex auto-assemblaggio cinetica in tempo reale (millisecondi risoluzione) attraverso QD-FRET risposte all'interno di un chip semplice microfluidica.
- QD-mediata FRET fornisce un'indicazione molto sensibile e quantitativa l'insorgenza di interazioni molecolari e in tutto il processo di self-assembly, mentre microfluidica offre una ben controllata microambiente per analizzare spazialmente il processo durante la sintesi del DNA nanocomplex.
- L'integrazione di microfluidica e nanofotonica suggerisce un approccio nuovo e interessante per indagare su qualunque tipo di reazioni di complessazione.
- La risultante QD-FRET nanocomplexes DNA polimerici potrebbe essere facilmente applicato per stabilire relazioni struttura-funzione. 1,2
Divulgazioni
The authors have nothing to disclose.
Riconoscimenti
Finanziamento supporto fornito dal NIH concedere HL89764, borse di NSF 0546012, 0730503 e 0725528.
Materiali
| Name | Company | Catalog Number | Comments |
| SU-8 | MicroChem Corp. | SU-8 2025 | |
| PDMS | Dow Corning | Sylgard 184 | |
| Plasmid DNA | Clontech Laboratories | pEGFP-C1 | 4.9 kb, MW = 3.3 x 106 |
| LabelIT Biotin Labeling Kit | Mirus Bio LLC | MIR 3400 | Standard protocol yields labeling efficiency of approximately one label every 20-60 bp of double-stranded DNA, The density of labeling was adjusted in this work. |
| Streptavidin 605QD | Invitrogen | Qdot® 605 ITK® Streptavidin Conjugate | |
| Cy5-NHS Ester | Amersham | PA15101 | |
| Chitosan | Vanson | 390 kDa | 83.5% deacetylated |
| Cover Glass | Fisher Scientific | 12-545C | No. 1; Size: 40 x 22mm |
| Gastight Glass Syringe | Hamilton Co | TLL series | 50μL to 500μL depending on sample volume |
| Tygon Tubing | Small Parts, Inc. | 0.02 ID, 100ft | Tygon Tubes Microbore, 0.02 ID, 100ft |
| Connector | Small Parts, Inc. | HTX-23R | Customized in length of 0.750" |
| Syringe Pump | Harvard Apparatus | PHD-2000 | |
| CCD | QImaging | Intensified Retiga Cooled | |
| Microscope | Olympus Corporation | BX-51 | 100W mercury arc lamp |
| ImageJ | National Institutes of Health | v1.36b | http://rsb.info.nih.gov/ij |
| Origin Pro8 | OriginLab | Student Version | |
| Microscope Filter sets | Omega Optical | 475AF40 | Excitation filter in both channels |
| Microscope Filter sets | Omega Optical | 595AF60 | Emission filter in 605QD channel |
| Microscope Filter sets | Omega Optical | 670DF40 | Emission filter in QD-FRET channel |
| Microscope Filter sets | Omega Optical | 500 DRLP | Long pass dichroic in 605QD channel |
| Microscope Filter sets | Omega Optical | 595DRLP | Long pass dichroic in QD-FRET channel |
Riferimenti
- Ho, Y. P., Chen, H. H., Leong, K. W., Wang, T. H. Evaluating the intracellular stability and unpacking of DNA nanocomplexes by quantum dots-FRET. J Control Release. 116, 83-89 (2006).
- Chen, H. H. Quantitative comparison of intracellular unpacking kinetics of polyplexes by a model constructed from quantum dot-FRET. Mol. Ther. 16, 324-332 (2008).
- Zhang, C. Y., Yeh, H. C., Kuroki, M., Wang, T. H. Single-Quantum-Dot-Based DNA Nanosensor. Nat Mat. 4, 826-831 (2005).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon