Method Article
バーコード酵母ライブラリーの競争力のあるゲノム画面
* これらの著者は同等に貢献しました
要約
我々は包括的、公平なゲノムワイドな遺伝子薬剤と遺伝子 - 環境相互作用を理解するために画面を開発しました。これらの変異体のコレクションをスクリーニングするための方法が提示されます。
要約
次世代シーケンシング技術の進歩のおかげで、我々は、ほぼ毎日新しいゲノム配列へのアクセス権を持っている。これらの進歩のテンポがより大きな深さと幅広さを約束し、加速しています。これらの飛躍的進歩を踏まえて、遺伝子の機能を定義するために、高速、並列メソッドの必要性がますます重要になります。酵母やE.におけるゲノムワイドな欠失変異体のコレクション大腸菌の遺伝子の機能の機能解析のための主力を務めているが、この方法はスケーラブルではない、現在の遺伝子欠失のアプローチは、ゲノムを構成する数千の遺伝子のそれぞれを削除し、検証する必要があります。この作業が完了した後にのみ、我々は、高スループットの表現型解析を挟むこともできます。過去10年間で、私たちの研究室では競争力のある、小型化、ハイスループットのゲノムワイドな並列実行できるアッセイのポートフォリオを改良しました。この並列化では、バーコードは、突然変異のプロキシとして機能しているためDNA"タグ"、または各変異体に"バーコード"を含めることが可能であり、1は変異型フィットネスを評価するためにバーコードの存在量を測定することができます。本研究では、我々はDNA配列やバーコード変異体コレクションの間のギャップを埋めるために求めている。これを達成するために我々は、新たに配列決定するまでのパラレルバーコードのアッセイを開きますが、悪い微生物を特徴とする複合トランスポゾン混乱 - バーコードのアプローチを紹介する。我々は新しいカンジダバーコード混乱のコレクションを発表し、どのように両方のマイクロアレイベースの、次世代シーケンシングベースのプラットフォームを記述するこのアプローチを説明するために10,000を集めるために使用することができます-一回の実験で1,000,000遺伝子-遺伝子と薬剤遺伝子の相互作用。
プロトコル
1。背景情報
バーコードタグを運ぶ変異体を生成するには、いくつかの方法があります。現在のゴールドスタンダードは、ラボのコンソーシアムによって作成され、2002年1年に完成した酵母のノックアウト(YKO)コレクションです。オリジナルYKOが導入されて以来、他の酵母のコレクションが生成されている。異なる菌株の背景に、過剰発現構築物を使用して、そしてそのようなEと他の微生物に大腸菌 2。並行して、バーコードshRNAライブラリを作成するための努力が急速に進んでおり、そして実際に、これらの哺乳類のコレクションのための設計原理の多くは、酵母から採用されている。バーコードトランスポゾンは、体系的変異体のコレクションを作成するための迅速、広範囲に適用可能な戦略となることができる方法を示すために、我々は最近、人間の真菌病原体、 カンジダアルビカンスで作成したコレクションに焦点を当てる。カンジダについて私たちの仕事はS.でバーコード画面の成功に基づいていた出芽酵母 、および例の生物としてここで使用されています。サンプルプロトコルは、マイナーな修正を加えて懸濁培養で成長させることができる任意の生物をスクリーニングするために使用することができます。いくつかの生物は、変換の必要な高い率と完全な欠失変異体を作成するために必要な効率的な有糸分裂組換えを持っているので、それに応じて我々は、ゲノムDNAライブラリーを突然変異を起こさせるためにin vitroでトランスポゾン突然変異誘発を使用するプロトコルを開発し、Candida albicansの 3にこれらのバーコードゲノム断片を変換、4。オリジナルYKOコレクションの成功に触発され、遺伝子ネットワークの性質についての基本的な発見で、その役割5-8、ゲノムワイドなハプロ不全9、創薬ターゲットとアクション10,11のメカニズム、及びゲノムの全遺伝子の本質我々は他の微生物にこのアプローチを拡大する予測12は、非常に実りあるでしょう。
以下のプロトコルは、所望の変異は、コレクションの作成 (例:YKOまたはカンジダ混乱のコレクション)と個別にアーカイブされた菌株として利用してしていることを前提としています。ひずみ建設の詳細な説明については1,13,14を参照してください。
2。単一のプールに個々の変異体を組み合わせる
- 一週間は、プールされた細胞のアリコートを(-80 ° Cで無期限に保存できる)を生成することができます。
- 完全に興味のある株について凍結グリセロールストックを融解が、細胞が> 2時間のために解凍のままさせてください。
- 2 70%エタノール浴中のディップ(例えばピペットチップボックスの蓋)、火炎ピンのツールと1分間クールに続いて96ウェルピンツール、残りの細胞を除去するために水でピンツールを浸漬し、滅菌する。ピンツール離れてエタノール浴から炎に注意してください。エタノール浴のレベルは、すべてのキャリーオーバー細胞が燃え上がると削除されるように、水浴中でレベルを上回っている必要があります。すべての4〜6 pinningsは、水を交換してください。
- 軽く解凍した96ウェルプレート、スワールに滅菌96ウェルピンのツールを挿入し、適切な抗生物質を含むYPD寒天を含むヌンクオムニトレイにセルを転送する。彼らは30 ° C(2 - 3D)で最大サイズに達するまでコロニーを生育させます。プレートを節約するために、我々は、〜384株で、単一のオムニトレイに4枚の96ウェルプレートを統合することが最も便利です。
- コロニーが成長した後、万一、不足や成長の遅い株のことに注意してくださいとでこれらをレーピン〜株の残りの部分として、細胞塊を2倍。
- 微生物の環境(炎と無菌実験機器と、5〜10 mlのメディアとの洪水のプレートでの作業、セルスプレッダーで5分間再懸濁しますコロニーを浸す。50 mlコニカル遠心チューブに液体を加えた細胞を注ぎ、とにグリセロールを追加する7%から15%DMSO(vol / vol)の。
- プールのOD 600を測定し、15%グリセロールまたは7%のDMSOを含む培地で50 OD 600 / mlの最終濃度に(希釈または遠心分離によって)調整する。
- -80℃でPCRストリップチューブや凍結で40μlの量で分注し℃に
3。実験的なプールの成長
この手順は、 図1で概説されます。
- 氷上で解凍プールのアリコート(PCRチューブで)。ロボットを使用していない場合、手順5に進みます。
- すぐに48ウェルプレートに700μlの総体積で0.0625のOD 600で接種、薬剤または選択の状態とメディアへ(gently!)プールを希釈する。プレート上の少なくとも1つの適切な溶媒コントロールが含まれています。 (よく1より大きい値をすなわち)成長の5世代を超えて拡張実験のために、隣接するメディアまたは選択の条件と井戸が、NOの細胞を埋める。
- プラスチック製のプレートシールでシール、条件は好気性の成長(例えば、非発酵性炭素源を)必要とする場合、各ウェルの側に向かって膜のシールでピアスの穴に21ゲージ針を使用。
- 実験的にdetermiで30℃で振盪、分光光度計で成長するNEDが揺れレジメン(例えば揺れ再開、最高の設定(または温度制御シェーカー)、読み取り井戸で14分を横に振る)。細胞懸濁液の一部は、ユーザー定義の生成時のポイントは、通常5、10、15と成長の20世代でロボット甲板上でコールドプレート上にロボットが収穫して保存することができます。 (詳細はお問い合わせC. NislowまたはG.イェーヴァー用http://med.stanford.edu/sgtc/technology/access.html、)。
- 手動の細胞増殖のために、250mlの培養フラスコに0.002の開始OD 600で50 mlの培養液に接種する。細胞が成長の〜10世代のために2.0の最終OD 600を ( サッカロマイセスまたはカンジダ用)に到達するまで、250 rpmで30℃で振とうする。成長の追加の世代には新鮮なフラスコに0.02に戻って2.0のOD600で希釈細胞によって得ることができます。
- セイフ-ロックのマイクロチューブに各サンプル/時間ポイントのための細胞の約2 OD 600単位以上の収穫。
注:必ず、説明したように1.5 mlチューブや処理に直接冷凍庫のアリコートからプール1-2 OD 600を追加することで、新しく作成されたプール内の初期ひずみの表現を評価するための出発細胞サンプル(例:"T0の時点")を収集以下に。
4。ゲノムDNA抽出、PCRやマイクロアレイハイブリダイゼーションまたはシーケンシング
- (議定書私は酵母のDNAを使用している場合)、または関心の生物(固有の別の適当な方法アルコール沈殿が続く標準のフェノール/クロロホルム抽出の仕組みは、メーカーの指示に従ってZymo研究YeaStarキットを用いて細胞のOD 600〜2からゲノムDNAを精製するよく多様な微生物のため)。 YeaStarキットを使用している場合、プロトコルで指定された1 × TE 60μLの代わりに、0.1 × TE300μlでDNAを溶出させる。ゲノムDNAは、-80℃で無期限に保存することができます。
- 33μLのddH 2 O、MgCl 2をせずに6μlの10 × PCR緩衝液、3μlの50mMのMgCl 2を 、1.2μL:次のように反応条件と、各サンプルに2つのPCR反応は、uptagsとdowntags用に一つずつを、設定する15μlの10 mMのdNTPsを、1.2μlの50μMUpまたはDownプライマーミックス、0.6μlの5 U /μlのTaqポリメラーゼ、〜0.1μgのゲノムDNA。総体積は60μlです。その後、72℃3分、そして4℃で保持℃、94℃で3分間、94 30サイクル° C 30秒、55℃30秒、72℃30秒:次の条件の下でThermocycleゲル上で得られたPCR産物をチェックし、両方のPCRのための60 bpの製品は、バーコードのシークエンシング用のアンプリコン)のためのハイブリダイゼーションおよび130bpのために使用されるアンプリコンのために期待されています。 PCR産物は、-80℃で無期限で保存することができる。
- 42〜Prewarmハイブリダイゼーションオーブンの温度℃、氷 - 水スラリーを含む沸騰水浴と氷のバケツを設定する。
- 徐々に120μlの1 ×ハイブリダイゼーション用緩衝液で充填してプリウェット配列。
- 42でハイブリダイゼーション緩衝液中でインキュベート℃、10分間20回転。
- 75μlの2Xハイブリダイゼーション緩衝液、0.5μlのB213コントロールのオリゴヌクレオチド(0.2 FM /μl)を、12μlの混合オリゴヌクレオチド(12.5分/μL)、μlの3を次のように、ハイブリダイゼーションサンプルあたりのミックスに加えて、バッファとして余分な1個の90μlを準備するロック上位0.5 mlチューブで50倍デンハルト溶液)。
- 150μLの全体積に対して120μlのハイブリダイゼーション混合物に、30μlのuptag PCRおよび30μlのdowntag PCRを追加。 2分間煮て、少なくとも2分間氷水に設定する。簡単に言えば、使用前にチューブをスピン。
- 配列からのプレハイブリダイゼーションバッファーを除去し、90μlのハイブリダイゼーション/ PCRミックスを追加してください。蒸発を防ぐために、タフスポットを持つ配列のガスケットをカバーしています。 42℃16時間ハイブリダイズ° C、20 rpmで。
- 次のようにたては、600サンプルごとμLビオチンラベリングミックスに加えて余分なものを準備します。180μlの20X SSPE、12μlの50Xデンハルト、6μlの1パーセントのTween 20(vol / vol)の、1μlの1 mg / mLのストレプトアビジン - フィコエリスリン、401μlをのddH 2 O暗闇の中ですべてのストレプトアビジン- PEのサンプルを保管してください。一定分量を2 mlのチューブに600μlの。チップからタフスポットを削除します。
- ゆっくりとピペットで配列からハイブリダイゼーションミックスを削除し、120μlの洗浄A.内閣総理アフィメトリクス流体の駅でマイクロアレイを埋める。
- (1サイクル、2ミックス)染色する前に、Bの温度42 ° Cの代わりを洗うウォッシュ1余分なステップ:次の点を変更"遺伝子Flex_Sv3_450"プロトコルを使用して、製造元の指示に従ってアフィメトリクス流体ステーションを使用してアレイを洗う40 ° C、° Cの代わりに25℃、42℃染色それは、(参照15頁396参照)後に手動でのハイブリダイゼーションの洗浄、ビオチンの染色、およびポスト染色の洗浄を行うことも可能です。流体操作の後、流体の駅"SHUTDOWN_450"プロトコルを実行してください。
- 洗浄に続いて、に気泡が存在していないことを確認してください。泡が消えるまでに必要なゆっくりと90μlの洗浄とピペットを追加した場合。いかなる商標またはSMがある場合アレイ表面上udgesは、イソプロパノールでガラス窓をきれいにし、糸くずの出ないティッシュ。ガスケット/セプタムを介して新鮮なタフスポットを適用すると、スキャナでのアレイを配置。
- 560nmの発光波長でアフィメトリクスGeneArrayスキャナでスキャン。
5。アレイ解析(Candida albicansの混乱のコレクションを使用して得られた例については、図2を参照)
- 外れ値のマスキング:アフィメトリクスTAG4の配列が含まれているので5がランダムにマスクして破棄することができます複製から逸脱するいかなるバーのプローブを分散した各バーコードの補体の複製。 4他のと比較して、その信号に基づいて、外れ値と思われる各配列の機能では、この機能を複製のためにこれを行うには、、当社のソフトウェアは、最初の容疑者で25の機能のマトリックスを生成する、疑わしい異常値を囲む5の機能を調べ、中央の機能。この地域の> 25分の13のプローブが10%以上彼らの個々の彼らのトリミングされた複製の平均値の各(中間にある三つの複製の平均値、複製最高と最低を除く)と異なる場合は、このプローブは、さらなる分析から破棄されます。そのような外れ値が最も頻繁にポストハイブリダイゼーションの洗浄の不整合の結果なので、疑わしいプローブを含む領域を、我々は展開したり、"パッド"。などで定義されている5 -プローブの半径内のすべてのプローブを含むことにより、パッドなどのプローブ、((X 1 - X 2)2 +(Y 1 - Y 2)2)½ <6ここで、X 1、X 2、Y 1、およびY 2は、2つの機能のためのx座標とy座標です。最後に、標準機能のピクセル/機能のピクセルを平均の偏差(Affymetrix社のアレイ用の。CELファイルに含まれる)のための機能を捨てる。外れ値を除去した後、残りのすべての平均強度値は複製されます。
- 使用不能タグの削除:低強度の値でタグと低品質の結果が得られますし、削除する必要があります。これらの低強度のプローブの除外カットオフは、次のように計算できます。
- 配列の任意の治療制御のペアの場合は、I Cは制御の強さである、I Tは、治療の強さであり、aとb、それぞれのタグのためにログインする2(( 私はC - B G)/(I T - B G))を計算gは割り当てられていないタグプローブの平均輝度です。
- 株によるuptagとdowntag比率をペアにし、それぞれのタグのペアに対して、2つのサンプルで二つのタグのための最小強度を取る。この最小強度が比のペアを並べ替えます。
- ランク比率のペアで50のスライディングウィンドウのサイズを使用して、ウィンドウ内比率のペアをuptagとdowntagの相関を計算する。また、前の手順で計算された最小強度の平均値を計算する。
- 25ペアでウィンドウをスライドさせ、すべてのペアが交差されるまで、前の手順を繰り返します。
- すべてのウィンドウでuptag - downtag相関対平均最小強度をプロットします。
- 最後に、強度のしきい値を選択して、一般的に我々は相関関係が最初にその最大レベルの80%に達する強度の値を使用してください。フラグとさらなる解析から、このカットオフより下のタグを削除。
- 彩度補正:バーコードマイクロアレイ上の各機能が飽和状態になる可能性があるため、TAG4アレイ上の信号が直線的にタグの濃度に関連していない。この飽和は、リファレンス16に記載のプロトコルに従うために修正する。
- 配列の正規化:各配列に対して、個別にuptagsとdowntagsを正規化する。正規化変位値に、増加する強さの順にuptagsとdowntags各配列から取得した値をランク付けする。 uptagsとdowntagsのセットごとに正規化を意味すると、平均値で割ります。すべてのアレイの平均正規化は、それぞれの配列の平均値に生データを変換する第二段階の次のようになります。
- 制御処理の比較のために感度のスコアを計算する:感度の指標としてログ2比を使用するには:それぞれの菌株については、ログに記録2((μC - B G)/(μT - B g)を )計算し、ここで、μcは対照サンプルの強度を意味し、μtは治療のサンプルの平均強度であり、aとbのgは割り当てられていないプローブの平均輝度です。ポジティブログ2比を有する株は、治療に敏感である、と耐性があるものは、負の対数2比を持っている。
6。シークエンシングによってバーコード酵母株の適応度の評価
- としてマイクロアレイのために説明する削除プールからDNAを分離する。
- (イルミナプライマーとアンプリコンの図については、図3の表を参照)イルミナフローセルへのハイブリダイゼーションに必要な一般的なバーコードのプライマーの配列との配列から成る複合的プライマーを用いて、各20 - merのuptagのバーコードを増幅する。これらのプライマーは、追加の精製なしで脱塩に使用することができます。 PCRは、以下の条件でインビトロジェンプラチナPCRスーパーミックス(カタログ番号11306から016)を用いて、100μLで実行されます:95℃/ 3分、94の25サイクル° C/30秒、55℃C/30秒、68 ° C/30秒、68℃C/10分が続く。
- キアゲンアプライ96 UF PCR精製キット(カタログ番号28051)でPCR産物(〜130bp)を精製する。
- PCR精製に続いて、インビトロジェンクワント- ITのdsDNA BRアッセイキット(カタログ番号Q32853)でDNAを定量化する。 280分の260の測定値に依存しないようにしてください!
- 正規化されたDNAの10μg/mlとプール等しいボリュームにDNA濃度を正規化する。
- 使用電圧に応じて3-4時間12%ポリアクリルアミドTBEゲル上で別のプールされたDNA。
- 30分間エチジウムブロマイド(SYBRグリーンも同様に動作するはずです)でゲルを染色する。
- (適切な顔面保護を着用)長波長の紫外線ライトで目的のバンドを見つけ、それを切り出し、ときめきを使用してDNAを抽出し、エタノール沈殿に続く方法17を浸す。
- 適切なサイズのDNA(130bp)が分離され、そのプライマーは、アジレントバイオアナライザの高感度DNAキット(カタログ番号5067から4626)を使用して削除されていることを確認してください。
- サンプルのシーケンシング:
- イルミナGAIIxプラットフォーム:
- CBOTとシングルリードクラスタ生成のキット(カタログ番号GD - 300 - 1001)を使用してシングルリードフローセル上でクラスタを生成する。リード1、UPおよびDOWN -タグは、配列決定用プライマーを変更するため100uMストック濃度でプールし、ストリップ、チューブ(120μlをHT1の各100uMシークエンシングプライマーの0.6μL)に追加されます。レシピSR_Amp_Block_StripTubeHyb_v7.0は、R1のクラスタを生成するために使用されます。
- ゲノム解析IIxはオンシーケンス。 18シーケンスサイクルに続いて、ペアエンドモジュールは、イルミナR1を(下)を使用して、合成された第一鎖を除去し、フローセルをrehybridizeするために使用されます。クラスタを再生成し、インデックスタグをキャプチャするために5サイクルのために配列決定されています。
- イルミナGAIIxプラットフォーム:
- インデックスタグ配列は、実験的なビンにビンのシーケンスに使用されます。
- 各実験のビンの中で、酵母のバーコードシーケンスは、各バーコードのカウントの合計数を算出するために集計されます。
- カウントは、それぞれの実験は、同じ数の分布を持つように正規化変位値です。バーコードマイクロアレイフィットネス実験、各菌株に対する適合の不良率から類推して、ログ2比(制御/治療)として計算して表現されます。肯定的なフィットネスの欠陥の点数は、薬物治療中のひずみの存在量の減少を意味し、その歪みに削除された遺伝子の野生型バージョンはその薬や阻害剤に対する耐性に必要であることを示唆している。
注:アレイハイブリダイゼーションの代替としてバー- seqを考慮。ハイスループットシーケンシングのコストは、タグの豊かさの読み出しとハイスループットシーケンシングを使用して、低下すると、実現可能な、多くの場合になってきている18、費用効率が向上します。このように、増幅されたPCR産物は、直接"カウント"としてではなくとして配列にハイブリダイズしたシグナルの強度として測定されます。これは、タグのクロスコンタミネーション、彩度、または非常に高いまたは非常に低い信号強度に起因する問題から生じる偽陰性と陽性を排除。さらに、複数の実験は4〜8塩基のDNAインデックス19の添加によりシーケンス処理の前に組み合わせることができます。酵母のバーコードが20 bpのなので、26から28塩基から読んで一つの、2ステップでは、極端な100 +の多重化を可能にする、多重インデックスと一意のバーコードの両方をキャプチャします。この記事の執筆時点では、バー- seqはバーコードのマイクロアレイ上のコスト優位性を提供しており、さらに、バー- seqは読み取り/実行数が増加するにつれて、多重化のレベルはさらにコスト削減に増加することができるように本質的に柔軟性があり。大手プラットフォームメーカーのすべてから、いくつかの"中容量"シーケンサーはさらに選択の読み出しになる可能性がシーケンシングと、バー- seqを民主化するだろう。
このプロトコルはまた、イルミナHiSeq2000に検証されています。
サッカロマイセスセレビシエの成長制御における基本的な生物学的問題に対処するためにバー- seqの使用の優秀なデモンストレーションは、グレシャムらによる最近の研究で提示されます。いくつかの重要な実験的なデザインと解釈のガイドラインを概説20。
7。プールされたスクリーニングデータの検証
どんな機能ゲノミクス画面から結果が隔離栽培における個々の菌株を用いて検証する必要があります。各実験は、感受性株の数の点で異なるので、確認するために候補株の数を選択すると、ある程度任意です。ガイドとして、(通常は2〜3標準デに変換されるログ2比またはZスコアが最も感受性株のランキングと候補者のトップ25から50パーセントをテストプール内のすべての株の平均値からviationsは)コストとベネフィットのバランスです。個々の確認はどのフラスコで行わが、我々は( 図4を参照 )おきに測定値が15分を取って、握手分光光度計でのメディアの100μl中でOD 600が 0.06の開始接種を用いて96ウェルプレートでの成長の5世代のためにこれらのテストを行うことができます。 。
8。代表的な結果
ゲノムワイドな画面は、完了、および配列は、正規化され、各菌株の動作は、データが最も容易に遺伝子を持つExcelファイルで操作される制御処理(マイクロアレイの強度を比較するか、カウント/ひずみをシーケンシングするなど)と比較したら制御/実験のlog2比によってランク付け。このように、負はlog2比が大きく、より高感度、特定の株は、試験条件になります。これらのExcelファイルは、ソフトウェアパッケージをグラフの様々なプロットすることができます。我々はそれが最も単純なY軸とX軸上の遺伝子またはORF名にはlog2比をプロットしてください。図2aに示す例では、クロトリマゾール治療(既知の抗真菌剤)のようなプロットが表示されます。 2のは、log2比による治療にかなり敏感なすべての菌株は赤で強調表示され、そして我々は通常、薬剤の同じ濃度の存在下でそれぞれの変異体の個々の成長のアッセイにおけるこのような菌株の多くを確認するでしょう。この例では、4株は、NCP1、ERG2とERG11の2つの独立した対立遺伝子、クロトリマゾールの既 知のタンパク質のターゲットを強調表示されます。これら4遺伝子の各々は、直接エルゴステロールの生合成、コレステロールの酵母と同等に関与している。例えば、NCP1はエルゴステロールの生合成に関与しているとErg11に関連付けられていると協調的に調節されるNADP -シトクロムP450還元酵素をコードしている。この例では、既知の薬剤ターゲット(Erg11が)この公平な画面で識別されているという事実だけでなく、ターゲットの経路のいくつかの他の主要なコンポーネントを示します。最後に、赤で強調表示された遺伝子のいくつかは、エルゴステロールの生合成にまたは別個の生物学的プロセスに関与する可能性がある遺伝子を表します。前述したように、プールされた画面内の機密として検出された各菌株は、個々の増殖アッセイで敏感として確認する必要があります。図2bに示す例では、4株は、野生型親菌株、BWP17との相対的な減少の成長に基づいて、クロトリマゾールに感受性であることが確認されています。これらの個々の成長曲線は、プールされた遺伝子の薬物画面の重要な特徴を強調、それは必ずしも感度のその正確なレベルを反映していない特定の株の絶対的なランクです。さらに、図2bは、各遺伝子に複数の対立遺伝子を持っていることの価値を示す、この場合、2つのerg11破壊変異体は、わずかに感度が異なっている。感受性の程度とこれらの混乱の性質を相関させる作用の薬物のメカニズムに追加の洞察を提供することができます。
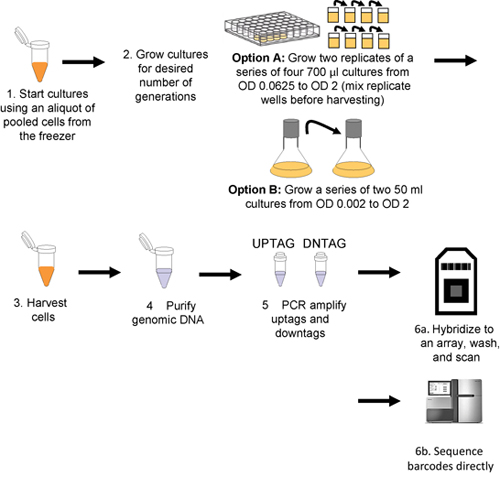
図1。プールされた増殖アッセイとバーコードの検出のためのワークフロー。文化は、プールされた細胞(ステップ1)の融解したアリコートを接種し、その後の世代の希望数の成長(ステップ2)のどちらかロボット制御(オプション)または手動(オプションB)されています。細胞を遠心分離(ステップ3)とゲノムDNAが採取される細胞を回収する(ステップ4)から分離し、uptagsとdowntagsは独立して(ステップ5)の増幅、および配列(ステップ6aまたは直接6bをステップシークエンス)にハイブリダイズされる。
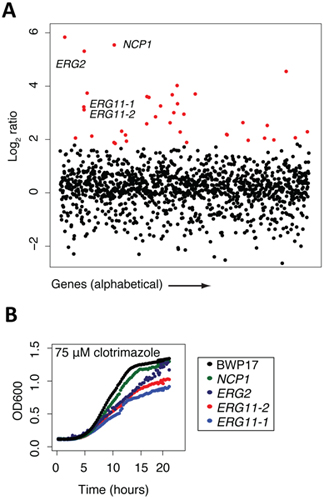
図2。サンプルデータは、プロトコルの特定のポイントで採取。スクリーニングの結果(13から適応)から(A)サンプルデータ。タグ付けされた変異体のプールは、クロトリマゾールとDMSO(対照)の存在下で20世代のために増殖させた。 2比(制御の強度/治療強度)をログに記録することは計算され、遺伝子の関数としてプロットした。機密性の高い菌株(赤)クロトリマゾール、ERG11pの既知のターゲットが含まれています。このアッセイは、頻繁に化合物の実際のターゲットに加えて、他の感受性変異株をただちに検出することに注意してください。一般に、これらの変異体がターゲットと総合的に作用するものであり、一般的なストレス/治療応答の一部であるもの、または確認するために失敗した偽陽性です。確認データの(B)の例(13から適応)。プールされた増殖アッセイの結果は、個々の文化でひずみを成長させることによって検証され、野生型成長(黒)と比較することができます。
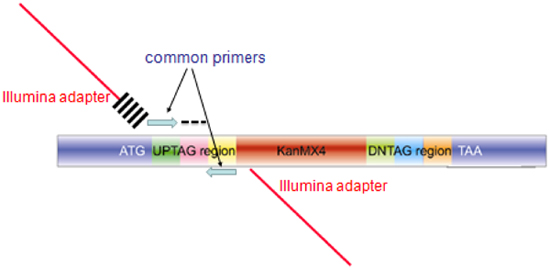
図3。マイクロアレイのハイブリダイゼーションまたはバーコードシーケンシングのためにプールされたバーコードのアッセイから生成アンプリコンの構造。各変異体のために作らリコン私n個のコレクションは、統合のためのゲノム(ATGとTAAのラベルの付いた青色の領域)、ユニークなバーコード(AGというラベルと黒のダッシュで示される)に相同性が含まれています。マイクロアレイハイブリダイゼーションのために、青色の共通プライマーは、マイクロアレイのハイブリダイゼーションのために60bpのプローブを増幅するために使用されます。バーコードシーケンシングのため、拡張されたプライマーは、イルミナアダプタ(赤いバー)、および6basesクロスハッチのインデックス)と上流のプライマーのための青色の一般的なプライマーをコードする配列から成る、PCR反応で使用され、同じコンポジットプライマー(マイナスされています第二のプライマーのための6ベースインデックス)。
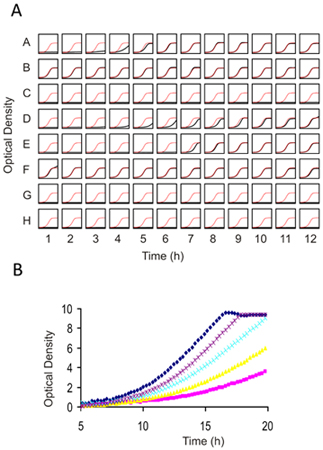
図4 1個人の成長アッセイ)は、ゲノムワイドなスクリーニングと2の適切な用量を決定するために、野生型酵母に対して化合物をプレスクリーニング)ゲノムワイド画面から結果を確認。 (A)96ウェル平底プレートには0.062のODで細胞懸濁液100μlで満たされている。各ウェルには、同じ株(用量決定のための)か、別の菌株と薬剤の組み合わせを(確認のアッセイ用)を含めることができます。化合物(一般的にDMSOに溶解)の2μlを添加し、細胞を一定の30℃で16〜20時間振盪しながら成長している℃にDMSOの最終濃度が2%を超えないようにしてください。この例では、プレートの各ウェルに成長曲線を赤で制御成長曲線のプロットに対して黒でプロットされます。互いの上に重ね合わせ例の薬を用いて得られたいくつかのprescreensの(B)より高い解像度のイメージ。この滴定のシリーズでは、IC 10-15紫の用量で取得され、削除のプロファイリング(HIPとHOP)に適しているでしょう。高い光学密度での非直線性に起因する、テカン(または任意の同様のプレートリーダー)ODSは、"従来の"1ミリメートル経路長のキュベットで得られたものを使用して校正する必要があります。
ディスカッション
ここでは、簡単にタグ付けされた変異体のコレクションを作成するためにさまざまな微生物のバーコード変異体コレクションの既存のコレクションの広い範囲に適合させることができる、ささやかな変更で、プロトコルを概説。我々は病原性酵母C.のタグトランスポゾン突然変異誘発のプロトコルを報告している間、我々はそれを強調するアルビカンス 、非常によく似たプロトコルは、単細胞の菌類の多様なに適応することができます。変更された、このプロトコルは、細菌13には適切に動作し、現在追加の真菌や細菌ゲノムの数のコレクションが建設中です。現時点では、このアッセイは、遺伝子-小分子の相互作用のための唯一の包括的な、ゲノムワイドな画面を提供します。アッセイの一つ、特に魅力的な機能は、遺伝子や低分子化合物の事前知識を必要としないことです。これらのアッセイの範囲とパワーにもかかわらず、他のラボへの譲渡は、結果の分析のための初期設備投資と情報ツールによって幾分妨げられてきた。分析のための堅牢なツールと組み合わせて次世代シーケンスの読み出しの採用により、我々は彼らの採用が増加すると予想。
開示事項
利害の衝突は宣言されません。
謝辞
我々は、議論や助言のためにトロント大学のロンデイビス、アダムDeutschbauer、および全体のHIP HOPの研究室に感謝します。 CNは、米国立ヒトゲノム研究所(助成番号HG000205)、RO1 HG003317、CIHR MOP - 84305、およびカナダの癌協会(#020380)からの補助金によってサポートされています。 JOは、スタンフォードゲノムのトレーニングプログラム(米国立ヒトゲノム研究所から助成番号T32 HG00044)と国立衛生研究所(助成番号P01 GH000205)によってサポートされていました。 GGは、博士へのイノベーションのためのカナダの財団からの助成金によって部分的にサポートされているNHGRI RO1 HG003317とカナダの癌協会、グラント#020380、TDとドネリーシーケンシングセンターによってサポートされています。ブレンダアンドリュースとジャックグリーンブラット。 AMSは、トロントオープンフェローシップの大学によってサポートされています。
資料
| Name | Company | Catalog Number | Comments |
| 抗生物質 | ベンダーとカタログ番号 | ||
| カルベニシリン | シグマ、一部#C1613 | ||
| カナマイシン | シグマ、一部#K1876 | ||
| スペクチノマイシン | シグマ、一部#S0692 | ||
| クロラムフェニコール | シグマ、一部#C0378 | ||
| DNAクリーンアップと濃縮キット | |||
| QIAprep Spin Miniprepキット | キアゲン、一部#27106 | ||
| ハイスピードプラスミドマキシキット | キアゲン、一部#12663 | ||
| QIAクイックPCR精製キット | キアゲン、一部#28106 | ||
| QIAquickゲル抽出キット | キアゲン、一部#28704 | ||
| PCRと電気泳動試薬 | |||
| Taq DNAポリメラーゼ(MG -フリー)バッファ | ニューイングランドバイオラボ、一部#M0320L | ||
| デオキシヌクレオチド溶液のミックス | ニューイングランドバイオラボ、一部#N0447L | ||
| 25 mMのMgCl 2の | シグマ、一部#63036 | ||
| アガロース、ローディング色素、および核酸は、ゲル電気泳動に適した染色 | 様々な | ||
| 10X TAEバッファー | シグマ、一部#T8280 | ||
| 1 KBプラスDNAラダー | インビトロジェン、一部#10787026 | ||
| YPD培養液 | |||
| 酵母エキス10 gの | シグマ、一部#Y1625 | ||
| 20グラムバクトペプトン | BDバイオサイエンス、一部#211677 | ||
| デキストロース20gの | シグマ、一部#D9434 | ||
| 実験機器 | |||
| マルチチャンネルピペット(1000、200、および20μL) | 様々な | ||
| 使い捨てピペット貯水池 | 様々な | ||
| 15〜50 mLの遠心管 | 様々な | ||
| 96 - と384ウェルディープウェルプレート | Axygen科学、一部#P - 2ML - SQ - CS&P - 384240SQCS | ||
| 96ウェルおよび384ウェルPCRプレートとシールのフィルム | 様々な | ||
| マルチウェルプレートをシールするためのプレートのローラー | シグマ、一部#R1275 | ||
| 30℃と37℃のプレート上に細菌や酵母の成長のために、チューブのインキュベーターを揺すって | 様々な | ||
| in vitroでトランスポゾン変異誘発の | |||
| EZ - Tn5が知られトランスポザーゼ | シャーロットのバイオテクノロジー、一部#TNP92110 | ||
| ハイスループット変換 | |||
| Seqprep 96 HTプラスミドプレップキット | エッジバイオシステムズ、一部#84359 | ||
| ポリエチレングリコール、分子量3350 | 様々な | ||
| リチウム酢酸 | シグマ、一部#517992 | ||
| 6ウェルプレート、滅菌 | コーニング、一部#3335 | ||
| 50 mg / mLのウリジン | シグマ、一部#U3750 | ||
| 100Xトリス- EDTA緩衝液 | シグマ、一部#T9285 | ||
| 1X TE/0.1M LiOAc | 様々な | ||
| サケ精巣DNA | シグマ、一部#1626 | ||
| バーコードのコレクションの成長 | |||
| 48ウェルプレート、プレート中で培養を成長した場合 | グレイナー、一部#M9437 | ||
| 粘着板のシール | ABgene、一部#AB - 0580 | ||
| 200mLの培養フラスコ | 様々な | ||
| 吸光度が可能な分光光度計 | 様々な | ||
| 250mlのフラスコや揺れ分光光度計用の温度制御シェーカー | 様々な | ||
| セイフ - ロック微量遠心チューブに、2 mLの | エッペンドルフ、一部#0030 120.094 | ||
| ハイブリダイゼーション装置 | |||
| ハイブリダイゼーションオーブン640 | アフィメトリクス、一部#800138 | ||
| 搭載されたGeneChip流体駅450 | アフィメトリクス、部分#00から0079 | ||
| GeneArrayスキャナ3000 | アフィメトリクス、部分#00から0212 | ||
| フローティングラックと沸騰水浴 | 様々な | ||
| ハイブリダイゼーションの消耗品 | |||
| Genflexタグ16K配列v2の | Affymetrix、一部#511331 | ||
| デンハルト溶液、50倍濃縮 | シグマ、一部#D2532 | ||
| ストレプトアビジン、R -フィコエリスリン共役(SAPE) | インビトロジェン、一部#S866 | ||
| セイフ - ロックマイクロ遠心チューブ、0.5 mLの | エッペンドルフ、一部#0030 123.301 | ||
| 小さなタフスポット | 多様なバイオテック、一部#LTTM - 1000 | ||
| 0.5 M EDTA | バイオラッド、一部#161-0729 | ||
| 10%のTween | シグマ、一部#T2700 | ||
| MES遊離酸一水和物 | シグマ、一部#M5287 | ||
| MESのナトリウム塩 | シグマ、一部#M5057 | ||
| 5 M NaClを | シグマ、一部#71386 | ||
| 20X SSPE | シグマ、一部#S2015 | ||
| 分子生物学グレードの水 | シグマ、一部#W4502 | ||
| ハイブリダイゼーションのプライマー | 様々なサプライヤー(標準脱塩) | ||
| Uptag | 5'GATGTCCACGAGGTCTCT 3' | ||
| Buptagkanmx4 | 5'ビオチン- GTCGACCTGCAGCGTACG 3' | ||
| Dntag | 5'CGGTGTCGGTCTCGTAG 3' | ||
| Bdntagkanmx4 | 5'ビオチン- GAAAACGAGCTCGAATTCATCG 3' | ||
| B213 | 5'ビオチン- CTGAACGGTAGCATCTTGAC 3' | ||
| Uptagkanmx | 5'GTCGACCTGCAGCGTACG 3' | ||
| Dntagkanmx | 5'GAAAACGAGCTCGAATTCATCG 3' | ||
| Uptagcomp | 5'AGAGACCTCGTGGACATC 3' | ||
| Dntagcomp | 5'CTACGAGACCGACACCG 3' | ||
| Upkancomp | 5'CGTACGCTGCAGGTCGAC 3' | ||
| Dnkancomp | 5'CGATGAATTCGAGCTCGTTTTC 3' | ||
| シーケンシングプライマー:イルミナのプラットフォーム | 様々なサプライヤー | ||
| UpTagフォワード(100uM) | 5'CAA GCA GAA GAC GGC ATA CGA GCT CTT CCG ATC T GAT GTC CAC GAG GTC TCT 3' | ||
| UpTagリバース(100uM) | 5'AAT GAT ACG GCG ACC ACC GAC ACT CTT TCC CTA CAC GAC GCT CTT CCG ATC T NNNNN GTC GAC CTG CAG CGT ACG 3' | ||
| DownTagフォワード(100uM) | 5'CAA GCA GAA GAC GGC ATA CGA GCT CTT CCG ATC T GAA AAC GAG CTC GAA TTC ATC G 3' | ||
| DownTagリバース(100uM) | 5'AAT GAT ACG GCG ACC ACC GAC ACT CTT TCC CTA CAC GAC GCT CTT CCG ATC T NNNNN CGG TGT CGG TCT CGT AG 3` | ||
| 1を読んUP -タグ配列のプライマー(100uM) | 5'GTC GAC CTG CAG CGT ACG 3' | ||
| 1 DOWN -タグ配列のプライマー(100uM)をお読みください | 5'CGG TGT CGG TCT CGT AG 3' | ||
| 2インデックスシーケンスプライマー(標準イルミナR1のプライマー)(100uM)をお読みください | 5'AC ACT CTT TCC CTA CAC GAC GCT CTT CCG ATC T 3' | ||
| 追加のシークエンシング試薬/装置 | |||
| キアゲンアプライ96 UF PCR精製キット | キアゲン、一部#29051 | ||
| 真空ポンプ | 任意のベンダー | ||
| マシュレ-ナーゲル真空マニホールド | マシュレ-ナーゲル、一部#740 681 | ||
| インビトロジェンクワント- ITのdsDNA BRアッセイキット | インビトロジェン、一部#Q32853 | ||
| インビトロジェン量子ビットのアッセイチューブ | インビトロジェン、一部#Q32856 | ||
| 40%アクリルアミドプラス1%N、N' -メチレン - ビス - アクリルアミド、37.5:1 | バイオラッド、一部#161-0148 | ||
| トリスベース | シグマ、一部#T1503 - 1KG | ||
| ホウ酸 | シグマ、一部#B6768 - 500G | ||
| 0.5M EDTA、pH8.0の | Teknova、一部#E0306 | ||
| 過硫酸アンモニウム | シグマ、一部#A3678 - 25G | ||
| TEMED | Bioshop、一部#TEM001.25 | ||
| 臭化エチジウム溶液 | Bioshop、一部#ETB444.10 | ||
| 0.5M酢酸アンモニウム | Teknova、一部#A2000 | ||
| 10mMの酢酸マグネシウム四水和物 | シグマ、一部#M0631 - 100G | ||
| 1mMのEDTA、pH8.0の | 0.5M EDTA、pHが8.0を参照してください。 | ||
| エタノール | 様々な | ||
| 酢酸ナトリウム、pH 5.2 | Teknova、一部#S0297 | ||
| 速度の真空 | 様々な | ||
| シングルリードクラスタ生成キット | イルミナ、一部#GD - 300 - 1001 | ||
| v4の36Cシークエンシングキット | イルミナ、一部#FC - 104 - 4002 | ||
10X TBEのレシピ
| 額 | 試薬 |
| 108グラム | トリスベース |
| 55グラム | ホウ酸 |
| 40mlの | 0.5M EDTA(pH 8.0)を |
| 1LのマークにのdH 2 Oを追加 | |
12%ポリアクリルアミドゲルのレシピ
| ボリューム | 試薬 |
| 5.8ミリリットル | 40%アクリルアミドプラス1%N、N' -メチレン - ビス - アクリルアミド、37.5:1 |
| 12ミリリットル | のdH 2 O |
| 2ミリリットル | 10X TBE |
| 140μL | 10%の過硫酸アンモニウム |
| 総量:20ミリリットル |
プライマーミックスをUptag:
その後再懸Uptagと100μMでのddH 2 OでBuptagkanmx4それぞれ、50μMの各終濃度に対して1:1の比率で混合する。 -20℃で保存
プライマーミックスをDowntag:
再懸DntagとBdntagkanmx4100μMでのddH 2 Oのそれぞれは、その後50μMの各終濃度に対して1:1の比率で混ぜる。 -20℃で保存
混合オリゴヌクレオチド:
100μMでのddH 2 Oで次の8つのオリゴ(標準脱塩)のそれぞれを再懸濁します。
Uptag、Dntag、Uptagkanmx、Dntagkanmx、Uptagcomp、Dntagcomp、Upkancomp、Dnkancomp。
12.5μM各終濃度8つのオリゴヌクレオチドの等量を混ぜる。
12X MES在庫:
10ミリリットルの場合は、8ミリリットル分子生物学グレードの水に0.7グラムMES遊離酸一水和物と1.93グラムMESのナトリウム塩を溶かす。よく混合した後、pHをチェックし、pHを6.5から6.7に、必要に応じて調整します。 10ミリリットルの合計体積に水を追加。滅菌濾過し、4℃での光(例えば、ホイルで管を包む)から保護。解決策が目に見え黄色または6ヶ月後になった場合に交換してください。
2Xハイブリダイゼーション緩衝液:
50ミリリットルの場合は、12X MES株式の8.3ミリリットル(2.9.14から)、5 M NaClを、0.5 M EDTA、10%Tween 20を0.1 mlの(vol / vol)の4.0 mlおよび19.9ミリリットルのddHフィルタリングの17.7 mlを混ぜて2 O滅菌フィルタします。
ミックス300ミリリットル20X SSPE、1 mlの10%トゥイーン(vol / vol)の、699ミリリットルのddH 2 O:ウォッシュ滅菌フィルタします。
ウォッシュB:ミックス150ミリリットル20X SSPE、1 mlの10%トゥイーン(vol / vol)の、849ミリリットルのddH 2 O滅菌フィルタします。
バーコードシーケンシングプライマー
UP -タグのプライマー配列の5'末端には、( 太字 )FとRプライマーに組み込まイルミナ特定のアダプターの配列である。変数の配列( イタリック体)インデックス/多重読み出しに使用される5 - merのインデックス作成のタグを表します。 3'末端には(下線)uptagのバーコードを隣接の一般的なプライマーを表しており、酵母のバーコードを増幅するために必要です。
DOWN - tag内のプライマーは5しかし、3'末端が(下線)を増幅するために使用される共通のプライマーで置き換えられる、UP -タグプライマー(イルミナ特定のシーケンス)の尾"尾は5と同一である'配列DOWNをタグのバーコード。
参考文献
- Giaever, G. Functional profiling of the Saccharomyces cerevisiae genome. Nature. 418, 387-391 (2002).
- Baba, T. Construction of Escherichia coli K-12 in-frame, single-gene knockout mutants: the Keio collection. Molecular systems biology. 2, 2006.0008-2006.0008 (2006).
- Claus, H., Frosch, M., Vogel, U. Identification of a hotspot for transformation of Neisseria meningitidis by shuttle mutagenesis using signature-tagged transposons. Mol Gen Genet. 259, 363-371 (1998).
- Hava, D. L., Camilli, A. Large-scale identification of serotype 4 Streptococcus pneumoniae virulence factors. Molecular microbiology. 45, 1389-1406 (2002).
- Costanzo, M. The Genetic Landscape of a Cell. Science. 327, 425-431 (2010).
- Tong, A. H. Systematic genetic analysis with ordered arrays of yeast deletion mutants. Science. 294, 2364-2368 (2001).
- Pan, X. A robust toolkit for functional profiling of the yeast genome. Molecular cell. 16, 487-496 (2004).
- Schuldiner, M. Exploration of the Function and Organization of the Yeast Early Secretory Pathway through an Epistatic Miniarray Profile. Cell. 123, 507-519 (2005).
- Deutschbauer, A. M. Mechanisms of haploinsufficiency revealed by genome-wide profiling in yeast. Genetics. 169, 1915-1925 (2005).
- Giaever, G. Chemogenomic profiling: identifying the functional interactions of small molecules in yeast. Proceedings of the National Academy of Sciences of the United States of America. 101, 793-798 (2004).
- Lum, P. Y. Discovering modes of action for therapeutic compounds using a genome-wide screen of yeast heterozygotes. Cell. 116, 121-137 (2004).
- Hillenmeyer, M. E. The chemical genomic portrait of yeast: uncovering a phenotype for all genes. Science. 320, 362-365 (2008).
- Oh, J. A universal TagModule collection for parallel genetic analysis of microorganisms. Nucleic acids research. 38, e146-e146 (2010).
- Oh, J. Gene annotation and drug target discovery in Candida albicans with a tagged transposon mutant collection. PLoS pathogens. 6, (2010).
- Nislow, C., Giaever, G., Stark, I., Stansfields, M. J. R. Chapter 387. Yeast Gene Analysis. , 387-414 (2007).
- Pierce, S. E., Davis, R. W., Nislow, C., Giaever, G. Genome-wide analysis of barcoded Saccharomyces cerevisiae gene-deletion mutants in pooled cultures. Nature protocols. 2, 2958-2974 (2007).
- Sambrook, J., Russell, D. W. . Molecular cloning : a laboratory manual. , (2001).
- Smith, A. M. Quantitative phenotyping via deep barcode sequencing. Genome Res. , (2009).
- Hamady, M., Walker, J. J., Harris, J. K., Gold, N. J., Knight, R. Error-correcting barcoded primers for pyrosequencing hundreds of samples in multiplex. Nature. 5, 235-237 (2008).
- Gresham, D. System-Level Analysis of Genes and Functions Affecting Survival During Nutrient Starvation in Saccharomyces cerevisiae. Genetics. 187, 299-317 (2011).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved