Method Article
Competitive Genomic Screens von Barcoded Hefe Bibliotheken
* Diese Autoren haben gleichermaßen beigetragen
In diesem Artikel
Zusammenfassung
Wir verfügen über umfassende, unvoreingenommene genomweiten Screens entwickelt, um Gen-Medikament und Gen-Umwelt-Interaktionen zu verstehen. Methoden für das Screening dieser mutierten Sammlungen präsentiert werden.
Zusammenfassung
Durch Fortschritte in der Sequenzierung der nächsten Generation Technologien haben wir Zugang zu neuen Genom-Sequenzen fast täglich. Das Tempo dieser Fortschritte ist die Beschleunigung, vielversprechende größere Tiefe und Breite. In Anbetracht dieser außergewöhnlichen Fortschritte, wird die Notwendigkeit für eine schnelle, parallele Methoden zur Gen-Funktion zu definieren immer mehr an Bedeutung. Sammlungen von genomweiten Deletionsmutanten in Hefe und E. coli wurden als Arbeitstiere für die funktionelle Charakterisierung der Genfunktion serviert, aber dieser Ansatz ist nicht skalierbar, erfordern aktuelle Gen-Deletion Ansätze jeder der Tausenden von Genen, die ein Genom zu löschen und überprüft werden sollen, umfassen. Erst nachdem diese Arbeit abgeschlossen ist können wir verfolgen, High-Throughput-Phänotypisierung. In den vergangenen zehn Jahren hat unser Labor ein Portfolio von wettbewerbsfähigen, miniaturisierte, High-Throughput-genomweite Tests, die in parallel ausgeführt werden können, verfeinert. Diese Parallelisierung ist wegen der Aufnahme von DNA 'Tags' oder 'Barcodes, "in jeder Mutante möglich, mit dem Barcode dient als Proxy für die Mutation, und man kann den Barcode Fülle Maßnahme zur mutierten Fitness zu bewerten. In dieser Studie wollen wir die Lücke zwischen DNA-Sequenz und Barcode-Mutante Sammlungen zu füllen. Um dies zu erreichen führen wir eine kombinierte Störung Transposon-Barcoding Ansatz, der sich öffnet parallel Barcode-Assays neu sequenziert, aber schlecht charakterisiert Mikroben. Zur Veranschaulichung dieses Ansatzes präsentieren wir eine neue Candida albicans Barcode Störungen Sammlung und beschreiben, wie beide Microarray-basierte und Sequenzierung der nächsten Generation-Plattformen verwendet werden kann auf 10.000 zu sammeln - 1.000.000 Gen-Gen-und Drogen-Gen-Interaktionen in einem einzigen Experiment.
Protokoll
1. Hintergrundinformationen
Es gibt mehrere Möglichkeiten, um Mutanten, die Barcode-Etiketten tragen zu generieren. Der derzeitige Goldstandard ist die Hefe KnockOut (YKO) Sammlung von einem Konsortium von Labors erstellt und abgeschlossen in 2002 1. Da die ursprüngliche YKO eingeführt wurde, andere Hefe Sammlungen generiert wurden, in anderen Stamm Hintergründen, mit Überexpression Konstrukte und in andere Mikroben wie E. coli 2. Parallel dazu ist das Bemühen, mit Barcode shRNA Bibliotheken erstellen rasch voran, und in der Tat, viele der Design-Prinzipien für diese Säuger Sammlungen aus Hefe verabschiedet worden. Um zu demonstrieren, wie Barcode-Transposons können eine schnelle, breit anwendbare Strategie zum Aufbau systematischer Sammlungen mutierten werden wir auf einer Sammlung, die wir vor kurzem in der menschlichen Pilzpathogen, Candida albicans erstellt konzentrieren. Unsere Arbeit an Candida war auf den Erfolg von Barcode-Bildschirmen in S. basierte cerevisiae, und wird hier als ein Beispiel Organismus verwendet. Die Probe-Protokoll, können mit geringfügigen Änderungen verwendet werden, um jeden Organismus, der in Suspensionskultur gezüchtet werden können Bildschirm. Da nur wenige Organismen die erforderlichen hohen Transformation und effiziente mitotische Rekombination benötigt, um perfekte Deletionsmutanten erstellt haben, entsprechend haben wir ein Protokoll, das Transposon-Mutagenese verwendet in vitro zu einer genomischen DNA-Bibliothek mutagenisieren, und dann verwandelte diesen Barcode Bruchstücke in Candida albicans 3 , 4. Inspiriert durch den Erfolg des Originals YKO Sammlung und ihre Rolle in grundlegende Entdeckungen über die Natur der Gen-Netzwerke 5-8, genomweite Haploinsuffizienz 9, Target und Wirkmechanismus 10,11, und der Wesentlichkeit aller Gene im Genom 12 Wir erwarten Ausweitung dieses Konzepts auf andere Mikroben wird als äußerst fruchtbar.
Das Protokoll wird vorausgesetzt, dass die gewünschte mutierte Sammlung erstellt wurde (z. B. YKO oder Candida albicans Störung Sammlung) und ist als individuell archiviert Stämme. Für eine detaillierte Beschreibung der Konstruktion von Stämmen sehen, 1,13,14.
2. Kombinieren einzelnen Mutanten in einem einzigen Pool
- Lassen Sie eine Woche gebündelt Zellaliquote (unbegrenzt bei -80 ° C gelagert werden) zu generieren.
- Auftauen gefrorenen Glycerin Aktien für die Stämme von Interesse vollständig, sondern lassen Sie sich nicht Zellen bleiben aufgetaut für> 2 Stunden.
- Sterilisieren einer 96-Loch Pin-Werkzeug, tauchen die Pin-Werkzeug in Wasser, um alle verbleibenden Zellen, die durch 2 Dips in 70% Ethanol Bäder (zB Pipettenspitze box Deckel), Flamme der Stift-Werkzeug und cool für 1 Minute zu entfernen. Achten Sie darauf, Flamme der Stift-Werkzeug weg von der Ethanol-Bäder. Die Höhe der Ethanol Bäder über dem Niveau in das Wasserbad, um sicherzustellen, all carry-over-Zellen geflammt und entfernt werden. Ersetzen Wasser alle 4-6 Pinnings.
- Legen Sie die sterile 96-Well-Pin-Werkzeug in einen aufgetauten 96-Well-Platte, vorsichtig schwenken und Transfer-Zellen zu einer Nunc Omni Tray mit YPD-Agar mit dem entsprechenden Antibiotikum. Wachsen Kolonien, bis sie maximale Größe bei 30 ° C erreichen (2-3d). Zur Schonung der Platten, finden wir es sehr nützlich zu vier 96-Well-Platten auf einem einzigen Omni-Tablett mit ~ 384 Stämme zu konsolidieren.
- Nach Kolonien gewachsen sind, beachten Sie fehlende oder langsam wachsende Linien repin diese bei ~ 2X die Zellmasse, wie der Rest der Stämme.
- Die Arbeit in einem Mikrobiologie-Umgebung (mit Flamme und sterilen Laborprodukte, Überschwemmung Platte mit 5-10 ml Medium, für 5 min und Resuspendieren Kolonien einweichen mit einer Zelle Streuer. Gießen Sie die Flüssigkeit plus Zellen in eine 50 ml konische Zentrifugenröhrchen, und fügen Glycerin zu 15% oder DMSO auf 7% (vol / vol).
- Messen Sie die OD 600 auf den Pool und anpassen (durch Verdünnung oder Zentrifugation) zu einer Endkonzentration von 50 OD 600 / ml mit Medium mit 15% Glycerin oder 7% DMSO.
- Aliquot in 40 ul in PCR-Streifen-Röhrchen und frieren bei -80 ° C.
3. Experimental-Pool Wachstum
Dieses Verfahren ist in Abbildung 1 dargestellt.
- Thaw Pool Aliquots (in PCR-Röhrchen) auf Eis. Wenn nicht mit Hilfe von Robotern, fahren Sie mit Schritt 5 fort.
- Unmittelbar verdünnen (gently!) am Pool in Medien mit Drogen oder den Zustand der Wahl, Impfen bei einer OD 600 von 0,0625 in einem Gesamtvolumen von 700 ul in einem 48-Well-Platte. Mindestens ein geeignetes Lösungsmittel Kontrolle auf der Platte. Für Experimente, die über 5 Generationen des Wachstums erweitern (dh mehr als 1 gut), füllen benachbarten Vertiefungen mit den Medien oder den Zustand der Wahl, aber keine Zellen.
- Seal mit einer Kunststoffplatte Dichtung; wenn die Bedingung erfordert aerobe Wachstum (z. B. nicht vergärbaren Kohlenstoffquellen), verwenden Sie eine 21-Gauge-Nadel zu durchbohren die Löcher in der Membran-Dichtung in Richtung der Seite eines jeden gut.
- Wachsen in einem Spektralphotometer, Schütteln bei 30 ° C mit einer experimentell Bestimmungned Schütteln Therapie (zB schütteln 14 min bei der höchsten Einstellung (oder temperaturgeführte Schüttler), lesen Brunnen, wieder Zittern). Ein Teil der Zellsuspension kann durch den Roboter geerntet werden und auf eine kalte Platte auf den Roboter-Deck benutzerdefinierte Generation Zeitpunkten, in der Regel 5, 10, 15 und 20 Generationen des Wachstums gespeichert. (Http://med.stanford.edu/sgtc/technology/access.html, für Details kontaktieren C. Nislow oder G. Giaever).
- Für die manuelle Zellwachstum, impfen einer 50 ml Kultur bei einem Ausgangs-OD 600 von 0,002 in einer 250 ml Kulturflasche. Schütteln bei 30 ° C bei 250 rpm, bis die Zellen eine endgültige OD 600 von 2,0 (für Saccharomyces oder Candida) erreichen für ~ 10 Generationen des Wachstums. Zusätzliche Generationen von Wachstum kann durch Verdünnung Zellen bei einer OD600 von 2,0 bis 0,02 zurück in eine frische Flasche erhältlich.
- Ernte mindestens ~ 2 OD 600 Einheiten von Zellen für jede Probe / Zeit-Punkt in Safe-Lock Reaktionsgefäße.
Hinweis: Immer sammeln Ausgangspunkt Zellprobe (dh ein "T0 Zeitpunkt"), um Ausgangsstamm Vertretung in allen neu erstellten Pool, indem 1-2 OD 600 von Pool direkt aus dem Gefrierschrank Aliquots einer 1,5-ml-Tube und Verarbeitung wie beschrieben zu bewerten unten.
4. Genomic DNA-Extraktion, PCR und Microarray-Hybridisierung oder Sequenzierung
- Purify genomischer DNA aus ~ 2 OD 600 von Zellen mit dem Zymo Forschung YeaStar Kit nach Herstellerangaben (Protokoll I bei Verwendung von Hefe-DNA) oder ein anderes geeignetes Verfahren speziell auf den Organismus von Interesse (Standard Phenol / Chloroform-Extraktion durch Alkohol-Fällung folgte Werke auch für diverse Mikroben). Bei Verwendung des YeaStar Kit eluieren DNA mit 300 ul 0,1 × TE statt der 60 &mgr; l 1X TE im Protokoll angegeben. Genomic DNA kann auf unbestimmte Zeit bei -80 ° C gelagert werden
- Richten Sie zwei PCR-Reaktionen für jede Probe, eine für die uptags und eine für die downtags, mit den Reaktionsbedingungen wie folgt: 33 ul ddH 2 O, 6 ul 10x PCR-Puffer ohne MgCl 2, 3 ul 50 mM MgCl 2, 1,2 ul 10 mM dNTPs, 1,2 ul 50 uM Up-oder Down-Primer-Mix, 0,6 ul 5 U / ul Taq-Polymerase, ~ 0,1 ug genomische DNA in 15 ul. Das Gesamtvolumen beträgt 60 ul. Thermocycle unter den folgenden Bedingungen: 94 ° C 3 min, 30 Zyklen von 94 ° C 30s, 55 ° C 30s, 72 ° C 30s, dann 72 ° C 3min, und halten bei 4 ° C. Überprüfen Sie die resultierenden PCR-Produkte auf einem Gel, eine 60 bp-Produkt für beide PCRs für Amplikons für Hybridisierung und 130bp für Amplikons für Barcode-Sequenzierung) eingesetzt erwartet. Die PCR-Produkte können dann bei -80 ° C unbegrenzt haltbar.
- Vorwärmen Hybridisierungsofen Temperatur auf 42 ° C und die Einrichtung eines kochenden Wasserbad und Eis Eimer mit einem Eis-Wasser-Schlamm.
- Vornässen Arrays durch langsames Befüllen mit 120 ul 1X Hybridisierungspuffer.
- Inkubieren im Hybridisierungspuffer bei 42 ° C und 20 rpm für 10 Minuten.
- Bereiten Sie 90 ul Hybridisierungsmix pro Probe, plus ein extra als Puffer, wie folgt: 75 ul 2X Hybridisierungspuffer, 0,5 ul B213 Kontroll-Oligonukleotid (0,2 fm / ul), 12 ul gemischt Oligonukleotide (12.05 / ul), 3 ul 50X Denhardt-Lösung) in Lock-Spitze 0,5 ml Röhrchen.
- Zugabe von 30 ul uptag PCR und 30 ul downtag PCR bis 120 ul Hybridisierung Mix für ein Gesamtvolumen von 150 ul. Kochen für 2 Minuten und stellen in Eis-Wasser für mindestens 2 Minuten. Kurz Spin die Rohre vor dem Gebrauch.
- Entfernen Sie die Pre-Hybridisierungspuffer aus den Arrays und fügen 90 ul Hybridisierung / PCR-Mix. Um zu verhindern, Verdunstung, decken Sie die Array-Dichtungen mit Tough-Spot. Hybridisierung für 16 Stunden bei 42 ° C, 20 Umdrehungen pro Minute.
- Frisch vorbereiten 600 ul Biotin Labeling Mix pro Probe plus eine extra, wie folgt: 180 ul 20x SSPE, 12 ul 50X Denhardt, 6 ul 1% Tween 20 (vol / vol), 1 ul 1 mg / ml Streptavidin-Phycoerythrin, 401 ul ddH 2 O. Speichern Sie alle Streptavidin-PE-Proben im Dunkeln. Aliquot 600 ul in 2 ml Röhrchen. Entfernen Tough-Spots von Chips.
- Langsam entfernen Hybridisierung Mix aus den Arrays mit einer Pipette und füllen Microarrays mit 120 ul Wash A. Prime die Affymetrix Fluidics Station.
- Waschen Sie die Arrays mit einem Affymetrix Fluidics Station nach den Anweisungen des Herstellers mit "Gene-Flex_Sv3_450"-Protokoll mit folgenden Änderungen: 1 extra Schritt mit Wash A (1 Zyklus, 2 Mixe) vor der Färbung, Wash B Temperatur 42 ° C statt 40 ° C, bei 42 ° C Fleck anstelle von 25 ° C. Es ist auch möglich, die Post-Hybridisierung zu waschen, die Biotin-Färbung, und die post-Färbung waschen manuell durchführen (siehe S. 396 in Bezug 15). Nach Fluidik-Operationen, führen Sie die Fluidik-Station "SHUTDOWN_450"-Protokoll.
- Nach dem waschen, damit keine Luftblasen vorhanden sind. Falls notwendig, 90 ul Wash A und Pipette langsam, bis die Blasen verschwinden. Wenn es irgendwelche Marken oder smudges auf der Array-Oberfläche, reinigen Sie die Glasscheibe mit Isopropanol und einem fusselfreien Tuch. Bewerben frischen Tough-Spots über die Dichtungen / Septen und Ort der Arrays in den Scanner.
- Scan in einem Affymetrix GeneArray Scanner bei einer Emissionswellenlänge von 560 nm auf.
5. Array-Analyse (siehe Abbildung 2 zeigt ein Beispiel unter Verwendung des Candida albicans Störung Sammlung)
- Ausreißer Maskierung: Da die Affymetrix Tag4 Array enthält 5 Replikate jeder Barcode ergänzen verteilt nach dem Zufallsprinzip einer Barcode-Sonde, die aus der Wiederholungen kann maskiert und verworfen werden abweicht. Um dies zu tun, für jedes Array-Funktion, scheint ein Ausreißer nach Signal basiert im Vergleich zu den 4 anderen Replikate von diesem Feature unserer Software prüft zunächst die 5 Funktionen, die der Verdächtige Ausreißer Surround, wodurch eine Matrix von 25 Merkmalen mit dem verdächtigen Funktion in der Mitte. Wenn> 13/25 Sonden in dieser Region aus jedem ihrer individuellen ihren getrimmt replizieren mean (Mittelwert der mittleren drei Wiederholungen unter Ausschluss des höchsten und niedrigsten repliziert) unterscheiden sich um mehr als 10% ist diese Sonde dann von der weiteren Analyse verworfen. Da solche Ausreißer meist sind das Ergebnis der Post-Hybridisierung Waschen Inkonsistenzen, erweitern wir oder "pad" der Region, die vermuten Sonden. Pad solche Sonden durch die Einbeziehung aller Sonden innerhalb einer 5-Sonde Radius, wie definiert durch ((x 1-x 2) 2 + (y 1-y 2) 2) ½ <6, wo x 1, x 2, y 1, und y 2 sind die x-und y-Koordinaten für die beiden Funktionen. Schließlich verwerfen Funktionen, für die Standardabweichung (in der. Cel-Datei für Affymetrix-Arrays) von Feature-Pixel / Mittelwert-Funktion Pixel. Nach dem Entfernen von Ausreißern, repliziert Durchschnitt der Helligkeitswerte für alle übrigen.
- Entfernen unbrauchbar tags: Tags mit niedriger Intensität Werte geben schlechte Qualität der Ergebnisse und müssen entfernt werden. Ein Ausschluss Cutoff für diese niedrige Intensität Sonden können wie folgt berechnet:
- Für jede Behandlung-Kontrolle Paar von Arrays, berechnen log 2 ((i c-b g) / (i t-b g)) für jeden Tag, wo i, c ist die Kontrolle Intensität i t ist die Behandlung der Intensität und b g ist die mittlere Intensität der zugewiesenen Tags Sonden.
- Koppeln Sie die uptag und downtag Verhältnisse durch den Stamm und für jede Tag-Paar, nehmen Sie die minimale Intensität für die beiden tags in den beiden Proben. Sortieren Verhältnis Paaren, die durch diese minimale Intensität.
- Verwenden Sie ein Schiebefenster Größe von 50 auf dem Platz Verhältnis Paare, die Berechnung der Korrelation von uptag und downtag Verhältnis Paare innerhalb des Fensters. Auch die Berechnung der Mittelwert der minimalen Intensitäten im vorherigen Schritt berechnet.
- Schieben Sie das Fenster mit 25 Paar, und wiederholen Sie den vorherigen Schritt, bis alle Paare gekreuzt haben gewesen.
- Plot der durchschnittlichen Mindest-Intensität gegen die uptag-downtag Korrelation für alle Fenster.
- Schließlich wählen Sie eine Intensität Schwelle; in der Regel nutzen wir die Intensität Wert, bei dem die Korrelation gelangt zunächst 80% seiner maximalen Wert. Flag und entfernen Sie aus der weiteren Analyse keine Tags unterhalb dieser Grenzfrequenz.
- Sättigung-Korrektur: Da jede Funktion auf dem Barcode-Microarray kann gesättigt werden, das Signal auf der Tag4 Array ist nicht linear um Schlagwort Konzentration verwandt. Um dies zu korrigieren Sättigung folgen das Protokoll in Referenz 16 beschrieben.
- Array Normalisierung: Für jedes Array, normalisieren die uptags und downtags getrennt. Um Quantil Normalisierung Rang der Werte aus den einzelnen Array für uptags und downtags in der Reihenfolge zunehmender Intensität gewonnen. Um meine normalisieren für jeden Satz von uptags und downtags, durch den Mittelwert verteilen. Die mittlere Normalisierung aller Arrays wird in einem zweiten Schritt folgt die Umwandlung der Rohdaten mit dem Mittelwert der einzelnen Arrays.
- Berechnung Empfindlichkeit Noten für die Steuerung der Behandlung Vergleiche: Um log 2-Verhältnisse als Metrik Empfindlichkeit zu verwenden: Für jeden Stamm, berechnen log 2 ((μ c-b g) / (μ t-b g)), wobei μ c ist die mittlere Intensität für die Kontrollproben, ist μ t die mittlere Intensität für die Behandlung Proben und b g ist die mittlere Intensität der zugewiesenen Sonden. Stämme mit einem positiven log 2-Verhältnis empfindlich auf die Behandlung, und solche, die resistent sind negative log 2-Verhältnisse.
6. Die Beurteilung der Eignung Barcode Hefestämme durch Sequenzierung
- Isolieren Sie DNA aus der Löschung Pools für Mikroarrays beschrieben.
- Amplify jeweils 20-mer uptag Barcode mit Composite-Primern der Sequenzen der gängigen Barcode-Primer und die Sequenzen für die Hybridisierung der Illumina Durchflusszelle erforderlich umfasste (Siehe Tabelle der Illumina-Primer und Abbildung 3 für ein Diagramm des Amplikons). DiesePrimer verwendet entsalzt ohne weitere Reinigung werden. PCR ist in 100 &mgr; l durchgeführt, wobei Invitrogen Platinum PCR Supermix (Art.-Nr. 11306-016) mit den folgenden Bedingungen: 95 ° C / 3 min, 25 Zyklen von 94 ° C/30 sec, 55 ° C/30 sec, 68 ° C/30 sec, gefolgt von 68 ° C/10 min.
- Purify das PCR-Produkt (~ 130bp) mit Qiagen MinElute 96 UF PCR Purification Kit (Best. Nr. 28051).
- Nach PCR Aufreinigung, Quantifizierung der DNA mit dem Invitrogen Quant-iT dsDNA BR Assay Kit (Kat. Nr. Q32853). Verlassen Sie sich nicht auf 260/280 Lesungen!
- Normalize DNA-Konzentration auf 10μg/ml und Pool gleiche Volumina der normalisierten DNA.
- Separate gepoolte DNA auf einem 12% Polyacrylamid-TBE-Gel für 3-4 Stunden, je nach Spannung verwendet.
- Stain Gele mit Ethidiumbromid (Sybr Grün sollte auch funktionieren) für 30 Minuten.
- Suchen Sie das Band von Interesse an einem langwelligen UV-favoriten (Tragen geeigneter Gesichtsschutz), schneiden Sie es aus und extrahiert die DNA mit dem Gedränge und genießen Methode 17 durch Ethanolfällung folgte.
- Bestätigen Sie, dass die entsprechende Größe DNA (130bp) isoliert wurde und dass Primer wurden entfernt mit dem Agilent Bioanalyzer High Sensitivity DNA-Kit (Art.-Nr. 5067-4626).
- Probe-Sequenzierung:
- Illumina GAIIx Plattform:
- Generieren Cluster auf einem Single-Read Durchflusszelle mit der CBOT und Single-Read Cluster-Generation Kit (Kat. Nr. GD-300 bis 1001). Für Read 1, UP-und DOWN-tag geändert Sequenzierung Primer sind in einem Lager 100 um Konzentration gebündelt und zu einem Band-tube (0.6μL jedes 100um Sequenzierprimers in 120 ul HT1). Rezept SR_Amp_Block_StripTubeHyb_v7.0 wird verwendet, um R1-Cluster zu erzeugen.
- Die Sequenzierung der Genome Analyzer IIx. Nach 18-Sequenzierung Zyklen ist die Paired-End-Modul verwendet, um die synthetisierten Erststrang Streifen und rehybridize der Durchflusszelle, mit dem Illumina R1 (unten). Cluster sind regeneriert und sequenziert für 5 Zyklen, um den Index-Tag zu erfassen.
- Illumina GAIIx Plattform:
- Der Index-Tag-Sequenz ist, bin Sequenzen in experimentellen Fächer eingesetzt.
- Innerhalb der einzelnen experimentellen bin, sind die Hefe Barcode-Sequenzen ausgezählt zu einer Gesamtzahl von Zählungen für jeden Barcode geben.
- Die Zählungen werden Quantil normalisiert, so dass jeder Versuch hat die gleiche Anzahl Distribution. In Analogie mit Barcode-Microarray-Experimente Fitness, Fitness-Mangel-Verhältnissen für jeden Stamm berechnet und als log 2-Verhältnis (Kontrolle / Behandlung) ausgedrückt. Positive Fitness Defekt Noten bedeuten eine Abnahme der Belastung Fülle während der Behandlung und lassen vermuten, dass das Wildtyp-Version des Gens in diesen Stamm gelöscht für die Resistenz gegen das Medikament oder Inhibitor ist nicht erforderlich.
Hinweis: Unter Berücksichtigung Bar-seq als Alternative zum Array Hybridisierung. Mit den Kosten der Sequenzierung mit hohem Durchsatz sinkt, mit Hochdurchsatz-Sequenzierung als ein Auslesen der Tag-Fülle wird immer möglich und in vielen Fällen kostengünstiger ist 18. Auf diese Weise ist amplifizierte PCR-Produkt direkt als "zählt" und nicht als Signalintensität als ein Array hybridisiert gemessen. Dies eliminiert falsche Negative und Positive, das von Tag Kreuzkontamination, Sättigung oder Fragen im Zusammenhang mit sehr hohen oder sehr niedrigen Signalintensitäten entstehen. Darüber hinaus können mehrere Experimente vor kombiniert werden, um durch die Zugabe von einer 4-8 Basis DNA Index 19 Sequenzierung. Da die Hefe Barcodes sind 20 bp, ein einziges, 2-stufig von 26-28 Basen lesen erfasst sowohl Multiplex-Index und eindeutigen Barcode, so dass für extreme 100 + Multiplexing. Zum Zeitpunkt des Schreibens dieses Artikels, bietet Bar-seq einen Kostenvorteil gegenüber Bar-Code-Microarrays, und darüber hinaus ist Bar-seq inhärent flexibel, so dass die Anzahl der Lese / run erhöht, das Niveau der Multiplex-Verfahren kann weiter die Kosten senken . Mehrere "mid-Kapazität" Sequenzer aus allen wichtigen Plattform-Hersteller weiter zu demokratisieren Bar-seq, mit Sequenzierung wahrscheinlich das Auslesen der Wahl geworden.
Dieses Protokoll wurde auch auf der Illumina HiSeq2000 validiert.
Eine ausgezeichnete Vorführung der Verwendung von Bar-seq eine grundlegende biologische Frage in Saccharomyces cerevisiae Wachstumskontrolle Adresse ist in einer aktuellen Studie von Gresham et al. 20 Personen, einige wichtige experimentelle Design und die Auslegung Richtlinien zu skizzieren.
7. Validierung von Screening-Daten gepoolt
Die Ergebnisse aus allen funktionellen Genomik Bildschirm sollte überprüft mit Hilfe der einzelnen Stämme in isolierten Kultur. Da jedes Experiment wird im Hinblick auf die Zahl der sensiblen Stämme unterscheiden, die Auswahl der Kandidaten-Stämme, um zu bestätigen ist etwas willkürlich. Als Richtwert Ranking der empfindliche Stämme von den log 2-Verhältnis oder z-Score und Test der oberen 25-50% der Kandidaten (in der Regel übersetzt auf 2-3 Standard deAbweichungen vom Mittelwert aller Stämme in den Pool) ist eine gute Balance zwischen Kosten und Nutzen. Individuelle Bestätigungen können in jeder Flasche durchgeführt werden, aber wir führen diese Tests seit 5 Generationen des Wachstums in 96-Well-Platten mit einem Start Inokulum von 0,06 OD 600 in 100 ul Medium in einem Schütteln Spektralphotometer, das Maßnehmen alle 15 Minuten (siehe Abbildung 4) .
8. Repräsentative Ergebnisse
Nach einer genomweiten Screen abgeschlossen ist, und die Arrays wurden normalisiert und das Verhalten von jedem Stamm eine Kontrolle der Behandlung verglichen werden (zB durch Vergleich Microarray-Intensitäten oder Sequenzierung counts / Stamm) die Daten am leichtesten in eine Excel-Datei mit Genen manipuliert Rang der log2-Verhältnisse von control / Experiment. Auf diese Weise ist, desto größer die negative log2-Verhältnis, desto empfindlicher ist, dass besondere Belastung auf die Probe Zustand. Diese Excel-Dateien können in einer Vielzahl von Grafik-Software-Pakete dargestellt werden. Wir finden es am einfachsten, die log2 Verhältnisse auf der Y-Achse und das Gen oder ORF Namen auf der X-Achse zeichnen. In dem Beispiel in Abbildung 2a gezeigt, ist ein solches Grundstück von Clotrimazol-Behandlung (ein bekanntes Fungizid) gezeigt. Alle Stamm, deutlich empfindlicher auf die Behandlung mit einem log2-Verhältnis von 2 sind, werden rot hervorgehoben, und wir würden in der Regel überprüfen viele dieser Stämme in einzelnen Wachstums-Assays jeder Mutante in Gegenwart der gleichen Konzentration des Medikaments. In diesem Beispiel werden 4 Stämme markiert, NCP1, ERG2 und 2 unabhängige Allele ERG11 die bekannten Zielprotein von Clotrimazol. Jede dieser 4 Gene direkt in Ergosterolbiosynthese, die Hefe Gegenwert von Cholesterin beteiligt. Zum Beispiel codiert NCP1 ein NADP-Cytochrom P450-Reduktase, die in Ergosterol-Biosynthese beteiligt ist, und die damit verbundenen und koordiniert reguliert mit ERG11. Dieses Beispiel unterstreicht die Tatsache, dass die bekannten Angriffspunkt für Medikamente (ERG11) in diesem unvoreingenommene Bildschirm identifiziert, sowie mehrere andere wichtige Komponenten der Ziel-Pfad. Schließlich stellen einige der markierten Gene in rot Gene, die in Ergosterolbiosynthese oder in unterschiedlichen biologischen Prozessen beteiligt sein können. Wie bereits erwähnt, sollte jeder Stamm als sensibel in einer gepoolten Bildschirm erkannt, wie seine Sensibilität in einzelnen Wachstums-Assay überprüft werden. In dem Beispiel in Abbildung 2b dargestellt, sind vier Stämme bestätigt, sensibel mit Clotrimazol auf ihre verminderte Wachstum gegenüber dem Wildtyp-Stammkultur, BWP17 basiert. Diese individuellen Wachstumskurven Highlight ein wichtiges Merkmal solcher gebündelt Gen-Drogen-Bildschirme, das ist der absolute Rang eines bestimmten Stamms nicht unbedingt die exakte Maß an Sensibilität. Darüber hinaus Abbildung 2b zeigt auch den Wert der mit multipler Allele für jedes Gen, in diesem Fall haben die beiden ERG11 Störung Mutanten leicht unterschiedlichen Empfindlichkeiten. Die Korrelation der Natur dieser Störungen mit dem Grad der Empfindlichkeit kann einen zusätzlichen Einblick in die Drogen Wirkmechanismus bieten.
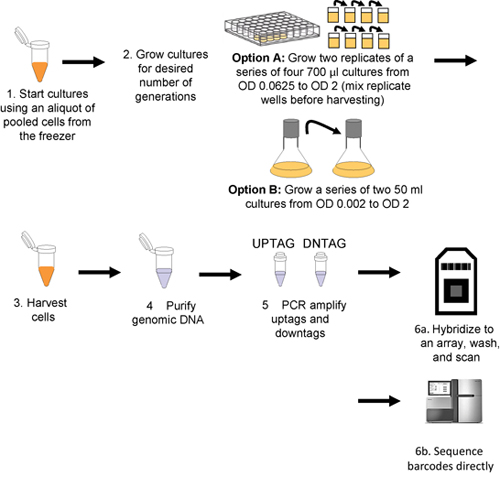
Abbildung 1. Workflow für gepoolte Wachstum Test-und Barcode-Erkennung. Die Kulturen werden mit aufgetauten Aliquots von gepoolten Zellen (Schritt 1) geimpft, und dann für die gewünschte Anzahl von Generationen (Schritt 2) entweder maschinell angebaut (Option A) oder manuell (Option B). Zellen durch Zentrifugation geerntet (Schritt 3) und genomische DNA wird dann aus den geernteten Zellen (Schritt 4) isoliert werden uptags und downtags unabhängig verstärkt (Schritt 5), und hybridisiert, um ein Array (Schritt 6a oder sequenziert direkt Schritt 6b).
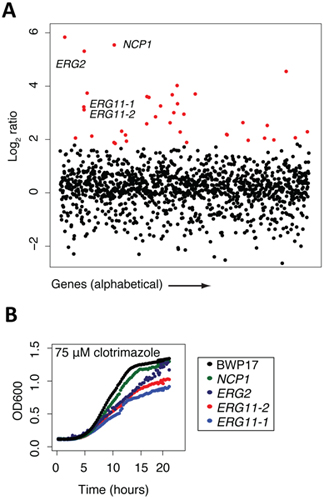
Abbildung 2. Sample-Daten an bestimmten Punkten des Protokolls gesammelt. (A) Beispiel Daten von Screening-Ergebnisse (in Anlehnung an 13). Der Pool von markierten Mutanten wurde für 20 Generationen in Gegenwart von Clotrimazol und DMSO (Kontrolle) gewachsen. Log 2-Verhältnis (Kontrollintensität / Behandlung Intensität) wurde berechnet und als Funktion des Gens. Hoch empfindliche Stämme (rot) enthalten die bekannten Ziel von Clotrimazol, ERG11p. Beachten Sie, dass dieser Test häufig deckt anderen Mutanten neben der Verbindung der eigentliche Ziel. Im Allgemeinen sind diese Mutanten diejenigen, die synthetisch wirken mit dem Ziel sind, sind diejenigen, die Teil einer allgemeinen Stress / Ansprechen auf die Behandlung, oder Fehlalarmen, die bestätigen, scheitern. (B) Beispiel für die Bestätigung von Daten (aus 13). Ergebnisse aus den gepoolten Wachstum Assays können, indem die Belastung in den einzelnen Kultur validiert und verglichen Wildtyp-Wachstum (schwarz).
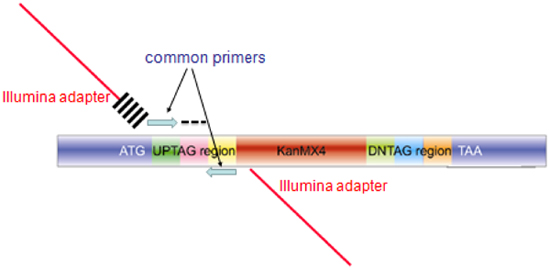
Abbildung 3. Struktur des Amplikons aus gepoolten Barcode-Assays für die Microarray-Hybridisierung oder Sequenzierung Barcode erzeugt. Das Amplikon für jede Mutante produziert in enthält die Sammlung Homologie zum Genom für die Integration (blau Regionen gekennzeichnet ATG und TAA), einzigartige Barcodes (mit AG und gekennzeichnet durch einen schwarzen Strich). Für Microarray-Hybridisierung, sind die blauen gemeinsamen Primer verwendet, um eine 60bp-Sonde für die Microarray-Hybridisierung zu verstärken. Für Barcode-Sequenzierung, erweitert werden Primer in der PCR-Reaktion eingesetzt, bestehend aus Sequenzen, die Illumina-Adapter (roter Balken), und der Index der 6bases cross-Luken) und die blaue gemeinsamen Primer für die Upstream-Primer und die gleichen zusammengesetzten Primer (minus die 6 Basis-Index) für das zweite Primer.
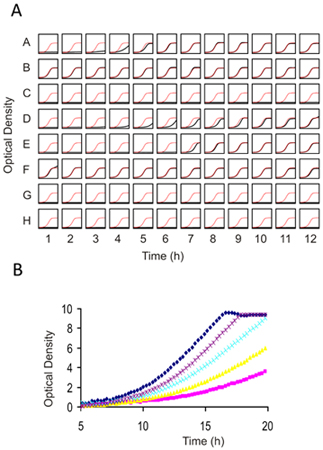
Abbildung 4. Individual Growth-Assays für 1) Prescreening Verbindungen gegen Wildtyp-Hefe zu einer angemessenen Dosis für genomweite Screening und 2 zu bestimmen) bestätigen Ergebnisse aus genomweiten Screens. (A) A 96 auch flache Bodenplatte ist mit 100 ul Zellsuspension bei einer OD von 0,062 gefüllt. Jedes der gut können die gleichen Stamm (Dosis-Bestimmung) oder anderen Stamm-und Medikamenten-Kombinationen (zur Bestätigung Assays). 2 ul der Verbindung (typischerweise gelöst in DMSO) zugegeben und die Zellen werden unter ständigem Schütteln für 16-20 h bei 30 ° C. Die Endkonzentration von DMSO sollte 2% nicht überschreiten. In diesem Beispiel, in jede Vertiefung der Platte der Wachstumskurve ist in schwarz gegen eine graphische Darstellung der Kontrolle Wachstumskurve in rot dargestellt. (B) Höhere Auflösung von mehreren prescreens mit einem Beispiel Medikament auf-einander überlagert erhalten. In dieser Titration Serie, ist ein IC 10-15 mit dem lila Dosis erhalten und angemessen wäre für die Löschung Profiling (HIP und HOP). Aufgrund der Nichtlinearität bei höheren optischen Dichten, müssen Tecan (oder eine ähnliche Platte Leser) ODs kalibriert diejenigen mit einem "traditionellen" 1mm Weglänge Küvette gewonnen werden.
Diskussion
Hier erörtern wir ein Protokoll, mit bescheidenen Modifikation, leicht zu einer Vielzahl von bestehenden Sammlungen von Barcode-Mutante Sammlungen von verschiedenen Mikroorganismen getaggt mutierten Sammlungen erstellen angepasst werden kann. Wir betonen, dass wir zwar ein Protokoll über den markierten Transposon-Mutagenese haben berichtet, für den humanpathogenen Hefepilz C. albicans, ein sehr ähnliches Protokoll könnte zu einer Vielzahl von einzelligen Pilze angepasst werden. Geändert arbeitet dieses Protokoll auch in Bakterien 13, und derzeit Kollektionen für eine Reihe von zusätzlichen Pilz-und bakterielle Genome sind im Bau. Derzeit bietet dieser Test die einzige umfassende, genomweite unvoreingenommene Bildschirm für die Gen-kleine Molekül-Wechselwirkungen. Eine besonders nützliche Funktion des Tests ist, dass keine Vorkenntnisse des Gens oder kleines Molekül erforderlich ist. Trotz des Umfangs und der Macht dieser Tests hat ihre Übertragbarkeit auf andere Labors wurde ein wenig durch die Erstinvestition und Informatik-Werkzeuge für die Analyse der Ergebnisse erschwert. Mit der Verabschiedung der nächsten Generation Reihenfolge Auslesen mit robuster Werkzeuge für die Analyse kombiniert, erwarten wir ihre Annahme zu erhöhen.
Offenlegungen
Keine Interessenskonflikte erklärt.
Danksagungen
Wir bedanken uns bei Ron Davis, Adam Deutschbauer, und die ganze HIP HOP Labor an der University of Toronto für Gespräche und Beratung. CN wird durch Zuschüsse aus dem National Human Genome Research Institute (Grant Number HG000205), RO1 HG003317, CIHR MOP-84305, und der kanadischen Krebsgesellschaft (# 020380) unterstützt. JO wurde von der Stanford Genome Training Program (Grant Number T32 HG00044 vom National Human Genome Research Institute) und die National Institutes of Health (Grant Number P01 GH000205) unterstützt. GG wird durch die NHGRI RO1 HG003317 und der kanadischen Krebsgesellschaft, Grant # 020380, TD und der Donnelly Sequencing Center unterstützt wird zum Teil durch Zuschüsse aus dem kanadischen Stiftung für Innovation zu Drs unterstützt. Brenda Andrews und Jack Greenblatt. AMS wird von der Universität von Toronto öffnen Fellowship unterstützt.
Materialien
| Name | Company | Catalog Number | Comments |
| Antibiotika | Hersteller und Katalog-Nummern | ||
| Carbenicillin | Sigma, Teil # C1613 | ||
| Kanamycin | Sigma, Teil # K1876 | ||
| Spectinomycin | Sigma, Teil # S0692 | ||
| Chloramphenicol | Sigma, Teil # C0378 | ||
| DNA Clean-up und die Konzentration Kits | |||
| QIAprep Spin Miniprep Kit | Qiagen, Teil # 27106 | ||
| HiSpeed Plasmid Maxi Kit | Qiagen, Teil # 12663 | ||
| QIAquick PCR Purification Kit | Qiagen, Teil # 28106 | ||
| QIAquick Gel Extraction Kit | Qiagen, Teil # 28704 | ||
| PCR und Elektrophorese-Reagenzien | |||
| Taq DNA Polymerase (Mg-free) Buffer | New England Biolabs, Teil # M0320L | ||
| Desoxynukleotid Lösung Mix | New England Biolabs, Teil # N0447L | ||
| 25 mM MgCl 2 | Sigma, Teil # 63036 | ||
| Agarose, Loading-Farbstoff und Nukleinsäure-Fleck geeignet für Gel-Elektrophorese | Verschiedene | ||
| 10x TAE-Puffer | Sigma, Teil # T8280 | ||
| 1 Kb Plus-DNA Ladder | Invitrogen, Teil # 10787026 | ||
| YPD Brühe | |||
| 10 g Hefe-Extrakt | Sigma, Teil # Y1625 | ||
| 20 g Bacto Pepton | BD Biosciences, Teil # 211677 | ||
| 20 g Dextrose | Sigma, Teil # D9434 | ||
| Labware | |||
| Mehrkanalpipetten (1000, 200, und 20 ul) | Verschiedene | ||
| Einweg Pipettieren Stauseen | Verschiedene | ||
| 15 und 50 mL Zentrifugenröhrchen | Verschiedene | ||
| 96 - und 384-Well Deep Well Platten | Axygen Scientific, Teil # P-2ML-SQ-CS & P-384240SQCS | ||
| 96-well und 384-well PCR-Platten und Dichtung Film | Verschiedene | ||
| Plattenroller zur Abdichtung Multi-Well-Platten | Sigma, Teil # R1275 | ||
| 30 ° C und 37 ° C Schüttelinkubatoren für den Anbau von Bakterien und Hefen auf Platten und in Rohren | Verschiedene | ||
| In-vitro-Transposon-Mutagenese | |||
| EZ-Tn5 Transposase | Epicentre Biotechnologies, Teil # TNP92110 | ||
| High-Throughput-Transformation | |||
| Seqprep 96 HT Plasmid Prep Kit | Edge-Biosystems, Teil # 84359 | ||
| Polyethylenglykol, Molekulargewicht 3350 | Verschiedene | ||
| Lithiumacetat | Sigma, Teil # 517992 | ||
| 6-Well-Platten, steril | Corning, Teil # 3335 | ||
| 50 mg / mL Uridin | Sigma, Teil # U3750 | ||
| 100X Tris-EDTA-Pufferlösung | Sigma, Teil # T9285 | ||
| 1X TE/0.1M LiOAc | Verschiedene | ||
| Lachs testis DNA | Sigma, Teil # 1626 | ||
| Das Wachstum der Barcode-Sammlungen | |||
| 48-Well-Platten, wenn wachsenden Kulturen in Platten | Greiner, Teil # M9437 | ||
| Klebeplatte Dichtungen | ABgene, Teil # AB-0580 | ||
| 200 ml Kulturflaschen | Verschiedene | ||
| Spektrophotometer für die Absorption | Verschiedene | ||
| Temperaturgeregelte Schüttler für 250 ml Flaschen oder Schütteln Spektralphotometer | Verschiedene | ||
| Safe-Lock Mikrozentrifugenröhrchen, 2 mL | Eppendorf, Teil # 0030 120.094 | ||
| Hybridisierung Ausrüstung | |||
| Hybridisierungsofen 640 | Affymetrix, Teil # 800138 | ||
| GeneChip Fluidic-Station 450 | Affymetrix, Teil # 00-0079 | ||
| GeneArray Scanner 3000 | Affymetrix, Teil # 00-0212 | ||
| Kochenden Wasserbad mit schwimmenden Rack | Verschiedene | ||
| Hybridisierung Verbrauchsmaterialien | |||
| Genflex Tag 16K Array v2 | Affymetrix, Teil # 511331 | ||
| Denhardt-Lösung, 50X Konzentrat | Sigma, Teil # D2532 | ||
| Streptavidin, R-Phycoerythrin-Konjugat (SAPE) | Invitrogen, Teil # S866 | ||
| Safe-Lock Mikrozentrifugenröhrchen, 0,5 mL | Eppendorf, Teil # 0030 123.301 | ||
| Teeny Tough-Spots | Diversified Biotech, Teil # LTTM-1000 | ||
| 0,5 M EDTA | BioRad, Teil # 161-0729 | ||
| 10% Tween | Sigma, Teil # T2700 | ||
| MES free-Monohydrat | Sigma, Teil # M5287 | ||
| MES Natriumsalz | Sigma, Teil # M5057 | ||
| 5 M NaCl | Sigma, Teil # 71386 | ||
| 20X SSPE | Sigma, Teil # S2015 | ||
| Molekularbiologie Wasser | Sigma, Teil # W4502 | ||
| Hybridisierung Primer | Verschiedene Anbieter (Standard Entsalzung) | ||
| Uptag | 5 'GATGTCCACGAGGTCTCT 3' | ||
| Buptagkanmx4 | 5 'Biotin-GTCGACCTGCAGCGTACG 3' | ||
| Dntag | 5 'CGGTGTCGGTCTCGTAG 3' | ||
| Bdntagkanmx4 | 5 'Biotin-GAAAACGAGCTCGAATTCATCG 3' | ||
| B213 | 5 'Biotin-CTGAACGGTAGCATCTTGAC 3' | ||
| Uptagkanmx | 5 'GTCGACCTGCAGCGTACG 3' | ||
| Dntagkanmx | 5'GAAAACGAGCTCGAATTCATCG 3 ' | ||
| Uptagcomp | 5'AGAGACCTCGTGGACATC 3 ' | ||
| Dntagcomp | 5'CTACGAGACCGACACCG 3 ' | ||
| Upkancomp | 5'CGTACGCTGCAGGTCGAC 3 ' | ||
| Dnkancomp | 5'CGATGAATTCGAGCTCGTTTTC 3 ' | ||
| Sequenzierungsprimern: Illumina Plattform | Verschiedene Anbieter | ||
| UpTag Sturm (100um) | 5 'CAA GCA GAA GAC GGC ATA CGA GCT CTT CCG ATC T GAT GTC CAC GAG GTC TCT 3' | ||
| UpTag Reverse (100um) | 5 'AAT GAT ACG GCG ACC ACC GAC ACT CTT TCC CTA CAC GAC GCT CTT CCG ATC T NNNNN GTC GAC CTG CAG CGT ACG 3' | ||
| DownTag Sturm (100um) | 5 'CAA GCA GAA GAC GGC ATA CGA GCT CTT CCG ATC T GAA AAC GAG CTC GAA TTC ATC G 3 " | ||
| DownTag Reverse (100um) | 5 'AAT GAT ACG GCG ACC ACC GAC ACT CTT TCC CTA CAC GAC GCT CTT CCG ATC T NNNNN CGG TGT CGG TCT CGT AG 3 ` | ||
| Lesen Sie 1 UP-tag seq-Primer (100 um) | 5 'GTC GAC CTG CAG CGT ACG 3' | ||
| Lesen Sie 1 AB-tag seq-Primer (100 um) | 5 'CGG TGT CGG TCT CGT AG 3' | ||
| Lesen Sie 2 Index-Sequenzierung Primer (Standard Illumina R1 Primer) (100 um) | 5 'AC ACT CTT TCC CTA CAC GAC GCT CTT CCG ATC T 3' | ||
| Zusätzliche Sequencing Reagenzien / Equipment | |||
| Qiagen MinElute 96 UF PCR Purification Kit | Qiagen, Teil # 29051 | ||
| Vakuumpumpe | Jeder Anbieter | ||
| Macherey-Nagel Vacuum Manifold | Macherey-Nagel, Teil # 740 681 | ||
| Invitrogen Quant-iT dsDNA BR Assay Kit | Invitrogen, Teil # Q32853 | ||
| Invitrogen Qubit Teströhrchen | Invitrogen, Teil # Q32856 | ||
| 40% Acrylamid plus 1% N, N'-Methylen-bis-Acrylamid, 37,5:1 | Bio Rad, Teil # 161-0148 | ||
| Tris Base | Sigma, Teil # T1503-1KG | ||
| Borsäure | Sigma, Teil # B6768-500G | ||
| 0,5 M EDTA, pH 8,0 | Teknova, Teil # E0306 | ||
| Ammoniumpersulfat | Sigma, Teil # A3678-25G | ||
| TEMED | BioShop, Teil # TEM001.25 | ||
| Ethidiumbromidlösung | BioShop, Teil # ETB444.10 | ||
| 0,5 M Ammoniumacetat | Teknova, Teil # A2000 | ||
| 10 mM Magnesium-Acetat-Tetrahydrat | Sigma, Teil # M0631-100G | ||
| 1mM EDTA, pH 8,0 | Siehe 0,5 M EDTA, pH 8,0 | ||
| Ethanol | Verschiedene | ||
| Natriumacetat, pH 5,2 | Teknova, Teil # S0297 | ||
| Speed-Vakuum | Verschiedene | ||
| Single-Read Cluster-Generation Kit | Illumina, part # GD-300-1001 | ||
| 36c Sequencing Kit v4 | Illumina, part # FC-104-4002 | ||
10x TBE Rezept
| Beträge | Reagenzien |
| 108 Gramm | Tris Base |
| 55 Gramm | Borsäure |
| 40mL | 0,5 M EDTA (pH 8,0) |
| Add dH 2 O auf 1L markieren | |
12% Polyacrylamidgel Rezept
| Volumes | Reagenzien |
| 5,8 ml | 40% Acrylamid plus 1% N, N'-Methylen-bis-Acrylamid, 37,5:1 |
| 12 ml | dH 2 O |
| 2 ml | 10x TBE |
| 140 ul | 10% Ammoniumpersulfat |
| Gesamtvolumen: 20 ml |
Uptag Primer-Mix:
Resuspendieren Uptag und Buptagkanmx4 jeweils in ddH 2 O bei 100 nM, dann in einem Verhältnis von 1:1 mischen für eine Endkonzentration von 50 uM jeder. Lagerung bei -20 ° C.
Downtag Primer-Mix:
Resuspendieren Dntag und Bdntagkanmx4 jeweils in ddH 2 O bei 100 nM, dann in einem Verhältnis von 1:1 mischen für eine Endkonzentration von 50 uM jeder. Lagerung bei -20 ° C.
Mixed-Oligonukleotide:
Resuspendieren jedem der folgenden acht Oligos (Standard entsalzt) in ddH 2 O bei 100 uM:
Uptag, Dntag, Uptagkanmx, Dntagkanmx, Uptagcomp, Dntagcomp, Upkancomp, Dnkancomp.
Mix ein gleiches Volumen der acht Oligonukleotide für eine Endkonzentration von 12,5 uM jeder.
12X MES Lager:
Für 10 ml, lösen sich 0,7 g MES free-Monohydrat und 1,93 g MES Natriumsalz in 8 ml Molekularbiologie Wasser. Nach dem Mischen gut, pH-Wert prüfen und einstellen, wenn bis zu einem pH von 6,5 bis 6,7 erforderlich. Fügen Sie Wasser bis zu einem Gesamtvolumen von 10 ml. Filter sterilisiert und bei 4 ° C vor Licht (zB Wickeln Sie das Rohr in Folie) geschützt. Ersetzen Sie, wenn Lösung wird sichtbar gelb oder nach 6 Monaten.
2X Hybridisierungspuffer:
Für 50 ml, Mix 8,3 ml 12X MES Lager (ab 2.9.14), 17,7 ml 5 M NaCl, 4,0 ml 0,5 M EDTA, 0,1 ml 10% Tween 20 (vol / vol) und 19,9 ml gefiltert ddH 2 O. Filter sterilisieren.
Wash A: 300 ml 20X SSPE, 1 mL 10% Tween (vol / vol), 699 ml ddH 2 O. Filter sterilisieren.
Wash B: Mix 150 ml 20X SSPE, 1 mL 10% Tween (vol / vol), 849 ml ddH 2 O. Filter sterilisieren.
Barcode Sequenzierungsprimern
In UP-tag Primersequenzen die 5 'tail (fett) sind Illumina Adapter-Sequenzen in der F-und R-Primer enthalten. Die variable Sequenz (kursiv) stellt die 5-mer Indizierung Tag in Multiplexen / index Auslesen verwendet. Die 3 'tail (unterstrichen) vertritt die gemeinsamen Primer flankieren die uptag Barcode und wird benötigt, um die Hefe Barcodes zu verstärken.
In AB-tag Primersequenzen der 5 'Schwanz ist identisch mit 5' Schwanz des UP-tag-Primer (Illumina bestimmten Reihenfolge), aber die 3 'tail (unterstrichen) ist mit dem Gemeinsamen Primer, die verwendet werden, um die Verstärkung ersetzt DOWN- tag Barcodes.
Referenzen
- Giaever, G. Functional profiling of the Saccharomyces cerevisiae genome. Nature. 418, 387-391 (2002).
- Baba, T. Construction of Escherichia coli K-12 in-frame, single-gene knockout mutants: the Keio collection. Molecular systems biology. 2, 2006.0008-2006.0008 (2006).
- Claus, H., Frosch, M., Vogel, U. Identification of a hotspot for transformation of Neisseria meningitidis by shuttle mutagenesis using signature-tagged transposons. Mol Gen Genet. 259, 363-371 (1998).
- Hava, D. L., Camilli, A. Large-scale identification of serotype 4 Streptococcus pneumoniae virulence factors. Molecular microbiology. 45, 1389-1406 (2002).
- Costanzo, M. The Genetic Landscape of a Cell. Science. 327, 425-431 (2010).
- Tong, A. H. Systematic genetic analysis with ordered arrays of yeast deletion mutants. Science. 294, 2364-2368 (2001).
- Pan, X. A robust toolkit for functional profiling of the yeast genome. Molecular cell. 16, 487-496 (2004).
- Schuldiner, M. Exploration of the Function and Organization of the Yeast Early Secretory Pathway through an Epistatic Miniarray Profile. Cell. 123, 507-519 (2005).
- Deutschbauer, A. M. Mechanisms of haploinsufficiency revealed by genome-wide profiling in yeast. Genetics. 169, 1915-1925 (2005).
- Giaever, G. Chemogenomic profiling: identifying the functional interactions of small molecules in yeast. Proceedings of the National Academy of Sciences of the United States of America. 101, 793-798 (2004).
- Lum, P. Y. Discovering modes of action for therapeutic compounds using a genome-wide screen of yeast heterozygotes. Cell. 116, 121-137 (2004).
- Hillenmeyer, M. E. The chemical genomic portrait of yeast: uncovering a phenotype for all genes. Science. 320, 362-365 (2008).
- Oh, J. A universal TagModule collection for parallel genetic analysis of microorganisms. Nucleic acids research. 38, e146-e146 (2010).
- Oh, J. Gene annotation and drug target discovery in Candida albicans with a tagged transposon mutant collection. PLoS pathogens. 6, (2010).
- Nislow, C., Giaever, G., Stark, I., Stansfields, M. J. R. Chapter 387. Yeast Gene Analysis. , 387-414 (2007).
- Pierce, S. E., Davis, R. W., Nislow, C., Giaever, G. Genome-wide analysis of barcoded Saccharomyces cerevisiae gene-deletion mutants in pooled cultures. Nature protocols. 2, 2958-2974 (2007).
- Sambrook, J., Russell, D. W. . Molecular cloning : a laboratory manual. , (2001).
- Smith, A. M. Quantitative phenotyping via deep barcode sequencing. Genome Res. , (2009).
- Hamady, M., Walker, J. J., Harris, J. K., Gold, N. J., Knight, R. Error-correcting barcoded primers for pyrosequencing hundreds of samples in multiplex. Nature. 5, 235-237 (2008).
- Gresham, D. System-Level Analysis of Genes and Functions Affecting Survival During Nutrient Starvation in Saccharomyces cerevisiae. Genetics. 187, 299-317 (2011).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten