Method Article
光活性化単一分子追跡を使用してライブ細菌細胞におけるタンパク質-DNA相互作用を可視化する
要約
単一分子追跡と組み合わさ光活性化局在化顕微鏡(PALM)は、生大腸菌細胞におけるタンパク質-DNA相互作用の直接観察及び定量化を可能にする。
要約
タンパク質-DNA相互作用は、多くの基本的な細胞プロセスの中心にあります。例えば、DNA複製、転写、修復、および染色体の組織は、特定のDNA構造または配列を認識するDNA結合タンパク質によって支配される。 インビトロ実験はまだ、DNA結合タンパク質の多くのタイプの機能のための詳細なモデルを生成するために貢献している、生きた細胞の複雑な環境でのこれらのプロセス、組織の正確なメカニズムははるかに理解残る。我々は最近、単一分子追跡と組み合わせた光活性化ローカライゼーション顕微鏡(PALM)を使用して、ライブ大腸菌細胞内DNA修復活動を定量化する方法を紹介しました。私たちの一般的なアプローチは、染色体との関連の際に単一のタンパク質の移動度の変化によって、個々のDNA結合事象を識別します。結合した分子の割合は、タンパク質の行為のための直接の定量的測定を提供基板や単一細胞レベルでの結合部位のivityと豊かさ。ここでは、方法の概念を説明し、サンプル調製、データ収集、およびデータ分析手順を示す。
概要
このプロトコルは、 大腸菌生細胞におけるタンパク質-DNA相互作用の直接測定を記載する。それは染色体( 図1)に結合するような技術は、単一の蛍光標識されたタンパク質の拡散係数の変化を利用する。我々は、DNAポリメラーゼラギング鎖の複製と除去修復経路1におけるDNAのギャップを埋め、I(POL1)、原型のDNA結合タンパク質を使用する方法を実証すること。
超解像蛍光顕微鏡の出現は、ナノメートル分解能での細胞内の分子構造の可視化を可能にする。光活性化ローカライゼーション顕微鏡(PALM)は、蛍光状態( 図2)に、初期暗状態から起動することができ、蛍光タンパク質を採用しています。すべての標識された分子のサブセットのみのtは、独立に、逐次的にそれらの位置を決定するために、任意の時点で活性化される彼は、サンプル2で標識された分子の濃度を合計。分子当たりの定位の精度は、主に蛍光点広がり関数(PSF)のサイズに依存し、収集された光子の数、およびバックグラウンド信号3。この方法の多くのアプリケーションでは、細胞構造の改良可視化に焦点を当てています。追跡Palmは単一分子と組み合わせることができます実現が4を直接生きた細胞内の標識されたタンパク質の任意の数の動きに追従するために新たな道を開いた。増加した感度と蛍光顕微鏡の時間分解能は現在、細菌の細胞質5に、単一の拡散の蛍光タンパク質の追跡を可能にします。
ここでは、PAmCherry、不可逆的に405 nmの光6の照射により蛍光状態に初期非蛍光状態から変換して設計された蛍光タンパク質を用いている。活性化したPAmCherry蛍光プローブは、イメージすることができます561 nmで励起し、光退色するまで数フレームで追跡することによってD。私たちは、POL 1とPAmCherryの融合を使用して、単一のタンパク質の一過DNA結合事象を同定する方法の能力を実証する。メタンスルホン酸メチル(MMS)による細胞の処理は、塩基除去修復酵素によってギャップのDNA基質になっているDNAメチル化損傷を引き起こす。本手法は、MMSダメージ7に対応して、単一のPOL 1分子の明確な結合を示す。
プロトコル
1。細胞培養
無菌培養チューブとピペットチップを使用してください。 E.大腸菌株 AB1157 のpolA-PAmCherryは POL 1のC末端PAmCherry融合を運ぶ。融合は、Datsenko らに記載のようにラムダ-レッド組換えを用いて野生型遺伝子を置換することにより天然の染色体位置に挿入した。DNA損傷剤メチルに対する細胞増殖速度および感度によって判断されるように、融合タンパク質の8の機能を確認したメタンスルホネート(MMS)。細胞株の構築についての詳細はUphoff らに記載されています。7、Datsenkoら 8、および レイエス-ラモスら 9細胞培養物の自己蛍光を低減し、顕微鏡スライド上のバックグラウンド粒子を避けるために、M9最少培地で増殖させる。あるいは、栄養豊富な定義された培地を、10を用いることができる。
- ストリークE.大腸菌菌株AB1157選択的抗生物質(ここでは、25μg/ mlのカナマイシン)とルリアブロス(LB)アガロースプレート上の凍結グリセロールストックからのpolA-PAmCherryとは、37℃で一晩インキュベートする。
- 単一細胞コロニーから5ミリリットルのLB培地に接種し、3時間220 rpmで振盪37℃で成長する。
- 5ミリリットル中に培養1:10,000に希釈する最少培地(M9培地、MEMアミノ酸がプロリン+、MEMビタミン、0.2%グリセロール)と37℃で一晩220rpmで振とうでインキュベートする。
- 翌朝、分光光度計を用いて光学濃度(OD)を測定し、OD 0.025に5ミリリットル新鮮な最小培地を文化を希釈する。初期の指数期(OD 0.1)に220 rpmで振盪37℃で2時間、成長する。
- 5分間2300×gで遠心分離することによって1.5 mlのマイクロ遠心チューブ内の細胞の1ミリリットルを集中。上清を除去し、20μlの残留培地および渦に細胞ペレットを再懸濁します。
2。顕微鏡スライドPreparatイオン
- のdH 2 Oを1.5%の低蛍光アガロース溶液を調製溶液が透明になるまで、アガロースを溶融するために電子レンジを使用しています。穏やかにピペッティングして、数回上下2X最小培地500μlで、溶融したアガロース溶液500μlを混合します。
- 顕微鏡カバースリップ(ノー1.5厚さ)の中央に均等にアガロース溶液を広げた。これは、アガロースが冷却する前に気泡を避け、迅速に行わなければならない。
- 第二のカバーガラス(ノー1.5厚さ)でパッドを平ら。バックグラウンドの蛍光体粒子を除去するために、カバースリップは、以前は1時間500℃の炉内で燃焼した。焼かカバースリップをアルミ箔で覆われ、室温で数週間保存することができる。
- DNA損傷実験のために、100 mMのMMSを含むアガロースパッドを準備します。ステップ2.1から2.3の手順に従いますが、100 mMのMの最終濃度、溶融された1.5%アガロースを500μlと混合する前にM9培地500μlに8.3μlのMMSを追加MS。 ( 注意!MMSは毒性と変異原性であり、手袋、マスク、ゴーグル、そして白衣を処理する必要があります)。
- パッドから一番上のスライドを削除し、パッド上に濃縮した細胞懸濁液の1を添加する。未使用焼かカバースリップ(ノー1.5厚さ、顕微鏡対物指定に一致)でパッドを覆うことにより、スライド上で、非常に軽く押して、細胞を固定。細胞は、アガロースパッドが乾燥する前に、固定化の45分以内に画像化されるべきである。長い実験中に乾燥を防ぐために、アガロースパッドは、シリコンガスケットを用いて封止することができる。
- DNA損傷実験のために、撮像の前に室温で加湿された容器内で20分間、100mMのMMSを含有するアガロースのパッド上に固定化された細胞をインキュベートする。
3。顕微鏡データ収集の準備
Palmは検出と単一の蛍光タンパク質の正確な位置に依存しています。感度との最適なアラインメント顕微鏡は、データ品質のために重要である。単一分子蛍光顕微鏡は、典型的には、カバーガラス表面上の薄い部分内のみフルオロフォアを励起することによって信号対雑音比を高めるために全内部反射(TIR)照明を採用する。ここでは、E.内部イメージング大腸菌は非常にわずかに励起光の角度を減少させることによって、TIRF顕微鏡で達成することができる傾斜照明11を必要とする。 PAmCherryイメージングはさらに、405 nmの光活性化レーザーおよび561 nmの励起レーザーを必要とする。蛍光発光は、114.5ナノメートル/ピクセルのピクセル長さが得られる倍率でCCD(EMCCD)カメラの電子増倍に記録される。最適なローカリゼーション精度のために、画素サイズは約あまりにも多くのピクセル上に信号を拡散することなく、十分なサンプリングを確保するために、PSFの標準偏差の幅と一致している必要があります。 図3は、最小限のPALMセットアップの模式図を示す。 動画1特注の顕微鏡構築プロセスの印象を与える;楽器の詳細については、Uphoff ら 7を参照してください。
- ルーチン顕微鏡合わせを行う。 405 nmおよび目的の前に561 nmの連続波レーザー強度を測定します。 3.5 MW(〜400 W / cm 2)をし、10μW(〜1 W / cm 2)を 405 nmの強度に561 nmの強度を調整します。 0-10μWから405 nmの強度が徐々に調整することができる連続可変NDフィルターホイールを使用してください。実験の開始まで、レーザー照明のスイッチを切る。
- 顕微鏡ステージ上のサンプルを配置し、透過光顕微鏡モード( 図4A)で焦点に細胞をもたらす。 EMCCDカメラゲインが白とびして、カメラへの損傷を防止するためにスイッチオフされなければならない。
- データサイズを小さくし、カメラの読み出し速度を増加させるためにトリミングさFOVを規定する。
- 周囲の光とSWITからサンプルをカバーEMCCDカメラゲインにCH。
- (0.26ミリカメラの読み出し時間を含む)15.26ミリ秒/フレームにフレームレートを設定します。ディスカッションセクションの「露出時間と励起強度」を参照してください。
- 暗い背景信号( 図4B)を確認するためにカメラデータを表示します。
- 561 nmのレーザーのスイッチを入れ、励起バックグラウンド信号( 図4C)を確認します。
- POL 1-PAmCherry融合タンパク質の光活性化のために405 nmのレーザーのスイッチを入れ、蛍光のPSFが表示されるまで強度を上げる。
- カバーガラスの表面に近いサンプルだけ薄い部分を照明するために励起ビームの角度を調整します。
- このために、レーザビームは100X 1.4 NA対物レンズの後焦点面( 図3)に集光される。ビームに対して垂直に焦点レンズを平行移動すると、離れて角度で目標を終了するには、ビームを引き起こす目的の中心からフォーカスを移動します。
- 人工知能蛍光強度を最大化し、バックグラウンド信号を最小にするmである。厳格なTIR励起がカバースリップ表面の100ナノメートル内の画像蛍光団が最適であることに注意してください、しかし、画像化DNA結合タンパク質を大腸菌に関連付けられている大腸菌核様体は、0.8ミクロンまでの深い照明を必要とします。
4。データ収集
ここでは、Palmの映画を取得するための一般的なプロトコルを記述します。同じ手順が損傷を受けていないE.でPOL 1-PAmCherry融合タンパク質を画像化するために適用される大腸菌の細胞およびMMSに連続DNA損傷治療中。セルごとに異なる分子量またはコピー数の融合タンパク質に対する法の適用(ディスカッションのセクションを参照)別の取得設定が必要になります。
- 透過光顕微鏡モードでのセルの新しい視野(FOV)を見つけて、画像の焦点を合わせる。セルの輪郭( 図4A)を記録するために、カメラのスナップショットを取る。
- 周囲の光からサンプルをカバーし、EMCCDカメラゲインに切り替えます。
- 561 nmのレーザーのスイッチを入れ、データ収集を開始する前に、数秒間、カバースリップ上の細胞の自家蛍光や背景スポットを漂白。細胞増殖させ、M9培地で画像化し、非常に少ない蛍光バックグラウンドは通常存在したカバーガラスを焼い使用しているが、予備漂白することは、LBなどの豊富な増殖培地中で細胞を画像化に有用である可能性があります。強烈な照明がとても予備漂白細胞に対して毒性であることに注意して最小限に抑える必要があります。
- 15.26ミリ秒/フレームで連続561 nmの励起下PALMムービーの取得を開始します。
- 405 nmのレーザーのスイッチを入れ、徐々に、映画の過程で強度を高め、1 W / cm 2の最大に達した。携帯の自家蛍光を引き起こす高い405 nmの強度を避けてください。蛍光分子の密度に注意を払う - それは低い活性化率を維持することが重要であるのPSFが明確になるように各フレーム( 図4D-F)で単離した。
- 万フレーム/ムービー(セルごとに画像化される分子の数に応じて)を記録し、1映画は、通常、2〜3分かかり、FOVの大きさに応じてハードディスクの空き容量0.5〜1 GBのが必要です。
- 複数の視野のための取得手順を繰り返します。 PAmCherryフルオロフォアは、光活性化して不可逆的に漂白付いているので、各FOVは一度だけ撮影することができることに注意してください。
5。データ解析
自動化され、堅牢なデータ解析フレームワークは、方法の性能および効率のために必須である。我々は、MATLABで記述されたカスタム·ソフトウェアを使用しています。
- 14。クロッカーら 12、ホールデンら 13、HoldenI らに記載のアルゴリズムを用いて局在化分析を実行し、ウィーザーら 15のPSFは、第7画素のガウスカーネルを用いたバンドパスフィルタ画像において識別されるの径( 図5A)。候補位置は、バックグラウンド信号( 図5B)の4.5倍の標準偏差上記ピーク画素強度とのPSFに対応しています。候補PSFあたりのローカルで最も明るいピクセルが楕円ガウス関数( 図5C)を装着するための初期推定として機能します。無料のフィットパラメータは次のとおりです。x位置、y位置、X幅、Y幅、回転角度、大きさ、背景のオフセット。楕円形のガウスマスクは、PSFをぼかして変形露光時間中に分子を占めています。
- 同じFOVの透過光顕微鏡画像上にPALM映画のすべてのフレームからの結果(X、Y)版をプロットします。 POL 1-PAmCherryのローカライズは、Eの中央領域内に表示されます大腸菌細胞 ( 図6A)。多くのローカライズは、細胞の外に表示される場合は、ローカライズのしきい値の設定が低すぎる、またはサンプルは、バックグラウンド蛍光粒子を含んでいた。
- 自動追跡分析のために、クロッカーらに記載されたアルゴリズムのMATLABの実装がら 12(ディスカッションセクションの「拡散分析」を参照)を使用することができる。ユーザ定義のトラッキング·ウィンドウ内の後続のフレームに表示される位置は、軌道を形成するように接続される。複数のローカライゼーションは、同じウィンドウで発生した場合には、トラックは一意ステップ長の合計を最小化することによって割り当てられる。単一粒子追跡データから拡散係数を算出する際に様々な検討事項の詳細な議論については、ウィーザーら 15を参照
- このアルゴリズムは、トラックの間に過渡的な点滅または逃したローカライズを考慮して、メモリー·パラメータを使用しています。ここでは、1フレームメモリにパラメータを設定し、より高い値は、長寿命の暗状態でのフルオロフォアを追跡するために使用することができる。
- 次のキャリブレーションの手順に基づいて、適切なトラッキング·ウィンドウを選択します。 POL 1のために、我々は0.57ミクロン(5ピクセル)を使用します。
- ウィンドウ·パラメータを追跡する範囲の追跡アルゴリズムを実行します。トラック( 図6B)を分割していない可能な限り最小のトラッキング·ウィンドウを識別するための追跡窓の関数として、セル毎に測定トラック数を計算します。
- 細胞内の分子の動きの空間分布を視覚化するために同一のFOVの透過光顕微鏡画像上で得られたトラックをプロットする。 POL 1トラックは、単一の細胞( 図6C-D)内に閉じ込められた拡散が表示されるはずです。
- トラックの一部が細胞間を横切るように見える場合、これはトラッキング·ウィンドウは大きすぎ、および/ または光活性化率は( 図6E)が高すぎた選ばれたため、別々の分子が誤ってリンクされていたことを示唆している。
- 連続したローカリゼーション( 図6F)との間にステップ長の累積分布をプロットします。曲線は上昇し、十分に大きな追跡窓スムーズに飽和ウィンドウが小さすぎて選択された場合しかし、カットオフエッジを示しています。
- POL 1の拡散特性を分析するために、N個のステップの合計で各トラックの連続したローカライズ版の間の平均二乗変位(MSD))を計算します。
MSD = 1 /(N-1)Σi = 1から N-1 (X I 1 - X I)2 +(Y I 1 - Y i)を 2。
唯一MSD値の統計的な不確実性を減らすために、少なくとも4段階(N≥5ローカライズ)の付いている曲があります。 - 複数のフレーム( 図6G)を介して変位を計算することにより、遅延時間の範囲にわたり、MSD値の曲線をプロットします。 MSDカーブの形状が観察された分子の動き( 図6H)を分類するのに役立ちます。
- MSDからトラックごとの見かけの拡散係数D *を計算します。
D * = MSD /(4ΔT) - LOC 2 /Δtがσ。
第2のTERMは、その定位誤差を補正する(ここでは、LOCσ= 40 nmおよびΔT= 15.26ミリ秒、ウィーザーらを参照してください。15)。 - FOV( 図7A)のすべてのトラックから測定D *値のヒストグラムをプロットします。
- トラック毎に測定D *値に基づいて、染色体に結合して表示され、個々のPOL 1分子を同定。バウンドの集団別の(を中心としたシャープな分布をD *〜0程度2 /秒)とD * <0.15ミクロン2 /秒のしきい値を設定することで、自由に拡散する分子(広い分布は、Dを中心とした*〜0.9μmの2 /秒)( 図7Aおよび7Dに赤いバー)。
- 損傷を受けていない細胞( 図7A-C)の中およびMMS( 図7D-E)でDNA損傷治療の下で細胞内の局在性、追跡、およびPOL 1のための拡散分析を行う。バインドされたトラックの割合は、DNの直接的な定量的な測定を提供生体内で POL 1の修復活性。
結果
インビボでのタンパク質-DNA相互作用を研究するために追跡光活性化単分子の概念を図1に示されている。 PAmCherry融合タンパク質は、生きたE.で検出され当時のセル当たり未満1分子の周波数で405 nmの光で確率的に単一の分子を光活性化することによって順次で大腸菌細胞 。活性化された分子は、連続561 nm励起の下で撮像される。セル内の分子運動は、不可逆的退色するまで、一連のフレーム内の近くの局在を接続することにより追跡することができる。 DNA結合タンパク質の拡散が染色体に結合する際に減速されるので、トラックごとに得られた見かけの拡散係数D *が直接個々のタンパク質-DNA相互作用について報告する。
図2は、ライブEにPOL 1-PAmCherry融合タンパク質の光活性化を実証大腸菌細胞 。 O 405nmで強度の影響n個の蛍光分子の密度は、 図4に見ることができる。密度はもっぱら405 nmの強度ではなく、追加的に活性化のために利用可能な分子の数によって決定されていないことに注意してください。残りの分子のプールは、PALM映画の過程で枯渇している。
局在化分析は、 図5に示すようPALMムービーのフレーム毎に行われる。私たちは、生きた細胞内の固定された細胞または結合した分子で不動の分子を使用してローカライズ精度を測定した。当社の取得設定は、理論予測3と一致して、σのLOC = 40nmのローカリゼーション精度を与えた。
結果POL 1のローカライズは、大きく分けてEの空間的な組織化をrecapitulating、細胞( 図6A)の中央部を占める大腸菌は 7を核様体。損傷を受けていない細胞内のPOL 1トラックの大半はDIF表示図6Cに示すように、融合。典型的な細胞は、 大腸菌あたり約400 POL 1分子のコピー数と一致して、数百POL 1トラック( 図6D)が含まれています大腸菌細胞1 図6Bおよび6E-Fは、適切なトラッキング·ウィンドウ·パラメータを選択するためのガイダンスを提供する-トラッキングウィンドウが大きすぎる場合、異なる分子が誤ってトラックに連結さになる可能性が高い。トラッキングウィンドウが小さすぎる場合、曲を長いステップで分割されます。 POL 1のためのMSD曲線は細胞閉じ込めた( 図6G)による、より長い遅延時間の短い遅延時間と飽和するために直線的に上昇する。分子運動の種類はMSD分析により同定することができる。監督の動きが放物線を与え、ブラウン運動を直線が特徴です。閉じ込められた拡散曲線がプラトーに達した。不動の粒子について、MSD曲線のオフセットLを表し、ocalizationの不確実性( 図6H)。単一粒子の追跡とトラブルシューティングのヒントに関する追加情報は、アルノーら 16に記載されています
我々は以前に、外因性DNAアルキル化7損傷に応答してPOL 1のDNA修復活性を測定する方法を適用した。 POL 1のD *のヒストグラムが分子( 図7A-C)の拡散が支配的集団を示す無傷の細胞内で追跡する。 2.7%バウンドPOL 1分子のごく一部は、おそらくラギング鎖の複製と内因性DNA損傷の修復に関与している。連続100 mMのMMSの損傷の下では、13.8%( 図7D)にD *〜0程度2 /秒に増加したトラックの人口。これらのトラックは、塩基除去修復経路の一部として、一塩基ギャップを埋めるためにDNA修復合成を行う個々のPOL1分子を表す。バインドされたトラックの位置は、個々の位置を示しDNA損傷と修復のサイト( 図7E-F)。
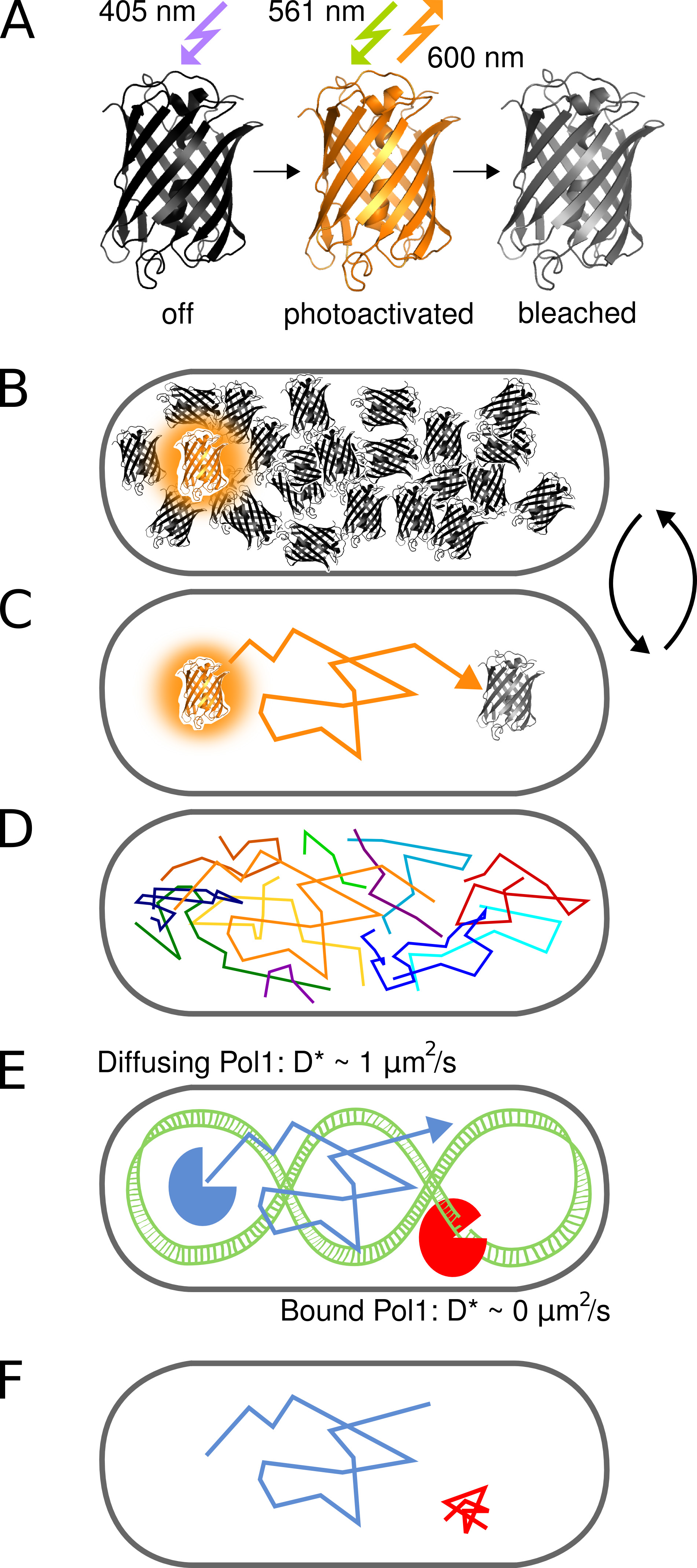
図1。方法のグラフ表示。(A)蛍光タンパク質PAmCherry 405 nmの光照射により初期非蛍光状態から光活性化することができる。明状態では561 nmで励起され、不可逆的に発蛍光漂白剤まで、600付近の蛍光を発する。 (B)光活性化速度を制御するイメージングつだけは、確率的に、まだアクティブにされていないか、すでに暗い状態のままで漂白された分子の任意の大きさのプールながらいつでもセル当たりPAmCherry融合タンパク質を活性化することを可能にする。 (C)蛍光分子の位置は、単離されたPSFとのtrの中心から決定される退色するまで数フレーム肯定応答。多くの分子の(D)トラックが順次記録される。 (EF)は、染色体標的配列または構造を有するDNA結合タンパク質の相互作用は、ランダムな拡散運動を停止させる。結合および非結合分子は、D *シングルトラックから抽出された見かけの拡散係数によって区別されます。結合した分子の結果の割合は、 生体内で DNA結合タンパク質の活性のための定量的な尺度を提供します。 拡大画像を表示するにはここをクリックしてください。
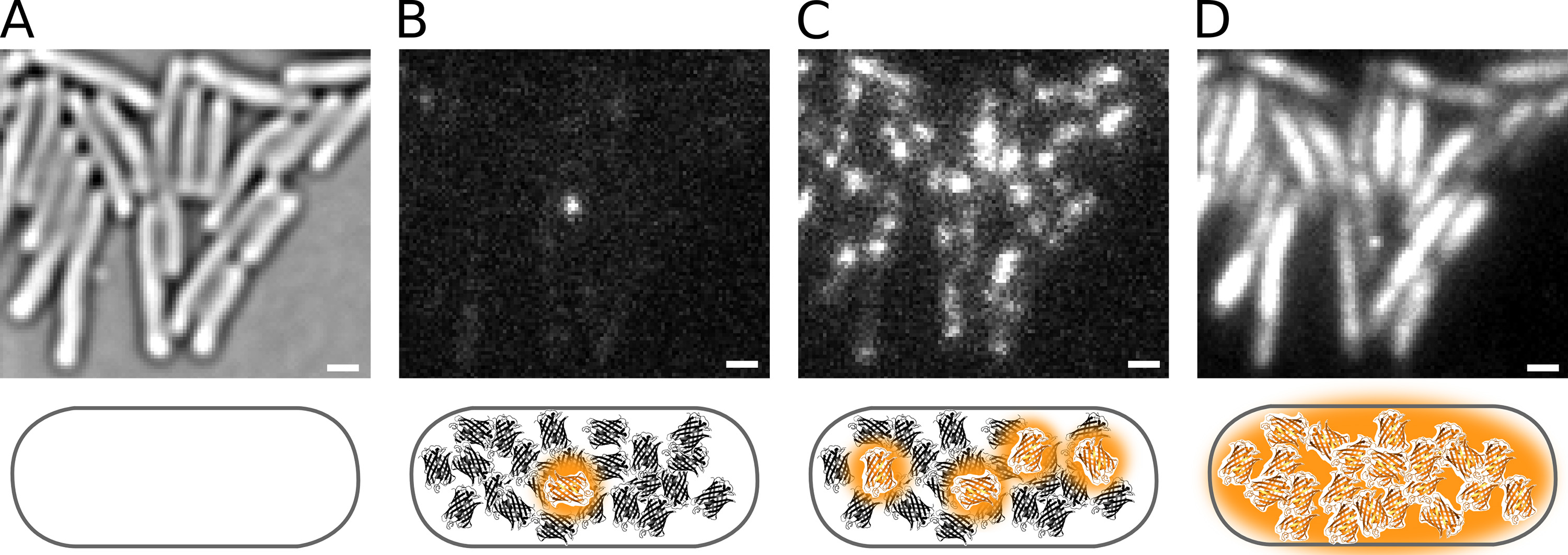
図2。ライブEのPOL 1-PAmCherryの光活性化大腸菌細胞 。スケールバー:1μmである。回路図は、各パネルの下に表示されます。 ( A)アガロースパッドの上に固定化された細胞の光学顕微鏡画像を送信した。 (b)1つのセル内の単一のPAmCherryの蛍光体をPhototactivating。 (C)より高い光活性化速度は、蛍光分子の数を増加させる。 (D)PALMの映画から統合PAmCherry蛍光。 拡大画像を表示するにはここをクリックしてください。
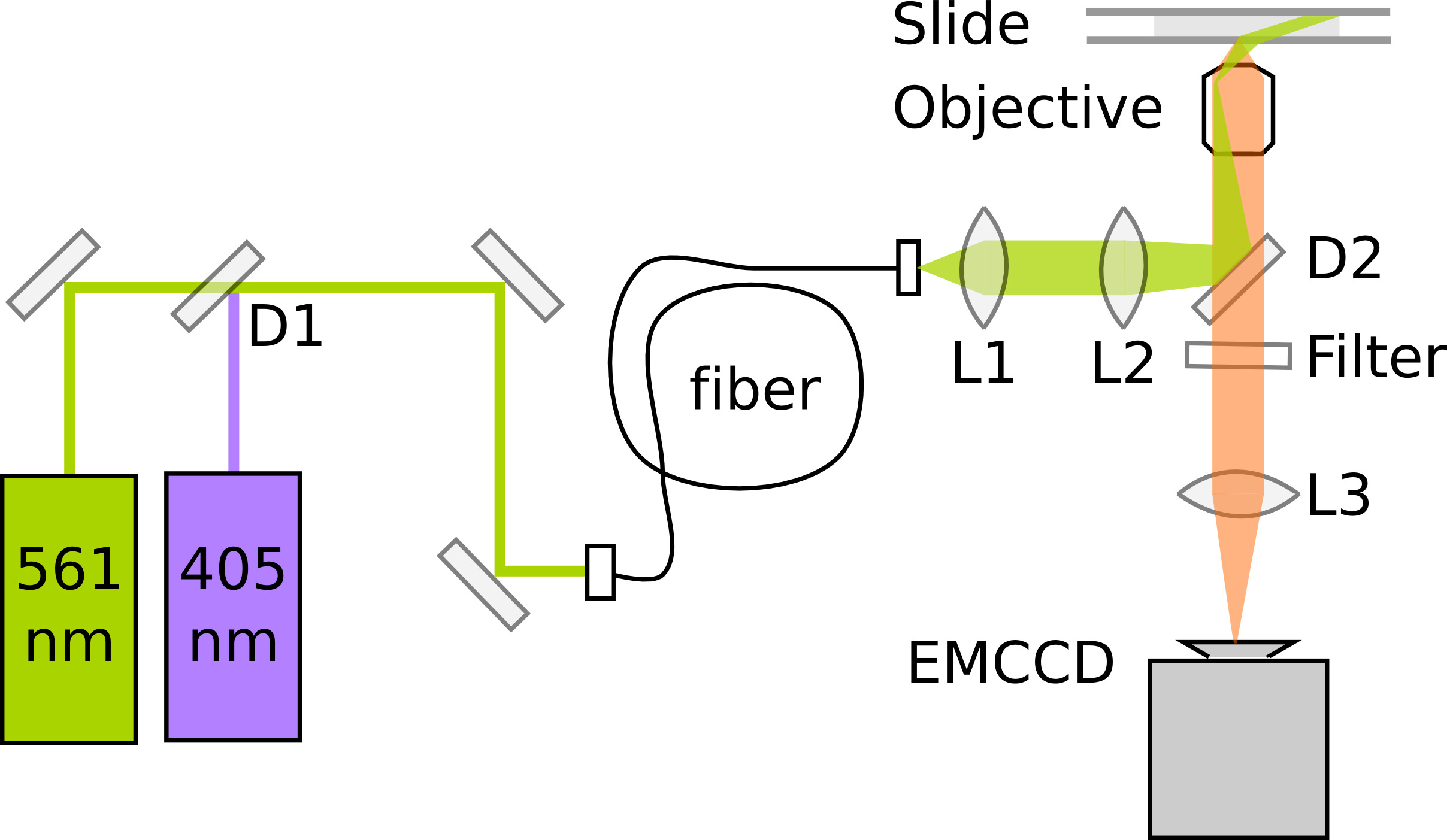
図3。光活性化およびイメージングPAmCherry融合タンパク質のための最小限のPALMセットアップの概略D1:。ダイクロイックミラー( 例えば 550nmのロングパス)。 D2:ダイクロイックミラー( 例えば 570nmのロングパス)。 L1:コリメートレンズ。 L2:TIRレンズ。 L3:チューブレンズ。1177/51177fig3highres.jpg "ターゲット=" _blank ">拡大画像を表示するにはここをクリックしてください。

図4。 15.26ミリ秒/フレームパームの映画から代表的な画像スケールバー:1μmである。 (A)は、アガロースパッド上に固定化された細胞の光像を送信します。 (B)レーザによるEMCCDカメラで測定された暗い背景画像はオフ。光活性化の前に連続561 nmの励起下(C)励起の背景画像。 (DF)の増加405nmの強度は、561 nmの連続的な励起の下で撮像さPAmCherryの高い光活性化率をもたらす。箱入りの領域は、以下に拡大表示されます。 (D)低405 nmの強度(<1μW)活性物質視野あたりの非常に少数の蛍光分子。 (E)中405ローカリゼーションおよび追跡、分析のために良いPSF密度の波長の強度(〜2μW)光活性化の結果。 (F)より高い405 nmの強度(〜10μW)の局在と、トラッキング解析を不明瞭いくつかのセルに複数の蛍光分子を活性化する。 拡大画像を表示するにはここをクリックしてください。

図5。ローカリゼーション解析のイラスト。スケールバー:1μmの(A)バンドパスフィルタリングは、スプリアス画素ノイズを除去し、FOVにわたる強度勾配を平坦化する。 (B)候補のPSFは、偽陽性および偽陰性検出を最小化するように選択されたユーザ定義の閾値に基づいてフィルタリングされた画像内で識別される。ゲートしldは、バックグラウンドの標準偏差(信号対雑音比、SNR)で割った候補画素の最小の強度に対応する。 (C)閾値は、二次元の楕円ガウスフィットのための初期ローカライゼーション推測(オレンジ断面)となる局所的に明るい画素を通過する。スケールバー:0.5ミクロン。その結果、超解像の局在(ブルークロス)= 40 nmのσのLOCの平均精度を持っています。 拡大画像を表示するにはここをクリックしてください。
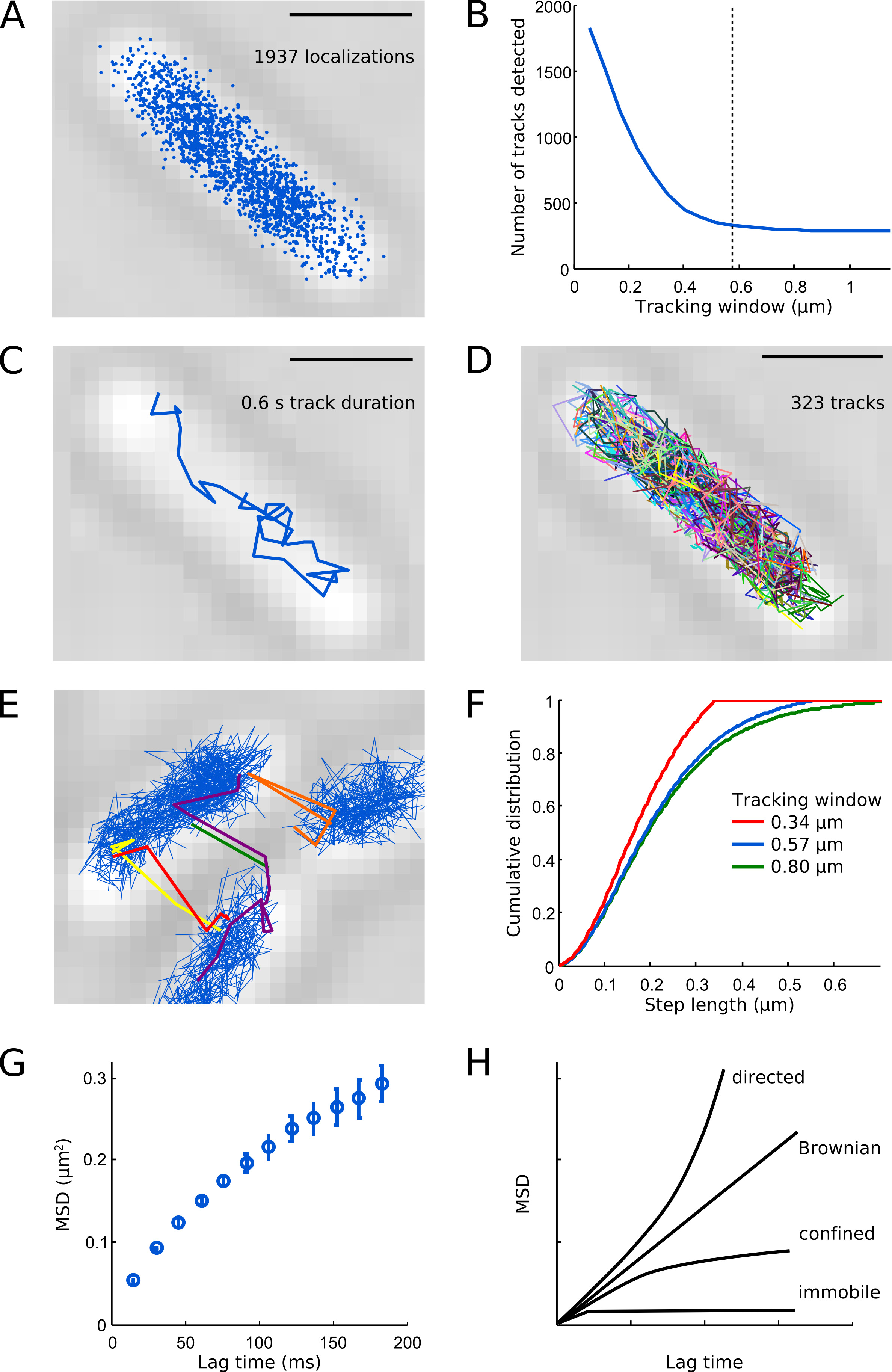
図6のトラッキング解析のイラストスケールバー:1μmである。 (a)すべてのは、例えば、細胞内のPOL 1-PAmCherryの局在を検出しました。 (B)トラック数を検出トラッキングウィンドウの関数として、例えば、細胞内編。小さな追跡窓はトラックの高い数を人為的につながる分子軌道を、分割します。破線はトラッキング·ウィンドウ·パラメータ(0.57ミクロン、5ピクセル)のための我々の選択を示している - これはステップの完全な分布を検出し、無傷の異なる分子の軌道を維持するとの間の良好な妥協点を提供します。 (C)シングルPOL 1-PAmCherry分子の例トラック。 (D)ランダムな色で示されているすべての測定された曲。トラッキングウィンドウが選択された場合(E)トラッキングアーチファクト(ここでは0.8ミクロン、7ピクセル)又はフレーム当たりのPSFの密度が高すぎる大きすぎる。ウィンドウを追跡するためのステップ長の(F)の累積分布:0.34ミクロン(3ピクセル、赤い線)、0.57ミクロン(5ピクセル、青線)、および0.80ミクロン(7画素、緑色の線)。 0.34ミクロンのトラッキングウィンドウは0.34ミクロンより長い手順を遮断することに注意してくださいこれを明確にステップの完全な分配を切り捨てます。 0.80ミクロンの追跡ウィンドウが行うように0.57ミクロントラッキング·ウィンドウには、ステップのほぼ同じ分布を検出する。 (G)、MSD曲線はPOL 1の閉じ込められた拡散を示しています。 (H)監督の運動、ブラウン運動、限られた拡散、および不動粒子の回路図MSDカーブを。 拡大画像を表示するにはここをクリックしてください。
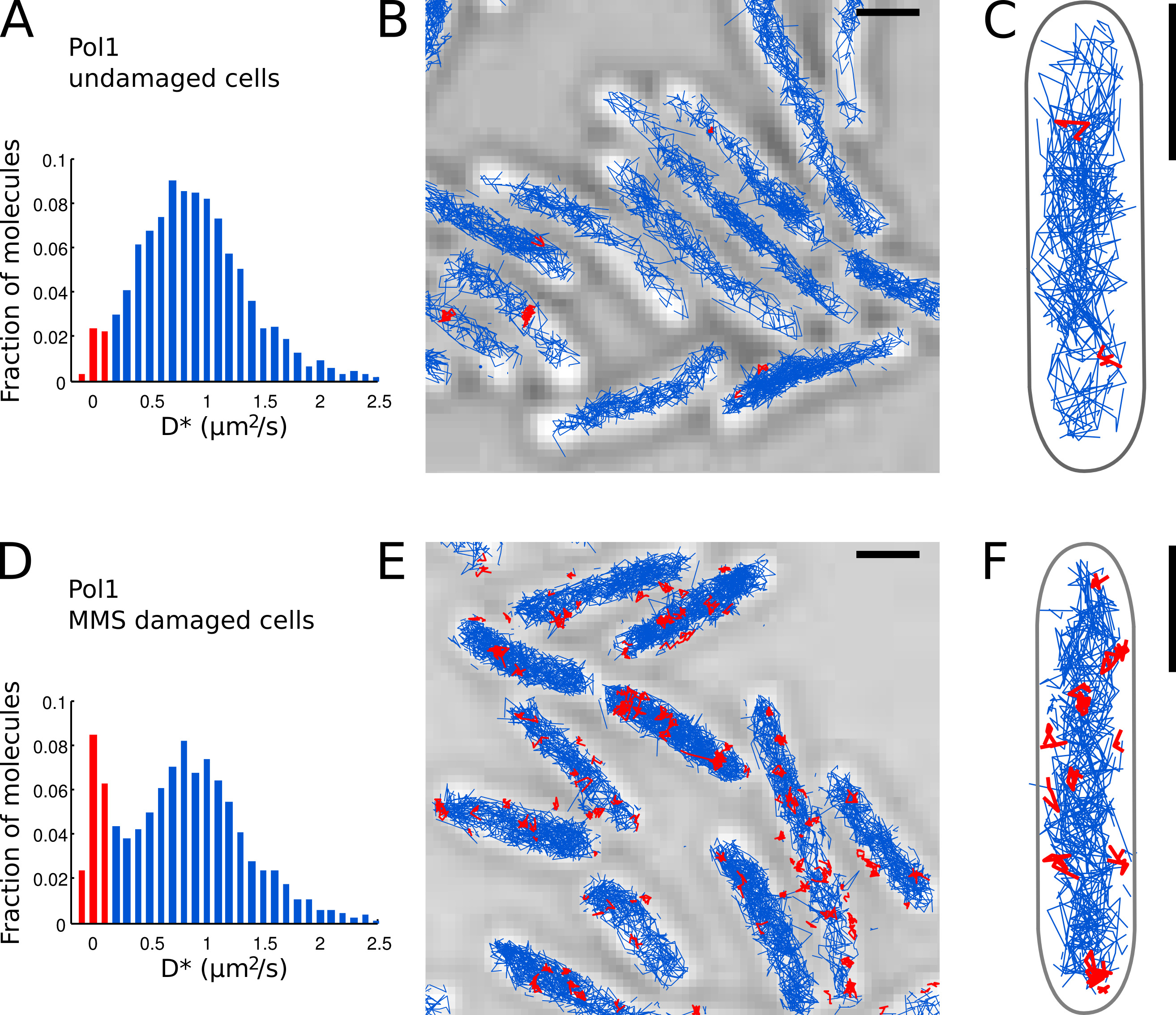
図7。ライブEのPOL 1のDNA修復活性を直接測定大腸菌細胞スケールバー:1μmである。 (A)損傷を受けていない細胞(n = 4162曲)のFOV内の4つ以上のステップの全てのトラックのための見かけの拡散係数D *のヒストグラム。 Bに分類分子の集団 ound赤で強調表示されます。 POL 1-PAmCherryの(BC)トラックは透過光顕微鏡画像に表示されます。その拡散係数に応じて結合すると分類トラックは赤で表示されます。 (D)D *(N = 2,128曲)を画像化する前に、100mMのMMSアガロースパッド上に固定化し、20分間インキュベートした細胞において測定POL 1トラックのヒストグラム。 DNA修復に従事して結合した分子の集団が赤で表示されている。 (EF)POL 1-PAmCherry赤文字の一つのPOL 1のDNA修復イベントのトラックを示す透過光顕微鏡画像上で追跡します。 拡大画像を表示するにはここをクリックしてください。

映画の1。オーダーメイドのPALMのセットアップを構築する 。JoVE_Uphoff_Movie1.avi "ターゲット=" _blank ">ビデオを見るにはここをクリックしてください。
ディスカッション
私たちは、実験の成功のためのいくつかの重要な考慮事項について説明します。
選択と蛍光融合タンパク質の発現:光活性化とphotoswitchable蛍光タンパク質17の大規模なパレットがあります。具体的な選択は、顕微鏡の特性、利用可能な特にレーザーとフィルタに依存します。 405 nmおよび561 nmでの組み合わせは、一般的な光活性化蛍光タンパク質に最適です。我々は、それが単量体であるのでPAmCherry 6を選択し、細胞内で全く凝集を示さなかった。さらに、不可逆的光活性化は、細胞あたりのタンパク質のコピー数を測定するために活性化フルオロフォアの数を数えることができる。代わりにプラスミドから融合タンパク質を発現する、我々は、野生型遺伝子座での融合タンパク質をコードする遺伝子の染色体挿入を好む。これは蛍光で目的のタンパク質の完全な置き換えをVE確実にrsionおよび野生型発現レベルを維持する。
光活性化率:これは、平均で細胞あたり未満の分子が、ムービーの任意のフレームで蛍光状態にあるように、光活性化速度を調整することが重要である。これは、405 nmの強度および活性化されたままの分子の数に依存する。非常に低い画像密度で、しかし、すべての分子が、映画の終わりの前に画像化されるか、非常に長いムービーは、取得する必要があります。映画ごとに記録されたフレーム数は、細胞あたりの融合タンパク質のコピー数および使用される励起条件でPAmCherryの寿命を光退色の平均に依存する。 POL 1のコピー数は、〜400分子/細胞1で、指数関数的な光退色寿命分布の平均値は約4フレームでした。徐々に405 nmの強度を増加させることによって、活性化は、均等に映画館10,000フレームにわたって分配される。したがって、各セルはoccupある万フレームの映画の中でのPSFとトラッキング合併症のほとんど重複を保証する、〜1600フレームの合計のために蛍光分子でIED。
露出時間と励起強度は:まず、露光時間は少し動きボケとのシャープのPSFを観察するのに十分に短いことが必要である。しかし、フレームレートが定位不確かさを超えて連続したフレーム間の観察可能な分子運動を生じるように選択されるべきで、それ以外の場合は重大な光子は、トラックをオーバーサンプリングすることによって浪費される。非結合分子の運動は、ローカリゼーションの不確実性のために結合した分子の見かけの動きとは明確に区別できるように、十分に長い時間間隔でサンプリングされなければならない。露光時間が設定されると、PSF強度が調整されるべきである。 PSFの定位の精度は、フレームの持続時間にわたって検出された光子の数と共に増加する。より高い励起強度は、光子放出の速度を増加させる府Tも光退色率とバックグラウンド信号。希望のローカライズ精度が得られ、最も低い励起強度を使用してください。 POL 1-PAmCherryのために我々は15.26ミリ秒/フレームと3.5 mWの561 nm励起(400 W / cm 2)を選んだ。これは、(Uphoff ら 7に補足情報を参照)、データ収集の前および後の細胞増殖および形態を監視することにより、特定の撮像条件についての細胞生存率を確認することが重要である。
POL 1は、in vivo 7 におけるギャップDNA基質に〜2秒の結合時間を示し、したがって、我々は、分子の大部分がトラックの全期間のどちらか結合または非結合状態にあることを期待しています。染色体部位が拡散係数を持っているので、結合した分子は、細胞質中のPOL 1拡散(〜1μmの2以上(エルモアら 18、〜10 -5程度2 /秒)数桁低く、本質的に不動現れる /秒)。
拡散分析:*は、個々のトラックのMSDから算出される見かけの拡散係数D、統計誤差を低減するために4つのステップ(5フレーム)の最小にわたって平均。 〜分子の75%が記載の撮像条件に対して5未満のフレーム内で漂白することに留意されたい。このような短いトラックが結合および非結合分子を区別するために十分な統計的な確実性を提供していない。しかし、タンパク質の活性を報告結合および非結合分子の相対的な画分を分析した総トラック数とは無関係である。
不確実性は、分子15の各ローカライズには明らかにランダムステップが追加されますので、それは、D *の計算にPSFのローカライズエラー(σLOC)を説明するために役立ちます。
バウンドして拡散する分子の分類を向上させるために、我々は、D *ボットを計算することをお勧めします2つのフレームの経時的な単一工程の変位及び変位の時間。のD *(15ミリ秒)<0.15ミクロン2 /秒とD *(30ミリ秒)<0.075ミクロン2 /秒:これは、2つの別々のD *のしきい値を設定することが可能である。
D *は、露光時間中に拡散によるボケトラックや動きの細胞の閉じ込めに影響される見かけの拡散係数であることに留意されたい。正確な公平な拡散係数を抽出するためには、確率的ブラウン運動モデル5,7に基づいて、シミュレートされたデータに観測された動きを比較するために有用であることがわかった。シミュレートされたデータは、データ分析手順を試験するために使用することができる。
この方法の潜在的なアプリケーション:我々 は、染色体に結合すると、タンパク質の移動度の変化により、生体内でのタンパク質-DNA相互作用を可視化し、定量化するための一般的なアプローチを説明した。 DNA-または活動修復に関与するRNA-結合タンパク質は、複製、転写、および染色体維持は、光学回折限界以下の空間分解能を有する単一細胞レベルでリアルタイムで追跡することができる。光活性化単一分子追跡は、セルあたり数標識分子に制限されている従来の追跡方法を拡張します。 生体内での分子拡散を測定別の方法は、(FRAP)を光退色後の蛍光回復です。 FRAPが大きいセルのグローバル拡散特性を測定するために非常に有用であるが、それは特に小さい細菌細胞のために、空間的に異機種環境で異なる移動度を持つ複数の分子種を解決する能力が制限されている。
我々は、DNA結合活性およびEの異なるタンパク質の範囲の細胞内局在を測定するために追跡する光活性化単一分子を適用しているPOL1、DNAリガーゼ、FISタンパク質、DNAを含む大腸菌ポリメラーゼIII 7だけでなく、染色体タンパク質MukB、E、Fの19の構造維持。我々は、この方法はまた、他の細胞タイプに適合させることができると予想している。
開示事項
著者は、彼らが競合する金融利害関係はありません宣言します。
謝辞
私たちは、ローカリゼーションソフトウェアの特注顕微鏡とシェイマス·ホールデンの建設に助けをジャスティン·ピンクニーとヨハネスHohlbeinを認める。ロドリゴ·レイエス·ラモスは、Eを提供するために感謝している大腸菌菌株。研究はANKに、欧州委員会第七次フレームワーク·プログラム·グラントFP7/2007-2013健康F4-2008から201418、英国バイオテクノロジー·生物科学研究会議助成BB/H01795X/1、欧州研究会議助成261227によって資金を供給された。 DJSは、ウェルカムトラストプログラムグラントWT083469によって資金を供給された。 SUは、MathWorksの博士フェローシップによってサポートされていました。
資料
| Name | Company | Catalog Number | Comments |
| E. coli strain carrying a chromosomal insertion for a PAmCherry DNA-binding fusion protein | created by Lambda-Red recombination | ||
| MEM amino acids | Invitrogen | 11130-051 | minimal medium supplement |
| MEM vitamins | Invitrogen | 11120-052 | minimal medium supplement |
| Agarose | BioRad | 161-3100 | certified molecular biology grade |
| Microscope coverslips No 1.5 thickness | Menzel | BB024060SC | remove background particles by heating slides in furnace at 500 °C for 1 hr |
| Methyl methanesulfonate (MMS) | Sigma-Aldrich | 129925 | CAUTION: toxic |
| PALM setup | home-built | described in Uphoff et al.7 | |
| MATLAB | MathWorks | for data analysis and visualization | |
| Localization software | custom-written, available online | http://www.physics.ox.ac.uk/Users/kapanidis/Group/Main.Software.html | MATLAB and C++ software package that can be adapted for localization analysis. Cite Holden et al.13 if used in publication. |
| Tracking software | available online | http://physics.georgetown.edu/matlab/ | MATLAB implementation by Blair and Dufresne. Cite Crocker & Grier12 if used in publication. |
参考文献
- Friedberg, E. C. . DNA Repair and Mutagenesis. , (2006).
- Betzig, E., et al. Imaging intracellular fluorescent proteins at nanometer spatial resolution. Science. 313 (5793), 1642-1645 (2006).
- Mortensen, K. I., Churchman, L. S., Spudich, J. A., Flyvbjerg, H. Optimized localization analysis for single-molecule tracking and super-resolution microscopy. Nat. Methods. 7 (5), 377-381 (2010).
- Manley, S., et al. High-density mapping of single-molecule trajectories with photoactivated localization microscopy. Nat. Methods. 5 (2), 155-157 (2008).
- English, B. P., et al. Single-molecule investigations of the stringent response machinery in living bacterial cells. Proc. Natl. Acad. Sci. U.S.A. 108 (31), (2011).
- Subach, F. V., et al. Photoactivatable mCherry for high-resolution two-color fluorescence microscopy. Nat. Methods. 6 (2), 153-159 (2009).
- Uphoff, S., Reyes-Lamothe, R., Garza de Leon, F., Sherratt, D. J., Kapanidis, A. N. Single-molecule DNA repair in live bacteria. Proc. Natl. Acad. Sci. U.S.A. 110 (20), 8063-8068 (2013).
- Datsenko, K. A., Wanner, B. L. One-step inactivation of chromosomal genes in Escherichia coli K-12 using PCR products. Proc. Natl. Acad. Sci. U.S.A. 97 (12), 6640-6645 (2000).
- Reyes-Lamothe, R., Possoz, C., Danilova, O., Sherratt, D. J. Independent positioning and action of Escherichia coli replisomes in live cells. Cell. 133 (1), 90-102 (2008).
- Neidhardt, F. C., Bloch, P. L., Smith, D. F. Culture medium for enterobacteria. J. Bacteriol. 119 (3), 736-747 (1974).
- Tokunaga, M., Imamoto, N., Sakata-Sogawa, K. Highly inclined thin illumination enables clear single-molecule imaging in cells. Nat. Methods. 5 (2), 159-161 (2008).
- Crocker, J. C., Grier, D. G. Methods of digital video microscopy for colloidal studies. J. Colloid. Interface. Sci. 179 (1), 298-310 (1996).
- Holden, S. J., et al. Defining the limits of single molecule FRET resolution in TIRF microscopy. Biophys. J. 99 (9), 3102-3111 (2010).
- Holden, S. J., Uphoff, S., Kapanidis, A. N. DAOSTORM: an algorithm for high-density super-resolution microscopy. Nat. Methods. 8 (4), 279-280 (2011).
- Wieser, S., Schütz, G. J. Tracking single molecules in the live cell plasma membrane-Do's and Don't's. Methods. 46 (2), 131-140 (2008).
- Arnauld, S., Nicolas, B., Hervé, R., Didier, M. Multiple-target tracing (MTT) algorithm probes molecular dynamics at cell surface. Protoc. Exch. , (2008).
- Lippincott-Schwartz, J., Patterson, G. H. Photoactivatable fluorescent proteins for diffraction-limited and super-resolution imaging. Trends Cell Biol. (11), 555-565 (2009).
- Elmore, S., Müller, M., Vischer, N., Odijk, T., Woldringh, C. L. Single-particle tracking of oriC-GFP fluorescent spots during chromosome segregation in Escherichia coli. J. Struct. Biol. 151 (3), 275-287 (2005).
- Badrinarayanan, A., Reyes-Lamothe, R., Uphoff, S., Leake, M. C., Sherratt, D. J. In vivo architecture and action of bacterial Structural Maintenance of Chromosome proteins. Science. 338 (6106), 528-531 (2012).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved