Method Article
Визуализация взаимодействий белок-ДНК в живых бактериальных клеток с использованием отслеживания одиночных молекул фотоактивируемые
В этой статье
Резюме
Фотоактивируемые локализации микроскопии (PALM) в сочетании с отслеживанием одиночных молекул позволяет прямое наблюдение и количественное определение взаимодействий белок-ДНК в живых кишечной клетки Escherichia.
Аннотация
Взаимодействия белок-ДНК находятся в центре многих фундаментальных клеточных процессов. Например, репликация ДНК, транскрипция, ремонт и организации хромосом регулируются ДНК-связывающих белков, которые распознают специфические структуры ДНК или последовательности. В экспериментах пробирке помогли генерировать подробные модели для функции многих видов ДНК-связывающих белков, но , точные механизмы этих процессов и их организации в сложных условиях живой клетки остаются гораздо менее понятны. Мы недавно представила метод для количественного репарации ДНК деятельность в живых кишечной палочки клеток с помощью фотоактивируемые локализации микроскопии (PALM) в сочетании с отслеживанием одиночных молекул. Наш общий подход идентифицирует отдельные события ДНК-связывающий изменением подвижности одного белка при ассоциации с хромосомой. Доля связанных молекул обеспечивает прямую количественную меру для акта белкаivity и обилие субстратов или сайтов связывания на уровне одноклеточного. Здесь мы описываем концепцию метода и продемонстрировать пробоподготовки, сбора данных, а также процедуры анализа данных.
Введение
Этот протокол описывает прямое измерение взаимодействий белок-ДНК в живой кишечной палочки клетки. Методика использует изменение коэффициента диффузии одного флюоресцентно меченого белка, как он связывается хромосомы (рис. 1). Для демонстрации метода мы используем ДНК-полимеразы I (Pol1), прототипом ДНК-связывающего белка, который заполняет пробелы ДНК в отстающей репликации нитей и иссечение ремонт путей 1.
Появление супер-разрешением флуоресцентной микроскопии позволяет визуализацию молекулярных структур в клетках с нанометровым разрешением. Фотоактивируемые Локализация микроскопии (PALM) использует флуоресцентные белки, которые могут быть активированы из начального темной состояния в флуоресцентного состояния (рис. 2). Только подмножество всех меченых молекул активирована в любое время, чтобы определить свои позиции в последовательном порядке, независимо от тон суммарной концентрации меченых молекул в образце 2. Точность локализации на молекулу в основном зависит от размера флуоресцентного функции рассеяния точки (ФРТ), количество собранных фотонов, а фоновый сигнал 3. Многие приложения этого метода сосредоточиться на улучшение визуализации клеточных структур. Осознание того, что PALM можно комбинировать с одной молекуле отслеживания 4 открывает новые возможности для прямого следовать за движением произвольным количеством меченых белков в живых клетках. Повышенная чувствительность и временное разрешение флуоресцентных микроскопов в настоящее время позволяют отслеживать отдельных диффундирующих флуоресцентных белков в бактериальной цитоплазме 5.
Здесь мы используем PAmCherry, проектированный флуоресцентный белок, который необратимо преобразует от начального нефлуоресцентный состояния в флуоресцентного состояния при облучении 405 нм света 6. Активированный PAmCherry флуорофоры может быть изображениег возбуждением на 561 нм и не найдена в течение нескольких кадров до фотообесцвечивания. Мы продемонстрировать способность метода для выявления переходных ДНК-связывающих события одного белков с использованием слияние Pol1 и PAmCherry. Обработка клеток с метилметансульфоната (MMS) вызывает повреждение ДНК метилирования, которая включена в гэп субстратов ДНК по базового эксцизионной репарации ферментов. Наш метод показывает ясно связывание одиночных молекул Pol1 в ответ на MMS повреждения 7.
протокол
1. Культура клеток
Используйте стерильные культуры трубки и наконечники. Е. штамм АВ1157-Пола-PAmCherry несет С-концевой PAmCherry слияние Pol1. Слитый был вставлен в родном месте хромосомной путем замены гена дикого типа с помощью лямбда-красный рекомбинации, как описано в Даценко и соавт. 8 функциональность гибридного белка было подтверждено чем можно судить по сотовой темпы роста и чувствительности к ДНК повреждающего агента метил метансульфонат (MMS). Более подробную информацию о построении клеточной линии, можно найти в Uphoff соавт. 7, Даценко и соавт. 8, и Reyes-Lamothe соавт. 9 Клеточные культуры выращивают на минимальной среде М9, чтобы уменьшить аутофлюоресценция и избежать фоновых частиц на предметное стекло микроскопа. В качестве альтернативы, богатой питательными веществами среде определенного могут быть использованы 10.
- Подряд Е. Штамм AB1157-Пола-PAmCherry из замороженного раствора в глицерине на Лурии бульон (LB) агарозном пластины с селективных антибиотиков (здесь, 25 мкг / мл канамицина) и инкубировать при 37 ° С в течение ночи.
- Инокуляции культуры 5 мл LB из колонии клеток одного и расти при 37 ° С на качалке при 220 оборотах в минуту в течение 3 часов.
- Развести культуру 1:10000 в 5 мл минимальной среде (среде М9, MEM аминокислоты + пролин, MEM витаминов, 0,2% глицерин) и инкубируют при 37 ° С на качалке при 220 оборотах в минуту в течение ночи.
- На следующее утро, измерить оптическую плотность (ОП) с помощью спектрофотометра и разбавить культуры в 5 мл свежего минимальная среда для OD 0,025. Расти в течение 2 часов при 37 ° С на качалке при 220 оборотах в минуту в начале экспоненциальной фазы (OD 0.1).
- Концентрат 1 мл клеток в 1,5 мл трубки микроцентрифужных путем центрифугирования при 2300 мкг в течение 5 мин. Удалить супернатант и ресуспендируют осадок клеток в 20 мкл остаточной среды и вихря.
2. Микроскоп Презентация Preparatион
- Подготовьте 1,5% с низким уровнем флуоресценции раствора агарозы в дН 2 O. Используйте микроволновую печь для плавления агарозы, пока раствор не станет прозрачным. Смешайте 500 мкл расплавленного раствора агарозы с 500 мкл 2х минимальной среде, осторожно пипеткой вверх и вниз несколько раз.
- Распространение раствора агарозы равномерно по центру микроскопа покровное (Нет 1,5 толщины). Это должно быть сделано быстро, пока агарозном охлаждает, избегая пузырьков.
- Свести площадку со вторым покровным (Нет 1,5 толщины). Для удаления фонового флуоресцентные частицы, покровные ранее сжигают в печи при 500 ° С в течение 1 часа. Сгоревшие покровные можно хранить в течение недели при комнатной температуре, покрытой алюминиевой фольгой.
- Для экспериментов повреждения ДНК, подготовить агарозном площадку, содержащий 100 мМ MMS. Выполните процедуру шагов 2.1-2.3, но добавить 8,3 мкл MMS до 500 мкл среде М9 перед смешиванием с 500 мкл расплавленного 1,5% агарозы, для конечной концентрации 100 мМ ММС. (Внимание! MMS является токсичным и мутагенным и должны быть обработаны с перчатки, маски, защитные очки, и халате).
- Снимите верхнюю слайд от блокнота и добавить 1 мкл концентрированной суспензии клеток на площадку. Остановите клетки, покрывая площадку с неиспользованным сожгли покровного (Нет 1,5 толщины, соответствующие объектива микроскопа спецификации) и, нажав очень мягко на слайде. Клетки должны быть отображены в 45 мин иммобилизации до агарозном площадку сохнет. Для предотвращения высыхания при длительных экспериментов, агарозные колодки могут быть запечатаны с помощью кремниевых прокладки.
- Для экспериментов повреждения ДНК, инкубировать клетки, иммобилизованные на агарозном площадку, содержащем 100 мМ MMS в течение 20 мин в увлажненной контейнере при комнатной температуре перед визуализации.
3. Подготовка микроскопии сбора данных
PALM опирается на выявление и точной локализации отдельных флуоресцентных белков. Чувствительность и оптимальная расстановкамикроскоп имеет решающее значение для качества данных. Одиночных молекул флуоресцентных микроскопов обычно используют полного внутреннего отражения (ПВО) освещения для повышения отношения сигнал-шум, возбуждая только флуорофорами внутри тонкой части над поверхностью покровного. Здесь, изображения внутри Е. палочка требует высокой наклонной подсветки 11, которая может быть достигнута на TIRF микроскопа, слегка уменьшении угла возбуждающего света. Томография PAmCherry далее требует фотоактивации лазер 405 нм и лазерного возбуждения в 561 нм. Флуоресценции регистрируется на электрон умножения CCD (EMCCD) камеру при увеличении в результате чего длина пикселя 114,5 нм / пиксель. Для оптимальной точности локализации, размер пикселя должен примерно соответствовать стандартной ширине отклонение PSF, чтобы обеспечить достаточную выборку, не распространяя сигнал на слишком многих точек. Рисунок 3 показана схема минимальной настройки PALM. Фильм 1дает представление о заказ процесса микроскоп потенциала; см. Uphoff др. 7 для подробного описания прибора..
- Выполнение программы выравнивания микроскопа. Измерьте 405 нм и 561 нм непрерывного излучения лазера интенсивности в перед объективом. Отрегулируйте интенсивность 561 нм до 3,5 мВт (~ 400 Вт / см 2) и 405 интенсивности нм до 10 мкВт (~ 1 Вт / см 2). Используйте бесступенчатую фильтр нейтральной плотности колесо, что позволяет постепенную корректировку 405 интенсивности нм от 0-10 мкВт. не Выключить лазерного излучения до начала эксперимента.
- Поместите образец на предметный столик микроскопа и принести клетки в центре внимания в проходящем свете режиме микроскопии (рис. 4А). Усиление камера EMCCD должен быть выключен, чтобы предотвратить повреждение камеры, передержки.
- Определите обрезки FOV для уменьшения размера данных и увеличить камеры считывания скорости.
- Накройте образец из окружающего света и Switч от усиления камеры EMCCD.
- Установите частоту кадров для 15,26 мс / кадр (в том числе 0,26 мс камеры время считывания). См. "Выдержка и возбуждения интенсивности" в разделе обсуждения.
- Отображение данных камеры, чтобы проверить темный фоновый сигнал (рис. 4б).
- Включите 561 нм лазера на и проверить фона возбуждения сигнал (рис. 4в).
- Включите 405 нм лазера на для фотоактивации слитых белков Pol1-PAmCherry и увеличить интенсивность, пока не появятся флуоресценции НПФ.
- Отрегулируйте угол луча возбуждения для освещения только тонкий срез образца близко к поверхности покровного.
- С этой целью, лазерный луч фокусируется в задней фокальной плоскости 100X Н.А. 1.4 цели (рис. 3). Перевод фокусирующей линзы, перпендикулярной пучку перемещает фокус от центра объектива, вызывающего луч для выхода цели под углом.
- Искусственный интеллектм, чтобы максимизировать интенсивность флуоресценции и свести к минимуму фоновый сигнал. Обратите внимание, что строгое возбуждения МДП является оптимальным для изображения флуорофорами в пределах 100 морских миль от поверхности покровного, однако, визуализации ДНК-связывающие белки, связанные с Е. палочка нуклеоид требует более глубокого освещения до 0,8 мкм.
4. Сбор данных
Здесь мы опишем общую протокол для приобретения кино PALM. Такой же порядок применяется для работы с изображениями слитые белки Pol1-PAmCherry в неповрежденной Е. палочка клетки и при непрерывном лечении повреждений ДНК с MMS. Применение метода к слитых белков различной молекулярной массы или числа копий на клетку будет требовать различных установок на приобретение (см. раздел Обсуждение).
- Найти новое поле зрения (FOV) клеток в проходящем свете режиме микроскопии и сфокусировать изображение. Возьмите камеру снимок для записи сотовые очертания (рисунок 4А).
- Обложка образца от окружающего света и включите усиления камеры EMCCD.
- Включите 561 нм лазера на и отбелить сотовой аутофлюоресценция и фоновые пятна на покровного стекла на несколько секунд, прежде чем начать сбор данных. Для клеток выращивали и отображается в среде М9 и использованием сожгли покровные там, как правило очень мало фоновой флуоресценции, однако предварительном отбеливании может быть полезным для работы с изображениями клетки в богатой среде для роста, таких как LB. Следует отметить, что интенсивное освещение является токсичным к клеткам так предварительном отбеливании должно быть сведено к минимуму.
- Начните приобретение кино PALM под непрерывном возбуждении 561 нм на 15,26 мс / кадр.
- Включите 405 нм лазера на и постепенно увеличивать интенсивность в течение фильма, доходя до 1 Вт / см 2. Избегайте высоких 405 нм интенсивности, которые вызывают клеточную аутофлюоресценция. Обратите внимание на плотность флуоресцентных молекул - важно держать ставки активации низкой, так что НПФ явноизолированы в каждом кадре (фиг. 4D-F).
- Запишите 10000 кадров / фильм (в зависимости от числа молекул для включения в образ на ячейку); один фильм, как правило, занимает 2-3 мин и требует 0,5-1 Гб места на жестком диске в зависимости от размера поля зрения.
- Повторите процедуру приобретения для многократного поля зрения. Заметим, что каждый угол обзора могут быть отображены только один раз, потому что PAmCherry флуорофоры получить фотоактивируемые и беленой необратимо.
5. Анализ данных
Автоматизированная и надежная база для анализа данных имеет важное значение для производительности и эффективности метода. Мы используем специальное программное обеспечение, написанный на MATLAB.
- Проведение анализа локализации с использованием алгоритмов, описанные в Crocker и соавт. 12, Holden и соавт. 13, HoldenI соавт. 14 и Визер и соавт. 15 PSFs, сначала идентифицируют в полосовой отфильтрованного изображения с использованием Gaussian ядро с 7 пикселяс Диаметр (рис. 5А). Кандидаты позиции соответствуют PSFs с интенсивностью пика пиксельных выше в 4,5 раза стандартного отклонения фонового сигнала (рис. 5б). Локально ярких пикселей на кандидата PSF служит начального приближения для установки эллиптическую функции Гаусса (рис. 5C). Свободные параметры пригодные являются: координаты Х, Y-положение, х ширины, у ширины, угол поворота, амплитуда и смещение фон. Эллиптическая гауссова маска приходится молекулы за время экспозиции, которая размывает и деформирует PSF.
- Участок полученные (х, у) локализации от всех кадрах фильма PALM на проходящем свете микроскопии образу и подобию аналогичного поля зрения. Локализации из Pol1-PAmCherry должны появиться в центральной части Е. палочки клетки (рис. 6а). Если многие локализации появляться за пределами клетки, порог локализации был установлен слишком низким или образец содержал фоновые флуоресцентные частицы.
- Для автоматизированного анализа отслеживания, реализация MATLAB алгоритма, описанного в Крокер и др. 12 может быть использован (см. "Diffusion анализ" в разделе обсуждения). Аль. Позиции, которые появляются в последующих кадрах в пользовательское окно слежения подключены, чтобы сформировать траекторию. В случае, когда несколько локализации происходить в том же окне, дорожки однозначно назначается минимизации суммы длин шага. Для детального обсуждения различных соображений при расчете коэффициентов диффузии по данным слежения одночастичных см. Визер и др. 15.
- Алгоритм использует параметр памяти для учета переходных миганием или пропущенных локализаций во время пути. Здесь мы установить параметр памяти до 1 кадра; более высокие значения могут быть использованы для отслеживания флуорофоры долгоживущими темных государств.
- Выберите подходящее окно отслеживания на основе следующих шагов калибровки. Для Pol1, мы используем 0,57 мкм (5 пикселей).
- Запустите алгоритм отслеживания для диапазона отслеживания параметров окна. Рассчитать количество измеряемых треков на клетку в зависимости от отслеживания окна, чтобы определить наименьший возможный окно отслеживания, который не разделен дорожек (рис. 6б).
- Участок получившиеся треки на проходящем свете микроскопии образу и подобию аналогичного FOV визуализировать пространственное распределение движения молекул внутри клеток. Pol1 треков должен отображать диффузии ограничивается в пределах одной клетки (рис. 6С-D).
- Если доля композиций по-видимому, пересекают между клетками это говорит о том, что отдельные молекулы были ошибочно связаны, потому что окно было выбрано отслеживание слишком большой и / или скорость фотоактивация была слишком высокой (рис. 6E).
- Участок кумулятивное распределение длин шаг между соседними локализаций (рис. 6F). Кривая поднимается и насыщает гладко при достаточно больших окон слеженияно показывает среза края, если окно было выбрано слишком маленьким.
- Для анализа диффузии характеристики Pol1, вычислить среднеквадратичной смещение (MSD) между последовательными локализаций для каждого трека с в общей сложности N шагов):
MSD = 1 / (N-1) Σ = 1 N-1 (Х я +1 - х я) 2 + (у я +1 - у я) 2.
Включить только отслеживает, по крайней мере 4 шага (N ≥ 5 локализаций), чтобы уменьшить статистическую неопределенность в значениях MSD. - Построить кривую значений MSD в диапазоне от времени запаздывания путем расчета смещения на нескольких кадров (рис. 6 г). Форма кривой MSD может помочь классифицировать наблюдаемую молекулярное движение (рис. 6H).
- Рассчитайте кажущуюся коэффициент диффузии D * на дорожку от MSD:
D * = MSD / (4 Δt) - σ ос 2 / Δt.
Вторая тхм корректирует расчетной ошибки локализации (здесь, σ LOC = 40 нм и T = 15,26 мс см. Визер и др.. 15). - Постройте гистограмму измеренных D * значений всех треков в поле зрения (рис. 7а).
- Определить отдельные молекулы Pol1, которые появляются связан с хромосомой на основании измеренного значения D * на дорожку. Отделите популяции оценки (резкого распределения с центром в D * ~ 0 мкм 2 / с) и свободно диффундирующих молекул (широкое распределение с центром в D * ~ 0,9 мкм 2 / с), установив порог D * <0,15 мкм 2 / с ( красные бары в фиг.7А и 7D).
- Провести локализацию, отслеживания и диффузии анализ для Pol1 в неповрежденных клеток (7А-C) и в клетках, которые проходят лечение повреждения ДНК с MMS (Цифры 7D-E). Фракцию, связанных дорожек обеспечивает прямое количественную меру DNРемонт активность Pol1 в естественных условиях.
Результаты
Понятие фотоактивированного одной молекуле отслеживания для изучения взаимодействий белок-ДНК в естественных условиях показан на рисунке 1. Слитые белки PAmCherry обнаруживаются в живом Е. палочки клетки в последовательном порядке по фотоактивирующего одиночных молекул стохастически с 405 нм света на частоте менее одной молекулой в клетке в то время. Активированные молекулы изображаются при непрерывном возбуждении 561 нм. Молекулярная движение в клетке могут быть отслежены по соединяясь поблизости локализации в серии кадров до необратимого фотообесцвечивания. Поскольку диффузия ДНК-связывающих белков замедляется при связывании хромосомы, очевидно, коэффициент диффузии D * получены на дорожку непосредственно сообщает на отдельных взаимодействий белок-ДНК.
На рисунке 2 показаны фотоактивации слитых белков Pol1-PAmCherry в живом Е. палочки клетки. Влияние интенсивности 405 нм он плотность флуоресцентных молекул можно видеть на фигуре 4. Следует отметить, что плотность определяется не только интенсивностью 405 нм, но дополнительно на количество молекул, которые доступны для активации; пул остальных молекул будет исчерпан в течение фильма ладони.
Анализ локализации выполняется для каждого кадра фильма PALM, как показано на рисунке 5. Мы измерили точность локализации с помощью неподвижных молекул в основной клеток или связанных молекул в живых клетках. Наши настройки приобретение дал точность локализации σ ос = 40 нм, что согласуется с теоретическим предсказанием 3.
Полученные Pol1 локализации занимают центральную область клетки (рис. 6а), широко обобщал пространственную организацию Е. палочка нуклеоид 7. Большинство Pol1 отслеживает в неповрежденных клеток дисплей дифслияние как показано на рисунке 6С. Типичный ячейка содержит несколько сотен Pol1 дорожек (рис. 6г), в соответствии с числом копий около 400 молекул Pol1 на Е. . палочка ячейка 1 рисунках 6B и 6E-F служить руководством по выбору соответствующего параметра окна слежения - если отслеживание окно является слишком большим, разные молекулы с большей вероятностью стать ошибочно связаны с дорожки, а если отслеживание окно слишком мал, композиций с более длинными шагами будут разделены. Кривая MSD для Pol1 растет линейно для коротким временем запаздывания и насыщается более длительные периоды времени в связи с клеточной камере (рис. 6G). Различные типы молекулярного движения могут быть определены с помощью анализа MSD. Направленное движение дает параболическую кривую; броуновское движение характеризуется прямой линии; ограничивается кривой диффузии достигает плато; смещение кривой MSD для неподвижных частиц представляет собой Localization неопределенность (рис. 6H). Дополнительная информация о одночастичных отслеживания и устранению неисправностей можно найти в Арно и др. 16.
Мы ранее применил метод для измерения ДНК-ремонтные работы из Pol1 в ответ на экзогенный повреждения алкилирования ДНК 7. D * гистограмма Pol1 отслеживает в неповрежденных клетках показывает доминирующую популяцию диффузии молекул (7А-С). Небольшая часть 2,7% связанных молекул Pol1, скорее всего, участвовать в отстающей репликации и репарации эндогенного повреждения ДНК пряди. При непрерывном 100 мМ MMS повреждения, население треков с D * ~ 0 мкм 2 / с увеличивается до 13,8% (рис. 7D). Эти треки представляют отдельные молекулы Pol1 выполняющих синтез репарации ДНК, чтобы заполнить пробелы однонуклеотидных в рамках базового иссечение ремонта пути. Позиции связанных треков показать местоположение индивидуальныйПовреждение ДНК и ремонт сайты (Рисунок 7E-F).
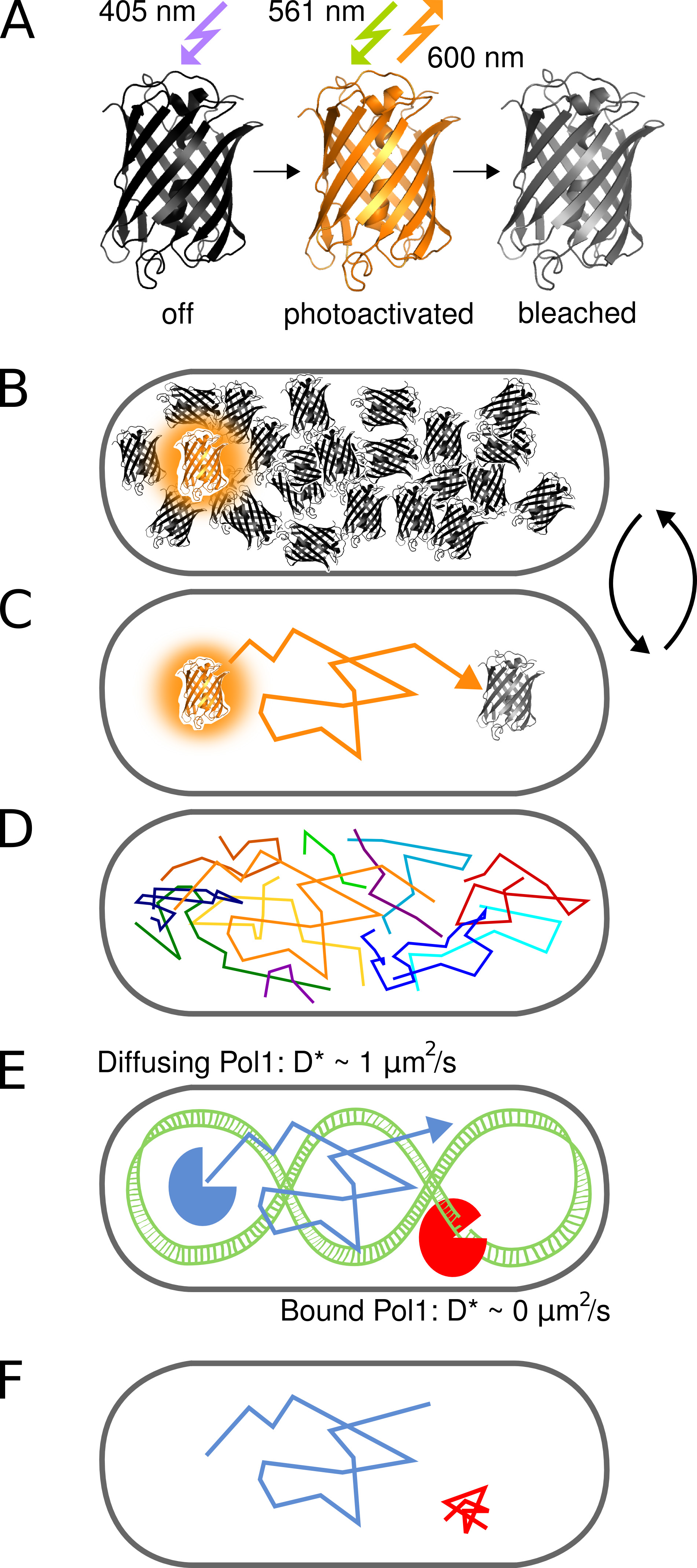
Рисунок 1. Графическое представление метода. (A) флуоресцентный белок PAmCherry может быть фотоактивированного от начального состояния нефлуоресцирующих при облучении светом 405 нм. Яркий состояние возбуждается на 561 нм и не излучает флуоресценцию вокруг 600 нм до флуорофорных отбеливатели необратимо. (B) управления скоростью фотоактивация позволяет визуализации только один стохастически активированный PAmCherry слитый белок на клетку в любой момент произвольно большой пул молекул, которые еще не были активированы или уже отбеленной остатки в темном состоянии. (C) Положение флуоресцентного молекулы определяется от центра выделенного PSF и трне acked для нескольких кадров до фотообесцвечивания. (D) Следы многих молекул записываются в последовательном порядке. (EF) Взаимодействие ДНК-связывающего белка с последовательностью-мишенью или структуры хромосомного останавливает случайное диффузионное движение. Связанных и несвязанных молекул отличаются кажущейся коэффициента диффузии D *, извлеченной из одного трека. В результате доля связанных молекул дает количественную меру для активности ДНК-связывающего белка в естественных условиях. Кликните здесь, чтобы посмотреть увеличенное изображение.
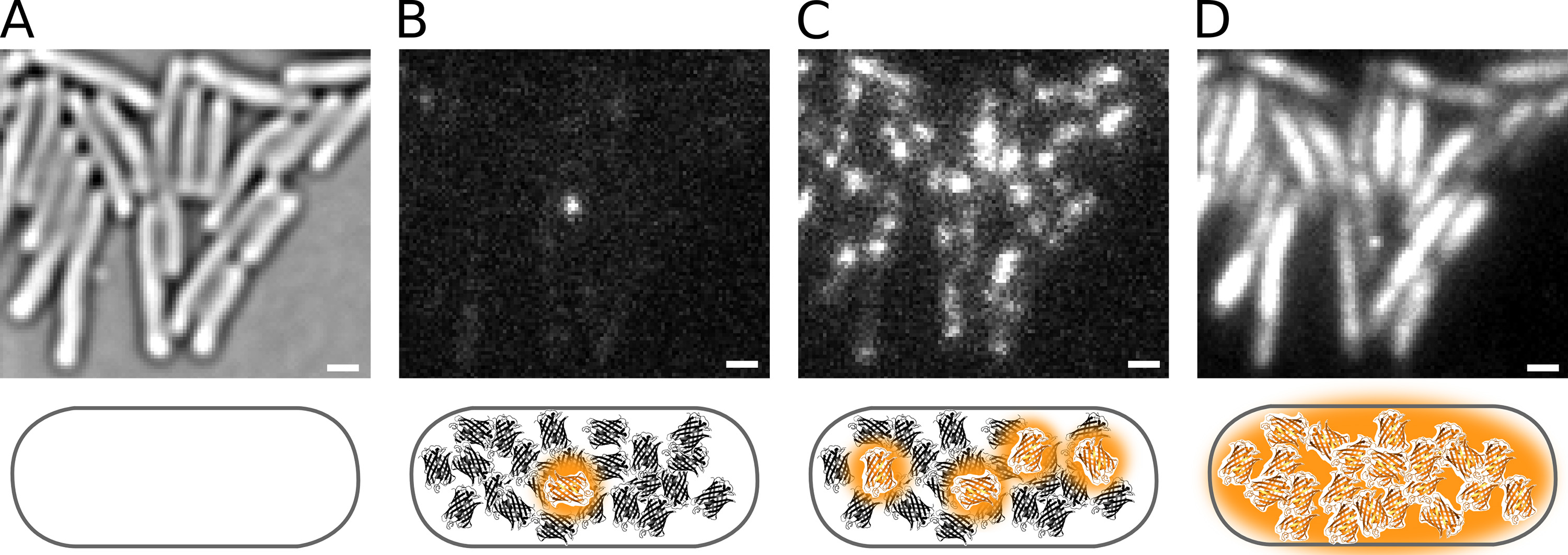
Рисунок 2. Фотоактивация Pol1-PAmCherry в живом Е. палочки клетки. Масштабные бары: 1 мкм. Схемы приведены под каждой панели. ( А) Передано световой микроскопии изображение клеток, иммобилизованных на агарозном площадку. (B) Phototactivating одну PAmCherry флуорофор в одной ячейке. (C) выше скорость фотоактивация увеличивает количество флуоресцентных молекул. (D) Встроенная PAmCherry флуоресценции из фильма PALM. Кликните здесь, чтобы посмотреть увеличенное изображение.
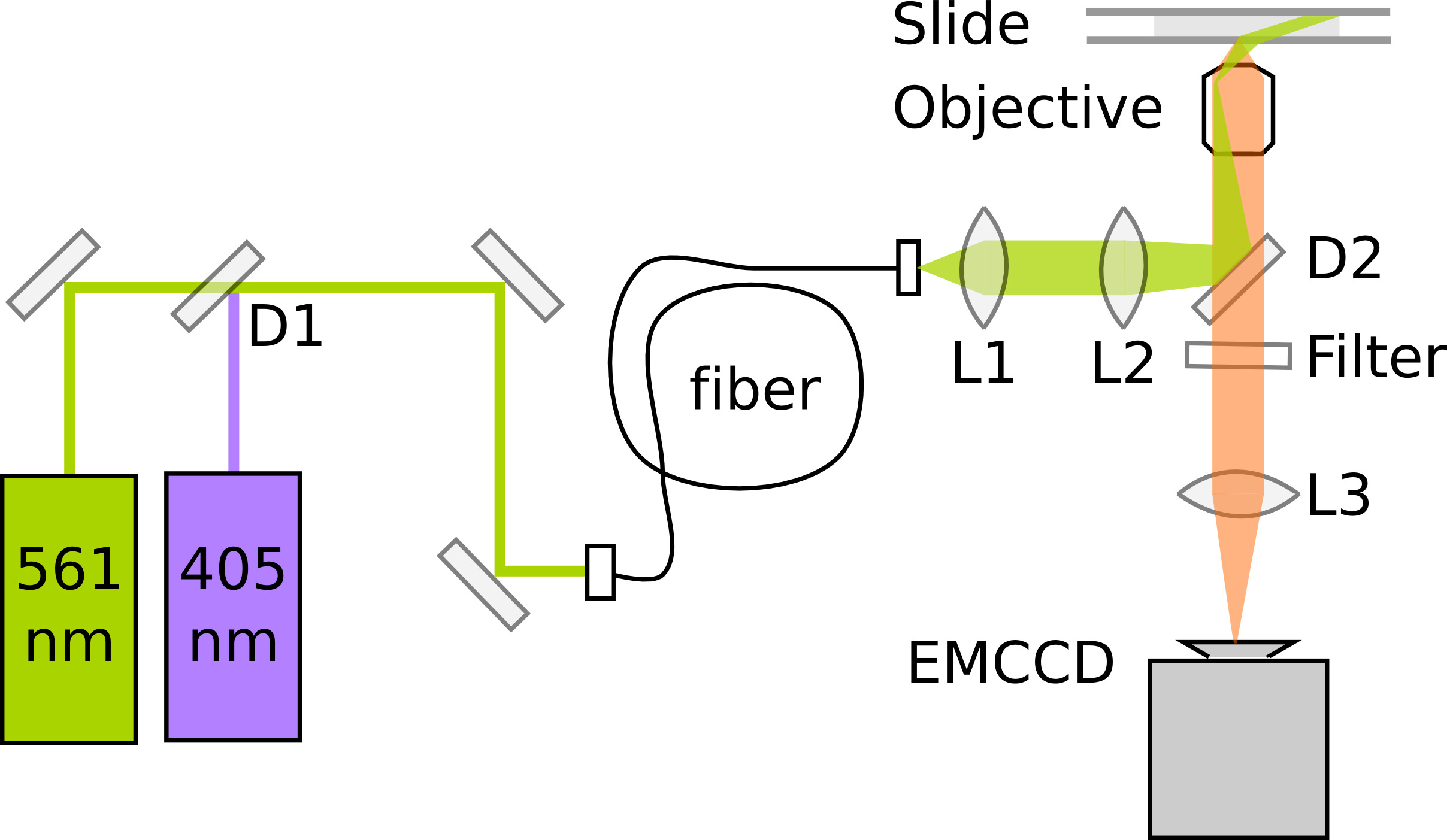
Рисунок 3. Схема минимальной настройки PALM для фотоактивирующего и обработки изображений PAmCherry слитых белков D1:. Дихроичный зеркало (например, 550 нм длинный-проход). D2: зеркальные (например 570 нм длинный-проход). L1: Коллиматорные линзы. L2: TIR объектив. L3: Труба объектив.1177/51177fig3highres.jpg "целевых =" _blank "> Нажмите здесь, чтобы посмотреть увеличенное изображение.

Рисунок 4. Представитель изображения из фильма PALM с 15.26 мс / кадр баров Масштаб:. 1 мкм. (A) проходящем свете изображение клеток, иммобилизованных на агарозном площадку. (В) Темный фон изображения измеряется на EMCCD камерой с лазерами выключен. (C) Возбуждение фоновое изображение при непрерывном возбуждении 561 нм до фотоактивации. (DF) Увеличение интенсивности 405 нм приводит к более высоким ставкам фотоактивации PAmCherry, отображаемого при непрерывном возбуждении 561 нм. В коробках области показаны в увеличенном ниже. (D) Низкая интенсивность 405 нм (<1 мкВт) активы очень мало флуоресцентных молекул на FOV. (Е) Средний 405Результаты нм интенсивность (~ 2 мкВт) фотоактивации в хорошем плотности PSF для локализации и отслеживания анализа. (F) Высшее интенсивности 405 нм (~ 10 мкВт) активирует более одного флуоресцентные молекулы в некоторых клетках, которая затемняет локализации и отслеживания анализ. Кликните здесь, чтобы посмотреть увеличенное изображение.

Рисунок 5. Иллюстрация анализа локализации. Масштабные бары: 1 мкм (А) фильтрации Полосовой удаляет паразитные шумы пикселей и объединяет градиенты интенсивности через поле зрения.. (В)-кандидаты НПФ выявляются в отфильтрованного изображения на основе определяемого пользователем порога, который выбран, чтобы минимизировать ложноположительные и ложноотрицательные обнаружений. ThreshoLD соответствует минимальной интенсивности пиксела-кандидата, разделенной на фоне стандартного отклонения (отношение сигнал-шум, SNR). (С) локально ярких пикселей, который проходит порог служит начального приближения локализации (оранжевый крест) для двумерной эллиптической Гаусса нужным. Масштабные бары: 0,5 мкм. В результате супер-разрешение локализации (синий крест) имеет среднюю точность σ ос = 40 нм. Кликните здесь, чтобы посмотреть увеличенное изображение.
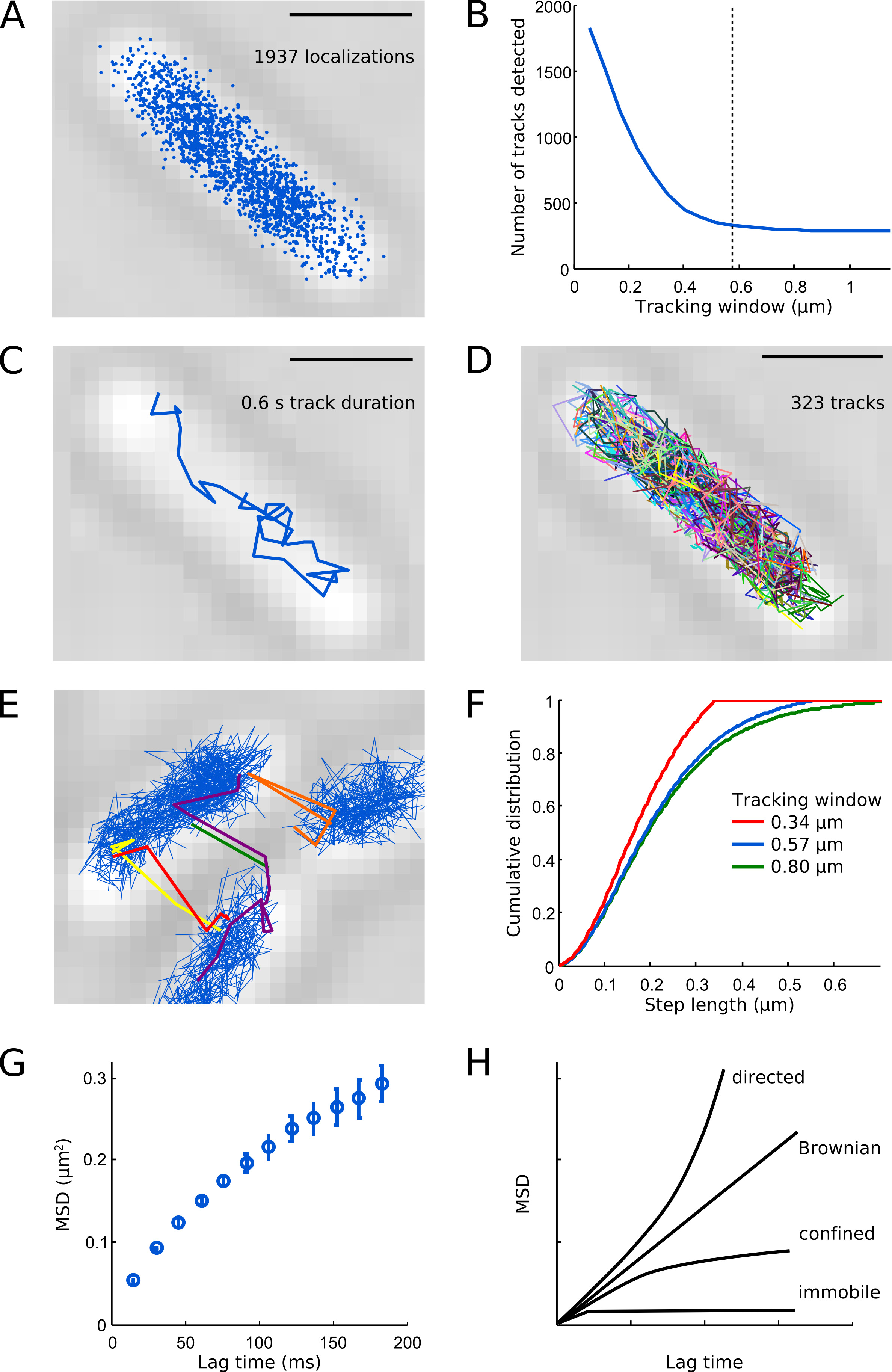
.. Рисунок 6 Иллюстрация анализа отслеживания Масштабные бары: 1 мкм. (А) Все обнаруженные локализации Pol1-PAmCherry в примере клетки. (В) Количество композиций обнаружитьред в примере клетки в зависимости от окна слежения. Маленькие окна слежения разделить молекулы траектории, что приводит к артефактом большое количество треков. Пунктирная линия указывает наш выбор для параметра окна слежения (0,57 мкм, 5 точек) - это дает хороший компромисс между обнаружения полное распределение шагов и держать траектории различных молекул нетронутыми. (С) Пример след одной молекулы Pol1-PAmCherry. (D) Все измеренные треков показаны в случайных цветах. (E) Наблюдают артефакты, если отслеживание окно выбрана слишком велико (здесь 0,8 мкм, 7 пикселей) или плотность PSFs в кадре слишком высока. (F) полезных распределения длин шага для отслеживания окон: 0,34 мкм (3 пикселей, красная линия), 0,57 мкм (5 пикселей, синяя линия) и 0,80 мкм (7 пикселей, зеленая линия). Обратите внимание, что сокращает отслеживая окно 0,34 мкм от действия дольше, чем 0,34 мкм, которые четкоусекает полное распределение шагов. Окно отслеживания 0,57 мкм обнаруживает почти такое же распределение шагов, как это делает отслеживание окно 0,80 мкм. (G) MSD кривая показывает ограниченную диффузию Pol1. (Н) Схема MSD кривые для направленного движения, броуновского движения, ограниченном диффузии и неподвижных частиц. Кликните здесь, чтобы посмотреть увеличенное изображение.
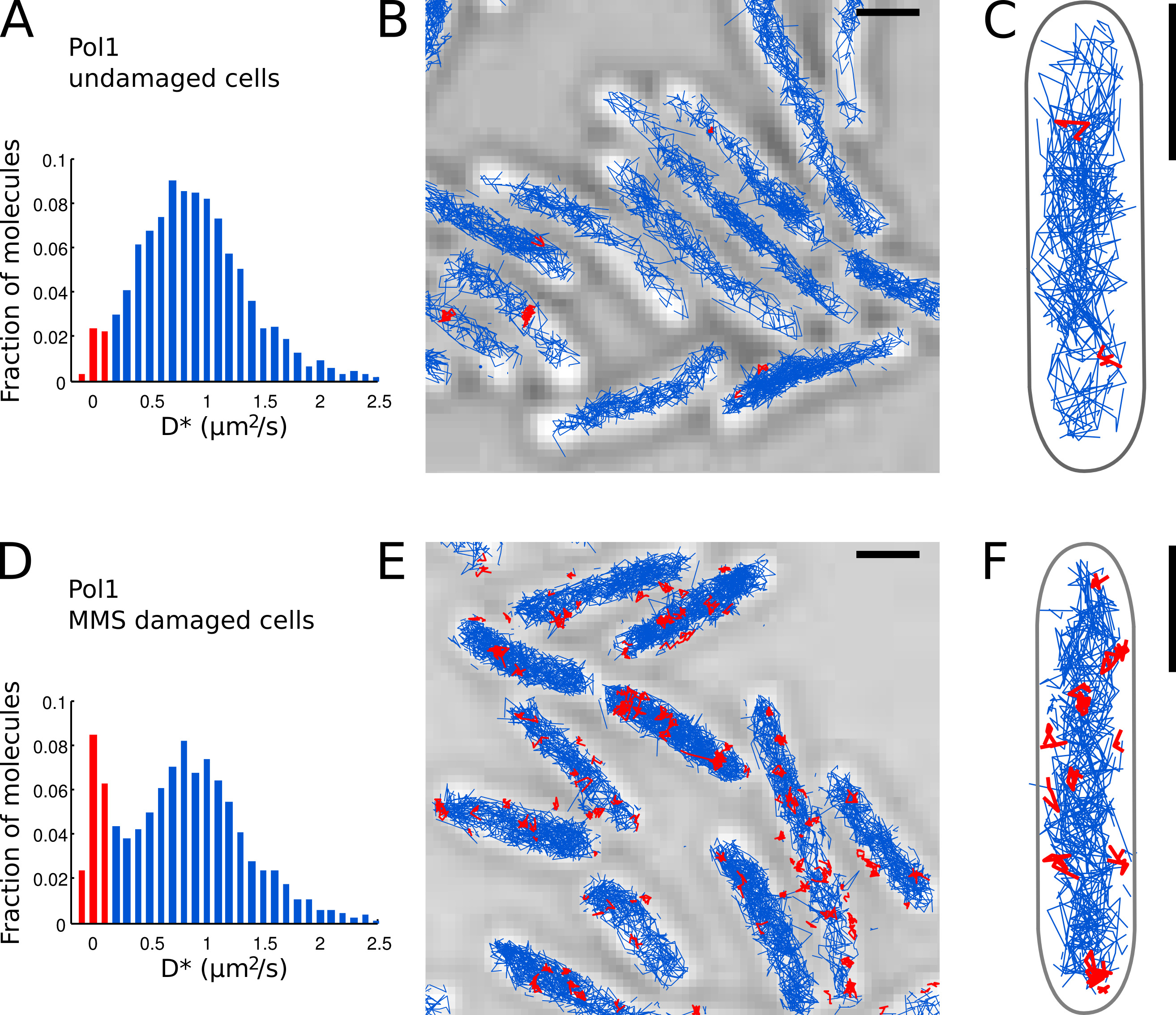
Рисунок 7. Прямое измерение репарации ДНК деятельности Pol1 в живом Е. . палочка клетки Масштабные бары: 1 мкм. (А) Гистограмма кажущейся коэффициентом диффузии D * для всех треках 4 или более шагов в поле зрения неповрежденных клеток (N = 4162 треков). Население молекул, классифицированных как б круглый подсвечивается красным цветом. (BC) Следы Pol1-PAmCherry показаны на проходящем свете микроскопии изображения. Дорожки, классифицированные как связаны в соответствии с их коэффициентом диффузии показаны красным цветом. (D) D * Гистограмма для Pol1 треков измеренных в клетках иммобилизованных на агарозном площадку с 100 мМ MMS и выдерживают в течение 20 мин до визуализации (N = 2128 треков). Население связанных молекул, участвующих в репарации ДНК отображается красным цветом. (EF) Pol1-PAmCherry отслеживает на проходящем свете микроскопии изображение, показывающее следы единичных событий Pol1 репарации ДНК в красный цвет. Кликните здесь, чтобы посмотреть увеличенное изображение.

Фильм 1. Строительство на заказ установку PALM.JoVE_Uphoff_Movie1.avi "целевых =" _blank "> Нажмите здесь для просмотра ролика.
Обсуждение
Мы обсуждаем несколько ключевых соображений для успеха эксперимента.
Выбор и выражение флуоресцентного белка слияния: Существует большая палитра фотоактивируемых и фотопереключаемых флуоресцентных белков 17. Конкретный выбор зависит от микроскопических характеристик, в частности лазеров и фильтров, доступных. Сочетание 405 нм и 561 нм идеально подходит для общих фотоактивируемых флуоресцирующих белков. Мы выбрали PAmCherry 6, потому что это мономерный и не показали агрегацию в клетках. Кроме того, необратимый фотоактивация позволяет подсчета количества активированных флуорофоров для измерения числа копий белка в клетке. Вместо того, чтобы выразить слитый белок из плазмиды, мы предпочитаем введение хромосомную ген, кодирующий слитого белка в дикого типа локуса. Это гарантирует полную замену интересующего белка с флуоресцентный веrsion и поддержания уровня экспрессии дикого типа.
Фотоактивация ставка: Важно регулировать скорость фотоактивации такой, что в среднем меньше, чем одна молекула на клетку находится в флуоресцентного состояния в любом кадре из фильма. Это зависит от интенсивности 405 нм и число молекул слева быть активирован. При очень низких плотностей изображений, однако, не все молекулы будут отображены до конца фильма или фильмов очень длинные должны быть приобретены. Количество кадров, записываемых в фильме зависит от числа копий слитых белков в клетке и среднего фотообесцвечивания жизни PAmCherry в условиях возбуждения используются. Количество копий Pol1 составляет ~ 400 молекул / ячейка 1 и среднее значение экспоненциального распределения жизни фотообесцвечивание составляла ~ 4 кадра. Постепенно увеличивая интенсивность волны 405 нм, активация равномерно распределяется по 10000 кадров фильма. Таким образом, каждая ячейка является OccupСВУ на флуоресцентных молекул в общей сложности ~ 1600 кадров, обеспечения мало перекрытие PSFs и сопровождение осложнений в кино 10000 кадров.
Выдержка и возбуждения интенсивности: Прежде всего, время экспозиции должны быть достаточно коротким, чтобы наблюдать резкие протяженности точки с небольшим движением размытие. Тем не менее, частота кадров должна быть выбрана, чтобы получить наблюдаемый молекулярное движение между последовательными кадрами за пределами неопределенности локализации, в противном случае решающими фотоны впустую передискретизации трек. Движение несвязанных молекул, должна проверяться при достаточно больших временах, чтобы четко отличаться от видимого движения связанных молекул в связи с неопределенностью локализации. Когда устанавливается время экспозиции, интенсивность PSF должна быть скорректирована. Точность локализации ФСФ возрастает с увеличением числа фотонов, обнаруженных в течение длительности кадра. Более высокие интенсивности возбуждения увеличить бу скорости излучения фотоновт и скорость фотообесцвечивание и фоновый сигнал. Используйте низкой интенсивности возбуждения, которая дает нужную точность локализации. Для Pol1-PAmCherry мы выбрали 15.26 мс / кадр и 3,5 мВт 561 нм возбуждение (400 Вт / см 2). Важно, чтобы подтвердить жизнеспособность клеток для конкретных условий обработки изображений путем мониторинга роста клеток и морфологию до и после сбора данных (см. Дополнительную информацию в Uphoff соавт. 7).
Pol1 проявляет связывания время ~ 2 сек до пропуском субстрата ДНК в естественных условиях 7, поэтому мы ожидаем, что в течение всего времени трека большинство молекул быть либо в связанном или свободном состоянии. Связанные молекулы появляются в основном неподвижны, потому что сайты хромосом имеют коэффициент диффузии на несколько порядков меньше (~ 10 -5 мкм 2 / с, Элмор и др.. 18), чем Pol1 диффузии в цитоплазме (~ 1 мкм 2 / сек).
Диффузия анализ: кажущийся коэффициент диффузии D * рассчитывается из MSD отдельных треков, усредненное по минимум 4 шагов (5 кадров), чтобы уменьшить статистическую погрешность. Обратите внимание, что ~ 75% молекул отбелить менее чем 5 кадров для условий обработки изображений, описанных. Такие короткие треки не обеспечивают достаточную статистическую достоверность различать связанных и несвязанных молекул. Однако относительные доли связанных и несвязанных молекул, которые сообщают по активности белка являются независимыми от общего числа дорожек проанализированы.
Это полезно для учета ошибки локализации PSF (σ LOC) при расчете D *, потому что неопределенность добавляет кажущуюся случайной шаг к каждой локализации молекулы 15.
Для улучшения классификацию связанных и диффундирующих молекул, мы рекомендуем расчета D * ботач от одноступенчатых перемещений и смещений за время двух кадров. В этом случае можно установить два отдельных D * пороги: D (15 мсек) <0,15 мкм 2 / с и D * (30 мсек) <0,075 мкм 2 / сек.
Обратите внимание, что D * является очевидным коэффициент диффузии, который зависит от клеток удержания дорожек и движения размывания за счет диффузии за время экспозиции. Чтобы извлечь точные беспристрастные коэффициентов диффузии, она доказала полезно сравнить наблюдаемую движение модельных данных на основе стохастического броуновского модели движения 5,7. Имитация данные также могут быть использованы для тестирования процедур анализа данных.
Потенциальные применения данного способа: мы описали общий подход к визуализации и количественного взаимодействия белок-ДНК в естественных условиях изменением подвижности белка при связывании с хромосоме. Деятельность ДНК-илиРНК-связывающие белки, участвующие в ремонте, репликации, транскрипции и, таким образом, обслуживание хромосома может следовать в реальном времени на уровне одной клетки с пространственным разрешением ниже предела оптической дифракции. Отслеживания одной молекулы фотоактивируемые расширяет традиционные методы слежения, которые ограничены до нескольких меченых молекул на клетку. Альтернативный метод, который измеряет молекулярную диффузию в естественных условиях является флуоресценции восстановления после фотообесцвечивания (FRAP). В то время как FRAP очень полезно для измерения глобальные характеристики диффузии в крупных клеток, он ограничен в своей способности разрешать несколько молекулярных частиц с различными подвижности в пространственно неоднородной среде, особенно для малых бактериальных клеток.
Мы применили фотоактивируемые одиночных молекул отслеживания для измерения деятельности ДНК-связывающих и субклеточных локализаций диапазоне различных белков в Е. палочка включая Pol1, ДНК-лигазы, Fis белка, ДНКполимеразной III 7, а также Структурная обслуживание хромосом белков MukB, E, и F 19. Мы предполагаем, способ также может быть приспособлен к другим типам клеток.
Раскрытие информации
Авторы заявляют, что они не имеют конкурирующие финансовые интересы.
Благодарности
Мы признаем, Джастин Пинкни и Йоханнес Hohlbein за помощь в строительстве заказ микроскопа и Симус Holden для программного обеспечения локализации. Родриго Рейес-Ламот объявлена благодарность за предоставление E. Штамм. Исследование финансировалось Европейской комиссии Седьмая Рамочная программа Грант FP7/2007-2013 ЗДОРОВЬЯ-F4-2008-201418, Великобритании биотехнологии и биологических наук исследовательский совет Грант BB/H01795X/1, и Европейский исследовательский совет Грант 261227 в АНК. DJS финансировалось за счет Целевого программы Wellcome Грант WT083469. SU была поддержана MathWorks докторской общении.
Материалы
| Name | Company | Catalog Number | Comments |
| E. coli strain carrying a chromosomal insertion for a PAmCherry DNA-binding fusion protein | created by Lambda-Red recombination | ||
| MEM amino acids | Invitrogen | 11130-051 | minimal medium supplement |
| MEM vitamins | Invitrogen | 11120-052 | minimal medium supplement |
| Agarose | BioRad | 161-3100 | certified molecular biology grade |
| Microscope coverslips No 1.5 thickness | Menzel | BB024060SC | remove background particles by heating slides in furnace at 500 °C for 1 hr |
| Methyl methanesulfonate (MMS) | Sigma-Aldrich | 129925 | CAUTION: toxic |
| PALM setup | home-built | described in Uphoff et al.7 | |
| MATLAB | MathWorks | for data analysis and visualization | |
| Localization software | custom-written, available online | http://www.physics.ox.ac.uk/Users/kapanidis/Group/Main.Software.html | MATLAB and C++ software package that can be adapted for localization analysis. Cite Holden et al.13 if used in publication. |
| Tracking software | available online | http://physics.georgetown.edu/matlab/ | MATLAB implementation by Blair and Dufresne. Cite Crocker & Grier12 if used in publication. |
Ссылки
- Friedberg, E. C. . DNA Repair and Mutagenesis. , (2006).
- Betzig, E., et al. Imaging intracellular fluorescent proteins at nanometer spatial resolution. Science. 313 (5793), 1642-1645 (2006).
- Mortensen, K. I., Churchman, L. S., Spudich, J. A., Flyvbjerg, H. Optimized localization analysis for single-molecule tracking and super-resolution microscopy. Nat. Methods. 7 (5), 377-381 (2010).
- Manley, S., et al. High-density mapping of single-molecule trajectories with photoactivated localization microscopy. Nat. Methods. 5 (2), 155-157 (2008).
- English, B. P., et al. Single-molecule investigations of the stringent response machinery in living bacterial cells. Proc. Natl. Acad. Sci. U.S.A. 108 (31), (2011).
- Subach, F. V., et al. Photoactivatable mCherry for high-resolution two-color fluorescence microscopy. Nat. Methods. 6 (2), 153-159 (2009).
- Uphoff, S., Reyes-Lamothe, R., Garza de Leon, F., Sherratt, D. J., Kapanidis, A. N. Single-molecule DNA repair in live bacteria. Proc. Natl. Acad. Sci. U.S.A. 110 (20), 8063-8068 (2013).
- Datsenko, K. A., Wanner, B. L. One-step inactivation of chromosomal genes in Escherichia coli K-12 using PCR products. Proc. Natl. Acad. Sci. U.S.A. 97 (12), 6640-6645 (2000).
- Reyes-Lamothe, R., Possoz, C., Danilova, O., Sherratt, D. J. Independent positioning and action of Escherichia coli replisomes in live cells. Cell. 133 (1), 90-102 (2008).
- Neidhardt, F. C., Bloch, P. L., Smith, D. F. Culture medium for enterobacteria. J. Bacteriol. 119 (3), 736-747 (1974).
- Tokunaga, M., Imamoto, N., Sakata-Sogawa, K. Highly inclined thin illumination enables clear single-molecule imaging in cells. Nat. Methods. 5 (2), 159-161 (2008).
- Crocker, J. C., Grier, D. G. Methods of digital video microscopy for colloidal studies. J. Colloid. Interface. Sci. 179 (1), 298-310 (1996).
- Holden, S. J., et al. Defining the limits of single molecule FRET resolution in TIRF microscopy. Biophys. J. 99 (9), 3102-3111 (2010).
- Holden, S. J., Uphoff, S., Kapanidis, A. N. DAOSTORM: an algorithm for high-density super-resolution microscopy. Nat. Methods. 8 (4), 279-280 (2011).
- Wieser, S., Schütz, G. J. Tracking single molecules in the live cell plasma membrane-Do's and Don't's. Methods. 46 (2), 131-140 (2008).
- Arnauld, S., Nicolas, B., Hervé, R., Didier, M. Multiple-target tracing (MTT) algorithm probes molecular dynamics at cell surface. Protoc. Exch. , (2008).
- Lippincott-Schwartz, J., Patterson, G. H. Photoactivatable fluorescent proteins for diffraction-limited and super-resolution imaging. Trends Cell Biol. (11), 555-565 (2009).
- Elmore, S., Müller, M., Vischer, N., Odijk, T., Woldringh, C. L. Single-particle tracking of oriC-GFP fluorescent spots during chromosome segregation in Escherichia coli. J. Struct. Biol. 151 (3), 275-287 (2005).
- Badrinarayanan, A., Reyes-Lamothe, R., Uphoff, S., Leake, M. C., Sherratt, D. J. In vivo architecture and action of bacterial Structural Maintenance of Chromosome proteins. Science. 338 (6106), 528-531 (2012).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеСмотреть дополнительные статьи
This article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены