Method Article
Visualiser les interactions protéine-ADN dans des cellules bactériennes vivantes en utilisant photoactivés suivi unique molécule
Dans cet article
Résumé
Microscopie localisation photoactivé (PALM) combiné avec suivi molécule unique permet l'observation directe et la quantification d'interactions protéine-ADN dans des cellules vivantes d'Escherichia coli.
Résumé
Interactions protéine-ADN sont au cœur de nombreux processus cellulaires fondamentaux. Par exemple, la réplication de l'ADN, la transcription, la réparation et l'organisation des chromosomes sont régies par des protéines de liaison à l'ADN qui reconnaissent des structures d'ADN spécifiques ou des séquences. Des expériences in vitro ont permis de générer des modèles détaillés pour la fonction de nombreux types de protéines de liaison à l'ADN, encore , les mécanismes exacts de ces processus et leur organisation dans l'environnement complexe de la cellule vivante demeurent beaucoup moins connues. Nous avons récemment introduit une méthode pour quantifier les activités réparation de l'ADN dans des cellules d'Escherichia coli en direct grâce photoactivés localisation de microscopie (PALM) combinée avec un suivi en une seule molécule. Notre approche générale identifie les événements de liaison à l'ADN individuels par la variation de la mobilité d'une protéine unique sur liaison avec le chromosome. La fraction de molécules liées fournit une mesure quantitative directe de l'acte de protéineivité et l'abondance des substrats ou des sites de liaison au niveau d'une seule cellule. Ici, nous décrivons le concept de la méthode et de démontrer la préparation de l'échantillon, l'acquisition de données, et les procédures d'analyse des données.
Introduction
Ce protocole décrit la mesure directe des interactions protéine-ADN dans les cellules d'Escherichia coli vivante. La technique utilise la variation du coefficient de diffusion d'une protéine marquée par fluorescence unique car elle se lie au chromosome (figure 1). Pour démontrer la méthode que nous utilisons l'ADN polymérase I (Pol1), une protéine de liaison à l'ADN prototypique qui comble les lacunes de l'ADN dans la réplication de brin tardif et voies réparation par excision de 1.
L'avènement de la microscopie de fluorescence super-résolution permet de visualiser les structures moléculaires dans les cellules avec une résolution nanométrique. Photoactivée localisation de microscopie (PALM) emploie des protéines fluorescentes qui peuvent être activées à partir d'un état sombre initial à un état fluorescent (Figure 2). Seul un sous-ensemble de toutes les molécules marquées est activée à tout moment pour déterminer leurs positions de manière séquentielle, indépendamment de til la concentration totale de molécules marquées dans l'échantillon 2. La précision de la localisation, par molécule, dépend principalement de la taille de la fluorescence fonction de flou (PSF), le nombre de photons collectés, et le signal de fond 3. De nombreuses applications de cette méthode se concentrent sur l'amélioration de la visualisation des structures cellulaires. La réalisation PALM peut être combiné avec une seule molécule de suivi 4 ouvert de nouvelles voies à suivre directement le mouvement des numéros arbitraires de protéines marquées dans les cellules vivantes. Augmentation de la sensibilité et une résolution temporelle de microscopes à fluorescence permettent désormais suivi de certaines protéines fluorescentes qui diffusent dans le cytoplasme bactérien 5.
Ici, nous employons PAmCherry, une protéine fluorescente d'ingénierie qui convertit de façon irréversible à partir d'un état initial non fluorescent à l'état fluorescent lors de l'irradiation avec 405 nm de lumière 6. Fluorophores PAmCherry activés peuvent être l'imaged par excitation à 561 nm et suivis pendant plusieurs cadres jusqu'à photoblanchiment. Nous démontrons la capacité de la méthode pour identifier les événements de liaison à l'ADN transitoire de protéines simples en utilisant une fusion de Pol1 et PAmCherry. Le traitement des cellules avec le méthanesulfonate de méthyle (MMS) provoque des lésions de la méthylation de l'ADN qui est tourné dans des substrats d'ADN par des enzymes avec gaps base excision repair. Notre méthode montre clairement la liaison de molécules pol1 simples en réponse aux dommages MMS 7.
Protocole
Une. Culture cellulaire
Utilisez des tubes de culture stériles et embouts de pipette. Le E. coli souche AB1157 polA-PAmCherry porte un PAmCherry fusion C-terminale de Pol1. La fusion a été inséré à l'emplacement chromosomique natif en remplaçant le gène de type sauvage en utilisant la lambda-Rouge recombinaison, comme décrit dans Datsenko et al. 8 de fonctionnalité de la protéine de fusion a été confirmée comme jugé par les taux de croissance cellulaire et de la sensibilité à l'agent endommageant l'ADN méthyle méthanesulfonate (MMS). Plus d'informations sur la construction de la souche cellulaire peut être trouvée dans Uphoff et al. 7, Datsenko et al. 8, et Reyes-Lamothe et al. 9 cultures de cellules sont cultivées en milieu minimum M9 pour réduire l'autofluorescence et éviter que des particules de fond sur la lame de microscope. Alternativement, un milieu défini riche en nutriments peut être utilisé 10.
- Série E. coli souche AB1157 polA-PAmCherry d'un stock de glycérol congelés sur une Luria Broth (LB) plaque d'agarose avec des antibiotiques sélectifs (ici, 25 pg / ml de kanamycine) et incuber à 37 ° C pendant la nuit.
- Inoculer une culture de 5 ml de LB à partir d'une colonie de cellules isolées et de croître à 37 ° C sous agitation à 220 tours par minute pendant 3 heures.
- Diluer la culture de 1:10000 dans 5 ml de milieu minimal (milieu M9, MEM acides aminés + proline, de vitamines MEM, 0,2% de glycerol) et incuber à 37 ° C sous agitation à 220 tours par minute pendant une nuit.
- Le lendemain matin, mesurer la densité optique (DO) à l'aide d'un spectrophotomètre et diluer la culture dans 5 ml de milieu minimum frais de OD 0,025. Croître pendant 2 heures à 37 ° C sous agitation à 220 rpm à la phase exponentielle précoce (OD 0,1).
- Concentré 1 ml de cellules dans un tube de 1,5 ml par centrifugation à 2300 xg pendant 5 min. Eliminer le surnageant et remettre en suspension le culot cellulaire dans 20 ul de milieu résiduel et vortex.
2. Lame Preparation
- Préparer une solution d'agarose à faible fluorescence de 1,5% en dH 2 O. Utilisez un micro-ondes pour faire fondre l'agarose jusqu'à la solution est claire. Mélanger 500 pi de la solution d'agarose fondu avec 500 pi de milieu minimum 2x par pipetage de haut en bas plusieurs fois.
- Répartir la solution d'agarose uniformément sur le centre d'une lamelle couvre-objet de microscope (n ° d'épaisseur 1,5). Cela doit être fait rapidement avant l'agarose se refroidit, en évitant les bulles.
- Aplatir le tampon avec une seconde lamelle (n ° d'épaisseur 1,5). Pour enlever les particules fluorescentes de fond, des lamelles ont été préalablement brûlé dans un four à 500 ° C pendant 1 heure. Lamelles brûlées peuvent être stockées pendant des semaines à la température ambiante recouvert de papier d'aluminium.
- Pour les expériences de lésions de l'ADN, préparer un tampon d'agarose contenant mM MMS 100. Suivre la procédure décrite dans les étapes 2.1 à 2.3, mais en ajoutant 8,3 ul de MMS à 500 ul de milieu M9 avant le mélange avec 500 ul de 1,5% d'agarose fondu, pour une concentration finale de 100 mM MMS. (Attention MMS est toxique et mutagène et doit être manipulé avec des gants, un masque, des lunettes et une blouse de laboratoire).
- Retirer la lame supérieure du tampon et ajouter 1 l de suspension cellulaire concentrée sur le tampon. Immobiliser les cellules en recouvrant le tampon avec une lamelle utilisé brûlé (Pas d'épaisseur 1,5, correspondant à la spécification objectif de microscope) et en appuyant très légèrement sur la lame. Les cellules doivent être visualisés dans 45 min d'immobilisation avant le séchage de plaquettes agarose. Pour éviter le dessèchement lors des expériences plus longues, coussins d'agarose peuvent être scellés avec joints en silicone.
- Pour les expériences lésions de l'ADN, incuber des cellules immobilisées sur la plate-agarose contenant mM MMS 100 pendant 20 min dans un récipient humide à température ambiante avant de l'imagerie.
3. Préparation d'acquisition de données de microscopie
PALM repose sur la détection et la localisation précise des protéines fluorescentes simples. La sensibilité et d'un alignement optimal desle microscope est critique pour la qualité des données. Une seule molécule microscopes à fluorescence utilisent typiquement réflexion totale interne (TIR) d'éclairage pour améliorer le rapport signal-sur-bruit en excitant uniquement fluorophores dans une section mince au-dessus de la surface de la lamelle. Ici, l'imagerie à l'intérieur E. coli nécessite très illumination inclinée 11, qui peut être réalisée sur un microscope TIRF en diminuant légèrement l'angle de la lumière d'excitation. Imagerie PAmCherry nécessite en outre un laser de photoactivation 405 nm et un laser d'excitation de 561 nm. L'émission de fluorescence est enregistrée sur une multiplication d'électrons CCD (EMCCD) appareil photo à un grossissement résultant en une longueur de pixel de 114,5 nm / pixel. Pour optimiser la précision de localisation, la taille des pixels doit correspondre à peu près à la largeur de l'écart type de la PSF à assurer un échantillonnage suffisant sans propagation du signal sur trop de pixels. Figure 3 montre un schéma d'une installation de PALM minime. Film 1donne une impression du processus de construction de microscope sur mesure; voir Uphoff et al 7 pour une description détaillée de l'instrument..
- Exécution d'alignement de microscope routine. Mesurer la 405 nm et 561 nm onde continue intensités laser en face de l'objectif. Régler l'intensité de 561 nm à 3,5 mW (~ 400 W / cm 2) et 405 nm à une intensité de 10 mW (~ 1 W / cm 2). Utilisez une roue à variation continue de densité neutre filtre qui permet un réglage progressif de 405 nm intensité de 0-10 mW. Eteindre l'éclairage laser jusqu'à ce que le début de l'expérience.
- Placer l'échantillon sur la platine du microscope et d'amener les cellules au point en mode de microscopie lumière transmise (figure 4A). Le gain de la caméra EMCCD doit être désactivé pour éviter d'endommager l'appareil photo par une surexposition.
- Définir un FOV recadrée pour réduire la taille des données et augmenter la vitesse de lecture caméra.
- Couvrir l'échantillon de la lumière ambiante et Switch sur le gain de la caméra EMCCD.
- Réglez la vitesse de défilement de 15,26 ms / frame (y compris 0,26 caméra ms temps de lecture). Voir «temps d'exposition et d'excitation intensités" dans la section Discussion.
- Afficher les données de la caméra pour vérifier le signal de fond sombre (figure 4B).
- Allumez le laser 561 nm et vérifier le signal d'excitation de fond (figure 4C).
- Allumez le laser à 405 nm pour la photoactivation des protéines de fusion Pol1-PAmCherry et augmenter l'intensité jusqu'à ce que le SP fluorescence apparaissent.
- Ajuster l'angle du faisceau d'excitation pour illuminer seulement une section mince de l'échantillon à proximité de la surface de la lamelle.
- A cet effet, le faisceau laser est focalisé dans le plan focal arrière d'un objectif 100X 1,4 NA (figure 3). La traduction de la lentille de focalisation perpendiculaire au faisceau se déplace l'accent mis sur le centre de l'objectif amener le faisceau de sortir l'objectif sous un angle.
- Aim afin de maximiser l'intensité de fluorescence et de minimiser le signal de fond. Notez que TIR strict excitation est optimale pour l'image fluorophores à moins de 100 nm de la surface de la lamelle couvre-objet, cependant, les protéines de liaison d'ADN associées à l'imagerie E. coli nucléoïde nécessite un éclairage plus profond jusqu'à 0,8 um.
4. Acquisition de données
Ici, nous décrivons le protocole général pour l'acquisition d'un film PALM. La même procédure s'applique pour l'imagerie des protéines de fusion Pol1-PAmCherry en bon état E. coli cellules et sous traitement continu de lésions de l'ADN avec le MMS. Application de la méthode de protéines de fusion de poids ou nombre de copies par cellule moléculaire différente, il faudra différents paramètres d'acquisition (voir la section Discussion).
- Trouver un nouveau champ de vision (FOV) de cellules en mode microscopie lumière transmise et focaliser l'image. Prendre un instantané de l'appareil photo pour enregistrer l'contours cellulaires (figure 4A).
- Couvrir l'échantillon de la lumière ambiante et basculer sur le gain de la caméra EMCCD.
- Allumez le laser 561 nm et blanchir l'autofluorescence cellulaire et taches de fond sur la lamelle pendant quelques secondes avant de commencer l'acquisition des données. Pour les cellules cultivées et imagée dans un milieu M9 et en utilisant des lamelles brûlé il ya généralement très peu de fond de fluorescence, mais préblanchiment pourrait être utile pour l'imagerie des cellules dans un milieu de croissance riche comme LB. Notez que l'éclairage intense est toxique pour les cellules afin de préblanchiment devraient être réduits au minimum.
- Lancement de l'acquisition d'un film sous PALM continue excitation 561 nm à 15,26 ms / cadre.
- Mettre en marche le laser à 405 nm et à augmenter graduellement l'intensité au cours du film, atteignant jusqu'à 1 W / cm 2. Évitez supérieur 405 nm intensités qui causent autofluorescence cellulaire. Faites attention à la densité de molécules fluorescentes - il est important de maintenir les taux d'activation faible tels que les SP sont clairementisolé dans chaque trame (figures 4D-F).
- Enregistrer 10 000 images / vidéo (en fonction du nombre de molécules à imagées par cellule), un film prend généralement 2-3 min et nécessite 0,5-1 Go d'espace disque en fonction de la taille du champ de vision.
- Répétez la procédure d'acquisition de multiples FOV. Notez que chaque champ de vision ne peut être reproduite fois parce fluorophores PAmCherry obtenir photoactivée et blanchi de manière irréversible.
5. Analyse des données
Un cadre d'analyse de données automatisé et robuste est essentiel pour la performance et l'efficacité de la méthode. Nous utilisons un logiciel personnalisé écrit en MATLAB.
- Effectuer l'analyse de la localisation en utilisant les algorithmes décrits dans Crocker et al. 12, Holden et al. 13, HoldenI et al. 14, et Wieser et al. 15 PSF sont d'abord identifiées en une image filtrée passe-bande à l'aide d'un noyau gaussien de 7 pixelss diamètre (figure 5A). les positions correspondent à des candidats PSF avec des intensités de pixel de crête au-dessus de 4,5 fois l'écart type du signal d'arrière-plan (figure 5B). La place la plus brillante pixel par PSF candidat sert estimation initiale de mise en place d'une fonction gaussienne elliptique (figure 5C). Les paramètres d'ajustement sont libres: position X, position Y, X largeur, y-largeur, angle de rotation, l'amplitude et de décalage de fond. Représente le masque gaussien elliptique pour molécule pendant le temps de l'exposition, qui brouille et déforme la PSF.
- Tracer les (x, y) des localisations résultant de toutes les images du film PALM sur l'image de microscopie lumière transmise de la même FOV. Localisations de Pol1-PAmCherry devraient apparaître dans la zone centrale de E. cellules de E. coli (figure 6A). Si de nombreuses localisations apparaissent à l'extérieur des cellules, le seuil de la localisation a été trop faible ou si l'échantillon contient des particules fluorescentes de fond.
- Pour une analyse de suivi automatisé, la mise en œuvre MATLAB de l'algorithme décrit dans Crocker et al. 12 peut être utilisé (voir «analyse de Diffusion" dans la section Discussion). Les positions qui apparaissent dans les images suivantes à l'intérieur d'une fenêtre de poursuite définie par l'utilisateur sont connectés pour former une trajectoire. Dans le cas où se produisent des localisations multiples dans la même fenêtre, les pistes sont attribués de manière unique en réduisant au minimum la somme des longueurs de pas. Pour une discussion détaillée des diverses considérations lors du calcul des coefficients de diffusion de données de suivi à particule unique, voir Wieser et al. 15
- L'algorithme utilise un paramètre de mémoire pour tenir compte de clignotement transitoire ou localisations manqués au cours d'une piste. Ici, nous définissons le paramètre de la mémoire à 1 image; des valeurs plus élevées peuvent être utilisées pour le suivi des fluorophores avec les Etats sombres à long terme.
- Choisissez une fenêtre de suivi approprié basé sur les étapes d'étalonnage suivantes. Pour Pol1, nous utilisons 0,57 um (5 pixels).
- Exécutez l'algorithme de suivi pour une gamme de paramètres de suivi de la fenêtre. Calculer le nombre de pistes de mesure par la cellule en fonction de la fenêtre de poursuite pour identifier la fenêtre de poursuite la plus petite possible qui n'est pas divisé titres (Figure 6B).
- Tracer les pistes résultantes sur l'image de microscopie à lumière transmise de la même FOV de visualiser la répartition spatiale de la molécule de mouvement dans les cellules. pistes pol1 devraient afficher diffusion confinée dans des cellules individuelles (figures 6C-D).
- Si une fraction de pistes apparaît à franchir entre les cellules ce qui suggère que les molécules séparées sont reliées par erreur, car la fenêtre de suivi de piste a été choisi trop grand et / ou le taux de photoactivation est trop grande (figure 6E).
- Tracer la distribution cumulative des longueurs de pas entre localisations successives (figure 6F). La courbe monte et sature en douceur pour suffisamment grandes fenêtres de suivimais montre un bord de coupure si la fenêtre a été choisie trop petite.
- Pour analyser les caractéristiques de diffusion de Pol1, calculer le déplacement quadratique moyenne (MSD) entre localisations consécutives pour chaque piste avec un total de N étapes):
TMS = 1 / (N-1) Σ i = 1 N-1 (X i +1 - x i) 2 + (y i +1 - y i) 2.
Inclure uniquement les pistes avec au moins 4 étapes (n ≥ 5 localisations) pour réduire l'incertitude statistique des valeurs de TMS. - Tracer la courbe des valeurs de TMS sur une plage de temps de latence en calculant les déplacements sur plusieurs cadres (figure 6G). La forme de la courbe de TMS peut aider à classer le mouvement moléculaire observé (figure 6H).
- Calculer le coefficient apparent de diffusion D * par piste de la TMS:
D * = MSD / (4 At) - σ loc 2 / At.
Le second teuh corrige l'erreur de localisation estimée (ici, σ loc = 40 nm et At = 15.26 ms, voir Wieser et al. 15). - Tracer un histogramme des valeurs de D * mesurées de toutes les pistes dans le champ de vision (figure 7A).
- Identifier des molécules d'pol1 individuels qui apparaissent lié au chromosome sur la base du D * valeur mesurée par titre. Séparer les populations de limite (distribution étroite centrée à D * ~ 0 pm 2 / s) et des molécules diffusant librement (large distribution centrée à D * ~ 0,9 um 2 / sec) en fixant un seuil D * <0,15 um 2 / s ( barres rouges sur les figures 7A et 7D).
- Effectuer la localisation, le suivi et l'analyse de diffusion pour Pol1 dans des cellules intactes (figures 7A-C) et dans les cellules en cours de traitement par l'ADN endommagé par MMS (figures 7D-E). La fraction de titres consolidés fournit une mesure quantitative directe de la DNUne activité de réparation de Pol1 in vivo.
Résultats
Le concept de photoactivé de suivi pour étudier les interactions protéine-ADN in vivo molécule unique est illustré sur la figure 1. Des protéines de fusion PAmCherry sont détectés en temps réel dans E. cellules de E. coli d'une manière séquentielle par photoactivation molécules uniques de manière stochastique à 405 nm de la lumière à une fréquence de moins d'une molécule par cellule à la fois. Molécules activées sont visualisés sous continu 561 nm excitation. Mouvement moléculaire dans la cellule peut être suivi en connectant localisations proximité dans une série d'images jusqu'à photoblanchiment irréversible. Étant donné que la diffusion des protéines de liaison à l'ADN est ralentie lors de la liaison du chromosome, le coefficient de diffusion apparent D * obtenue par voie relève directement sur les interactions protéine-ADN individuels.
La figure 2 démontre la photoactivation de protéines de fusion-Pol1 PAmCherry en direct E. cellules de E. coli. L'influence de l'intensité à 405 nm on la densité des molécules fluorescentes peut être vu sur la figure 4. A noter que la densité n'est pas uniquement déterminée par l'intensité à 405 nm, mais en outre par le nombre de molécules qui sont disponibles pour l'activation, la piscine de molécules restantes est épuisé au cours d'un film PALM.
l'analyse de la localisation est effectuée pour chaque image d'un film PALM comme illustré sur la Figure 5. Nous avons mesuré la précision de localisation en utilisant des molécules immobiles dans les cellules fixes ou molécules liées à des cellules vivantes. Nos paramètres d'acquisition ont une précision de localisation de σ loc = 40 nm, en accord avec la prédiction théorique 3.
Les localisations POL1 résultant occupent la zone centrale de la cellule (figure 6A), récapitulant largement l'organisation spatiale de l'E. coli nucléoïde 7. La majorité des Pol1 pistes dans des cellules intactes afficher diffusion tel que représenté sur la figure 6C. Une cellule typique contient plusieurs centaines de titres pol1 (Figure 6D), en conformité avec le nombre de copies d'environ 400 molécules pol1 par E. . cellule coli 1 Figures 6B et 6E-F fournir des conseils sur le choix d'un paramètre approprié de la fenêtre de suivi - si la fenêtre de poursuite est trop grande, différentes molécules sont plus susceptibles de devenir à tort lié à une piste, si la fenêtre de poursuite est trop petit, les pistes avec de plus longues étapes seront répartis. La courbe des TMS pour l'Pol1 augmente de façon linéaire pour les temps de latence court et sature à de plus longs temps de latence dus à l'isolement cellulaire (figure 6G). Différents types de mouvement moléculaire peuvent être identifiés par l'analyse des TMS. Réalisé mouvement donne une courbe parabolique, le mouvement brownien est caractérisé par une ligne droite, la courbe de diffusion confinée atteint un plateau, un décalage de la courbe de TMS pour les particules immobiles représente la localization incertitude (figure 6H). Des renseignements supplémentaires sur une particule de suivi et des conseils de dépannage peuvent être trouvées dans Arnauld et al. 16
Nous avons déjà appliqué la méthode pour mesurer l'activité de réparation de l'ADN Pol1 en réponse à dommages à l'ADN exogène d'alkylation 7. Le D * histogramme de Pol1 pistes dans des cellules intactes montre une population dominante de diffusion des molécules (figures 7A-C). Une petite fraction de 2,7% liés molécules pol1 est probablement impliqué dans la réplication de brin tardif et la réparation des lésions de l'ADN endogène. Sous continu dommages 100 mM MMS, la population de pistes avec D * ~ 0 pm 2 / sec augmente à 13,8% (figure 7D). Ces titres représentent des molécules pol1 individuels effectuant la synthèse de réparation d'ADN pour combler les lacunes d'un seul nucléotide dans le cadre de la voie de base excision repair. Les positions de titres consolidés montrent les emplacements des particuliersdommages à l'ADN et les sites de réparation (Figure 7E-F).
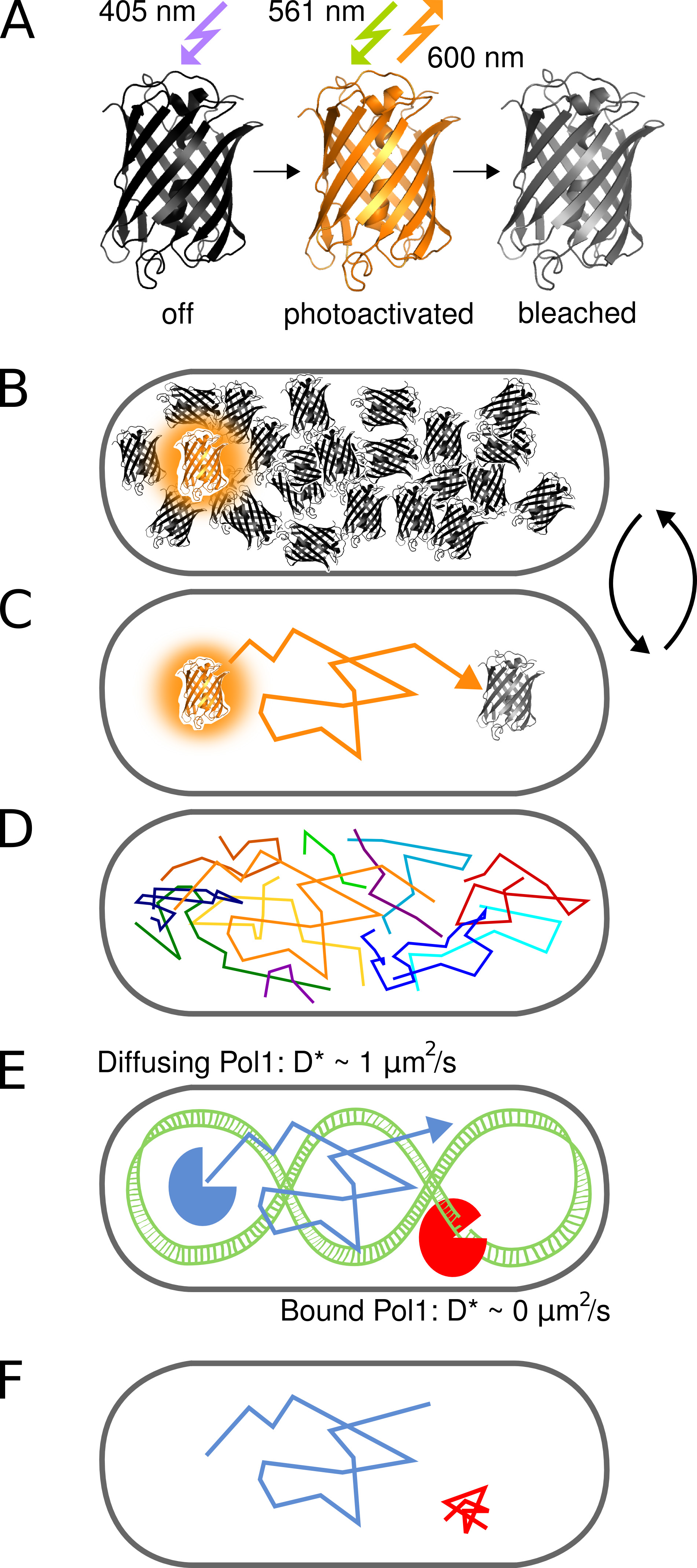
Figure 1. Représentation graphique de la méthode. (A) Le PAmCherry protéine fluorescente peut être photoactivé à partir d'un état initial non fluorescent lors de l'irradiation avec 405 nm lumière. L'état lumineux est excité à 561 nm et émet de la fluorescence à 600 nm jusqu'à ce que les agents de blanchiment fluorophores irréversible. (B) Contrôle du taux de photo-activation permet l'imagerie d'un seul stochastique activated protein PAmCherry de fusion par cellule à tout moment pendant la arbitrairement grande piscine de molécules qui n'ont pas encore été activées ou ont déjà été blanchis reste dans un état sombre. (C) La position de la molécule fluorescente est déterminée à partir du centre de la PSF et tr isoléacked pour plusieurs trames jusqu'à photoblanchiment. (D) Les Titres de nombreuses molécules sont enregistrées de manière séquentielle. (EF) L'interaction d'une protéine de liaison à l'ADN avec une séquence cible ou de la structure chromosomique arrête le mouvement diffusif aléatoire. Molécules liées et non liées se distinguent par le coefficient apparent de diffusion D * extrait de singles. La fraction résultant de molécules liées donne une mesure quantitative de l'activité d'une protéine de liaison à l'ADN in vivo. Cliquez ici pour agrandir l'image.
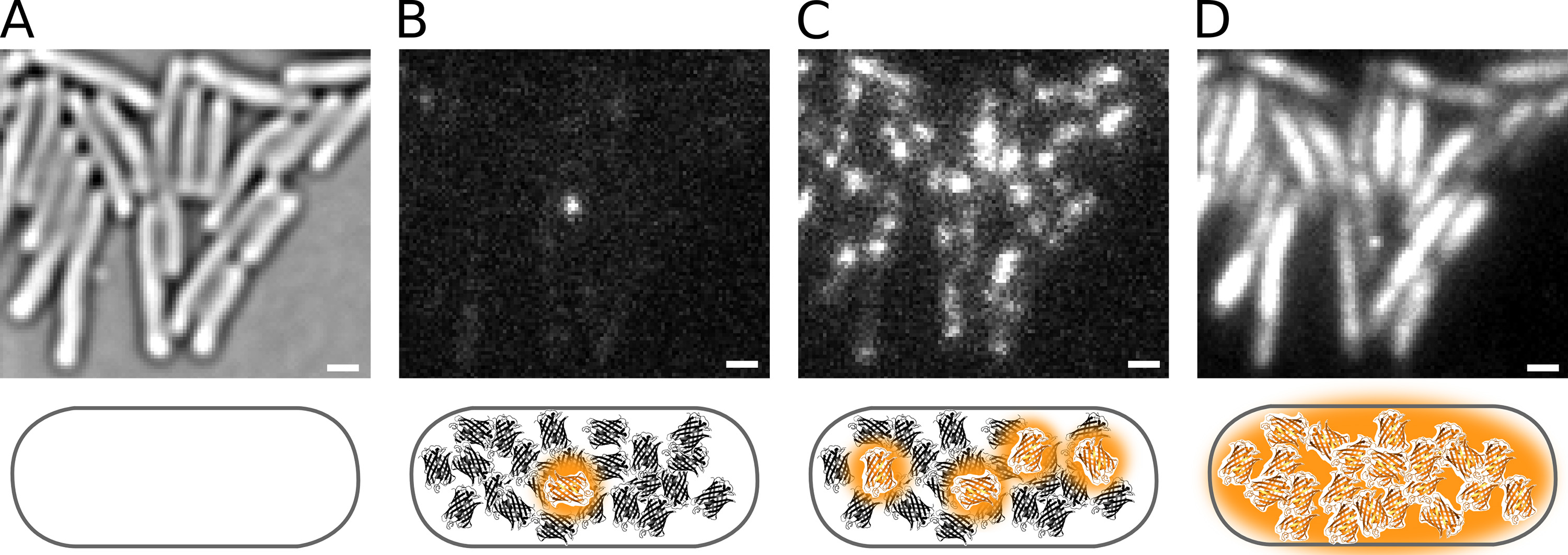
Figure 2. Photoactivation de Pol1-PAmCherry en direct E. cellules de E. coli. Barres d'échelle: 1 um. Schémas sont indiqués sous chaque panneau. ( A) l'image de microscopie optique des cellules immobilisées sur un pad agarose transmissibles. (B) Phototactivating un seul fluorophore PAmCherry dans une cellule. (C) Un taux de photo-activation plus élevée augmente le nombre de molécules fluorescentes. (D) PAmCherry intégré fluorescence d'un film PALM. Cliquez ici pour agrandir l'image.
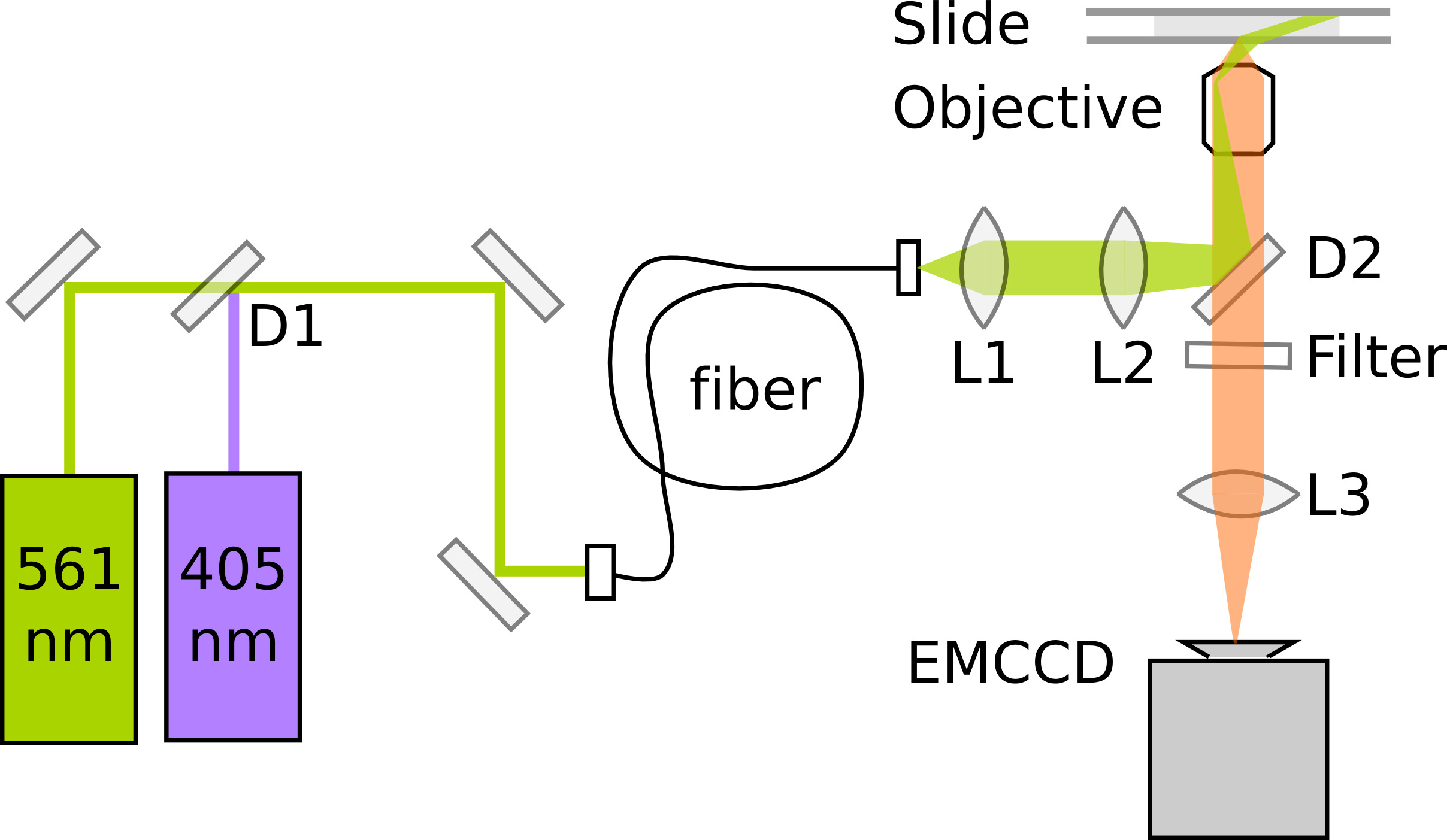
Figure 3. Schéma d'une installation de PALM minimale pour les protéines photoactivante et imagerie PAmCherry fusion D1:. Miroir dichroïque (par exemple 550 passe à long nm). D2: Le miroir dichroïque (par exemple 570 passe à long nm). L1: collimateur. L2: lentille TIR. L3: lentille de métro.1177/51177fig3highres.jpg "target =" _blank "> Cliquez ici pour agrandir l'image.

Figure 4. Images représentatives d'un film PALM avec 15,26 ms / cadre barres d'échelle: 1. Pm. (A) image lumineuse de cellules immobilisées sur un tampon d'agarose transmissibles. (B) l'image d'arrière-plan foncé mesurée sur la caméra EMCCD avec les lasers éteint. (C) Excitation image de fond sous excitation continue 561 nm avant photoactivation. (DF) Augmentation 405 nm intensité conduit à des taux plus élevés de photoactivation de PAmCherry, imagées sous continu 561 nm excitation. Les zones encadrées sont présentés ci-dessous agrandie. (D) faible intensité de 405 nm (<1 mW) actifs très peu de molécules fluorescentes par FOV. (E) moyen 405résultats intensité nm (~ 2 mW) de photoactivation dans une bonne densité de PSF pour la localisation et l'analyse de suivi. (F) supérieur 405 de l'intensité de la nm (~ 10 mW) active plus d'une molécule fluorescente dans certaines cellules, qui obscurcit la localisation et l'analyse de suivi. Cliquez ici pour agrandir l'image.

Figure 5. Illustration de l'analyse de la localisation. Barres d'échelle: 1 um (A) filtrage passe-bande élimine le bruit de pixels parasites et aplatit gradients d'intensité à travers le champ de vision.. (B) PSF candidats sont identifiés dans l'image filtrée en fonction d'un seuil défini par l'utilisateur qui est choisi pour minimiser les fausses détections positifs et faux négatifs. Le threshold correspond à l'intensité minimale d'un pixel candidat divisée par l'écart-type de fond (rapport signal-sur-bruit, SNR). (C) Le pixel localement plus brillants qui passe le seuil sert d'estimation de localisation initiale (croix orange) pour un ajustement gaussien elliptique en deux dimensions. Barres d'échelle: 0,5 um. La super-résolution localisation résultant (croix bleue) a une précision moyenne de σ loc = 40 nm. Cliquez ici pour agrandir l'image.
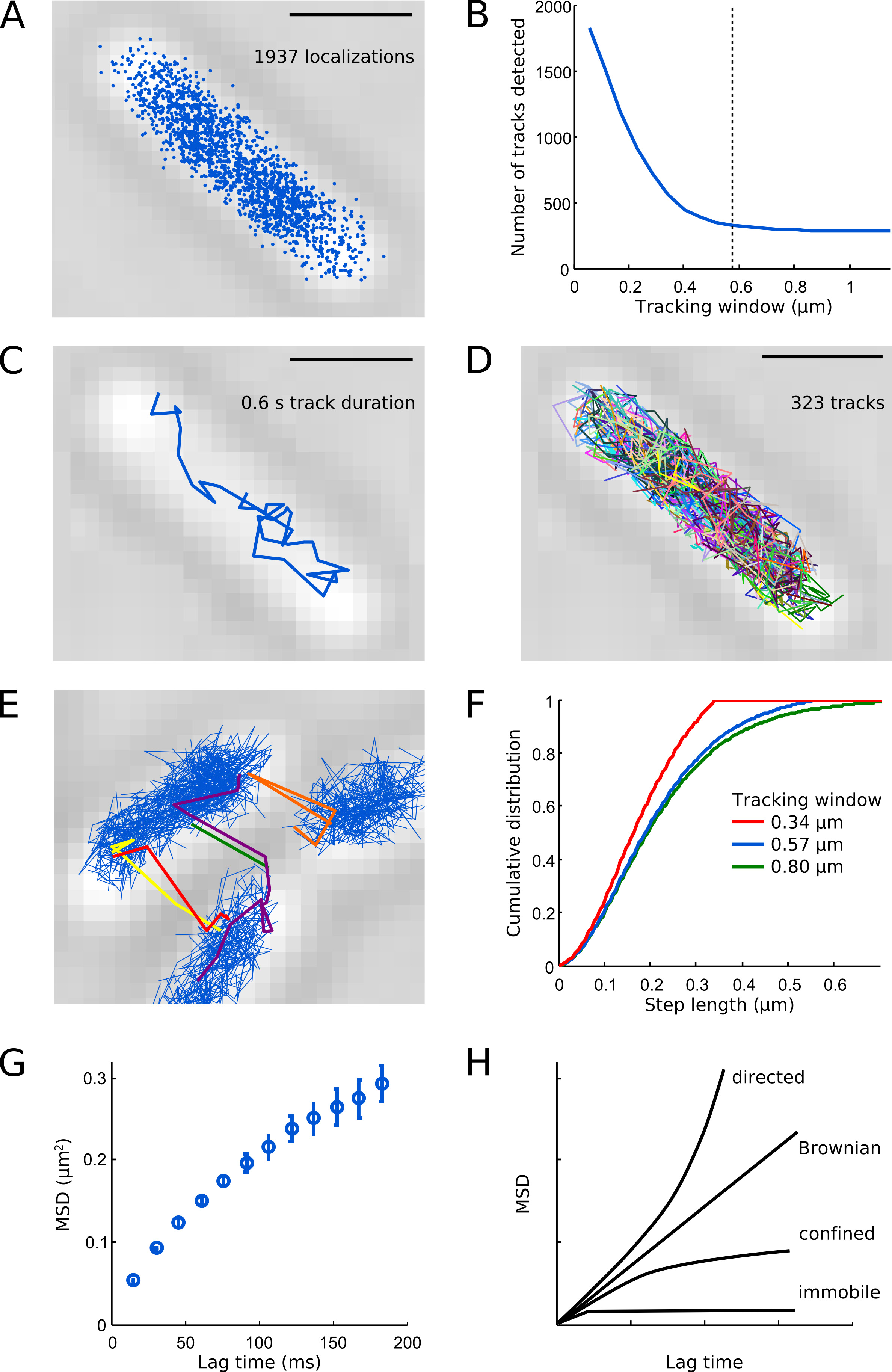
.. Figure 6 Illustration de l'analyse de suivi des barres d'échelle: 1 um. (A) Toutes les localisations détectées de Pol1-PAmCherry dans une cellule d'exemple. (B) Nombre de titres détectered dans l'exemple de cellule en fonction de la fenêtre de suivi. Windows Small de suivi répartis trajectoires de molécules, ce qui conduit à un artefact nombre élevé de pistes. La ligne en pointillés indique notre choix pour le paramètre de la fenêtre de poursuite (0,57 pm, 5 pixels) - ce qui donne un bon compromis entre la détection de la distribution complète d'étapes et en gardant les trajectoires des différentes molécules intactes. (C) Exemple piste d'une seule molécule Pol1-PAmCherry. (D) Toutes les pistes mesurées indiquées dans des couleurs aléatoires. (E) des objets de suivi si la fenêtre de poursuite est choisi trop grand (ici 0,8 um, 7 pixels) ou la densité de PSF par image est trop élevé. (F) Distributions cumulées des longueurs de pas pour les fenêtres de suivi: 0,34 um (3 pixels, ligne rouge), 0,57 um (5 pixels, ligne bleue), et 0,80 um (7 pixels, ligne verte). Notez que la fenêtre de suivi de 0,34 um coupe étapes de plus de 0,34 um qui a clairementtronque la distribution complète d'étapes. La fenêtre de 0,57 um de suivi détecte pratiquement la même distribution des étapes comme le fait la fenêtre de suivi de 0,80 um. Courbe (G) MSD montre diffusion limitée de Pol1. Courbes (H) Schéma de TMS pour le mouvement dirigé, mouvement brownien, la diffusion limitée, et les particules immobiles. Cliquez ici pour agrandir l'image.
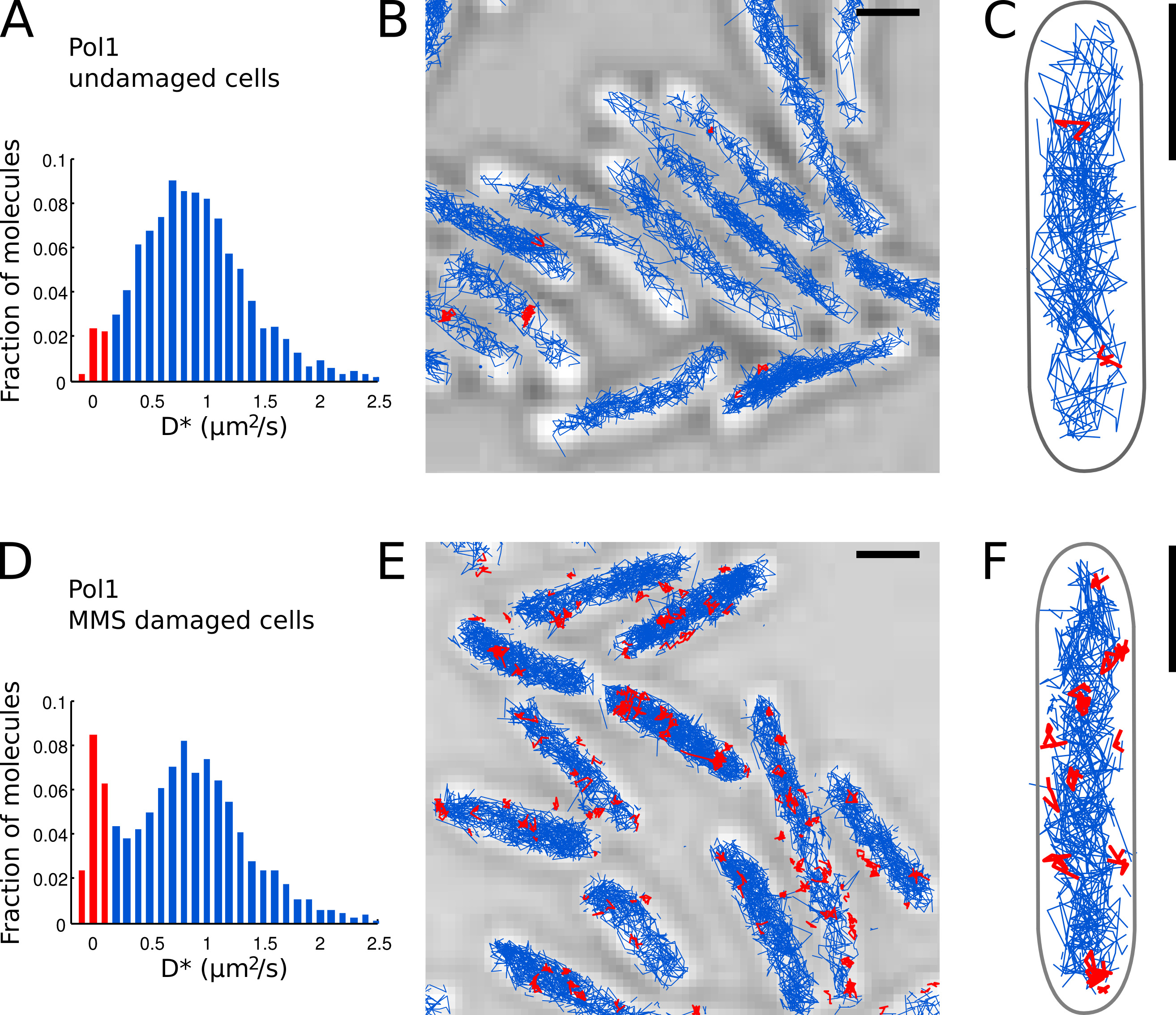
Figure 7. La mesure directe de l'activité de réparation d'ADN de Pol1 en direct E. coli. cellules barres d'échelle: 1 um. (A) de l'histogramme de la coefficient apparent de diffusion D * pour toutes les pistes de 4 ou plusieurs étapes dans un FOV de cellules intactes (N = 4162 titres). La population de molécules classées comme b des plaies est mis en évidence en rouge. (Colombie-Britannique) Titres de Pol1-PAmCherry sont présentés sur une image lumineuse transmise de microscopie. Titres classés comme lié en fonction de leur coefficient de diffusion sont indiquées en rouge. (D) D * histogramme des pistes pol1 mesurées dans les cellules immobilisées sur un pad agarose mM MMS 100 et incubées pendant 20 min avant l'imagerie (N = 2128 pistes). La population de molécules liées engagés dans la réparation d'ADN est représenté en rouge. (EF) Pol1-PAmCherry pistes sur l'image de microscopie lumière transmise montrant les traces des événements Pol1 de réparation d'ADN simples en rouge. Cliquez ici pour agrandir l'image.

Film 1. Construire une installation de PALM sur mesure."Target =" _blank JoVE_Uphoff_Movie1.avi "> Cliquez ici pour voir la vidéo.
Discussion
Nous discutons de plusieurs éléments clés pour la réussite de l'expérience.
Choix et d'expression de la protéine de fusion fluorescente: Il ya une grande palette de protéines fluorescentes photoactivables et photoswitchable 17. Le choix précis dépend des caractéristiques microscopiques, en particulier les lasers et les filtres disponibles. La combinaison de 405 nm et 561 nm est idéal pour des protéines fluorescentes photoactivables communs. Nous avons choisi PAmCherry 6 car il est monomère et montré aucune agrégation dans les cellules. En outre, la photoactivation irréversible permet de compter le nombre de fluorophores activés pour mesurer des nombres de copies de protéine par cellule. Au lieu d'exprimer la protéine de fusion à partir d'un plasmide, nous préférons l'insertion chromosomique du gène codant pour la protéine de fusion au niveau du locus de type sauvage. Ceci assure le remplacement complet de la protéine d'intérêt avec le fluorescente version et le maintien du niveau d'expression de type sauvage.
taux de photoactivation: Il est important d'ajuster le taux de photo-activation de telle sorte que, en moyenne, moins d'une molécule par cellule est dans l'état fluorescent dans n'importe quelle image de la vidéo. Cela dépend de l'intensité à 405 nm et le nombre de molécules de gauche à être activé. À des densités d'imagerie très faibles, cependant, pas toutes les molécules seront imagé avant la fin du film ou de très longues films doivent être acquis. Le nombre d'images enregistrées par film dépend du nombre de copies des protéines de fusion par la cellule et la durée de vie moyenne de photoblanchiment PAmCherry les conditions d'excitation utilisés. Le nombre de copies est de Pol1 ~ 400 molécules / cellule 1 et la valeur moyenne de la distribution de la durée de vie photoblanchiment exponentielle était ~ 4 trames. En augmentant l'intensité à 405 nm progressivement, l'activation est répartie uniformément sur les 10 000 images du film. Par conséquent, chaque cellule est occupied par des molécules fluorescentes pour un total de ~ 1600 cadres, assurant peu de chevauchement de PSF et les complications de suivi dans un film de 10 000 cadres.
temps d'exposition et d'excitation intensités: Tout d'abord, le temps d'exposition doivent être suffisamment courte pour observer PSF nettes avec peu de mouvement flou. Cependant, la vitesse de défilement doit être choisie pour fournir le mouvement moléculaire observable entre trames successives au-delà de l'incertitude de localisation, sinon photons cruciales sont gaspillées par suréchantillonnage la piste. Le mouvement des molécules non liées doivent être échantillonnés à des intervalles suffisamment longue de temps pour se distinguer clairement de mouvement apparent de molécules liées en raison de l'incertitude de localisation. Lorsque la durée d'exposition est réglé, l'intensité de la PSF doit être ajustée. La précision de la localisation d'une PSF augmente avec le nombre de photons détectés au cours de la durée d'une trame. Intensités d'excitation plus élevées augmentent les bu de taux d'émission de photonst également le taux de photoblanchiment et de signal d'arrière-plan. Utiliser la plus faible intensité d'excitation qui donne la précision de localisation souhaitée. Pour Pol1-PAmCherry nous avons choisi 15,26 ms / cadre et 3,5 mW 561 nm excitation (400 W / cm 2). Il est important de confirmer la viabilité des cellules pour les conditions d'imagerie particulier en surveillant la croissance des cellules et la morphologie avant et après l'acquisition de données (voir Renseignements supplémentaires dans Uphoff et al. 7).
Pol1 présente un temps de liaison de ~ 2 sec sur un substrat d'ADN in vivo lacunaire 7, nous attendons donc la majorité des molécules peut se faire soit à l'état lié ou non lié pendant toute la durée d'une piste. Molécules liées apparaissent essentiellement immobile à cause des sites chromosomiques ont un coefficient de diffusion de plusieurs ordres de grandeur plus faibles (~ 10 -5 2 um / sec, Elmore et al. Pol1 18) de diffusion dans le cytoplasme (~ 1 um 2 / sec).
analyse de Diffusion: Le coefficient apparent de diffusion D * est calculée à partir de la MSD de pistes individuelles, en moyenne sur une période minimale de 4 étapes (5 images) pour réduire l'erreur statistique. A noter que ~ 75% des molécules de chlore en moins de cinq trames pour les conditions d'imagerie décrites. Ces courtes pistes ne fournissent pas de certitude statistique suffisante pour distinguer les molécules liées et non liées. Toutefois, les fractions relatives des molécules liées et non liées qui rendent compte de l'activité de la protéine sont indépendants du nombre total de pistes analysés.
Il est utile de tenir compte de l'erreur de localisation PSF (σ loc) dans le calcul de D *, car l'incertitude ajoute une étape au hasard apparent pour chaque localisation d'une molécule 15.
Pour améliorer la classification des molécules liées et de diffusion, nous vous recommandons de calcul D * both à partir des déplacements à une seule étape et les déplacements au cours de la période de deux trames. Il est alors possible de définir deux seuils distincts D *: D * (15 ms) <0,15 um 2 / s et D * (30 ms) <0,075 um 2 / sec.
Notez que D * est un coefficient de diffusion apparent qui est affectée par le confinement des cellules de la voie ferrée et le mouvement de flou dû à la diffusion au cours du temps d'exposition. Pour extraire précises des coefficients de diffusion impartiales, il s'est avéré utile de comparer le mouvement observé à des données simulées basées sur un modèle de mouvement brownien 5,7 stochastique. Des données simulées peuvent également être utilisés pour tester les procédures d'analyse des données.
Les applications potentielles de cette méthode: Nous décrit une approche générale pour la visualisation et la quantification des interactions protéine-ADN in vivo par le changement de la mobilité d'une protéine lors de la liaison au chromosome. Les activités de l'ADN oudes protéines liant l'ARN impliquées dans la réparation, la replication, la transcription et la maintenance des chromosomes peuvent ainsi suivre en temps réel au niveau d'une seule cellule avec une résolution spatiale inférieure à la limite de diffraction optique. Photoactivée suivi une seule molécule s'étend méthodes de suivi classiques qui sont limités à quelques molécules marquées par cellule. Une autre méthode qui mesure la diffusion moléculaire in vivo est Fluorescence Recovery après photoblanchiment (FRAP). Bien que FRAP est très utile pour mesurer des caractéristiques globales de diffusion dans de grandes cellules, il est limité dans sa capacité à résoudre plusieurs espèces moléculaires avec des mobilités différentes dans un environnement spatialement hétérogène, en particulier pour les petites cellules bactériennes.
Nous avons appliqué photoactivée seule molécule de suivi pour mesurer les activités de liaison à l'ADN et localisations subcellulaires d'une gamme de différentes protéines dans E. coli y compris Pol1, l'ADN ligase, la protéine Fis, ADNpolymérase III 7, ainsi que de maintenance, construction de chromosomes protéines MukB, E, et F 19. Nous prévoyons que le procédé peut également être adapté à d'autres types de cellules.
Déclarations de divulgation
Les auteurs déclarent qu'ils n'ont aucun intérêt financier concurrents.
Remerciements
Nous reconnaissons Justin Pinkney et Johannes Hohlbein de l'aide à la construction du microscope sur mesure et Seamus Holden pour le logiciel de localisation. Rodrigo Reyes-Lamothe est remercié pour fournir l'E. coli souche. La recherche a été financé par le septième programme-cadre européen Commission du programme Grant FP7/2007-2013 SANTÉ-F4-2008-201418, Royaume-Uni biotechnologie et sciences biologiques du Conseil de recherches Grant BB/H01795X/1, et le Conseil européen de la recherche Grant 261227 à ANK. DJS a été financé par une subvention de WT083469 Programme Wellcome Trust. SU a été soutenue par une bourse de doctorat MathWorks.
matériels
| Name | Company | Catalog Number | Comments |
| E. coli strain carrying a chromosomal insertion for a PAmCherry DNA-binding fusion protein | created by Lambda-Red recombination | ||
| MEM amino acids | Invitrogen | 11130-051 | minimal medium supplement |
| MEM vitamins | Invitrogen | 11120-052 | minimal medium supplement |
| Agarose | BioRad | 161-3100 | certified molecular biology grade |
| Microscope coverslips No 1.5 thickness | Menzel | BB024060SC | remove background particles by heating slides in furnace at 500 °C for 1 hr |
| Methyl methanesulfonate (MMS) | Sigma-Aldrich | 129925 | CAUTION: toxic |
| PALM setup | home-built | described in Uphoff et al.7 | |
| MATLAB | MathWorks | for data analysis and visualization | |
| Localization software | custom-written, available online | http://www.physics.ox.ac.uk/Users/kapanidis/Group/Main.Software.html | MATLAB and C++ software package that can be adapted for localization analysis. Cite Holden et al.13 if used in publication. |
| Tracking software | available online | http://physics.georgetown.edu/matlab/ | MATLAB implementation by Blair and Dufresne. Cite Crocker & Grier12 if used in publication. |
Références
- Friedberg, E. C. . DNA Repair and Mutagenesis. , (2006).
- Betzig, E., et al. Imaging intracellular fluorescent proteins at nanometer spatial resolution. Science. 313 (5793), 1642-1645 (2006).
- Mortensen, K. I., Churchman, L. S., Spudich, J. A., Flyvbjerg, H. Optimized localization analysis for single-molecule tracking and super-resolution microscopy. Nat. Methods. 7 (5), 377-381 (2010).
- Manley, S., et al. High-density mapping of single-molecule trajectories with photoactivated localization microscopy. Nat. Methods. 5 (2), 155-157 (2008).
- English, B. P., et al. Single-molecule investigations of the stringent response machinery in living bacterial cells. Proc. Natl. Acad. Sci. U.S.A. 108 (31), (2011).
- Subach, F. V., et al. Photoactivatable mCherry for high-resolution two-color fluorescence microscopy. Nat. Methods. 6 (2), 153-159 (2009).
- Uphoff, S., Reyes-Lamothe, R., Garza de Leon, F., Sherratt, D. J., Kapanidis, A. N. Single-molecule DNA repair in live bacteria. Proc. Natl. Acad. Sci. U.S.A. 110 (20), 8063-8068 (2013).
- Datsenko, K. A., Wanner, B. L. One-step inactivation of chromosomal genes in Escherichia coli K-12 using PCR products. Proc. Natl. Acad. Sci. U.S.A. 97 (12), 6640-6645 (2000).
- Reyes-Lamothe, R., Possoz, C., Danilova, O., Sherratt, D. J. Independent positioning and action of Escherichia coli replisomes in live cells. Cell. 133 (1), 90-102 (2008).
- Neidhardt, F. C., Bloch, P. L., Smith, D. F. Culture medium for enterobacteria. J. Bacteriol. 119 (3), 736-747 (1974).
- Tokunaga, M., Imamoto, N., Sakata-Sogawa, K. Highly inclined thin illumination enables clear single-molecule imaging in cells. Nat. Methods. 5 (2), 159-161 (2008).
- Crocker, J. C., Grier, D. G. Methods of digital video microscopy for colloidal studies. J. Colloid. Interface. Sci. 179 (1), 298-310 (1996).
- Holden, S. J., et al. Defining the limits of single molecule FRET resolution in TIRF microscopy. Biophys. J. 99 (9), 3102-3111 (2010).
- Holden, S. J., Uphoff, S., Kapanidis, A. N. DAOSTORM: an algorithm for high-density super-resolution microscopy. Nat. Methods. 8 (4), 279-280 (2011).
- Wieser, S., Schütz, G. J. Tracking single molecules in the live cell plasma membrane-Do's and Don't's. Methods. 46 (2), 131-140 (2008).
- Arnauld, S., Nicolas, B., Hervé, R., Didier, M. Multiple-target tracing (MTT) algorithm probes molecular dynamics at cell surface. Protoc. Exch. , (2008).
- Lippincott-Schwartz, J., Patterson, G. H. Photoactivatable fluorescent proteins for diffraction-limited and super-resolution imaging. Trends Cell Biol. (11), 555-565 (2009).
- Elmore, S., Müller, M., Vischer, N., Odijk, T., Woldringh, C. L. Single-particle tracking of oriC-GFP fluorescent spots during chromosome segregation in Escherichia coli. J. Struct. Biol. 151 (3), 275-287 (2005).
- Badrinarayanan, A., Reyes-Lamothe, R., Uphoff, S., Leake, M. C., Sherratt, D. J. In vivo architecture and action of bacterial Structural Maintenance of Chromosome proteins. Science. 338 (6106), 528-531 (2012).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon