Method Article
生細胞内のタンパク質断片相補アッセイ(PCA)によるゲノムワイドなタンパク質間相互作用のスクリーニング
要約
Proteins interact with each other and these interactions determine in a large part their functions. Protein interaction partners can be identified at high-throughput in vivo using a yeast fitness assay based on the dihydrofolate reductase protein-fragment complementation assay (DHFR-PCA).
要約
Proteins are the building blocks, effectors and signal mediators of cellular processes. A protein’s function, regulation and localization often depend on its interactions with other proteins. Here, we describe a protocol for the yeast protein-fragment complementation assay (PCA), a powerful method to detect direct and proximal associations between proteins in living cells. The interaction between two proteins, each fused to a dihydrofolate reductase (DHFR) protein fragment, translates into growth of yeast strains in presence of the drug methotrexate (MTX). Differential fitness, resulting from different amounts of reconstituted DHFR enzyme, can be quantified on high-density colony arrays, allowing to differentiate interacting from non-interacting bait-prey pairs. The high-throughput protocol presented here is performed using a robotic platform that parallelizes mating of bait and prey strains carrying complementary DHFR-fragment fusion proteins and the survival assay on MTX. This protocol allows to systematically test for thousands of protein-protein interactions (PPIs) involving bait proteins of interest and offers several advantages over other PPI detection assays, including the study of proteins expressed from their endogenous promoters without the need for modifying protein localization and for the assembly of complex reporter constructs.
概要
タンパク質相互作用ネットワーク(ピン)は、タンパク質が機能的にセル1に編成されている方法の低解像度マップを提供します。各物理的な2つのタンパク質間の接続、またはタンパク質 - タンパク質相互作用(PPI)は、そのようなタンパク質複合体の中に見られるように、時間的に安定して関連付けを表すことができ、それは細胞の構造的組織化に寄与する。これらの接続には、活性、安定性、局在化および2のパートナーの相互作用を調節する過渡関連を表すことができる。与えられたタンパク質の物理的相互作用パートナーを識別すること、したがってそのタンパク質2,3の機能と規制に関する豊富な情報を提供しています。これらの理由から、大きな努力が大腸菌 4-6、 シロイヌナズナ7、サッカロマイセス·セレビシエ(Saccharomyces cerevisiae)8-12、キイロショウジョウバエ<などのモデル生物でのPINのマッピングに向けて置かれている/ em>の13、線虫は、14と15 サピエンスホモエレガンス 。これらの研究は、タンパク質が細胞内で編成されているかを重要な洞察および未知の機能を有するタンパク質上のため、重要な情報を提供してきた。
いくつかの戦略は、ピンを研究するために長年にわたって開発されてきた。これらの技術は広く(16-18に概説)はPPIの上に提供する情報の種類に基づいて、三つのカテゴリーに分類することができる。最初の1は、酵母ツーハイブリッドおよびその誘導体19に基づいています。これらの技術は、バイナリのネットワークを構築することができ、タンパク質のペアの間の直接的な関連、上の情報を提供します。第二のファミリーは、ベイトタンパク質のアフィニティー精製および質量分析20に続くアフィニティー精製などのそれらの関連パートナーの同定に基づく。これらのアプローチは、直接であるタンパク質のグループを識別するまたは間接的に、一般的に安定した方法で、関連する、およびタンパク質複合体を同定するために非常に強力である。第三のアプローチは、タンパク質断片相補アッセイ(PCAは)11,21に基づいています。それは、タンパク質間の直接および近位の関連付けを検出することができますように、このアプローチは、二人の元のアプローチの解像度の中間レベルを提供します。最近18を見直しとして各技術は、独自の長所と短所があります。
そのプロテオームは、他のモデル真核生物のものよりも比較的複雑であるため、とのPPIを検出するためのハイスループットアッセイは、最初にアッセイされ、より効率的であるたために最善のような真核生物のPINは、部分的には、これまでで出芽酵母Saccharomyces cerevisiaeの一つですこのモデル生物9-12で実装。酵母系に特に強力な方法は、ジヒドロ葉酸還元酵素タンパク質断片相補アッセイ(DHFR-PCA)、されているアッセイである標準と摂動条件11,22-26における酵母PINを研究するために別のコンテキストで使用。この方法は、定量的に27で内因性発現レベルにおける所与のベイトタンパク質と相互作用パートナー11,21の天然の細胞内の局在化のための直接近直接PPIの検出を可能にする生存アッセイに依存している。このアッセイ(高密度コロニーアレイ上すなわちコロニーサイズ)を使用して得られた信号は、このように、野生型細胞のいずれかとほぼ同等の細胞環境におけるベイトとプレイとの間に形成されるタンパク質複合体の量を反映する。アッセイは、その2つのタンパク質が相互作用するときに目的の2つのタンパク質に融合されるDHFRの2つの相補断片が接近することにより、葉酸代謝に関与するレポーター酵素、ジヒドロ葉酸レダクターゼ(DHFR)の再構成に基づいている今度は酵素活性1の可逆的再構成につながる1とメトトレキサート(MTX; 図1)を含む培地上で株の増殖。この化合物は、内因性DHFR酵素ではなく、アッセイ28で使用される突然変異したものを阻害する。 PCA株の二つのコレクションを含む1 マタで株〜4300 ORFはDHFR Fに融合した[1,2]フラグメントおよびORFはDHFR [3]断片に融合、に購入することができた株α〜4800 MATを含む1すべての実験室での小規模または大規模にDHFR-PCAを実装する。ここでは、1ベイトタンパク質と、このアッセイを用いて〜4800獲物タンパク質との間のPPIをスクリーニングするための一般的なが、詳細なプロトコルを記述します。
プロトコル
1.建設/ベイト株の検証
- 関心の餌株はのMATa DHFR F [1,2]コレクションで使用可能な場合はステップ1.1.1で説明したように、ステップ1.1.2で説明したようにそうでない株を構築、コレクションからそれを取り出す。
注:プロトコルは、ここで説明するこのコレクションは、DHFR F [1,2]コレクションより多くの株が含まれているように、獲物として餌およびDHFR F [3]コレクションとしてDHFR F [1,2]菌株を使用しています。一つはインタラクトームの高いカバレッジを必要とする場合ベイト株はDHFR F [3]コレクション又は両方の向きでのみ利用可能である場合は、それは逆に画面を行うことができる。- 氷上で1時間ベイト株を含んでグリセロールストックプレートを解凍する。 95%エタノールを用いてプレートを覆うアルミホイルを滅菌する。ピアース選択酵母エキスペプトン上のセルとグリセロールストックの連勝2-3μlの再懸濁するために上下に無菌の先端、ピペットを用いて箔単一のコロニーを単離するために、デキストロース(YPD)+100μg/ mlのヌーセオスリシン(ナット)。 30℃で2日間インキュベートする。
- 餌PCA株の構築( のMATa DHFR F [1,2])。
- 高忠実度ポリメラーゼと標準的なPCRプロトコルを使用して、DHFR Fを増幅プラスミドpAG25 - リンカー-DHFR Fから[1,2]カセット[1,2] -ADHtermすると、オーバーハングを有するオリゴヌクレオチドを使用すると、ORFの最後の40bpのに相同な終了停止コドン(フォワードプライマー)および遺伝子の3'-UTR(リバースプライマー)( 図1A)の最初の40塩基対に除く3 '末端。
- 29( 図1A)のような標準LiOAc / PEG酵母形質転換プロトコルを用いて(通常BY4741株)のコンピテント酵母細胞中にPCR産物を形質転換する。
- 陽性形質転換体を単離するための選択的YPD +ナット媒体上のプレート。
- 適切なDHFR F [1,2]の融合を確認するために、単離したコロニーの診断コロニーPCRを行います。 PRを使用しimers遺伝子コード配列(フォワードオリゴ)DHFR融合の上流約100 bpおよび2)カセット(オリゴリバース)( 図1B)のADHターミネータ中で)1をアニールする。
- 適切な遺伝子融合を確認するために、サンガー配列決定によってPCR産物をシーケンスする。
- -80℃で25%グリセロール中で確認された餌株をアーカイブします。
注:このプロトコルは、この段階で一時停止することができます。
2.ピン·ツール滅菌と印刷手順
注:後述の滅菌手順はBM3-BC(S&Pロボティクス)ロボットプラットフォームによって操作ピンツール用に最適化されたが、他のプラットフォームに適合させることができる。このセクションでは、プロトコルの残りのために別の培地から細胞を転送するために使用されるピンツール殺菌、印刷手順を記載している。社内でこれらのルーチンを実行するために使用されるスクリプトは、要求に応じて得ることができる。 THAに注意してくださいtはすべてのステップは、手動、ピンツール30を用いて、ロボットプラットフォームを必要とせずに行うことができる。
- ロボットプラットフォーム上の適切なピンツールをマウントします。
- 次のようにクリーニングとウェットステーションを準備します。
- 水浴ステーション滅菌水500mlを加える。
- ブラシステーション内の滅菌水320ミリリットルを追加します。
- 寒天プレートに液体培養を含むマイクロタイタープレートから複製するときに寒天プレートに、または400ミリリットル(固化した培地の35ミリリットルを含む、86×128 mmのomnitray)寒天プレートから複製するときに超音波処理器内の70%エタノール380ミリリットルを追加します。
- (無菌の空のomnitrayからなる)、ウェットステーション内の滅菌水35ミリリットルを追加します。
- ロボットプラットフォームを必要とする各一日の開始時に、超音波処理器浴中で1分間のピンツールを5回滅菌する。一方、ロボットの筐体を滅菌する5分間のUVランプをオンにします。
注:ロブ場合、これは必須ではありませんOTは、無菌のボンネットの下に収納されている。 - 各ピンツール複製ラウンドの間に、次のようにピンツールを殺菌:
- 細胞塊を除去するために水浴局において10秒間のピンツールを5回浸す。
- ブラシステーションで二度前後にピンツールを浸す。
注:回転ブラシは、残留細胞を除去します。 - 超音波処理器ステーションで20秒間二回のピンツールを浸す。
注:ピンの残りの細胞を超音波処理することにより除去またはエタノールによって殺される。 - ピンの浸漬深さは適切な滅菌を確実にするためにそれぞれの連続バスで増加することを確認してください。
- 25秒間空気乾燥機ステーション内のピンツールを乾かします。
- ソースプレート上の細胞を取る前に、omnitrayに滅菌水35ミリリットルを含んで湿った駅でピンを濡らす。
- ソースプレートからコロニーで二回のピンを浸し。
- AGに触れて、新鮮に注ぎ目的プレートに印刷アル表面倍(以下、「印刷」配列のアクションと呼ばれる)。
自動ロボットプラットフォームを使用して1536アレイではDHFR F [3]コレクションの3凝縮
- 氷上で解凍DHFR F [3]コレクション(60 96ウェルプレート)、およびL-DHFR F [3]コントロール株DHFR Fが含まれていた( 図1C)、(3)断片で埋め、追加の96ウェルプレートと上流のリンカはネガティブコントロールとして単独で発現。
注:原則として、この断片は、任意のDHFRフラグメント融合タンパク質と相互作用してはならない(詳細についての説明を参照)。 - 井戸の中で交差汚染のリスクを回避するためにアルミホイルを取り外す前に、遠心機のプレート(クイックスピン)。
- 384株( 図2A)の16のアレイ上にコレクションを凝縮。これを行うには、それぞれ384アレイに対して、選択的YPD + 250 / mlのハイグロマイシンB(HygB)の4象限上の印刷4グリセロールプレートが目を使用してomnitray(ここでは、384配列は、それぞれ、2×2マトリックス·レイアウト内96箇所からなる、4つの等しいinterspaced象限に分割することができます)E 96ピンツール。正確に4 1536のアレイを埋める64プレートの最終セットを持つために60他のプレートの間にL-DHFR F [3]ネガティブコントロールを含む4 96ウェルプレートを挿入します。ステップ2で説明したように、各複製サイクルの間にピンツールを滅菌する。
注:4最後の1536アレイのそれぞれに1つそのようなプレートを持っているために、L-DHFR F [3]のプレートを挿入します。 - 30℃で2日間、プレートをインキュベートする。注:この段階では、DHFRのコレクションは、寒天プレート上で1ヶ月まで4℃で384フォーマットで格納することができる。
- 1536株( 図2A)の4つの配列にコレクションを凝縮。これを行うには、それぞれ1536アレイに対して、384ピン·ツールを使用してステップ3.2のように、同じ培地のプレートの4象限に384株の4つのアレイを印刷する(ここでは、1536アレイは、4(e)に細分することができる各2×2のマトリクスレイアウトで384箇所からなるqually interspaced象限)。
- 30℃で2日間、プレートをインキュベートする。注:この段階では、DHFRのコレクションは、寒天プレート上で1ヶ月まで4℃で1536形式で保存することができる。
- 1536ピンツールを使用してコロニーサイズを標準化するために、同じ培地上で4つの配列を複製。
- 30℃で2日間プレートをインキュベートします。
4.ハイスループットDHFR-PCA手順
- 50ミリリットルチューブ( 図2B)に液体YPD +ナット20mlのステップ1.1.1または1.1.2から得餌株(DHFR F [1,2]の融合)の培養物に接種。
- 培養物が飽和に達するように、250rpmで振盪しながら30℃で2日間インキュベートする。
- インキュベーションの2日後、YPD +のNAT omnitray上の文化のプレート5ミリリットル。細胞は5〜10分間の表面に吸着しましょうと余分な液体( 図2B)を取り外します。繰り返しトン3回の反復を作るためwice。
- 30℃で2日間インキュベートする。
- 4倍を超えない各細胞ローンを使用して、1536ピンツールを使って(DHFR F [3]コレクション×3回の反復の4枚のプレートを交配するための十分な)ステップ4.3および4.4 12上のYPDプレートから餌株を印刷します。
- 1536ピン·ツール( 図2C)を使用して、餌細胞の上にDHFR F [3]コレクションの適切な配列を出力します。
- 株は、30℃で2日間インキュベートすることによって嵌合う。
- YPD + HygB +ナット( 図2C)を含むomnitrays上のコロニーを印刷して二倍体細胞を選択します。
- 30℃で2日間インキュベートする。
- ステップ4.8および4.9( 図2B)で説明したように二倍体の選択を繰り返します。
- それは毒性化合物であるようにMTXを扱う場合は注意が常に手袋を着用し、prote:。メディアはこれらのステップ21(注意翌日使用前にMTX(MTX培地)を含有するプレートを準備しますctionメガネと白衣それを扱う)( 図2C):
- 10倍-Lysを準備/脱イオン水に/ ADEアミノ酸ドロップアウトに会った。フィルター滅菌し、大量に必要に応じて、0.2μmの滅菌シリンジフィルターを使用してドロップアウト滅菌ボトル内に、または、0.2μmのボトルトップ濾過漏斗( 表1)。
- ジメチルスルホキシド(DMSO)中に10mg / mlのMTXストック溶液を準備します。溶液の調製後すぐに使用し、-20℃で、残りを凍結。それが感光性であるように、光から保護する。それを解凍した後、MTX再び凍結しないでください。
- 次のようにメディアを準備し(培地の成分および量はTarassov ら 11で用いたものと同じである。)。
- 培地1リットルについては、二つの別々のフラスコ内で混合:1)アミノ酸を含まない硫酸アンモニウムなしの酵母窒素塩基の6.69グラムおよび脱イオン水330 mlで2)貴金属寒天および500mlの脱イオン水を25g。
- 20分間121℃でオートクレーブのフラスコ。
- 少なくとも1時間55℃の水浴中で温度を平衡化する。
- 2つのフラスコを混合し、滅菌40%グルコース、10倍滅菌ドロップアウトを100ml、及びMTX 10mg / mlの20mlを50mlのを追加する。
- 35ミリリットルを注ぎomnitrays媒体の(下記注参照)。少なくとも時間半固化してみましょう。光から遮光板。
注:ここでは、omnitrayあたりのメディアの35ミリリットルを注ぎ、すべてのダウンストリームのステップのために重要であると等しい板厚を確保することが重要です。
- ロボットプラットフォームまたは均一なプレート照明との定期的なデジタルカメラで二倍体選択の第2ラウンドからの画像プレート。下流の分析を実行するときにアレイ上の空の位置を特定するためにこれらの写真を使用してください。カメラのパラメータが常に同じとロボットライトがオンになっていることであることを確認してください。
- MTXメディアUSI上の二倍体細胞を印刷1536ピンツールをngの。
- 乾燥を防ぐためにプラスチックバッグに30℃で4日間インキュベートする。
- ステップ4.11で説明したようにMTX培地を含有するomnitraysの第二のバッチを準備します。
- インキュベーションの4日後、ロボットプラットフォームまたは通常のデジタルカメラを用いた画像プレート。カメラのパラメータが常に同じとロボットライトがオンになっていることであることを確認してください。
- MTXメディアの第二のバッチ上の細胞を複製することによってMTX選択の第二ラウンドを実行します。
注:これはPCA株のバックグラウンド増殖を減少させ、定量的な解像度を増加させる。 - 乾燥を防ぐためにプラスチックバッグに30℃で4日間インキュベートする。
- などの画像プレートはステップ4.16で説明した
5.画像解析
- カスタムImageJの31スクリプトでコロニーアレイの画像を分析したり、そのような植民、Htのコロニーグリッド·アナライザ、セル·プロファイラ、コロニーイメージとして公開ソフトウェアを使用してR、ScreenMill、YeastXtractとギッター(32でコンパイル)。画像解析は、出力1または各配列の各位置のためのコロニーの大きさを含むいくつかのスプレッドシートは、すべてのダウンストリームの分析のためにこれらのコロニーのサイズを使用する必要があります。
注:この研究では、カスタムを使用ImageJのLeducqに記述されたスクリプトら 33(詳細については議論のセクションを参照)。
6.データ解析
注記:画像解析の結果は、エクセルまたはR 34のようなスクリプト言語を使用するものとしてタブで処理することができる。次の手順では、カスタムImageJの31スクリプトを使用して手順を説明します。
- カスタムスクリプトを使用して、画像解析からの出力ファイルを連結し、プレートと各列に注釈を付けると補足表1のように情報株。
- ここで、列"IntDenBackSub&amp;ログ2は、コロニーサイズ値(統合密度またはコロニー面積を変換する8221。補足表1)を用いた。
注:値の分布は、図3(a)のようになります。 - 各プレートの中央値を減算することにより、これらの値を正規化する。
注:自動画像取得における不平等なメディア量や変動から生じるプレートバイアスのためのこのステップを制御し、およびインター複製の分散( 図3B)を低減します。 - の反復実験の再現性を評価するために( 図3C)互いに相関することを確認します。
- 非相互作用ベイト獲物対から相互作用区別するために、L-DHFR F [3]のコントロールの分布の95 パーセンタイルに対応する高信頼閾値を設定する。
注記:この実験では、これは3.39( 図3D)に対応する。あるいは、そのようなバイオグリッド35で報告されているような既知の物理的な相互作用物質との重なりに基づいてしきい値を使用することができる。詳細については、説明を参照してください。 - どんな餌のために、フィルターのプレイは(詳細については説明を参照)DHFR-PCA画面で偽陽性の相互作用に関与すると補足表2(「フィルタリング」の欄に「1」として識別される)に記載されているように同定した。
- 各相互作用(補足表2の欄「平均スコア」)の3回の反復ののLog2正規化されたコロニーのサイズを平均。
物理インターアクターの7.検証小規模実験を用いて
注:上記のスコアまたは固体または液体MTX培地上での増殖アッセイを使用して、小規模な実験計画にDHFR-PCAアッセイを使用して検証することができる適用閾値に近いを有する特定の興味のある任意のPPI。以下の手順は、手動で二倍PCA株を構築し、MTX媒体上のスポットアッセイを実行するための手順を示している。実験者は、すべてのnecessarのためにこれらのステップを実行する必要がありますYコントロール(ベイト-DHFR F [1,2]のx L-DHFR F [3]、ジッパー - リンカー - DHFR倍体菌株とリンカ-DHFR二倍体菌株)。
- 1.1.2.6で生成された餌株のグリセロールストックのプレート2-3μL)、L-DHFR F [3]コントロール株、YPD +ナット、YPD +上のジッパー - リンカー-DHFRおよびリンカー-DHFR倍体菌株HygBと二回YPD +ナット+ HygBメディア、それぞれ。
- 代わりにYPD +ナット媒体のYPD + HygB培地の負担をDHFR F [3]コレクション内の関心の獲物を取得して、ステップ1.1.1の指示に従いますが、ストリーク。
- 獲物軌跡とシーケンス製品でDHFR F [3]の融合を確認するために、1.1.2.4のように診断PCRを行います。
- 交尾(ベイトX獲物、ベイトのx L-DHFR F [3]コントロール)と二倍体を形成させ、30℃で少なくとも2日成長して一倍体株と液体YPD培地の1ミリリットルを接種する。
- 固体YPD + HygB +ナット媒体に7.4での培養の4-5μlのストリーキングによって二倍体を選択します。 30℃で二日間成長する。
- 割当可成長アッセイを実行するために液体培養中で一晩(1ミリリットル)をトンつの単離コロニーと成長する。
- 使用前に一日MTXおよびDMSO(MTX培地と同じ成分が、MTXを含まない)のプレートを準備します。詳細は、ステップ4.11を参照してください。
- コントロール(DMSO)と選択プレート(MTX)で、異なる文化の段階希釈をスポットすることにより成長アッセイを実行します。
- OD = 1に前培養を希釈します。
- 滅菌96ウェルプレートに(625の希釈係数まで)5倍希釈を行う。
- スポットPCAメディア(DMSOおよびMTX)上の各希釈液4μlの。
- 乾燥を防ぐためにプラスチックバッグに30°Cでインキュベートする。
- ロボットプラットフォームまたは通常のデジタルカメラを使用して1日のインキュベーションの7の画像プレート。
結果
補足表2は、Nup82はDHFR F餌として[1,2]断片に融合した酵母タンパク質を用いて得られた代表的な結果の一例である。 L-DHFR Fで定義されたしきい値は、[3]のコントロールは、高い信頼ヒット( 図3Dおよび3E)を決定するために経験的な閾値として使用することができる。代替的に、スコアランキングは、遺伝子オントロジー濃縮度を実行するために使用することができ、または他の機能は、金基準37に基づいて、36を解析する。餌の既知の物理的相互作用因子はバイオグリッド35のようなデータベースから検索したデータ( 図3E&3F)に重ね合わせることができる。この例では、5つの8のうち、高い信頼のヒットは、以前にNup82の相互作用因子として報告されていると2はNup82の複体、Nup116とNup159( 図3F&3G)の一部である。複雑な、NSP1の他のメンバーは、すべてのインタラックが表示されない我々の実験でる。二つの餌食、Ade17とTEF2( 図3Fには示されていない)が、ハード閾値以上のスコアが適用されていたが、これらは、彼らが我々は(未発表の結果)を行っているPCAの画面のほぼすべての餌タンパク質と相互作用するように偽陽性である可能性が高い。一方、Pex30はNup82の新規物理的相互作用因子を表すことができ、我々は、低スループット( 図3G)でのDHFR-PCAを使用して、この相互作用を確認することができました。 Pex30はペルオキシソーム膜タンパク質であり、数の直接的な相互作用は、核膜孔複合体(NPC)とこの細胞小器官の間で報告されている。ツーハイブリッドスクリーンはPex30 38の物理的な相互作用因子として、他の二つのNPCタンパク質、Nup53とAsm4(Nup59)を同定し、そしてPex30とNup170間の遺伝的相互作用は、39を報告されている。他の二つの相互作用パートナーは、検出されNup120とNup85( 図3F&3G)、能力を示す、Nup82サブ複合体の一部ではありませんDHFR-PCAの中で、より大きな複合体11でsubcomplexes間の相互作用を検出する。
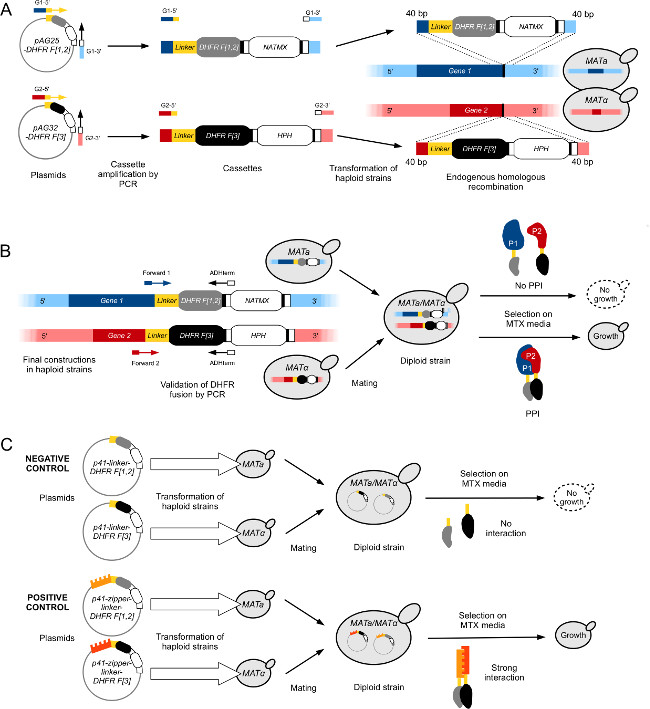
図1:半数体のMATaマットα株のハイスループットDHFR-PCAのためのエンジニアリング酵母株(。Leducqらから適応図2012 33)(A)建設は、DHFRとGene1(G1)とGene2(G2)を融合させF [1,2] -NatMXおよびDHFR F [3] -HPHカセット 、それぞれ。カセットは、その後、[3]の順方向でG1-5」とG2-5を'プライマープラスミドpAG25-DHFR F [1,2]とpAG32-DHFR Fから増幅、およびリバースプライマーG1-3'とG2-3」、とされている相同組換えにより標的遺伝子の3 '末端のゲノムに挿入する。得られたタンパク質は、P1及びP2は、それぞれDに融合されるHFR F [1,2]断片( のMATa)と柔軟なリンカーを介してDHFR F [3]断片(MATα)。(B)構造の検証中(A)はGene1のとGene2者のORFとの間の接合部を配列決定することにより行われるDHFRカセット。接合型の対向構築PCA株はその後、二倍体を形成するために交配させる。二つの相補的DHFR断片がDHFR酵素の活性を再構成P1とP2との間の相互作用によって近接させている場合、二倍体株は、MTX培地上で成長する。(C)建設管理の二倍体PCAは、DHFR-PCAスクリーニングのため株。陰性対照(L-DHFR)は、プラスミドP41リンカーDHFR F [1,2]およびP41リンカーDHFR F [3] 11のそれぞれに別々に半数体のMATa及びMATα株を形質転換することによって構築される。 2株はDHFR断片ができないもので、陰性対照二倍体株ではその結果交配するお互いに(上)を補完する。ポジティブコントロールは、ネガティブコントロールの場合と同じアプローチを使用して構築されているが、プラスミドは、一倍体株(P41-ジッパー-リンカー-DHFR F [1,2](P41-ZL-DHFR F [1,2])とP41で変換リンカー-DHFR--zipper F [3](P41-ZL-DHFR F [3]))、底部(DHFR活性の再構成に強力かつ構成的な相互作用をもたらす、相補的DHFR断片に融合した2 GCN4平行ロイシンジッパーフラグメントを含む)。 この図の拡大版をご覧になるにはこちらをクリックしてください。
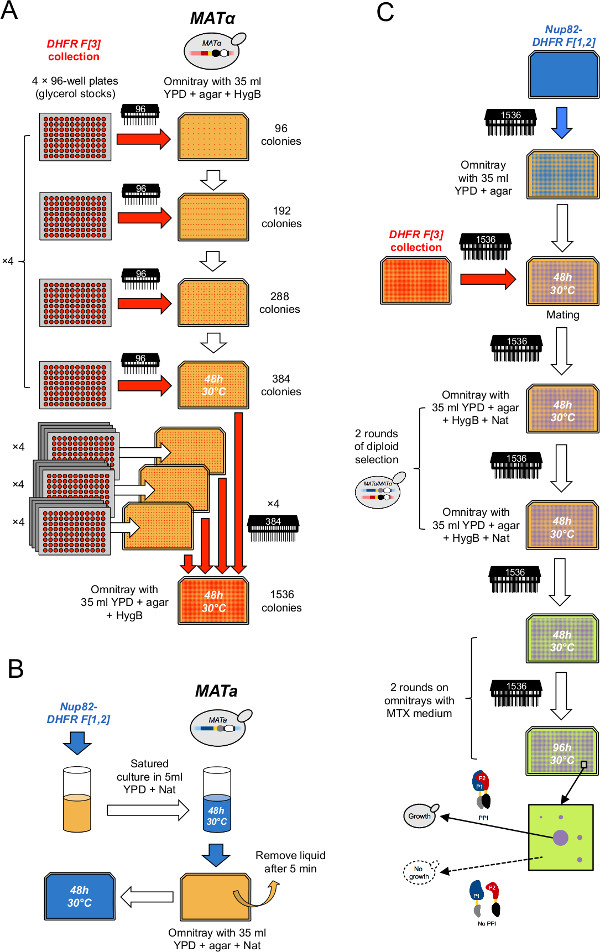
図2:ハイスループットDHFR-PCA手順(Leducq らから適応図33。)(A)MATαDHFR F [3]コレクション。結露の2つの連続ラウンドを1536形式で凝縮されている。まず、グリセロールストックプレート96、ピンツールを使用して、384フォーマットで選択的YPD + HygB媒体上の4つの基によって結合される。第二に、384のアレイ384のピンツールを使用して、1536形式の選択YPD + HygB媒体上の4つの基によって結合される。 のMATa PCAベイト株(B)細胞芝生はベイト株の飽和培養物を増殖させることによって調製されるYPD +ナットomnitray。(C)これらの芝生で培養めっき選択YPD +ナット媒体とは、YPD培地上DHFR F [3]コレクションとベイト株を交配するために使用される。細胞は、連続して二倍体を選択し、二回MTX媒体にPCAを実行するためにYPD + HygB +ナット媒体に二回転送されます。 DHFR断片をベイトおよびプレイタンパク質との間の相互作用の後、お互いを補完した場合、成長は、MTX媒体上に観察される。e.jpg「ターゲット= "_空白">この図の拡大版をご覧になるにはこちらをクリックしてください。
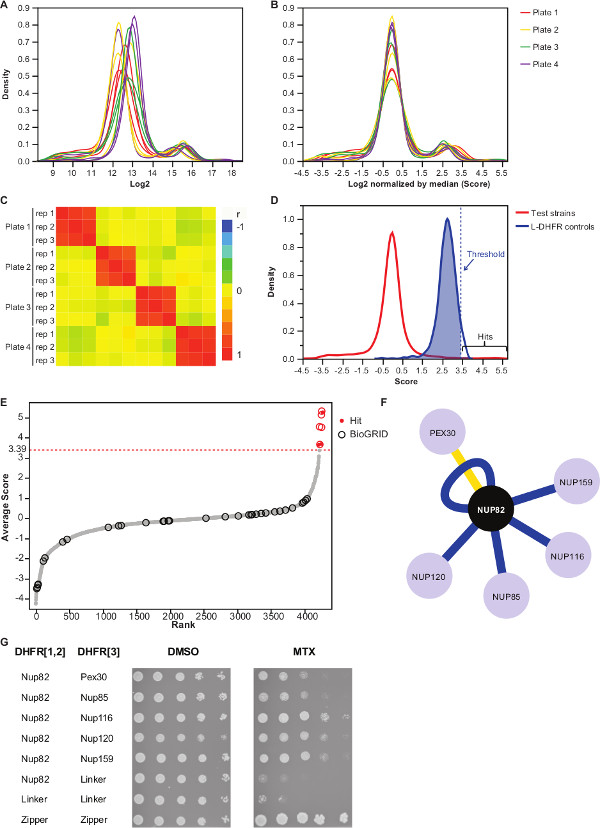
図3:各画像の中央値によるデータは、正規化の工程を経て正の相互作用の有意性閾値と識別の決定を分析する(A)は 、各プレート上のコロニーの大きさの密度分布(2ログ)(B)正規化は、バイアスを補正するプレート間の副作用を伴う。(C)ヒートマップは、手順の再現性を確認し、プレート間のスピアマンの相関係数を示す。テストのPPIのスコアの(D)の分布とL-DHFR F [3]を制御する。ハード閾値は点線の垂直表される([3]の分布の高い信頼性のPPIを識別するためにL-DHFR Fの95 パーセンタイル値に設定することができ各獲物の平均スコアの行)。(E)順位分布。バイオグリッド35 Nup82pの以前に報告され、物理的相互作用パートナーは、円によって識別され、この研究で報告されたものは赤い点で識別されます。 (D)で定義された閾値は点線で示されている。この研究で同定高い組み合わせのPPIを示した(F)ネットワーク。ブルーエッジが以前に報告されたショーの物理的相互作用因子(バイオグリッド35)、イエローエッジがPex30と以前に報告されていない相互作用を示す。(G)Nup82-DHFR F [1,2]と獲物-DHFR F [3を含むPCA二倍体株のスポット希釈アッセイ]ペアは、本研究におけるNup82pの物理的な相互作用因子として同定した。増殖アッセイは、DMSO媒体(MTX溶媒、左パネル)およびMTX培地(右パネル)で行った。 [3]とリンカ-DHFR F [1,2]リンカ-DHFR F - - [1,2] Nup82-DHFR Fからなる負の対照リンカ-DHFR F [3]及びTからなるポジティブコントロール2ロイシンジッパー部分間の彼の強力な相互作用(ジッパー-DHFR F [1,2] - ジッパー-DHFR F [3])が含まれていた。 MTX培地でネガティブコントロールに優れた細胞増殖を、物理的相互作用として解釈されるべきである。 この図の拡大版をご覧になるにはこちらをクリックしてください。
| アミノ酸 | 量(g)に |
| アデニン硫酸塩* | 0.2 |
| L-トリプトファン | 0.4 |
| L-チロシン | 0.3 |
| L-フェニルアラニン | 0.5 |
| Lグルタミン酸(ナトリウム塩) | 1.0 |
| L-アスパラギン | 1.0 |
| Lバリン | 1.5 |
| L-スレオニン | 2.0 |
| Lセリン | 3.75 |
| ウラシル | 0.2 |
| L-ヒスチジン塩酸 | 0.2 |
| L-アルギニン塩酸 | 0.2 |
| L-メチオニン* | 0.2 |
| L-リジン* | 0.2 |
| L-ロイシン | 0.6 |
| *(このプロトコルのように)標準的なPCAを実行する際に廃止が、他の目的のために添加することができる。 | |
表1:MTX培地で10倍-Lys / MET / ADEドロップアウトの構成数量は10倍、ドロップアウトの1リットル用です。アスタリスクは、標準的なMTX媒体として引き出されるべき化合物を示すが、それは、他の目的のために添加することができる。
補足表1:12試験板から合成されたデータは、表1に連結されたDAが含まれていますImageJの解析からのTAは、各行がそれぞれ1536アレイ上の単一の位置に対応するカスタムImageJのスクリプトを使用して実行。また、各行は、画像ファイル、DHFR収集プレート、複製、ORFおよびタンパク質名に関する情報で注釈されている。
補足表2:菌株ごとの平均正規化された成長表2は、標準偏差と一緒に、コレクションの各株についての平均対数2正規化された成長が含まれています。フィルタ処理餌食、ヒットと既知の物理的相互作用因子は、別の列で識別されている。
ディスカッション
我々は、高スループットでの任意のベイトタンパク質のための物理的な相互作用因子の系統的な同定を可能にするDHFR-PCAアッセイに基づくプロトコルを記載している。このプロトコルは、より多くの餌をスクリーニングすることによって適合され、この複製の任意の所望のレベルにすることができる。 Nup82:私たちは、核膜孔複合体に関与ベイトタンパク質のための物理的な相互作用パートナーの同定により、このプロトコルの信頼性を実証する。我々の分析は、酵母タンパク質のインタラクトームを研究する方法の能力を強調し、5以前に報告されたインターアクターと1以前に報告されていない相互作用因子( 図3F&3G)を見つけることができました。
ここで説明するプロトコルは、実験者が注意を払う必要があります先のいくつかの重要なステップを含む。私たちは、餌DHFR Fは[1,2]融合が( 図1B)が正しいことを確認してください)1にお勧めします。これは、建設およびmeasurを配列決定することによって達成することができる抗DHFR F [1,2]または抗DHFR F [3]抗体を用いて、適切なタンパク質発現をる。 2)画面を開始する前に、それが関心のある任意の餌は、PCAの画面で無差別な相互作用を発揮するかどうかを確認することをお勧めします。これは、適切なL-DHFR制御または手動で適切なL-DHFRコントロールで餌を交配し、MTXの培地中での増殖アッセイを実行することによって交差餌とコントロール画面を実行することによって行うことができる。水分が印刷プロセス中に寒天表面上の細胞接着のために最適であるように、それらが使用される前に3)プレート日注がれるべきである。 4)ソースプレートは、先プレートに十分な細胞を転送するための4倍以上を使用すべきではない。目的プレートのコピー数の増加は、膨張の連続工程( - > 16コピー- > 64コピー、例えば 4コピー)することによって行うことができる。あるいは、細胞は、芝生または複製の異なるラウンド間コロニーの異なる位置に撮像することができる。 5)もし、いくつかのpositionsは二倍体の選択(複数可)の後に不足している、ソースプレートは(4.5〜4.7ステップ)嵌合段階で何度も使用されなかったことを確認してください。 6)MTX媒体は右の濃度ですべての必須成分が含まれていることを確認。全く成長がMTX培地上で観察されていない場合には相互作用は、目的のタンパク質についてPCAによって検出されないため、またはMTX媒体が適切に準備されなかったため、実際には、のいずれかであり得る。媒体はDHFRが相補性断片を示す株の増殖を可能にすることを保証するために、構成的な相互作用は、コレクションの空の位置に追加され、そのようなロイシンジッパー部分33( 図1C)に融合DHFR断片陽性対照として使用することができる。リンカ-DHFR断片またはジッパー - リンカー - DHFR断片を用いた並列テストがDESCRとして、偽陽性の相互作用をする傾向があるすべての細胞が成長することを可能にする条件(低MTX濃度またはベイトタンパク質を区別できるようになります全ての株(培地中に欠けているMTX濃度が高すぎるか、または本質的な成分)の増殖を防止するための条件は、以下アイベッド);および例えば、ピンツールは、複製ラウンドおよび/または最後の水との間で適切に滅菌されていない、場合7)PCAを別の媒体から複製の連続ラウンドを介して行われることを考えると、異なるプレート間の株の間で交差汚染が発生することが滅菌手順のバス( すなわちウェットステーション)は、前回の複製ラウンドのコロニーによって汚染されている。アレイ上のいくつかの位置は空であり、従って、全く成長が交差汚染を検出するために観察されないはずである制御位置として使用することができる。
画像解析は、いくつかの公開されたソフトウェア(プロトコルのセクション5を参照)、または任意のカスタムスクリプトを使用して行うことができる。本研究では、カスタムスクリプトは、次の手順を実行します。1)スクリプトが共存するために、各プレートの値をピクセルに空のプレートの画素値を減算し、照明バイアスを修正してください。 2)スクリプトがエッジコロニー上の矩形を重ねることによって決定された各プレートの各1536位置については、)10. 3の画素値のしきい値を使用してバイナリに各バックグラウンド補正画像に変換し、スクリプトはImageJのが「粒子を分析し実行します...円形の選択で」機能。円形の選択は、2つの位置の±10画素間の間隔に等しい半径が設定される。 4)スクリプトは、選択範囲の中心から最も近い粒子を選択し、その場所が離れて選択範囲の中心から2コロニー間の半分以上の間隔でない場合は植民地としてそれを確認する。 5)スクリプトは、バックグラウンド補正画像上の選択された粒子の画素値を測定する。 6)さらに、残りの背景照明の偏りを修正するには、スクリプトがコロニーの画素値に対する粒子の一部ではない円形選択からすべての画素の平均値を減算する。これらの補正画素値の和補足表1の欄「IntDenBackSub」に格納されているのは、コロニーの大きさの尺度として使用される。
解析部内の重要なステップは、有意性の閾値の選択である。ここでは、負のL-DHFR F [3]のコントロールの分布に基づいて閾値を選択したが、画面の目的に応じて、そのような閾値は、厳しすぎるかもしれない。相補的な断片は、自然発生的に互いに補完してもよく、これらは、このようにほとんどのタンパク質の発現を代表するものではないことを確かに、L-DHFR F [3]コントロールが過剰発現される(強いTEFプロモーター)など。これは、L-DHFR Fの分布は、[3]のコントロールは、バックグラウンド増殖(図3D)の平均値よりも高いという事実によって強調される。したがって、バックグラウンド増殖分布の外側に明確に定められているいくつかのこの厳しい閾値を下回るスコアを有する相互作用が、は、例えば、一過性または弱間を表すことが、推定ヒットとして考えることができるアクション。例えば、二つのタンパク質をL-DHFRのコントロールのようなDHFR断片の自発的な相補性を可能にすることができるレベルで発現されていない、場合は、これらをさらに研究し、クロス検証できます。別の方法として、1は偽陽性上の真陽性の割合を最大化するために、バイオグリッド35のようなデータベースで報告された物理的な相互作用因子との重複の割合に基づいて、有意性の閾値を設定することができます。例えば、既知の物理的な相互作用物質の数が十分に高くない場合は、L-DHFR分布の使用とは異なり、この代替は必ずしも可能でないかもしれません。また、有意な閾値の選択は、最終的なデータセットで偽陽性と偽陰性の割合に影響を与える。前述のように、例えば、タンパク質が非常に豊富である場合には確かに、他のPPI検出アッセイのように、偽陽性がDHFR融合タンパク質とのタンパク質の非特異的相互作用から生じ得る。このいくつかのプレイ体系PCA画面内のすべてのベイトタンパク質と相互作用し、従って、分析11( 例えば、TEF2とAde17及び補足表2)から除去する必要があるという事実が挙げられる。 ([3] F [1,2]またはF)自発的なDHFRを呈する餌と獲物を識別するために、この問題は、適切なL-DHFRコントロールに対して2コレクションの制御PCA画面を回避するには、相補性は、特定の条件で行うことができ断片各画面の。与えられた餌の機能が分かっている場合、また、遺伝子オントロジー濃縮解析を行うことは、データの信頼性を高めることができる。一方、DHFR-PCAは、いくつかの理由のために偽陰性を生じさせることができる:1)全てのタンパク質は、これらのタンパク質を不安定化させる場合、または、例えば、それらの局在を変更することができるようにDHFR断片に融合させることができない、とDHFR融合C末端は、局在化シグナルと干渉する。いくつかの細胞区画メートルで2)DHFR再構成例えば、葉酸合成に必須の前駆体が利用できない場合などのy葉酸を生成しない。 DHFR相補11を発生するための8nmの距離内にあるように3)C末端の必要性。それらのC末端は、空間に十分近くに配置されていないこのように、よく知られた相互作用が検出されないことがある。これは、間接的であるほとんどがデータベースに報告Nup82物理的相互作用の大部分は、我々のアッセイでは検出されなかったという事実によってここで例示されている。同様に、C末端が膜に対してトランスであるための膜タンパク質間の相互作用は、DHFRにはつながらない相補性を断片化し、11が検出されることはありません。制限は、1)と3)は、タンパク質のN末端にDHFR断片を融合することにより、比較的簡単に回避することができる。そうするC末端の近くに局在化シグナルを妨害することが防止でき、NおよびC末端のシス相対している膜タンパク質の間の相互作用を検出することを可能にする膜へ。
いくつかの課題は、(2,3で検討)ピンの調査に残る。これまでに製造されたPINのマップは、主にそれぞれの種のための単一の実験条件に記載されて、したがって、タンパク質ネットワークが組織されることがありますどのように単一のスナップショットを提供しています。開発または以下の突然変異を横切って、PINが環境の変化に応じて再編成することができる方法を特定の刺激を見るために、他の実験条件の探索が必要とされている。これらの課題は、生細胞中で、彼らは研究室の大きなコミュニティによって使用できるように現在の技術を適合させることにより、リアルタイムでのPPIを問い合わせるための新しい技術の開発によって克服される。 DHFR相補複合体27の量の変化を検出することができる定量的な技術として、DHFR-PCAは、これらの課題を克服するために適合させることができるとのPPIは、DNA損傷剤22によってどのように影響されるかを研究するために使用されている、化学剤25、遺伝子欠失23,26または他の酵母種およびそれらの混成33。これらの新しい次元を探るPINの動的を明らかにすることがますます重要になるであろう。
開示事項
この記事のオープンアクセス出版料の一部は、S&Pロボティクスによって支払われた。
謝辞
この作品は、ヘルスリサーチ(CIHR)のカナダの研究所によってサポートされていました自然科学とカナダのディスカバリーの助成金とCRLへのヒューマン·フロンティア·サイエンス·プログラムの助成金の工学研究評議会、191597、299432と324265を付与します。 CRLはCIHR新しい研究者である。ギヨームディスはテオのフェローシップでサポートされています。サミュエル·ロシェットはNSERCとFRQNTフェローシップでサポートされています。
資料
| Name | Company | Catalog Number | Comments |
| BioMatrix Robot, Bench-top Configuration | S&P Robotics Inc. | BM5-BC | |
| 96-format Pin-tool | S&P Robotics Inc. | PH-96-10 | Standard 96-format Pin-tool with 96 high-precision floating pins |
| 384-format Pin-tool | S&P Robotics Inc. | PH-384-10 | Standard 384-format Pin-tool with 384 high-precision floating pins |
| 1536-format Pin-tool | S&P Robotics Inc. | PH-1536-05 | Custom 1536-format Pin-tool with 0.5mm high-precision floating pins |
| Automated imaging module | S&P Robotics Inc. | IMG-02 | |
| Methotrexate | Bioshop Canada Inc. | MTX440 | CAUTION: toxic compound |
| Hygromycin B | Bioshop Canada Inc. | HYG003 | |
| Nourseothricin dihydrogen sulfate | Werner BioAgents | 5010000 | |
| Yeast-Interactome Collection | Thermo Scientific | YSC5849 | |
| Omni Tray w/lid sterile | Thermo Scientific | 242811 | |
| Anti-DHFR F[1,2] antibody | Sigma-Aldrich | D1067 | |
| Anti-DHFR F[3] antibody | Sigma-Aldrich | D0942 |
参考文献
- Alberts, B. The cell as a collection of protein machines: preparing the next generation of molecular biologists. Cell. 92, 291-294 (1998).
- Diss, G., et al. Integrative avenues for exploring the dynamics and evolution of protein interaction networks. Curr Opin Biotechnol. 24, 775-783 (2013).
- Vidal, M., Cusick, M. E., Barabasi, A. L. Interactome networks and human disease. Cell. 144, 986-998 (2011).
- Hu, P., et al. Global functional atlas of Escherichia coli encompassing previously uncharacterized proteins. PLoS biology. 7, e96 (2009).
- Arifuzzaman, M., et al. Large-scale identification of protein-protein interaction of Escherichia coli K-12. Genome Res. 16, 686-691 (2006).
- Rajagopala, S. V., et al. The binary protein-protein interaction landscape of Escherichia coli. Nature biotechnology. 32, 285-290 (2014).
- Arabidopsis-Interactome-Mapping-Consortium. Evidence for network evolution in an Arabidopsis interactome map. Science. 333, 601-607 (2011).
- Babu, M., et al. Interaction landscape of membrane-protein complexes in Saccharomyces cerevisiae. Nature. 489, 585-589 (2012).
- Krogan, N. J., et al. Global landscape of protein complexes in the yeast Saccharomyces cerevisiae. Nature. 440, 637-643 (2006).
- Gavin, A. C., et al. Functional organization of the yeast proteome by systematic analysis of protein complexes. Nature. 415, 141-147 (2002).
- Tarassov, K., et al. An in vivo map of the yeast protein interactome. Science. 320, 1465-1470 (2008).
- Uetz, P., et al. A comprehensive analysis of protein-protein interactions in Saccharomyces cerevisiae. Nature. 403, 623-627 (2000).
- Guruharsha, K. G., et al. A protein complex network of Drosophila melanogaster. Cell. 147, 690-703 (2011).
- Li, S., et al. A map of the interactome network of the metazoan C. elegans. Science. 303, 540-543 (2004).
- Rual, J. F., et al. Towards a proteome-scale map of the human protein-protein interaction network. Nature. 437, 1173-1178 (2005).
- Landry, C. R., Levy, E. D., Abd Rabbo, D., Tarassov, K., Michnick, S. W. Extracting insight from noisy cellular networks. Cell. 155, 983-989 (2013).
- Berggard, T., Linse, S., James, P. Methods for the detection and analysis of protein-protein interactions. Proteomics. 7, 2833-2842 (2007).
- Wodak, S. J., Vlasblom, J., Turinsky, A. L., Pu, S. Protein-protein interaction networks: the puzzling riches. Current opinion in structural biology. 23, 941-953 (2013).
- Fields, S., Song, O. A novel genetic system to detect protein-protein interactions. Nature. 340, 245-246 (1989).
- Dunham, W. H., Mullin, M., Gingras, A. C. Affinity-purification coupled to mass spectrometry: basic principles and strategies. Proteomics. 12, 1576-1590 (2012).
- Michnick, S. W., Ear, P. H., Landry, C., Malleshaiah, M. K., Messier, V. A toolkit of protein-fragment complementation assays for studying and dissecting large-scale and dynamic protein-protein interactions in living cells. Methods Enzymol. 470, 335-368 (2010).
- Rochette, S., Gagnon-Arsenault, I., Diss, G., Landry, C. R. Modulation of the yeast protein interactome in response to DNA damage. Journal of proteomics. 100, 25-36 (2014).
- Diss, G., Dube, A. K., Boutin, J., Gagnon-Arsenault, I., Landry, C. R. A systematic approach for the genetic dissection of protein complexes in living cells. Cell Rep. 3, 2155-2167 (2013).
- Gagnon-Arsenault, I., et al. Transcriptional divergence plays a role in the rewiring of protein interaction networks after gene duplication. Journal of proteomics. 81, 112-125 (2013).
- Schlecht, U., Miranda, M., Suresh, S., Davis, R. W., St Onge, R. P. Multiplex assay for condition-dependent changes in protein-protein interactions. Proceedings of the National Academy of Sciences of the United States of America. 109, 9213-9218 (2012).
- Lev, I., et al. Reverse PCA, a systematic approach for identifying genes important for the physical interaction between protein pairs. PLoS Genet. 9, e1003838 (2013).
- Freschi, L., Torres-Quiroz, F., Dube, A. K., Landry, C. R. qPCA: a scalable assay to measure the perturbation of protein-protein interactions in living cells. Mol Biosyst. 9, 36-43 (2013).
- Pelletier, J. N., Campbell-Valois, F. X., Michnick, S. W. Oligomerization domain-directed reassembly of active dihydrofolate reductase from rationally designed fragments. Proceedings of the National Academy of Sciences of the United States of America. 95, 12141-12146 (1998).
- Gietz, R. D., Woods, R. A. Transformation of yeast by lithium acetate/single-stranded carrier DNA/polyethylene glycol method. Methods Enzymol. 350, 87-96 (2002).
- Schuldiner, M., Collins, S. R., Weissman, J. S., Krogan, N. J. Quantitative genetic analysis in Saccharomyces cerevisiae using epistatic miniarray profiles (E-MAPs) and its application to chromatin functions. Methods. 40, 344-352 (2006).
- Schneider, C. A., Rasband, W. S., Eliceiri, K. W. NIH Image to ImageJ: 25 years of image analysis. Nature methods. 9, 671-675 (2012).
- Wagih, O., Parts, L. gitter: A Robust and Accurate Method for Quantification of Colony Sizes From Plate Images. G3 (Bethesda). 4 (3), 547-552 (2014).
- Leducq, J. B., et al. Evidence for the robustness of protein complexes to inter-species hybridization. PLoS Genet. 8, e1003161 (2012).
- . . Development Core Team: A language and environment for statistical computing. , (2008).
- Stark, C., et al. BioGRID: a general repository for interaction datasets. Nucleic Acids Res. 34, D535-D539 (2006).
- Vinayagam, A., et al. Protein complex-based analysis framework for high-throughput data sets. Science signaling. 6, rs5 (2013).
- Jansen, R., Gerstein, M. Analyzing protein function on a genomic scale: the importance of gold-standard positives and negatives for network prediction. Current opinion in microbiology. 7, 535-545 (2004).
- Ito, T., et al. A comprehensive two-hybrid analysis to explore the yeast protein interactome. Proceedings of the National Academy of Sciences of the United States of America. 98, 4569-4574 (2001).
- Costanzo, M., et al. The genetic landscape of a cell. Science. 327, 425-431 (2010).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved