Method Article
L'échelle du génome interaction protéine-protéine Le dépistage par Protein-fragment complémentation Assay (PCA) dans les cellules vivantes
Dans cet article
Résumé
Proteins interact with each other and these interactions determine in a large part their functions. Protein interaction partners can be identified at high-throughput in vivo using a yeast fitness assay based on the dihydrofolate reductase protein-fragment complementation assay (DHFR-PCA).
Résumé
Proteins are the building blocks, effectors and signal mediators of cellular processes. A protein’s function, regulation and localization often depend on its interactions with other proteins. Here, we describe a protocol for the yeast protein-fragment complementation assay (PCA), a powerful method to detect direct and proximal associations between proteins in living cells. The interaction between two proteins, each fused to a dihydrofolate reductase (DHFR) protein fragment, translates into growth of yeast strains in presence of the drug methotrexate (MTX). Differential fitness, resulting from different amounts of reconstituted DHFR enzyme, can be quantified on high-density colony arrays, allowing to differentiate interacting from non-interacting bait-prey pairs. The high-throughput protocol presented here is performed using a robotic platform that parallelizes mating of bait and prey strains carrying complementary DHFR-fragment fusion proteins and the survival assay on MTX. This protocol allows to systematically test for thousands of protein-protein interactions (PPIs) involving bait proteins of interest and offers several advantages over other PPI detection assays, including the study of proteins expressed from their endogenous promoters without the need for modifying protein localization and for the assembly of complex reporter constructs.
Introduction
réseaux d'interactions protéiques (NIP) offrent une faible carte de la résolution de la façon dont les protéines sont fonctionnellement organisés dans la cellule 1. Chaque connexion physique entre deux protéines, ou de l'interaction protéine-protéine (PPI), peuvent représenter une liaison qui est stable dans le temps, tels que ceux trouvés dans des complexes de protéines et qui contribuent à l'organisation structurelle de la cellule. Ces connexions peuvent également représenter les associations transitoires qui régulent l'activité, la stabilité, la localisation et les interactions des deux partenaires. Identifier les partenaires d'interaction physiques d'une protéine donnée fournit donc des informations riches sur la fonction et la régulation de cette protéine 2,3. Pour ces raisons, de gros efforts ont été mis à la cartographie des codes PIN dans des organismes modèles, y compris Escherichia coli 4-6, Arabidopsis thaliana 7, Saccharomyces cerevisiae 12.8, Drosophila melanogaster </ Em> 13, Caenorhabditis elegans 14 et 15 Homo Sapiens. Ces études ont fourni des informations importantes sur la façon dont les protéines sont organisés dans la cellule et de l'information ainsi clé sur les protéines avec des fonctions jusque-là inconnues.
Plusieurs stratégies ont été développées au cours des années à étudier NIP. Ces technologies peuvent être regroupés en trois catégories en fonction de la nature des informations qu'ils fournissent sur les IPP (revue dans 16-18). La première est basée sur deux hybrides de levure et 19 ses dérivés. Ces technologies fournissent des informations sur l'association directe entre des paires de protéines, ce qui permet la construction de réseaux binaires. La seconde famille est basée sur la purification par affinité de protéines d'appât et l'identification de leurs partenaires associés, tels que la purification par affinité suivie par spectrométrie de masse 20. Ces approches identifier des groupes de protéines qui sont directementou indirectement associée, généralement d'une manière stable, et sont extrêmement puissants pour identifier des complexes de protéines. La troisième approche est basée sur des essais de complémentation protéines fragment (APC) 11,21. Cette approche fournit un niveau de résolution intermédiaire entre les deux approches anciennes, car elle permet de détecter des liens directs entre les protéines et proximale. Chaque technique a ses propres forces et faiblesses, comme l'a récemment examiné 18.
Le code PIN eucaryote mieux décrit est de loin celui de la levure bourgeonnante Saccharomyces cerevisiae, en partie parce que son protéome est relativement moins complexes que ceux des autres eucaryotes modèles et parce que des essais à haut débit pour détecter les IPP ont d'abord été testés et sont plus efficacement mis en œuvre dans cet organisme modèle 9-12. Un procédé particulièrement efficace pour le système de levure est la dihydrofolate réductase protéine-fragment complémentation dosage (DHFR-PCA), un essai qui a étéutilisé dans différents contextes d'étudier le code PIN de levure dans des conditions standard et perturbés 11,22-26. Cette méthode repose sur une analyse de survie qui permet la détection des IPP directs et quasi-directs pour une protéine appât donnée à ces deux niveaux d'expression endogènes et localisations subcellulaires indigènes des partenaires d'interaction 11,21 de manière quantitative 27. Le signal obtenu en utilisant ce dosage (ce est à dire de la taille des colonies sur les tableaux de colonies de haute densité) reflète ainsi la quantité de complexes protéiques formés entre l'appât et proie dans un environnement cellulaire presque équivalente à celle des cellules de type sauvage. Le dosage est basé sur la reconstitution d'une enzyme rapporteur impliqué dans le métabolisme des folates, le dihydrofolate reductase (DHFR), de sorte que deux fragments complémentaires de la DHFR qui sont fusionnés aux deux protéines d'intérêt sont mis en proximité lorsque les deux protéines interagissent, ce qui à son tour, conduit à la reconstitution de l'activité réversible de l'enzyme 1Et une croissance de la souche sur un milieu contenant du methotrexate (MTX; Figure 1). Ce composé inhibe l'enzyme DHFR endogène, mais pas celui muté utilisé dans l'essai 28. Deux collections de souches CPA, contenant une ~ 4300 souches MATa avec une ORF fusionné à la DHFR F [1,2] et un fragment contenant ~ 4800 MAT α souches avec un ORF fusionné à la DHFR [3] fragment, peuvent être achetés à mettre en œuvre DHFR-PCA à petite ou grande échelle dans tout laboratoire. Ici, nous décrivons un protocole général mais détaillée à l'écran pour les IPP entre une protéine appât et ~ 4800 protéines proies à l'aide de ce test.
Protocole
1. Construction / vérification de Bait souches
- Si la souche de l'appât d'intérêt est disponible dans le MATa DHFR F [1,2] collecte, récupérer de la collection comme décrit dans l'étape 1.1.1, sinon construire la souche comme décrit à l'étape 1.1.2.
REMARQUE: Le protocole décrit ici utilise une DHFR F [1,2] souche comme un appât et l'DHFR F [3] collection que proies, que cette collection contient plusieurs souches que l'DHFR F [1,2] collection. Cependant, il est possible d'effectuer l'écran dans l'autre sens si la souche de l'appât ne est disponible que dans le DHFR F [3] collecte ou dans les deux orientations si l'on exige une couverture plus élevé de l'interactome.- Décongeler le stock de glycérol plaque qui contient la souche d'appât sur la glace pendant une heure. Stériliser la feuille d'aluminium recouvrant la plaque en utilisant 95% d'éthanol. Pierce la feuille avec un embout stérile, une pipette de haut en bas pour remettre en suspension les cellules et série 2-3 ul de glycérol stock sélective peptone extrait de levuredextrose (YPD) + 100 pg / ml nourséothricine (Nat) afin d'isoler des colonies isolées. Incuber pendant deux jours à 30 ° C.
- Construction d'une souche appât PCA (MATa DHFR F [1,2]).
- Utilisation de haute fidélité polymérase et un protocole de PCR standard, amplifier la DHFR F [1,2] cassette du plasmide pAG25-lieur-DHFR F [1,2] -ADHterm utilisant des oligonucléotides avec en surplomb se termine homologue à la 40 dernières pb de l'ORF extrémité 3 'à l'exclusion du codon d'arrêt (amorce avant) et au 40 premières pb de la 3'-UTR du gène (amorce inverse) (figure 1A).
- Transformer le produit de PCR dans des cellules de levure compétentes (habituellement dans une souche BY4741) en utilisant LiOAc / PEG protocole de transformation de levure selon la norme 29 (figure 1A).
- Plate sur YPD + milieu sélectif Nat pour isoler des transformants positifs.
- Effectuer une colonie PCR de diagnostic sur des colonies isolées de confirmer le bon DHFR F [1,2] fusion. Utilisez prIMERS recuit 1) dans la séquence du gène codant (Forward oligo) environ 100 pb en amont de la fusion DHFR et 2) dans le terminateur ADH de la cassette (oligo inverse) (figure 1B).
- Séquencer le produit de PCR par séquençage Sanger pour confirmer gène de fusion approprié.
- Archiver la souche d'appât confirmé dans 25% de glycerol à -80 ° C.
NOTE: Le protocole peut être suspendu à cette étape.
2. Pin-outils de stérilisation et d'impression Procédures
NOTE: La procédure de stérilisation décrit ci-dessous a été optimisé pour les pin-outils manipulés par le BM3-BC (S & P Robotics) plate-forme robotique, mais peut être adapté à d'autres plateformes. Cette section décrit les procédures de stérilisation et d'impression Pin-outils qui sont utilisés pour transférer des cellules d'un milieu à un autre pour le reste du protocole. Son exécution en interne utilisés pour effectuer ces routines peuvent être obtenus sur demande. Remarque that toutes les mesures peuvent être effectuées sans la nécessité d'une plate-forme robotique en utilisant une pin-outil manuel 30.
- Montez la broche-outil approprié sur la plate-forme robotique.
- Préparer nettoyage et stations humides comme suit:
- Ajouter 500 ml d'eau stérile dans la station de bain d'eau.
- Ajouter 320 ml d'eau stérile dans la station de brosse.
- Ajouter 380 ml d'éthanol à 70% dans l'appareil à ultrasons lors de la réplication de plaque de gélose (86 x 128 mm OmniTray, contenant 35 ml de milieu solidifié) de plaque de gélose, ou 400 ml lors de la réplication à partir d'une plaque de microtitrage contenant des cultures liquides à une plaque de gélose.
- Ajouter 35 ml d'eau stérile dans la station humide (composé d'un OmniTray vide stérile).
- Au début de chaque journée qui nécessite la plate-forme robotique, stériliser les outils pin-cinq fois pendant une minute dans le bain de sonication. Pendant ce temps, mettez la lampe UV pendant cinq minutes pour stériliser l'enceinte du robot.
NOTE: Ce ne est pas nécessaire si le robOT est logé sous une hotte stérile. - Entre chaque broche-outil autour de réplication, stériliser la broche-outil comme suit:
- Faire tremper la broche-outil cinq fois pendant 10 secondes dans la station de bain de l'eau pour enlever amas de cellules.
- Faire tremper la broche-outil à deux reprises avant et en arrière dans la station de brosse.
NOTE: La brosse rotative sera éliminer les cellules résiduelles. - Faire tremper la broche-outil à deux reprises pendant 20 secondes dans la station de traitement ultrasonique.
REMARQUE: les cellules restant sur les broches sera supprimé par sonication ou tué par de l'éthanol. - Assurez-vous que la profondeur des broches de trempage augmente à chaque bain successive pour vous assurer une bonne stérilisation.
- Séchez la broche-outil dans la station de séchage d'air pendant 25 s.
- Avant de prendre des cellules sur les plaques de source, mouiller les épingles dans la station humide, qui contient 35 ml d'eau stérile dans un OmniTray.
- Plonger les repères de deux fois dans les colonies de la plaque d'origine.
- Imprimer sur une plaque de destination fraîchement coulé en touchant l'agar surface de deux fois (ci-après dénommé l'action de "l'impression" un tableau).
3. La condensation de la DHFR F [3] Collection en 1536 deux tableaux en utilisant une plate-forme robotique automatisé
- Décongélation sur de la glace la DHFR F [3] collection (plaques 60 à 96 puits) et une plaque de 96 puits supplémentaire remplie de la DHFR L-F [3] souche témoin (figure 1C), qui contient le DHFR F [3] fragment et l'agent de liaison en amont exprimé seul comme témoin négatif.
NOTE: En principe, ce fragment ne devrait pas interagir avec ne importe quelle protéine de fusion DHFR fragment (voir la discussion pour plus de détails). - Plaques centrifugeuse (tour rapide) avant d'enlever la feuille d'aluminium pour éviter les risques de contamination croisée entre les puits.
- Condenser la collecte sur 16 tableaux de 384 souches (figure 2A). Pour ce faire, pour chaque 384 tableau, quatre plaques d'impression de glycérol sur les quatre quadrants d'un YPD sélective + 250 pg / ml d'hygromycine B (hygB) OmniTray utilisant ee 96 pin-outil (ici, un réseau 384 peut être subdivisé en quatre quadrants également espacées, composées chacune de 96 positions dans une matrice 2 x schéma 2). Insérez quatre plaques à 96 puits contenant le [3] contrôle de la L-DHFR F négative entre les 60 autres plaques afin d'avoir un ensemble final de 64 plaques qui remplissent exactement quatre tableaux 1536. Stériliser la broche-outil entre chaque cycle de réplication comme décrit à l'étape 2.
REMARQUE: Insérez le L-DHFR F [3] plaques afin d'avoir une telle plaque sur chacun des quatre derniers 1536 tableaux. - Incuber les plaques pendant deux jours à 30 ° C. NOTE: A ce stade, la collection DHFR peut être stocké dans un format 384 à 4 ° C pendant un mois sur les plaques d'agar.
- Condenser la collection en quatre tableaux de 1536 souches (figure 2A). Pour ce faire, pour chaque 1536 tableau, imprimer quatre tableaux de 384 souches sur les quatre quadrants d'une plaque du même milieu que dans l'étape 3.2 en utilisant la broche-outil 384 (ici, une gamme 1536 peut être divisée en quatre eQually espacées quadrants constitués chacun de 384 positions dans une matrice 2 x schéma 2).
- Incuber les plaques pendant deux jours à 30 ° C. NOTE: A ce stade, la collection DHFR peut être stocké dans un format 1 536 à 4 ° C pendant un mois sur les plaques d'agar.
- Répliquer les quatre tableaux sur le même support de normaliser la taille des colonies à l'aide d'une broche-outil 1536.
- Incuber les plaques pendant deux jours à 30 ° C.
4. Procédure DHFR-PCA à haut débit
- Inoculer une culture de la souche d'appât (DHFR F [1,2] de fusion) obtenu à l'étape 1.1.1 ou 1.1.2 dans 20 ml de YPD + Nat liquide dans un tube de 50 ml (figure 2B).
- Incuber pendant deux jours à 30 ° C sous agitation à 250 tours par minute pour permettre à la culture d'atteindre la saturation.
- Après deux jours d'incubation, la plaque 5 ml de la culture sur un YPD + Nat OmniTray. Laissez cellules adsorbées à la surface pendant 5-10 minutes et enlever l'excès de liquide (figure 2B). Répétez twice faire trois répétitions.
- Incuber pendant deux jours à 30 ° C.
- Imprimer la souche de l'appât des étapes 4,3 et 4,4 sur 12 plaques de YPD (assez pour se accoupler quatre plaques de la DHFR F [3] collecte × trois répétitions) avec une broche-outil 1536 l'utilisation de chaque tapis cellulaire ne est plus de quatre fois.
- Imprimer le tableau approprié de la DHFR F [3] collection sur le dessus des cellules d'appât à l'aide du 1536 pin-outil (figure 2C).
- Laissez les souches se accouplent par incubation pendant deux jours à 30 ° C.
- Sélectionner des cellules diploïdes en imprimant des colonies sur milieu YPD contenant omnitrays + + Nat hygB (figure 2C).
- Incuber pendant deux jours à 30 ° C.
- Répétez sélection diploïde tel que décrit dans les étapes 4.8 et 4.9 (figure 2B).
- Préparer des plaques avec les médias contenant MTX (milieu MTX) un jour avant l'utilisation suivant ces étapes 21 (ATTENTION:. Être prudent lorsqu'on travaille avec le MTX que ce est un composé toxique Toujours porter des gants, proteverres ction et une blouse de laboratoire lors de la manipulation) (figure 2C):
- Préparer les 10x Lys / MET / ade acide aminé abandon dans l'eau déminéralisée. Filtre-stériliser l'abandon dans une bouteille stérile en utilisant un filtre à seringue stérile de 0,2 um ou, le cas échéant en grandes quantités, un entonnoir de filtration supérieure à 0,2 um de bouteille (tableau 1).
- Préparer une solution stock MTX ml 10 mg / dans le diméthylsulfoxyde (DMSO). Utiliser immédiatement après préparation de la solution et de geler le reste à -20 ° C. Protéger de la lumière comme il est photosensible. Ne pas congeler MTX nouveau après décongélation.
- Préparer le support de la manière suivante (les ingrédients et les quantités moyennes sont les mêmes que ceux utilisés dans Tarassov 11 et al.):
- Pour un litre de milieu, mélanger dans deux flacons distincts: 1) 6,69 g de base azotée de levure sans acides aminés et sans sulfate d'ammonium et 330 ml d'eau déminéralisée; 2) 25 g de ml nobles agar et 500 d'eau déminéralisée.
- Flacons autoclave à 121 ° C pendant 20 min.
- Équilibrer la température dans un bain-marie à 55 ° C pendant au moins une heure.
- Mélanger les deux flacons et ajouter 50 ml de 40% de glucose stérile, 100 ml de 10x stérile abandon, et 20 ml de MTX 10 mg / ml.
- Verser 35 ml (voir note ci-dessous) de la moyenne omnitrays. Laisser se solidifier pendant au moins une heure et demie. plaques de blindage de la lumière.
REMARQUE: Ici, verser 35 ml de milieu par OmniTray est essentielle pour assurer épaisseur de la plaque égale, ce qui est important pour toutes les étapes en aval.
- plaques d'image de la deuxième ronde de sélection diploïde avec la plate-forme robotique ou avec un appareil photo numérique régulière avec éclairage de plaque uniforme. Utilisez ces photos pour identifier les positions vides sur les tableaux lors de l'exécution de l'analyse en aval. Assurez-vous que les paramètres de la caméra sont toujours les mêmes et que la lumière de robot est mis en marche.
- Imprimer cellules diploïdes sur MTX usi médiatiqueng la broche-outil 1536.
- Incuber pendant quatre jours à 30 ° C dans des sacs en plastique pour éviter le dessèchement.
- Préparer un deuxième lot de omnitrays contenant du milieu MTX comme décrit à l'étape 4.11.
- Après quatre jours d'incubation, les plaques d'image en utilisant la plate-forme robotique ou appareil photo numérique régulière. Assurez-vous que les paramètres de la caméra sont toujours les mêmes et que la lumière de robot est mis en marche.
- Effectuer un deuxième tour de sélection MTX en reproduisant les cellules sur le deuxième lot de MTX médias.
NOTE: Cela réduira la croissance de fond des souches APC et augmenter la résolution quantitative. - Incuber pendant quatre jours à 30 ° C dans des sacs en plastique pour éviter le dessèchement.
- plaques d'image comme décrit dans l'étape 4.16.
Analyse 5. Image
- Analyser des images de tableaux colonies avec ImageJ personnalisé 31 scripts ou en utilisant des logiciels tels que publiés colonisateur, Ht grille de colonie analyseur, Cell profileur, Colony imager, ScreenMill, YeastXtract et Gitter (compilé en 32). L'analyse d'image devrait sortie une ou plusieurs feuilles de calcul contenant la taille des colonies pour chaque position de chaque tableau, utiliser ces formats de colonies pour toutes les analyses en aval.
NOTE: Dans cette étude, nous avons utilisé un script ImageJ personnalisé décrit dans Leducq et al 33 (voir la section de discussion pour plus de détails)..
6. Analyse des données
REMARQUE: Les résultats de l'analyse d'image peuvent être traités dans une tabulatrice comme Excel ou en utilisant un langage de script tels que R 34. Les étapes suivantes décrivent la procédure en utilisant une coutume ImageJ 31 script.
- Utilisation d'un script personnalisé, concaténer des fichiers de sortie de l'analyse d'image et d'annoter chaque rangée avec la plaque et filtrer l'information que dans le tableau supplémentaire 1.
- Log 2 transformer les valeurs de taille de la colonie (densité intégré ou de la zone de la colonie; ici, la colonne "IntDenBackSub & #8221; du tableau supplémentaire 1 a été utilisé).
REMARQUE: la distribution de la valeur se penchera comme dans la figure 3A. - Normaliser les valeurs en soustrayant la valeur médiane de chaque plaque.
NOTE: Ce contrôles étape pour le biais de la plaque pouvant résulter de la quantité de médias inégale ou variation de l'acquisition automatique de l'image, et réduit la variance inter-répliquer (figure 3B). - Vérifiez que répétitions en corrélation avec l'autre (figure 3C) pour évaluer la reproductibilité des expériences.
- Pour différencier l'interaction de paires non-interaction appât-proie, fixer un seuil de haute confiance correspondant aux 95 e percentile de la distribution de la L-DHFR F [3] contrôles.
NOTE: Dans cette expérience, ce qui correspond à 3,39 (Figure 3D). En variante, un seuil sur la base du chevauchement avec interacteurs physiques connus tels que ceux rapportés dans la BioGRID 35 peut être utilisé.Voir la discussion pour plus de détails. - Pour tout appât, filtre proies identifié comme impliquées dans les interactions faux positifs dans les écrans DHFR-APC (voir la discussion pour plus de détails) et énumérés dans le tableau complémentaire 2 (identifié comme "1" dans la colonne "filtrée").
- La moyenne des LOG2 taille des colonies normalisée des trois répétitions de chaque interaction (colonne "Note moyenne" dans le tableau complémentaire 2).
7. Validation des Interactiens physique à l'aide expériences à petite échelle
REMARQUE: Tous les PPI d'un intérêt particulier ayant un score supérieur ou proche du seuil appliqué peut être validée en utilisant le test DHFR-PCA dans un modèle expérimental à petite échelle en utilisant un dosage de croissance sur milieu de MTX solide ou liquide. Les étapes suivantes montrent la procédure pour construire manuellement des souches de l'APC diploïdes et effectuer des essais sur place sur un milieu MTX. L'expérimentateur doit effectuer ces étapes pour tous nécessacontrôles y (Bait-DHFR F [1,2] x L-DHFR F [3], Zipper-lieur-DHFR souches diploïdes et linker-DHFR souche diploïde).
- Plate 2-3 ul du stock de glycérol de la souche d'appât généré en 1.1.2.6), le L-DHFR F [3] souche témoin, le Zipper-lieur-DHFR et linker-DHFR souches diploïdes sur YPD + Nat, YPD + hygB et deux fois YPD + Nat + hygB médias, respectivement.
- Récupérer la proie d'intérêts dans le DHFR F [3] collecte et suivez les instructions à l'étape 1.1.1, mais la souche série sur le milieu YPD + hygB place de milieu YPD + Nat.
- Effectuer un diagnostic par PCR comme dans 1.1.2.4 pour confirmer la DHFR F [3] la fusion au niveau du locus de proie et la séquence du produit.
- Inoculer 1 ml de milieu YPD liquide avec les souches haploïdes de se accoupler (Bait x Prey, Bait x L-DHFR F [3] le contrôle) et de croître au moins deux jours à 30 ° C pour permettre les diploïdes pour former.
- Sélectionnez diploïdes en stries 4-5 ul de la culture en milieu solide 7,4 sur YPD + hygB + Nat. Cultiver deux jours à 30 ° C.
- Select d'une colonie isolée et croître pendant une nuit dans une culture liquide (1 ml) pour réaliser le dosage de croissance.
- Préparer plaques MTX et DMSO (mêmes Ingrédients que le milieu MTX, mais sans MTX) un jour avant l'utilisation. Voir l'étape 4.11 pour plus de détails.
- Effectuer test de croissance en déposant des dilutions en série des cultures différentes sur la commande (DMSO) et des plaques de sélection (MTX).
- Diluer précultures à DO = 1.
- Effectuer des dilutions quintuples (jusqu'à un facteur de dilution de 625) dans une plaque de 96 puits stérile.
- Coin 4 ul de chaque dilution sur le support PCA (DMSO et MTX).
- Incuber à 30 ° C dans des sacs en plastique pour éviter le dessèchement.
- plaques d'image de jour 1-7 d'incubation à l'aide de la plate-forme robotique ou un appareil photo numérique régulière.
Résultats
Tableau supplémentaire 2 est un exemple de représentation des résultats obtenus en utilisant la protéine de levure fusionné à la Nup82 DHFR F [1,2] fragment comme un appât. Le seuil défini avec le L-DHFR F [3] contrôles peut être utilisé comme un seuil empirique pour déterminer coups de confiance élevé (figures 3D et 3E). Sinon, le classement de score peut être utilisé pour effectuer des enrichissements Gene Ontology ou autre analyse fonctionnelle 36 basé sur les normes d'or 37. Les interacteurs physiques connues de l'appât peuvent être récupérés à partir de bases de données comme BioGRID 35 et superposées sur les données (figures 3E et 3F). Dans cet exemple, cinq des huit coups haute de confiance ont été précédemment rapporté que interacteurs Nup82 et deux font partie de la sous-complexe Nup82, Nup116 et Nup159 (Figure 3F et 3G). L'autre membre du complexe, NSP1, ne montre aucune Interaction dans notre expérience. Deux proies, Ade17 et Tef2 (non représenté dans la figure 3F), avaient des scores supérieurs au seuil dure appliquée, mais ceux-ci sont susceptibles d'être de faux positifs car ils interagissent avec presque ne importe quelle protéine appât dans les écrans de l'APC nous avons effectuées (résultats non publiés). D'autre part, Pex30 peut représenter un interacteur physique roman Nup82 et nous avons été en mesure de confirmer cette interaction en utilisant DHFR-PCA à bas-débit (figure 3G). Pex30 est une protéine de membrane peroxysomale et quelques interactions directes ont été signalées entre le complexe du pore nucléaire (NPC) et cet organite. Un écran double-hybride identifié deux autres protéines PNJ, Nup53 et Asm4 (Nup59), que interacteurs physiques de Pex30 38, et une interaction génétique entre Pex30 et Nup170 a été rapporté 39. Deux autres partenaires d'interaction détectée, Nup120 et Nup85 (Figure 3F et 3G), ne font pas partie de la sous-Nup82 complexe, illustrant la capacitéde la DHFR-PCA pour détecter les interactions au sein et entre Subcomplexes dans 11 grands complexes.
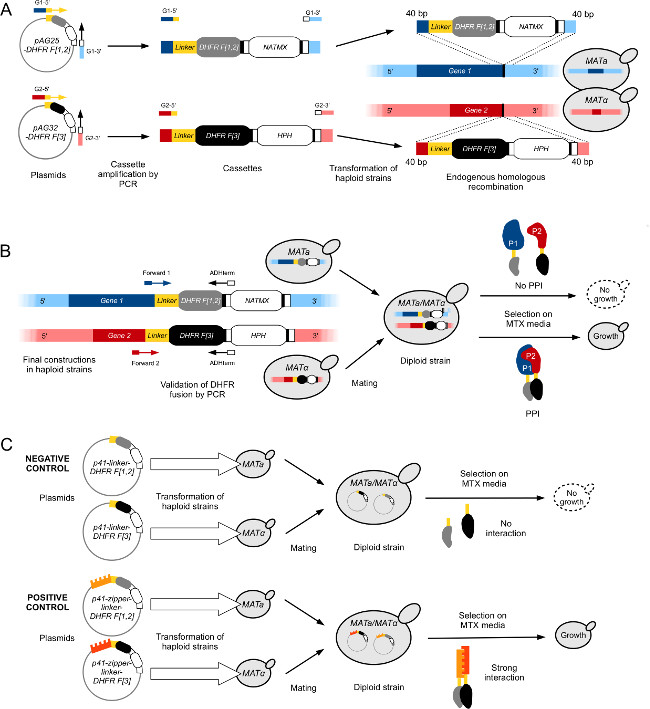
Figure 1:. Souches de levure d'ingénierie pour à haut débit DHFR-PCA (. Figure adaptée de Leducq et al 2012 33) (A) Construction de souches Mata et MAT α haploïdes de fusionner Gene1 (G1) et Gene2 (G2) avec la DHFR F [1,2] -NatMX et DHFR F [3] -HPH cassettes, respectivement. Les cassettes sont amplifiés à partir de plasmides pAG25 DHFR-F [1,2] et pAG32 DHFR-F [3] avec amorces sens G1-5 'et G2-5', et les amorces inverse G1-3 'et G2-3', puis inséré dans le génome à l'extrémité 3 'du gène cible par recombinaison homologue. Les protéines résultantes, P1 et P2, respectivement sont fusionnés à la DHFR F [1,2] fragment (MATa) et la DHFR F [3] fragment (MATa) via un lieur flexible. (B) de vérification de la construction en (a) est réalisée par séquençage des jonctions entre Gene1 de et les ORF de Gene2 et les cassettes DHFR. Les souches APC construits de se opposer types d'accouplement sont ensuite accouplés pour former un diploïde. Souches diploïdes poussent sur milieu MTX si les deux fragments DHFR complémentaires sont mises en proximité par une interaction entre P1 et P2, qui reconstitue l'activité de l'enzyme DHFR. (C) Construction de souches de contrôle diploïde PCA pour DHFR-écrans APC. Les contrôles négatifs (L-DHFR) sont construits en transformant souches Mata Mata et haploïdes séparément avec des plasmides p41-lieur-DHFR F [1,2] et p41-lieur-DHFR F [3] 11, respectivement. Les deux souches sont accouplés pour résultat une souche diploïde de contrôle négatif dans lequel les fragments de la DHFR sont incapablespour compléter l'autre (en haut). Les contrôles positifs sont construits en utilisant la même approche que pour les contrôles négatifs, mais les plasmides transformés dans les souches haploïdes (p41-zipper-lieur-DHFR F [1,2] (p41-ZL-DHFR F [1,2]) et p41 -Fermeture à glissière-lieur-DHFR F [3] (p41-ZL-DHFR F [3])) contenir leucine parallèle fragments de fermeture à glissière deux de GCN4 fusionnés aux fragments DHFR complémentaires, ce qui conduit à une interaction forte et constitutive qui reconstitue l'activité de DHFR (en bas ). Se il vous plaît cliquer ici pour voir une version plus grande de cette figure.
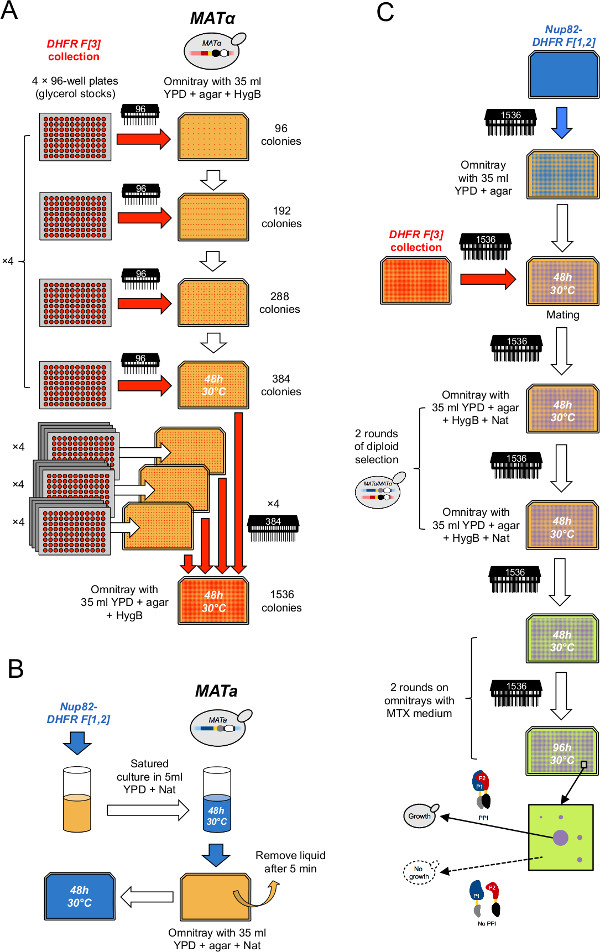
Figure 2: procédure à haut débit DHFR-PCA (figure adaptée de Leducq et al 33.) (A) Le MAT α DHFR F [3] collection. est condensé dans un format 1536 par deux cycles successifs de condensation. Tout d'abord, le glycérol plaques d'achat d'actions sont combinées par groupes de quatre sur sélective milieu YPD + hygB dans un format 384 en utilisant la broche-outil 96. Ensuite, 384 des tableaux sont combinés en groupes de quatre sur milieu sélectif YPD + hygB dans un format 1536 en utilisant la broche-outil 384. (B) tapis cellulaires de la souche d'appât MATa PCA sont préparés en cultivant une culture saturée de la souche d'appât dans YPD sélective + support Nat et le placage de la culture sur un YPD + Nat OmniTray. (C) Ces pelouses sont utilisés pour accoupler la souche d'appât à la DHFR F [3] collection sur milieu YPD. Les cellules sont transférées successivement deux fois sur un milieu YPD + hygB + Nat pour sélectionner les diploïdes et deux fois sur un milieu MTX pour effectuer PCA. La croissance ne sera observée sur un milieu MTX si les fragments DHFR se complètent mutuellement suivant une interaction entre l'amorce et les protéines de proies.e.jpg "target =" _ blank "> Se il vous plaît cliquer ici pour voir une version plus grande de cette figure.
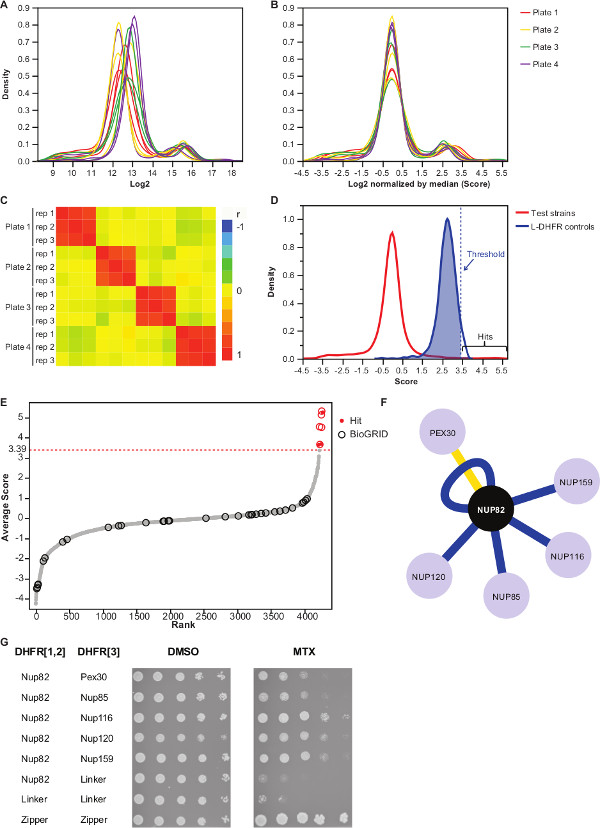
Figure 3:.. Les analyses de données à travers les étapes de la normalisation, de la détermination d'un seuil de signification et l'identification des interactions positives (A) la distribution de la densité de la taille des colonies sur chaque plaque (log 2) (B) Normalisation par la médiane de chaque image corrige les biais associés à des effets plaque-to-plate. (C) Heatmap montrant le coefficient de corrélation de Spearman entre les plaques, ce qui confirme la reproductibilité de la procédure. (D) Répartition des scores pour les IPP testé et L-DHFR F [3] contrôles. Un seuil dur peut être réglé sur les 95 e percentile de la L-DHFR F [3] Distribution d'identifier IPP haute confiance (représenté par une verticale en pointillésla distribution de la note moyenne de chaque proie ligne). (E) Rang de commande. Partenaires d'interaction physiques précédemment rapportés de Nup82p dans le BioGRID 35 sont identifiés par des cercles et ceux rapportés dans cette étude sont identifiés par des points rouges. Le seuil défini en (D) est représenté par une ligne pointillée. (F) Réseau montrant les IPP haute confiance identifiés dans cette étude. Bords blue Voir précédemment déclaré interacteurs physiques (BIOGRID 35) et les bords jaunes montrent une interaction non déclarés antérieurement avec Pex30. (G) Spot-dilution dosage des APC souches diploïdes impliquant Nup82-DHFR F [1,2] et la proie-DHFR F [3 ] paires identifiés comme interacteurs physiques de Nup82p dans la présente étude. dosage de croissance a été réalisée en milieu DMSO (solvant MTX, panneau de gauche) et moyenne MTX (panneau de droite). Les témoins négatifs constitués de Nup82 DHFR-F [1,2] - Lieur-F DHFR [3] et Lieur-F DHFR [1,2] - Lieur-F DHFR [3] et un témoin positif consistant en tqu'il forte interaction entre deux fractions de glissière à leucine (glissière-DHFR F [1,2] - fermeture éclair DHFR F [3]) ont été inclus. La croissance cellulaire supérieure aux contrôles négatifs en milieu MTX devrait être interprété comme une interaction physique. Se il vous plaît cliquer ici pour voir une version plus grande de cette figure.
| acides aminés | Quantité (g) |
| Adénine sulfate * | 0,2 |
| L-tryptophane | 0,4 |
| L-Tyrosine | 0,3 |
| L-phénylalanine | 0,5 |
| l'acide L-glutamique (sel monosodique) | 1.0 |
| L-Asparagine | 1.0 |
| L-Valine | 1,5 |
| L-Thréonine | 2.0 |
| L-sérine | 3,75 |
| Uracile | 0,2 |
| chlorhydrate de L-histidine | 0,2 |
| chlorhydrate de L-arginine | 0,2 |
| L-méthionine * | 0,2 |
| L-Lysine * | 0,2 |
| L-Leucine | 0,6 |
| * Retrait lors de l'exécution norme PCA (comme dans ce protocole), mais peuvent être ajoutés à d'autres fins. | |
Tableau 1:. Composition de la Lys 10X / MET / ade abandon dans le milieu MTX quantités sont pour un litre de 10x abandon. Les astérisques indiquent les composés qui devraient être retirés pour le milieu MTX standard, mais qui peuvent être ajoutés à d'autres fins.
Tableau supplémentaire 1:. Les données combinées des plaques 12 d'essai Tableau 1 contient la concaténation data de l'analyse ImageJ effectuée en utilisant un script de ImageJ personnalisé avec chaque ligne correspondant à une position unique sur chaque 1536 tableau. En outre, chaque ligne a été annoté avec les informations sur le fichier d'image, DHFR plaque de collection, reproduire, ORF et le nom de protéine.
Tableau complémentaire 2:. La croissance moyenne normalisée par souche tableau 2 contient le journal 2 croissance normalisée moyenne pour chaque souche de la collection ainsi que l'écart type. Proies filtré, hits et interacteurs physiques connues sont identifiés dans des colonnes distinctes.
Discussion
Nous décrivons un protocole basé sur le dosage DHFR-PCA permettant l'identification systématique des interacteurs physiques pour toute protéine appât donnée à haut débit. Ce protocole peut être adapté par le dépistage de plus appâts, et ce, à ne importe quel niveau de la réplication souhaité. Nous démontrons la fiabilité de ce protocole par l'identification de partenaires d'interaction physiques pour une protéine appât impliqué dans le complexe pore nucléaire: Nup82. Notre analyse a permis de trouver cinq interacteurs signalés précédemment, et une Interactor non signalés précédemment (figures 3F et 3G), soulignant la capacité de la méthode pour étudier l'interactome des protéines de levure.
Le protocole décrit ici comprend plusieurs étapes critiques à laquelle l'expérimentateur devrait prêter attention. Nous recommandons à 1) Assurez-vous que la DHFR appât F [1,2] fusion est correcte (figure 1B); cela peut être réalisé par séquençage de la construction et mesuring expression de la protéine appropriée en utilisant un anti-DHFR F [1,2] ou anti-DHFR F [3] anticorps; 2) Avant de commencer l'écran, il est recommandé de vérifier si tout appât d'intérêt présente interactions promiscuité dans les écrans de l'APC. Cela peut être fait en effectuant les écrans de contrôle avec des appâts croisées avec la L-DHFR contrôle approprié ou manuellement par l'appât accouplement avec la L-DHFR contrôle approprié et en effectuant un essai de croissance dans un milieu MTX. 3) Les plaques doivent être versé le jour avant leur utilisation de sorte que l'humidité est optimale pour l'adhérence des cellules sur la surface de la gélose au cours du processus d'impression; 4) des plaques de source doivent pas être utilisés plus de quatre fois pour transférer suffisamment de cellules sur la plaque de destination. Augmenter le nombre de copies de la plaque de destination peut être effectuée par des étapes successives de l'expansion (par exemple de 4 copies -> 16 copies -> 64 copies). Alternativement, les cellules peuvent être ramassés à différentes positions sur les pelouses ou dans la colonie entre les différents cycles de réplication; 5) Si plusieurs pOSTES sont portées disparues après la sélection diploïde (s), assurez-vous que les plaques de source ne ont pas été utilisés trop souvent dans l'étape d'accouplement (étapes 4.5 à 4.7); 6) Se assurer que le milieu MTX contient tous les ingrédients essentiels à la bonne concentration. En effet, si aucune croissance du tout est observée sur le support MTX, il peut être soit parce qu'aucune interaction est détectable par l'APC pour les protéines d'intérêt ou parce que le milieu MTX ne était pas préparé correctement. Pour se assurer que le milieu permet la croissance de souches présentant DHFR complémentation de fragments, une interaction constitutive peut être ajouté à des positions vides, de la collecte et utilisée comme témoin positif tel que DHFR fragments fusionnés à fermeture éclair à leucine fragments 33 (figure 1C). Tests parallèles utilisant les fragments lieur-DHFR ou les fragments glissière-lieur-DHFR permettront de discriminer entre les conditions qui permettent toutes les cellules de croître (concentration de MTX à faible ou protéine appât qui ont tendance à rendre les interactions faux positifs, comme le described ci-dessous) et des conditions qui empêchent la croissance de toutes les souches (MTX ingrédient concentration trop élevée ou essentiel manquant dans le milieu); 7) Compte tenu de l'APC est réalisée par des cycles successifs de répétitions d'un milieu à un autre, la contamination croisée entre les souches entre les différentes plaques peut se produire si, par exemple, la broche-outil ne est pas stérilisé correctement entre les cycles de réplication et / ou la dernière eau bain (poste-à-dire l'état humide) dans la procédure de stérilisation est contaminé par des colonies de cycles de réplication précédentes. Plusieurs positions sur les tableaux sont vides et peuvent donc être utilisés comme postes de contrôle où doit être observé aucune croissance pour détecter les contaminations croisées.
L'analyse d'image peut être effectuée en utilisant plusieurs logiciels publiés (voir l'article 5 du protocole) ou tout script personnalisé. Dans cette étude, le script personnalisé exécute les étapes suivantes: 1) Le script soustrait les valeurs de pixels d'une assiette vide au pixel des valeurs de chaque plaque afin de correct les biais d'éclairage. 2) Le script convertit chaque image de fond corrigée en binaire en utilisant un seuil de 10. 3 de valeur de pixel) Pour chaque 1536 positions de chaque plaque, déterminée en superposant un rectangle sur les colonies de pointe, le script se exécute ImageJ "Analyser particules ... fonction "dans une sélection circulaire. La sélection est réglée circulaire avec un rayon égal à l'intervalle entre deux positions moins 10 pixels. 4) Le script sélectionne la particule la plus proche du centre de la sélection et confirme comme une colonie si son emplacement ne est pas plus de la moitié de l'intervalle entre deux colonies du centre de la sélection. 5) Le script mesure des valeurs de pixel de la particule sélectionnée sur l'image de fond corrigée. 6) pour corriger en outre des distorsions restantes fond d'illumination, le script soustrait la valeur moyenne de tous les pixels de la sélection circulaire ne faisant pas partie d'une particule de valeurs de pixel de la colonie. La somme de ces valeurs de pixel corrigés, stockées dans la colonne "IntDenBackSub" du tableau supplémentaire 1, sont utilisés comme une mesure de la taille de la colonie.
Une étape critique dans la partie d'analyse est le choix du seuil de signification. Ici, nous avons choisi un seuil basé sur la distribution du négatif L-DHFR F [3] contrôles, mais en fonction de l'objectif de l'écran, ce seuil peut être trop strictes. En effet, la L-DHFR F [3] commandes sont surexprimé (fort promoteur de TEF) de telle sorte que les fragments complémentaires peuvent spontanément se complètent l'un l'autre et ceux-ci ne sont donc pas représentatifs de l'expression de la plupart des protéines. Ceci est mis en évidence par le fait que la répartition de la L-DHFR F [3] contrôles est supérieure à la moyenne de la croissance d'arrière-plan (figure 3D). Ainsi, certaines interactions ayant des notes inférieures à ce seuil strictes mais qui sont clairement en dehors de la distribution de la croissance de fond peut être considéré comme résultats putatifs qui peuvent représenter, par exemple, entre transitoire ou faibleactions. Ceux-ci peuvent être en outre étudiés et contre-validées que si, par exemple, les deux protéines ne sont pas exprimés à des niveaux qui peuvent permettre la complémentation spontanée des fragments DHFR comme les contrôles L-DHFR. Comme alternative, on pourrait fixer un seuil de signification sur la base de la proportion de chevauchement avec interacteurs physiques signalées dans des bases de données comme BioGRID 35 afin de maximiser la proportion de vrais positifs sur les faux positifs. Cependant, contrairement à l'utilisation de la distribution L-DHFR, cette solution ne est pas toujours possible si, par exemple, le nombre d'interacteurs physiques connues ne est pas suffisamment élevée. De plus, le choix du seuil de signification a une incidence sur la proportion de faux positifs et de faux négatifs dans l'ensemble de données final. En effet, comme tout autre essai de détection de PPI, les faux positifs peuvent résulter d'une interaction non spécifique d'une protéine avec la protéine DHFR-fusion si, par exemple, la protéine est très abondante comme mentionné plus haut. Cetteest illustré par le fait que certaines proies interagir systématiquement avec toutes les protéines d'appâts dans les écrans de l'APC et, par conséquent, doivent être retirés de l'analyse 11 (par exemple Tef2 et Ade17 et le tableau complémentaire 2). Pour contourner ce problème, un écran de contrôle de PCA des deux collections contre le contrôle L-DHFR approprié (F [1,2] ou F [3]) pour identifier les appâts et proies présentant spontanée DHFR fragmente complémentation peut être effectuée dans les conditions spécifiques de chaque écran. En outre, la réalisation d'une analyse de l'enrichissement Gene Ontology peut augmenter la confiance dans les données si la fonction d'un appât donnée est connue. D'autre part, DHFR-PCA peut donner lieu à des faux négatifs pour plusieurs raisons: 1) toutes les protéines peuvent être fusionnés aux fragments DHFR comme ceux-ci peuvent déstabiliser les protéines ou modifier leur localisation si, par exemple, la fusion à la DHFR C-terminale interfère avec un signal de localisation; 2) la reconstitution de la DHFR dans certains compartiments cellulaires may pas produire de folate si, par exemple, un précurseur essentiel à la synthèse de l'acide folique ne est pas disponible; 3) C-terminales besoin d'être dans un rayon de 8 nm pour DHFR complémentation de se produire 11. Ainsi, une interaction bien connue peut ne pas être détecté si leur C-terminales sont pas assez proche dans l'espace. Cela est illustré ici par le fait qu'une grande partie des interactions physiques Nup82 rapportés dans les bases de données, dont la plupart sont indirect, ne ont pas été détectée dans notre essai. De même, les interactions entre protéines membranaires dont les C-terminales sont en trans par rapport à la membrane ne conduiront pas à DHFR fragmente complémentation et ne sera pas détectée 11. Limitations 1) et 3) peut être contournée relativement simplement par fusion du fragment de DHFR à l'extrémité N-terminale de la protéine. Cela peut empêcher d'interférer avec un signal de localisation près de la C-terminales et peut permettre de détecter une interaction entre protéines membranaires dont N et C-terminale sont en cis par rapportà la membrane.
Plusieurs défis restent à relever dans l'étude des codes PIN (revue dans 2,3). Les cartes de broches produits jusqu'ici ont été largement décrit dans une seule condition expérimentale pour chaque espèce et d'offrir ainsi un instantané unique de la façon dont les réseaux de protéines pourraient être organisées. Il ya donc un besoin pour l'exploration d'autres conditions expérimentales de voir comment les codes PIN peuvent être réorganisées en réponse aux changements environnementaux, des stimuli spécifiques, à travers le développement ou des mutations suivantes. Ces défis seront surmontés par le développement de nouvelles technologies pour interroger IPP en temps réel, dans les cellules vivantes et en adaptant les techniques actuelles afin qu'ils puissent être utilisés par une grande communauté de laboratoires. Comme une technique quantitative qui permet de détecter les changements dans la quantité de DHFR complémentation complexes 27, DHFR-PCA peut être adapté pour surmonter ces défis et a été utilisé pour étudier comment les IPP sont affectés par un agent de l'ADN 22 endommager , 25 agents chimiques, la délétion de gènes 23,26 ou dans d'autres espèces de levures et leurs hybrides 33. L'étude de ces nouvelles dimensions deviendra de plus en plus important de révéler la dynamique du code PIN.
Déclarations de divulgation
Une partie des frais de publication ouverts accès pour cet article ont été payés par S & P Robotics.
Remerciements
Ce travail a été soutenu par l'Institut canadien de recherche en santé du Canada (IRSC) Subventions 191 597, 299 432 et 324 265, du Conseil de recherches en génie du Canada subvention Discovery sciences naturelles et en une subvention Human Frontier Science Program à CRL. CRL est un nouveau chercheur des IRSC. Guillaume Diss est soutenu par une bourse PROTEO. Samuel Rochette est appuyé par le CRSNG et FRQNT bourses.
matériels
| Name | Company | Catalog Number | Comments |
| BioMatrix Robot, Bench-top Configuration | S&P Robotics Inc. | BM5-BC | |
| 96-format Pin-tool | S&P Robotics Inc. | PH-96-10 | Standard 96-format Pin-tool with 96 high-precision floating pins |
| 384-format Pin-tool | S&P Robotics Inc. | PH-384-10 | Standard 384-format Pin-tool with 384 high-precision floating pins |
| 1536-format Pin-tool | S&P Robotics Inc. | PH-1536-05 | Custom 1536-format Pin-tool with 0.5mm high-precision floating pins |
| Automated imaging module | S&P Robotics Inc. | IMG-02 | |
| Methotrexate | Bioshop Canada Inc. | MTX440 | CAUTION: toxic compound |
| Hygromycin B | Bioshop Canada Inc. | HYG003 | |
| Nourseothricin dihydrogen sulfate | Werner BioAgents | 5010000 | |
| Yeast-Interactome Collection | Thermo Scientific | YSC5849 | |
| Omni Tray w/lid sterile | Thermo Scientific | 242811 | |
| Anti-DHFR F[1,2] antibody | Sigma-Aldrich | D1067 | |
| Anti-DHFR F[3] antibody | Sigma-Aldrich | D0942 |
Références
- Alberts, B. The cell as a collection of protein machines: preparing the next generation of molecular biologists. Cell. 92, 291-294 (1998).
- Diss, G., et al. Integrative avenues for exploring the dynamics and evolution of protein interaction networks. Curr Opin Biotechnol. 24, 775-783 (2013).
- Vidal, M., Cusick, M. E., Barabasi, A. L. Interactome networks and human disease. Cell. 144, 986-998 (2011).
- Hu, P., et al. Global functional atlas of Escherichia coli encompassing previously uncharacterized proteins. PLoS biology. 7, e96 (2009).
- Arifuzzaman, M., et al. Large-scale identification of protein-protein interaction of Escherichia coli K-12. Genome Res. 16, 686-691 (2006).
- Rajagopala, S. V., et al. The binary protein-protein interaction landscape of Escherichia coli. Nature biotechnology. 32, 285-290 (2014).
- Arabidopsis-Interactome-Mapping-Consortium. Evidence for network evolution in an Arabidopsis interactome map. Science. 333, 601-607 (2011).
- Babu, M., et al. Interaction landscape of membrane-protein complexes in Saccharomyces cerevisiae. Nature. 489, 585-589 (2012).
- Krogan, N. J., et al. Global landscape of protein complexes in the yeast Saccharomyces cerevisiae. Nature. 440, 637-643 (2006).
- Gavin, A. C., et al. Functional organization of the yeast proteome by systematic analysis of protein complexes. Nature. 415, 141-147 (2002).
- Tarassov, K., et al. An in vivo map of the yeast protein interactome. Science. 320, 1465-1470 (2008).
- Uetz, P., et al. A comprehensive analysis of protein-protein interactions in Saccharomyces cerevisiae. Nature. 403, 623-627 (2000).
- Guruharsha, K. G., et al. A protein complex network of Drosophila melanogaster. Cell. 147, 690-703 (2011).
- Li, S., et al. A map of the interactome network of the metazoan C. elegans. Science. 303, 540-543 (2004).
- Rual, J. F., et al. Towards a proteome-scale map of the human protein-protein interaction network. Nature. 437, 1173-1178 (2005).
- Landry, C. R., Levy, E. D., Abd Rabbo, D., Tarassov, K., Michnick, S. W. Extracting insight from noisy cellular networks. Cell. 155, 983-989 (2013).
- Berggard, T., Linse, S., James, P. Methods for the detection and analysis of protein-protein interactions. Proteomics. 7, 2833-2842 (2007).
- Wodak, S. J., Vlasblom, J., Turinsky, A. L., Pu, S. Protein-protein interaction networks: the puzzling riches. Current opinion in structural biology. 23, 941-953 (2013).
- Fields, S., Song, O. A novel genetic system to detect protein-protein interactions. Nature. 340, 245-246 (1989).
- Dunham, W. H., Mullin, M., Gingras, A. C. Affinity-purification coupled to mass spectrometry: basic principles and strategies. Proteomics. 12, 1576-1590 (2012).
- Michnick, S. W., Ear, P. H., Landry, C., Malleshaiah, M. K., Messier, V. A toolkit of protein-fragment complementation assays for studying and dissecting large-scale and dynamic protein-protein interactions in living cells. Methods Enzymol. 470, 335-368 (2010).
- Rochette, S., Gagnon-Arsenault, I., Diss, G., Landry, C. R. Modulation of the yeast protein interactome in response to DNA damage. Journal of proteomics. 100, 25-36 (2014).
- Diss, G., Dube, A. K., Boutin, J., Gagnon-Arsenault, I., Landry, C. R. A systematic approach for the genetic dissection of protein complexes in living cells. Cell Rep. 3, 2155-2167 (2013).
- Gagnon-Arsenault, I., et al. Transcriptional divergence plays a role in the rewiring of protein interaction networks after gene duplication. Journal of proteomics. 81, 112-125 (2013).
- Schlecht, U., Miranda, M., Suresh, S., Davis, R. W., St Onge, R. P. Multiplex assay for condition-dependent changes in protein-protein interactions. Proceedings of the National Academy of Sciences of the United States of America. 109, 9213-9218 (2012).
- Lev, I., et al. Reverse PCA, a systematic approach for identifying genes important for the physical interaction between protein pairs. PLoS Genet. 9, e1003838 (2013).
- Freschi, L., Torres-Quiroz, F., Dube, A. K., Landry, C. R. qPCA: a scalable assay to measure the perturbation of protein-protein interactions in living cells. Mol Biosyst. 9, 36-43 (2013).
- Pelletier, J. N., Campbell-Valois, F. X., Michnick, S. W. Oligomerization domain-directed reassembly of active dihydrofolate reductase from rationally designed fragments. Proceedings of the National Academy of Sciences of the United States of America. 95, 12141-12146 (1998).
- Gietz, R. D., Woods, R. A. Transformation of yeast by lithium acetate/single-stranded carrier DNA/polyethylene glycol method. Methods Enzymol. 350, 87-96 (2002).
- Schuldiner, M., Collins, S. R., Weissman, J. S., Krogan, N. J. Quantitative genetic analysis in Saccharomyces cerevisiae using epistatic miniarray profiles (E-MAPs) and its application to chromatin functions. Methods. 40, 344-352 (2006).
- Schneider, C. A., Rasband, W. S., Eliceiri, K. W. NIH Image to ImageJ: 25 years of image analysis. Nature methods. 9, 671-675 (2012).
- Wagih, O., Parts, L. gitter: A Robust and Accurate Method for Quantification of Colony Sizes From Plate Images. G3 (Bethesda). 4 (3), 547-552 (2014).
- Leducq, J. B., et al. Evidence for the robustness of protein complexes to inter-species hybridization. PLoS Genet. 8, e1003161 (2012).
- . . Development Core Team: A language and environment for statistical computing. , (2008).
- Stark, C., et al. BioGRID: a general repository for interaction datasets. Nucleic Acids Res. 34, D535-D539 (2006).
- Vinayagam, A., et al. Protein complex-based analysis framework for high-throughput data sets. Science signaling. 6, rs5 (2013).
- Jansen, R., Gerstein, M. Analyzing protein function on a genomic scale: the importance of gold-standard positives and negatives for network prediction. Current opinion in microbiology. 7, 535-545 (2004).
- Ito, T., et al. A comprehensive two-hybrid analysis to explore the yeast protein interactome. Proceedings of the National Academy of Sciences of the United States of America. 98, 4569-4574 (2001).
- Costanzo, M., et al. The genetic landscape of a cell. Science. 327, 425-431 (2010).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon