Method Article
Genomweite Protein-Protein-Wechselwirkungen durch Screening Protein-Fragment Komplementierungsassay (PKA) in lebenden Zellen
In diesem Artikel
Zusammenfassung
Proteins interact with each other and these interactions determine in a large part their functions. Protein interaction partners can be identified at high-throughput in vivo using a yeast fitness assay based on the dihydrofolate reductase protein-fragment complementation assay (DHFR-PCA).
Zusammenfassung
Proteins are the building blocks, effectors and signal mediators of cellular processes. A protein’s function, regulation and localization often depend on its interactions with other proteins. Here, we describe a protocol for the yeast protein-fragment complementation assay (PCA), a powerful method to detect direct and proximal associations between proteins in living cells. The interaction between two proteins, each fused to a dihydrofolate reductase (DHFR) protein fragment, translates into growth of yeast strains in presence of the drug methotrexate (MTX). Differential fitness, resulting from different amounts of reconstituted DHFR enzyme, can be quantified on high-density colony arrays, allowing to differentiate interacting from non-interacting bait-prey pairs. The high-throughput protocol presented here is performed using a robotic platform that parallelizes mating of bait and prey strains carrying complementary DHFR-fragment fusion proteins and the survival assay on MTX. This protocol allows to systematically test for thousands of protein-protein interactions (PPIs) involving bait proteins of interest and offers several advantages over other PPI detection assays, including the study of proteins expressed from their endogenous promoters without the need for modifying protein localization and for the assembly of complex reporter constructs.
Einleitung
Protein-Interaktionsnetzwerke (PIN) bieten eine niedrige Auflösung der Karte, wie Proteine funktionell in der Zelle 1 organisiert. Jeder physikalische Verbindung zwischen zwei Proteinen oder Protein-Protein-Wechselwirkung (PPI) kann eine Verbindung, die zeitlich stabil ist, wie sie in Proteinkomplexe gefunden repräsentieren und die in die strukturelle Organisation der Zelle beitragen. Diese Verbindungen können auch stellen vorübergehenden Vereinigungen, die die Aktivität, Stabilität, Lokalisierung und Interaktionen der beiden Partner zu regeln. Die Ermittlung der physikalischen Interaktionspartner eines bestimmten Proteins bietet daher reichen Informationen über die Funktion und Regulation dieses Proteins 2,3. Aus diesen Gründen haben große Anstrengungen zur Kartierung von PINs in Modellorganismen wie Escherichia coli 4-6, Arabidopsis thaliana 7, Saccharomyces cerevisiae 8-12, Drosophila melanogaster worden/ Em> 13, Caenorhabditis elegans 14 und Homo sapiens 15. Diese Studien haben wichtige Erkenntnisse darüber, wie Proteine in der Zelle und damit wichtige Informationen über Proteine mit bisher unbekannten Funktionen organisiert ist.
Mehrere Strategien sind in den letzten Jahren zu studieren PINs entwickelt. Diese Technologien lassen sich grob in drei Kategorien auf der Grundlage der Art der Informationen, die sie auf PPI bieten (in 16 bis 18 überprüft) gruppiert werden. Die erste ist auf die Hefe-Zwei-Hybrid und seine Derivate 19 basiert. Diese Technologien liefern Informationen über die direkte Verbindung zwischen Paaren von Proteinen, die die Konstruktion binärer Netzwerke ermöglicht. Die zweite Familie basiert auf der Affinitätsreinigung von Köderproteine und die Identifizierung ihrer zugehörigen Partner wie Affinitätsreinigung gefolgt von Massenspektrometrie 20 basiert. Diese Ansätze identifiziert Gruppen von Proteinen, die direktoder indirekt verbunden sind, im allgemeinen in einer stabilen Art und Weise und sind extrem leistungsfähig, um die Proteinkomplexe zu identifizieren. Der dritte Ansatz beruht auf Protein-Fragment Komplementierungsassays (PKA) 11,21 basiert. Dieser Ansatz bietet ein mittleres Niveau der Auflösung zwischen den beiden früheren Ansätzen, denn sie ermöglicht Erfassung direkter und proximalen Assoziationen zwischen Proteinen. Jede Technik hat ihre eigenen Stärken und Schwächen, wie vor kurzem überprüft 18.
Die beste beschriebenen eukaryotischen PIN ist bei weitem die eines der Bäckerhefe Saccharomyces cerevisiae, zum Teil wegen seiner Proteom ist relativ weniger komplex als bei anderen Modell Eukaryoten und wegen Hochdurchsatz-Assays zum Nachweis PPI haben zunächst untersucht worden und effizienter sind in diesem Modellorganismus 9-12 implementiert. Eine besonders leistungsfähige Methode für die Hefe-System ist das Dihydrofolatreduktase-Protein-Fragment Komplementierungsassay (DHFR-PCA) ist ein Test, der gewesen istin unterschiedlichen Zusammenhängen verwendet, um die Hefe-PIN in Standard- und gestörte Bedingungen 11,22-26 studieren. Dieses Verfahren beruht auf einem Überlebens-Assay, der die Detektion von direkt und nahezu direkte PPI für eine gegebene Köderprotein sowohl endogenen Expressionsniveaus und nativen subzellulären Lokalisation der Interaktionspartner 11,21 auf quantitative Weise 27 ermöglicht. Die unter Verwendung dieses Tests (dh Koloniegröße auf High-Density-Kolonie-Arrays) erhaltene Signal spiegelt somit die Menge an Proteinkomplexen zwischen Köder und Beute in einer zellulären Umgebung fast gleichbedeutend mit dem eines Wildtyp-Zellen gebildet. Der Test basiert auf der Rekonstitution eines Reporterenzym in Folatstoffwechsel berücksichtigt, die Dihydrofolatreduktase (DHFR), wobei zwei komplementäre Fragmente von DHFR, die an die beiden Proteine von Interesse fusioniert sind, in die Nähe gebracht wird, wenn die zwei Proteine interagieren, die wiederum zu einer reversiblen Rekonstitution der Enzymaktivität 11 und das Wachstum des Stammes auf einem Medium, das Methotrexat (MTX; Abbildung 1). Diese Verbindung hemmt die endogene DHFR-Enzym, nicht aber das mutierte im Test 28 verwendet. Zwei Sammlungen von PCA-Stämmen, von denen eine ~ 4300 MATa Stämme mit ein ORF fusioniert mit dem DHFR-F [1,2] Fragment und eine mit ~ 4800 MAT α-Stämme mit einem ORF fusioniert mit dem DHFR [3] Fragment kann erworben werden Umsetzung DHFR-PCA bei kleinen oder großen Maßstab in jedem Labor. Hier beschreiben wir eine allgemeine, aber ausführliches Protokoll, um Bildschirm für die PPI zwischen einem Köderprotein und ~ 4800 Beuteproteine unter Verwendung dieses Tests.
Protokoll
1. Aufbau / Überprüfung der Bait-Stämme
- Wenn der Köder Stamm von Interesse ist in der MATa DHFR F [1,2] Sammlung zur Verfügung, rufen Sie sie aus der Sammlung, wie in Schritt 1.1.1, die Belastung auf andere Weise zu konstruieren, wie in Schritt 1.1.2 beschrieben.
HINWEIS: Die hier beschriebene Protokoll verwendet eine DHFR-F [1,2] Belastung als Köder und das DHFR-F [3] Sammlung als Beute, wie diese Sammlung enthält mehr Stämme als die DHFR F [1,2] Sammlung. Es ist jedoch möglich, den Bildschirm umgekehrt durchführen, wenn der Köder Stamm ist in der DHFR-F [3] Sammlung oder in beiden Orientierungen nur verfügbar, wenn eine höhere Abdeckung des Interaktom erfordert.- Auftauen Glycerolstammlösung Platte, die den Köder Belastung auf Eis für eine Stunde enthält. Sterilisieren Sie die Aluminiumfolie bedeckt die Platte mit 95% igem Ethanol. Pierce die Folie mit einem sterilen Spitze Pipette auf und ab, um die Zellen und streifen 2-3 ul Glycerin Lager auf selektiven Hefeextrakt-Pepton resuspendierenDextrose (YPD) + 100 ug / ml Nourseothricin (Nat), um einzelne Kolonien zu isolieren. Inkubieren für zwei Tage bei 30 ° C.
- Bau eines Köders PCA Stamm (MATa DHFR F [1,2]).
- Mit High-Fidelity-Polymerase und eine Standard-PCR-Protokoll, verstärken das DHFR F [1,2] Kassette aus dem Plasmid pAG25-Linker-DHFR F [1,2] -ADHterm unter Verwendung von Oligonukleotiden mit überhängenden Enden homolog zu der letzten 40 bp des ORF 3'-Ende außer dem Stopcodon (Forward-Primer) und den ersten 40 bp des Gens der 3'-UTR (Rückwärts-Primer) (1A).
- Transformieren des PCR-Produkts in kompetente Hefezellen (üblicherweise in einem BY4741 Stamm) unter Verwendung von Standard LiOAc / PEG Hefe-Transformationsprotokoll, wie in 29 (1A).
- Platte auf selektive YPD + Nat mittlere bis positive Transformanten zu isolieren.
- Führen Sie eine Diagnose-Kolonie-PCR auf isolierten Kolonien, um die richtige DHFR F [1,2] Fusion zu bestätigen. Verwenden prIMers Tempern 1) in der Gen-kodierenden Sequenz (Forward oligo) etwa 100 bp stromaufwärts von der DHFR-Fusion und 2) in der ADH-Terminator der Kassette (Rückwärts-Oligo) (1B).
- Sequenzierung des PCR-Produkts durch Sanger-Sequenzierung, um die ordnungsgemäße Genfusion zu bestätigen.
- Archivieren Sie das bestätigt Köder Stamm in 25% Glycerin bei -80 ° C.
HINWEIS: Das Protokoll kann in diesem Schritt angehalten werden.
2. Pin-Werkzeug Sterilisation und Vorgehensweise beim Drucken
HINWEIS: Die nachfolgend beschriebene Sterilisationsverfahren wurde für die pin-Tools durch die BM3-BC (S & P Robotics) Roboterplattform manipuliert optimiert, kann jedoch auf anderen Plattformen als auch angepasst werden. Dieser Abschnitt beschreibt die Pin-Werkzeug Sterilisation und Druckverfahren, die verwendet werden, um Zellen von einem Medium auf ein anderes für den Rest des Protokolls zu übertragen. In-house verwendet, um diese Routinen Skripte können auf Anfrage erhältlich. Hinweis that alle Schritte können ohne die Notwendigkeit eines Roboterplattform über eine manuelle Nadelwerkzeug 30 durchgeführt werden.
- Montieren Sie den entsprechenden Stift-Werkzeug auf der Roboterplattform.
- Bereiten Reinigung und Nassstationen wie folgt:
- Fügen Sie 500 ml sterilem Wasser in das Wasserbad entfernt.
- Fügen Sie 320 ml sterilem Wasser in der Bürstenstation.
- Fügen Sie 380 ml 70% Ethanol im Ultraschallgerät beim Replizieren von Agar-Platte (86 x 128 mm OmniTray, mit 35 ml verfestigtes Medium) auf Agar-Platte oder 400 ml bei der Replikation von einer Mikrotiterplatte, die Flüssigkulturen auf einer Agar-Platte.
- Fügen Sie 35 ml steriles Wasser in der nassen Bahn-Station (bestehend aus einer sterilen leeren OmniTray).
- Zu Beginn eines jeden Tages, die die Roboterplattform benötigt, Sterilisieren der Stift-Werkzeuge fünfmal für eine Minute im Ultraschallbad. In der Zwischenzeit, schalten Sie die UV-Lampe für fünf Minuten, um den Roboter Gehäuse zu sterilisieren.
HINWEIS: Dies ist nicht, ob die rob erforderlichot unter einer sterilen Haube untergebracht. - Zwischen jedem Pin-Werkzeug Replikationsrunde, sterilisieren Sie die Pin-Tool wie folgt:
- Weichen Sie das Stift-Werkzeug fünfmal für 10 Sekunden in das Wasserbad Station Zellklumpen zu entfernen.
- Weichen Sie das Stift-Werkzeug zweimal hin und her in der Bürstenstation.
HINWEIS: Die rotierende Bürste wird Restzellen zu entfernen. - Weichen Sie das Stift-Werkzeug zweimal für 20 Sekunden in der Beschallungsgerät Station.
HINWEIS: verbleibenden Zellen an den Zapfen wird durch Ultraschallbehandlung entfernt oder durch Ethanol getötet werden. - Stellen Sie sicher, dass die Eintauchtiefe der Stifte erhöht bei jedem aufeinanderfolgenden Bad, um eine ordnungsgemäße Sterilisation gewährleistet.
- Trocknen Sie das Stift-Werkzeug in der Lufttrocknerstation für 25 Sekunden.
- Vor der Einnahme von Zellen auf den Quellenplatten, die Stifte zu benetzen im Nassstation, die 35 ml steriles Wasser in einem OmniTray enthält.
- Die Stifte Tauchen Sie zweimal in den Kolonien, die von der Source-Platte.
- Drucken auf einem frisch gezapften Zielplatte, indem Sie die agar Fläche zweimal (im Folgenden als die Wirkung der "Drucken" ein Array bezeichnet).
3. Kondensation des DHFR-F [3] Sammlung in 1536 Arrays mit Hilfe eines automatisierten Roboter-Plattform
- Thaw auf Eis das DHFR-F [3] Sammlung (60 96-Well-Platten) und ein zusätzliches 96-Well-Platte mit dem L-DHFR-F [3] Kontrollstamm (1C), die das DHFR-F enthält [3] Fragment aufgefüllt und der stromaufwärts Linker allein als negative Kontrolle ausgedrückt.
HINWEIS: Im Prinzip sollte dieses Fragment nicht mit einem DHFR-Fragment-Fusionsprotein (siehe Diskussion für Details) interagieren. - Zentrifugenplatten (schnelle Runde), bevor Sie die Aluminiumfolie um das Risiko von Kreuzkontaminationen bei den Brunnen zu vermeiden.
- Kondensieren die Sammlung auf 16 Feldern von 384 Stämmen (Abbildung 2A). Um dies zu tun, für jede 384-Array, Druck vier Glycerin Platten an den vier Quadranten eines selektiven YPD + 250 ug / ml Hygromycin B (HygB) OmniTray mit the 96 pin-Tool (hier ein Array 384 kann in vier gleich beabstandeten Quadranten unterteilt werden, die jeweils aus 96 Positionen in einem 2 x 2-Matrix-Layout). Legen vier Platten mit 96 Vertiefungen, die das L-DHFR F [3] Negativkontrolle zwischen den anderen Platten 60, um einen endgültigen Satz von 64 Platten, die genau vier 1.536 Anordnungen füllen. Sterilisieren Sie den Stift-Werkzeug zwischen jedem Replikationszyklus wie in Schritt 2 beschrieben.
HINWEIS: Legen Sie die L-DHFR-F [3] Platten, um eine solche Platte auf jeder der vier letzten 1536 Arrays. - Inkubation erfolgt für zwei Tage bei 30 ° C. HINWEIS: In diesem Stadium ist die DHFR Sammlung kann in einem 384-Format bei 4 ° C für bis zu einem Monat auf Agarplatten gespeichert.
- Kondensieren die Sammlung in vier Arrays von 1536 Stämme (Abbildung 2A). Um dies zu tun, für jedes Array 1.536 Drucken vier Arrays von 384 Stämmen an den vier Quadranten einer Platte aus dem gleichen Medium wie in Schritt 3.2 mit der 384-Pin-Werkzeug (hier kann ein 1.536 Array in vier E unterteilt werdenQually beabstandet Quadranten, die jeweils aus 384 Positionen in einer 2 x 2-Matrix-Layout).
- Inkubation erfolgt für zwei Tage bei 30 ° C. HINWEIS: In diesem Stadium ist die DHFR Sammlung kann in einem 1.536-Format bei 4 ° C für bis zu einem Monat auf Agarplatten gespeichert.
- Replizieren Sie die vier Arrays auf dem gleichen Medium, um Koloniegröße Standardisierung mit Hilfe eines 1536 Pin-Werkzeug.
- Inkubation erfolgt für zwei Tage bei 30 ° C.
4. Hochdurchsatz-DHFR-PCA Verfahren
- Impfen Sie eine Kultur des aus Schritt 1.1.1 oder 1.1.2 in 20 ml flüssigem YPD + Nat in einem 50-ml-Tube (2B) erhalten Köder Stamm (DHFR F [1,2] Fusion).
- Inkubieren für zwei Tage bei 30 ° C unter Schütteln bei 250 rpm, damit die Kultur bis zur Sättigung zu erreichen.
- Nach zwei Tagen Inkubation Platte 5 ml der Kultur auf einem YPD + Nat OmniTray. Lassen Zellen adsorbiert auf der Oberfläche für 5-10 Minuten und entfernen Sie die überschüssige Flüssigkeit (2B). Wiederholen twice zu drei Wiederholungen zu machen.
- Inkubieren für zwei Tage bei 30 ° C.
- Drucken Sie den Köder Stamm aus den Schritten 4.3 und 4.4 am 12. YPD-Platten (genug, um Paarung vier Platten des DHFR-F [3] Sammlung × drei Wiederholungen) mit einem 1536 Pin-Werkzeug mit jeder Zellrasen nicht mehr als das Vierfache.
- Drucken Sie den entsprechenden Array des DHFR-F [3] Sammlung oben auf den Köder Zellen unter Verwendung des 1536 Stift-Werkzeug (2C).
- Lassen Sie die Belastungen paaren durch Inkubation für 2 Tage bei 30 ° C.
- Wählen diploiden Zellen durch Drucken Kolonien auf OmniTrays enthaltenden YPD + HygB + Nat (2C).
- Inkubieren für zwei Tage bei 30 ° C.
- Wiederholen diploiden Auswahl wie in den Schritten 4.8 und 4.9 (2B) beschrieben.
- Bereiten Platten Medien mit MTX (MTX-Medium) am Tag vor der Verwendung die folgenden Schritte 21 (ACHTUNG:. Vorsicht beim Umgang mit MTX, da es eine toxische Verbindung Tragen Sie immer Handschuhe, protection Brille und Laborkittel beim Umgang mit es) (2C):
- Bereiten Sie die 10-fach-Lys / met / ade Aminosäure-drop-out in VE-Wasser. Filter-Sterilisation des Drop-out in eine sterile Flasche mit einem 0,2 um sterile Spritzenfilter oder, wenn in großen Mengen benötigt wird, eine 0,2 um Flaschenverschluss Filtertrichter (Tabelle 1).
- Bereiten Sie eine 10 mg / ml MTX Stammlösung in Dimethylsulfoxid (DMSO). Unmittelbar nach dem Herstellen der Lösung und Gefriert der Rest bei -20ºC. Schützen Sie es vor Licht, da sie lichtempfindlich ist. MTX wieder Nicht einfrieren nach dem Auftauen es.
- Vorbereiten der Medien wie folgt (mittel Bestandteile und Mengen sind die gleichen wie diejenigen in Tarassov et al 11 verwendet.):
- Für einen Liter des Mediums, mischen in zwei getrennten Kolben: 1) 6,69 g Hefe-Stickstoffbase ohne Aminosäuren und ohne Ammoniumsulfat und 330 ml entionisiertem Wasser; 2) 25 g Agar und edlen 500 ml VE-Wasser.
- Autoklaven-Kolben bei 121 ° C für 20 min.
- Äquilibrieren Temperatur in einem Wasserbad bei 55 ° C für mindestens eine Stunde.
- Man vermischt die beiden Kolben miteinander und 50 ml sterile 40% Glucose, 100 ml 10X sterile drop-out, und 20 ml MTX 10 mg / ml.
- Gießen Sie 35 ml (siehe Anmerkung unten) von mittel OmniTrays. Lassen Sie erstarren für mindestens eine Stunde und eine Hälfte. Schutzplatten vor Licht.
HINWEIS: Hier gießt 35 ml Medium pro OmniTray ist entscheidend für die gleiche Blechdicke, die für alle nachfolgenden Schritte ist zu gewährleisten.
- Bildplatten von der zweiten Runde der diploiden Auswahl mit der Roboter-Plattform oder mit einer normalen Digitalkamera mit einheitlichen Schildbeleuchtung. Verwenden Sie diese Bilder zu leeren Positionen auf den Arrays zu identifizieren, wenn die Durchführung der Downstream-Analyse. Sicherstellen, dass die Parameter der Kamera immer die gleichen sind, und dass der Roboter Licht eingeschaltet ist.
- Drucken diploiden Zellen auf MTX Medien using des 1536 Stift-Werkzeug.
- Inkubieren für vier Tage bei 30 ° C in Plastikbeuteln um ein Austrocknen zu verhindern.
- Bereiten Sie eine zweite Charge von OmniTrays enthält MTX Medium wie in Schritt 4.11 beschrieben.
- Nach vier Tagen Inkubation, Bildplatten mit Hilfe der Roboterplattform oder normalen Digitalkamera. Sicherstellen, dass die Parameter der Kamera immer die gleichen sind, und dass der Roboter Licht eingeschaltet ist.
- Durchführen einer zweiten Runde der MTX-Selektion durch die Replikation der Zellen in der zweiten Charge von MTX Medien.
Hinweis: das wird das Hintergrundwachstum von PCA-Stämme zu verringern und die quantitative Auflösung. - Inkubieren für vier Tage bei 30 ° C in Plastikbeuteln um ein Austrocknen zu verhindern.
- Bildplatten, wie in Schritt 4.16 beschrieben.
5. Bildanalyse
- Analysieren Sie Bilder der Kolonie-Arrays mit benutzerdefinierten ImageJ 31 Skripte oder mit veröffentlichten Software wie Colonizer, Ht Kolonie Grid-Analysator, Cell Profiler Colony Bildr, ScreenMill, YeastXtract und gitter (in 32 zusammengestellt). Die Bildanalyse ist die Gesamtproduktion einer oder mehrerer Tabellen, die Koloniegrößen für jede Position jedes Array, verwenden Sie diese Kolonie Größen für alle Downstream-Analysen.
HINWEIS: In dieser Studie verwendeten wir ein in Leducq et al benutzerdefinierte ImageJ Skript 33 (siehe Diskussion Abschnitt für weitere Details)..
6. Datenanalyse
HINWEIS: Die Ergebnisse der Bildanalyse in einem Tabulator wie Excel oder mit Hilfe einer Skriptsprache wie R 34 verarbeitet werden. Die folgenden Schritte beschreiben das Verfahren mit einem benutzerdefinierten Skript ImageJ 31.
- Mit einem benutzerdefinierten Skript, verketten Ausgabedateien von Bildanalyse und kommentieren jede Zeile mit der Platte und belasten Informationen wie in Tabelle 1 Zusatz.
- Melden Sie 2 verwandeln die Koloniegröße Werte (Integrated Dichte oder Kolonie Bereich, hier Spalte "IntDenBackSub & #8221; Ergänzende von Tabelle 1 verwendet wurde).
HINWEIS: Wertverteilung wird wie in 3A zu suchen. - Normalisierung dieser Werte durch Subtrahieren des Medianwerts von jeder Platte.
Anmerkung: Dieser Schritt steuert für Platten Vorspannung, die aus ungleichen Medienmenge bzw. Variation der automatischen Bildaufnahme ergeben können, und reduziert die inter replicate Varianz (3B). - Überprüfen Sie, ob Wiederholungen miteinander korrelieren (3C), um die Reproduzierbarkeit der Versuche zu bewerten.
- Zur Unterscheidung Interaktion von nicht-wechselwirkenden Köder-Beute-Paare, legen großen Vertrauen Schwelle, die den 95. Perzentil der Verteilung der L-DHFR-F [3] Kontrollen.
HINWEIS: Bei diesem Versuch, dies entspricht 3,39 (3D). Alternativ kann ein Schwellwert auf der Grundlage der Überlappung mit bekannten physikalischen Interaktoren, wie sie in der BioGrid 35 gemeldet werden.Siehe Diskussion für weitere Details. - Für alle Köder, Beuten-Filter identifiziert wie in falsche positive Wechselwirkungen in DHFR-PCA-Bildschirme beteiligt (siehe Diskussion für weitere Details) und Ergänzende Tabelle 2 (in der Spalte "1" identifiziert "gefiltert") aufgeführt.
- Der Mittelwert der normalisierten Log2 Koloniegrößen der drei Wiederholungen jeder Interaktion (Spalte "Durchschnitt" im Ergänzungs Tabelle 2).
7. Überprüfung der Physical Interacter Mit Kleines Experimente
Hinweis: alle PPI von besonderem Interesse, die eine Punktzahl über oder dicht an der angewendet Schwelle kann unter Verwendung des DHFR-PCA-Test in einer kleinen Versuchsanordnung mit einem Wachstumstest auf festem oder flüssigem MTX Medium geprüft. Die folgenden Schritte zeigen die Prozedur manuell zu erstellen diploiden PCA Stämme und führen vor Ort Tests auf MTX Medium. Der Versuchsleiter sollten diese Schritte für alle notwen führeny Kontrollen (Bait-DHFR F [1,2] x L-DHFR-F [3], Zipper-Linker-DHFR diploiden Stämme und Linker-DHFR diploide Stamm).
- Platte 2-3 ul der Glycerolstammlösung des Köders Belastung in 1.1.2.6 generiert), die L-DHFR-F [3] Kontrollstamm, der Zipper-Linker-DHFR und Linker-DHFR diploiden Stämme auf YPD + Nat, YPD + HygB und zweimal YPD + Nat + HygB Medien auf.
- Rufen Sie die Beute von Interesse in der DHFR-F [3] Sammlung und folgen Sie den Anweisungen in Schritt 1.1.1, aber Streifen die Belastung auf YPD + HygB Medium statt YPD + Nat Medium.
- Führen Sie eine diagnostische PCR wie in 1.1.2.4, um die DHFR F bestätigen [3] Fusion an der Beute Locus und Folge das Produkt.
- Impfen Sie 1 ml Flüssigkeit YPD-Medium mit den haploiden Stämmen zu paaren (Bait x Prey, Bait x L-DHFR-F [3] Steuerung) und wachsen mindestens zwei Tage bei 30 ° C, damit die Diploide zu bilden.
- Wählen Diploiden durch Ausstreichen 4-5 ul der Kultur in 7,4 auf festem YPD + HygB + Nat Medium. Wachsen 2 Tage bei 30 ° C.
- Select eine isolierte Kolonie und über Nacht wachsen in Flüssigkultur (1 ml), um den Wachstumstest durchzuführen.
- Bereiten MTX und DMSO (gleichen Inhaltsstoffe wie MTX Medium, aber ohne MTX) Platten am Tag vor dem Gebrauch. Siehe Schritt 4.11 für weitere Details.
- Führen Wachstumstest durch Spotten Reihenverdünnungen der verschiedenen Kulturen auf die Kontrolle (DMSO) und Selektionsplatten (MTX).
- Verdünnen Vorkulturen auf OD = 1.
- Führen fünffache Verdünnungen (bis zu einem Verdünnungsfaktor von 625) in einer sterilen 96-Well-Platte.
- Spot 4 ul jeder Verdünnung auf der PCA Medien (DMSO und MTX).
- Inkubation bei 30 ° C in Plastiktüten, um ein Austrocknen zu verhindern.
- Bildplatten aus den Tagen 1 bis 7 der Inkubation mit dem Roboterplattform oder einen normalen Digitalkamera.
Ergebnisse
Ergänzende Tabelle 2 ist ein Beispiel für repräsentative Ergebnisse mit dem Hefeprotein Nup82 verschmolzen mit dem DHFR-F [1,2] Fragment als Köder erhalten. Die mit der L-DHFR F definierte Schwelle [3] Steuerelemente können als empirische Schwelle verwendet werden, um ein hohes Vertrauen Hits (Abbildungen 3D u 3E) zu bestimmen. Alternativ kann der Core-Ranking verwendet werden, um Gene Ontology Anreicherungen führen oder andere funktionelle Analysen 36 basierend auf Goldstandards 37. Die bekannten physikalischen Interaktoren der Köder kann von Datenbanken wie BIOGRID 35 abgerufen und überlagert von den Daten (3E und 3F). In diesem Beispiel sind fünf von acht hohen Vertrauens Treffer wurden zuvor als Interaktoren Nup82 gemeldet und zwei sind Teil der Nup82 Subkomplex, Nup116 und Nup159 (Abbildung 3F 3G). Das andere Mitglied des Komplexes, nsP1, keinerlei Wechselwirkung nicht zeigention in unserem Experiment. Zwei Beute, Ade17 und TEF2 (nicht in 3F gezeigt), hatten Werte über der Fest Schwelle angewendet, aber mit Fehlmeldungen, wie sie mit fast jeder Köderprotein in PCA-Bildschirme haben wir (unveröffentlichte Ergebnisse) durchgeführt zu interagieren sind. Auf der anderen Seite kann Pex30 eine neuartige physikalische Interaktor der Nup82 stellen, und wir waren in der Lage, diese Interaktion mit DHFR-PCA bei niedrigen Durch (Abbildung 3G) zu bestätigen. Pex30 eine peroxisomale Membranprotein und ein paar direkte Wechselwirkungen zwischen dem Kernporenkomplexes (NPC) und dieser Organelle gemeldet. Ein Zwei-Hybrid-Screen identifiziert beiden anderen NPC Protein Nup53 und ASM4 (Nup59), als physikalische Interaktoren von Pex30 38 und eine genetische Interaktion zwischen Pex30 und Nup170 wurde berichtet, 39. Zwei weitere Interaktionspartner erkannt Nup120 und Nup85 (Abbildung 3F 3G) sind nicht Teil der Nup82 Unterkomplex, welches die Fähigkeitdes DHFR-PCA zu Wechselwirkungen innerhalb und zwischen den Unterkomplexe in größeren Komplexen 11 zu erkennen.
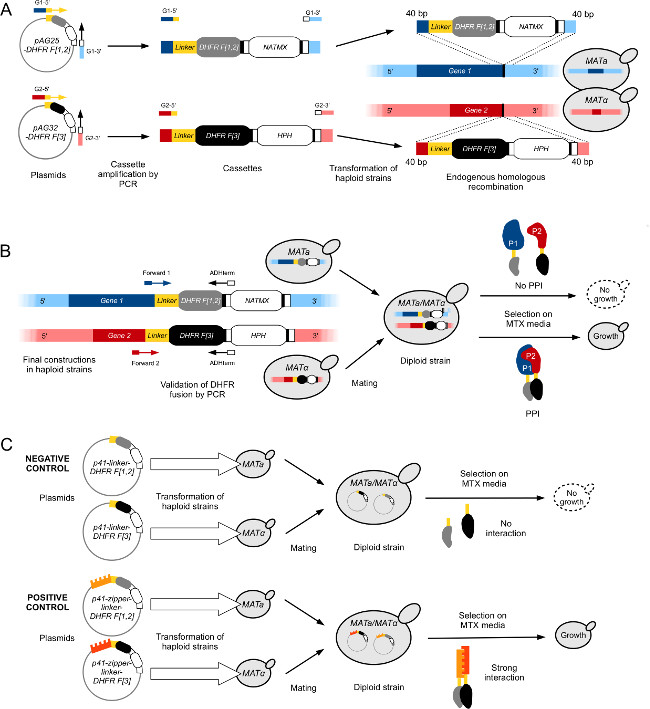
Abb. 1: Entwicklung Hefestämme für Hochdurchsatz-DHFR-PCA (. Figur aus Leducq et al angepasst 2012 33) (A) Bau von haploiden MATa und MAT α Stämme Gene1 (G1) und Gene2 (G2) mit dem DHFR-Sicherung F [1,2] -NatMX und das DHFR F [3] -HPH Kassetten auf. Kassetten aus den Plasmiden verstärkt pAG25-DHFR F [1,2] und pAG32-DHFR-F [3] mit Vorwärtsprimer G1-5 'und G2-5' und Reverse-Primer G1-3 'und G2-3', und dann in das Genom am 3'-Ende des Zielgens durch homologe Rekombination inseriert. Die resultierenden Proteine, P1 und P2 sind jeweils mit dem D verschmolzenHFR F [1,2] Fragment (MATa) und das DHFR F [3] Fragment (MATa) über einen flexiblen Linker. (B) Überprüfung der Konstruktion in (A) wird durch Sequenzierung der Verbindungsstellen zwischen Gene1 und Gene2 des ORFs und durch die DHFR-Kassetten. Die konstruierten PCA Stämme von gegnerischen Paarungstypen werden dann zusammengefügt, um einen diploiden zu bilden. Diploiden Stämme wachsen auf MTX Medium, wenn die zwei komplementären DHFR-Fragmente werden durch eine Wechselwirkung zwischen P1 und P2, die die Aktivität des Enzyms DHFR rekonstituiert in unmittelbare Nähe gebracht werden. (C) Konstruktion von Steuer diploiden Stämme PCA für DHFR-PCA-Bildschirme. Die Negativkontrollen (L-DHFR) durch separat haploiden MATa und MATa Stämme mit Plasmiden p41-Linker-DHFR F [1,2] und p41-Linker-DHFR-F [3] 11 bzw. Umwandlung errichtet. Die beiden Stämme werden gepaart, was zu einer negativen Kontrolle diploiden Stamm, in dem das DHFR-Fragmente sind nichtsich gegenseitig (oben) zu ergänzen. Positive Kontrollen werden mit dem gleichen Ansatz wie für die Negativkontrollen hergestellt, aber die Plasmide in haploiden Stämmen (p41-Reißverschluss-Linker-DHFR F [1,2] (p41-ZL-DHFR F [1,2]) und p41 verwandelt -Reißverschluss-Linker-DHFR F [3] (p41-ZL-DHFR F [3])) zwei parallel GCN4-Leucin-Zipper-Fragmente mit den komplementären DHFR Fragmente fusioniert, was zu einem starken und konstitutiven Interaktion, die DHFR-Aktivität rekonstituiert führt (bottom ). Bitte klicken Sie hier, um eine größere Version dieses Bild anzuzeigen.
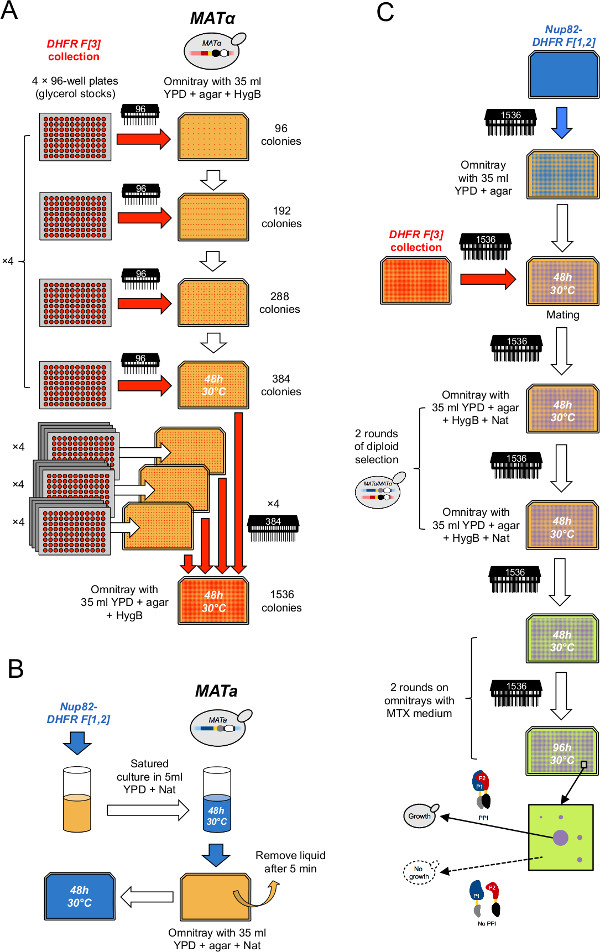
Abbildung 2: Hochdurchsatz-DHFR-PCA-Verfahren (Figur aus Leducq et al 33 angepasst.) (A) Die MAT α DHFR F [3] Sammlung. befindet sich in einem 1536-Format durch zwei aufeinanderfolgende Runden Kondensation kondensiert. Zunächst werden Glycerolstammlösung Platten von einer Gruppe von vier auf selektiven YPD + HygB Medium in einem 384-Format mit dem 96 Pin-Tool kombiniert. Zweitens werden 384 Arrays von Gruppen von vier auf selektiven YPD + HygB Medium in einem 1536-Format mit Hilfe der 384 Pin-Tool kombiniert. (B) Zellrasen der MATa PCA Köder Belastung werden, indem eine gesättigte Kultur des Köders Belastung in vorbereitet selektive YPD + Nat mittel- und Galvanisieren der Kultur auf einem YPD + Nat OmniTray. (C) Diese Rasenflächen verwendet werden, um den Köder Belastung mit dem DHFR-F [3] Sammlung auf YPD-Medium passen. Die Zellen werden nacheinander zweimal auf YPD + HygB + Nat-Medium überführt, um für Diploiden und zweimal auf MTX mittlere bis PCA durchführen wählen. Wachstum nur auf MTX Medium beobachtet, wenn die DHFR-Fragmenten ergänzen einander nach einer Wechselwirkung zwischen Köder und Beute-Proteine werden.e.jpg "target =" _ blank "> Bitte klicken Sie hier, um eine größere Version dieses Bild anzuzeigen.
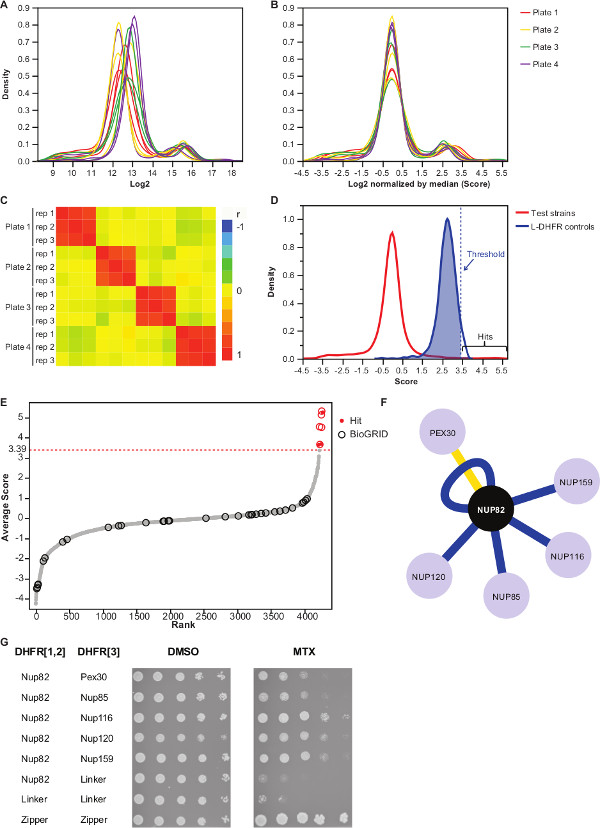
Fig. 3:. Datenanalyse durch die Schritte der Normalisierung, die Bestimmung einer Signifikanzschwelle und Identifizierung von positiven Wechselwirkungen (A) Dichteverteilung der Koloniegröße auf jeder Platte (log 2) (B) Normalisierung durch die Median jedes Bildes korrigiert Vorspannungen mit Platte-zu-Platte Wirkungen verbunden. (C) Heatmap zeigt die Spearman Korrelationskoeffizient zwischen den Platten, bestätigt die Reproduzierbarkeit des Verfahrens. (D) Verteilung der Noten für die getesteten PPI und die L-DHFR-F [3] Kontrollen. Eine harte Schwelle kann auf die 95. Perzentil der eingestellt werden die L-DHFR-F [3] Verteilung an Hoch Vertrauen PPI identifizieren (durch eine gestrichelte vertikale vertretenLinie). (E) Rang um Verteilung der Durchschnittsnote von jeder Beute. Zuvor berichtete, physische Interaktionspartner von Nup82p im BIOGRID 35 sind durch Kreise gekennzeichnet und die in dieser Studie berichtet, sind durch rote Punkte gekennzeichnet. Die in (D) definierte Schwelle wird als gestrichelte Linie dargestellt. (F) Netzwerk, das die Hoch Vertrauen PPI in dieser Studie identifiziert. Blaue Kanten zeigen bereits berichtet körperliche Interaktoren (BIOGRID 35) und gelben Rändern zeigen einen zuvor unbekannten Interaktion mit Pex30. (G) Spot-Verdünnungsanalyse der PCA diploiden Stämme mit Nup82-DHFR F [1,2] und Beute-DHFR-F [3 ] Paare als physische Interaktionspartner von Nup82p in der vorliegenden Studie identifiziert. Wachstumstest wurde in DMSO-Medium (MTX Lösungsmittel, links) und MTX-Medium (rechts) durchgeführt. Negative Kontrollen aus Nup82-DHFR F [1,2] - Linker-DHFR-F [3] und Linker-DHFR F [1,2] - Linker-DHFR-F [3] und eine positive Kontrolle, bestehend aus ter starke Wechselwirkung zwischen zwei Leucin-Zipper-Einheiten (Reißverschluss-DHFR F [1,2] - Reißverschluss-DHFR-F [3]) waren eingeschlossen. Das Zellwachstum in MTX Medium besser als die Negativkontrollen sollten als eine physikalische Wechselwirkung interpretiert werden. Bitte klicken Sie hier, um eine größere Version dieses Bild anzuzeigen.
| Aminosäure | Menge (g) |
| Adeninsulfat * | 0.2 |
| L-Tryptophan | 0.4 |
| L-Tyrosin | 0.3 |
| L-Phenylalanin | 0,5 |
| L-Glutaminsäure (Mononatriumsalz) | 1.0 |
| L-Asparagin | 1.0 |
| L-Valin | 1.5 |
| L-Threonin | 2.0 |
| L-Serin | 3,75 |
| Uracil | 0.2 |
| L-Histidin-HCl | 0.2 |
| L-Arginin HCl | 0.2 |
| L-Methionin * | 0.2 |
| L-Lysin * | 0.2 |
| L-Leucin | 0.6 |
| * Zurückgezogen beim Ausführen Standard PCA (dem in diesem Protokoll), können aber auch für andere Zwecke zugegeben werden. | |
Tabelle 1:. Zusammensetzung des 10X -Lys / met / ade Drop-out in der MTX Mittelmengen sind für einen Liter 10x Drop-out. Sternchen bezeichnen die Verbindungen, die für die Standard-MTX Medium entzogen werden sollte, aber das kann für andere Zwecke zugegeben werden.
Ergänzende. Tabelle 1: Kombinierte Daten aus den 12 Testplatten In Tabelle 1 sind die verketteten data von der ImageJ-Analyse unter Verwendung eines kundenspezifischen ImageJ Skript jede Zeile entspricht einer einzelnen Position auf jedem 1536-Arrays. Darüber hinaus hat jede Zeile mit der Information über die Bilddatei, DHFR Sammelteller annotiert, zu replizieren, ORF und Proteinnamen.
Ergänzende Tabelle 2:. Durchschnittliche normalisierte Wachstums pro Stamm Tabelle 2 enthält die durchschnittliche Log 2 normalisierte Wachstums für jeden Stamm der Sammlung zusammen mit der Standardabweichung. Gefiltert Beute, Hits und bekannter physikalischer Interaktionspartner werden in separaten Spalten identifiziert.
Diskussion
Wir beschreiben ein Protokoll auf Basis des DHFR-PCA-Test ermöglicht die systematische Ermittlung von physikalischen Interaktoren für einen bestimmten Köderprotein bei hohem Durchsatz. Dieses Protokoll kann durch Screenen auf mehr Köder ausgelegt werden, und dies in jeder gewünschten Höhe der Replikation. Wir demonstrieren die Zuverlässigkeit dieses Protokoll durch die Identifizierung von physischen Interaktionspartner für eine Köderprotein in der Kernporenkomplex beteiligt: Nup82. Unsere Analyse aktivieren, damit Sie fünf bereits berichtet Interaktoren und einer zuvor unbekannten Interaktor (3F und 3G) zu finden, Hervorhebung der Fähigkeit des Verfahrens, um die Hefe Protein Interaktom studieren.
Das hier beschriebene Protokoll enthält einige wichtige Schritte, denen der Experimentator sollte darauf achten. Wir empfehlen 1) Stellen Sie sicher, dass der Köder DHFR F [1,2] Fusion korrekt (Abbildung 1B) ist; dies kann durch Sequenzierung der Konstruktion und measur erreicht werdening korrekte Proteinexpression mit Hilfe eines Anti-DHFR F [1,2] oder Anti-DHFR F [3] Antikörper; 2) Vor dem Beginn des Bildschirms ist, wird empfohlen, um zu überprüfen, ob jeder Köder von Interesse aufweist Promiscuous Wechselwirkungen in PCA-Bildschirme. Dies kann durch die Steuerung Bildschirme mit Ködern mit den entsprechenden L-DHFR-Steuerung oder durch manuelle Paarung den Köder mit dem entsprechenden L-DHFR-Steuerung und Durchführung einer Wachstumstest in MTX Medium gekreuzt erfolgen. 3) Die Platten sollten gegossen werden der Tag, bevor sie verwendet werden, so dass die Feuchtigkeit optimal für Zellhaftung auf der Agar-Oberfläche während des Druckvorganges; 4) Quellplatten sollten nicht mehr als vier Mal verwendet, um genügend Zellen auf der Zielplatte zu übertragen. Eine Erhöhung der Anzahl der Kopien der Zielplatte durch aufeinanderfolgende Schritte der Expansion (-> 16 Kopien -> 64 Kopien zB 4 Kopien) erfolgen. Alternativ können die Zellen an unterschiedlichen Positionen auf dem Rasen oder in der Kolonie zwischen verschiedenen Replikationsrunden abgeholt werden; 5) Sind mehrere positions werden nach der diploiden Auswahl (en), stellen Sie sicher, dass die Quellplatten wurden nicht zu oft in der Paarungsschritt verwendet fehlt (Schritte von 4,5 bis 4,7); 6) Stellen Sie sicher, dass die MTX Medium enthält alle wesentlichen Bestandteile in den richtigen Konzentrationen. Wenn nämlich kein Wachstum bei allen auf der MTX Medium beobachtet, es sein kann, entweder weil keine Interaktion durch PCA nachweisbaren für die Proteine von Interesse oder weil MTX Medium wurde nicht richtig vorbereitet. Um sicherzustellen, daß das Medium erlaubt das Wachstum von Stämmen, welche DHFR-Fragmenten Komplementierung kann eine konstitutive Wechselwirkung an leeren Positionen der Sammlung hinzugefügt werden und als eine positive Kontrolle, wie DHFR-Fragmenten an Leucin-Zipper-Einheiten 33 (1C) fusioniert werden. Parallelversuche mit den Linker-DHFR-Fragmente oder die Reißverschluss-Linker-DHFR-Fragmenten wird es ermöglichen, zwischen den Bedingungen, die alle Zellen wachsen lassen (low MTX-Konzentration oder Köderprotein, die zu falsch positiven Wechselwirkungen machen neigen zu unterscheiden, wie described unten) und Bedingungen, die das Wachstum von allen Stämmen (MTX-Konzentration zu hoch oder wesentlicher Bestandteil in das Medium fehlt) zu verhindern; 7) Da PCA wird durch aufeinanderfolgende Runden von Replikationen von einem Medium zu einem anderen durchgeführt wird, kann eine Kreuzkontamination zwischen den Belastungen zwischen den verschiedenen Platten auftreten, wenn zum Beispiel der Pin-Werkzeug nicht richtig zwischen Replikationsrunden und / oder das letzte Wasser sterilisiert Bad (dh feuchte Station) in der Sterilisationsverfahren wird von Kolonien der vorherigen Replikationsrunden kontaminiert. Mehrere Positionen auf den Arrays sind leer und kann somit als Steuerpositionen, wo kein Wachstum zu beachten, um Kreuzkontaminationen zu erkennen sein werden.
Die Bildanalyse kann unter Verwendung von mehreren veröffentlichten Software (siehe Abschnitt 5 des Protokolls) oder ein benutzerdefiniertes Skript werden. In dieser Studie, die benutzerdefiniertes Skript führt die folgenden Schritte: 1) Das Skript subtrahiert Pixelwerte einem leeren Teller auf Werte von jeder Platte, um gemeinsam PixelRRECT für die Beleuchtung Vorurteile. 2) Das Skript wandelt jedes grundkorrigierte Bild in dual mit einem Pixelwert Schwelle von 10. 3) Für je 1536 Positionen jeder Platte, durch Überlagerung ein Rechteck auf den Rand Kolonien bestimmt, wird das Skript ImageJ "Analyze Partikel ... "Funktion in einer Kreisauswahl. Die kreisförmige Auswahl mit einem Radius gleich dem Abstand zwischen zwei Positionen minus 10 Pixel eingestellt. 4) Das Skript wählt die am besten Teilchen von der Mitte der Auswahl und bestätigt sie als Kolonie, wenn die Lage nicht mehr als die Hälfte des Intervalls zwischen zwei Kolonien von der Mitte der Markierung. 5) Das Skript misst Pixelwerte des ausgewählten Teilchen auf dem Hintergrund-korrigierten Bild. 6), um alle verbleibenden Hintergrundbeleuchtung spannt weiter zu korrigieren, subtrahiert das Skript den Mittelwert aller Pixel von der Kreisauswahl, die nicht Teil eines Teilchens in Pixelwerte der Kolonie waren. Die Summe dieser korrigierten Pixelwerts, in der Spalte "IntDenBackSub" des Ergänzungs Tabelle 1 gespeichert sind, werden als Maß für die Koloniegröße verwendet.
Ein kritischer Schritt in dem Analyseteil die Wahl der Signifikanzschwelle. Hier haben wir uns für einen Schwellenwert basierend auf der Verteilung des negativen L-DHFR F [3] steuert, aber je nach dem Ziel auf dem Bildschirm kann eine solche Schwelle zu streng sein. Tatsächlich L-DHFR F [3] steuert überexprimiert werden (starke TEF-Promotor), so dass die komplementären Fragmente können spontan ergänzen einander und sind deshalb nicht repräsentativ für die Expression der meisten Proteine. Dies wird durch die Tatsache, dass die Verteilung der L-DHFR F [3] steuert ist höher als der Mittelwert der Hintergrund-Wachstum (Figur 3D) hervorgehoben. So dass Wechselwirkungen mit Noten unterhalb dieser Schwelle strengere, aber das sind eindeutig außerhalb des Hinterwachstumsverteilung kann als mögliche Treffer, die darstellen können, zum Beispiel, vorübergehende oder schwache inter betrachtenAktionen. Diese können weiter untersucht werden und eine Kreuzvalidierung, wenn beispielsweise die beiden Proteine nicht ausreichte, um die spontane Komplementation der DHFR-Fragmente, wie die L-DHFR Steuerelemente ermöglichen kann ausgedrückt. Alternativ könnte man eine Signifikanzschwelle bezogen auf den Anteil der Überlappung mit berichteten physischen Interaktoren in Datenbanken wie BIOGRID 35, um den Anteil der wahren Positiven über falsche Positive zu maximieren. Im Gegensatz zu der Verwendung des L-DHFR Verteilung Diese Alternative kann nicht immer möglich sein, wenn, zum Beispiel, ist die Anzahl von bekannten physikalischen Interaktoren nicht ausreichend hoch. Darüber hinaus hat die Wahl der Signifikanzschwelle einen Einfluss auf den Anteil der falsch-positive und falsch-negative Ergebnisse in der letzten Datensatz. Tatsächlich, wie jede andere PPI-Nachweistest, Fehlalarme aus unspezifischen Wechselwirkung eines Proteins mit dem DHFR-Fusionsprotein führen, wenn beispielsweise sehr reichlich das Protein wie zuvor erwähnt. Diesewird durch die Tatsache, dass einige Beute systematisch alle Köder-Proteine interagieren in PCA-Bildschirme und so veranschaulicht, müssen vor der Analyse 11 (zB TEF2 und Ade17 und Ergänzungs Tabelle 2) entfernt werden. Um dieses Problem zu, ein Steuer PCA Bildschirm der beiden Sammlungen gegenüber dem entsprechenden L-DHFR Kontrolle zu umgehen (F [1,2] oder F [3]), um Köder und Beute zeigen spontane DHFR-Fragmente Ergänzung in den spezifischen Bedingungen durchgeführt werden, zu identifizieren auf jedem Bildschirm. Außerdem Durchführung eines Gene Ontology Bereicherung Analyse kann das Vertrauen in die Daten zu erhöhen, wenn die Funktion eines bestimmten Köder bekannt ist. Andererseits kann DHFR-PCA zu falschen Negativen aus mehreren Gründen: 1 zu ergeben) nicht alle Proteine können an die DHFR-Fragmenten, da diese die Proteine zu destabilisieren oder deren Lokalisation verändern, wenn beispielsweise verschmolzen werden, das DHFR-Fusion an die C-Terminus stört eines Lokalisierungssignals; 2) DHFR Rekonstitution in einigen zellulären Kompartimenten may keine Folsäure zu erzeugen, wenn zum Beispiel ein wesentlicher Vorläufer für Folat-Synthese ist nicht verfügbar; 3) C-Termini, ob innerhalb einer Distanz von 8 nm für DHFR Komplementation auftreten 11. So kann eine bekannte Interaktion nicht erkannt werden, wenn ihre C-Termini sind nicht nah genug im Raum. Dies wird hier durch die Tatsache, dass ein großer Anteil der Nup82 physikalische Wechselwirkungen in Datenbanken, von denen die meisten indirekten gemeldet werden, wurden in unserem Test nachgewiesen exemplifiziert. Ähnlich wird membrangebundene Proteine, für die die C-Termini sind in trans relativ zu der Membran nicht zu der DHFR-Fragmenten Komplementierung und nicht erkannt wird 11. Einschränkungen 1) und 3) kann relativ einfach durch Fusionieren des DHFR-Fragments mit dem N-Terminus des Proteins, umgangen werden. Andernfalls kann es zu verhindern, mit einem Lokalisierungssignal in der Nähe des C-Terminus beeinträchtigen und ermöglichen eine Interaktion zwischen Membranproteine, deren N- und C-Terminus zu erkennen sind in cis gegenauf die Membran.
Einige Herausforderungen bleiben in der Studie von PINs (in 2,3 überprüft). Die Karten von PINs bisher produziert wurden weitgehend in einem einzigen experimentellen Bedingungen für die einzelnen Arten beschrieben worden und bieten somit eine einzelne Momentaufnahme, wie Protein-Netzwerken organisiert werden könnte. Es besteht daher ein Bedarf an der Erforschung der anderen experimentellen Bedingungen, wie PINs können als Reaktion auf Umweltveränderungen, bestimmte Reize, in die Entwicklung oder die folgenden Mutationen reorganisiert werden. Diese Herausforderungen werden durch die Entwicklung neuer Technologien für die Abfrage von PPI in Echtzeit, in lebenden Zellen und durch Anpassung der aktuellen Techniken, so dass sie von einer größeren Gemeinschaft von Laboratorien verwendet werden, überwunden werden. Als quantitatives Verfahren, das Änderungen in der Menge des verwendeten DHFR Komplementation erfassen kann Komplexe 27 kann DHFR-PCA geeignet ist, diese Herausforderungen zu überwinden und wurde verwendet, um zu untersuchen, wie EPI durch einen DNA-schädigenden Mittel 22 sind betroffen , Chemikalien 25. Gendeletionen 23,26 oder in anderen Hefearten und ihre Hybriden 33. Erforschung dieser neuen Dimensionen wird mehr und mehr an Bedeutung gewinnen, um die Dynamik der PIN offenbaren.
Offenlegungen
Ein Teil der Open-Access-Publikationskosten für diesen Artikel wurden von S & P Robotics bezahlt.
Danksagungen
Diese Arbeit wurde vom Canadian Institute of Health Research (CIHR) Gewährt 191.597, 299.432 und 324.265, unterstützt ein Natur- und Ingenieurwissenschaften Research Council of Canada Discovery-Zuschuss und ein Human Frontier Science Program Stipendium CRL. CRL ist ein CIHR New Investigator. Guillaume Diss wird von einem PROTEO Gemeinschaft unterstützt. Samuel Rochette wird von NSERC und FRQNT Stipendien unterstützt.
Materialien
| Name | Company | Catalog Number | Comments |
| BioMatrix Robot, Bench-top Configuration | S&P Robotics Inc. | BM5-BC | |
| 96-format Pin-tool | S&P Robotics Inc. | PH-96-10 | Standard 96-format Pin-tool with 96 high-precision floating pins |
| 384-format Pin-tool | S&P Robotics Inc. | PH-384-10 | Standard 384-format Pin-tool with 384 high-precision floating pins |
| 1536-format Pin-tool | S&P Robotics Inc. | PH-1536-05 | Custom 1536-format Pin-tool with 0.5mm high-precision floating pins |
| Automated imaging module | S&P Robotics Inc. | IMG-02 | |
| Methotrexate | Bioshop Canada Inc. | MTX440 | CAUTION: toxic compound |
| Hygromycin B | Bioshop Canada Inc. | HYG003 | |
| Nourseothricin dihydrogen sulfate | Werner BioAgents | 5010000 | |
| Yeast-Interactome Collection | Thermo Scientific | YSC5849 | |
| Omni Tray w/lid sterile | Thermo Scientific | 242811 | |
| Anti-DHFR F[1,2] antibody | Sigma-Aldrich | D1067 | |
| Anti-DHFR F[3] antibody | Sigma-Aldrich | D0942 |
Referenzen
- Alberts, B. The cell as a collection of protein machines: preparing the next generation of molecular biologists. Cell. 92, 291-294 (1998).
- Diss, G., et al. Integrative avenues for exploring the dynamics and evolution of protein interaction networks. Curr Opin Biotechnol. 24, 775-783 (2013).
- Vidal, M., Cusick, M. E., Barabasi, A. L. Interactome networks and human disease. Cell. 144, 986-998 (2011).
- Hu, P., et al. Global functional atlas of Escherichia coli encompassing previously uncharacterized proteins. PLoS biology. 7, e96 (2009).
- Arifuzzaman, M., et al. Large-scale identification of protein-protein interaction of Escherichia coli K-12. Genome Res. 16, 686-691 (2006).
- Rajagopala, S. V., et al. The binary protein-protein interaction landscape of Escherichia coli. Nature biotechnology. 32, 285-290 (2014).
- Arabidopsis-Interactome-Mapping-Consortium. Evidence for network evolution in an Arabidopsis interactome map. Science. 333, 601-607 (2011).
- Babu, M., et al. Interaction landscape of membrane-protein complexes in Saccharomyces cerevisiae. Nature. 489, 585-589 (2012).
- Krogan, N. J., et al. Global landscape of protein complexes in the yeast Saccharomyces cerevisiae. Nature. 440, 637-643 (2006).
- Gavin, A. C., et al. Functional organization of the yeast proteome by systematic analysis of protein complexes. Nature. 415, 141-147 (2002).
- Tarassov, K., et al. An in vivo map of the yeast protein interactome. Science. 320, 1465-1470 (2008).
- Uetz, P., et al. A comprehensive analysis of protein-protein interactions in Saccharomyces cerevisiae. Nature. 403, 623-627 (2000).
- Guruharsha, K. G., et al. A protein complex network of Drosophila melanogaster. Cell. 147, 690-703 (2011).
- Li, S., et al. A map of the interactome network of the metazoan C. elegans. Science. 303, 540-543 (2004).
- Rual, J. F., et al. Towards a proteome-scale map of the human protein-protein interaction network. Nature. 437, 1173-1178 (2005).
- Landry, C. R., Levy, E. D., Abd Rabbo, D., Tarassov, K., Michnick, S. W. Extracting insight from noisy cellular networks. Cell. 155, 983-989 (2013).
- Berggard, T., Linse, S., James, P. Methods for the detection and analysis of protein-protein interactions. Proteomics. 7, 2833-2842 (2007).
- Wodak, S. J., Vlasblom, J., Turinsky, A. L., Pu, S. Protein-protein interaction networks: the puzzling riches. Current opinion in structural biology. 23, 941-953 (2013).
- Fields, S., Song, O. A novel genetic system to detect protein-protein interactions. Nature. 340, 245-246 (1989).
- Dunham, W. H., Mullin, M., Gingras, A. C. Affinity-purification coupled to mass spectrometry: basic principles and strategies. Proteomics. 12, 1576-1590 (2012).
- Michnick, S. W., Ear, P. H., Landry, C., Malleshaiah, M. K., Messier, V. A toolkit of protein-fragment complementation assays for studying and dissecting large-scale and dynamic protein-protein interactions in living cells. Methods Enzymol. 470, 335-368 (2010).
- Rochette, S., Gagnon-Arsenault, I., Diss, G., Landry, C. R. Modulation of the yeast protein interactome in response to DNA damage. Journal of proteomics. 100, 25-36 (2014).
- Diss, G., Dube, A. K., Boutin, J., Gagnon-Arsenault, I., Landry, C. R. A systematic approach for the genetic dissection of protein complexes in living cells. Cell Rep. 3, 2155-2167 (2013).
- Gagnon-Arsenault, I., et al. Transcriptional divergence plays a role in the rewiring of protein interaction networks after gene duplication. Journal of proteomics. 81, 112-125 (2013).
- Schlecht, U., Miranda, M., Suresh, S., Davis, R. W., St Onge, R. P. Multiplex assay for condition-dependent changes in protein-protein interactions. Proceedings of the National Academy of Sciences of the United States of America. 109, 9213-9218 (2012).
- Lev, I., et al. Reverse PCA, a systematic approach for identifying genes important for the physical interaction between protein pairs. PLoS Genet. 9, e1003838 (2013).
- Freschi, L., Torres-Quiroz, F., Dube, A. K., Landry, C. R. qPCA: a scalable assay to measure the perturbation of protein-protein interactions in living cells. Mol Biosyst. 9, 36-43 (2013).
- Pelletier, J. N., Campbell-Valois, F. X., Michnick, S. W. Oligomerization domain-directed reassembly of active dihydrofolate reductase from rationally designed fragments. Proceedings of the National Academy of Sciences of the United States of America. 95, 12141-12146 (1998).
- Gietz, R. D., Woods, R. A. Transformation of yeast by lithium acetate/single-stranded carrier DNA/polyethylene glycol method. Methods Enzymol. 350, 87-96 (2002).
- Schuldiner, M., Collins, S. R., Weissman, J. S., Krogan, N. J. Quantitative genetic analysis in Saccharomyces cerevisiae using epistatic miniarray profiles (E-MAPs) and its application to chromatin functions. Methods. 40, 344-352 (2006).
- Schneider, C. A., Rasband, W. S., Eliceiri, K. W. NIH Image to ImageJ: 25 years of image analysis. Nature methods. 9, 671-675 (2012).
- Wagih, O., Parts, L. gitter: A Robust and Accurate Method for Quantification of Colony Sizes From Plate Images. G3 (Bethesda). 4 (3), 547-552 (2014).
- Leducq, J. B., et al. Evidence for the robustness of protein complexes to inter-species hybridization. PLoS Genet. 8, e1003161 (2012).
- . . Development Core Team: A language and environment for statistical computing. , (2008).
- Stark, C., et al. BioGRID: a general repository for interaction datasets. Nucleic Acids Res. 34, D535-D539 (2006).
- Vinayagam, A., et al. Protein complex-based analysis framework for high-throughput data sets. Science signaling. 6, rs5 (2013).
- Jansen, R., Gerstein, M. Analyzing protein function on a genomic scale: the importance of gold-standard positives and negatives for network prediction. Current opinion in microbiology. 7, 535-545 (2004).
- Ito, T., et al. A comprehensive two-hybrid analysis to explore the yeast protein interactome. Proceedings of the National Academy of Sciences of the United States of America. 98, 4569-4574 (2001).
- Costanzo, M., et al. The genetic landscape of a cell. Science. 327, 425-431 (2010).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten