Method Article
肉認証にペプチド応用のMRM質量分析法を用いて混合物中の種の決意と定量
要約
We present a protocol for identifying and quantifying the components in mixtures of species possessing similar proteins. Mass spectrometry detects peptides for identification, and gives relative quantitation by ratios of peak areas. As a tool food for fraud detection, the method can detect 1% horse in beef.
要約
We describe a simple protocol for identifying and quantifying the two components in binary mixtures of species possessing one or more similar proteins. Central to the method is the identification of 'corresponding proteins' in the species of interest, in other words proteins that are nominally the same but possess species-specific sequence differences. When subject to proteolysis, corresponding proteins will give rise to some peptides which are likewise similar but with species-specific variants. These are 'corresponding peptides'. Species-specific peptides can be used as markers for species determination, while pairs of corresponding peptides permit relative quantitation of two species in a mixture. The peptides are detected using multiple reaction monitoring (MRM) mass spectrometry, a highly specific technique that enables peptide-based species determination even in complex systems. In addition, the ratio of MRM peak areas deriving from corresponding peptides supports relative quantitation. Since corresponding proteins and peptides will, in the main, behave similarly in both processing and in experimental extraction and sample preparation, the relative quantitation should remain comparatively robust. In addition, this approach does not need the standards and calibrations required by absolute quantitation methods. The protocol is described in the context of red meats, which have convenient corresponding proteins in the form of their respective myoglobins. This application is relevant to food fraud detection: the method can detect 1% weight for weight of horse meat in beef. The corresponding protein, corresponding peptide (CPCP) relative quantitation using MRM peak area ratios gives good estimates of the weight for weight composition of a horse plus beef mixture.
概要
The European horse meat scandal of 2013, in which undeclared horse meat was found in a number of supermarket beef products1, highlights the need for testing methods capable of detecting and measuring food fraud in meat. Several technologies have been explored, especially enzyme-linked immunosorbent assay (ELISA) and DNA-based methods2. An alternative route, based on mass spectrometry, targets species-specific peptides which in turn arise from species-specific proteins. Here we outline one such peptide-based approach that offers both identification and relative quantitation of the adulterant species in a meat mixture3.
The protocol is framed in the context of red meats and the desire to determine the presence of one in another at the level of 1% by weight, the level considered by some to represent fraudulent food adulteration as opposed to contamination4. The method relies in the first instance on identifying a protein which is nominally 'the same' in all target meats. Myoglobin, the protein responsible for the red color of meat, is a good candidate since it is abundant, relatively heat tolerant and water soluble, and has been used for species determination of meat previously5,6. The myoglobins for beef (Bos Taurus), pork (Sus scrofa), horse (Equus caballus) and lamb (Ovis aries)3, for instance, are nominally the same, as required, but their sequences are not identical. Such groups of 'similar but different' proteins, like these four myoglobins, can conveniently be described as 'corresponding proteins'. The sequence differences in these four myoglobins are species-specific: for example, the full myoglobin proteins for beef and horse, P02192 and P68082 respectively, each comprise 154 amino acids with 18 sequence differences between the two. Subject to proteolysis using trypsin these proteins produce two sets of peptides, some of which are identical, and some which show one or more species-specific amino acid differences: corresponding proteins therefore give rise to corresponding peptides.
The CPCP approach, therefore, seeks first to identify proteins from two or more species where these proteins exhibit limited species-specific sequence variants. These are corresponding proteins. Following proteolysis, corresponding proteins give rise to peptides, some of which likewise display species-specific sequence variants inherited from the parent protein. These are corresponding peptides. The CPCP approach can be used to compare levels of two corresponding proteins in a mixed species sample by monitoring the levels of corresponding peptides.
The natural technology for the detection of known peptides is multiple reaction monitoring mass spectrometry, or MRM-MS7. Species-specific peptides yield precursor ions, which along with their mass spectrometry fragment ions, are easily itemized in advance by software tools. These lists are then used to instruct the mass spectrometer to record only specific precursor plus fragment ion pairs, called transitions. A particular target peptide is therefore identified not only by its retention time in the chromatography preceding the mass spectrometer, but also by a set of transitions sharing a common precursor ion. This is a highly selective means of detecting known peptides that makes efficient use of the mass spectrometer resource.
Other authors have used mass spectrometry to test for meat adulteration via peptide markers but from disparate proteins8-14. Using the corresponding proteins, corresponding peptides (CPCP) scheme, however, means experimental conditions can be optimized, aiding identification of the species in the mixture from known species-specific transitions. In addition, corresponding proteins and peptides will generally behave similarly in the extraction, proteolysis and detection stages. Since transition peak areas are quantitative and reproducible, ratios of peak areas arising from pairs of corresponding peptides from different species provide a direct estimate of the relative quantities of two meats in a mixture. In contrast, more traditional quantitation routes exploit calibrations based on reference materials to establish absolute quantitation14,15.
Though the protocol is outlined in the context of myoglobin and meat, proteins other than myoglobin could be used for identification and relative quantitation via the CPCP strategy in meat mixtures, though potentially with modifications to the protocol. In addition the strategy is also applicable to binary mixtures of other species sharing one or more corresponding proteins.
The starting point for the protocol is purified 'reference' myoglobin, which for some species can be purchased but which for others must be prepared by conventional size-exclusion chromatography. The procedure for preparing reference myoglobin is not included in the protocol, but is described elsewhere3. Software tools16 are used to list candidate peptides and transitions arising from myoglobins of interest. Each reference myoglobin is subjected to proteolysis and the resultant peptides analyzed by liquid chromatography electrospray ionization tandem mass spectrometry (LC-ESI-MS/MS) to discover which of the candidate precursor ions and transitions are most useful, and to determine the matching peptide retention times. The outcome of this stage is a revised list of target peptides with their transitions, suitable for species determination, and a list of CPCP pairs, suitable for relative quantitation. To test real meats, sample extractions are prepared then subjected to proteolysis to generate peptides both from myoglobin and other extraneous proteins. The myoglobin-based peptides are then monitored by LC-ESI-MS/MS based on their listed transitions. The species present in a mixture are identified by the transition peaks associated with marker peptides. Estimates of the relative amounts of two meats in a binary mixture are calculated using ratios of transition peak areas. A set of test mixtures of pairs of meats will allow the ratio of peak areas for a given pair of transitions to be checked and calibrated against actual mixtures.
プロトコル
1.タンパク質分解およびリファレンスミオグロビンの分析
- 参照ミオグロビンのタンパク質分解
- 精製された参照ミオグロビンのソリューション準備(範囲0.2から25 mMの重炭酸アンモニウム中の0.5mg / ml)を3。
- 2 mlの遠心チューブに移し、各サンプルの1mlアリコートを。
- 熱的に30分間95℃の熱ブロック中でサンプルを加熱することによって抽出されたタンパク質を変性させます。それが室温になるまで約15分間、試料を冷却します。混ぜた後、消化を高めるために、尿素(最終濃度0.5 M)30mgを追加します。
- トリプシンのタンパク質分解
- 25mMの重炭酸アンモニウム中のトリプシンの1mg / ml溶液を調製し、必要に応じて、氷上で保存。穏やかにボルテックスにより混合し、3で一晩タンパク分解するすることができ、その後、抽出したタンパク質の単位/ mg、最終的な酵素活性は420 BAEE(N-ベンゾイル-L-アルギニンエチルエステル塩酸塩)であるように、トリプシンの十分な量を追加します。7°C。
- タンパク質分解の完全性を実証するために、ドデシル硫酸ナトリウムポリアクリルアミドゲル電気泳動(SDS-PAGE)17を行います。
- ポストタンパク質分解サンプルの脱塩
- 2 V:水とVサンプル1に希釈します。
- 1%ギ酸を1ml添加することによって、カートリッジを平衡化し、次いで、1mlのメタノールを添加することにより、30mgのRPの材料で充填されたポリマー性逆相(RP)カートリッジを活性化します。
- 重力下でのカートリッジにサンプルをロードします。
- 重力下で5%のメタノール/ 1%ギ酸1mlで洗浄します。
- 5μlのジメチルスルホキシド(DMSO)があらかじめ入力2ミリリットルのマイクロチューブに重力下;:アセトニトリル/水の1ミリリットル(0.1%ギ酸V 90:10 v)を有するペプチドを溶出します。
- その後、250μlのアセトニトリル/水で残留物を再溶解、120分間遠心エバポレーターを用いて50℃の真空下で溶媒を取り外します(3:97 V:V; 0.1%ギ酸)。
- 移転低ボリュームオートサンプラーバイアルを解決。
注:サンプルは、液体クロマトグラフィー質量分析(LC / MS)分析の準備ができるまで4℃で保存することができます。
- MRMの移行リストの生成
- UniProtのデータベースとは別の肉のためのミオグロビンのシーケンスを探します。
- ペプチドと遷移予測ソフトウェア( 例えば 、スカイライン)の「ターゲット」ボックスにミオグロビンのシーケンスを入力します。必要であれば、そのフラグメントリストを明らかにするために、ペプチド上にカーソルを置きます。
- 「設定」をクリックし、「ペプチド設定」を選択します。入力消化の環境( すなわち、トリプシン)と切れ残りの数(0)。特に、追加のパラメータに必要な選択を入力し、ペプチド長(6から25)、N末端の除外(0)およびアミノ酸修飾(なし)を想定。
- 「設定」をクリックし、[トランジションの設定」を選択します。以下のための環境設定を選択しますLC / MS分析のために使用される機器の種類。
- [エクスポート]をクリックして、生成されたMRMトランジションとパラメータを含むスプレッドシートを作成するには、「トランジションリスト」を選択します。
- LC / MSによる分析
- オートサンプラーを高速液体クロマトグラフ(HPLC)、C18コアシェルHPLCカラム(10センチメートルX 2.1ミリメートル:バイナリ勾配のシステム(V水(A)及びアセトニトリル(B)、0.1%ギ酸vでそれぞれ)を設定MRM検出正エレクトロスプレーモードで操作三連四重極質量分析計に接続された、2.6ミクロンの粒子サイズ)。
- データ収集ソフトウェア( 例えば 、アナリスト)では、「ファイル」と「新規」を選択し、ポップアップボックスに「取得メソッド」をクリックし、次に「OK」をクリックします。
注:これは、新しいLC / MS法のセットアップを可能にする接続されたデバイスのリストが含まれている機器のメソッドエディタを開きます。 - 「バイナリポンプ」をクリックすると私流量値(300μL/分)、テーブルの勾配時間、22分かけて30%Bに3%Bのバイナリー勾配プロファイルを設定し、5分間の洗浄を23分で100%Bまで増加NPUTさらに6分間の初期条件と再平衡に戻る前に。
- 「オートサンプラー」をクリックし、注入量(5μl)を挿入します。 「ニードル洗浄サイクル」を有効にし、「ウォッシュ時間」(30秒)を入力し、「フラッシュポート」を選択します。
- 「カラム恒温コントローラ」と「カラムオーブンプロパティ」が「左温度」と「適温」(40°C)を設定中にをクリックします。
- 「質量分析計」をクリックして、原料ガスの条件を入力するには、[パラメータの編集」をクリックします。 「正」としての「MRM(MRM)」と「極性」としての「検索の種類」を選択します。 「期間の概要」に移動し、「継続時間」を入力し、LC分析ANの合計時間dは平衡(35分)。
- テーブル内を右クリックして、テーブルにこれらの列を追加するには、「デクラスタリングポテンシャル(DP)」と「衝突エネルギー(CP)」を選択します。遷移リストで作成された単一の肉種、(ステップ1.4.5を参照)のために、Q1、Q3、時間(ミリ秒)、ID、DPおよび遷移のすべてのためのCEの値を入力します。
注:時間(ミリ秒)は滞留時間を指す、質量分析計は、各遷移のスキャンに費やす時間は、の合計が3秒を超えてはなりません。 - 取得方法ファイル(ファイル拡張子.dam)を保存します。
注: - 1.5.8各肉種のために繰り返される必要が1.5.2手順。これは、以下の分析の準備のためにスクリーンモードで各肉種のための単一のメソッドファイルを作成します。 - データ収集ソフトウェアでは、「獲得」をクリックして、「平衡化」を選択します。開くボックスで、楽器の平衡を開始するために必要な取得方法を選択します。
- ARにサンプルバイアルを置きオートサンプラにackを。
- 「ファイル」をクリックし、「新規」、次に「取得バッチ」を選択します。 「サンプル」タブの「設定の追加」を選択し、 'サンプルを追加します」。分析すべきサンプル数を挿入し、「OK」をクリックします。 「買収」ボックスで、ドロップダウンメニューから、分析のために使用されるメソッドファイルを選択します。
- 表では、「プレートコード」を選択し、ドロップダウンメニューから適切なトレイ構成を選択します。右クリックを列ヘッダ「コードプレート」を選択し「ダウンフィル」で左クリックします。 「バイアルポジション」で行のオートサンプラー内の各サンプルの位置を入力します。
- 「データファイル」で取得のためにファイル名を入力し、列ヘッダーの後、左クリック、右クリックが続くと「ダウン塗りつぶし」を選択します。 「サンプル名」で分析すべき試料のそれぞれのアイデンティティを挿入します。取得バッチファイルとして保存します(ファイルEXTensionで.dab)。
- [送信]タブをクリックした後、LC / MSで分析する必要があるサンプルをハイライトします。 [送信]をクリックします。分析を開始するには、「サンプル開始 ''買収 'とをクリックします。
注:各取得方法は、単一の肉種のためのクロマトグラフの全長にわたってMRMトランジションをスキャンします。 MRM取得のための質量分析計の設定は、楽器の種類やペプチドに応じて変化します。 - データ閲覧ソフトを使用して生成されたデータファイルを表示します。 XIC(抽出イオン)にし、単一の前駆体(Q1)のためのすべてのフラグメント(Q3値)をハイライト表示し、ドロップダウンリストをクリックします。新しいペインには、それが唯一の選択遷移を示し開きます。
- これらは、単一のペプチドに対応するので、同時遷移のグループのための保持時間(R t)を記録します。
- それぞれについて、それぞれのペプチドにピークを割り当てるために、遷移のセットごとに、前の2つの手順を繰り返し肉種。
- 相対的定量に適した対応する対を形成マーカー一緒にそれらの保持時間で、(馬のための例えば 、ペプチドHPGDFGADAQGAMTK、前駆体のm / z = 752、R T = 12.0分)種の同定を提供するのに適しているペプチド、およびメモを記録。
注:例えば、馬のマーカーペプチド(前駆体のm / z = 752)が対応する牛肉ペプチド、HPSDFGADAQAAMSK(前駆体のm / z = 767、R tの= 13.2分)を有しています。 - 肉種のすべてを受け入れ、単一の動的メソッドを作成するためには、データの閲覧ソフトウェアで、順番に各肉種について、(1.5.8内の特定のペプチドに割り当てられた)それぞれの前駆体のためのXIC遷移データを開きます。
- 左クリックで選択された保持時間にピーククラスタにズームインし、クラスタの下にカーソルをドラッグします。 (ピークラベルを右クリックして)最も強い遷移を特定します。
- 手動でトランジションを記録スプレッドシート内および保持時間。
- LC / MSソフトウェア上の新しい動的メソッドなどのパラメータを入力するには、「質量分析計」をクリックし、原料ガスの条件を入力するには、[パラメータの編集」をクリックします。 「正」としての「MRM(MRM)」と「極性」としての「検索の種類」を選択します。
- 「期間の概要」に移動し、(LC分析及び平衡化のための総時間として設定)継続時間を入力します。テーブル内を右クリックして、テーブルにこれらの列を追加するには、「デクラスタリングポテンシャル(DP)」と「衝突エネルギー(CP)」を選択します。
注:「時間」欄は、現在各遷移の予想保持時間(分)を指します。 - LC / MSデータ収集ソフトウェアの「パラメータの編集]セクションで、「スケジュールMRM」チェックボックスをオンにします。 Q1、Q3、時間(分)、ID、スプレッドシート(1.5.21)で作成された遷移のためのDPとCE値と取得方法を保存(FIL入力E拡張.dam)。
注:この方法は、典型的には、MRMの数は、各ペプチドについて4最も強烈に遷移し、改善された感度およびデータの品質を与え、唯一のそれぞれのペプチドピークの保持時間窓にわたって走査減少します。 「動的」方法は「ガイド付きリテンションタイムウィンドウ」方式とも呼ばれるスケジューリングです。
2.キャリブレーションサンプルの調製および分析
- 肉混合物の抽出
- 15ミリリットルのプラスチック遠心管に以前その後冷凍肉肉のそれぞれの量を計量することにより、肉混合物の範囲を準備し、粉末に粉砕(約300mgの総質量)を使用します。
- 抽出緩衝液4ml(pH6.5の0.15Mの塩化カリウム+ 0.15 Mリン酸緩衝液)を加えます。 30秒間ボルテックス。 250サイクル/分で2時間室温で実験室シェーカー上で抽出します。
注:サイクル/分の振動運動を指します。 - の2ミリリットルを転送2ミリリットルのマイクロチューブに抽出します。 17000×gで4℃で5分間の遠心分離。
- 2ミリリットル遠心管に(2.2を参照して、タンパク質アッセイのために少量を確保)し、上清の200μlのアリコートを移し、遠心エバポレーター(予め設定されたプログラム:通気および120分の期間なしで50°C)を用いて乾燥させます。
- タンパク質測定
- 96ウェルプレートのウェルに三連で予約上清の転送7μlのアリコート(2.1.4を参照)。
- 転送三重でのタンパク質標準の一連の7μlのアリコートを、範囲0から1.0 mg / mlウシ血清アルブミン(BSA)、同じ96ウェルプレートに。
- 各ウェルにクマシープラスタンパク質アッセイ試薬200μlのを追加します。
- 視覚校正標準の範囲内にあるサンプルを確認するために、タンパク質標準とサンプルウェルの色を比較します。必要であれば、それは範囲内になるように、希釈されたサンプルで繰り返します。
- 目にプレートを残しますそして3分間。
- 皮下注射針で形成されている任意の気泡をバースト。
- 595ナノメートルの波長での標準的なエンドポイントプロトコルを使用して、プレートリーダーでプレートを分析します。
- タンパク質標準からのキャリブレーションデータを用いて、サンプルのタンパク質濃度を決定します。
注:これは、トリプシン消化物に使用されるトリプシンの量の算出のために必要とされます。
- 肉混合物のタンパク質分解
- 25mMの重炭酸アンモニウム溶液1mlステップ2.1.4からの乾燥した残留物を再溶解。 rotamixerによく混ぜます。
- ステップ1.1.3から1.3.7へのプロトコルに従ってください。
- LC / MSによる分析
- 以前に(ステップ1.5.1)のように、LC / MSを設定します。
- 前に概説したように新規取得バッチを作成します(1.5.9ステップ - 1.5.14)を、肉種のすべてを組み合わせるダイナミックLC / MS法を使用しています1.5.24でのステップで作成した取得方法を選択し、ためのデータを取得しますdigesテッド肉サンプル。
- データ閲覧ソフトで完全なクロマトグラムを表示します。順番に設定された各遷移にXICを表示します。視覚的に各クラスタは、それによって選択されたペプチドの存在を確認し、予想保持時間でベル型のピークの必要な数が含まれています確認してください。
- ナビゲーションバーの[定量法を構築する」をダブルクリックして、目的の遷移のそれぞれについて、ピーク面積を統合するためのソフトウェアを表示したデータを使用して定量化を行います。
- 「選択サンプル」ペインで「データファイル」と「サンプル」を選択する「分析物」テーブルを生成するために分析されます。
- 統合されるように遷移する(分析物)の最初に表示する「統合」タブをクリックします。
- トランジションのドロップダウンリストを表示するには、「検体」ボックスをクリックします。それを表示し、視覚的に正しいピークを統合するために選択されていることを確認するために順番に各遷移を選択します。メートルへodifyまたは強制的な統合を、左クリックと(これは緑色で強調表示されます)目標ピークの上にカーソルをドラッグします。 「選択ピーク」ボタンをクリックして、[適用]をクリックします。
- メソッドファイル(.qmf)としてワークスペースを保存します。
注:これは、サンプルのピーク面積のその後の計算のための定量方法ファイルを作成します。 - ナビゲーションバーでダブルクリックして「定量ウィザード」。 「選択サンプル」ウィンドウで、その後、一つ以上の「使用可能なサンプル」を単一の「データファイル」を選択して「定量セット」を作成。 「[設定]を選択し、[クエリ]ボックスを表示するには、[次へ]を選択します。 「選択メソッド」を表示するには、[次へ]を選択し、デフォルト値を使用しておきます。 「メソッド 'ドロップダウンボックスからステップ2.4.8で作成した「積分法」ファイルを選択し、「完了」を選択します。
注:これは肉の混合物から発生する遷移のピーク面積を含め、「結果表」を作成します。 - 「結果表」(ファイル拡張子.RDB)、テキストファイルとしてエクスポートファイル(.txt)を保存し、データを確認するには、スプレッドシートで開きます。
- 2フラグメントが含まれているものを例に焦点を当てて対応するペプチドから選択された遷移のために選択MRMのための2つの肉の測定された割合(w / w)の対別の内の1つの肉の(遷移ピーク面積によって)割合のプロットグラフ、 C末端から数えたアミノ酸の数が同じ。
注:同一の断片化サイトと同一の断片は、最適な結果を与えます。 - 上記2.4.11からプロットを調べます。いずれか視覚的に、またはプロットパッケージのトレンドラインツールを使用して、線形および類似の勾配の両方であるプロットのグループを識別します。本物の肉試料中のキャリブレーションのためにこれらのCPCPプラスフラグメントの組み合わせのいずれか一つ以上を使用してください。
注意:異常な勾配を示すプロットは、信号STREにおける必然的な減少を有するペプチドまたはフラグメントの抑制のいずれかを示すことができますngth。非線形プロットが悪いピーク検出または他の問題を示すことができます。
3.肉のサンプル
- 目標肉試料からのタンパク質の抽出
- スパチュラを用いて、試料から該当する場合には、消費税外来の非肉材料。例えば、チルドラザニアからソースとパスタを削り取ります。
- 金属ビーカーに肉の20グラムの重量を量ります。
- pH6.5の0.15Mの塩化カリウムの100ミリリットル/ 0.15 M一リン酸カリウムバッファーを追加します。
- 1分間高速ホモジナイザーで肉をブレンドすることにより、タンパク質を抽出します。
- 2.3.2 - ステップ2.1.4からのプロトコルに従ってください。
- LC / MSによるサンプルの分析
- ダイナミックLC / MS法を使用してデータを取得するための手順を繰り返し2.4.2。
- ステップ2.4.3で実行されるよう、各肉ミオグロビン由来のペプチドを特定します。
- 定量のために、関心対象の各遷移のピーク面積を統合するために定量ソフトウェアを使用ステップ2.4.9で概説されました。
- 混合物中の種の同定のために、これらの遷移のためノイズに遷移し、信号の番号のための合意された基準を満たすものをマーカーペプチドを記録。
- ステップ2.4.12から合意として定量のために、統合された遷移ピーク面積を使用すると、遷移ピーク面積によってパーセンテージを使用して、混合物中の2種からミオグロビンの割合を計算します。
- サンプル中に存在する2肉の相対的なw / wの量を推定するために、肉における可能性ミオグロビンレベルの文献18から事前の知識を使用してください。
結果
単一の動的モードMRM実験では、各プログラムの移行が指定された保持時間ウィンドウにわたって(検出器秒あたりのカウント、CPSとして)別々に記録されています。したがって、1つの実験において収集されたすべてのデータから、各遷移のピーク強度は、個々に抽出することができます。そして、唯一の有限の信号は、その遷移に設定保持時間枠のためです。窓の外では、信号は定義によりゼロです。例えばいずれかの遷移のための信号、馬から752→1269(質量1,501.66ダルトンは、前駆体イオンの m / z 751.84ダルトンは、状態= 2、フラグメントイオンyの13を充電するモノアイソトピックペプチド)は、典型的には、測定ノイズとないとだけ競うために持っていますおそらく他の種に由来する可能性がある他の遷移ピークから。出力は、したがって、共通の前駆体イオンを共有し、それらの遷移のための共通の保持時間できれいなピークは、遷移ごとに1、のセットです。
図1は、1%の混合物のための4つのトランジション752→(1269、706、248、1366)のセットの出力/重量を示しています牛肉で馬。表示された4遷移が馬に関連付けられており、純粋な牛肉、羊肉や豚肉のサンプルに存在しないされているので、これらのピークは、馬の存在を意味します。ロバスト性の基準に応じて、ノイズレベルにある特定の信号を超える二つ以上の遷移の組それぞれは、識別を確立します。この図は、したがって、牛肉で馬ワット/ wの1%の混合物中の馬の存在を確立します。
時折、単一の孤立遷移が検出されます。これは、質量分析計にシステムから期待され、プログラムしたもので、おそらく無関係なタンパク質から、前駆イオンと単一フラグメントの偶然の一致を示します。予期しない保持時間における特異ピークの性質、及びその発生は、Siであります無視することができ、偶発的移行のgnature。
各遷移のピーク下の面積を個別に計算することができます。適切なフラグメントに基づいて、牛肉の遷移ピーク面積に対する馬の割合は、例えば、767→1299へ→1269年752(馬)(牛肉)は、混合物中の実際の肉の割合に比例します。 図2は、Aを示し牛肉と馬との混合物中の馬の重量のための重量百分率対これらの2つの遷移のピーク面積百分率のプロット。パーセンテージ遷移ピーク面積は、肉の重量のための重量パーセントと一致した場合、傾きはこのプロットにおける傾きがこれらの遷移とCPCP対について、遷移ピーク面積は、相対的な量の信頼性の尺度を与える、ことを示す、1.03 1です。混合物中の2つの肉の。他の要因変わらずで、試料中の馬の肉は、その後の傾斜牛肉としてミオグロビンで二倍の豊富だった場合行は、1よりも大きいであろう。
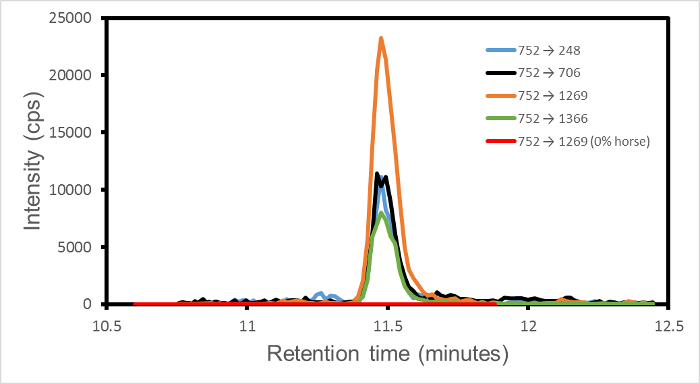
牛肉で馬w / wの1%の保持時間に対する 図1. MRM遷移強度を。遷移はそれぞれ、黒、青と緑、オレンジ色で示され、752→(1269、706、248、1366)です。マーカーペプチドはHPGDFGADAQGAMTKです。 4遷移フラグメントはここで、yはそれぞれ、Y 13、Y 7、Y 2及びY 14で表さNペプチドのC末端からN個のアミノ酸で計数表すことができます。信号対雑音比は、4つの遷移上で23から53まで変化します。追加の赤い線は0%馬、比較のために100%ビーフのための752→1269推移を示しています。保持時間の唯一の非ゼロの領域が表示されます。この図は、ワトソンらから変更されている。3。エス/ ftp_upload / 54420 / 54420fig1large.jpg "ターゲット=" _空白 ">この図の拡大版をご覧になるにはこちらをクリックしてください。
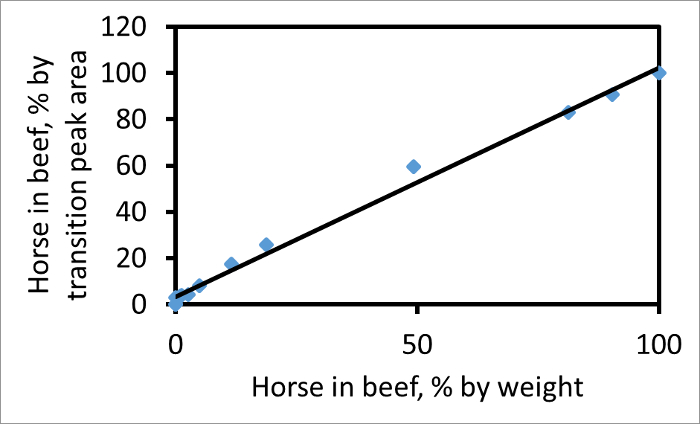
パーセント遷移ピーク面積として牛肉で馬対重量のパーセント重量などの牛肉で馬の 図2. プロット、。プロットは、ペプチド牛肉(767)と馬(752)との両方のためのyの13フラグメントイオンのペアを使用しています。 Aはピーク面積を表す場合には、縦軸が100 A、H /(H + B)です。最良適合線(R 2 = 0.99)の傾きは1.03です。この図は、ワトソンら 3から変更されている。 この図の拡大版をご覧になるにはこちらをクリックしてください。
ディスカッション
好適な標的タンパク質の選択が重要です。優れた標的タンパク質は、目的の種の対応する形態は、十分な種依存性配列変異、種特異性を有し、生物内でアクセス可能な量で存在する必要があります。処理を受けた混合物(例えば、熱処理)を評価するため、その処理に比較的免疫配列を有するタンパク質であることが望ましいです。ミオグロビンは、調理された赤身肉などの赤肉、のための良い候補であるが、唯一の可能性ではありません。標的タンパク質が決定されると、プロトコルの最も重要な部分は、タンパク質のタンパク質分解です。ミオグロビンは異なるタンパク質がよく、代替タンパク質分解プロトコルを要求することができます。
記載されているようにプロトコルは、基準精製タンパク質に基づいてセグメントを含みます。これは、保持時間ウィンドウと、適切な前駆体およびフラグメントイオンを発見することを目指しています。このセグメントは非常に有用であるが、必須ではありません。
、それは時々、配列の差が消化プロファイルに劇的な影響を有している場合です。例えば、ペプチドペアVLGFHG(牛肉)とELGFQG(馬)( 図2の1未満の勾配としてマニフェスト)異常 な定量結果を与えます。後者のペプチドは、混合物中の馬のレベルの過少見積もりを引き起こし、比較的抑制KE切断から生じるためです。異なるアミノ酸で始まる対応するペプチドは、したがって、最高の回避されます。多くの場合、対応する二つのペプチドのフラグメントは、同一のアミノ酸配列を有し、行儀されているが、これは常にそうではなく、方法の開発中にチェックする必要があります。種の同定は、相対定量よりも、これらの問題に非常に敏感ではありません。プロトコルは、4つの赤肉のために実証されています秒3。あまりにも多くのマーカーペプチドが共溶出場合、遷移ピーク形状の品質を効果的に滞留時間を減少させ、最終的に相対的な定量推定値を分解、劣化するかもしれないが、追加の肉種が、含まれ得ます。すでに利用可能な改善された機器は、これを改善します。関連する問題ではない、すべての肉は、異なるミオグロビンを有することです。例えば、馬、ロバとシマウマミオグロビンは同一であり、したがって、厳密方法を言えば牛肉に馬やロバやシマウマを検出することができるだけです。いくつかのケースでは、ミオグロビンが同一ではないにもかかわらず、いくつかの重要なペプチドは、することができます。例えば、いくつかのラムミオグロビン由来のマーカーペプチドはまた、ヤギに表示されます。
このおよび他のタンパク質ベースの定量法が直面している合併症は、タンパク質またはペプチドのレベルは、混合物中の肉のレベルに自明と同一視することがある場合、タンパク質レベルは、全ての種にわたって一定であると仮定しなければならないことです。ミオグロビンと4赤メートルのためにこれは普遍的真実ではありません食べます。一般的にレベルは豚肉は4の最低レベルを示すと、依存種です。また、ミオグロビンレベルは肉カットと動物の年齢に応じて変化します。遷移のピーク面積の比が、ミオグロビンの比率に確実にマッピングがので、実際の肉の割合へのマッピングは、混合物中の肉の可能性ソースに関する仮定の推定図です。
この作業で概説したアプローチは、他の公開の貢献から、いくつかの方法で異なっています。より典型的な経路は、異なる種のマーカーが互いに8-12,14,19とは特定の関係を有していない、その場合には、様々な異種の種依存性マーカーペプチドを同定するプロテオミクスの方法を使用することです。これとは対照的に、我々は3バリアント 、種依存性シーケンスまで関心のあるすべての種に共通するタンパク質を選択しました。別に私たちの相対定量戦略の中心であることから、これはそのサンプル利点を有しています調製ストラテジーを最適化することができます。さらに、このような対応するタンパク質は、抽出、またはそのような調理または缶詰などのサンプルの商業的処理、例えば、同様に挙動することが予想されるかもしれません。種の同定は、その後、通常、典型的には、1つまたは2配列の差異を有する密接に関連するペプチドの検出によりCPCPアプローチ種の識別が進む中で、一方、異種のマーカーペプチドの検出を介して進行します。最後に、他に1種の重量%を推定するタンパク質の定量は、従来別々に既知の標準7,14,15に基づいて、各タンパク質の絶対定量を介して進行することがあります。しかし、較正方法を必要としないCPCP法を用いて。代わりに、相対的なレベルは、完全に絶対測定ステージをバイパスし、両種からの対応する二つのペプチドの信号強度を比較することによって推定されます。究極の目標は、あの、1つの種の重量%であることからTHER、相対定量、その後CPCPは両方のより直接的かつ2つの絶対定量測定値を比較するよりも簡単です。これらの機能は、食品の不正検出の分野における急速な監視ツールとしての技術が有用なもの、洗練されたプロトコルを用いて、約2時間であることが予想される短い実験時間、に変換されます。
開示事項
The authors have nothing to disclose.
謝辞
We acknowledge financial support from Institute of Food research BBSRC Core Strategic Grant funds, BBSRC Project BB/J004545/1.
資料
| Name | Company | Catalog Number | Comments |
| Uniprot database | www.uniprot.org | Freely accessible database of protein sequences | |
| Skyline software | www.skyline.gs.washington.edu | Free software to download that enables the creation of targeted methods for proteomic studies, peptide and fragment prediction | |
| Ammonium bicarbonate | Sigma-Aldrich Co Ltd, Gillingham, UK www.sigmaaldrich.com | O9830 | |
| Methanol, HPLC grade | Fisher Scientific, Loughoborough, UK www. fisher.co.uk | 10674922 | |
| Acetonitrile, HPLC grade | Fisher Scientific, Loughoborough, UK www. fisher.co.uk | 10010010 | |
| Urea | Sigma-Aldrich Co Ltd, Gillingham, UK www.sigmaaldrich.com | U5378 | |
| Trypsin(from bovine pancreas treated with TPCK) | Sigma-Aldrich Co Ltd, Gillingham, UK www.sigmaaldrich.com | T1426 | |
| Formic acid | Sigma-Aldrich Co Ltd, Gillingham, UK www.sigmaaldrich.com | F0507 | |
| Coomassie Plus Protein Assay Reagent | Thermo Fisher Scientific www.thermofisher.com | 1856210 | |
| Protein standard | Sigma-Aldrich Co Ltd, Gillingham, UK www.sigmaaldrich.com | P0914 | |
| Ultra Turrax homogeniser T25 | Fisher Scientific, Loughoborough, UK www. fisher.co.uk | 13190693 | |
| Edmund and Buhler KS10 lab shaker | |||
| Heraeus Fresco 17 Centrifuge | Thermo Fisher Scientific www.thermoscientific.com | 75002420 | |
| Vacuum centrifuge RC 1022 | Jouan | ||
| Plate Reader | |||
| Strata-X 33u polymeric reversed-phase cartridges 60 mg/3 ml tubes | Phenomenex, Macclesfield, UK | 8B-S100-UBJ | |
| 4000 QTrap triple-quadrupole mass spectrometer | AB Sciex, Warrington, UK www.sciex.com | ||
| 1200 rapid resolution LC system | Agilent, Stockport, UK | ||
| XB C18 reversed-phase capillary column (100 mm x 2.1 mm, 2.6 µm particle size) | Phenomenex, Macclesfield, UK www.phenomenex.com | ||
| Analyst 1.6.2 software | AB Sciex, Warrington, UK www.sciex.com | QTrap data acquisition and analysis, including peak area integration | |
| Autosampler vials |
参考文献
- O'Mahony, P. J. Finding horse meat in beef products-a global problem. QJM-An Int. J. Med. 106, 595-597 (2013).
- Sentandreu, M. A., Sentandreu, E. Authenticity of meat products: Tools against fraud. Food Res. Int. 60, 19-29 (2014).
- Watson, A. D., Gunning, Y., Rigby, N. M., Philo, M., Kemsley, E. K. Meat Authentication via Multiple Reaction Monitoring Mass Spectrometry of Myoglobin Peptides. Anal. Chem. 87, 10315-10322 (2015).
- Food Standards Agency. . Report of the investigation by the Food Standards Agency into incidents of adulteration of comminuted beef products with horse meat and DNA. , (2013).
- Taylor, A. J., Linforth, R., Weir, O., Hutton, T., Green, B. Potential of electrospray mass-spectrometry for meat pigment identification. Meat Science. 33, 75-83 (1993).
- Ponce-Alquicira, E., Taylor, A. J. Extraction and ESI-CID-MS/MS analysis of myoglobins from different meat species. Food Chem. 69, 81-86 (2000).
- Gallien, S., Duriez, E., Domon, B. Selected reaction monitoring applied to proteomics. J. Mass Spectrom. 46, 298-312 (2011).
- Orduna, A. R., Husby, E., Yang, C. T., Ghosh, D., Beaudry, F. Assessment of meat authenticity using bioinformatics, targeted peptide biomarkers and high-resolution mass spectrometry. Food Addit. Contam. Part A-Chem. 32, 1709-1717 (2015).
- Claydon, A. J., Grundy, H. H., Charlton, A. J., Romero, M. R. Identification of novel peptides for horse meat speciation in highly processed foodstuffs. Food Addit. Contam. Part A-Chem. 32, 1718-1729 (2015).
- von Bargen, C., Brockmeyer, J., Humpf, H. U. Meat Authentication: A New HPLC-MS/MS Based Method for the Fast and Sensitive Detection of Horse and Pork in Highly Processed Food. J. Agric. Food Chem. 62, 9428-9435 (2014).
- von Bargen, C., Dojahn, J., Waidelich, D., Humpf, H. U., Brockmeyer, J. New Sensitive High-Performance Liquid Chromatography Tandem Mass Spectrometry Method for the Detection of Horse and Pork in Halal Beef. J. Agric. Food Chem. 61, 11986-11994 (2013).
- Montowska, M., Alexander, M. R., Tucker, G. A., Barrett, D. A. Authentication of processed meat products by peptidomic analysis using rapid ambient mass spectrometry. Food Chem. 187, 297-304 (2015).
- Montowska, M., Alexander, M. R., Tucker, G. A., Barrett, D. A. Rapid detection of Peptide markers for authentication purposes in raw and cooked meat using ambient liquid extraction surface analysis mass spectrometry. Anal. Chem. 86, 10257-10265 (2014).
- Sentandreu, M. A., Fraser, P. D., Halket, J., Patel, R., Bramley, P. M. A Proteomic-Based Approach for Detection of Chicken in Meat Mixes. J. Proteome Res. 9, 3374-3383 (2010).
- Elliott, M. H., Smith, D. S., Parker, C. E., Borchers, C. Current trends in quantitative proteomics. J. Mass Spectrom. 44, 1637-1660 (2009).
- MacLean, B., et al. Skyline: an open source document editor for creating and analyzing targeted proteomics experiments. Bioinformatics. 26, 966-968 (2010).
- Keeton, J. T., Ellerbeck, S. M., Nunez de Gonzalez, M. T., Devine, C., Dikeman, M. . Encyclopedia of Meat Sciences. 1, 235-243 (2014).
- Montowska, M., Alexander, M. R., Tucker, G. A., Barrett, D. A. Rapid Detection of Peptide Markers for Authentication Purposes in Raw and Cooked Meat Using Ambient Liquid Extraction Surface Analysis Mass Spectrometry. Anal. Chem. 86, 10257-10265 (2014).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved