Method Article
VirusMapperと超解像顕微鏡のためのオープンソースの単一粒子分析
要約
この原稿は、ナノスケールの構造の正確なモデルを生成するために、超解像顕微鏡画像に単一粒子の分析を適用するVirusMapperフィジーベースのオープン・ソース・ソフトウェア・パッケージを使用しています。
要約
超解像蛍光顕微鏡は、現在、細胞生物学の研究に革命を起こしています。約300ナノメートルの分解能の限界を打破する能力は、ナノスケールの生物学的複合体およびプロセスの日常イメージングが可能になります。解像度の増加はまた、単一粒子分析のような電子顕微鏡において一般的な方法は、容易に超解像蛍光顕微鏡に適用することができることを意味します。超解像光の撮像と、この分析手法を組み合わせることにより、準安定構造内の分子素子の構造のマップを生成するために、蛍光顕微鏡の分子に特異的な標識能力を活用することが可能となります。この目的のために、我々は新しいアルゴリズムを開発した - VirusMapper - 使いやすい、高性能、および高スループットのImageJのプラグインとしてパッケージ化。この記事では、生物学的メートル新規な構造的特徴を発見する能力を披露し、このソフトウェアへの綿密なガイドを提示しますolecular複合体。ここでは、互換性のあるデータをアセンブルし、超解像画像に単一粒子分析を適用するには、このアルゴリズムを使用する方法のステップバイステップのプロトコルを提供する方法を提示します。
概要
超解像(SR)顕微鏡は、それらを理解するのに重要な分子特異的な標識と一緒に画像のキー分子プロセスに能力を提供することにより、細胞生物学に大きな影響を与えています。 SRは、現在、このような画像生細胞1、2の電位としての光学顕微鏡の主要な利点を保持しつつ、電子顕微鏡(EM)により以前にのみ達成可能解像度(20〜150 nm)を接近する光学顕微鏡を可能にします。さらに、ナノスケールレベルで見出される構造的保存は、SRデータの単一粒子分析(SPA)、電子顕微鏡3で広く使用される概念の応用を可能にします。 SPAを使用して、構造体の多くの高度に保存されたコピーは、画像化され、解像度、精度、または信号対ノイズ可視化オブジェクトのを改善するために一緒に平均化することができます。 SRと組み合わせて使用すると、SPAは、高Pのための強力なツールであることが実証されています核膜孔複合体4,5の構成要素、中心体6、及びそのようなHIV 7及びHSV-1~8などのウイルスのrecisionマッピング。
しかし、SRおよびSPAのルーチンを組み合わせたアプリケーションは、利用可能なソフトウェアの欠如によって挑戦されています。このような理由から、我々はVirusMapper、人気の画像処理ソフトImageJを/フィジー9のプラグインを開発しました。これは、SR顕微鏡で画像化された構造の速い、ユーザーフレンドリーな、マルチチャンネルナイーブ平均を提供するために設計された蛍光画像10と一般SPAの最初の自由に利用可能なソフトウェアパッケージです。ウイルスのために設計されているが、それは、異なる分子種が、撮像された識別され、そして局在化することができる任意の高分子複合体にも適用することができます。
VirusMapperは、高精度の分子を生成するために使用することができます平均寸法および他のパラメータの計算を可能にする任意の既知の構造モデル。アルゴリズム設計は、明確な方向性や異なる形態学的状態の決意を提供する、構造体の集団を分離するために特に有用なものにします。また、マルチチャネルイメージングは、これにより参照ベース構造の発見を可能にする、基礎となる構造はよく知られている場合には、基準チャネルを使用するために使用することができます。ソフトウェアをダウンロードし、インストールするための命令が上に設けられているhttps://bitbucket.org/rhenriqueslab/nanoj-virusmapper 。例データもあり見つけることができ、ユーザーは自分のに適用しようとする前に、例えば、データ上のソフトウェアを使用して練習することをお勧めします。
ここでは、生データからのSPAモデルを生成するために、このプラグインを使用するための手順が説明されています。ソフトウェアは、シングルOを含むRAW画像を取ります入力としてマルチ標識構造はRとなります。これは、ソフトウェアが実行されるように調整されるパラメータの数、撮像された構造内の標識された成分の平均分布を示すSPAモデルの対象返します。
このプロトコルの目的は、図1に概説パイプラインに係る撮像された構造内の構成要素の平均局在を与える正確なSPAモデルを生成することです。 図1に示すように、ソフトウェア・ワークフローは有用3つの段階に分けられます。第一段階は、各チャンネルの粒子の積み重ね、その結果、セグメントの拡大イメージです。これらの粒子は、モデルを作成し、モデル生成のためのシードを生成するために平均化される単位です。第二段階は、最終段階での粒子のセット全体を登録するために使用されるシード画像を生成することです。これは、基準チャネルを選択し、手動のに貢献するこのチャネル内の粒子を選択することによって行われますEEDS。種子は、この基準チャネルに選択されるが、全てのチャンネルのために生成することができます。粒子は、最初にこのチャネルで2次元ガウシアンをフィットさせることによって再調整されます。選択及び再整列されたすべての粒子は、次いで、種子を生産するために平均化されます。モデル化されるデータに見られる各共通構造のために、粒子は、明確かつ正確にその構造を表すシードとして選択されるべきです。この段階でのインタフェースはまた、そのような構造のためのデータをスキャンするために有用です。
最終段階は、テンプレートマッチングを用いてモデルを生成することです。これは、本来相互相関によって、前のセクションで生成されたシード画像に抽出した粒子の登録によって達成されます。登録された粒子のサブセットが一緒に平均化され、そしてプロセスはさらに、所望であれば、平均二乗誤差モデルを減少させるために繰り返されます。このサブセットは、満たされなければならない種子に対して最小の類似度を設定することによって決定されます。モデルを作成する場合複数のチャンネルで同時にsは、関節の類似性、又は各チャネルの類似度の平均値が用いられます。それらに貢献し、得られたモデルと登録された粒子は、その後、さらに分析することができます。
プロトコル
注:このプロトコルとビデオは、より詳細にソフトウェアパッケージを記述したオリジナル紙10を補足します。読者は、ソフトウェアの使用に関する追加のガイダンスのために慎重にこれを確認することをお勧めします。三つの主要な段階がある:粒子の抽出、個々の粒子へのセグメントの拡大イメージは、一般的な構造は、データで識別され、最終段階で使用されている種子を生成するために整列されるシードの選択、。テンプレートマッチングは、これらの種に基づくモデルの生成は、抽出された粒子と、平均化をSPAモデルを生成するためにサブセットを整列させます。
ソフトウェアパッケージを実行する前に1.セットアップ
- カバースリップ上または関連する実験条件で研究中の構造のサンプルを準備します。
- 画像ような構造化照明顕微鏡(SIM)11又は刺激に対するような超解像蛍光顕微鏡による試料テッド・エミッションの枯渇(STED)12の顕微鏡。
注:正確な準備方法の詳細と画像サンプルは、調査対象の構造物の性質に大きく依存するため、関連する文献は、相談する必要があります。一例として、そのようなここで使用されるものとして、ワクシニアウイルスのサンプルを調製し、画像化するための正確な方法は、代表的な結果のセクションに記載されています。 - 構造体又は粒子、好ましくは数千の独立したコピーの多数を示す複数の視野の画像を作成します。可能な限り互いに分離し、画像が汚れや関心のない他の蛍光構造の自由であることを確認されている画像の粒子。
- フィジーのツールバーにファイルをドラッグするか>「開く」「ファイル」を選択することで、フィジーの粒子を含むすべての画像を開きます。
- 連結する「イメージ」>「スタック」>「ツール」>「連結し」を選択シングルスタックに画像。結果の画像がHyperstackであればその後、「イメージ」>「Hyperstacks」>「スタックにHyperstack」を選択することで、スタックにそれを回します。
注:最後のスタックがチャンネルをインターカレートしている必要があります。 2つのチャネルがある場合、スタック内の最初のスライスは最初の画像からチャンネル1であるべきで、第二のスライスは、対応するチャネル2でなければならない、第三のスライスは2番目の画像からチャンネル1であり、そしてべきです。
2.粒子を抽出
- セグメントに画像を選択し、「ウイルスの構造を抽出する」を選択します。抽出された粒子を保存し、「解凍ウイルス構造」を表示する場所を選択してください ダイアログ( 図2)。
- 以下のように「ウイルスの構造を抽出」ダイアログにそれらを入力することにより、初期推定と抽出パラメータを割り当てます。画像分割をプレビューした後、これらのパラメータを微調整。
- 麻痺を設定します。撮像された異なる蛍光チャネルの数( 例えば、2)のようなデータセット内のチャネルのER。
- 粒子がチャネルの選択の数を入力することにより、そのチャネル内のピークの検出によって抽出される基準チャネルを設定します。最も一貫性のあるチャンネルを選択します。それは、ほとんどの粒子が同じ外観を持っているチャンネルです。
注:可能な場合は、このチャネル中の粒子は中央の最大値を持つことになります。 - 事前に検出ガウスぼかしを適用するかどうかを選択します。何のぼかしを適用しない場合は、0にプリ検出ガウスぼかしを設定します。この値を大きくすると、所定の半径のガウシアンぼかしフィルタは、極大値の検出の前に適用されます。基準チャネルは、中央最大値( 例えば、リング形状)を有していない場合は、この機能を使用します。ボケが1の外観を誘導します。
注:この機能は使用のみであるとして関心のセグメント化された領域(ROIが)、それらに適用されるガウスぼかしを持っていませんDは、基準チャネル内のROIを位置決めします。 - ピクセル(各極大値付近に設定される)ROIの半径を設定します。その関心領域は、 図3のように最大の粒子よりもわずかに大きくなるように値を選択してください。最大粒子(眼によって概算)約30画素の直径を有するように見える場合、例えば、次に20個のピクセルにROIの半径を設定します。
- 100以下最初、比較的小さな値にフレーム当たりに使用するROIの数を設定します。
- 最大ROIオーバーラップを設定します。粒子が十分に分離されている場合は、この小さなを保ちます。粒子がクラスタ化された場合、重なるように関心領域を可能にするために、これを増やします。
- 「番組プレビュー」を選択します。
注:これは、選択された場合、現在のフレームに関連付けられた参照チャネルのための抽出のROIが表示されます。 - ROI半径、ROIの数、できるだけ多くの粒子の周囲に適当な大きさの関心領域を有するように、最大ROIの重なりを調整します、 図3のように。
- セグメンテーションを実行するために、「OK」を選択します。画像およびROIマネージャを閉じます。
注:パーティクルセットのファイルの名前を変更しないでください。次のセクションのために - これらの名前は、フォーマット「channelXウイルス粒子」でなければなりません。
3.種子
- 、「シードを生成」を選択し、抽出粒子が保存されているフォルダを選択し、「種子の生成」ダイアログやウィンドウ( 図4)を表示します。
- 以下のように、「シードを生成」ダイアログにそれらを入力することにより、初期シード-選択パラメータを割り当てます。
- すべてのチャネルを整列し、中央に装着される基準チャネルを設定します。ほとんどの粒子が同一の外観を有し、可能な場合には、中央の最大値を有する、チャネルを選択してください。
- シードが生成する必要のあるすべてのチャネルのボックスを選択します。
- 種子はrotatあるべき場合に選択します90°編。他のモデルと一致アライメントを持つように、この機能を使用します
- プリアライメントガウスぼかしを適用するかどうかを選択します。何ぼかしを適用しないように0に各チャンネルのぼかし半径を設定します。再編成の前に与えられた半径のガウスぼかしフィルタを適用するには、この値を増やします。粒子は中央の最大を持っていない場合は、この機能を使用します。ボケが1の外観を誘導します。
注:この機能は唯一の一貫性のあるアライメントを得ることであると種子は、ブラーが適用されていません。 - チェックや各チャンネル毎に「シフト補正」ボックスをオフにして、それらを回転させていないが、別途非参照チャンネルの種を中心にシフト補正を使用するかどうかを選択します。
注:チャンネルはよく互いに整列されていない場合は、これを使用します。それはまた、シフト補正なしで、単に基準に応じて、他のチャネルを整列させるために有用であり得ます。
- シードとして使用するための粒子を選択してください。トンを検索彼はモデル化し、フレーム番号を記録する構造に似ている粒子を見つけるために、シーケンスを粒子。
- ボックス「を使用するフレーム」に数値を入力し、複数のウィンドウが( 図5)が表示されます。カンマで区切られた複数のフレーム番号を入力します。
- 表示されるウィンドウでシード枠と結果の平均種を見ます。
注:ログは平均に似た種を提案します。 - 見つかった種フレームの数は、同様の外観を有するようにシード選択プロセスを最適化するために、基準チャネル、ガウシアンぼかし半径、及びシフト補正のオプションを調整します。平均種子が十分にモデル化するデータで見られる構造に似ていることに作成されるまで、追加の種を続行します。
- 種子やどこにに保存されます名前を付けて、モデル生成で後で使用するために、最終的なシードの画像を保存するために「OK」を選択します。
4.モデルを生成
- セLECT、「シードに基づいてモデルを生成」を抽出粒子が保存されているフォルダを選択し、「モデルの生成」ダイアログ( 図6)を表示します。
- チェックボックスを使用して、各チャンネルの種をロードします。
注:種子を「シードを生成」ダイアログで指定されたサブフォルダに保存されます。デフォルト名は「分析」です。シード平均値ではなく、また、参考のために保存されているフレームを選択するように注意してください。 - 以下のように、「モデルを作成」ダイアログにそれらを入力することにより、初期モデルの生成パラメータを割り当てます。これらのパラメータ後で微調整。
- 唯一の基準チャネルから翻訳及び回転を計算し、すべてのチャンネルに適用し位置合わせのための基準チャネルを使用するかどうかを選択します。このオプションを使用する場合は、リファレンスとして使用するチャンネルを選択します。
注:このオプションは、具体的REFEREように、1つのチャネルを使用するように選択する必要がありますこのような基準に基づく構造の発見をしているかのようにNCE、。それ以外の場合は、あまり正確なモデルになります。チャンネルはよく、このオプションを使用する場合は整列されなければなりません。 - テンプレートマッチングの際に画像強度を二乗するかどうかを選択します。これは小さな違いを強調するので、特に微妙な特徴を持つモデルを作成する際に、これを使用します。
- 「最小類似種に対する」スライダを使用するか、ボックスに数値を入力することにより、種子に対して最小の類似度を選択します。
注:のみ使用されます。このカットオフよりも大きな種子への類似性を有する粒子。 60から80パーセントは、一般的に開始するには良い選択肢です。 - 、1回の反復の最大数を設定シードに対して最小の類似性を最適化し、必要に応じて、後でそれを高めます。
- 表示されるモデル生成プロセスの要素を選択するボックスを選択します。
注:「ショーの種」ボクセでロードされた種が表示されますダイアログの上部にあるの。 「表示モデルは、」シードに対して最小の類似性を満たしている粒子の部分集合を平均から作成されたモデルのすべての反復を表示します。 「ショーMSEは、」最も可変であるモデルの領域を強調した平均二乗誤差(MSE)の画像が表示されます。 「表示粒子」と表示されたモデルの最高の繰り返しに応じて登録されたモデルを作成するために使用された粒子のサブセットを、表示されます。
- 唯一の基準チャネルから翻訳及び回転を計算し、すべてのチャンネルに適用し位置合わせのための基準チャネルを使用するかどうかを選択します。このオプションを使用する場合は、リファレンスとして使用するチャンネルを選択します。
- 選択のプレビューモデルを生成し、結果を表示する「プレビューを表示」。
注:これはプロセスの最も計算集約的なステップです。デスクトップPC上の画素の数十の直径を持つ数千の粒子の集合のために実行している時間は約10分でなければなりません。計算時間が問題である場合、ユーザは、第1のデータの小さいサブセットにアルゴリズムを試みるべきか、可能な場合、ステップ2.2.4においてより小さいROI半径を使用します。 - 生成見ますDモデルとパラメータ、種子に対して特に最小の類似性を最適化します。モデル化された形態の唯一の真の粒子は、モデルに含まれるまでシードに対して最小の類似度を増加させます。
- スライダー「反復の最大数」を使用するか、ボックスに番号を入力することにより、反復の最大数を増やすと、モデル生成プロセスを反復することを可能にします。最大の反復のために10前後の値を使用してください。
- モデルに名前を付け、最終モデル生成プロセスのすべての反復が含まれているモデルの進化スタックを保存するために「OK」を選択します。
注:シードに対する最小類似性は全く粒子が、この類似性を持っていないことが非常に高い場合には、何も更新されません。プラグインが凍結していると思われる場合は、最小の類似度が高すぎるという可能性を考えます。
結果
ここでは、モデルのポックスウイルス、ワクシニアウイルスのソフトウェアを示しています。最も複雑な哺乳動物ウイルス、ワクシニアパッケージ350 X 270 X 250 NM 3煉瓦状粒子13、14内の約80の異なるタンパク質の一つ。三つの部分構造は電子顕微鏡によって認識されている:二本鎖DNAゲノムを含む中心コア;コアに隣接する横体と呼ばれる2つのタンパク質性構造は、。単一のプロテオリ二重層エンベロープ15。大型、複雑な構造、および組換え蛍光タンパク質タグに対する従順は、ワクシニアVirusMapperワークフローを実証するための優れたシステム作ります。
ここで説明するようにソフトウェアを使用して、ワクシニアウイルス粒子上の様々なタンパク質の分布をモデル化することができます。タンパク質を標識し、コンビに、おそらく、画像化しました記載されているように参照として既知の分布の他のタンパク質との国、およびソフトウェアは、粒子上のタンパク質の局在化の平均モデルを生成するために使用しました。この例では、2つのタンパク質は、内側コアタンパク質L4、および主要な横方向の本体部品F17をモデル化しました。
mCherryを16でタグ付けされたF17は、GFP及びL4でタグ付けされた組換えワクシニアウイルスを使用しました。精製されたウイルスは、1 mMトリス、pHが9に希釈し、室温で30分間、それらを被覆することにより洗浄し、高性能カバーガラスに結合させました。次いで、試料を20分間、PBS中の4%ホルムアルデヒドを適用することにより固定しました。カバーガラスを抗退色マウンティング培地中でスライドに直ちにマウントしました。イメージングは、市販のSIM顕微鏡でSIMによって行われました。視野は5つの位相シフトと561ナノメートルと3つのグリッドの回転(32ミクロン格子PEを用いて取得されたウイルスおよび画像の数百を含む選択されましたriod)および488 nmの(32μmの格子周期)レーザ。画像はsCMOSカメラを用いて取得し、顕微鏡用ソフトウェアを用いて処理しました。チャネルは、同一の画像取得設定で撮像されたマルチカラービーズスライドに基づいて整列させました。 SIM再構成およびチャネルアラインメントイメージ後フィジーで開かれた単一の画像スタックに連結。
これらの粒子は、中央最大値を有するようにウイルス粒子を、基準として任意のガウスぼかしを適用することなく、L4チャネルを用いて画像から抽出しました。周りの15,000粒子は、この実験で抽出しました。
ワクの形状に、横方向の体はウイルスの向きに基づいて明らかに異なる外観を持っています。私たちは、1つまたは2人の横方向の体のいずれかを区別することができている2つの方向を可視化しました。私たちは、正面と矢状としてこれらの姿勢に敬意を言及しましたively。
正面およびサジタル方向のための別個の種が「シードを生成」段階で粒子リストを検索して選択した( 図4および5)。一方向または他に明らかにした粒子を選択しました。 L4チャネルは互いにシードを整列させるために、基準チャネルとして使用しました。ここでも、ガウスぼかしが必要ではなかったです。各方向のための5つの粒子は、選択された種子を生産するために平均しました。
モデルは、これらの種子をもとに各方向のために作成しました。基準チャネルも二乗強度値のいずれも使用しました。反復の最大数は1に初期設定し、最小類似性は各方向のための一貫性のある外観を与え、それぞれの場合において、約1,000粒子を含むように設定しました。反復の最大数は、その後に増加しましたモデルの収束を可能にします。モデルは、このように二つのチャンネルに2つの向き( 図7)のために生成されました。
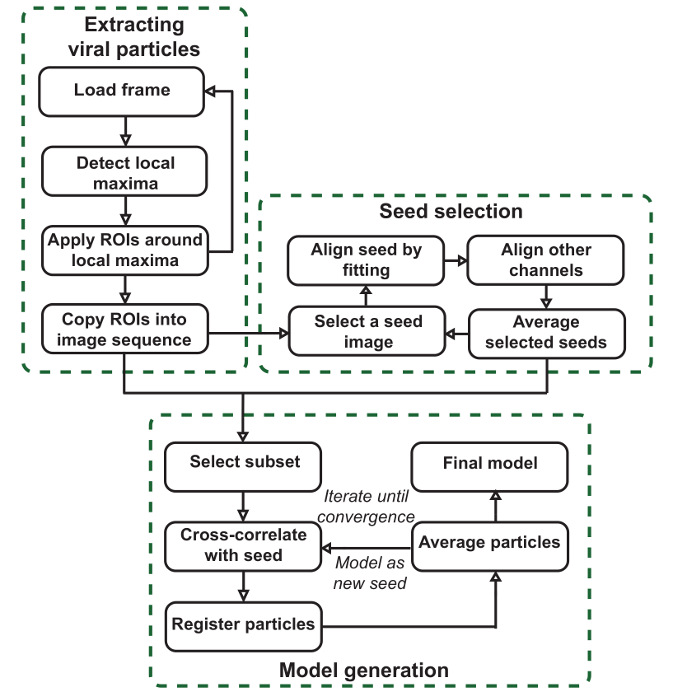
図1:VirusMapper ワークフロー。プラグインは、三つの主要な段階に編成されます。ウイルス粒子は、拡大イメージ、テンプレート画像または種子から抽出されたデータから、半手動で選択され、そして最終的なSPAモデルは種子を参照して、データから生成されます。 この図の拡大版をご覧になるにはこちらをクリックしてください。
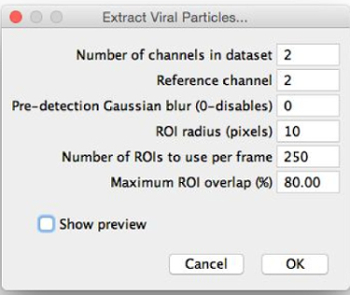
図2: ダイアログを「ウイルス構造を抽出」。選択する場合、「ウイルスStructurを抽出 ES」、このダイアログが表示されます。パラメータが最適なセグメンテーションのための初期推定値で満たされる必要がある。 『を表示するプレビュー』、その後のROIをプレビューすることとパラメータを微調整することができるように、選択することができます。 ご覧になるにはこちらをクリックしてください。この図の拡大版。

図3: 抽出パラメータを設定します。抽出されたROI、ROI半径、ROIの数、および最大ROI重複をプレビューした後、このような状況を達成するように調整されます。関心領域は、全ての粒子は、ROIに含まれ、関心領域は、クラスタ化された粒子を分離することができるように十分にオーバーラップすることができ、粒子よりもわずかに大きいです。ANK ">この図の拡大版をご覧になるにはこちらをクリックしてください。
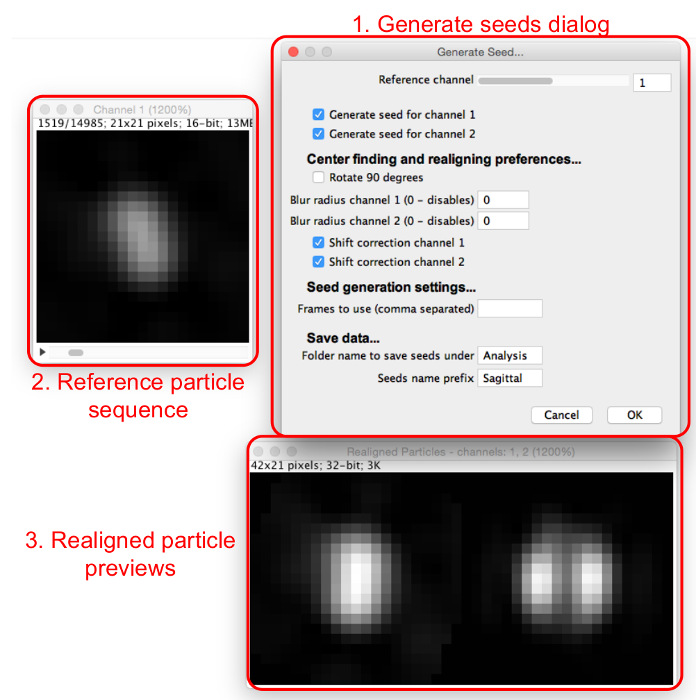
図4: テンプレートマッチングの種を生成します。 「シードを生成」ダイアログ(1)が割り当てられるパラメータを設定します。参照粒子配列(2)ユーザが基準チャネル中の粒子を走査することを可能にします。粒子は、基準粒子配列に見た場合、すべてのチャネルの再整列粒子が再整列粒子のプレビュー(3)で見ることができます。 この図の拡大版をご覧になるにはこちらをクリックしてください。
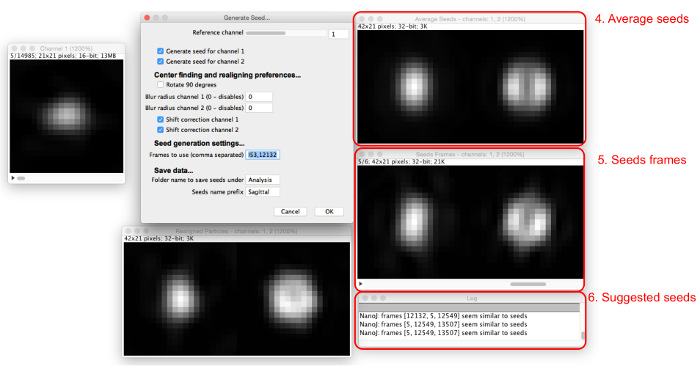
図5: シード・イメージを追加します。種子はに追加されますボックス「フレームを使用する」、関与する全ての種子(4)の平均値とフレーム(5)が表示されています。現在の平均種子に類似している粒子は、ダイアログ・ボックス(6)で提案されています。 この図の拡大版をご覧になるにはこちらをクリックしてください。
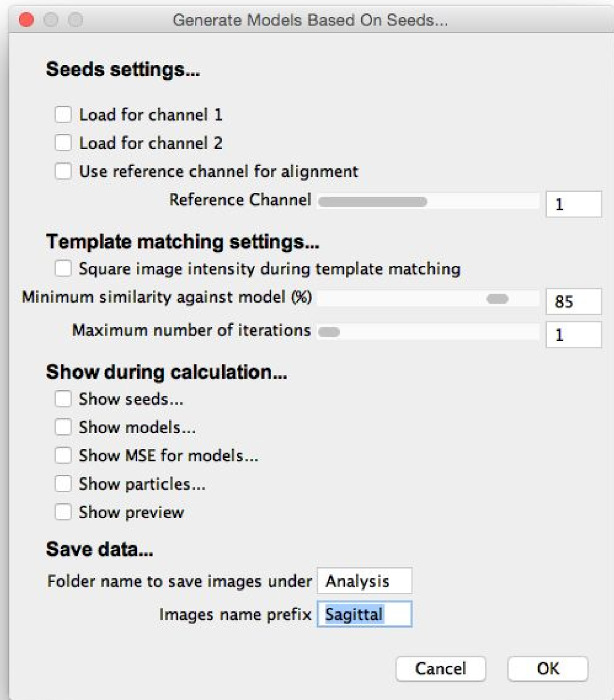
図6: ダイアログ「モデルの生成」。選択する場合は、「シーズに基づくモデルを生成し、」このダイアログが表示されます。パラメータは、最適なモデル生成のための初期推定値で充填されるべきであり、計算中に示されるべきモデル生成手順の要素が選択されるべきです。 「ショーのプレビューは、」その後、モデル生成プロセスを実行できるように、選択することができ、パラメータを微調整します。ftp_upload / 55471 / 55471fig6large.jpg」ターゲット= 『_空白』>この図の拡大版をご覧になるにはこちらをクリックしてください。
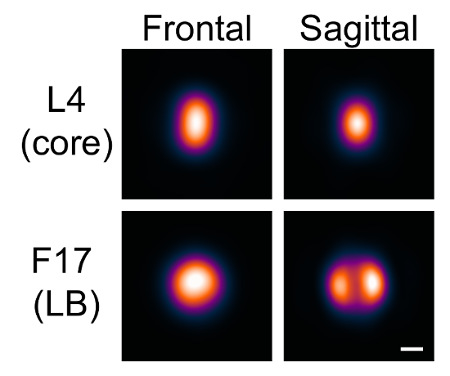
図7:VirusMapper で生成されたモデル。 mCherryを用いてタグ付けされたL4コアタンパク質およびEGFPでタグ付けされたF17横体タンパク質とワクシニアビリオンは、SIMを使用して画像化しました。プロトコールに記載されているようなモデルは、次に、ソフトウェアで生成されました。 2つの向き、正面と矢状は、横体の出現によって区別されます。スケールバー= 100 nmの。 この図の拡大版をご覧になるにはこちらをクリックしてください。
ディスカッション
この方法で、研究者は、ウイルスおよび他の高分子複合体のタンパク質構造の高精度、多チャンネルの2次元モデルを生成するために、SPA及びSR顕微鏡の力を結合するために装備されています。しかし、いくつかの重要な考慮事項が考慮されるべきです。
種子は、一貫して見られる構造を表現するために選択されるべきです。種子が選択される前にこのように、生のデータを慎重に検査しなければなりません。これは、バイアスされたモデルを防止するために重要です。選択肢は、モデル中の粒子の特定の数を含むのに必要な最小の類似性閾値の検査によって検証することができます。明らかに、シードの選択のために、より高いこのしきい値は、粒子の所与の数、構造データには明らかであることがよりためする必要があります。
データの不均一がある場合には、テンプレートマッチングのコンセプトは特に便利です。 viのあるすべての異なる構造sibleが特定されなければならないと異なったモデルは、それぞれの場合のために作成します。一つのチャンネルでの異種構造を分離するが、同時に第二のチャネルでモデルを作成することにより、パターンがそれはすぐに明らかにされていませんでし出現することがあります。
このアルゴリズムを使用する際に注意すべきもう1つの考慮事項は、反復手順が確率的非対称性を最大化することです。 2つの対称の最大値を有する構造をモデル化する場合、例えば、最大値の間のすべてのわずかな非対称性は、反復中に互いに整列され、最終的なモデルは、このように最大限に非対称です。これは、モデル化された構造で知られている対称性を反映していない場合、これは考慮すべきです。現在、この最大化を回避するための唯一の方法は、VirusMapperは、モデル生成プロセスに対称軸を組み込むことのために潜在的な開発が可能だろうが、1回の反復の数を制限することです。 VirusMapperのすべての新しいバージョンでは、のAvaIになります参照ウェブサイト上でlable( 材料表を参照してください)。また、ユーザーは任意の一般的なクエリに答えるために、ここでよくある質問を検索します。
説明したようにソフトウェアにより、ユーザは、モデル化することを望む特徴を視覚化するために十分な解像度で画像化することができる任意の構造に適用可能です。 SPAは、分解能を向上させることができるが、それは明らかにそうでないと見えない特徴の視認性を改善しないであろう。このプロトコルは、従って、データの品質を向上させる方法ではありません。任意の技術と同様に、イメージング戦略の慎重なサンプル調製および最適化は、クリーンデータと最良の結果のモデルを提供します。
SRイメージングモダリティの選択は、一般的には、手元にあるサンプルに依存し、また重要であると。 、VirusMapperはSIMとSTED 10でうまく動作することが確認されており、それはまた、高品質な局在化顕微鏡のデータを使用することができますが、注意が、この場合には注意が必要ですスパースとして標識は非対称性の最大化と同様の問題を引き起こす可能性があります。
現在、VirusMapperは、蛍光画像の単一粒子分析のみ汎用2D SPA平均化ソフトウェアのための唯一の自由に利用可能なアルゴリズムです。同じ原則4、6の使用をした他の研究では、8は、それぞれ特定の研究に特化したカスタムソフトウェアを使用していました。何のソフトウェアが提供されなかったが、3Dデータの再構築のための汎用アルゴリズムは、5、18を発表されています。
この記事に記載されているように使用される場合、VirusMapperは、ウイルスやその他の複合体の高分子タンパク質構造の、正確な正確、かつ堅牢なモデルを生成するために使用することができます。これらのモデルでは、研究者は、構造体の平均寸法の正確な測定を行うことができます検討中のURESは、潜在的に彼らがそうでなければ不可能でした生物学的な結論に到達することを可能にします。
また、この技術のマルチチャネル機能では、複合体中のタンパク質および成分を無制限にマッピングし、新規タンパク質組織を発見することが可能です。そのようなウイルスのライフサイクルの各段階として、異なる生物学的に関連する条件、ナノスケール構造の変化を調べると、生物学に貴重な洞察を提供する可能性を秘めています。
開示事項
著者は、開示することは何もありません。
謝辞
私たちは、VirusMapperのオリジナルの開発と検証への貢献のためコリーナ・ビアリ、ジェジー・サモールジ、ペドロ・マトス・ペレイラ、クリストファー・ブレック、とカトリン・シーラー感謝したいと思います。また、原稿の彼の重要な読書のためアルトゥール・ヤカムービック感謝したいと思います。この作品は、バイオテクノロジー・生物科学研究会議(BB / M022374 / 1)(RH)からの助成金によって賄われていました。分子細胞生物学のためのMRC研究所のコア資金、ユニバーシティ・カレッジ・ロンドン(JM)。欧州研究評議会(649101-UbiProPox)(JM)。医療研究評議会(MR / K015826 / 1)(RHとJM)。 RGは、工学・物理科学研究会議(EP / M506448 / 1)によって運営されています。
資料
| Name | Company | Catalog Number | Comments |
| Fiji | Open-source image analysis software | ||
| NanoJ-VirusMapper | developed by the Henriques lab | Open source-Fiji plugin (https://bitbucket.org/rhenriqueslab/nanoj-virusmapper) | |
| VectaShield antifade mounting medium | Vector Labs | H-100 | |
| Elyra PS1 | Zeiss | ||
| ZEN BLACK | Zeiss | Image processing software for SIM | |
| High performance coverslip | Zeiss | 474030-9000-000 | |
| TetraSpeck beads | ThermoFisher | T7279 |
参考文献
- Henriques, R., Griffiths, C., Rego, E. H., Mhlanga, M. M. PALM and STORM: Unlocking live-cell super-resolution. Biopolymers. 95 (5), 322-331 (2011).
- Gustafsson, N., Culley, S., et al. Fast live-cell conventional fluorophore nanoscopy with ImageJ through super-resolution radial fluctuations. Nat Commun. 7, 12471 (2016).
- Cheng, Y., Grigorieff, N., Penczek, P. A., Walz, T. A Primer to Single-Particle Cryo-Electron Microscopy. Cell. 161 (3), 438-449 (2015).
- Szymborska, A., de Marco, A., Daigle, N., Cordes, V. C., Briggs, J. A. G., Ellenberg, J. Nuclear pore scaffold structure analyzed by super-resolution microscopy and particle averaging. Science. 341 (6146), 655-658 (2013).
- Broeken, J., Johnson, H., et al. Resolution improvement by 3D particle averaging in localization microscopy. Methods Appl Fluoresc. 3 (1), 14003 (2015).
- Burns, S., Avena, J., Unruh, J., Yu, Z. Structured illumination with particle averaging reveals novel roles for yeast centrosome components during duplication. Elife. , (2015).
- Lelek, M., Di Nunzio, F., Henriques, R., Charneau, P., Arhel, N., Zimmer, C. Superresolution imaging of HIV in infected cells with FlAsH-PALM. Proc Nat Acad Sci U S A. 109 (22), 8564-8569 (2012).
- Laine, R. F., Albecka, A., Svan de Linde, ., Rees, E. J., Crump, C. M., Kaminski, C. F. Structural analysis of herpes simplex virus by optical super-resolution imaging. Nat Commun. 6, 5980 (2015).
- Schindelin, J., Arganda-Carreras, I., et al. Fiji: an open-source platform for biological-image analysis. Nat Methods. 9 (7), 676-682 (2012).
- Gray, R. D. M., Beerli, C. VirusMapper: open-source nanoscale mapping of viral architecture through super-resolution microscopy. Sci Rep. 6, 29132 (2016).
- Gustafsson, M. G. L. Surpassing the lateral resolution limit by a factor of two using structured illumination microscopy. J of Micros. 198 (2), 82-87 (2000).
- Hell, S. W., Wichmann, J. Breaking the diffraction resolution limit by stimulated emission: stimulated-emission-depletion fluorescence microscopy. Opt Let. 19 (11), 780 (1994).
- Chung, C. -. S., Chen, C. -. H., Ho, M. -. Y., Huang, C. -. Y., Liao, C. -. L., Chang, W. Vaccinia virus proteome: identification of proteins in vaccinia virus intracellular mature virion particles. J Virol. 80 (5), 2127-2140 (2006).
- Moss, B. Poxviridae: the viruses and their replication. Fields virology. , (2010).
- Condit, R. C., Moussatche, N., Traktman, P. In a nutshell: structure and assembly of the vaccinia virion. Adv Virus Res. 66 (6), 31-124 (2006).
- Schmidt, F. I., Bleck, C. K. E., et al. Vaccinia virus entry is followed by core activation and proteasome-mediated release of the immunomodulatory effector VH1 from lateral bodies. Cell Rep. 4 (3), 464-476 (2013).
- . Nanoj-virusmapper Available from: https://bitbucket.org/rhenriqueslab/nanoj-virusmapper (2016)
- Fortun, D., Guichard, P., Unser, M. Reconstruction From Multiple Poses in Fluorescence Imaging: Proof of Concept. IEEE J Sel Topics Signal Process. 10 (1), 61-70 (2016).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved