Method Article
CRISPRコンカテマーを用いたマウス小腸オルガノイドにおける複数の遺伝子ノックアウトのためのプロトコル
要約
このプロトコルは、CRISPR / Cas9技術を用いたマルチ遺伝子ノックアウトの作成に特に有用な、1つのガイドRNAコンカテマーベクターに複数の単一ガイドRNAをクローニングするための工程を記載する。腸オルガノイドにおける二重ノックアウトの生成は、この方法の適用可能性として示されている。
要約
CRISPR / Cas9技術は遺伝子機能を理解する上で不可欠な機能喪失研究の実現可能性とスピードを大幅に改善しました。高等真核生物では、パラロガス遺伝子は遺伝子の損失を補うことによって潜在的な表現型を隠蔽することができ、単一の遺伝子ノックアウトに依存する遺伝子研究から得られる情報を制限する。我々は、マウス小腸オルガノイドの一回トランスフェクション後にマルチ遺伝子ノックアウトを作製するために、ガイドRNA(gRNA)コンカテマーの新規な迅速なクローニング法を開発した。我々の戦略は、アニーリングされたgRNAオリゴとあらかじめ設計されたレトロウイルスベクターとのゴールデンゲートシャフリング反応を1回実行することにより、最大4つの個々のgRNAを単一のベクターにコンカテマー化することができます。これにより、最大4つの異なる遺伝子の同時ノックアウト、または複数のgRNAによる1つの遺伝子の標的化後のノックアウト効率の増加が可能になる。このプロトコルでは、詳細を示しますどのようにして複数のgRNAをレトロウイルスCRISPRコンカテマーベクターに効率的にクローン化するか、そしてどのように腸オルガノイドにおいて非常に効率的なエレクトロポレーションを達成するか。一例として、我々は、Wntシグナル伝達経路(Axin1 / 2およびRnf43 / Znrf3)の負の調節因子をコードする2対の遺伝子の同時ノックアウトが、腸オルガノイドを主要な成長因子の回収に耐性にすることを示す。
概要
逆遺伝学アプローチは、遺伝子の機能を調べるために広く用いられている方法である。特に、遺伝子の破壊が表現型変化を引き起こす機能喪失研究は、生物学的プロセスの理解を構築する上で重要な役割を果たす。 CRISPR / Cas9法は、ゲノム工学技術における最新の進歩を表し、細胞および生物における現在の遺伝学の実践に革命をもたらしました。 Cas9は、gRNAに相補的な特定のDNA配列に結合し、二本鎖切断(DSB)を生じるRNA誘導エンドヌクレアーゼである。このDSBは、相同組換えのためのDNA鋳型の非存在下で切断されたDNA鎖を誤りがちな非相同末端結合を介して再連結するDNA修復機構を採用し、その結果、ヌクレオチドの挿入または欠失が生じ、フレームシフト突然変異を引き起こす1 。
CRISPR / Cas9 apprの大きな容易性と多様性oachは、未知の遺伝子機能2、3の解明を目的としたゲノムスケールノックアウトスクリーンのため非常に魅力的なツールになりました。それにもかかわらず、冗長機能を有する複数のパラログが存在する場合、単一遺伝子ノックアウト手法は限られた用途に過ぎない。したがって、1つの遺伝子をアブレーションすることは、パラログによる可能性のある報酬を与えられた場合、その遺伝子の機能を決定するには不十分であり、表現型の変化がほとんどまたは全くない4 。したがって、遺伝的補償の影響を克服するために、異なるパラロガス遺伝子を標的とする複数のgRNAベクターを送達することによってパラログを同時にノックアウトすることが重要である。
CRISPR / Cas9のパラロガス遺伝子ノックアウトへの使用を拡大するために、我々は最近、最大4つのプレアニーリングされたgRNAを単一のレトロウイルスベクターにクローニングする迅速なワンステップクローニング法を開発した5 。 CRISPRコンカテマーと名づけられたバックボーンは、反復性のgRNA発現カセットを含むMSCVレトロウイルスプラスミド上に置いた。各カセットには、Type IIS制限酵素Bbs Iの2つの逆位認識部位が含まれています。これは、単管6でゴールデンゲートシャフリング反応を使用してオーバーハングが一致するアニーリングされたgRNAオリゴで不可逆的に置き換えられます。このクローニング方法は、消化および連結の反復サイクルからなり、 Bbs Iによって生成された異なるオーバーハング配列を利用することにより、複数のDNAフラグメントの同時アセンブリを可能にする。この酵素の一意性は、例えば、その認識外で非対称切断を行う能力シーケンス;したがって、各カセットは、 Bbs Iコア部位に隣接するカスタマイズされたオーバーハングを有する異なる配列を有することができ、このようにして、各gRNAをコンカテマーベクターの特定の位置および配向にクローニングすることができる。
原則の証明として、我々はこの戦略の使用をマウスエレクトロポ5の一周で同時にWnt経路のパラロガス負の調節因子の二対を破壊することによって腸のオルガノイド。
過去数年の間、多くの他のグループは、ヒト細胞株8、9、ゼブラフィッシュ10及び大腸菌(Escherichia coli)などの様々なモデル系、多遺伝子ノックアウトを達成するために7をシャッフルゴールデンゲートを用いて構成された多重gRNA発現ベクターに基づく同様の戦略を開発しました11 。それらのプロトコルでは、gRNAを最初に個々の中間ベクターにクローン化し、次いで一緒に組み立てて1つの最終産物にする。対照的に、CRISPRコンカテマー戦略の主な利点は、単一のBbs Iシャフリングクローニングステップの利便性です。他のgRNAコンカテマーと同様に、本発明者らの方法は、最大4つの同時ノックアウト異なる遺伝子または複数のgRNAを有する1つまたは2つの遺伝子の標的化後のCRISPRノックアウト効率の増加( 図1 )。
このプロトコールでは、gRNA設計からゴールデンゲート反応まで、そして成功したクローニングの確認まで、CRISPRコンカテマーベクターの生成におけるすべてのステップを詳細に記述する。我々はまた、エレクトロポレーションおよびその後の成長因子回収実験によって、CRISPRコンカテマーのマウス小腸オルガノイドへのトランスフェクションのための非常に効率的なプロトコールを提供する。
プロトコル
1. CRISPR-コンカテマーベクターのためのgRNA設計
注:このセクションの目的は、最適なターゲティング戦略を選択する方法、およびCRISPRコンカテマーベクターの特定のオーバーハングを含むgRNAを設計する方法を説明することです。
- 選択したCRISPR gRNA設計ツールを使用して目的の遺伝子に対するgRNAを設計する。例については表の表を参照してください。
注:1組のパラロガス遺伝子を標的とする場合、1遺伝子につき1つのgRNAを設計することは可能ですが、遺伝子ごとに2つのgRNAを設計してダブルノックアウトを達成する可能性を高めることが推奨されます ( 図1 )。 - 制限地図作成ツールを使用してgRNAにBbs I認識部位が含まれていないことを確認します(例の表を参照)。
- 表1に示すように、各オリゴに特異的CRISPR-コンカテマーベクターオーバーハングを付加する。
表1:CRISPRコンカテマーベクターの各カセットのオーバーハング。
2.CRISPR-コンカテマーベクターへのgRNAのクローニング
- オリゴのリン酸化およびアニーリング
注:この手順は、どのようにto各gRNAオリゴのトップストランドおよびボトムストランドをアニールする方法、および単一の反応でそれらの末端をリン酸化する方法。- 以下の手順に従って、オリゴをリン酸化し、氷上で上鎖および下鎖をアニーリングするための反応混合物を調製する。
注:すべてのオリゴを1つの反応にプールすることができます。例えば、4gRNAコンカテマーベクターの場合、8オリゴを一緒にプールする。 - 3つのコンカテマーについて、3.0μLのgRNAトップ鎖(各gRNAから1.0μL、10μM、1μL/ gRNA)、3.0μLのgRNAボトム鎖(各gRNAから1.0μL;10μM、1μL/ gRNA)、2.0μLのT4 DNAリガーゼ緩衝液(10×)、1.0μLのT4 PNK、および総容量20.0μLまでH 2 Oを加える。
- ピペットでよく混合し、以下の設定を使用してサーモサイクラーでこれを実行します:37℃で30分間、95℃で5分間、25℃で0.3℃/分で下降、4℃で保持。
- 以下の手順に従って、オリゴをリン酸化し、氷上で上鎖および下鎖をアニーリングするための反応混合物を調製する。
- Bbs私はシャッフル反応
注:このセクションでは、プレアニーリングされたgRNAオリゴは、消化とライゲーションのサイクルを交互に繰り返すことによって、コンカテマーベクターの適切な位置に1ステップで取り込まれます。- DNase / RNaseを含まない水で1:100の反応液を希釈して、3および4gRNAコンカテマーベクターを作製する。
注:2 gRNAコンカテマーをクローニングする場合、このステップは必要ありません 。 - 以下の手順に従って、氷上で反応をシャフリングするBbsを組み立てます。ベクターのみを含むネガティブコントロールを含めます。
- 100μgのCRISPRコンカテマーベクター、10.0μLのオリゴ混合物、1.0μLのBSA含有制限酵素緩衝液(10倍)、 1.0μLDTT (10mM)、 1.0μLATP (10mM)、 1.0μLBbs I、 1.0μLT7リガーゼ、およびH 2 Oを総量20.0μLまで含む。
- ピペット操作でよく混合し、以下の設定を使用してサーモサイクラーでこれを実行する:3および4gRNA-コンカテマーをクローニングするために50サイクルを実行し、 保持する、2gRNA-コンカテマーについて25サイクル以上。
- DNase / RNaseを含まない水で1:100の反応液を希釈して、3および4gRNAコンカテマーベクターを作製する。
- エキソヌクレアーゼ処理
注:線状化したDNAの痕跡を取り除いてクローニングの効率を上げるため、この手順を強くお勧めします。- 次のように、 Bbs Iシャフリング反応をDNAエキソヌクレアーゼ( 表の表を参照)で処理する。
- 前のステップ(2.2.3)から11.0μLのライゲーションミックスを取り出し、1.5μLのエキソヌクレアーゼ緩衝液(10×)、1.5μLのATP(10mM)、1.0μLのDNAエキソヌクレアーゼを添加し、総容量を水で15.0μLにする。 37℃で30分間インキュベートし、続いて70℃で30分間インキュベートする。
注:このステップでは、混合物中の残留線状化DNAが除去され、クローニング効率が向上します。 - 2μLの反応混合物を用いて化学的に変換する熱ショックによるテント大腸菌 12 。
注:あるいは、-20℃で最大1週間反応を保存することができます。
- 制限消化
注:この手順の目的は、制限消化によってクローニング手順の成功を評価することです。- CRISPR-コンカテマーベクター中のgRNAインサートの存在を確認するために、接種ループを有する4〜8個の細菌コロニーを選択し、4mLのLB培地中で37℃で一晩オービタルシェーカーで各クローンを増殖させる。製造業者の指示に従ってプラスミドミニプレップキットを用いてDNAを抽出する( 表の表を参照)。
- 細菌インキュベーター内で37℃で3時間インキュベートすることにより、10μLの反応液中に10UのEcoRI + 5UのBglIIを含む200ngのDNAを消化する。サイズ比較のための陽性対照として対応する元のベクターと別の反応混合物を含める。
注:ティこれらの2つの制限酵素が全コンカテマーを排出するため、すべてのコンカテマーが存在するかどうかを確認する。これらは、各コンカテマーの予想されるサイズであり、2gRNAコンカテマー(800bp)、3gRNAコンカテマー(1.2kbp)、および4gRNAコンカテマー(1.6kbp)である。 - 1%アガロースゲル上で90Vで約20分間消化反応を行う。
- UVトランスイルミネーターを使用してゲルを視覚化する。正確な挿入サイズのクローンを、そのバンドパターンが元のベクターのものと一致すること、およびDNAラダーを使用して各フラグメントが予想されるサイズを有することを確認することによって同定する。
- 選択したクローンを5U Bbs Iで37℃で3時間、細菌インキュベーターで消化します。対照として対応する元のベクターと別の反応混合物を含める。
注:この追加の消化ステップは、gRNAが正しい位置にクローニングされ、結果としてすべてのBbs I認識部位が失われていることを確認することです。 - 消化反応を実行する1%アガロースゲル(90V、約20分間)。彼らがgRNAだけを含んでいるので、 Bbs Iによって切断されていないベクターだけを修正すると考えてください。
- ベクターシーケンシング
注:このステップの目的は、制限消化分析によって正しいと同定されたベクター中のgRNA配列の存在を確認することである。- 以下のプライマーを用いてSanger配列決定13により陽性コンカテマーベクターを確認する:
フォワード:TCAAGCCCTTTGTACACCCTAAG(最初のgRNAカセットをチェックするため)
Linker1_Forward:GACTACAAGGACGACGATGACAA(2番目のgRNAカセットをチェックするため)
Linker2_Reverse:GGCGTAGTCGGGCACGTCGTAGGGGT(第2のgRNAカセットをチェックするため)
Linker2_Forward:ACCCCTACGACGTGCCCGACTACGCC(第3のgRNAカセットをチェックするため)
Linker3_逆:TCCTCCTCTGAGATCAGCTTCTGCAT(第3のgRNAカセットをチェックするため)
逆:AGGTGGCGCGAAGGGGCCACCAAAG(最後のgRNAをチェックするため)カセット) - シークエンシングリードで配列を検索することにより、すべてのgRNAの存在を確認します。
- 以下のプライマーを用いてSanger配列決定13により陽性コンカテマーベクターを確認する:
3.エレクトロポレーションによる腸オルガノイドのトランスフェクション
注:この手順は、Fujii et al 。によって発行されたプロトコルに基づいています。 2015年には、マウス小腸オルガノイド培養液14のための適応を持ちます。
- エレクトロポレーション前
注:このセクションでは、エレクトロポレーションの前にマウスの腸オルガノイドを調製する方法について説明します。これは、エレクトロポレーションの間の起こりうる毒性作用を防止する。- トランスフェクション手順の0日目に、1:2の比率でオルガノイドを分割する。
注:腸のオルガノイド培養物は、以前に確立されたプロトコールに従って陰窩分離を行うことによって得ることができる15 。を参照してください - 電気穿孔のためにオルガノイドを分割する場合、トランスフェクション当たり48ウェルプレートの最低6ウェルを種にする。
- 20μL、基底行列滴でオルガノイドを播種し、37℃でWENR +のNIC媒体(のWnt + EGF +ノギン+ Rスポンジン+ニコチンアミド)でそれらを増殖、加湿インキュベーター中5%CO 2(としては、以前15に記載) 。
- トランスフェクション手順の0日目に、1:2の比率でオルガノイドを分割する。
- 2日目にWENR + Nicを抗生物質を含まないEN(EGF + Noggin)+ CHIR99021(グリコーゲン合成酵素キナーゼ-3阻害剤)+ Y-27632(ROCK阻害剤)250μLに置き換えて培地を交換する( 表2参照)。
注:すべてのステップで、48ウェルプレートの各ウェルに添加する培地の量は250μLです。 - 3日目に、オルガノイド培地を抗生物質を含まないEN + CHIR99021 + Y-27632 + 1.25%v / vジメチルスルホキシド(DMSO)に交換する。
注意:ここでは、オルガノイドを機械的および化学的解離によって小細胞クラスターに分ける方法を説明します。これらの手順は、手順の成功に不可欠です。
- 4日目に、1mLのピペットチップを用いて基底マトリックスのドームを破壊し、オルガノイドを1.5mLチューブに移す。 48ウェルプレートの4つのウェルの内容物をチューブにプールする。
- P200ピペットで上下に約200回ピペット操作することにより、オルガノイドを小さな断片に機械的に破壊する。室温で遠心分離し、600×gで5分間遠心分離する。
- 培地を除去し、ペレットを細胞培養グレードの組換えプロテアーゼ(材料表参照)1 mLに再懸濁する。 37℃で最大5分間インキュベートし、4倍の対物レンズを備えた倒立光学顕微鏡下で50μLのサンプルを点検する。
注:エレクトロポレーション後の細胞生存を増加させるので、10〜15個の細胞のクラスターが望ましい。 - 細胞懸濁液を低結合15 mLチューブに移し、抗生物質を含まない9mLの基礎培地を添加することにより解離させた( 表2参照)。室温、600xgで5分間遠心分離し、上清を捨て、ペレットを1mLの還元血清培地( 表の表を参照)に再懸濁する。
- Bürkerのチャンバーで細胞数を数え、エレクトロポレーション反応あたり最低1 x 10 5個の細胞を使用する。還元血清培地9mLを15mLチューブに加え、室温で遠心分離し、400×gで3分間遠心分離する。
注:以下のセクションでは、エレクトロポレーションを実行し、後にオルガノイドを回復させる方法について説明します。
- すべての上清を除去し、ペレットをエレクトロポレーション溶液で再懸濁する( 表の表を参照)。総量10μgのDNAを細胞懸濁液に添加し、エレクトロポレーション溶液を最終容量100μLに加え、細胞DN氷上での混合物。 1:1の比でCas9発現プラスミド( 例えば、 Addgene#41815)と組み合わせてCRISPR-コンカテマーベクターを使用する。
注記:添加するDNAの総容量は、総反応量の10%以下である必要があります。 - トランスフェクション効率( 例: pCMV-GFP、Addgene#11153、または一般的なGFP発現プラスミド)を評価するために、GFPプラスミドを含む別のトランスフェクションミックスを含めます。
- 細胞-DNA混合物をエレクトロポレーションキュベットに加え、エレクトロポレーターチャンバーに入れる。エレクトロポレーターの適切なボタンを押してインピーダンスを測定し、それが0.030-0.055Ωであることを確認します。 表3に示す設定に従ってエレクトロポレーションを行います。
注:インピーダンス値が許容範囲外になる場合は、キュベット内の溶液量を調整してください。 - エレクトロポレーションバッファー+ Y-27632400μLをキュベットに加え、すべてを1.5mLチューブに移す。インカバ室温で30分間インキュベートして細胞を回収し、続いてそれらを室温で3分間400xgで回転させる。
- 上清を除去し、ペレットを20μL/ウェルの基底マトリックスに再懸濁する。種子は、約1×10 4〜1×10 5個の 48ウェルプレートにウェル当たり細胞およびEN + CHIR99021 + Y-27632 + 1.25%(v / v)のDMSO媒体を加えます。 37℃でインキュベートする。
- 5日目に、培地をEN + CHIR99021 + Y-27632に交換し、GFP発現を観察することによりトランスフェクション効率を確認する( 図2 )。オルガノイドを37°Cに保ち、2日後にEN + CHIR99021 + Y-27632培地を再懸濁する。
- 9日目に、培地をWENR + Nic + Y-27632に交換し、37℃でインキュベートする。
注:Y-27632は、7〜10日後(16〜19日目)に除去することができます。
| ポーリングパルス | 転送パルス | |
| 電圧 | 175V | 20V |
| パルス長 | 5ミリ秒 | 50msec |
| パルス間隔 | 50msec | 50msec |
| パルス数 | 2 | 5 |
| 減衰率 | 10% | 40% |
| 極性 | + | +/- |
表3:エレクトロポレーションの設定。
4.成長因子退出
注:ここでは、腸オルガノイドにおけるWnt経路の負のレギュレーターをノックアウトする際に、成長因子回収実験を行う方法を例示する。
- 10-14日後電気穿孔法を用いて、上記の手順(3.2.1-3.2.2)に従って48ウェルプレート中で1:3の比率でオルガノイドを分割する。
- オルガノイドペレットを20μLの基底膜マトリックスに再懸濁し、37℃で10分間固化させる。次に、標的遺伝子のノックアウトが達成されているかどうかを試験するために、250μLの増殖因子枯渇培地( 例えば、 EN)をオーバーレイする。5 。
- 野生型野生型(WT)対照オルガノイドおよび変異オルガノイド5、15の間の生存の差を確認するために3継代- 2の最小の成長因子欠乏条件下オルガノイドを分割します。
注:野生型オルガノイドは、2つの継代にわたって増殖因子欠乏培地で生存できないはずであるが、突然変異株は増殖できるはずである。
結果
コンカテマーベクター中に正しい数のgRNAインサートが存在することを確認するために、全てのgRNA発現カセットに隣接する酵素( EcoR I + Bgl II)を用いて制限消化を行う(各カセットのサイズは〜400bp、 図1 )。例えば、4gRNAコンカテマーベクターを作製する場合、アガロースゲル中のより低いバンドの予想サイズは約1.6Kbpであり;これより低いバンドは、4つのgRNAカセットのすべてがベクターに挿入されていないことを示しています( 図2A )。さらに、 Bbs I認識部位がすべて失われ、酵素がベクターを切断しないことを確認することが常に推奨されます( 図2B )。
構築物が確認されたら、それらをエレクトロポレーションによりマウス腸オルガノイドに送達して最適化を達成することができるGFPコントロール( 図3 )で示されるように、1レベルのトランスフェクション効率(最大70%)を達成します。
最後に、この戦略の効率を機能的に試験するために、Cas9でトランスフェクトした腸オルガノイドおよびAxin1 / 2およびRnf43 / Znrf3に対するコンカテマーベクターをEN(R-スポンジン離脱)およびEN + IWP2(R-スポンジンおよびWnt離脱、IWP2 :Porcupine阻害剤、2.5μM)培地で最低3継代培養した( 図4 )。トランスフェクトされていないWTオルガノイドは両方の条件下で死亡したが、Axin1 / 2ノックアウトオルガノイドはWnt経路の下流の活性化のために両方で生存した。さらに、Rnf43 / Znrf3変異オルガノイドは、R-スポンジンの非存在下で生存するが、IWP2の存在下では生き残ることができず、これは経路を活性化するWntの枯渇を引き起こす。まとめると、これらの観察は、これらのパラログの対のノックアウトが、t彼はオルガノイドの表現型を期待していた。これらの結果の詳細は、 Developmental Biology 5に掲載されています。
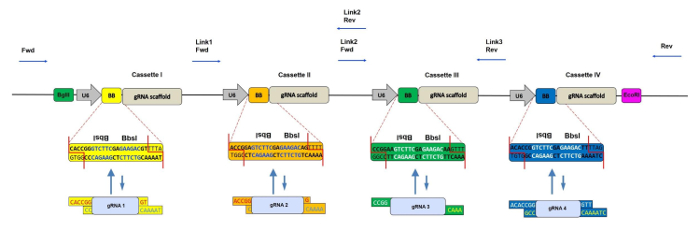
図1:4つのカセットを備えたCRISPRコンカテマーの模式図 U6プロモーター、2つの逆方向反復BbsI部位(BBとも示される)およびgRNA足場をこの順序で含む各400bpカセットを有する4つのgRNA-コンカテマーベクターのスキーム。シャッフリング反応の間、 BbsI部位は、 突き合わせに一致するgRNA断片によって置換され、結果として失われる。 gRNAオリゴの正しい挿入を確認するための配列決定プライマーの結合部位を青色矢印で示す。 Fwd =フォワードプライマー、Rev =リバースプライマー、Link 1/2/3 =リンカー領域1/2/3。 してくださいこの図のより大きなバージョンを見るにはここを舐めてください。

図2:コンカテマーベクターの代表的な消化パターン。 ( A ) EcoR IおよびBgl IIを用いた3および4gRNAコンカテマーベクターの二重消化。正確な消化パターンは緑色のダニでマークされていますが、1または2のgRNA挿入のみを持つベクターは赤い十字でマークされています。レーン1は、陽性対照として使用した4gRNA連結体親ベクターの消化を示す(「+」印)。同様に、レーン5は、3つのgRNAコンカテマー親ベクターの消化を示し、「+」で示す。 ( B )消化されたコンカテマーベクター(緑色のダニで示される)の正しいサイズを示す、 Bbs Iによる消化。 BbsI部位を欠失したgRNA含有コンカテマーベクターの消化を陽性対照として使用するdは "+"でマークされます。 この図の拡大版を見るには、ここをクリックしてください。
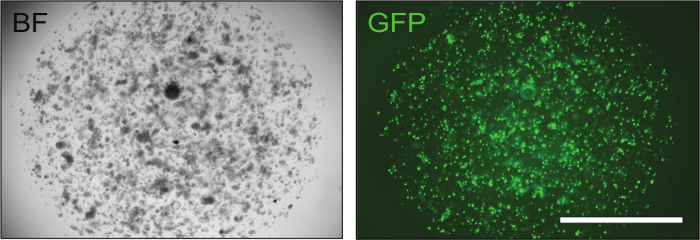
図3:成功したElectroporated腸オルガノイドの代表的なイメージ。トランスフェクション効率を評価するには、GFPプラスミドのトランスフェクションが重要です。エレクトロポレーションの約24時間後に、少数の細胞を含むオルガノイドが既に見え、エレクトロポレーション手順が成功した場合、それらの70%までが緑色蛍光を示す。 BF =明視野、GFP =緑色蛍光タンパク質。スケールバー=2,000μm。 この図の拡大版を見るには、ここをクリックしてください。
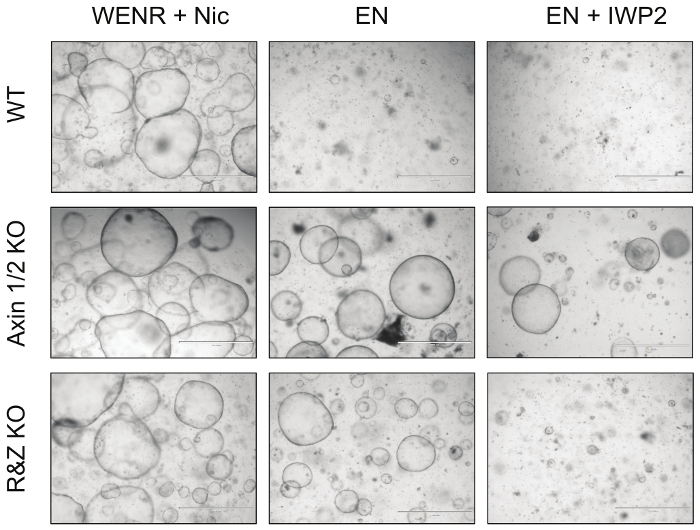
図4:変異腸オルガノイドの代表的な画像。 Wnt経路Axin1およびRnf43の負の調節因子のノックアウトは、それらのパラログと一緒に、腸オルガノイドを増殖因子欠乏に対して抵抗性にする。特に、Axin1 / 2ノックアウトオルガノイド(Axin1 / 2KO)は、R-スポンジン(EN:EGF +ノギン)とWnt(EN + IWP2:EN + Porcupineインヒビター)の両方の非存在下で増殖することができるのに対して、Rnf43 / Znrf3変異オルガノイド(R&Z KO)は、R-スポンジン(EN)の非存在下でのみ生き残ることができる。対照的に、WTオルガノイドは、対照培養条件、WENR + Nic(Wnt + EGF + Noggin + R-スポンジン+ニコチンアミド)においてのみ生存することができる。スケールバー=1,000μm。 この図の拡大版を見るには、ここをクリックしてください。
| 基礎培地 | コメント | |
| 4℃で4週間保存する | ||
| 細胞培養培地 | 500 mL | 材料の表を参照 |
| L-グルタミン100倍 | 5mL | |
| 緩衝剤1 M | 5mL | 材料の表を参照 |
| ペニシリンストレプトマイシン100倍 | 5mL | |
| WENR + Nic(Wnt + EGF +ノギン+ R-スポンジン+ニコチンアミド) | ||
| 4℃で2週間保存する | ||
| 基礎培地 | 最大50 mL | |
| ニューロン細胞無血清サプリメント(50倍) | 1 mL | maの表を参照テリアール |
| ニューロン細胞無血清サプリメント(100倍) | 500μL | 材料の表を参照 |
| n-アセチルシステイン(500mM) | 125μL | |
| マウスEGF(100μg/ mL) | 25μL | |
| マウスノギン(100μg/ mL) | 50μL | |
| R-スポンジン条件培地 | 5mL | |
| Wnt3a条件培地 | 25 mL | |
| ニコチンアミド(1 M) | 250μL | |
| EN + CHIR + Y-27632(EGF +ノギン+ CHIR + Y-27632) | ||
| 4℃で2週間保存する | ||
| ペニシリン・ストレプトマイシンを含まない基本培地 | 最大20 mL | |
| ニューロン細胞無血清サプリメント(50倍) | 400μL | 材料の表を参照 |
| ニューロン細胞無血清サプリメント(100倍) | 200μL | 材料の表を参照 |
| n-アセチルシステイン(500mM) | 50μL | |
| マウスEGF(100μg/ mL) | 10μL | |
| マウスノギン(100μg/ mL) | 20μL | |
| Y-27632(10μM) | 20μL | |
| CHIR99021(8μM) | 10μL | |
| EN(EGF +ノギン) | ||
| 4℃で4週間保存する | ||
| 基礎培地 | 最大50 mL | |
| ニューロン細胞無血清サプリメント(50倍) | 1 mL | 材料表を参照 |
| ニューロン細胞無血清サプリメント(100倍) | 500μL | 材料表を参照 |
| n-アセチルシステイン(500mM) | 125μL | |
| マウスEGF(100μg/ mL) | 25μL | |
| マウスノギン(100μg/ mL) | 50μL |
表2:オルガノイド培地の組成。
ディスカッション
このプロトコールでは、複数の遺伝子を同時にノックアウトするために、CRISPRコンカテマーを生成し、マウス腸オルガノイドにCRISPRコンカテマーを適用するために必要なすべてのステップを詳述します。前述のように、この戦略には、スピード、高効率、コスト効率などのいくつかの利点があります。
手順全体を首尾よく行うためには、考慮すべき重要な側面がいくつかあります。第1に、すべてのgRNAオリゴが適切にアニーリングされ、リン酸化されていることが必須であり、それはBbs Iクローニング反応のための出発物質であり、それ自体が非常に効率的であるからである。第2に、オルガノイドをエレクトロポレーションすると、条件ごとに細胞が多く使用されるほど、トランスフェクション効率が最大に高くなります。さらに、細胞解離後、小さな細胞クラスターが単一細胞より優勢であることも重要である。
それにもかかわらず、技術的な問題に遭遇する可能性があります最初にクローニングまたはトランスフェクションのいずれかを試みるときはレム(lems) gRNAのクローニング中に問題が発生した場合は、gRNAオリゴ配列を再度確認し、正確であれば、制限消化スクリーニング用に追加の細菌コロニーを選択することをお勧めします。トランスフェクション効率および細胞生存率がエレクトロポレーション後に低い場合、条件ごとにより多くの細胞を使用し、細胞解離の時間を3分に短縮することを推奨します。
CRISPRコンカテマーの生成は比較的安価で容易であるが、オルガノイド培養に関連するコストおよびその労働集約的性質によってスケールが制限されるので、オルガノイドにおいてより大きなスケールの遺伝子スクリーニングを行うことはできない。この場合、CRISPR-コンカテマー法は、HEK293およびマウス胚性幹細胞などの細胞系とも適合性があることに言及することは重要である。
セルラーシステムにかかわらず、このsの別の潜在的な欠点3つまたは4つの異なる遺伝子を同時にノックアウトすることを目指すと、怒りを感じることがあります。例えば、各gRNAは異なるターゲティング効率を有するであろうし、同時に全ての遺伝子をヒットする変化は比較的低くてもよい。この理由から、コンカテマーシステムを用いて同じ遺伝子に対して複数のgRNAを指向させることが望ましい。
同様にゴールデンゲートシャッフリングに基づく代替戦略は、多重gRNAベクトル7、8を生成するために、長年にわたって提案されてきました。しかし、我々の方法では、複数回のgRNAを1回のクローニングで単一のレトロウイルスベクターに直接組み立てることが可能であり、これにより、パラログを標的とするためのgRNAライブラリを生成するのに適している。
当社のCRISPRコンカテマーは、MSCVレトロウイルスベクターのバックボーンに組み込まれています。したがって、gRNAコンカテマー含有レトロウイルスは、過剰発現する安定な細胞株を生成するために使用され得るgRNAを分解する。 Cas9誘導性の系と組み合わせると、我々の系を用いて誘導性のパラログノックアウトを行うことができる。
要約すると、ここでは、最大4つの異なるgRNAを同じベクターに1ステップでクローン化する方法と、高いトランスフェクション効率でオルガノイド培養にこの戦略を適用する方法について説明します。さらに、私たちは、手順全体を通して成功のチャンスを最大にするために有用な提案を提供します。
開示事項
著者は何も開示することはない。著者らは、利益相反を宣言していない。
謝辞
私たちは、原稿の批判的な読み方についてクリストファー・ヒンドレーに感謝します。 AMは、Wntsapp(Marie Curie ITN)、AA-Rによってサポートされています。メディカルリサーチカウンシル(MRC)とBK.Kによってサポートされています。 Wellcome TrustとRoyal Society [101241 / Z / 13 / Z]のSir Henry Dale Fellowshipによってサポートされ、Wellcome TrustとMRCからWellcome Trust - MRC Cambridge Stem Cell Institute 。
資料
| Name | Company | Catalog Number | Comments |
| Optimized CRISPR Design Tool | Feng Zhang group | CRISPR gRNA design tool; http://crispr.mit.edu/ | |
| Webcutter 2.0 | restriction mapping tool; http://rna.lundberg.gu.se/cutter2/ | ||
| T4 PNK (Polynucleotide Kinase) | New England Biolabs | M0201L | |
| T4 DNA ligase buffer | New England Biolabs | M0202S | |
| T7 DNA Ligase | New England Biolabs | M0318L | |
| DTT (dithiothreitol) | Promega | P1171 | |
| ATP (adenosine triphosphate) | New England Biolabs | P0756S | |
| FastDigest BbsI (BpiI) | Thermo Fisher | FD1014 | |
| Tango buffer (BSA-containing restriction enzyme buffer) | Thermo Fisher | BY5 | |
| BglII | New England Biolabs | R0144 | |
| EcoRI | New England Biolabs | R0101 | |
| Plasmid-safe exonuclease | Cambio | E3101K | |
| Thermal cycler | Applied biosystems | 4359659 | |
| 10G competent E. coli bacteria | Cambridge Bioscience | 60108-1 | |
| Plasmid mini kit | Qiagen | 12125 | |
| Table top microcentrifuge | Eppendorf | UY-02580-01 | |
| Inoculating loops | Microspec | PLS5 | |
| Bacteria incubator | Sanyo | MIR-262 | |
| Luria-Bertani broth (LB) | Sigma-Aldrich | L3522 | |
| Agarose | Sigma-Aldrich | A4718 | |
| Agarose gel electrophoresis apparatus | Bioneer | A-7020 | |
| Advanced DMEM/F12(cell culture medium) | Invitrogen | 12634-034 | |
| Glutamax (L-Glutamine) 100x | Invitrogen | 35050-068 | |
| HEPES 1 M (buffering agent) | Invitrogen | 15630-056 | |
| Penicillin-streptomycin 100x | Invitrogen | 15140-122 | |
| B27 supplement (Neuronal cell serum-free supplement) 50x | Invitrogen | 17504-044 | |
| N2 supplement (Neuronal cell serum-free supplement) 100x | Invitrogen | 17502-048 | |
| n-Acetylcysteine 500 mM | Sigma-Aldrich | A9165-5G | |
| Mouse EGF 500 µg/mL | Invitrogen Biosource | PMG8043 | |
| Mouse Noggin 100 µg/mL | Peprotech | 250-38 | |
| Nicotinamide 1 M | Sigma | N0636 | |
| R-Spondin conditioned medium | n.a. | n.a. | Produced in house from HEK293 cells, for details see Sato and Clevers 2013 |
| Wnt conditioned medium | n.a. | n.a. | Produced in house from HEK293 cells, for details see Sato and Clevers 2013 |
| Y-27632 10 µM | Sigma-Aldrich | Y0503-1MG | |
| Standard BD Matrigel matrix | BD Biosciences | 356231 | |
| 48-well Plate | Greiner Bio One | 677980 | |
| CHIR99021 | Sigma-Aldrich | A3734-1MG | |
| IWP-2 | Cell Guidance Systems | SM39-10 | |
| TrypLE (recombinant protease) | Invitrogen | 12605-010 | |
| Opti-MEM (reduced serum medium ) | Life technologies | 51985-034 | |
| Electroporation Cuvettes 2mm gap | NepaGene | EC-002S | |
| Low binding 15 mL tubes | Sigma-Aldrich | CLS430791 | |
| Bürker’s chamber | Sigma-Aldrich | BR719520-1EA | |
| NEPA21 Super Electroporator | NepaGene | contact supplier | |
| Protein LoBind tubes low binding | Thermo Fisher | 10708704 | |
| BTXpress electroporation buffer | Harvard Apparatus | 45-0805 | |
| DMSO (Dimethyl sulfoxide) | AppliChem | A3672 |
参考文献
- Mali, P., et al. RNA-Guided Human Genome Engineering via Cas9. Science. 339 (6121), 823-826 (2013).
- Koike-Yusa, H., Li, Y., Tan, E. -. P., Velasco-Herrera, M. D. C., Yusa, K. Genome-wide recessive genetic screening in mammalian cells with a lentiviral CRISPR-guide RNA library. Nat Biotech. 32 (3), 267-273 (2014).
- Bassett, A. R., Kong, L., Liu, J. L. A Genome-Wide CRISPR Library for High-Throughput Genetic Screening in Drosophila Cells. J Genet Genomics. 42 (6), 301-309 (2015).
- Diss, G., Ascencio, D., DeLuna, A., Landry, C. R. Molecular mechanisms of paralogous compensation and the robustness of cellular networks. J Exp Zool B Mol Dev Evol. 322 (7), 488-499 (2014).
- Andersson-Rolf, A., Merenda, A., Mustata, R. C., Li, T., Dietmann, S., Koo, B. -. K. Simultaneous paralogue knockout using a CRISPR-concatemer in mouse small intestinal organoids. Dev Biol. 420 (2), 1-7 (2016).
- Ran, F. A., Hsu, P. P. D., Wright, J., Agarwala, V., Scott, D., Zhang, F. Genome engineering using the CRISPR-Cas9 system. Nat Protoc. 8 (11), 2281-2308 (2013).
- Engler, C., Gruetzner, R., Kandzia, R., Marillonnet, S. Golden gate shuffling: A one-pot DNA shuffling method based on type IIs restriction enzymes. PLoS ONE. 4 (5), (2009).
- Sakuma, T., Nishikawa, A., Kume, S., Chayama, K., Yamamoto, T. Multiplex genome engineering in human cells using all-in-one CRISPR/Cas9 vector system. Sci Rep. 4 (5), 5400 (2014).
- Kabadi, A. M., Ousterout, D. G., Hilton, I. B., Gersbach, C. A. Multiplex CRISPR/Cas9-based genome engineering from a single lentiviral vector. Nucleic Acids Res. 42 (19), 1-11 (2014).
- Yin, L., et al. Multiplex Conditional Mutagenesis Using Transgenic Expression of Cas9 and sgRNAs. Genetics. 200 (2), 431-441 (2015).
- Cress, B. F., Toparlak, O. D., et al. CRISPathBrick: Modular Combinatorial Assembly of Type II-A CRISPR Arrays for dCas9-Mediated Multiplex Transcriptional Repression in E. coli. ACS Synth Biol. 4 (9), 987-1000 (2015).
- Froger, A., Hall, J. E. Transformation of plasmid DNA into E. coli using the heat shock method. J Vis Exp. (6), e253 (2007).
- Zimmermann, J., Voss, H., Schwager, C., Stegemann, J., Ansorge, W. Automated Sanger dideoxy sequencing reaction protocol. FEBS Letters. 233 (2), 432-436 (1988).
- Fujii, M., Matano, M., Nanki, K., Sato, T. Efficient genetic engineering of human intestinal organoids using electroporation. Nat. Protoc. 10 (10), 1474-1485 (2015).
- Andersson-Rolf, A., Fink, J., Mustata, R. C., Koo, B. -. K. A video protocol of retroviral infection in primary intestinal organoid culture. J Vis Exp. (90), e51765 (2014).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved