Method Article
Um protocolo para múltiplo knockout de genes em ratos Organoids Intestinal Pequeno Usando um CRISPR-concatemer
Neste Artigo
Resumo
Este protocolo descreve as etapas para a clonagem de vários RNAs de guia único em um vetor de guia de RNA concatemer, que é de uso particular na criação de knockouts multi-genes usando a tecnologia CRISPR / Cas9. A geração de knockouts duplos em organoides intestinais é mostrada como uma possível aplicação deste método.
Resumo
A tecnologia CRISPR / Cas9 melhorou muito a viabilidade e a velocidade dos estudos de perda de função que são essenciais na compreensão da função genética. Em eucariotas superiores, genes paralogous podem mascarar um fenótipo potencial, compensando a perda de um gene, limitando assim a informação que pode ser obtida a partir de estudos genéticos que dependem de knockouts de genes únicos. Desenvolvemos um novo método de clonagem rápida para concatestores de guia RNA (gRNA) para criar knockouts de múltiplos genes após uma única rodada de transfecção em organoides de intestino delgado de mouse. Nossa estratégia permite a concatenação de até quatro gRNAs individuais em um único vetor, realizando uma única reação de arrastar Golden Gate com oligos de gRNA recozidos e um vetor retroviral pré-projetado. Isso permite o nocaute simultâneo de até quatro genes diferentes, ou o aumento da eficiência do knockout após o direcionamento de um gene por vários gRNAs. Neste protocolo, mostramos em detalhesComo clonar de forma eficiente múltiplos gRNAs no vetor retroviral CRISPR-concatemer e como conseguir eletroporação altamente eficiente em organoides intestinais. Como exemplo, mostramos que o nocaute simultâneo de dois pares de genes que codificam reguladores negativos da via de sinalização de Wnt (Axin1 / 2 e Rnf43 / Znrf3) torna os organoídeos intestinais resistentes à retirada dos principais fatores de crescimento.
Introdução
A abordagem de genética reversa é um método amplamente utilizado para investigar a função de um gene. Em particular, os estudos de perda de função, nos quais a interrupção de um gene causa alterações fenotípicas, desempenham um papel fundamental na construção de nossa compreensão dos processos biológicos. O método CRISPR / Cas9 representa o avanço mais recente na tecnologia de engenharia do genoma e revolucionou a prática atual de genética em células e organismos. Cas9 é uma endonuclease guiada por ARN que se liga a uma sequência de DNA específica complementar ao gRNA e gera uma ruptura de cadeia dupla (DSB). Este DSB recruta máquinas de reparo de DNA que, na ausência de um modelo de DNA para recombinação homóloga, voltarão a ligar a cadeia de DNA cortada através de uma união final não homóloga propensa a erros, o que pode resultar em inserções ou deleções de nucleotídeo (s) Causando mutações de deslocamento de quadro 1 .
A grande facilidade e versatilidade do CRISPR / Cas9 apprOach tornou-a uma ferramenta altamente atraente para telas de nocaute de escala genômica destinadas a desvendar funções de genes desconhecidas 2 , 3 . No entanto, as abordagens knockout de um único gene são de uso limitado se existem vários paralogues com funções redundantes. Assim, a ablação de um único gene pode não ser suficiente para determinar a função desse gene, dada a possível compensação por paralogues, resultando em pouca ou nenhuma alteração fenotípica 4 . Portanto, é importante eliminar paralogues em paralelo, fornecendo múltiplos vetores de gRNA visando os diferentes genes paralogous, a fim de superar a influência da compensação genética.
Para ampliar o uso de CRISPR / Cas9 para o knockout de genes paralogous, desenvolvemos recentemente um método de clonagem rápido e de um passo para clonar até quatro gRNAs pré-recozidos em um único vetor retroviral 5 . A espinha dorsal, chamada CRISPR-concatemer, é baseadaEm um plasmídeo retroviral MSCV contendo cassetes de expressão repetitivas de gRNA. Cada cassete contém dois locais de reconhecimento invertidos da enzima de restrição Tipo IIS Bbs I, que podem ser substituídos de forma irreversível por um oligo de gRNA recozido com saliências correspondentes usando uma reação de baralhamento de Golden Gate em um único tubo 6 . Este método de clonagem consiste em ciclos repetitivos de digestão e ligadura que permitem a montagem simultânea de múltiplos fragmentos de DNA, explorando as diferentes sucessões geradas por Bbs I. A singularidade dessa enzima é, por exemplo, a capacidade de realizar cortes assimétricos fora do seu reconhecimento seqüência; Portanto, cada cassete pode ter uma seqüência diferente com saliências personalizadas que flanqueiam o site central Bbs I e, desta forma, cada gRNA pode ser clonado em uma posição específica e orientação do vetor concatemer.
Como prova de princípio, demonstramos o uso desta estratégia emOrganoides intestinais do rato, interrompendo simultaneamente dois pares de reguladores negativos paralogous da via Wnt por uma rodada de eletroporação 5 .
Nos últimos anos, muitos outros grupos desenvolveram estratégias semelhantes baseadas em vários vetores de expressão de gRNA construídos usando o Golden Gate shuffling 7 para alcançar o knockout multi-genes em vários sistemas modelo, como linhas celulares humanas 8 , 9 , peixe-zebra 10 e Escherichia coli 11 . Em seus protocolos, os gRNAs são primeiro clonados em vetores intermediários individuais e depois são reunidos em um produto final. Em contrapartida, a principal vantagem de nossa estratégia CRISPR-concatemer é a conveniência de um único Bbs I baralhar, etapa de clonagem. Como outros concatestores de gRNA, nosso método possibilita o nocaute simultâneo de até quatroDiferentes genes ou aumentou a eficiência de nocaute CRISPR após a segmentação de um ou dois genes com múltiplos gRNAs ( Figura 1 ).
Neste protocolo, descrevemos em detalhes cada passo na geração de vetores CRISPR-concatemer, do design gRNA para a reação Golden Gate e para a confirmação da clonagem bem-sucedida. Nós também fornecemos um protocolo altamente eficiente para a transfecção de CRISPR-concatemers em organoides de intestino delgado de ratos por eletroporação e subseqüentes experimentos de retirada de fatores de crescimento.
Protocolo
1. Projeto gRNA para o vetor CRISPR-concatemer
Nota: O objetivo desta seção é explicar como optar pela melhor estratégia de segmentação e como criar gRNAs contendo saliências específicas para o vetor CRISPR-concatemer.
- Desenhe gRNAs contra os genes de interesse usando uma ferramenta de design CRISPR gRNA de escolha. Veja a Tabela de Materiais para um exemplo.
NOTA: Ao direcionar um par de genes paralogous, embora seja possível projetar um gRNA por gene, é aconselhável projetar dois gRNAs por gene para aumentar as chances de alcançar um duplo nocaute ( Figura 1 ). - Certifique-se de que os gRNAs não contenham o site de reconhecimento Bbs I usando uma ferramenta de mapeamento de restrição (consulte a Tabela de Materiais para um exemplo).
- Adicione saliências de vetores específicos de CRISPR-concatemer a cada oligo, como mostrado na Tabela 1 .
Tabela 1: Saliências para cada cassete do vetor CRISPR-concatemer.
2. Clonagem de gRNAs no CRISPR-concatemer Vector
- Fosforilação e recozimento de oligos
NOTA: Esta etapa ilustra como tO recozimento de fios de topo e inferior para cada oligo de gRNA e como fosforilar as extremidades em uma única reação.- Prepare a mistura de reação para oligos de fosforilação e recozimento de costas e costas em gelo, de acordo com as instruções abaixo.
NOTA: Todos os oligos podem ser agrupados em uma reação; Por exemplo, no caso de um vetor 4 gARNA-concatemer, agrupam 8 oligos. - Para 3 concatemers, use 3,0 μL de cadeia superior de gRNA (1,0 μL de cada gARN, 10 μM, 1 μL / gRNA), 3,0 μL de cadeia inferior de gRNA (1,0 μL de cada gARN, 10 μM, 1 μL / gRNA), 2,0 μL de T4 Tampão de ligase de ADN (10x), 1,0 μL de T4 PNK e adicione H 2 O até um volume total de 20,0 μL.
- Misture bem por pipetagem e execute isso em um termociclador usando as seguintes configurações: 37 ° C durante 30 min, 95 ° C durante 5 min, rampa até 25 ° C a 0,3 ° C / min, segure a 4 ° C.
- Prepare a mistura de reação para oligos de fosforilação e recozimento de costas e costas em gelo, de acordo com as instruções abaixo.
- Bbs Reagendo a reação
NOTA: Nesta seção, os oligos de gRNA pré-recozidos são incorporados na posição apropriada do vetor concatemer em um passo alternando ciclos de digestão e ligadura.- Diluir a mistura reaccional 1: 100 na água DNase / RNase-livre para gerar 3 e 4 vectores gARN-concatemer.
NOTA: Ao clonar 2 gRNA-concatemers, este passo não é necessário. - Montar a reação Bbs I shuffling no gelo, conforme as instruções abaixo. Inclua um controle negativo que contém apenas o vetor.
- Use 100 ng CRISPR-vetor concatemer, 10,0 μL de mistura de oligo, 1,0 μL de tampão de enzima de restrição contendo BSA (10x), 1,0 μL de DTT (10 mM), 1,0 μL de ATP (10 mM), 1,0 μL de Bbs I, 1,0 μL de T7 de ligase E H 2 O até um volume total de 20,0 μL.
- Misture bem por pipetagem e execute isso em um termociclador usando as seguintes configurações: Execute 50 ciclos para clonagem 3 e 4 gRNA-concatemers e 25 ciclos para 2 gARN-concatemers, ambos a 37 ° C durante 5 min, 21 ° C durante 5 min, mantenha a 37 ° C durante 15 min, depois 4 ° C para sempre.
- Diluir a mistura reaccional 1: 100 na água DNase / RNase-livre para gerar 3 e 4 vectores gARN-concatemer.
- Tratamento de exonuclease
NOTA: Esta etapa é altamente recomendada, pois aumenta a eficiência da clonagem, removendo qualquer vestígio de DNA linearizado.- Trate a reação Bbs I shuffling com uma exonuclease de DNA (veja Tabela de Materiais ) da seguinte maneira.
- Tome 11,0 μL de mistura de ligação do passo anterior (2.2.3), adicione 1,5 μL de tampão de exonuclease (10x), 1,5 μL de ATP (10 mM), 1,0 μL de exonuclease de DNA e aumente o volume total para 15,0 μL com água. Incubar a 37 ° C durante 30 min seguido por 70 ° C durante 30 min.
NOTA: Esta etapa remove qualquer DNA residual linearizado na mistura e, portanto, aumenta a eficiência de clonagem. - Use 2 μL da mistura de reação para transformação em quimicamente compeBactéria E. E. coli por choque térmico 12 .
NOTA: Alternativamente, a reação pode ser armazenada por até uma semana a -20 ° C.
- Digestão de restrição
NOTA: O objetivo deste passo é avaliar por digestão de restrição o sucesso do procedimento de clonagem.- Para confirmar a presença de inserções de gRNA no vetor CRISPR-concatemer, coloque as colônias bacterianas de 4 a 8 com um ciclo de inoculação, cresça cada clone em 4 mL de meio LB durante a noite a 37 ° C em um agitador orbital. Extraia o DNA usando um kit miniprep de plasmídeo de acordo com as instruções do fabricante (veja a Tabela de Materiais ).
- Digest ~ 200 ng de DNA com 10 U EcoR I + 5 U Bgl II em uma reação de 10 μL incubando-o a 37 ° C durante 3 h em uma incubadora de bactérias. Inclua uma mistura de reação separada com o vetor original correspondente como um controle positivo para comparação de tamanho.
NOTA: ThiS confirmará se todos os concatemers estão presentes, uma vez que estas duas enzimas de restrição irão consumir concatemers completos. Estes são os tamanhos esperados para cada concatemer: 2 gRNA-concatemer (800 pb), 3 gRNA-concatemer (1,2 kbp) e 4 gRNA-concatemer (1,6 kbp). - Executar reações de digestão em um gel de agarose a 1% a 90 V durante aproximadamente 20 min.
- Visualize o gel usando um transiluminador UV. Identifique os clones com o tamanho de inserção correto, assegurando que seu padrão de banda corresponda ao vetor original e que cada fragmento tenha o tamanho esperado usando uma escada de DNA.
- Digite os clones selecionados com 5 U Bbs I a 37 ° C durante 3 h em uma incubadora de bactérias. Inclua uma mistura de reação separada com o vetor original correspondente como controle.
NOTA: Este passo de digestão adicional é para confirmar que os gRNAs foram clonados na posição correta e, consequentemente, todos os sites de reconhecimento Bbs I foram perdidos. - Executar reações de digestão sobreUm gel de agarose a 1% a 90 V durante aproximadamente 20 min. Considere como correto apenas os vetores que não são cortados pelo Bbs I, pois eles apenas contêm gRNAs.
- Seqüenciamento vetorial
NOTA: O objetivo deste passo é confirmar a presença de sequências de gRNA nesses vetores identificados como corretos por análise de digestão de restrição.- Confirme os vetores de concatemer positivos pelo seqüenciamento de Sanger 13 usando os seguintes primers:
Avançar: TCAAGCCCTTTGTACACCCTAAG (para verificar a primeira cassete gRNA)
Linker1_Forward: GACTACAAGGACGACGATGACAA (para verificar a segunda cassete gRNA)
Linker2_Reverse: GGCGTAGTCGGGCACGTCGTAGGGGT (para verificar a segunda cassete gRNA)
Linker2_Forward: ACCCCTACGACGTGCCCGACTACGCC (para verificar a terceira cassete gRNA)
Linker3_Reverse: TCCTCCTCTGAGATCAGCTTCTGCAT (para verificar a terceira cassete gRNA)
Reverse: AGGTGGCGCGAAGGGGCCACCAAAG (para verificar o último gRNAcassete) - Verifique a presença de todos os gRNA pesquisando suas seqüências nas leituras de seqüências.
- Confirme os vetores de concatemer positivos pelo seqüenciamento de Sanger 13 usando os seguintes primers:
3. Transfecção de Organoides Intestinais por Electroporação
NOTA: Observe que este procedimento é baseado no protocolo publicado por Fujii et al . Em 2015, com adaptação para culturas de organo intestino intestinal do rato 14 .
- Pré-eletroporação
NOTA: Esta seção descreve como preparar os organoides intestinais do mouse antes da eletroporação removendo todos os antibióticos e meios condicionados do meio de cultura deles. Isso evitará possíveis efeitos tóxicos durante a eletroporação.- No dia 0 do procedimento de transfecção, separe os organoídios numa proporção de 1: 2.
NOTA: As culturas intestinais intestinais podem ser obtidas através do isolamento da cripta de acordo com protocolos previamente estabelecidos 15 . Por favor, consulte Tabela 2 para todas as composições de mídia.- Ao dividir organoides para eletroporação, semeie um mínimo de 6 poços de uma placa de 48 poços por transfecção.
- Semeje os organoídeos em 20 μL de gotas da matriz do porão e cresça-os em meio WENR + Nic (Wnt + EGF + Noggin + R-spondin + Nicotinamida) a 37 ° C, 5% de CO 2 em uma incubadora humidificada (como descrito anteriormente 15 ) .
- No dia 2, altere o meio substituindo WENR + Nic por 250 μL de EN (EGF + Noggin) + CHIR99021 (inibidor de Glycogen Synthase Kinase-3) + Y-27632 (inibidor de ROCK), sem antibióticos (ver Tabela 2 ).
NOTA: Em todas as etapas, a quantidade de meio adicionado a cada poço de uma placa de 48 poços é de 250 μL. - No dia 3, mude o meio organoide para EN + CHIR99021 + Y-27632 + 1,25% v / v Dimetilsulfóxido (DMSO), sem antibióticos.
- No dia 0 do procedimento de transfecção, separe os organoídios numa proporção de 1: 2.
- Preparação das células
NOTA:Aqui descrevemos como fragmentar organoids em grupos de células pequenas por dissociação mecânica e química. Essas etapas são fundamentais para o sucesso do procedimento.- No dia 4, interromper as cúpulas da matriz do porão usando uma ponta de pipeta de 1 mL e transferir organoides para um tubo de 1,5 mL. Conteúdo da piscina de quatro poços de uma placa de 48 poços em um tubo.
- Quebrar organicamente os organoids em pequenos fragmentos, pipetando para cima e para baixo com uma pipeta P200 aproximadamente 200 vezes. Centrifugar à temperatura ambiente, 5 min a 600 x g.
- Remova o meio e ressuspenda o grânulo em 1 mL de protease recombinante de grau de cultura celular (ver tabela de materiais). Incubar a 37 ° C durante um máximo de 5 min e verificar uma gota de amostra de 50 μL sob um microscópio de luz invertido com um objetivo de 4x.
NOTA: Clusters de 10 a 15 células são desejáveis, pois isso aumenta a sobrevivência celular após a eletroporação. - Transfira a suspensão celular para um tubo de 15 mL de baixa ligação eDissociação, adicionando 9 mL de meio basal sem antibióticos (ver Tabela 2 ). Centrifugação à temperatura ambiente, 5 min a 600 xg, depois descarte o sobrenadante e ressuspenda o sedimento em 1 mL de meio soro reduzido (ver Tabela de Materiais ).
- Contar o número de células com uma câmara de Bürker e usar um mínimo de 1 x 10 5 células por reação de eletroporação. Adicione 9 mL de meio de soro reduzido ao tubo de 15 mL e centrifugue à temperatura ambiente, 3 min a 400 x g.
- Electroporação
NOTA: As seções a seguir fornecem instruções sobre como realizar a eletroporação e fazer recuperação de organoides posteriormente.- Remova todo o sobrenadante e ressuspenda o sedimento em uma solução de eletroporação (ver Tabela de Materiais ). Adicione uma quantidade total de 10 μg de DNA à suspensão celular e adicione a solução de eletroporação a um volume final de 100 μL e mantenha a célula DNUma mistura em gelo. Use vetores CRISPR-concatamer em combinação com um plasmídeo de expressão Cas9 ( por exemplo, Addgene # 41815) em uma proporção de 1: 1.
NOTA: O volume total do DNA adicionado deve ser inferior ou igual a 10% do volume de reação total. - Incluir uma mistura de transfecção separada contendo um plasmídeo GFP para avaliar a eficiência de transfecção ( por exemplo, pCMV-GFP, Addgene # 11153 ou qualquer plasmídeo que expressa GFP genérico).
- Adicione a mistura célula-DNA à cuvete de eletroporação e coloque-a na câmara do eletroporador. Mude a impedância pressionando o botão apropriado no eletroporador e assegure-se de que seja 0.030-0.055 Ω. Execute a eletroporação de acordo com as configurações mostradas na Tabela 3 .
NOTA: Se o valor da impedância for inferior ao intervalo permitido, ajuste o volume da solução na cuvete. - Adicione 400 μL de tampão de eletroporação + Y-27632 à cuvete e, em seguida, transfira tudo para um tubo de 1,5 mL. IncubaÀ temperatura ambiente durante 30 min para permitir que as células se recuperem e depois giram a temperatura ambiente durante 3 min a 400 x g.
- Remova o sobrenadante e ressuspenda o sedimento em 20 μL / poço da matriz do porão. Semente aproximadamente 1 x 10 4 a 1 x 10 5 células por poço em uma placa de 48 poços e adicione o meio DMSO EN + CHIR99021 + Y-27632 + 1,25% v / v. Incubar a 37 ° C.
- No dia 5, altere o meio para EN + CHIR99021 + Y-27632 e verifique a eficiência de transfecção observando a expressão GFP ( Figura 2 ). Mantenha os organoides a 37 ° C e atualize o meio EN + CHIR99021 + Y-27632 após 2 dias.
- No dia 9, altere o meio para WENR + Nic + Y-27632 e incube a 37 ° C.
NOTA: Y-27632 pode ser removido após 7-10 dias (no dia 16-19).
- Remova todo o sobrenadante e ressuspenda o sedimento em uma solução de eletroporação (ver Tabela de Materiais ). Adicione uma quantidade total de 10 μg de DNA à suspensão celular e adicione a solução de eletroporação a um volume final de 100 μL e mantenha a célula DNUma mistura em gelo. Use vetores CRISPR-concatamer em combinação com um plasmídeo de expressão Cas9 ( por exemplo, Addgene # 41815) em uma proporção de 1: 1.
| Pulso de Poring | Pulso de transferência | |
| Voltagem | 175V | 20V |
| Comprimento do pulso | 5 ms | 50msec |
| Intervalo de pulso | 50msec | 50msec |
| Número de pulsos | 2 | 5 |
| Taxa de decaimento | 10% | 40% |
| Polaridade | + | +/- |
Tabela 3: Configurações de eletroporação.
4. Retirada do fator de crescimento
Nota: Aqui é exemplificado como conduzir um experimento de retirada do fator de crescimento ao eliminar os reguladores negativos da via Wnt em organoides intestinais.
- 10-14 dias depoisR na eletroporação, separe os organoides em uma proporção de 1: 3 em uma placa de 48 poços seguindo as etapas acima mencionadas (3.2.1 - 3.2.2).
- Ressuspender o sedimento de organoid em 20 μL de matriz de membrana basal e deixar solidificar a 37 ° C durante 10 min. Em seguida, sobreponha 250 μL de meio privado de fatores de crescimento ( por exemplo, EN) para testar se o knockout dos genes alvo foi alcançado 5 .
- Divida os organoides sob condições de fatores de crescimento para um mínimo de 2 - 3 passagens para ver uma diferença na sobrevivência entre organoids de controle de tipo selvagem selvagem (WT) e organoids mutantes 5 , 15 .
NOTA: Os organoídeos Wildtype não devem poder sobreviver em meio privado de fator de crescimento em duas passagens, enquanto as linhas mutantes devem poder crescer.
Resultados
Para confirmar a presença do número correto de inserções de gRNA no vetor concatemer, a digestão de restrição é realizada com enzimas ( EcoR I + Bgl II) flanqueando todas as cassetes expressoras de gRNA (cada tamanho da cassete é ~ 400 pb, Figura 1 ). Por exemplo, ao gerar um vector de 4 gARN-concatemer, o tamanho esperado da banda inferior no gel de agarose é de aproximadamente 1,6 Kbp; Qualquer banda menor do que isso indica que nem todas as 4 cassetes gRNA estão inseridas no vetor ( Figura 2A ). Além disso, é sempre recomendável verificar se todos os sites de reconhecimento Bbs I estão perdidos e a enzima não corta o vetor ( Figura 2B ).
Uma vez que as construções foram confirmadas, elas podem ser entregues aos organoides intestinais do mouse por eletroporação para alcançar a óptimaL níveis de eficiência de transfecção (até 70%), como mostrado pelo controle GFP ( Figura 3 ).
Finalmente, para testar funcionalmente a eficiência desta estratégia, os organoides intestinais transfectados com Cas9 e os vetores concatemer contra Axin1 / 2 e Rnf43 / Znrf3 foram cultivados em EN (retirada de R-spondin) e EN + IWP2 (retirada de R-spondin e Wnt, IWP2 : Inibidor de porco-espinho, 2,5 μM) para um mínimo de 3 passagens ( Figura 4 ). Enquanto os organoids WT não transfundidos morreram sob ambas as condições, os organoídeos knockout Axin1 / 2 sobreviveram em ambos devido à ativação a jusante da via Wnt; Além disso, os organoídios mutantes Rnf43 / Znrf3 sobrevivem na ausência de R-spondin, mas não podem sobreviver na presença de IWP2, o que provoca o esgotamento do Wnt que ativa o caminho. Tomados em conjunto, essas observações demonstram que o nocaute desses pares de paralogues é possível gerando tEle esperava o fenótipo de organoide. Os detalhes desses resultados foram publicados em Developmental Biology 5 .
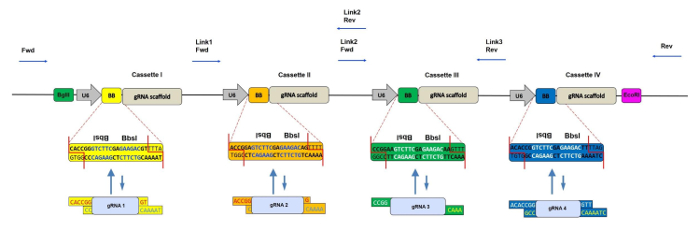
Figura 1: Representação esquemática do CRISPR-concatemer com 4 Cassettes. Esquema do vetor 4 gARN-concatemer com cada cassete de 400 pb contendo um promotor U6, dois locais Bbs I repetidos invertidos (também indicados como BB) e andaimes gRNA nesta ordem. Durante a reação de baralhar, os sites Bbs I são substituídos por fragmentos de gRNA com saliências correspondentes e, conseqüentemente, perdidos. Os locais de ligação dos iniciadores de seqüenciamento para verificar a inserção correta de oligos de gRNA são mostrados pelas setas azuis. Fwd = iniciador direto, Rev = primário reverso, Link 1/2/3 = regiões de ligação 1/2/3. Por favor cLambe aqui para ver uma versão maior dessa figura.

Figura 2: Padrões de digestão representativos de vetores Concatemer. ( A ) Digestão dupla de 3 e 4 vectores de gARN-concatemer com EcoR I e Bgl II. O padrão de digestão correto é marcado por um tiqueiro verde, enquanto vetores com apenas 1 ou 2 inserções de gRNA são marcados por uma cruz vermelha. A pista 1 mostra a digestão de um vetor parental 4 gRNA-concatemer usado como controle positivo (marcado por "+"); Da mesma forma, a pista 5 mostra a digestão de um vetor parental de 3 gARN-concatemer, marcado por "+". ( B ) Digestão com Bbs I, mostrando o tamanho correto de vetores de concatemer não digeridos (indicados pelos carrapatos verdes). A digestão de um vetor concatemer contendo gRNA que perdeu sites Bbs I é usado como um controle positivo eD é marcado por "+". Clique aqui para ver uma versão maior dessa figura.
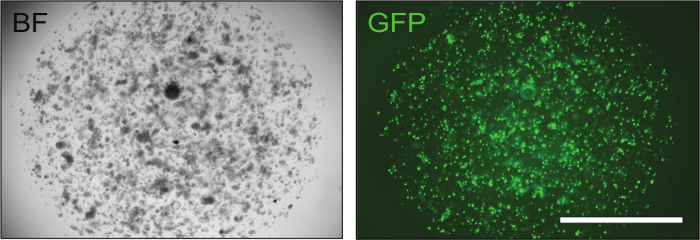
Figura 3: Imagem representativa de Organoids Intestinal Exitosamente Electroporados. A transfecção de um plasmídeo GFP é fundamental para avaliar a eficiência da transfecção. Aproximadamente 24 h após a eletroporação, os organoídeos contendo um pequeno número de células já estão visíveis e, se o procedimento de eletroporação tiver sido bem sucedido, até 70% deles apresenta fluorescência verde. BF = campo brilhante, GFP = proteína verde fluorescente. Barra de escala = 2,000 μm. Clique aqui para ver uma versão maior dessa figura.
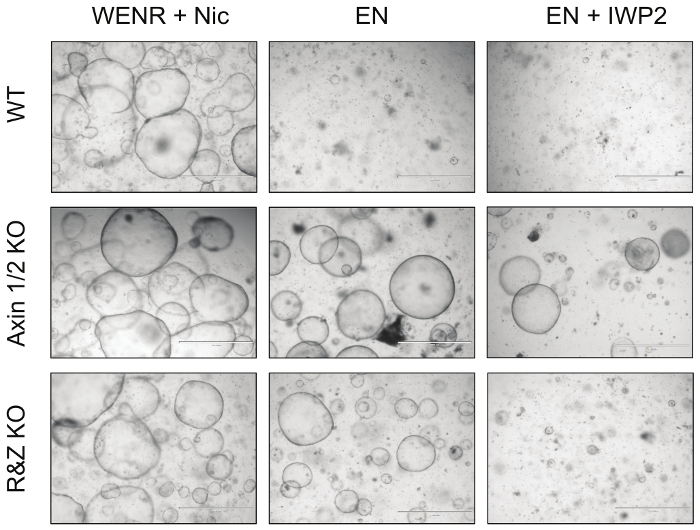
Figura 4: Imagens representativas de Organoids Intestinal Mutantes. Knockout dos reguladores negativos da via Wnt Axin1 e Rnf43, juntamente com seus paralogues, torna os organoídeos intestinais resistentes à privação de fatores de crescimento. Em particular, os organoids knockout Axin1 / 2 (Axin1 / 2 KO) podem crescer na ausência de R-spondin (EN: EGF + Noggin) e Wnt (EN + IWP2: EN + Inibidor de porco-espinho), enquanto os organoids mutantes Rnf43 / Znrf3 (R & Z KO) só pode sobreviver na ausência de R-spondin (EN). Em contraste, os organoícidos WT só podem sobreviver na condição de cultura de controle, WENR + Nic (Wnt + EGF + Noggin + R-spondin + Nicotinamida). Barras de escala = 1000 μm. Clique aqui para ver uma versão maior dessa figura.
| Meio basal | Comentários | |
| Armazenar a 4 ° C por 4 semanas | ||
| Meio de cultura celular | 500 mL | Veja tabela de materiais |
| L-Glutamina 100x | 5 mL | |
| Agente de buffer 1 M | 5 mL | Veja tabela de materiais |
| Penicilina Streptomicina 100x | 5 mL | |
| WENR + Nic (Wnt + EGF + Noggin + R-spondin + Nicotinamida) | ||
| Armazenar a 4 ° C por 2 semanas | ||
| Meio basal | Até 50 mL | |
| Suplemento de células sem neurônio celular sem soro (50x) | 1 mL | Veja a tabela de maTermal |
| Suplemento de células sem neurônio sem soro (100x) | 500 μL | Veja tabela de materiais |
| N-acetilcisteína (500 mM) | 125 μL | |
| EGF de ratinho (100 μg / mL) | 25 μL | |
| Rato Noggin (100 μg / mL) | 50 μL | |
| Meio condicionado R-Spondin | 5 mL | |
| Meio condicionado Wnt3a | 25 mL | |
| Nicotinamida (1 M) | 250 μL | |
| EN + CHIR + Y-27632 (EGF + Noggin + CHIR + Y-27632) | ||
| Armazenar a 4 ° C por 2 semanas | ||
| Meio basal sem penicilina Streptomicina | Até 20 mL | |
| Suplemento de células sem neurônio celular sem soro (50x) | 400 μL | Veja tabela de materiais |
| Suplemento de células sem neurônio sem soro (100x) | 200 μL | Veja tabela de materiais |
| N-acetilcisteína (500 mM) | 50 μL | |
| EGF de ratinho (100 μg / mL) | 10 μL | |
| Rato Noggin (100 μg / mL) | 20 μL | |
| Y-27632 (10 uM) | 20 μL | |
| CHIR99021 (8 μM) | 10 μL | |
| EN (EGF + Noggin) | ||
| Armazenar a 4 ° C por 4 semanas | ||
| Meio basal | Até 50 mL | |
| Suplemento de células sem neurônio celular sem soro (50x) | 1 mL | Ver tabela de materiais |
| Suplemento de células sem neurônio sem soro (100x) | 500 μL | Ver tabela de materiais |
| N-acetilcisteína (500 mM) | 125 μL | |
| EGF de ratinho (100 μg / mL) | 25 μL | |
| Rato Noggin (100 μg / mL) | 50 μL |
Tabela 2: Composição da mídia orgânica.
Discussão
Neste protocolo, detalhamos todas as etapas necessárias para gerar CRISPR-concatemers e para aplicar CRISPR-concatemers em organoids intestinais do mouse, a fim de eliminar simultâneamente vários genes. Como observado anteriormente, esta estratégia tem várias vantagens, como sua velocidade, alta eficiência e custo-efetividade.
Para executar com sucesso todo o procedimento, há alguns aspectos críticos a serem considerados. Em primeiro lugar, é essencial que todos os oligos de gRNA estejam adequadamente recozidos e fosforilados, pois representam o material de partida para a reação de clonagem Bbs I que em si é muito eficiente. Em segundo lugar, quando os organoides eletroportivos, quanto mais células são usadas por condição, maior a eficiência de transfecção máxima possível. Além disso, também é importante que, após a dissociação celular, predominam clusters de células pequenas em células isoladas.
No entanto, é possível encontrar problemas técnicosQuando tenta a clonagem ou a transfecção pela primeira vez; No caso de problemas durante a clonagem de gRNA, recomenda-se verificar a sequência de oligo de gRNA e, se correto, selecionar colônias bacterianas adicionais para seleção de digestão de restrição. Se a eficiência de transfecção e a viabilidade celular forem baixas após a eletroporação, é aconselhável repetir o protocolo usando mais células por condição e reduzindo o tempo de dissociação celular para 3 min.
Embora a geração de CRISPR-concatemers seja relativamente barata e fácil, a realização de telas genéticas de escala maior em organoides não é, pois a escala é limitada pelos custos associados à cultura organoidal e pela natureza intensiva em mão-de-obra. Vale ressaltar, neste caso, que o método CRISPR-concatemer também é compatível com linhas celulares, como HEK293 e células-tronco embrionárias de mouse.
Independentemente do sistema celular, outra desvantagem potencial desta sTrategy pode ser encontrado ao visar o nocaute simultâneo de três ou quatro genes diferentes. Por exemplo, cada gRNA terá uma eficiência de segmentação diferente e as mudanças de bater em todos os genes ao mesmo tempo podem ser relativamente baixas; Por esse motivo, é aconselhável empregar o sistema concatemer para direcionar mais de um gRNA contra o mesmo gene.
Estratégias alternativas baseadas de maneira semelhante em Golden Gate shuffling foram propostas ao longo dos anos para gerar vectores multiplex gRNA 7 , 8 . No entanto, no nosso método, é possível montar diretamente múltiplos gRNAs em um único vetor retroviral em uma única rodada de clonagem, o que o torna adequado para gerar bibliotecas gRNA para segmentar paralogues.
Nosso CRISPR-concatemer é construído no backbone do vetor retroviral MSCV. Assim, o retrovírus que contém concato de gRNA pode ser usado para gerar linhas celulares estáveis que sobreexistemGRNAs de execução. Quando combinado com um sistema Casu-indutível, pode-se realizar knockouts paralogeis induzíveis usando nosso sistema.
Em resumo, aqui descrevemos como clonar até quatro gRNAs diferentes no mesmo vetor em um passo e como aplicar essa estratégia à cultura organoidal com alta eficiência de transfecção. Além disso, fornecemos sugestões úteis para maximizar as chances de sucesso ao longo de todo o procedimento.
Divulgações
Os autores não têm nada a divulgar. Os autores não têm nenhum conflito de interesse declarado.
Agradecimentos
Agradecemos Christopher Hindley pela leitura crítica do manuscrito. AM é suportado pelo Wntsapp (Marie Curie ITN), AA-R. É apoiado pelo Medical Research Council (MRC) e pelo BK.K. E RM são apoiados por uma bolsa Sir Henry Dale da Wellcome Trust e da Royal Society [101241 / Z / 13 / Z] e recebem apoio através de uma bolsa básica do Wellcome Trust e do MRC para o Wellcome Trust - MRC Cambridge Stem Cell Institute .
Materiais
| Name | Company | Catalog Number | Comments |
| Optimized CRISPR Design Tool | Feng Zhang group | CRISPR gRNA design tool; http://crispr.mit.edu/ | |
| Webcutter 2.0 | restriction mapping tool; http://rna.lundberg.gu.se/cutter2/ | ||
| T4 PNK (Polynucleotide Kinase) | New England Biolabs | M0201L | |
| T4 DNA ligase buffer | New England Biolabs | M0202S | |
| T7 DNA Ligase | New England Biolabs | M0318L | |
| DTT (dithiothreitol) | Promega | P1171 | |
| ATP (adenosine triphosphate) | New England Biolabs | P0756S | |
| FastDigest BbsI (BpiI) | Thermo Fisher | FD1014 | |
| Tango buffer (BSA-containing restriction enzyme buffer) | Thermo Fisher | BY5 | |
| BglII | New England Biolabs | R0144 | |
| EcoRI | New England Biolabs | R0101 | |
| Plasmid-safe exonuclease | Cambio | E3101K | |
| Thermal cycler | Applied biosystems | 4359659 | |
| 10G competent E. coli bacteria | Cambridge Bioscience | 60108-1 | |
| Plasmid mini kit | Qiagen | 12125 | |
| Table top microcentrifuge | Eppendorf | UY-02580-01 | |
| Inoculating loops | Microspec | PLS5 | |
| Bacteria incubator | Sanyo | MIR-262 | |
| Luria-Bertani broth (LB) | Sigma-Aldrich | L3522 | |
| Agarose | Sigma-Aldrich | A4718 | |
| Agarose gel electrophoresis apparatus | Bioneer | A-7020 | |
| Advanced DMEM/F12(cell culture medium) | Invitrogen | 12634-034 | |
| Glutamax (L-Glutamine) 100x | Invitrogen | 35050-068 | |
| HEPES 1 M (buffering agent) | Invitrogen | 15630-056 | |
| Penicillin-streptomycin 100x | Invitrogen | 15140-122 | |
| B27 supplement (Neuronal cell serum-free supplement) 50x | Invitrogen | 17504-044 | |
| N2 supplement (Neuronal cell serum-free supplement) 100x | Invitrogen | 17502-048 | |
| n-Acetylcysteine 500 mM | Sigma-Aldrich | A9165-5G | |
| Mouse EGF 500 µg/mL | Invitrogen Biosource | PMG8043 | |
| Mouse Noggin 100 µg/mL | Peprotech | 250-38 | |
| Nicotinamide 1 M | Sigma | N0636 | |
| R-Spondin conditioned medium | n.a. | n.a. | Produced in house from HEK293 cells, for details see Sato and Clevers 2013 |
| Wnt conditioned medium | n.a. | n.a. | Produced in house from HEK293 cells, for details see Sato and Clevers 2013 |
| Y-27632 10 µM | Sigma-Aldrich | Y0503-1MG | |
| Standard BD Matrigel matrix | BD Biosciences | 356231 | |
| 48-well Plate | Greiner Bio One | 677980 | |
| CHIR99021 | Sigma-Aldrich | A3734-1MG | |
| IWP-2 | Cell Guidance Systems | SM39-10 | |
| TrypLE (recombinant protease) | Invitrogen | 12605-010 | |
| Opti-MEM (reduced serum medium ) | Life technologies | 51985-034 | |
| Electroporation Cuvettes 2mm gap | NepaGene | EC-002S | |
| Low binding 15 mL tubes | Sigma-Aldrich | CLS430791 | |
| Bürker’s chamber | Sigma-Aldrich | BR719520-1EA | |
| NEPA21 Super Electroporator | NepaGene | contact supplier | |
| Protein LoBind tubes low binding | Thermo Fisher | 10708704 | |
| BTXpress electroporation buffer | Harvard Apparatus | 45-0805 | |
| DMSO (Dimethyl sulfoxide) | AppliChem | A3672 |
Referências
- Mali, P., et al. RNA-Guided Human Genome Engineering via Cas9. Science. 339 (6121), 823-826 (2013).
- Koike-Yusa, H., Li, Y., Tan, E. -. P., Velasco-Herrera, M. D. C., Yusa, K. Genome-wide recessive genetic screening in mammalian cells with a lentiviral CRISPR-guide RNA library. Nat Biotech. 32 (3), 267-273 (2014).
- Bassett, A. R., Kong, L., Liu, J. L. A Genome-Wide CRISPR Library for High-Throughput Genetic Screening in Drosophila Cells. J Genet Genomics. 42 (6), 301-309 (2015).
- Diss, G., Ascencio, D., DeLuna, A., Landry, C. R. Molecular mechanisms of paralogous compensation and the robustness of cellular networks. J Exp Zool B Mol Dev Evol. 322 (7), 488-499 (2014).
- Andersson-Rolf, A., Merenda, A., Mustata, R. C., Li, T., Dietmann, S., Koo, B. -. K. Simultaneous paralogue knockout using a CRISPR-concatemer in mouse small intestinal organoids. Dev Biol. 420 (2), 1-7 (2016).
- Ran, F. A., Hsu, P. P. D., Wright, J., Agarwala, V., Scott, D., Zhang, F. Genome engineering using the CRISPR-Cas9 system. Nat Protoc. 8 (11), 2281-2308 (2013).
- Engler, C., Gruetzner, R., Kandzia, R., Marillonnet, S. Golden gate shuffling: A one-pot DNA shuffling method based on type IIs restriction enzymes. PLoS ONE. 4 (5), (2009).
- Sakuma, T., Nishikawa, A., Kume, S., Chayama, K., Yamamoto, T. Multiplex genome engineering in human cells using all-in-one CRISPR/Cas9 vector system. Sci Rep. 4 (5), 5400 (2014).
- Kabadi, A. M., Ousterout, D. G., Hilton, I. B., Gersbach, C. A. Multiplex CRISPR/Cas9-based genome engineering from a single lentiviral vector. Nucleic Acids Res. 42 (19), 1-11 (2014).
- Yin, L., et al. Multiplex Conditional Mutagenesis Using Transgenic Expression of Cas9 and sgRNAs. Genetics. 200 (2), 431-441 (2015).
- Cress, B. F., Toparlak, O. D., et al. CRISPathBrick: Modular Combinatorial Assembly of Type II-A CRISPR Arrays for dCas9-Mediated Multiplex Transcriptional Repression in E. coli. ACS Synth Biol. 4 (9), 987-1000 (2015).
- Froger, A., Hall, J. E. Transformation of plasmid DNA into E. coli using the heat shock method. J Vis Exp. (6), e253 (2007).
- Zimmermann, J., Voss, H., Schwager, C., Stegemann, J., Ansorge, W. Automated Sanger dideoxy sequencing reaction protocol. FEBS Letters. 233 (2), 432-436 (1988).
- Fujii, M., Matano, M., Nanki, K., Sato, T. Efficient genetic engineering of human intestinal organoids using electroporation. Nat. Protoc. 10 (10), 1474-1485 (2015).
- Andersson-Rolf, A., Fink, J., Mustata, R. C., Koo, B. -. K. A video protocol of retroviral infection in primary intestinal organoid culture. J Vis Exp. (90), e51765 (2014).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados