Method Article
Un protocole pour l'élimination des gènes multiples dans les petits organoïdes intestinaux de souris à l'aide d'un concatériste CRISPR
Dans cet article
Résumé
Ce protocole décrit les étapes pour le clonage de plusieurs ARN à guide unique dans un vecteur de vecteur de guide RNA, qui est particulièrement utilisé dans la création de knockouts multi-gènes utilisant la technologie CRISPR / Cas9. La génération de double knockouts dans les organoïdes intestinaux est une application possible de cette méthode.
Résumé
La technologie CRISPR / Cas9 a considérablement amélioré la faisabilité et la rapidité des études de perte de fonction qui sont essentielles à la compréhension de la fonction génétique. Dans les eucaryotes supérieurs, les gènes paraloges peuvent masquer un phénotype potentiel en compensant la perte d'un gène, limitant ainsi l'information qui peut être obtenue à partir d'études génétiques s'appuyant sur des knockouts à un seul gène. Nous avons développé une nouvelle méthode de clonage rapide pour les concatéristes guides d'ARN (gRNA) afin de créer des knock-out multi-gènes après un seul tour de transfection dans les organoïdes intestinales de souris. Notre stratégie permet la concatérisation de jusqu'à quatre gRNAs individuels en un seul vecteur en effectuant une seule réaction de mélange de Golden Gate avec des oligos de gARN recuits et un vecteur rétroviral pré-conçu. Cela permet soit le knockout simultané de jusqu'à quatre gènes différents, soit une augmentation de l'efficacité du knock-out suite au ciblage d'un gène par plusieurs gRNA. Dans ce protocole, nous montrons en détailComment cloner efficacement plusieurs gRNAs dans le vecteur CRCIR-concatemer rétroviral et comment obtenir une électroporation hautement efficace dans les organoïdes intestinaux. À titre d'exemple, nous montrons que le knock-out simultané de deux paires de gènes codant pour les régulateurs négatifs de la voie de signalisation Wnt (Axin1 / 2 et Rnf43 / Znrf3) rend les organoïdes intestinaux résistants au retrait des facteurs de croissance clés.
Introduction
L'approche génétique inverse est une méthode largement utilisée pour étudier la fonction d'un gène. En particulier, les études de perte de fonction, dans lesquelles la perturbation d'un gène provoque des altérations phénotypiques, jouent un rôle clé dans la compréhension des processus biologiques. La méthode CRISPR / Cas9 représente l'avancement le plus récent de la technologie d'ingénierie du génome et a révolutionné la pratique actuelle de la génétique dans les cellules et les organismes. Cas9 est une endonucléase guidée par un ARN qui se lie à une séquence d'ADN spécifique complémentaire à l'ARNg et génère une rupture à double brin (DSB). Cette DSB recrute des machines de réparation de l'ADN qui, en l'absence d'un modèle d'ADN pour la recombinaison homologue, re-ligateront le fil d'ADN coupé via une jointure terminale non homologue propice aux erreurs, ce qui peut ainsi entraîner des insertions ou des deletions de nucléotide (s) Provoquant des mutations de décalage de cadre 1 .
La grande facilité et la polyvalence de l'accord CRISPR / Cas9Oach l'a fait un outil très attrayant pour les écrans knock-out à l'échelle du génome visant à démêler les fonctions génétiques inconnues 2 , 3 . Néanmoins, les approches de knock-out à un seul gène sont limitées si des paralogues multiples avec des fonctions redondantes existent. Ainsi, l'ablation d'un seul gène pourrait ne pas être suffisant pour déterminer la fonction de ce gène étant donné une compensation possible par des paralogues, ce qui entraîne peu ou pas d'altération phénotypique 4 . Il est donc important d'éliminer les parallèles en parallèle en fournissant de multiples vecteurs d'ARNg ciblant les différents gènes paraloges afin de surmonter l'influence de la compensation génétique.
Pour étendre l'utilisation de CRISPR / Cas9 à un knockout de gène paralogé, nous avons développé récemment une méthode de clonage rapide et en une étape pour cloner jusqu'à quatre gRNAs pré-recuits dans un seul vecteur rétroviral 5 . L'épine dorsale, nommée CRISPR-concatemer, est baséeSur un plasmide rétroviral MSCV contenant des cassettes d'expression de gARN répétitives. Chaque cassette contient deux sites de reconnaissance inversés de l'enzyme de restriction Type IIS Bbs I, qui peut être remplacée irréversiblement par un oligo à l'ARNp recuit avec des surplombs correspondants à l'aide d'une réaction de brassage Golden Gate dans un seul tube 6 . Cette méthode de clonage consiste en des cycles répétitifs de digestion et de ligature qui permettent l'assemblage simultané de multiples fragments d'ADN en exploitant les différentes séquences de surtension générées par Bbs I. L'unicité de cette enzyme est, par exemple, la capacité d'effectuer des coupures asymétriques en dehors de sa reconnaissance séquence; Par conséquent, chaque cassette peut avoir une séquence différente avec des surplombs personnalisés flanquant le site central de Bbs I et de cette manière, chaque gRNA peut être clone dans une position spécifique et une orientation du vecteur concatemer.
En tant que preuve de principe, nous avons démontré l'utilisation de cette stratégie dansOrganoïdes intestinaux de souris en perturbant simultanément deux paires de régulateurs négatifs paraloges de la voie de Wnt par un cycle d'électroporation 5 .
Au cours des dernières années, de nombreux autres groupes ont développé des stratégies similaires basées sur de multiples vecteurs d'expression d'ARNg construits en utilisant Golden Gate shuffling 7 pour réaliser des gènes multi-gènes dans différents systèmes modèles, tels que les lignées cellulaires humaines 8 , 9 , le poisson zèbre 10 et Escherichia coli 11 . Dans leurs protocoles, les gRNA sont d'abord clones dans des vecteurs intermédiaires individuels puis assemblés ensemble en un produit final. En revanche, le principal avantage de notre stratégie CRISPR-concatemer est la commodité d'une seule étape de clonage Bbs I. Comme d'autres concatères gRNA, notre méthode rend possible soit le knock-out simultané jusqu'à quatreDifférents gènes ou une augmentation de l'efficacité du knock-out de CRISPR suite au ciblage d'un ou deux gènes avec des ARNm multiples ( figure 1 ).
Dans ce protocole, nous décrivons en détail toutes les étapes de la génération de vecteurs CRISPR-concatemer, du design gRNA à la réaction Golden Gate et à la confirmation du clonage réussi. Nous fournissons également un protocole hautement efficace pour la transfection de CRISPR-concatemers dans des organoïdes intestinaux de souris par électroporation et des expériences ultérieures de retrait de facteur de croissance.
Protocole
1. gRNA Design pour le CRISPR-concatemer Vector
Remarque: L'objectif de cette section est d'expliquer comment opter pour la meilleure stratégie de ciblage et comment concevoir des gRNA contenant des surplombs spécifiques pour le vecteur CRISPR-concatemer.
- Concevez des gRNA contre les gènes d'intérêt en utilisant un outil de conception CRISPR gRNA de choix. Voir la table des matières pour un exemple.
NOTE: Lors du ciblage d'une paire de gènes parallèles, bien qu'il soit possible de concevoir un gRNA par gène, il est conseillé de concevoir deux gRNA par gène pour augmenter les chances d'obtenir un double knock-out ( figure 1 ). - Assurez-vous que les gRNA ne contiennent pas le site de reconnaissance Bbs I en utilisant un outil de cartographie de restriction (voir la table des matières pour un exemple).
- Ajouter des surtensions spécifiques du vecteur conciseur CRISPR à chaque oligo, comme indiqué dans le tableau 1 .
Tableau 1: Surplomb pour chaque cassette du vecteur CRISPR-concatemer.
2. Le clonage des gRNA dans le vecteur CRISPR-concatemer
- Phosphorylation et recuit d'oligos
NOTE: Cette étape illustre comment tO recuit et inférieur des brins pour chaque oligo gRNA et comment phosphoryler leurs extrémités dans une seule réaction.- Préparer le mélange réactionnel pour les oligos phosphorylants et recuire les brins supérieurs et inférieurs sur la glace, selon les instructions ci-dessous.
NOTE: Tous les oligos peuvent être regroupés en une seule réaction; Par exemple, dans le cas d'un vecteur 4 gARN-concatemer, rassemblent ensemble 8 oligos. - Pour 3 concatemers, utiliser un brin supérieur de gRNA de 3,0 μL (1,0 μL de chaque ARNg, 10 μM, 1 μl / gRNA), 3,0 μL de brin de gRNA (1,0 μL de chaque ARNg, 10 μM, 1 μL / ARNg), 2,0 μL de T4 Le tampon de ligase d'ADN (10x), 1,0 μL de T4 PNK, et ajoute H 2 O jusqu'à un volume total de 20,0 μL.
- Bien mélanger en pipetant et exécuter cela dans un thermocycleur en utilisant les réglages suivants: 37 ° C pendant 30 min, 95 ° C pendant 5 min, rampe jusqu'à 25 ° C à 0,3 ° C / min, maintenir à 4 ° C.
- Préparer le mélange réactionnel pour les oligos phosphorylants et recuire les brins supérieurs et inférieurs sur la glace, selon les instructions ci-dessous.
- Bbs I shuffling reaction
NOTE: Dans cette section, les oligos de gARN pré-recuits sont incorporés dans la position appropriée du vecteur concatemer en une étape en alternant les cycles de digestion et de ligature.- Diluer le mélange réactionnel 1: 100 dans de l'eau exempte de DNase / RNase pour générer 3 et 4 vecteurs de galk-concatemer gRNA.
NOTE: Lors du clonage de 2 gRNA-concatemers, cette étape n'est pas nécessaire. - Assemblez la réaction Bbs I shuffling sur la glace, selon les instructions ci-dessous. Inclure un contrôle négatif qui ne contient que le vecteur.
- Utiliser 100 ng de vecteur conciseur CRISPR, 10,0 μL de mélange d'oligo, 1,0 μL de tampon d'enzyme de restriction contenant de la BSA (10x), 1,0 μL de DTT (10 mM), 1,0 μL d'ATP (10 mM), 1,0 μL de Bbs I, 1,0 μL de T7 de ligase , Et H 2 O jusqu'à un volume total de 20,0 μL.
- Mélangez bien en pipettant et exécutez-le dans un thermocycleur en utilisant les paramètres suivants: exécutez 50 cycles pour le clonage 3 et 4 gRNA-concatemers et 25 cycles pour 2 concédérants d'ARNg, à la fois à 37 ° C pendant 5 min, 21 ° C pendant 5 min, maintenez à 37 ° C pendant 15 minutes, puis 4 ° C pour l'éternité.
- Diluer le mélange réactionnel 1: 100 dans de l'eau exempte de DNase / RNase pour générer 3 et 4 vecteurs de galk-concatemer gRNA.
- Traitement de l'exonucléase
NOTE: Cette étape est fortement recommandée car elle augmente l'efficacité du clonage en supprimant toute trace d'ADN linéarisé.- Traiter la réaction Bbs I shuffling avec une exonucléase d'ADN (voir le tableau des matériaux ) comme suit.
- Prendre 11,0 μL de mélange de ligature de l'étape précédente (2.2.3), ajouter 1.5 μL de tampon d'exonucléase (10x), 1.5 μL d'ATP (10 mM), 1,0 μL d'exonucléase d'ADN et augmenter le volume total à 15,0 μL avec de l'eau. Incuber à 37 ° C pendant 30 min suivi de 70 ° C pendant 30 min.
NOTE: Cette étape supprime tout ADN résiduel linéarisé dans le mélange et augmente ainsi l'efficacité du clonage. - Utiliser 2 μl du mélange réactionnel pour une transformation en compétition chimiqueBactéries de E. coli contre les tiques par choc thermique 12 .
NOTE: Alternativement, la réaction peut être conservée jusqu'à une semaine à -20 ° C.
- Digestion par restriction
NOTE: L'objectif de cette étape est d'évaluer par la digestion par restriction le succès de la procédure de clonage.- Pour confirmer la présence d'inserts de gRNA dans le vecteur CRISPR-concatemer, choisissez 4 à 8 colonies bactériennes avec une boucle d'inoculation, développez chaque clone dans 4 mL de milieu LB pendant une nuit à 37 ° C dans un agitateur orbital. Extraire l'ADN en utilisant un kit miniprep plasmidique selon les instructions du fabricant (voir la Table des matériaux ).
- Digest ~ 200 ng d'ADN avec 10 U EcoR I + 5 U Bgl II dans une réaction de 10 μl en incubant à 37 ° C pendant 3 h dans un incubateur de bactéries. Inclure un mélange de réaction séparé avec le vecteur original correspondant comme un contrôle positif pour la comparaison des tailles.
REMARQUE: ThiS confirmera si tous les concatemères sont présents, puisque ces deux enzymes de restriction accèdent aux concurrents entiers. Ce sont les tailles attendues pour chaque concatemer: 2 gRNA-concatemer (800 pb), 3 gRNA-concatemer (1,2 kbp) et 4 gRNA-concatemer (1,6 kbp). - Effectuer des réactions de digestion sur un gel d'agarose à 1% à 90 V pendant environ 20 min.
- Visualisez le gel en utilisant un transilluminateur UV. Identifiez les clones avec la taille d'insertion correcte en assurant que leur motif de bande correspond à l'un des vecteurs originaux et que chaque fragment ait la taille attendue en utilisant une échelle d'ADN.
- Décrivez les clones sélectionnés avec 5 U Bbs I à 37 ° C pendant 3 h dans un incubateur de bactéries. Inclure un mélange de réaction séparé avec le vecteur original correspondant comme un contrôle.
REMARQUE: Cette étape de digestion supplémentaire consiste à confirmer que les gRNA ont été clonés dans la bonne position et, par conséquent, tous les sites de reconnaissance de Bbs I ont été perdus. - Effectuer des réactions de digestion surUn gel d'agarose à 1% à 90 V pendant environ 20 min. Considérons comme correct uniquement les vecteurs qui ne sont pas coupés par Bbs I car ils ne contiennent que des gRNA.
- Séquence vectorielle
NOTE: Le but de cette étape est de confirmer la présence de séquences d'ARNg dans ces vecteurs identifiés comme corrects par une analyse de digestion de restriction.- Confirmez les vecteurs de concatemer positifs par Sanger séquencing 13 en utilisant les amorces suivantes:
En avant: TCAAGCCCTTTGTACACCCTAAG (pour vérifier la première cassette gRNA)
Linker1_Forward: GACTACAAGGACGACGATGACAA (pour vérifier la seconde cassette gRNA)
Linker2_Reverse: GGCGTAGTCGGGCACGTCGTAGGGGT (pour vérifier la seconde cassette gRNA)
Linker2_Forward: ACCCCTACGACGTGCCCGACTACGCC (pour vérifier la troisième cassette gRNA)
Linker3_Reverse: TCCTCCTCTGAGATCAGCTTCTGCAT (pour vérifier la troisième cassette gRNA)
Inverser: AGGTGGCGCGAAGGGGCCACCAAAG (pour vérifier le dernier gRNAcassette) - Vérifiez la présence de tous les gRNA en recherchant leurs séquences dans les lectures séquentielles.
- Confirmez les vecteurs de concatemer positifs par Sanger séquencing 13 en utilisant les amorces suivantes:
3. Transfection des organoïdes intestinaux par électroporation
REMARQUE: Veuillez noter que cette procédure est basée sur le protocole publié par Fujii et al . En 2015, avec adaptation pour les cultures organoïdes intestinales intestinales de souris 14 .
- Pré-électroporation
REMARQUE: Cette section décrit comment préparer les organoïdes intestinaux de la souris avant l'électroporation en éliminant tous les antibiotiques et les milieux conditionnés de leur milieu de culture. Cela empêchera d'éventuels effets toxiques lors de l'électroporation.- Au jour 0 de la procédure de transfection, diviser les organoïdes en un rapport de 1: 2.
NOTE: Les cultures organo-intestinales peuvent être obtenues en effectuant l'isolement des cryptes selon les protocoles établis précédemment 15 . Prière de se référer à Tableau 2 pour toutes les compositions médiatiques.- Lors de la division des organoïdes pour l'électroporation, semer au moins 6 puits d'une plaque de 48 puits par transfection.
- Graisser les organoïdes dans des gouttes de matrice de sous-sol de 20 μL et les cultiver dans du milieu WENR + Nic (Wnt + EGF + Noggin + R-spondin + Nicotinamide) à 37 ° C, 5% de CO 2 dans un incubateur humidifié (comme décrit précédemment 15 ) .
- Le jour 2, changez de support en remplaçant WENR + Nic par 250 μL de EN (EGF + Noggin) + CHIR99021 (inhibiteur de Glycogen Synthase Kinase-3) + Y-27632 (inhibiteur de ROCK), sans antibiotiques (voir tableau 2 ).
REMARQUE: Dans toutes les étapes, la quantité de milieu ajoutée à chaque puits d'une plaque de 48 puits est de 250 μL. - Le jour 3, changez le milieu organoïde en EN + CHIR99021 + Y-27632 + 1,25% v / v Diméthylsulfoxyde (DMSO), sans antibiotiques.
- Au jour 0 de la procédure de transfection, diviser les organoïdes en un rapport de 1: 2.
- Préparation des cellules
REMARQUE:Nous décrivons ici comment fragmenter les organoïdes en petits groupes de cellules par dissociation mécanique et chimique. Ces étapes sont essentielles à la réussite de la procédure.- Le jour 4, perturber les dômes de la matrice du sous-sol à l'aide d'une pointe de pipette de 1 ml et transférer les organoïdes dans un tube de 1,5 ml. Contenu de la piscine de quatre puits d'une plaque de 48 puits dans un tube.
- Briser mécaniquement les organoïdes en petits fragments en pipetant vers le haut et vers le bas avec une pipette P200 environ 200 fois. Centrifuge à température ambiante, 5 min à 600 x g.
- Retirer le milieu et remettre à nouveau le culot dans 1 ml d'une protéase recombinante de culture de culture cellulaire (voir tableau de matériaux). Incuber à 37 ° C pendant 5 min maximum, puis vérifier une goutte d'échantillon de 50 μL sous un microscope optique inversé avec un objectif 4x.
NOTE: Des grappes de 10 à 15 cellules sont souhaitables, car cela augmente la survie cellulaire après l'électroporation. - Transférer la suspension cellulaire dans un tube de 15 mL à faible liaison et arrêter leDissociation en ajoutant 9 mL de milieu basal sans antibiotiques (voir tableau 2 ). Centrifuger à température ambiante, 5 min à 600 xg, puis jeter le surnageant et remettre à nouveau le culot dans 1 mL de milieu sérique réduit (voir Tableau des matériaux ).
- Comptez le nombre de cellules avec une chambre de Bürker et utilisez un minimum de 1 x 10 5 cellules par réaction d'électroporation. Ajouter 9 ml de milieu sérique réduit au tube de 15 ml et centrifuger à température ambiante, 3 min à 400 x g.
- Electroporation
REMARQUE: Les sections suivantes fournissent des instructions sur la façon d'effectuer l'électroporation et de récupérer les organoïdes par la suite.- Retirer tout le surnageant et remettre en suspension la pastille dans une solution d'électroporation (voir la Table des matières ). Ajouter une quantité totale de 10 μg d'ADN à la suspension cellulaire et ajouter une solution d'électroporation à un volume final de 100 μL et conserver le DN-cellulaireUn mélange sur glace. Utilisez des vecteurs CRATPR-concatamer en combinaison avec un plasmide d'expression Cas9 ( par exemple, Addgene # 41815) dans un rapport 1: 1.
NOTE: Le volume total de l'ADN ajouté doit être inférieur ou égal à 10% du volume total de la réaction. - Inclure un mélange de transfection séparé contenant un plasmide GFP pour évaluer l'efficacité de transfection ( par exemple pCMV-GFP, Addgene # 11153 ou tout plasmide générique contenant une GFP).
- Ajouter le mélange cellule-ADN à la cuvette d'électroporation et placez-le dans la chambre de l'électroporteur. Mesurez l'impédance en appuyant sur le bouton approprié sur l'électroporteur et assurez-vous qu'il est de 0,030-0,055 Ω. Effectuer l'électroporation selon les paramètres indiqués dans le tableau 3 .
REMARQUE: si la valeur d'impédance tombe à l'extérieur de la plage autorisée, ajustez le volume de la solution dans la cuvette. - Ajouter 400 μl de tampon d'électroporation + Y-27632 à la cuvette et ensuite transférer tout dans un tube de 1,5 mL. IncubaÀ température ambiante pendant 30 minutes pour permettre aux cellules de récupérer et ensuite les faire tourner à température ambiante pendant 3 min à 400 x g.
- Retirer le surnageant et remettre en suspension la pastille dans 20 μL / puits de matrice de sous-sol. Graisser environ 1 x 10 4 à 1 x 10 5 cellules par puits dans une assiette de 48 puits et ajouter EN + CHIR99021 + Y-27632 + 1,25% v / v de milieu DMSO. Incuber à 37 ° C.
- Le jour 5, changez le support en EN + CHIR99021 + Y-27632 et vérifiez l'efficacité de la transfection en observant l'expression GFP ( Figure 2 ). Maintenir les organoïdes à 37 ° C et actualiser EN + CHIR99021 + Y-27632 moyen après 2 jours.
- Le jour 9, changer le milieu à WENR + Nic + Y-27632 et incuber à 37 ° C.
REMARQUE: Y-27632 peut être supprimé après 7-10 jours (le jour 16-19).
- Retirer tout le surnageant et remettre en suspension la pastille dans une solution d'électroporation (voir la Table des matières ). Ajouter une quantité totale de 10 μg d'ADN à la suspension cellulaire et ajouter une solution d'électroporation à un volume final de 100 μL et conserver le DN-cellulaireUn mélange sur glace. Utilisez des vecteurs CRATPR-concatamer en combinaison avec un plasmide d'expression Cas9 ( par exemple, Addgene # 41815) dans un rapport 1: 1.
| Pouls de portage | Impulsion de transfert | |
| Tension | 175V | 20V |
| Longueur d'impulsion | 5 ms | 50 ms |
| Intervalle d'impulsion | 50 ms | 50 ms |
| Nombre d'impulsions | 2 | 5 |
| Taux de décroissance | dix% | 40% |
| Polarité | + | +/- |
Tableau 3: Paramètres de l'électroporation.
4. Retrait du facteur de croissance
Note: Ici, il est illustré comment mener une expérience de retrait du facteur de croissance lors de la suppression des régulateurs négatifs de la voie Wnt dans les organoïdes intestinaux.
- 10-14 jours aprèsR l'électroporation, diviser les organoïdes en un rapport 1: 3 dans une plaque de 48 puits suivant les étapes précitées (3.2.1 - 3.2.2).
- Remettre en suspension la pastille organoïde dans 20 μL de matrice de membrane basale et la laisser solidifier à 37 ° C pendant 10 min. Ensuite, recouvrez 250 μL de milieu dépourvu de facteur de croissance ( p. Ex. EN) pour vérifier si le knock-out des gènes cibles a été atteint 5 .
- Diviser les organoïdes dans des conditions privées de facteur de croissance pour un minimum de 2 à 3 passages pour voir une différence de survie entre les organoïdes témoins de type sauvage (WT) et les organoïdes mutants 5 , 15 .
REMARQUE: Les organoïdes Wildtype ne devraient pas pouvoir survivre dans un milieu dépourvu de facteur de croissance sur deux passages, alors que les chaînes mutantes devraient pouvoir se développer.
Résultats
Afin de confirmer la présence du nombre correct d'inserts de gRNA dans le vecteur concatemer, la digestion par restriction est effectuée avec des enzymes ( EcoR I + Bgl II) flanquant toutes les cassettes d'expression de gRNA (chaque taille de cassette est de ~ 400 pb, Figure 1 ). Par exemple, lors de la génération d'un vecteur concatérisme 4 gRNA, la taille attendue de la bande inférieure dans le gel d'agarose est d'environ 1,6 Kbp; Toute bande inférieure à celle-ci indique que toutes les 4 cassettes gRNA ne sont pas toutes insérées dans le vecteur ( Figure 2A ). En outre, il est toujours recommandé de vérifier que tous les sites de reconnaissance de Bbs I sont perdus et que l'enzyme ne coupe pas le vecteur ( Figure 2B ).
Une fois que les constructions ont été confirmées, elles peuvent être livrées à des organoïdes intestinaux de souris par électroporation pour atteindre l'optimaL niveaux d'efficacité de transfection (jusqu'à 70%), comme le montre le contrôle GFP ( Figure 3 ).
Enfin, pour tester efficacement l'efficacité de cette stratégie, les organoïdes intestinaux transfectés avec Cas9 et les vecteurs concaisers contre Axin1 / 2 et Rnf43 / Znrf3 ont été cultivés en EN (retrait de R-spondin) et EN + IWP2 (R-spondin et Wnt retrait, IWP2 : Inhibiteur de porc-épic, 2,5 μM) pour un minimum de 3 passages ( figure 4 ). Alors que les organoïdes WT non transférables sont morts dans les deux conditions, les organoïdes knockout Axin1 / 2 ont survécu dans les deux en raison de l'activation en aval de la voie Wnt; En outre, les organoïdes mutants Rnf43 / Znrf3 survivent en l'absence de R-spondin mais ne peuvent pas survivre en présence d'IWP2, ce qui provoque l'épuisement du Wnt qui active la voie. Ensemble, ces observations démontrent que le knock-out de ces paires de paralogues est possible en générant tIl s'attendait au phénotype organoïde. Les détails de ces résultats ont été publiés dans Developmental Biology 5 .
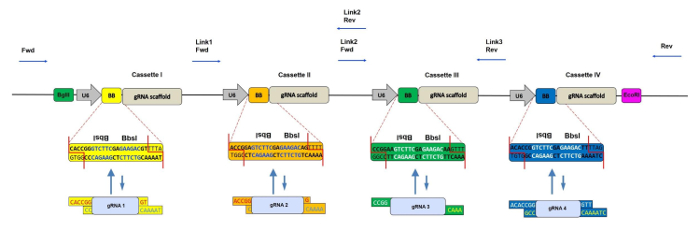
Figure 1: Représentation schématique du CRISPR-concatemer avec 4 Cassettes. Schéma du vecteur 4 gARN-concatemer avec chaque cassette de 400 pb contenant un promoteur U6, deux sites Bbs I répétés inversés (également désignés sous le nom BB) et échafaudage gRNA dans cet ordre. Au cours de la réaction de brassage, les sites Bbs I sont remplacés par des fragments d'ARNg avec des surplombs correspondants et par conséquent perdus. Les flèches bleues montrent les sites de liaison des amorces de séquençage pour vérifier l'insertion correcte des oligos de gRNA. Fwd = primaire avant, Rev = amorce inverse, Lien 1/2/3 = régions de liaison 1/2/3. S'il te plait CLèche ici pour voir une version plus grande de ce chiffre.

Figure 2: Patterns de digestion représentatifs des vecteurs Concatemer. ( A ) Double digestion de 3 et 4 vecteurs gARN-concatemer avec EcoR I et Bgl II. Le motif de digestion correct est marqué par une tache verte, alors que les vecteurs avec seulement 1 ou 2 insertions d'ARNg sont marqués par une croix rouge. La voie 1 montre la digestion d'un vecteur parent de 4 gRNA-concatemer utilisé comme témoin positif (marqué par "+"); De même, la voie 5 montre la digestion d'un vecteur parent de 3 gARN-concatemer marqué par "+". ( B ) Digestion avec Bbs I, montrant la taille correcte des vecteurs de concatemer non-digérés (indiqué par les tiques vertes). La digestion d'un vecteur concatérol contenant un gRNA qui a perdu des sites Bbs I est utilisée comme témoin positif etD est marqué par "+". Cliquez ici pour voir une version plus grande de ce chiffre.
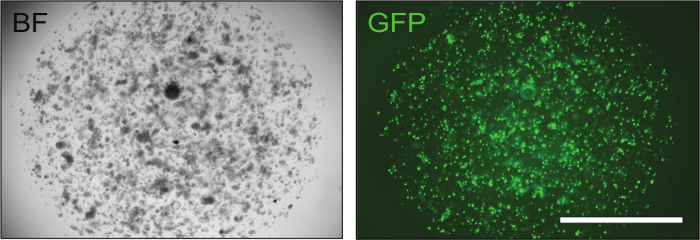
Figure 3: image représentative des organoïdes intestinaux électroporés avec succès. La transfection d'un plasmide GFP est essentielle pour évaluer l'efficacité de la transfection. Environ 24 heures après l'électroporation, des organoïdes contenant un petit nombre de cellules sont déjà visibles et, si la procédure d'électroporation a réussi, jusqu'à 70% d'entre elles présentent une fluorescence verte. BF = champ lumineux, GFP = protéine fluorescente verte. Barre d'échelle = 2 000 μm. Cliquez ici pour voir une version plus grande de ce chiffre.
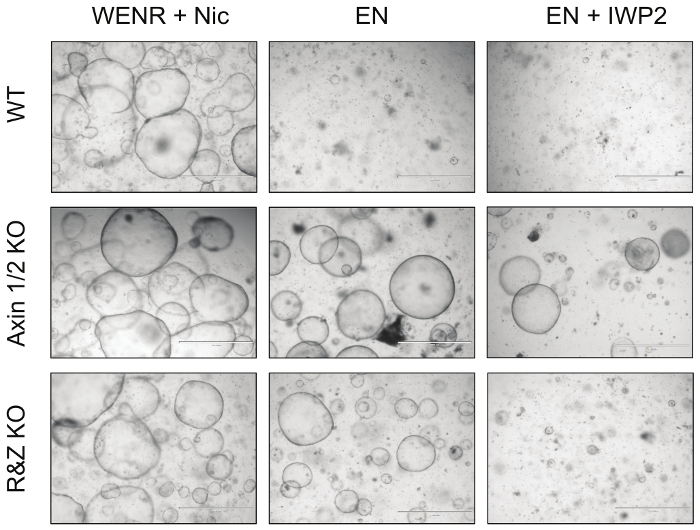
Figure 4: Images représentatives des organismes organiques mutants. Le knock-out des régulateurs négatifs de la voie Wnt Axin1 et Rnf43, ainsi que leurs paralogues, rend les organoïdes intestinaux résistants à la privation de facteur de croissance. En particulier, les organoïdes Knockout Axin1 / 2 (Axin1 / 2 KO) peuvent croître en l'absence de R-spondin (EN: EGF + Noggin) et Wnt (EN + IWP2: EN + inhibiteur de porc-épic), alors que les organoïdes mutants Rnf43 / Znrf3 (R & Z KO) ne peut survivre qu'en l'absence de R-spondin (EN). En revanche, les organoïdes WT ne peuvent survivre que dans l'état de culture témoin, WENR + Nic (Wnt + EGF + Noggin + R-spondin + Nicotinamide). Barres d'échelle = 1 000 μm. Cliquez ici pour voir une version plus grande de ce chiffre.
| Moyen basal | commentaires | |
| Conserver à 4 ° C pendant 4 semaines | ||
| Moyen de culture cellulaire | 500 mL | Voir le tableau des matériaux |
| L-Glutamine 100x | 5 mL | |
| Agent tampon 1 M | 5 mL | Voir le tableau des matériaux |
| Penicilline Streptomycine 100x | 5 mL | |
| WENR + Nic (Wnt + EGF + Noggin + R-spondin + Nicotinamide) | ||
| Conserver à 4 ° C pendant 2 semaines | ||
| Moyen basal | Jusqu'à 50 mL | |
| Supplément sans sérum neuronal cellulaire (50x) | 1 mL | Voir le tableau de maTeriel |
| Supplément exempt de cellules neuronales (100x) | 500 μL | Voir le tableau des matériaux |
| N-acétylcystéine (500 mM) | 125 μL | |
| EGF de souris (100 μg / mL) | 25 μL | |
| Noggin de souris (100 μg / mL) | 50 μL | |
| R-Spondin conditionné | 5 mL | |
| Wnt3a conditionné | 25 mL | |
| Nicotinamide (1 M) | 250 μL | |
| EN + CHIR + Y-27632 (EGF + Noggin + CHIR + Y-27632) | ||
| Conserver à 4 ° C pendant 2 semaines | ||
| Moyenne basale sans pénicilline Streptomycine | Jusqu'à 20 mL | |
| Supplément sans sérum neuronal cellulaire (50x) | 400 μL | Voir le tableau des matériaux |
| Supplément exempt de cellules neuronales (100x) | 200 μL | Voir le tableau des matériaux |
| N-acétylcystéine (500 mM) | 50 μL | |
| EGF de souris (100 μg / mL) | 10 μL | |
| Noggin de souris (100 μg / mL) | 20 μL | |
| Y-27632 (10 μM) | 20 μL | |
| CHIR99021 (8 μM) | 10 μL | |
| EN (EGF + Noggin) | ||
| Conserver à 4 ° C pendant 4 semaines | ||
| Moyen basal | Jusqu'à 50 mL | |
| Supplément sans sérum neuronal cellulaire (50x) | 1 mL | Voir la table des matières |
| Supplément exempt de cellules neuronales (100x) | 500 μL | Voir la table des matières |
| N-acétylcystéine (500 mM) | 125 μL | |
| EGF de souris (100 μg / mL) | 25 μL | |
| Noggin de souris (100 μg / mL) | 50 μL |
Tableau 2: composition organoïde des médias.
Discussion
Dans ce protocole, nous détaillons toutes les étapes nécessaires pour générer CRISPR-concatemers et pour appliquer CRISPR-concatemers dans les organoïdes intestinaux de souris afin d'éliminer simultanément plusieurs gènes. Comme indiqué précédemment, cette stratégie présente plusieurs avantages, tels que sa vitesse, son efficacité élevée et sa rentabilité.
Afin d'effectuer avec succès toute la procédure, il y a quelques aspects critiques à considérer. Tout d'abord, il est essentiel que tous les oligos d'ARNp soient correctement recuits et phosphorylés, car ils représentent le matériau de départ pour la réaction de clonage Bbs I qui en soi est très efficace. Deuxièmement, lorsque les organoïdes électroporiques, plus les cellules sont utilisées par condition, plus l'efficacité de transfection maximale possible est élevée. En outre, il est également important qu'après la dissociation cellulaire, les grappes de petites cellules prédominent sur des cellules individuelles.
Néanmoins, il est possible de rencontrer des problèmes techniquesLorsque vous tentez le clonage ou la transfection pour la première fois; Dans le cas de problèmes lors du clonage de l'ARNg, il est recommandé de vérifier la séquence d'oligo-gRNA et, s'il y a lieu, sélectionner des colonies bactériennes supplémentaires pour le dépistage de la digestion par restriction. Si l'efficacité de la transfection et la viabilité cellulaire sont peu post-électroporation, il est conseillé de répéter le protocole en utilisant plus de cellules par état et en réduisant le temps de dissociation cellulaire à 3 min.
Bien que la génération de CRISPR-concatemers soit relativement peu coûteuse et facile, l'exécution d'écrans génétiques à plus grande échelle dans les organoïdes n'est pas, car l'échelle est limitée par les coûts associés à la culture organoïde et par sa nature à forte intensité de main-d'œuvre. Il convient de mentionner dans ce cas que la méthode CRISPR-concatemer est également compatible avec les lignées cellulaires, telles que le HEK293 et les cellules souches embryonnaires de souris.
Indépendamment du système cellulaire, un autre inconvénient potentiel de cette sTrategy peut être rencontré en visant le knock-out simultané de trois ou quatre gènes différents. Par exemple, chaque gRNA aura une efficacité de ciblage différente et les changements de toucher tous les gènes au même moment peuvent être relativement faibles; Pour cette raison, il est conseillé d'utiliser le système concatemer pour diriger plus d'un gRNA contre le même gène.
D'autres stratégies, basées sur le brassage de Golden Gate, ont été proposées au cours des années pour générer des vecteurs d'ADNg multiplex 7 , 8 . Cependant, dans notre méthode, il est possible d'assembler directement plusieurs ARNg dans un seul vecteur rétroviral dans un seul cycle de clonage, ce qui le rend approprié pour générer des bibliothèques de gRNA pour cibler des paralogues.
Notre CRISPR-concatemer est construit dans le squelette du vecteur retroviral MSCV. Ainsi, le retrovirus contenant du concatéron de gRNA peut être utilisé pour générer des lignées cellulaires stables qui s'explosentRessemble à des gRNA. Lorsqu'il est combiné avec un système Cas9 inducible, on peut effectuer des knock-out parallèles inductibles à l'aide de notre système.
En résumé, nous décrivons ici comment cloner jusqu'à quatre gRNAs différents dans le même vecteur en une étape et comment appliquer cette stratégie à une culture organoïde avec une efficacité de transfection élevée. En outre, nous fournissons des suggestions utiles pour maximiser les chances de succès tout au long de la procédure.
Déclarations de divulgation
Les auteurs n'ont rien à dévoiler. Les auteurs n'ont aucun conflit d'intérêts déclaré.
Remerciements
Nous remercions Christopher Hindley pour la lecture critique du manuscrit. AM est pris en charge par Wntsapp (Marie Curie ITN), AA-R. Est soutenu par le Medical Research Council (MRC), et BK.K. Et RM sont soutenus par une bourse Sir Henry Dale de Wellcome Trust et la Royal Society [101241 / Z / 13 / Z] et reçoivent un soutien grâce à une subvention de base de Wellcome Trust et MRC au Wellcome Trust - MRC Cambridge Stem Cell Institute .
matériels
| Name | Company | Catalog Number | Comments |
| Optimized CRISPR Design Tool | Feng Zhang group | CRISPR gRNA design tool; http://crispr.mit.edu/ | |
| Webcutter 2.0 | restriction mapping tool; http://rna.lundberg.gu.se/cutter2/ | ||
| T4 PNK (Polynucleotide Kinase) | New England Biolabs | M0201L | |
| T4 DNA ligase buffer | New England Biolabs | M0202S | |
| T7 DNA Ligase | New England Biolabs | M0318L | |
| DTT (dithiothreitol) | Promega | P1171 | |
| ATP (adenosine triphosphate) | New England Biolabs | P0756S | |
| FastDigest BbsI (BpiI) | Thermo Fisher | FD1014 | |
| Tango buffer (BSA-containing restriction enzyme buffer) | Thermo Fisher | BY5 | |
| BglII | New England Biolabs | R0144 | |
| EcoRI | New England Biolabs | R0101 | |
| Plasmid-safe exonuclease | Cambio | E3101K | |
| Thermal cycler | Applied biosystems | 4359659 | |
| 10G competent E. coli bacteria | Cambridge Bioscience | 60108-1 | |
| Plasmid mini kit | Qiagen | 12125 | |
| Table top microcentrifuge | Eppendorf | UY-02580-01 | |
| Inoculating loops | Microspec | PLS5 | |
| Bacteria incubator | Sanyo | MIR-262 | |
| Luria-Bertani broth (LB) | Sigma-Aldrich | L3522 | |
| Agarose | Sigma-Aldrich | A4718 | |
| Agarose gel electrophoresis apparatus | Bioneer | A-7020 | |
| Advanced DMEM/F12(cell culture medium) | Invitrogen | 12634-034 | |
| Glutamax (L-Glutamine) 100x | Invitrogen | 35050-068 | |
| HEPES 1 M (buffering agent) | Invitrogen | 15630-056 | |
| Penicillin-streptomycin 100x | Invitrogen | 15140-122 | |
| B27 supplement (Neuronal cell serum-free supplement) 50x | Invitrogen | 17504-044 | |
| N2 supplement (Neuronal cell serum-free supplement) 100x | Invitrogen | 17502-048 | |
| n-Acetylcysteine 500 mM | Sigma-Aldrich | A9165-5G | |
| Mouse EGF 500 µg/mL | Invitrogen Biosource | PMG8043 | |
| Mouse Noggin 100 µg/mL | Peprotech | 250-38 | |
| Nicotinamide 1 M | Sigma | N0636 | |
| R-Spondin conditioned medium | n.a. | n.a. | Produced in house from HEK293 cells, for details see Sato and Clevers 2013 |
| Wnt conditioned medium | n.a. | n.a. | Produced in house from HEK293 cells, for details see Sato and Clevers 2013 |
| Y-27632 10 µM | Sigma-Aldrich | Y0503-1MG | |
| Standard BD Matrigel matrix | BD Biosciences | 356231 | |
| 48-well Plate | Greiner Bio One | 677980 | |
| CHIR99021 | Sigma-Aldrich | A3734-1MG | |
| IWP-2 | Cell Guidance Systems | SM39-10 | |
| TrypLE (recombinant protease) | Invitrogen | 12605-010 | |
| Opti-MEM (reduced serum medium ) | Life technologies | 51985-034 | |
| Electroporation Cuvettes 2mm gap | NepaGene | EC-002S | |
| Low binding 15 mL tubes | Sigma-Aldrich | CLS430791 | |
| Bürker’s chamber | Sigma-Aldrich | BR719520-1EA | |
| NEPA21 Super Electroporator | NepaGene | contact supplier | |
| Protein LoBind tubes low binding | Thermo Fisher | 10708704 | |
| BTXpress electroporation buffer | Harvard Apparatus | 45-0805 | |
| DMSO (Dimethyl sulfoxide) | AppliChem | A3672 |
Références
- Mali, P., et al. RNA-Guided Human Genome Engineering via Cas9. Science. 339 (6121), 823-826 (2013).
- Koike-Yusa, H., Li, Y., Tan, E. -. P., Velasco-Herrera, M. D. C., Yusa, K. Genome-wide recessive genetic screening in mammalian cells with a lentiviral CRISPR-guide RNA library. Nat Biotech. 32 (3), 267-273 (2014).
- Bassett, A. R., Kong, L., Liu, J. L. A Genome-Wide CRISPR Library for High-Throughput Genetic Screening in Drosophila Cells. J Genet Genomics. 42 (6), 301-309 (2015).
- Diss, G., Ascencio, D., DeLuna, A., Landry, C. R. Molecular mechanisms of paralogous compensation and the robustness of cellular networks. J Exp Zool B Mol Dev Evol. 322 (7), 488-499 (2014).
- Andersson-Rolf, A., Merenda, A., Mustata, R. C., Li, T., Dietmann, S., Koo, B. -. K. Simultaneous paralogue knockout using a CRISPR-concatemer in mouse small intestinal organoids. Dev Biol. 420 (2), 1-7 (2016).
- Ran, F. A., Hsu, P. P. D., Wright, J., Agarwala, V., Scott, D., Zhang, F. Genome engineering using the CRISPR-Cas9 system. Nat Protoc. 8 (11), 2281-2308 (2013).
- Engler, C., Gruetzner, R., Kandzia, R., Marillonnet, S. Golden gate shuffling: A one-pot DNA shuffling method based on type IIs restriction enzymes. PLoS ONE. 4 (5), (2009).
- Sakuma, T., Nishikawa, A., Kume, S., Chayama, K., Yamamoto, T. Multiplex genome engineering in human cells using all-in-one CRISPR/Cas9 vector system. Sci Rep. 4 (5), 5400 (2014).
- Kabadi, A. M., Ousterout, D. G., Hilton, I. B., Gersbach, C. A. Multiplex CRISPR/Cas9-based genome engineering from a single lentiviral vector. Nucleic Acids Res. 42 (19), 1-11 (2014).
- Yin, L., et al. Multiplex Conditional Mutagenesis Using Transgenic Expression of Cas9 and sgRNAs. Genetics. 200 (2), 431-441 (2015).
- Cress, B. F., Toparlak, O. D., et al. CRISPathBrick: Modular Combinatorial Assembly of Type II-A CRISPR Arrays for dCas9-Mediated Multiplex Transcriptional Repression in E. coli. ACS Synth Biol. 4 (9), 987-1000 (2015).
- Froger, A., Hall, J. E. Transformation of plasmid DNA into E. coli using the heat shock method. J Vis Exp. (6), e253 (2007).
- Zimmermann, J., Voss, H., Schwager, C., Stegemann, J., Ansorge, W. Automated Sanger dideoxy sequencing reaction protocol. FEBS Letters. 233 (2), 432-436 (1988).
- Fujii, M., Matano, M., Nanki, K., Sato, T. Efficient genetic engineering of human intestinal organoids using electroporation. Nat. Protoc. 10 (10), 1474-1485 (2015).
- Andersson-Rolf, A., Fink, J., Mustata, R. C., Koo, B. -. K. A video protocol of retroviral infection in primary intestinal organoid culture. J Vis Exp. (90), e51765 (2014).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon