このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
微生物細胞の成長に不可欠なプロセスを遵守する細胞蛍光顕微鏡をライブします。
要約
細菌の本質的なプロセスの機能を理解することは困難です。ターゲット固有の染料を蛍光顕微鏡は、微生物細胞の成長そして細胞周期の進行に重要な洞察を提供できます。ここでは、アグロバクテリウムは不可欠なプロセスの特性評価のための生きているセルイメージ投射のための方法を強調するモデル菌として使用されます。
要約
DNA 複製と分離、タンパク質合成、細胞壁の生合成と細胞分裂などのコア細胞プロセスは、細菌の生存に不可欠なタンパク質の機能に依存します。ターゲット固有の染料のシリーズよりプローブがこれらのプロセスを理解するように使用できます。膜構造の観察、脂質マイクロ ドメインの可視化および膜鉱物の検出を有効脂溶性染料で染色します。蛍光 d-アミノ酸酸ペプチドグリカン生合成のサイトを調査する (FDAAs) の使用は、細胞壁や細胞成長パターンの作成の潜在的な欠陥を示すことができます。最後に、核酸の汚れは、DNA の複製または染色体の分離の可能な欠陥を指定できます。シアニン DNA は、細胞ラベルの汚れ、微速度顕微鏡観察細胞増殖時核様体の形態のリアルタイム観測を有効にするために適しています。セルの分類のためのプロトコルは、膜構造、細胞壁の生合成、または染色体の欠陥を識別するために蛋白質の枯渇変異に適用できます。さらに、微速度顕微鏡観察は重要な蛋白質が削除され、タンパク質の機能に追加の洞察力を提供することができる形態学的変化の監視に使用できます。たとえば、重要な細胞分裂蛋白質の枯渇結果を細線または分岐、細胞成長タンパク質の枯渇が短いまたは円形になる細胞を引き起こす可能性があります一方。ここでは、細胞の成長、ターゲット固有のラベリングおよび微速度顕微鏡観察のためのプロトコルは、細菌植物病原菌アグロバクテリウムを提供しています。一緒に、ターゲット固有の染料と微速度顕微鏡観察に不可欠なプロセスの特性を有効にA. 根頭がんしゅ病菌。最後に、他の細菌に不可欠なプロセスを調査する提供するプロトコルを容易に変更できます。
概要
細菌の細胞周期の進行には、膜と細胞壁の生合成、DNA 複製と分離、細胞分裂など多くのプロセスの調整が必要です。細菌の細胞生物学の複雑さを完全に理解するには、これらの重要なイベントを研究する必要があります。しかし、これはこれらの経路の主要なコンポーネントは、突然変異誘発時にセル実行可能性が侵害されたので以外の些細なタスクであります。ターゲット固有の染料と相まって落射蛍光顕微鏡は、野生型と変異細菌株のこれらの重要なプロセスを調査する強力なアプローチです。
ペプチドグリカン固有染料など蛍光抗生物質 (バンコマイシン フロリダ州、bocillin FL) 蛍光 d アミノの酸 (たとえば、7-hydroxycoumarin-3-カルボン酸-3-アミノ-d-アラニン、波田; 4-chloro-7-nitrobenzofurazan-3-amino-d-アラニン、灘。tetramethylrhodamine-3-アミノ-d-アラニン;多田)。グラム陽性菌のペプチドグリカン生合成のサイトを調査する抗生物質類縁体蛍光の致死濃度の使用はペプチドグリカン挿入パターン1,2を明らかにするための効果的な戦略をされています。 3,4。外膜は一般的に蛍光を使用できないように関門を提供します蛍光バンコマイシンのラベリングは、固定のグラム陰性細菌5の挿入パターン、ペプチドグリカンに洞察力を得るために使用されています、一方生細胞におけるペプチドグリカン生合成用プローブとしての抗生物質。対照的に、蛍光 d アミノの酸または双直交官能と d-アミノ酸の短いパルスは共有生活細菌細胞6、7の広い範囲で最近のペプチドグリカンの挿入の領域をラベルします。合成 d-アミノ酸と観察されているペプチドグリカン挿入のパターンは、点状、中隔 (大腸菌と枯草) 極と中隔 (アグロバクテリウムとリステリア菌)、中隔のみ (黄色ブドウ球菌)、および根尖 (放線菌 venezuelae)6,7。これらの観察は、細菌が細胞壁の生合成の多様なパターンを示すことと成長パターンを検査するためのプローブとして合成 d-アミノ酸の利用は多くの細菌で貴重な戦略を示します。
細菌の染色体にラベルを付ける染料は、デオキシリボ核酸 (DNA) 特定のマイナーな溝バインダー (4, 6-diamidino-2-phenylindole;DAPI) と親和性の高いシアニン色素 (緑とオレンジ; 材料のリストを参照してください)。生きているセルの DAPI 染色、細菌生存率9を示すために使用されます環境試料8から細菌の列挙に役立ちます DAPI 染色固定セルの。対照的に、シアニン色素膜非透過性の「死んだ」細胞の非実行可能なセル9を列挙する汚れとオレンジと緑が頻繁に記述されているようです。驚くことに、これらの試薬はセル成長の間に細菌の核様体の形態を調査する際、DAPI、オレンジと緑すべてあることを示した膜側の透過物、生きているセル10のラベル付けが可能。住んでいる大腸菌の細胞、細胞質から自動蛍光によるびまん性表示され DAPI は DNA の染色、紫外線 (UV) の DAPI 染色性細胞の反復暴露は核構造10を摂動します。染色の大腸菌や枯草オレンジと明らかにこの色素が膜側の透過物です、染色体分配10 や DNA の複製、細胞の増殖に影響を与えることがなく生きている細胞内の DNA に結合する長期的な蛍光を提供します.これらの観察は、DNA のシアニン色素を多くの細菌の細胞の成長中に核様体の形態を監視する使用ことができることをお勧めします。
Phospholipid-specific stryl N-(3-triethylammoniumpropyl)-4-(6-(4-(diethylamino) フェニルなど染料) hexatrienyl) ピリジニウム臭化 (4-64; 材料リストを参照) は陽イオン性化合物、負荷電に優先的に関連付けるリン脂質カルジオリピンおよびホスファチジルグリセロール11など。4 64 を使用して別の細菌の膜をラベルするときは、明確なパターンが観察されます。大腸菌4-64 極、外側壁に沿ってバンドの濃縮し、後半の pre-divisional の部門サイト細胞12。枯草菌、4-64 の分類、脂質スパイラル13の可視化が可能です。アグロバクテリウムで 4 64 外膜をラベルし、成長ポールが14,15をラベリングを欠いている、特徴的な「馬蹄形」パターンで観察する.これらの観察は、これらの細菌が細胞の非対称性に寄与する脂質ドメインの存在による異種の脂質分布を示すことを示します。びまん性ラベリングの存在など 4 64 ラベリング パターンの変化、鉱物または小胞、陥入、または膜収縮が有益であろう分布または脂質の生合成に影響を与える突然変異体を特徴付けること。
細胞を染色、を超えて、不可欠なプロセスに参加しているタンパク質の機能を決定することは必要です。削除必須遺伝子と表現型の結果を研究することが可能ではないために、重要なタンパク質の解析は技術的に挑戦的です。したがって、タンパク質を破壊する方法が浮上しています。たとえば、そのネイティブのプロモーターではなく誘導性プロモーターの制御下に必須な遺伝子を置くことができます。誘導性プロモーターなどの小さな分子に敏感であります。16イソプロピル β-d-1-thiogalactopyranoside (IPTG)17,18,19,20,21, アラビノース22、バニリン酸17,23, 亜鉛します。キシロース23、従ってターゲット遺伝子のトランスクリプションを停止するためにおよび興味の蛋白質がなくなると、誘導が削除されたとき。興味の本質的な蛋白質を破壊のための代替アプローチは、CRISPR 干渉25,26 標的遺伝子の転写を阻害する低分子と RNA の相互作用を用いる合成 riboswitches24を含んでいます。ブロックの転写標的遺伝子と化学肥料プロテアーゼによる分解の標的タンパク質をペプチド タグを使用する誘導タンパク分解27,28に。したがって、タンパク質枯渇中に時間の経過とともに細胞の顕微鏡像は、特性評価のための強力なアプローチ、枯渇系統は、細胞生存率を失う前に特性の短時間だけを提供します。確かに、生きている細菌の細胞の顕微鏡観察は、区画29分泌、細胞形状維持のメカニズムを含む基本的な生物学的プロセスへの洞察を得るために研究者を可能にしました。
A. 根頭がんしゅ病菌細菌植物病原体30は、自然遺伝エンジニア31,32。したがって、機構、病原性に関連する宿主-病原体相互作用33,34,35など分泌36、ホスト変換30,31, 37広く検討されています。A. 根頭がんしゅ病菌による病気を防ぐため、植物形質転換を高める戦略を設計するには、 A. 根頭がんしゅ病菌の生存に不可欠なプロセスをよりよく理解する必要があります。ターゲット固有の染料を使用し、最近A. 根頭がんしゅ病菌18タンパク質枯渇戦略の不可欠なプロセスを調査するための手段を提供します。
ここでは、 A. 根頭がんしゅ病菌の野生型、変異体、および蛋白質の枯渇系統の顕微解析のための詳しいプロトコルが提供されます。最初の 2 つのプロトコルはセルを準備し、ターゲット固有の染料でそれらにラベルを付ける方法を説明します。第 3 のプロトコルでは、細菌の細胞 (図 2, , 図 3図 4) をイメージングとアガロース パッド (図 1) を準備するためのステップバイ ステップの指示を提供します。これらのプロトコルは、別のメディア条件、成長率、酸素の要件、および細胞構造を考慮して追加適応を持つ他の細菌に適した可能性があります。
プロトコル
1. A. 根頭がんしゅ病菌系統の成長
-
A. 根頭がんしゅ病菌菌株を培養
- 滅菌木製スティックまたはピペットの先端を使用すると、必要なひずみの単一コロニー ATGN 成長媒体 (レシピの材料リスト参照) の 1 mL を接種します。
注: A. 根頭がんしゅ病菌の枯渇株、ATGN に 1 mM IPTG 重要な蛋白質の生合成を維持するために誘導因子としてします。 - 225 rpm で振とうしながら 28 ° c ATGN で一晩A. 根頭がんしゅ病菌系統を成長します。
- 600 セルの光学密度を測定 nm (外径600) 分光光度計を使用しています。外径600に細胞培養を希釈 = ~0.2、外径 φ600まで成長し続ける = ~0.6 に達する。枯渇の緊張のため外径 φ600まで誘導成長し続ける = ~0.6 に達する。
- 外径600 A. 根頭がんしゅ病菌の野生型と変異体の系統を使用してターゲット固有の染色や微速度顕微鏡観察 (セクション 2 および 3.1 参照) の ~0.6 を =。枯渇株をウォッシュ アウト インデューサ (セクション 1.2 を参照)。
- 滅菌木製スティックまたはピペットの先端を使用すると、必要なひずみの単一コロニー ATGN 成長媒体 (レシピの材料リスト参照) の 1 mL を接種します。
-
A. 根頭がんしゅ病菌タンパク質枯渇系統の特性評価用インデューサの除去
- 指数文化の 1 mL にペレット (外径600 = ~0.4-0.6) 室温でデスクトップ、遠心分離機で 5 分 7,000 × g で遠心分離によって。
- 洗浄し、上清を除去し新鮮なメディアの誘導なしでペレットを再懸濁し、(1.2.1) 上記のようにセルをペレットします。新鮮なメディアで 3 回の合計セルを洗浄してください。
- 新鮮なメディアで最終的なペレットを再懸濁します、外径600細胞を集中 = ターゲット特定の染料 (セクション 2 および図 4 bD) または (セクション 3.2 と図 4 a) 微速度顕微鏡観察と即時の汚損のための ~0.8.また、成長しているメディアの染色と細胞のイメージングの前に誘導なし細胞でに時間の必要な量のセルを使い果たす前。
2. ターゲット固有A. 根頭がんしゅ病菌の細胞の染色
-
蛍光の d-アミノ酸酸細胞壁のラベリング
注: FDAAs は、 A. 根頭がんしゅ病菌ペプチドグリカンに容易に組み込まれている、非毒性の蛍光の d-アミノ酸酸です。この単純なラベリング手順は生きているセル6のパターニング成長をプローブします。4 FDAAs の合成のためのプロトコルは、使用可能な38です。- 指数文化を 500 μ l 添加ペレット (外径600 = ~0.4-0.6) デスクトップ、遠心分離機で 5 分 7,000 × g で遠心分離によって。新鮮なメディアの 100 μ L の細胞ペレットを再懸濁します。
- 5 mM に FDAA 洗浄し細胞を集中して、暗闇の中で 2 分間インキュベートの 5 μ L を追加します。
注意: 制限 FDAA ライトへの露出。潜伏の時は、株の成長率によって異なります。通常、インキュベーション時間は 2 倍時間6の 5-10% をする必要があります。 - 遠心分離によって細胞をペレットし、3 回リン酸緩衝生理食塩水 (PBS) 細胞ペレットを洗浄します。
- 〜 50 μ L の PBS でペレットを再懸濁します。
注: 再懸濁用 PBS のボリュームは、ペレット サイズに基づいて異なる場合があります。 - 0.8-1 適用 agarose パッドと落射蛍光顕微鏡すぐに (を参照してくださいセクション 3.1 と 3.2) を使用してイメージするセルの μ L。
- 冷たい 70% エタノールとセルを固定することによってさらにラベル定款または、停止します。冷たい 70% エタノール 1 mL の細胞ペレットを再懸濁し、氷で 15 分間インキュベートします。
- 遠心分離によって細胞を収集し、後でイメージングのため PBS の小さなボリュームで再懸濁します。48 時間以内に 4 ° C およびイメージで細胞を懸濁液を格納します。
-
DNA 染色 DAPI やオレンジ色の汚れ
注: DNA を観察する構造、細胞は DAPI またはオレンジ (死んだ細胞染色) が付いています。オレンジは側の透過物、古典的な「死んだ細胞染色」として記述されているがあると生きている細胞の DNA10をイメージングを有効にする、細菌の細胞の成長には影響しません。A. 根頭がんしゅ病菌のオレンジ ラベル作品は生きている細胞でよく、微速度顕微鏡観察 (図 3) に適しています。対照的に、DAPI 染色性細胞イメージングに必要な紫外線の (紫外線) 光の露出は光毒性 (図 3)、したがって DAPI 染色即時イメージングまたは固定セルを汚すのための生きた細胞を染色に最も適しています。- DAPI は細胞の染色
- 指数文化の 1 mL にペレット (外径600 = ~0.4-0.6) デスクトップ、遠心分離機で 5 分 7,000 × g で遠心分離によって。
- 必要に応じて前の染色にセルを修正する 70% 冷たいエタノール 1 mL に再細胞ペレット懸を濁します。10-15 分 2.2.1.1 記載として遠心分離によって細胞収集のため氷の上を孵化させなさい。
- DAPI 原液 (1 mg/mL; 参照材料表) の 1 μ L を含む PBS の 1 mL の細胞ペレットを再懸濁します。ピペットで穏やかに混合し、暗闇の中で 5 分間インキュベートします。
- 5 分間 7,000 × g で遠心分離によって細胞をペレットし、過剰な DAPI を削除する PBS の 1 mL で再懸濁します。セルを洗浄してさらに 2 回、50 μ L の PBS またはメディアにペレットを再懸濁します。
- 0.8-1 適用 agarose パッドとすぐに落射蛍光顕微鏡を用いた画像のセルの μ L。3.1 と 3.2 のセクションを参照してください。
注: このプロトコルは染色体のダイナミクス (図 3) を観察する微速度顕微鏡観察に最適ではありません。オレンジ染色 (2.2.2 を参照) の代替として使用してください。
- 生きているセルのオレンジ色染色
- 指数文化の 1 mL にペレット (外径600 = ~0.4-0.6) デスクトップ、遠心分離機で 5 分 7,000 × g で遠心分離によって。1 mL のオレンジ原液 1 μ L を含む PBS で細胞ペレットを再懸濁します (マテリアル リストを参照してください)。ピペットで穏やかに混合し、暗闇の中で 5 分間インキュベートします。
- 遠心分離によって細胞をペレットし、過剰なオレンジを削除する PBS の 1 mL で再懸濁します。セルを洗浄してさらに 2 回、50 μ L の PBS でペレットを再懸濁します。
- 0.8-1 適用 agarose パッドとすぐに落射蛍光顕微鏡を用いた画像のセルの μ L。3.1 と 3.2 のセクションを参照してください。
注: このプロトコルは生きているセルでうまく動作し、染色体のダイナミクス (図 3) を観察する微速度顕微鏡観察に適しています。すぐに画像に便利ではない、冷たい 70% エタノール (セクション 2.2.1.2 参照) でセルを修正可能性があります。
- DAPI は細胞の染色
-
膜のラベリング
注: 親脂性の styryl の蛍光染料 4 64 使用されています広範囲の細菌の細胞膜を観察します。多くの細菌とは対照的 4 64 標識A. 根頭がんしゅ病菌の細胞は均一に、ラベル付けはしないが頻繁ではなく「馬蹄形」のパターン14,15を展示。驚くことに、古いポールのラベルは強烈なに対し、成長極はほぼ汚れを欠いているです。したがって、4 64 はセル成長パターンとA. 根頭がんしゅ病菌における膜構造物の可視化を実現。 にします。- 指数段階の細胞培養の 1 mL 中 8 μ g/mL の最終的な集中に FM 4-64 を追加し、暗闇の中で 5 分間室温でインキュベートします。
- 7,000 x g でのデスクトップ、遠心分離機で 5 分間遠心分離による標識細胞のペレットし、1 mL の PBS で細胞ペレットを再懸濁します。セルを洗浄して余分な染料を削除する 3 回の合計。
- PBS の小さいボリュームをすぐにイメージする agarose パッド上のスポットで細胞を再懸濁します。(セクション 3.1 と 3.2 参照)
注意: セルをすぐにでイメージして特徴的なラベリング パターンを観察する必要があります。
3. A. 根頭がんしゅ病菌の細胞のイメージング
-
アガロース パッド準備
注:図 1には、動画像 (パネル) と微速度顕微鏡観察のために準備する典型的な agarose パッドの模式図 (パネル B) が含まれています。必要に応じて通常 Agarose パッドで用意します。- 3.1.1 ガイドとカット、22 mm × 22 mm 角研究室のフィルム エッジの周りのメスを実行して (材料のリストを参照してください) として観察を使用してください。
- 2 ~ 5 を残して研究所フィルムの中心の正方形をカット mm 罫線 agarose パッド用ガスケットとして使用します。中央の切り欠きを破棄します。
- ガラス スライド (75 mm × 25 mm) (材料のリストを参照)、アンモニア、アルコール フリーのクリーナーできれいにフィルムのガスケットを配置熱ガラス (図 1 aの上部のイメージ) にフィルムが溶けて少しまでとします。検査フィルムを溶かす、70 ° c の熱ブロック セットの端を使用して、または炎の上を軽く溶かします。
- ~0.075 g アガロースを混合することによってアガロース溶液を準備 (材料のリストを参照) を 5 mL の小さなフラスコ内のメディア。熱ソリューション ミックス agarose を溶解するまで、ソリューションを定期的な旋回と電子レンジでは明らかです。55-70 ° C でアガロース溶液を維持し、48 時間以内複数 agarose パッドの建設のため使用します。
- ガスケットの中心に 1.2 〜 1.5% の agarose を含むピペット メディア。
注: メディアのボリュームは 50-60 μ L ですがガスケットのサイズによって異なります。冷たいスライドにメディアがあまりにも速く固まるアガロースを引き起こす可能性が追加すると、このようにスライドに保管されるべき暖かい表面。ヒート ブロックのエッジを設定する 70 ° C の作品も。水アガロースまたはリン酸緩衝生理食塩水 (PBS) などのバッファーに格納された agarose のソリューションは、継続的な細胞増殖が必要ではないアプリケーションのアガロースを含むメディアの代わりに使用できます。インデューサ枯渇系統をイメージングすることができます提示またはメディアには存在しません。インデューサの不在は枯渇の表現型を明らかにするのに対し、インデューサの存在をコントロールとして使用できます。 - 場所ガスケット上 coverslip agarose を均等に分散します。
- アガロース パッドや細胞懸濁液の表面に適用されたときのプールでしわだらけの表面になるので、回避がオーバードライ agarose パッドの ~ 2 分を固めるにクールなレベルの表面にスライドを配置します。
- アガロース パッド coverslip を慎重にスライドさせます。
注意: この手順を急いでしないでください。アガロース パッドは簡単に引き裂くことができるし、不均一な agarose パッド イメージングを困難にすることができます。 - 許可空気に agarose パッドに 1-2 分乾燥室温でパッドの表面の乾燥を待ちます。メスを使用すると、空気のポケット (中央の画像、図 1 a); を作成する agarose の小さなストリップを削除します。空気のポケットは、〜 2 × 7-10 mm とA. 根頭がんしゅ病菌細胞エアポケット (図 1) に近い最高の成長する傾向があります。
注: 固定セルのまたは成長の監視されていない条件下ではイメージング、空気のポケットの必要はありません。 - スポット 0.8 1 アガロース パッドと新しい coverslip カバー上のセルの μ L。Coverslip をアガロース タッチパッドの表面細胞に分散する agarose のパッドの上にそっと置きます。
- 溶かされた VALAP を使用してカバーガラスのエッジをシール (レシピの材料表を参照してください。図 1 a下の画像)。すべてのエッジや、カバーガラスのコーナーに沿ってシール可能性イメージ投射の間の細胞の漂流につながる agarose のパッドの乾燥します。注: coverslip のシーリングは、長期のタイムラプス イメージング用のみ必要です。
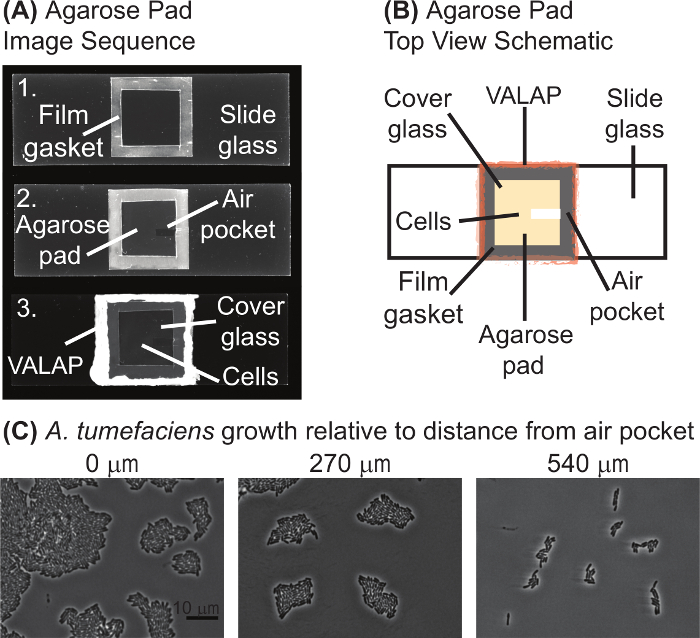
図 1: Agarose パッド準備します。(A) agarose パッドの準備のプロトコルのシーケンスをイメージします。画像 1 は、実験映画のガスケットでスライドです。画像 2 でアガロース パッドと空気のポケットを視覚化します。最後に、画像 3 は、coverglass の下のセルに完全な agarose パッドを示し、VALAP で密封します。微速度顕微鏡観察用アガロース パッドの (B) の回路図を提供しています。アガロース パッドの特長は、回路図にラベル付けされます。(C) 空気のポケットは、アガロース パッドにA. 根頭がんしゅ病菌の生育を推進しました。20 時間 agarose パッド上に成長した野生型A. 根頭がんしゅ病菌細胞の画像が表示されます。画像は、空気のポケットからはますます遠い位置で撮影されました。上の各画像のイメージの最も近い端から空気のポケットまでの距離が表示されます。スケール バー = 10 μ m.この図の拡大版を表示するのにはここをクリックしてください。
-
イメージング
注: 微分干渉コントラスト、位相コントラストや落射蛍光イメージング ハードウェア ベースのオート フォーカス、自動ステージ、標準フィルター、LED 光源、60 X オイルの液浸を搭載した倒立顕微鏡を実行です。位相コントラストまたは差動干渉の対照 (DIC) の目的 (1.4 NA) と 1 K 電子乗算電荷結合素子 (EMCCD) カメラです。顕微鏡は、恒温室内に置くべき。また、暖かいステージまたは商工会議所は、イメージング中に一貫した温度を維持するために使用できます。- 落射蛍光顕微鏡
注: 生細胞の蛍光イメージングのため必要があるイメージの買収の数を制限し、退色、光毒性を最小限に抑えるための露出時間を最適化するには各色素のイメージングは、十分な蛍光検出を提供していますが、退色または光毒性につながるしない最適な露出時間を見つけることをお勧めします。図 2-4、200 ms の露光時間はすべての蛍光画像に使用されていました。時間経過のシーケンスは、図 3に示すように、画像は 3 時間 10 分毎に買収されました。- 目的目標にイマージョン オイルを置き、スライド ホルダーに逆スライドをステージに配置します。フォーカシングのノブを使用すると、セルにフォーカスをもたらします。
- 段階 (または DIC) 画像と必要な蛍光フィルターを取得します。
注:図 2図 3、および図 4で使用される汚れの最大励起と放射の波長は次のとおり: 羽田 (405/460)、灘 (450/555)、多田 (555/570) 4-64 (515/640)、DAPI (360/460)、オレンジ (547/570)。
- 微速度顕微鏡観察
注: 微速度顕微鏡観察時に次の機能がコンピューター制御: x、y、および z の位置、シャッター、および蛍光フィルター。ハードウェア ベースのオート フォーカス システムは、タイムラプス イメージング中にフォーカスを維持するために最適です。また、ソフトウェアによるオート フォーカス ループを使用できます。- 目的目標にイマージョン オイルを置き、スライド ホルダーに逆スライドをステージに配置します。フォーカシングのノブを使用すると、セルにフォーカスをもたらします。
- 必要に応じて: 取得複数の (x, y) 位置。ランダムに agarose パッドの空気のポケット近くに細胞の 10 個のフィールドを選択します。
- 適切な時間間隔で位相または DIC のイメージに時間のシーケンス取得を設置しました。
注:図 4に示す時間経過のシーケンス、DIC 画像は 30 ms 露出 14 時間 10 分毎に買収されました。微速度顕微鏡観察における蛍光イメージングを使用している場合は、退色と光毒性を最小限に取得間隔と露出時間を調整します。
- 落射蛍光顕微鏡
結果
ターゲット固有の野生型のラベル付けA. 根頭がんしゅ病菌の細胞
その細胞の形態を説明するためには、洗浄の手順または (蛍光塗料を希釈するもの) 1 %dmso による治療によっては影響されません、セルでセルを洗浄後文化 (図 2 a、一番左のパネル) から直接イメージしました1.2 (図 2 a<...
ディスカッション
このプロトコルには、一連a. 根頭がんしゅ病菌野生型、変異体、および枯渇系統の調査のためのプロシージャにはが含まれています。それはすべてのプロトコル セクションに記載されている手順が成長媒体、温度、および成長率を考慮して追加の変更とその他の細菌の緊張のために容易に適応することができますは注目に値するです。
ターゲット固有の染料の使?...
開示事項
著者が明らかに何もありません。
謝辞
図 2と図 4で FDAAs の贈り物ありがとうマイケル ・ VanNieuwenhze (インディアナ大学)。この原稿の準備の間のフィードバックのためのブラウンの実験室のメンバーに感謝いたします。A. 根頭がんしゅ病菌の細胞増殖と分裂にブラウンの実験室の研究は、国立科学財団 (IOS1557806) によってサポートされます。
資料
| Name | Company | Catalog Number | Comments |
| Bacterial Strains | |||
| Agrobacterium tumefaciens C58 | ATCC | 33970 | Watson B, Currier TC, Gordon MP, Chilton MD, Nester EW. 1975. Plasmid required for virulence of Agrobacterium tumefaciens. J Bacteriol 123:255-264. |
| Agrobacterium tumefaciens C58ΔtetRA::mini-Tn7T-GM-Ptac-ctrA ΔctrA | Figueroa-Cuilan W, Daniel JJ, Howell M, Sulaiman A, Brown PJB. 2016. Mini-Tn7 insertion in an artificial attTn7 site enables depeltion of the essentail master regulator CtrA in the phytopathogen Agrobacterium tumefaciens. Appl Environ Microbiol. 82:5015-5025. | ||
| Name | Company | Catalog Number | Comments |
| Media Components | |||
| ATGN Minimal Medium | To 1 L of sterilized water add 50 ml 20X Buffer, 50 ml 20X Salts, 12.5 ml 40% glucose. For plates, add 15 g Bacto Agar to 1 L of water and autoclave. Cool to 55 °C and add 50 ml 20X Buffer, 50 ml 20X Salts, 12.5 ml 40% glucose. | ||
| 20X AT Buffer | Add 214 g/L KH2PO4 to water and adjust pH to 7.0 with sodium hydroxide. Autoclave. | ||
| NaOH | Fisher BioReagents | BP359 | |
| KH2PO4 | Fisher Chemical | P288 | |
| 20X AT Salts | Add 40 g/L (NH4)2SO4, 3.2 g/L MgSO4•7H2O, 0.2 g/L CaCl2•2H2O, and 0.024 g/L MnSO4•H2O to water. Autoclave. | ||
| (NH4)2SO4 | Fisher Chemical | A701 | |
| MgSO4•7H2O | Fisher BioReagents | BP213 | |
| CaCl2•2H2O | Fisher BioReagents | BP510 | |
| MnSO4•H2O | Fisher Chemical | M114 | |
| Glucose | Fisher Chemical | D16 | Prepare 40% stock in water. Filter sterilize. |
| Bacto Agar | Fisher BioReagents | BP1423 | Add 15 g to 1 L of water when preparing plates. |
| Name | Company | Catalog Number | Comments |
| Optional Media Additives | |||
| Kanamycin | GoldBio | K-120 | Prepare as a 100 mg/ml stock solution in water and filter sterilize. Use at final concentration of 200 µg/ml. |
| IPTG | GoldBio | I2481C5 | Prepare as a 1 M stock solution in water and filter sterilize. Use at final concentration of 1 mM as needed for induction. |
| Name | Company | Catalog Number | Comments |
| Microscopy Materials | |||
| Microscope Slides | Fisherbrand | 12-550D | 25 X 75 X 1.0 mm. Clean with Sparkle glass cleaner. |
| Microscope Cover Glass | Fisherbrand | 12-541-B | 22 X 22 mm. No. 1.5. Clean with Sparkle glass cleaner. |
| Sparkle Glass Cleaner | Home Depot | 203261385 | Ammonia and alcohol free. |
| Ultra Pure Agarose | Invitrogen | 16500-100 | Add to water, PBS, or media to a final concentration of 1 - 1.5%. Melt in microwave and place on 70 C |
| PBS | Fisher BioReagents | BP399500 | 10X solution to be diluted to 1X with sterile water. |
| Parafilm | Bemis | PM-999 | Laboratory film used as gasket in agarose pad preparation. |
| VALAP | Add equal weights of lanolin, parafin wax, and petroleum jelly to a conical tube. Heat tube in 70 °C dry, bead or water bath to melt and mix. Apply VALAP while still molten. | ||
| Lanolin Butter | SAAQIN | SQ-LAB-R1 | |
| Petroleum Jelly | Target Corp. | 06-17644 | |
| Paraffin Wax | Crafty Candles | 263012 | |
| Name | Company | Catalog Number | Comments |
| Target-specific dyes | |||
| DMSO | Fisher BioReagents | BP231-1 | Use to dilute stock solutions of dyes as needed. |
| FDAAs (NADA, HADA,TADA) | FDAAs can be synthesized or acquired through agreement with Mike VanNieuwenhze (Indiana University). Prepare 100 mM stock solution in DMSO. Use at a final concentration of 5 mM. | ||
| DAPI | ThermoFisher Scientific | 62247 | Prepare 1 mg/ml stock solution in DMSO. Use at final concentration of 1 µg/ml. |
| SYTOX Orange Nucleic Acid Stain | Invitrogen | S11368 | Stock concentration is 5 mM in DMSO. Use at final concentration of 5 µM. |
| FM4-64 | Invitrogen | T3166 | Prepare 8 mg/ml stock solution in DMSO. Use at final concentration of 8 µg/ml. |
| Name | Company | Catalog Number | Comments |
| Equipment | |||
| Dry bath | Sheldon Manufacturing, Inc. | 52120-200 | |
| Metallic thermal beads | Lab Armor | 42370-002 | |
| Epifluorescence microscope equipped with an EMCDD camera | Nikon Eclipse TiE equipped with a QImaging Rolera em-c2 1K electron-multiplying charge-coupled-device (EMCCD) camera is used in this work. |
参考文献
- Daniel, R. A., Errington, J. Control of cell morphogenesis in bacteria: two distinct ways to make a rod-shaped cell. Cell. 113 (6), 767-776 (2003).
- Tiyanont, K., et al. Imaging peptidoglycan biosynthesis in Bacillus subtilis with fluorescent antibiotics. Proc Natl Acad Sci U S A. 103 (29), 11033-11038 (2006).
- Turner, R. D., et al. Peptidoglycan architecture can specify division planes in Staphylococcus aureus. Nat Commun. 1, 26 (2010).
- Wheeler, R., Mesnage, S., Boneca, I. G., Hobbs, J. K., Foster, S. J. Super-resolution microscopy reveals cell wall dynamics and peptidoglycan architecture in ovococcal bacteria. Mol Microbiol. 82 (5), 1096-1109 (2011).
- Turner, R. D., Hurd, A. F., Cadby, A., Hobbs, J. K., Foster, S. J. Cell wall elongation mode in Gram-negative bacteria is determined by peptidoglycan architecture. Nat Commun. 4, 1496 (2013).
- Kuru, E., et al. In Situ probing of newly synthesized peptidoglycan in live bacteria with fluorescent D-amino acids. Angew Chem Int Ed Engl. 51 (50), 12519-12523 (2012).
- Siegrist, M. S., et al. (D)-Amino acid chemical reporters reveal peptidoglycan dynamics of an intracellular pathogen. ACS Chem Biol. 8 (3), 500-505 (2013).
- Kepner, R. L., Pratt, J. R. Use of fluorochromes for direct enumeration of total bacteria in environmental samples: past and present. Microbiol Rev. 58 (4), 603-615 (1994).
- Johnson, M. B., Criss, A. K. Fluorescence microscopy methods for determining the viability of bacteria in association with mammalian cells. J Vis Exp. (79), (2013).
- Bakshi, S., et al. Nonperturbative imaging of nucleoid morphology in live bacterial cells during an antimicrobial peptide attack. Appl Environ Microbiol. 80 (16), 4977-4986 (2014).
- Barak, I., Muchova, K. The role of lipid domains in bacterial cell processes. Int J Mol Sci. 14 (2), 4050-4065 (2013).
- Fishov, I., Woldringh, C. L. Visualization of membrane domains in Escherichia coli. Mol Microbiol. 32 (6), 1166-1172 (1999).
- Barak, I., Muchova, K., Wilkinson, A. J., O'Toole, P. J., Pavlendova, N. Lipid spirals in Bacillus subtilis and their role in cell division. Mol Microbiol. 68 (5), 1315-1327 (2008).
- Cameron, T. A., Anderson-Furgeson, J., Zupan, J. R., Zik, J. J., Zambryski, P. C. Peptidoglycan synthesis machinery in Agrobacterium tumefaciens during unipolar growth and cell division. MBio. 5 (3), e01219 (2014).
- Zupan, J. R., Cameron, T. A., Anderson-Furgeson, J., Zambryski, P. C. Dynamic FtsA and FtsZ localization and outer membrane alterations during polar growth and cell division in Agrobacterium tumefaciens. Proc Natl Acad Sci U S A. 110 (22), 9060-9065 (2013).
- Eberhardt, A., Wu, L. J., Errington, J., Vollmer, W., Veening, J. W. Cellular localization of choline-utilization proteins in Streptococcus pneumoniae using novel fluorescent reporter systems. Mol Microbiol. 74 (2), 395-408 (2009).
- Iniesta, A. A., Garcia-Heras, F., Abellon-Ruiz, J., Gallego-Garcia, A., Elias-Arnanz, M. Two systems for conditional gene expression in Myxococcus xanthus inducible by isopropyl-beta-D-thiogalactopyranoside or vanillate. J Bacteriol. 194 (21), 5875-5885 (2012).
- Figueroa-Cuilan, W., Daniel, J. J., Howell, M., Sulaiman, A., Brown, P. J. Mini-Tn7 Insertion in an Artificial attTn7 Site Enables Depletion of the Essential Master Regulator CtrA in the Phytopathogen Agrobacterium tumefaciens. Appl Environ Microbiol. 82 (16), 5015-5025 (2016).
- Jacob, F., Monod, J. Genetic regulatory mechanisms in the synthesis of proteins. J Mol Biol. 3, 318-356 (1961).
- Khan, S. R., Gaines, J., Roop, R. M., Farrand, S. K. Broad-host-range expression vectors with tightly regulated promoters and their use to examine the influence of TraR and TraM expression on Ti plasmid quorum sensing. Appl Environ Microbiol. 74 (16), 5053-5062 (2008).
- Yansura, D. G., Henner, D. J. Use of the Escherichia coli lac repressor and operator to control gene expression in Bacillus subtilis. Proc Natl Acad Sci U S A. 81 (2), 439-443 (1984).
- Guzman, L. M., Belin, D., Carson, M. J., Beckwith, J. Tight regulation, modulation, and high-level expression by vectors containing the arabinose PBAD promoter. J Bacteriol. 177 (14), 4121-4130 (1995).
- Thanbichler, M., Iniesta, A. A., Shapiro, L. A comprehensive set of plasmids for vanillate- and xylose-inducible gene expression in Caulobacter crescentus. Nucleic Acids Res. 35 (20), e137 (2007).
- Topp, S., et al. Synthetic riboswitches that induce gene expression in diverse bacterial species. Appl Environ Microbiol. 76 (23), 7881-7884 (2010).
- Peters, J. M., et al. A Comprehensive, CRISPR-based Functional Analysis of Essential Genes in Bacteria. Cell. 165 (6), 1493-1506 (2016).
- Qi, L. S., et al. Repurposing CRISPR as an RNA-guided platform for sequence-specific control of gene expression. Cell. 152 (5), 1173-1183 (2013).
- Griffith, K. L., Grossman, A. D. Inducible protein degradation in Bacillus subtilis using heterologous peptide tags and adaptor proteins to target substrates to the protease ClpXP. Mol Microbiol. 70 (4), 1012-1025 (2008).
- McGinness, K. E., Baker, T. A., Sauer, R. T. Engineering controllable protein degradation. Mol Cell. 22 (5), 701-707 (2006).
- Schneider, J. P., Basler, M. Shedding light on biology of bacterial cells. Philos Trans R Soc Lond B Biol Sci. 371 (1707), (2016).
- Escobar, M. A., Dandekar, A. M. Agrobacterium tumefaciens as an agent of disease. Trends Plant Sci. 8 (8), 380-386 (2003).
- Gelvin, S. B. Agrobacterium-mediated plant transformation: the biology behind the "gene-jockeying" tool. Microbiol Mol Biol Rev. 67 (1), 16-37 (2003).
- Nester, E. W. Agrobacterium: nature's genetic engineer. Front Plant Sci. 5, 730 (2014).
- Imam, J., Singh, P. K., Shukla, P. Plant Microbe Interactions in Post Genomic Era: Perspectives and Applications. Front Microbiol. 7, 1488 (2016).
- Subramoni, S., Nathoo, N., Klimov, E., Yuan, Z. C. Agrobacterium tumefaciens responses to plant-derived signaling molecules. Front Plant Sci. 5, 322 (2014).
- Yuan, Z. C., Williams, M. A really useful pathogen, Agrobacterium tumefaciens. Plant Cell. 24 (10), (2012).
- Alvarez-Martinez, C. E., Christie, P. J. Biological diversity of prokaryotic type IV secretion systems. Microbiol Mol Biol Rev. 73 (4), 775-808 (2009).
- Pitzschke, A. Agrobacterium infection and plant defense-transformation success hangs by a thread. Front Plant Sci. 4, 519 (2013).
- Kuru, E., Tekkam, S., Hall, E., Brun, Y. V., Van Nieuwenhze, M. S. Synthesis of fluorescent D-amino acids and their use for probing peptidoglycan synthesis and bacterial growth in situ. Nat Protoc. 10 (1), 33-52 (2015).
- Curtis, P. D., Brun, Y. V. Identification of essential alphaproteobacterial genes reveals operational variability in conserved developmental and cell cycle systems. Mol Microbiol. 93 (4), 713-735 (2014).
- Kim, J., Heindl, J. E., Fuqua, C. Coordination of division and development influences complex multicellular behavior in Agrobacterium tumefaciens. PLoS One. 8 (2), e56682 (2013).
- Jong, I. G., Beilharz, K., Kuipers, O. P., Veening, J. W. Live Cell Imaging of Bacillus subtilis and Streptococcus pneumoniae using Automated Time-lapse Microscopy. J Vis Exp. (53), (2011).
- Turnbull, L., et al. Super-resolution imaging of the cytokinetic Z ring in live bacteria using fast 3D-structured illumination microscopy (f3D-SIM). J Vis Exp. (91), e51469 (2014).
- Zeng, L., Golding, I. Following cell-fate in E. coli after infection by phage lambda. J Vis Exp. (56), e3363 (2011).
- Brown, P. J., et al. Polar growth in the Alphaproteobacterial order Rhizobiales. Proc Natl Acad Sci U S A. 109 (5), 1697-1701 (2012).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved