Bu içeriği görüntülemek için JoVE aboneliği gereklidir. Oturum açın veya ücretsiz deneme sürümünü başlatın.
Method Article
Temel işlemler sırasında mikrobiyal hücre büyümesini gözlemlemek için hücre Floresans mikroskobu canlı
Bu Makalede
Özet
Bakterilerde temel süreçleri işlevini anlamak zordur. Floresans mikroskobu hedef özel boyalar ile mikrobiyal hücre büyüme ve hücre döngüsü ilerleme içine anahtar anlayışlar sağlayabilir. Burada, Agrobacterium tumefaciens yöntemleri canlı hücre görüntüleme için gerekli işlemleri karakterizasyonu için vurgulamak için bir model bakteri kullanılmaktadır.
Özet
Temel hücresel süreçleri DNA ikileşmesi ve segregasyon, protein sentezi, hücre duvarı sentezi ve hücre bölünmesi gibi bakteriyel hayatta kalmak için gerekli proteinlerin işlevi güveniyor. Bu işlemler probları daha iyi anlamak gibi bir dizi hedef özel boyalar kullanılabilir. Lipofilik boyalar ile boyama membran yapısı gözlenmesi, lipid microdomains görselleştirme ve membran blebs olarak algılanmasını sağlar. Floresan-d-amino asitler (FDAAs) peptidoglikan biyosentezi sitelerin soruşturma için kullanımı hücre duvarı Dipnotlar veya hücre büyüme desenlendirme potansiyel kusurları işaret ediyor olabilir. Son olarak, nükleik asit lekeleri DNA çoğaltma veya Kromozom segregasyon olası hataları işaret ediyor olabilir. Siyanür DNA hücre yaşayan etiket lekeleri ve hızlandırılmış mikroskobu çevrili Morfoloji gerçek zamanlı gözlemleri sırasında hücre büyümesini etkinleştirme için uygundur. Hücre etiketleme için protokol protein tükenmesi mutantlar membran yapısı, hücre duvarı Biyogenez ya da kromozom segregasyon tanımlamak için uygulanabilir. Ayrıca, hızlandırılmış mikroskobu gibi gerekli bir protein kaldırılır ve ek protein işlevi kazandırabileceğini morfolojik değişiklikleri izlemek için kullanılabilir. Örneğin, hücre büyüme proteinler tükenmesi hücre daha kısa veya yuvarlak haline neden olabilir, ancak temel hücre bölünmesi proteinler tükenmesi filamentation veya dallanma, sonuçları. Burada, hücre büyümesi, hedef özel etiketleme ve hızlandırılmış mikroskobu protokollerde bakteriyel bitki patojeni Agrobacterium tumefacienssağlanır. Birlikte, hedef özel boyalar ve hızlandırılmış mikroskobu temel süreçleri karakterizasyonu etkinleştirmek A. tumefaciens. Son olarak, sağlanan iletişim kuralları diğer bakterilerde temel işlemleri soruşturma için kolayca değiştirilebilir.
Giriş
Bakteriyel hücre döngüsü boyunca ilerleme membran ve hücre duvarı sentezi, DNA ikileşmesi ve segregasyon ve hücre bölünmesi gibi birçok işlemi koordinasyon gerektirir. Tamamen bakteriyel hücre biyolojisi karmaşıklığını anlamak için bu temel olayları incelemek gereklidir; Başlıca bileşenleri bu yollar, mutagenized zaman hücre canlılığı tehlikeye beri Ancak, bu önemsiz bir görevdir. Hedef özel boyalar ile birleştiğinde Epifluorescence mikroskopi wildtype ve mutant bakteri suşları temel bu işlemlerde soruşturma için güçlü bir yaklaşımdır.
Peptidoglikan özel boyalar floresan antibiyotik (Vankomisin-FL, bocillin-FL) ve floresan d amino asitler (örneğin, 7-Hidroksikumarin-3-karboksilik asit-3-amino-d-alanin, HADA; 4-chloro-7-nitrobenzofurazan-3-amino-d-alanin içerir , NADA; tetramethylrhodamine-3-amino-d-alanin; TADA). Gram-pozitif bakterilerde peptidoglikan biyosentezi sitelerin soruşturma için floresan antibiyotik analogları sublethal konsantrasyonları kullanımı peptidoglikan ekleme desenleri1,2ortaya çıkarmak için etkili bir strateji olmuştur, 3,4. Floresan Vankomisin etiketleme ekleme desenleri sabit Ayagin Gram-negatif bakteri5peptidoglikan anlayışlar kazanmak için kullanılmıştır, dış membran genellikle floresan kullanımını engelleyen bir geçirgenliği bariyer sağlar antibiyotik olarak bir sonda peptidoglikan canlı hücreleri içinde biyosentezi için. Buna ek olarak, floresan-d-amino asit veya biorthogonal fonksiyonel gruplar ile d-amino asitler ve kısa darbeleri kovalent bölgelerinde yaşayan bakteri hücreleri6,7geniş bir alanda son peptidoglikan ekleme etiketleyin. Sentetik d amino asitler ile gözlenen desenleri peptidoglikan ekleme punctate ve septal içerir (Escherichia coli ve Bacillus subtilis), kutup ve septal (Agrobacterium tumefaciens ve Listeria Monositogenez), septal tek (Staphylococcus aureus) ve apikal (Streptomyces venezuelae)6,7. Bu gözlemler bakteri hücre duvarı dipnotlar farklı desenler sergi ve sentetik d amino asitler olarak büyüme desenlendirme incelenmesi için sonda kullanımı birçok bakteri içinde değerli bir stratejidir gösterir.
Bakteriyel kromozomlar etiket boya dahil deoksiribonükleik asit (DNA) belirli küçük groove cilt (4,6-diamidino-2-phenylindole; DAPI) ve yüksek afinite siyanür boyalar (yeşil ve turuncu malzeme listesini görmek;). Canlı hücrelerin DAPI boyama bakteriyel canlılığı9belirtmek için kullanılır, ancak sabit hücrelerin DAPI boyama numaralandırma çevre örnekleri8, bakterilerin yardımcı olur. Buna ek olarak, Sigara-hücrelerin9numaralandırmak için membran impermeant "ölü" hücre lekeleri gibi turuncu ve yeşil sık açıklandığı gibi siyanür boyuyor. Bu reaktifler hücre büyümesi sırasında bakteriyel çevrili morfolojisi soruşturma için kullanıldığında, dikkat çekici, DAPI, turuncu ve yeşil tüm membran permeant ve canlı hücreleri10etiketleme kabil olmak gösterilmiştir. E. coli hücreleri canlı, DAPI DNA'sı boyama sitoplazma auto-floresan nedeniyle yaygın görünür ve ultraviyole (UV) ışık için hücre DAPI lekeli tekrarlanan Etkilenmeler perturbs çevrili yapısı10. Bu boya membran permeant olup uzun süreli Floresans bağlama üzerine canlı hücrelerdeki DNA hücre büyümesi, DNA ikileşmesi veya Kromozom ayrımı10 etkilemeden sağlar boyama E. coli ya da B. subtilis ile portakal ortaya çıkarır . Bu gözlemler siyanür DNA boyalar nucleoids Morfoloji çoğu bakterinin hücre büyüme sırasında izlemek için kullanılabilir öneririz.
Phospholipid-Specific stryl boya N-(3-triethylammoniumpropyl)-4-(6-(4-(diethylamino) fenil gibi) hexatrienyl) pyridinium dibromide (4-64; bkz: malzeme listesi) Katyonik bileşiklerdir ve tercihen olumsuz ücret ilişkilendirmek fosfolipitler gibi cardiolipin ve phosphatidylglycerol11. 4-64 farklı bakteri membran etiketlemek için kullanıldığında farklı desenler gözlenir. Escherichia coli, 4-64 yan duvar boyunca bantlarında Polonyalılar zenginleştirilmiş ve bölünme siteler geç pre-divisional12hücreleri. Bacillus subtilisiçinde 4-64 etiketleme lipid spiraller13görselleştirme sağlar. Agrobacterium tumefaciens, 4-64 dış membran etiketleri ve karakteristik "nal" desen büyüme Kutbu14,15etiketleme yoksun olduğu görülmektedir. Bu gözlemler Bu bakterilerin varlığı nedeniyle heterojen lipid dağıtımları için hücresel asimetri katkıda lipid etki alanlarının sergi gösteriyor. Diffüz etiketleme varlığını gibi 4-64 etiketleme şekillerindeki değişiklikleri, blebs veya veziküller, invaginations veya membran büzülme dağıtım veya lipidler biyosentezi etkisi mutantlar karakterize bilgilendirici olabilir.
Hücre boyama ötesinde, proteinlerin temel süreçlerine katılan işlevi belirlenmesi gereklidir. Gerekli genler silmek ve fenotipik sonuçları çalışma mümkün değildir çünkü temel proteinler karakterizasyonu teknik olarak zordur. Böylece, protein tüketen alternatif yaklaşımlar ortaya çıkmıştır. Örneğin, temel bir gen bir indüklenebilir organizatörü tercihan--dan onun yerel organizatörü kontrolü altında konabilir. İndüklenebilir rehberleri gibi küçük moleküllere duyarlı vardır; 16, izopropil β-d-1-thiogalactopyranoside (IPTG)17,18,19,20,21, arabinoz22, vanillate17,23çinko, ve ksiloz23, böylece hedef gen transkripsiyonu sona erer ve uyarıcı kaldırıldığında faiz protein tükenmiştir. Küçük molekül RNA etkileşimleri hedef genlerin transkripsiyon CRISPR girişim25,26 engellemek için kullanılan sentetik riboswitches24 ilgi temel protein tüketen Alternatif yaklaşımları içerir hedef genlerin ve tarafından ClpXP proteaz bozulmayı peptid Etiketler hedef proteinler için kullandığı indüklenebilir protein yıkımı27,28 blok transkripsiyon için. Hücrelerin canlılık kaybetmeden tükenmesi suşları karakterizasyonu için sadece kısa bir süre sağlarsanız, bu nedenle, hücrelerin mikroskobik görüntüleme protein tüketimi sırasında zamanla karakterizasyonu için güçlü bir yaklaşım. Nitekim, mikroskobu canlı bakteri hücre hücre şekil bakım, salgı ve Bölünebilme29mekanizmaları da dahil olmak üzere temel biyolojik süreçlerin anlayışlar kazanmak araştırmacılar sağladı.
A. tumefaciens bir bakteriyel bitki patojeni30 ve doğal genetik mühendisi31,32olduğunu. Böylece, mekanizmalar patojen için ilgili ana bilgisayar-patojen etkileşimleri33,34,35, salgı36ve ana bilgisayar dönüşüm30,31, gibi 37 kapsamlı bir şekilde araştırıldı. A. tumefaciens aracılı hastalığı önlemek ya da bitki dönüşüm geliştirmek stratejiler tasarlamak için A. tumefaciens hayatta kalmak için gerekli işlemleri daha iyi anlaşılması gerekir. Hedef özel boya kullanımı ve son gelişmeler A. tumefaciens18 için bir protein tükenmesi stratejinin temel süreçleri araştırmak için bir yol sağlar.
Burada, A. tumefaciens wildtype, mutant ve protein tükenmesi suşları mikroskobik analizi için detaylı protokoller sağlanır. İlk iki protokol, hücreleri hazırlamak ve onları hedef özel boyalar ile etiket açıklanmıştır. Üçüncü protokol için özel yastıkları (şekil 1) hazırlama ve bakteri hücreleri (Şekil 2, şekil 3, şekil 4) görüntüleme hakkında adım adım yönergeler sağlar. Bu protokoller de farklı ortam koşulları, büyüme oranları, oksijen gereksinimleri ve hücre yapıları için hesabınıza ek uyarlamalar ile diğer bakteriler için uygun olabilir.
Protokol
1. A. tumefaciens suşlarının büyüme
-
A. tumefaciens suşları kültür
- Bir koloni istediğiniz baskı ile ATGN büyüme ortamının (tarifi için malzemeler listeye bakın) 1 mL aşılamak için steril bir tahta sopa ya da damlalıklı ipucu kullanın.
Not: A. tumefaciens tükenmesi suşları için ATGN 1 mM IPTG temel protein biyosentezi korumak için bir uyarıcı içermelidir. - A. tumefaciens suşları gecede ATGN 225 rpm'de sallayarak ile 28 ° C'de içinde büyümek.
- Hücreleri 600 optik yoğunluğunu ölçmek bir Spektrofotometre kullanarak nm (OD600). Hücre kültürü bir OD600 seyreltik ~0.2 = ve OD600 kadar büyümeye devam = ~0.6 ulaşıldığında. Tükenmesi suşları için OD600 kadar uyarıcı ile büyümeye devam = ~0.6 ulaşıldığında.
- A. tumefaciens wildtype ve mutant suşları OD600 kullanın = ~0.6 hedef özel boyama ve/veya hızlandırılmış mikroskobu (bkz: Bölüm 2 ve 3.1). Tükenmesi suşları için uyarıcı (bakınız Bölüm 1.2.) yıkamak
- Bir koloni istediğiniz baskı ile ATGN büyüme ortamının (tarifi için malzemeler listeye bakın) 1 mL aşılamak için steril bir tahta sopa ya da damlalıklı ipucu kullanın.
-
A. tumefaciens protein tükenmesi suşları karakterizasyonu için uyarıcı kaldırılması
- Üstel kültürünün 1 mL cips (OD600 ~0.4-0.6 =) tarafından Santrifüjü 7000 x g 5 dakika içinde oda sıcaklığında masaüstü santrifüj için de.
- Yıkamak için süpernatant kaldırın ve uyarıcı olmadan taze medya Pelet resuspend ve hücreleri (1.2.1) açıklandığı gibi cips. Hücreleri toplam taze medya 3 kez yıkayın.
- Son Pelet taze medyada resuspend ve konsantre bir OD600 hücrelere hemen hedef özel boyalar (Bölüm 2 ve şekil 4B-D) veya hızlandırılmış mikroskobu (Bölüm 3.2 ve şekil 4A) ile boyama için ~0.8 = . Alternatif olarak, hücreleri istediğiniz süreyi için medya olmadan önce boyama ve hücreleri Imaging uyarıcı hücrelerde büyüyen tarafından önceden tüketmek.
2. hedef özel boyama A. tumefaciens hücre
-
Floresan d-amino asit hücre duvarı etiketleme
Not: FDAAs kolayca A. tumefaciens peptidoglikan dahil edilmiştir toksik olmayan floresan d amino asitleri vardır. Bu basit etiketleme yordamı büyüme canlı hücreleri6' desenlendirme sondalar. Dört FDAAs sentezi için kullanılabilir38iletişim kurallarıdır.- Üstel kültürünün 500 µL cips (OD600 ~0.4-0.6 =) tarafından Santrifüjü 7000 x g 5 dakika içinde Masaüstü santrifüj için de. Hücre Pelet taze medya 100 µL içinde resuspend.
- 5 FDAA için yıkanmış ve hücrelerin konsantre ve karanlıkta 2 min için kuluçkaya mm 5 µL ekleyin.
Not: Sınırlamak FDAA ışığa maruz kalma. Kuluçka süresi zorlanma büyüme hızının bağlı olarak değişir. Genellikle, kuluçka kez katlama zaman%65-10 olmalıdır. - Hücreleri tarafından Santrifüjü cips ve hücre topakları fosfat tamponlu tuz (PBS) ile üç kez yıkayın.
- Pelet ~ 50 µL PBS içinde resuspend.
Not: PBS resuspension için kullanılan birim Pelet boyutuna göre değişebilir. - 0,8-1 uygulamak µL hücre bir özel defteri ve hemen epifluorescence mikroskobu (bkz: bölümler 3.1 ve 3.2) kullanarak görüntü için.
- Alternatif olarak, daha fazla etiket birleşme buz gibi % 70 etanol ile hücreleri düzelterek durdurmak. Hücre Pelet 1 mL buz gibi % 70 etanol resuspend ve 15 dakika boyunca buz üzerinde kuluçkaya.
- Santrifüjü tarafından hücreleri toplamak ve daha sonra görüntüleme için PBS küçük bir hacim içinde resuspend. Hücre süspansiyonlar 4 ° C ve görüntü 48 saat içinde saklayın.
-
DNA DAPI veya turuncu leke ile boyama
Not: DNA gözlemlemek için yapısı, hücre DAPI ya da portakal (ölü hücre leke) ile etiketlenir. Her ne kadar klasik bir "ölü hücre leke" açıklanan, turuncu permeant, photostable ve canlı hücre DNA10Imaging etkinleştirme bakteri hücrelerinin büyümesini etkilemez. A. tumefaciens, turuncu etiketleme işleri canlı hücreler içinde iyi ve hızlandırılmış mikroskobu (şekil 3) için uygundur. Buna ek olarak, ultraviyole (UV) ışık pozlama görüntüleme DAPI lekeli hücreler için gerekli Fototoksik (şekil 3) ve böylece DAPI boyama için anlık görüntüleme veya sabit hücreleri boyama canlı hücreleri boyama için en uygun olan.- DAPI hücreleri boyama
- Üstel kültürünün 1 mL cips (OD600 ~0.4-0.6 =) tarafından Santrifüjü 7000 x g 5 dakika içinde Masaüstü santrifüj için de.
- İsteğe bağlı olarak, hücre boyama önce düzeltmek için hücre Pelet 1 mL % 70 buz gibi etanol resuspend. 10-15 dk. içinde 2.2.1.1 hücreler Santrifüjü tarafından açıklanan toplamak için buz üzerinde kuluçkaya.
- Hücre Pelet 1 mL 1 µL DAPI hisse senedi çözüm (1 mg/mL; bkz: Malzemeler tablo) içeren PBS resuspend. Pipetting tarafından karışımı yavaşça ve 5 dakika içinde belgili tanımlık karanlık kuluçkaya.
- Hücreleri tarafından Santrifüjü 7000 x g 5 min için de cips ve 1 mL aşırı DAPI kaldırmak için PBS resuspend. Hücreleri iki kez daha yıkama ve Pelet 50 µL PBS veya medya resuspend.
- 0,8-1 uygulamak µL hücre bir özel defteri ve hemen epifluorescence mikroskobu kullanarak görüntü için. 3.1 ve 3.2 bölümlerine bakın.
Not: Bu protokol kromozom dynamics (şekil 3) gözlemlemek hızlandırılmış mikroskopi için en uygun değildir. Turuncu boyama (bakınız Bölüm 2.2.2) alternatif olarak kullanmayı düşünün.
- Turuncu canlı hücreleri boyama
- Üstel kültürünün 1 mL cips (OD600 ~0.4-0.6 =) tarafından Santrifüjü 7000 x g 5 dakika içinde Masaüstü santrifüj için de. Hücre Pelet 1 mL 1 µL turuncu hisse senedi çözüm içeren PBS resuspend (malzeme listeye bakın). Pipetting tarafından karışımı yavaşça ve karanlıkta 5 min için kuluçkaya.
- Hücreleri tarafından Santrifüjü cips ve 1 mL aşırı turuncu kaldırmak için PBS resuspend. Hücreleri iki kez daha yıkama ve Pelet 50 µL PBS resuspend.
- 0,8-1 uygulamak µL hücre bir özel defteri ve hemen epifluorescence mikroskobu kullanarak görüntü için. 3.1 ve 3.2 bölümlerine bakın.
Not: Bu protokol de canlı hücreleri ile çalışır ve hızlandırılmış mikroskobu kromozom dynamics (şekil 3) gözlemlemek uygundur. Hemen görüntüye uygun değilse, hücreler buz gibi % 70 etanol (bakınız Bölüm 2.2.1.2) sabit olabilir.
- DAPI hücreleri boyama
-
Membran etiketleme
Not: Lipofilik styryl floresan boya 4-64 yaygın bakteriyel hücre membran gözlemlemek için kullanılmıştır. Birçok bakteri, aksine 4-64 A. tumefaciens hücreleri etiketli düzgün etiket değil, ama oldukça sık sergi "nal" desen14,15. Dikkat çekici, büyüme Kutbu yaklaşık leke yoksun ise eski pole yoğun bir şekilde etiketlenmiştir. Böylece, 4-64 hücre büyüme desen ve A. tumefaciensmembran yapısı görselleştirme sağlar.- 8 µg/mL üssel faz hücre kültürü 1 ml nihai bir konsantrasyon FM 4-64 ekleyin ve 5 dakika içinde belgili tanımlık karanlık oda sıcaklığında kuluçkaya.
- 5 dakika içinde Masaüstü santrifüj için 7000 x g de Santrifüjü tarafından etiketli hücreleri cips ve hücre Pelet PBS 1 mL resuspend. Hücreleri aşırı boya kaldırmak için üç kez toplam yıkayın.
- PBS ve hemen görüntü için bir özel defteri yerinde küçük bir cilt hücrelerinde resuspend. (3.1 ve 3.2 bölümlerine bakın)
Dikkat: Hücreleri hemen karakteristik etiketleme desen gözlemlemek için yansıması gerekir.
3. A. tumefaciens hücreleri görüntüleme
-
Özel yüzey hazırlama
Not: Resim 1 resim silsilesi (Masası'nda) ve şematik (Masası B) hızlandırılmış mikroskopi için hazırlanan bir tipik özel ped içerir. Özel yastıkları genellikle gerektiği gibi hazırlanır.- 3.1.1. bir coverslip bir 22 x 22 mm laboratuvar kare film (malzeme listesini görmek) kenarlarda neşterden çalıştırarak bir rehber ve kesim olarak kullanın.
- ~ 2-5 bırakarak laboratuvar film Merkez dışında bir kare şeklinde bir kesik mm kenarlık için özel yastık bir conta olarak hizmet için. Merkezi kesme atmak.
- Bir amonyak ve alkol içermeyen temizleyici ile (bkz: malzeme listesi) temiz bir cam slayt (75 mm x 25 mm) üzerine film conta yeri ve film biraz cam ( şekil1a en iyi görüntü) üzerine erimiş kadar ısı. Laboratuvar film eritmek için 70 ° C ısı blok kümesine kenarına kullanın veya hafifçe bir ateşte eritin.
- Özel çözüm ~0.075 g özel karıştırarak hazırlayın (malzeme listesini görmek) küçük bir şişeye medyada 5 ml. Isı ile periyodik özel eriyene kadar karıştırın ve çözüm için dönen bir mikrodalga çözümde açıktır. Özel çözüm 55-70 ° C de tutabilir ve 48 saat içinde birden çok özel yastıkları inşaat için kullanabilirsiniz.
- 1.2-%1.5 özel conta ortasına içeren medya pipet.
Not: Medya hacmi genellikle 50-60 µL olmakla birlikte conta boyutuna göre değişir. Soğuk bir slayt için medya çok hızlı bir şekilde kuvvetlendirmek özel neden olabilir eklemek, böylece slayt sıcak bir yüzey üzerinde tutulmalıdır. Bir ısı blok kenarına ayarlayın 70 ° C çalışır iyi. Su özel veya fosfat tamponlu tuz (PBS) gibi arabelleğe alınan özel çözümler içeren özel uygulamalar sürekli hücre büyümesini gerekli değildir için medya yerine kullanılabilir. Tükenmesi suşları görüntüleme için uyarıcı olabilir sunmak da yok medyada. Uyarıcı yokluğu-ecek açığa vurmak tükenmesi fenotip ise uyarıcı varlığı bir denetim olarak kullanılabilir. - Bir coverslip özel olarak dağıtabilmenizi conta yerleştirin.
- Slayt bu buruşuk yüzeyinde özel defteri ve yüzeye uygulanan hücre süspansiyon havuzu da yol açar gibi ~ 2 dk. kaçının özel yastık •kızılötesi için kuvvetlendirmek için serin, düz bir yüzey üzerine yerleştirin.
- Dikkatle coverslip özel yastık kapalı kaydırın.
Dikkat: Bu adımı acele etmeyelim. Özel yastık kolayca gözyaşı ve düzensiz özel yastık görüntüleme zorlaştırabilir. - Ped yüzeyi kuru görünene kadar hava için özel defteri oda sıcaklığında 1-2 min için Kuru izin. Bir neşter kullanarak, özel hava boşluğuna (orta görüntü, şekil 1A); oluşturmak için a küçük soymak kaldırmak hava boşluğu genellikle ~ 2 x 7-10 mm ve A. tumefaciens hücreleri hava boşluğu (şekil 1 c) en iyi büyümeye eğilimindedir.
Not: Sabit hücre veya büyüme nerede izlenmiyor koşullar altında görüntüleme için hava boşluğuna gerekli değildir. - Nokta 0,8-1 µL özel defteri ve yeni bir coverslip ile kapak sayfasında bulunan bir hücreler. Yavaşça coverslip hücreleri özel ped yüzeyi boyunca dağıtmak için özel yastık tepesinden yerleştirin.
- Erimiş VALAP kullanarak coverslip kenarlarını kapatın (bkz: Malzemeler tablo için tarifi; Şekil 1A, alt resim). Tüm kenarları ve köşeleri coverslip mühür hücreleri görüntüleme sırasında sürüklenen götürecek özel ped kurutma neden olabilir. Not: Mühürleme coverslip sadece uzun vadeli zaman hata görüntüleme için gereklidir.
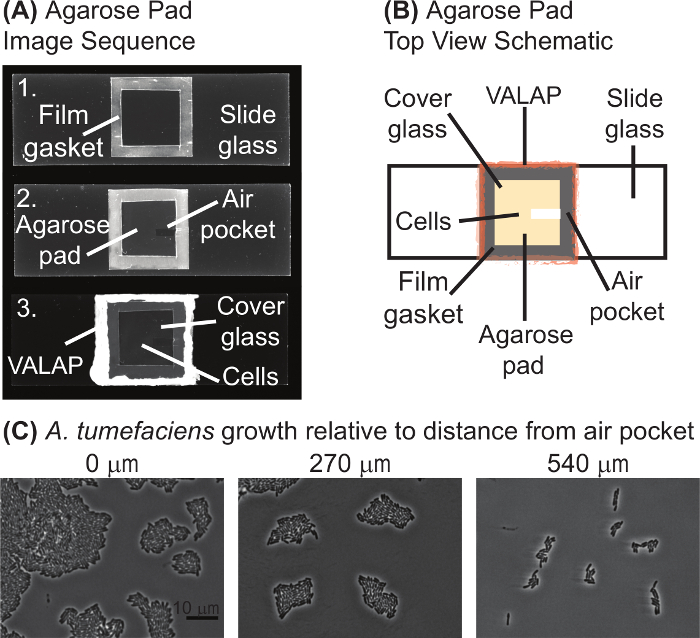
Şekil 1: özel yastık hazırlık. (A)görüntü özel yastık hazırlık protokolü sırasını. Resim 1 laboratuvar film conta ile bir slayttır. Resim 2, özel defteri ve hava boşluğu görüntülenir. Son olarak, Resim 3 tam özel yastık altında bir coverglass hücreleri ile gösterir ve VALAP ile mühürlenmiş. (B) A şematik bir özel ped hızlandırılmış mikroskopi için sağlanır. Anahtar şekil-in belgili tanımlık özel yastık şematik etiketlenir. (C) hava boşluğu A. tumefaciens büyüme özel yastıkları üzerinde terfi. Görüntüleri bir özel yastık üzerinde 20 saat boyunca yetiştirilen wildtype A. tumefaciens hücre gösterilir. Görüntüleri mevzilerine hava cebinden giderek uzak alındı. Görüntünün en yakın kenara uzaklığı hava boşluğu için yukarıdaki her görüntü gösterilir. Ölçek çubuğu 10 µm. = Bu rakam daha büyük bir versiyonunu görüntülemek için buraya tıklayınız.
-
Görüntüleme
Not: Fark girişim kontrast, faz kontrast ve epifluorescence görüntüleme yapılır donanım tabanlı autofocusing, otomatik bir sahne, standart filtrelere, bir LED ışık kaynağı, 60 X yağı daldırma ile donatılmış bir ters mikroskobu ile Hedefler (1.4 NA) faz kontrast veya fark girişim kontrast (DIC) ve 1K elektron teksir makinesi şarj-birleştiğinde-cihaz (EMCCD) kamera. Mikroskop bir sıcaklık kontrollü odada yer almalıdır. Alternatif olarak, bir sahne daha sıcak veya odası tutarlı sıcaklık görüntüleme sırasında korumak için kullanılabilir.- Epifluorescence mikroskobu
Not: floresan görüntüleme için canlı hücre, resim satın alma sayısını sınırlamak ve pozlama süresi photobleaching ve fototoksisite en aza indirmek için en iyi duruma getirmek için gerekli. Her boya görüntüleme için bu yeterli Floresans algılama sağlar ancak photobleaching veya fototoksisite yol değil bir en uygun çekim hızı bulmak için tavsiye edilir. Şekil 2-4' te, pozlama süreleri 200 MS tüm Floresans resimler için kullanılmıştır. Şekil 3' te gösterilen hızlandırılmış dizileri için her 10 min 3 h için görüntüleri elde.- Daldırma yağ istenen hedefte yerleştirin ve ters Slayt Slayt tutucusuna sahne alanı üzerine yerleştirin. Odaklama topuzlar hücreleri odak haline getirmek için kullanın.
- Aşama (ya da DIC) Albümdeki ve istenen floresan filtre edinme.
Not: Maksimal uyarma ve emisyon dalga boylarında Şekil 2, şekil 3ve şekil 4 kullanılan lekeler için aşağıdaki gibidir: HADA (405/460), NADA (450/555), TADA (555/570), 4-64 (515/640), DAPI (360/460), Orange (547/570).
- Hızlandırılmış mikroskobu
Not: bilgisayar kontrollü hızlandırılmış mikroskobu sırasında aşağıdaki özellikleri şunlardır: x, y ve z konum, panjurlar ve floresan filtreler. Bir donanım tabanlı otomatik odaklama sistemi odak zaman hata görüntüleme sırasında korumak için en iyi yöntemdir. Alternatif olarak, bir yazılım tabanlı otomatik odaklama döngü kullanılabilir.- Daldırma yağ istenen hedefte yerleştirin ve ters Slayt Slayt tutucusuna sahne alanı üzerine yerleştirin. Odaklama topuzlar hücreleri odak haline getirmek için kullanın.
- İsteğe bağlı olarak: Elde birden fazla (x, y) pozisyonlar. Rastgele 10 alan hücre yakın özel yastıkları hava boşluğu seçin.
- Sistem saat sıra satın görüntü için faz veya DIC istenilen zaman aralığında.
Not: şekil 4' te gösterilen hızlandırılmış sırası için 30 ms maruz her 10 min 14 h ile DIC görüntüleri elde. Floresans görüntüleme sırasında hızlandırılmış mikroskobu kullanırken, photobleaching ve fototoksisite en aza indirmek için satın alma aralığı ve pozlama zamanını ayarlayın.
- Epifluorescence mikroskobu
Sonuçlar
Hedef özgü wildtype etiketleme A. tumefaciens hücreleri
Bu hücre morfolojisi göstermek için yıkama adımları veya (hangi floresan boyalar sulandırmak için kullanılır) % 1 DMSO ile tedavi etkilenmez, hücreleri doğrudan kültür (şekil 2A, sol paneli), hücreler tarafından yıkadıktan sonra yansıma 1.2 (şekil 2A, sol panelde) veya % 1 DMSO 10 min iç...
Tartışmalar
Bu protokol A. tumefaciens tükenmesi wildtype ve mutant suşları incelenmesi için işlemler bir dizi içerir. Bu protokol bölümünde listelenen tüm yordamları büyüme medya, sıcaklık ve büyüme oranları için hesabınıza ek değişiklikler ile diğer bakteri suşları için kolayca adapte edilebilir dikkati çekiyor.
Hedef özel boyalar bakteri hücreleri hücre döngüsü olayları detaylı karakterizasyonu sağlamak için değerli bir araç kullanmaktır. Burada, peptid...
Açıklamalar
Yazarlar ifşa gerek yok.
Teşekkürler
Michael VanNieuwenhze (Indiana Üniversitesi) Resim 2 ve şekil 4kullanılan FDAAs hediye için teşekkür ediyoruz. Bu el yazması hazırlanması sırasında üye kahverengi laboratuarının geribildirim için teşekkür. A. tumefaciens hücre büyümesi ve bölünme kahverengi laboratuarında Araştırma Ulusal Bilim Vakfı (IOS1557806) tarafından desteklenmektedir.
Malzemeler
| Name | Company | Catalog Number | Comments |
| Bacterial Strains | |||
| Agrobacterium tumefaciens C58 | ATCC | 33970 | Watson B, Currier TC, Gordon MP, Chilton MD, Nester EW. 1975. Plasmid required for virulence of Agrobacterium tumefaciens. J Bacteriol 123:255-264. |
| Agrobacterium tumefaciens C58ΔtetRA::mini-Tn7T-GM-Ptac-ctrA ΔctrA | Figueroa-Cuilan W, Daniel JJ, Howell M, Sulaiman A, Brown PJB. 2016. Mini-Tn7 insertion in an artificial attTn7 site enables depeltion of the essentail master regulator CtrA in the phytopathogen Agrobacterium tumefaciens. Appl Environ Microbiol. 82:5015-5025. | ||
| Name | Company | Catalog Number | Comments |
| Media Components | |||
| ATGN Minimal Medium | To 1 L of sterilized water add 50 ml 20X Buffer, 50 ml 20X Salts, 12.5 ml 40% glucose. For plates, add 15 g Bacto Agar to 1 L of water and autoclave. Cool to 55 °C and add 50 ml 20X Buffer, 50 ml 20X Salts, 12.5 ml 40% glucose. | ||
| 20X AT Buffer | Add 214 g/L KH2PO4 to water and adjust pH to 7.0 with sodium hydroxide. Autoclave. | ||
| NaOH | Fisher BioReagents | BP359 | |
| KH2PO4 | Fisher Chemical | P288 | |
| 20X AT Salts | Add 40 g/L (NH4)2SO4, 3.2 g/L MgSO4•7H2O, 0.2 g/L CaCl2•2H2O, and 0.024 g/L MnSO4•H2O to water. Autoclave. | ||
| (NH4)2SO4 | Fisher Chemical | A701 | |
| MgSO4•7H2O | Fisher BioReagents | BP213 | |
| CaCl2•2H2O | Fisher BioReagents | BP510 | |
| MnSO4•H2O | Fisher Chemical | M114 | |
| Glucose | Fisher Chemical | D16 | Prepare 40% stock in water. Filter sterilize. |
| Bacto Agar | Fisher BioReagents | BP1423 | Add 15 g to 1 L of water when preparing plates. |
| Name | Company | Catalog Number | Comments |
| Optional Media Additives | |||
| Kanamycin | GoldBio | K-120 | Prepare as a 100 mg/ml stock solution in water and filter sterilize. Use at final concentration of 200 µg/ml. |
| IPTG | GoldBio | I2481C5 | Prepare as a 1 M stock solution in water and filter sterilize. Use at final concentration of 1 mM as needed for induction. |
| Name | Company | Catalog Number | Comments |
| Microscopy Materials | |||
| Microscope Slides | Fisherbrand | 12-550D | 25 X 75 X 1.0 mm. Clean with Sparkle glass cleaner. |
| Microscope Cover Glass | Fisherbrand | 12-541-B | 22 X 22 mm. No. 1.5. Clean with Sparkle glass cleaner. |
| Sparkle Glass Cleaner | Home Depot | 203261385 | Ammonia and alcohol free. |
| Ultra Pure Agarose | Invitrogen | 16500-100 | Add to water, PBS, or media to a final concentration of 1 - 1.5%. Melt in microwave and place on 70 C |
| PBS | Fisher BioReagents | BP399500 | 10X solution to be diluted to 1X with sterile water. |
| Parafilm | Bemis | PM-999 | Laboratory film used as gasket in agarose pad preparation. |
| VALAP | Add equal weights of lanolin, parafin wax, and petroleum jelly to a conical tube. Heat tube in 70 °C dry, bead or water bath to melt and mix. Apply VALAP while still molten. | ||
| Lanolin Butter | SAAQIN | SQ-LAB-R1 | |
| Petroleum Jelly | Target Corp. | 06-17644 | |
| Paraffin Wax | Crafty Candles | 263012 | |
| Name | Company | Catalog Number | Comments |
| Target-specific dyes | |||
| DMSO | Fisher BioReagents | BP231-1 | Use to dilute stock solutions of dyes as needed. |
| FDAAs (NADA, HADA,TADA) | FDAAs can be synthesized or acquired through agreement with Mike VanNieuwenhze (Indiana University). Prepare 100 mM stock solution in DMSO. Use at a final concentration of 5 mM. | ||
| DAPI | ThermoFisher Scientific | 62247 | Prepare 1 mg/ml stock solution in DMSO. Use at final concentration of 1 µg/ml. |
| SYTOX Orange Nucleic Acid Stain | Invitrogen | S11368 | Stock concentration is 5 mM in DMSO. Use at final concentration of 5 µM. |
| FM4-64 | Invitrogen | T3166 | Prepare 8 mg/ml stock solution in DMSO. Use at final concentration of 8 µg/ml. |
| Name | Company | Catalog Number | Comments |
| Equipment | |||
| Dry bath | Sheldon Manufacturing, Inc. | 52120-200 | |
| Metallic thermal beads | Lab Armor | 42370-002 | |
| Epifluorescence microscope equipped with an EMCDD camera | Nikon Eclipse TiE equipped with a QImaging Rolera em-c2 1K electron-multiplying charge-coupled-device (EMCCD) camera is used in this work. |
Referanslar
- Daniel, R. A., Errington, J. Control of cell morphogenesis in bacteria: two distinct ways to make a rod-shaped cell. Cell. 113 (6), 767-776 (2003).
- Tiyanont, K., et al. Imaging peptidoglycan biosynthesis in Bacillus subtilis with fluorescent antibiotics. Proc Natl Acad Sci U S A. 103 (29), 11033-11038 (2006).
- Turner, R. D., et al. Peptidoglycan architecture can specify division planes in Staphylococcus aureus. Nat Commun. 1, 26 (2010).
- Wheeler, R., Mesnage, S., Boneca, I. G., Hobbs, J. K., Foster, S. J. Super-resolution microscopy reveals cell wall dynamics and peptidoglycan architecture in ovococcal bacteria. Mol Microbiol. 82 (5), 1096-1109 (2011).
- Turner, R. D., Hurd, A. F., Cadby, A., Hobbs, J. K., Foster, S. J. Cell wall elongation mode in Gram-negative bacteria is determined by peptidoglycan architecture. Nat Commun. 4, 1496 (2013).
- Kuru, E., et al. In Situ probing of newly synthesized peptidoglycan in live bacteria with fluorescent D-amino acids. Angew Chem Int Ed Engl. 51 (50), 12519-12523 (2012).
- Siegrist, M. S., et al. (D)-Amino acid chemical reporters reveal peptidoglycan dynamics of an intracellular pathogen. ACS Chem Biol. 8 (3), 500-505 (2013).
- Kepner, R. L., Pratt, J. R. Use of fluorochromes for direct enumeration of total bacteria in environmental samples: past and present. Microbiol Rev. 58 (4), 603-615 (1994).
- Johnson, M. B., Criss, A. K. Fluorescence microscopy methods for determining the viability of bacteria in association with mammalian cells. J Vis Exp. (79), (2013).
- Bakshi, S., et al. Nonperturbative imaging of nucleoid morphology in live bacterial cells during an antimicrobial peptide attack. Appl Environ Microbiol. 80 (16), 4977-4986 (2014).
- Barak, I., Muchova, K. The role of lipid domains in bacterial cell processes. Int J Mol Sci. 14 (2), 4050-4065 (2013).
- Fishov, I., Woldringh, C. L. Visualization of membrane domains in Escherichia coli. Mol Microbiol. 32 (6), 1166-1172 (1999).
- Barak, I., Muchova, K., Wilkinson, A. J., O'Toole, P. J., Pavlendova, N. Lipid spirals in Bacillus subtilis and their role in cell division. Mol Microbiol. 68 (5), 1315-1327 (2008).
- Cameron, T. A., Anderson-Furgeson, J., Zupan, J. R., Zik, J. J., Zambryski, P. C. Peptidoglycan synthesis machinery in Agrobacterium tumefaciens during unipolar growth and cell division. MBio. 5 (3), e01219 (2014).
- Zupan, J. R., Cameron, T. A., Anderson-Furgeson, J., Zambryski, P. C. Dynamic FtsA and FtsZ localization and outer membrane alterations during polar growth and cell division in Agrobacterium tumefaciens. Proc Natl Acad Sci U S A. 110 (22), 9060-9065 (2013).
- Eberhardt, A., Wu, L. J., Errington, J., Vollmer, W., Veening, J. W. Cellular localization of choline-utilization proteins in Streptococcus pneumoniae using novel fluorescent reporter systems. Mol Microbiol. 74 (2), 395-408 (2009).
- Iniesta, A. A., Garcia-Heras, F., Abellon-Ruiz, J., Gallego-Garcia, A., Elias-Arnanz, M. Two systems for conditional gene expression in Myxococcus xanthus inducible by isopropyl-beta-D-thiogalactopyranoside or vanillate. J Bacteriol. 194 (21), 5875-5885 (2012).
- Figueroa-Cuilan, W., Daniel, J. J., Howell, M., Sulaiman, A., Brown, P. J. Mini-Tn7 Insertion in an Artificial attTn7 Site Enables Depletion of the Essential Master Regulator CtrA in the Phytopathogen Agrobacterium tumefaciens. Appl Environ Microbiol. 82 (16), 5015-5025 (2016).
- Jacob, F., Monod, J. Genetic regulatory mechanisms in the synthesis of proteins. J Mol Biol. 3, 318-356 (1961).
- Khan, S. R., Gaines, J., Roop, R. M., Farrand, S. K. Broad-host-range expression vectors with tightly regulated promoters and their use to examine the influence of TraR and TraM expression on Ti plasmid quorum sensing. Appl Environ Microbiol. 74 (16), 5053-5062 (2008).
- Yansura, D. G., Henner, D. J. Use of the Escherichia coli lac repressor and operator to control gene expression in Bacillus subtilis. Proc Natl Acad Sci U S A. 81 (2), 439-443 (1984).
- Guzman, L. M., Belin, D., Carson, M. J., Beckwith, J. Tight regulation, modulation, and high-level expression by vectors containing the arabinose PBAD promoter. J Bacteriol. 177 (14), 4121-4130 (1995).
- Thanbichler, M., Iniesta, A. A., Shapiro, L. A comprehensive set of plasmids for vanillate- and xylose-inducible gene expression in Caulobacter crescentus. Nucleic Acids Res. 35 (20), e137 (2007).
- Topp, S., et al. Synthetic riboswitches that induce gene expression in diverse bacterial species. Appl Environ Microbiol. 76 (23), 7881-7884 (2010).
- Peters, J. M., et al. A Comprehensive, CRISPR-based Functional Analysis of Essential Genes in Bacteria. Cell. 165 (6), 1493-1506 (2016).
- Qi, L. S., et al. Repurposing CRISPR as an RNA-guided platform for sequence-specific control of gene expression. Cell. 152 (5), 1173-1183 (2013).
- Griffith, K. L., Grossman, A. D. Inducible protein degradation in Bacillus subtilis using heterologous peptide tags and adaptor proteins to target substrates to the protease ClpXP. Mol Microbiol. 70 (4), 1012-1025 (2008).
- McGinness, K. E., Baker, T. A., Sauer, R. T. Engineering controllable protein degradation. Mol Cell. 22 (5), 701-707 (2006).
- Schneider, J. P., Basler, M. Shedding light on biology of bacterial cells. Philos Trans R Soc Lond B Biol Sci. 371 (1707), (2016).
- Escobar, M. A., Dandekar, A. M. Agrobacterium tumefaciens as an agent of disease. Trends Plant Sci. 8 (8), 380-386 (2003).
- Gelvin, S. B. Agrobacterium-mediated plant transformation: the biology behind the "gene-jockeying" tool. Microbiol Mol Biol Rev. 67 (1), 16-37 (2003).
- Nester, E. W. Agrobacterium: nature's genetic engineer. Front Plant Sci. 5, 730 (2014).
- Imam, J., Singh, P. K., Shukla, P. Plant Microbe Interactions in Post Genomic Era: Perspectives and Applications. Front Microbiol. 7, 1488 (2016).
- Subramoni, S., Nathoo, N., Klimov, E., Yuan, Z. C. Agrobacterium tumefaciens responses to plant-derived signaling molecules. Front Plant Sci. 5, 322 (2014).
- Yuan, Z. C., Williams, M. A really useful pathogen, Agrobacterium tumefaciens. Plant Cell. 24 (10), (2012).
- Alvarez-Martinez, C. E., Christie, P. J. Biological diversity of prokaryotic type IV secretion systems. Microbiol Mol Biol Rev. 73 (4), 775-808 (2009).
- Pitzschke, A. Agrobacterium infection and plant defense-transformation success hangs by a thread. Front Plant Sci. 4, 519 (2013).
- Kuru, E., Tekkam, S., Hall, E., Brun, Y. V., Van Nieuwenhze, M. S. Synthesis of fluorescent D-amino acids and their use for probing peptidoglycan synthesis and bacterial growth in situ. Nat Protoc. 10 (1), 33-52 (2015).
- Curtis, P. D., Brun, Y. V. Identification of essential alphaproteobacterial genes reveals operational variability in conserved developmental and cell cycle systems. Mol Microbiol. 93 (4), 713-735 (2014).
- Kim, J., Heindl, J. E., Fuqua, C. Coordination of division and development influences complex multicellular behavior in Agrobacterium tumefaciens. PLoS One. 8 (2), e56682 (2013).
- Jong, I. G., Beilharz, K., Kuipers, O. P., Veening, J. W. Live Cell Imaging of Bacillus subtilis and Streptococcus pneumoniae using Automated Time-lapse Microscopy. J Vis Exp. (53), (2011).
- Turnbull, L., et al. Super-resolution imaging of the cytokinetic Z ring in live bacteria using fast 3D-structured illumination microscopy (f3D-SIM). J Vis Exp. (91), e51469 (2014).
- Zeng, L., Golding, I. Following cell-fate in E. coli after infection by phage lambda. J Vis Exp. (56), e3363 (2011).
- Brown, P. J., et al. Polar growth in the Alphaproteobacterial order Rhizobiales. Proc Natl Acad Sci U S A. 109 (5), 1697-1701 (2012).
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiThis article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır