Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Vivre la microscopie de Fluorescence cellulaire afin d’observer les processus essentiels au cours de la croissance des cellules microbiennes
Dans cet article
Résumé
Comprendre la fonction des processus essentiels chez les bactéries est difficile. Microscopie à fluorescence avec des colorants spécifiques à la cible peut éclairer clé dans la progression de la croissance et cycle cellulaire des cellules microbiennes. Ici, Agrobacterium tumefaciens est utilisé comme une bactérie modèle pour mettre en évidence les méthodes d’imagerie de cellules vivantes pour la caractérisation des processus essentiels.
Résumé
Le processus cellulaire de base telles que la réplication de l’ADN et la ségrégation, la synthèse des protéines, la biosynthèse de la paroi cellulaire et la division cellulaire s’appuient sur la fonction des protéines qui sont essentielles pour la survie de bactéries. Une série de colorants spécifiques à la cible peut être utilisée comme sondes afin de mieux comprennent ces processus. Coloration avec colorants lipophiles permet l’observation de la structure de la membrane, la visualisation des microdomaines lipidiques et détection des bulles de la membrane. Utilisation des acides aminés fluorescentes-d (FDAAs) pour sonder les sites de la biosynthèse du peptidoglycane peut indiquer des défauts potentiels dans la biogenèse pariétale ou patrons de croissance cellulaire. Enfin, taches d’acide nucléique peuvent indiquer des défauts possibles en ségrégation réplication ou chromosomique d’ADN. Cyanine ADN taches étiquette vivant des cellules et sont adapté pour la microscopie Time-lapse permettant des observations en temps réel de nucléoïde morphologie au cours de la croissance cellulaire. Protocoles pour l’étiquetage de la cellule peuvent être appliqués à des mutants déplétion protéique à repérer les défauts dans la structure de la membrane, biogenèse pariétale ou ségrégation des chromosomes. Par ailleurs, Time-lapse microscopie peut être utilisée pour surveiller les changements morphologiques comme une protéine essentielle est supprimée et peut fournir des indications supplémentaires en fonction de la protéine. Par exemple, l’appauvrissement de la couche de protéines essentielles division cellulaire entraînant filamentation ou ramification, considérant que la déplétion des protéines de croissance cellulaire peut-être causer des cellules à devenir plus courts ou plus rond. Ici, les protocoles pour la croissance cellulaire, l’étiquetage spécifique à la cible et la microscopie Time-lapse sont fournis pour le phytopathogène bactéries Agrobacterium tumefaciens. Ensemble, colorants spécifiques à la cible et le Time-lapse microscopie permettent la caractérisation des processus essentiels dans a. tumefaciens. Enfin, les protocoles fournis peuvent être facilement modifiés pour sonder les processus essentiels chez d’autres bactéries.
Introduction
Progression dans le cycle cellulaire bactérienne nécessite la coordination de nombreux processus, y compris la biosynthèse de la membrane et la paroi cellulaire, réplication de l’ADN et la ségrégation et la division cellulaire. Pour bien comprendre la complexité de la biologie de la cellule bactérienne, il est nécessaire d’étudier ces phénomènes essentiels ; Cependant, c’est une tâche non triviale puisque la viabilité cellulaire est compromise lorsque les éléments clés de ces voies sont mutagénisées tant. Microscopie à épifluorescence couplée avec des colorants spécifiques à la cible est une approche puissante pour sonder ces processus essentiels dans le type sauvage et les souches bactériennes mutantes.
Colorants de peptidoglycane spécifique incluent antibiotiques fluorescents (vancomycine-FL, bocillin-FL) et des acides aminés fluorescentes-d (par exemple, 7-hydroxycoumarine-3-carboxylic acide 3-amino-d-alanine, HADA ; 4-chloro-7-nitrobenzofurazan-3-amino-d-alanine , NADA ; tétraméthylrhodamine-3-amino-d-alanine ; TADA). Chez les bactéries Gram-positives, l’utilisation de concentrations sublétales de fluorescents antibiotiques analogues aux sites de la biosynthèse du peptidoglycane de la sonde a été une stratégie efficace pour révéler le peptidoglycane insertion patrons1,2, 3,4. Alors que le marquage fluorescent à la vancomycine a été utilisé pour acquérir des connaissances sur le peptidoglycane modes d’insertion dans des bactéries Gram-négatives fixe5, la membrane externe fournit généralement une barrière de perméabilité qui empêche l’utilisation de la fluorescence antibiotiques comme une sonde pour la biosynthèse du peptidoglycane dans des cellules vivantes. En revanche, des impulsions courtes de fluorescent-d acides aminés ou des acides aminés d avec les groupes fonctionnels bi-orthogonale étiquette par covalence régions d’insertion récente de peptidoglycane dans un large éventail de la vie des cellules bactériennes6,7. Modes d’insertion de peptidoglycane qui ont été observées avec les acides aminés d synthétiques comprennent ponctuée et septale (Escherichia coli et Bacillus subtilis), polar et septale (Agrobacterium tumefaciens et Listeria monocytogenes), seule septale (Staphylococcus aureus) et apicale (Streptomyces venezuelae)6,7. Ces observations indiquent que les bactéries présentent divers patrons de paroi cellulaire biogenèse et que l’utilisation d’acides aminés d synthétiques comme sondes pour l’examen des motifs de croissance est une stratégie utile dans de nombreuses bactéries.
Les colorants qui étiquette les chromosomes bactériens comprennent le liant spécifique petit sillon de l’acide désoxyribonucléique (ADN) (4, 6-diamidino-2-phénylindole ; DAPI) et colorants cyanine de haute affinité (vert et Orange ; Voir la liste des matériaux). DAPI souillant des cellules fixes aide dénombrement des bactéries présentes dans les échantillons environnementaux8, tandis que la coloration DAPI de cellules vivantes est utilisée pour indiquer la viabilité bactérienne9. En revanche, cyanine dyes comme Orange et vert sont souvent décrits comme cellule « morte » imperméants membrane taches d’énumérer des cellules non viables9. Remarquablement, lorsque ces réactifs sont utilisés pour sonder la morphologie du nucléoïde bactérienne au cours de la croissance cellulaire, DAPI, Orange et vert ont été tous avéré membrane perméable et capable de marquage des cellules vivantes10. Dans vivre cellules d’Escherichia coli , DAPI, coloration de l’ADN apparaît diffuse en raison de l’auto-fluorescence du cytoplasme et de cellules colorés au DAPI, des expositions répétées aux rayons ultraviolets (UV) perturbe le nucléoïde structure10. Coloration d’e. coli ou de b. subtilis avec Orange révèle que ce colorant est membrane perméable et fournit la fluorescence longue durée lors de la liaison à l’ADN dans des cellules vivantes sans impact sur la croissance cellulaire, réplication de l’ADN ou ségrégation des chromosomes10 . Ces observations suggèrent que cyanine ADN colorants peuvent être utilisés pour surveiller la morphologie des nucléoïdes au cours de la croissance cellulaire dans beaucoup de bactéries.
Phospholipid-specific stryl colorants tels que N-(3-triethylammoniumpropyl)-4-(6-(4-(diethylamino) phényle) hexatrienyl) dibromure de pyridinium (4-64 ; Voir la liste des matériaux) sont des composés cationiques et associer préférentiellement avec une charge négative phospholipides tels que cardiolipine et phosphatidylglycérol11. Des profils distincts sont observées lorsque 4-64 est utilisé pour indiquer la membrane des bactéries différentes. Chez Escherichia coli, 4-64 est enrichi en polonais, en bandes le long de la paroi latérale et dans les sites de la division de la fin pre-divisional des cellules de12. Chez Bacillus subtilis, 4-64 étiquetage permet la visualisation des lipides spirales13. Agrobacterium tumefaciens, 4-64 étiquettes la membrane externe et on observe un motif caractéristique « fer à cheval » dans lequel le pôle de croissance est dépourvue d’étiquetage14,15. Ces observations indiquent que ces bactéries présentent des distributions de lipides hétérogène en raison de la présence de domaines lipidiques qui contribuent à l’asymétrie cellulaire. Changements dans 4-64 étiquetage telles que la présence de marquage diffus, bulles ou de vésicules, invaginations ou rétrécissement de la membrane peut être instructif pour la caractérisation des mutants qui touchent la distribution ou la biosynthèse des lipides.
Au-delà de la coloration des cellules, il est nécessaire de déterminer la fonction des protéines participant au processus essentiels. La caractérisation de protéines essentielles est techniquement difficile car il n’est pas possible de supprimer les gènes essentiels et d’étudier les conséquences phénotypiques. Ainsi, des approches alternatives qui appauvrissent la protéine ont vu le jour. Par exemple, un gène essentiel peut être mis sous le contrôle d’un promoteur inductible plutôt que son promoteur natif. Promoteurs inductibles répondent aux petites molécules telles que ; zinc16, isopropyl β-d-1-thiogalactopyranoside (IPTG)17,18,19,20,21, arabinose,22,23du vanillate17,, et xylose23, donc la transcription du gène cible cesse et la protéine d’intérêt est épuisée lorsque l’inducteur est supprimé. Les approches alternatives pour épuisement des protéines essentielles d’intérêt incluent riborégulateurs synthétique24 qui utilisent des interactions de petite molécule-RNA faire obstacle à la transcription de gènes cibles, CRISPR interférence25,26 à bloc la transcription des gènes cibles et protéine inductible dégradation27,28 , qui utilise des balises de peptides de protéines cibles pour la dégradation par la protéase ClpXP. Souches de déplétion ne fournissent que peu de temps pour la caractérisation avant les cellules perdent la viabilité, imagerie microscopique des cellules au fil du temps pendant la déplétion protéique est donc une approche puissante pour la caractérisation. En effet, la microscopie des cellules bactériennes vivantes a permis aux chercheurs de mieux comprendre les processus biologiques fondamentaux, y compris les mécanismes d’entretien de forme cellulaire, sécrétion et compartimentation29.
A. tumefaciens est une plante bactérienne pathogène30 et ingénieur en génétique naturelle31,32. Ainsi, des mécanismes associés à la pathogénicité, y compris host-pathogen interactions33,34,35, sécrétion36et hôte transformation30,31, 37 ont été étudiées. Pour concevoir des stratégies pour prévenir les maladies de a. tumefaciens médiation ou améliorer la transformation des plantes, les processus essentiels pour la survie d’a. tumefaciens doivent être mieux comprises. L’utilisation de colorants spécifiques à la cible et le développement récent d’une stratégie de déplétion protéique pour a. tumefaciens18 fournit un moyen d’enquêter sur les processus essentiels.
Ici, des protocoles détaillés pour analyse microscopique des souches de type sauvage, mutant et protéine appauvrissement d’a. tumefaciens sont fournis. Les deux premiers protocoles décrivent comment préparer les cellules et étiquetez-les avec des colorants spécifiques à la cible. Le troisième protocole fournit des directives étape par étape pour préparer les coussinets de gel d’agarose (Figure 1) et l’imagerie des cellules bactériennes (Figure 2, Figure 3, , Figure 4). Ces protocoles peuvent également être adaptés pour d’autres bactéries avec des adaptations supplémentaires pour tenir compte des conditions de milieux différents, les taux de croissance, besoins en oxygène et structures cellulaires.
Protocole
1. croissance des souches d’a. tumefaciens
-
Mise en culture des souches d’a. tumefaciens
- Utiliser une pointe en bois de bâton ou pipette stérile pour ensemencer 1 mL de milieu de culture ATGN (voir la liste des matériaux pour la recette) avec une seule colonie de la souche désirée.
NOTE : Pour les souches d’a. tumefaciens de l’appauvrissement, l’ATGN doit contenir 1 mM IPTG comme inducteur de maintenir la biosynthèse des protéines essentielles. - Cultiver les souches d’a. tumefaciens nuit à ATGN à 28 ° C sous agitation à 225 t/mn.
- Mesurer la densité optique des cellules à 600 nm (OD600) à l’aide d’un spectrophotomètre. Diluer la culture cellulaire à une OD600 = ~0.2 et continue de grandir jusqu'à OD600 = ~0.6 est atteint. Pour les souches de l’appauvrissement de la couche, continue de grandir avec inducteur jusqu'à OD600 = ~0.6 est atteint.
- Utiliser des souches de type sauvage et mutant d’a. tumefaciens OD600 = ~0.6 pour une coloration spécifique à la cible ou Time-lapse microscopie (voir les sections 2 et 3.1). Pour les souches de l’appauvrissement de la couche, lavent l’inducteur (voir la section 1.2).
- Utiliser une pointe en bois de bâton ou pipette stérile pour ensemencer 1 mL de milieu de culture ATGN (voir la liste des matériaux pour la recette) avec une seule colonie de la souche désirée.
-
Enlèvement d’inducteur pour la caractérisation des souches de déplétion protéique a. tumefaciens
- 1 mL de culture exponentielle de granule (OD600 = ~0.4-0.6) par centrifugation à 7 000 x g pendant 5 min dans une centrifugeuse Bureau à température ambiante.
- Pour laver, éliminer le surnageant et Resuspendre le culot dans les supports neufs sans inducteur et les cellules de granule, tel que décrit ci-dessus (1.2.1). Laver les cellules 3 fois dans les supports neufs.
- Resuspendre le culot final dans les supports neufs et concentrer les cellules à une OD600 = ~0.8 pour une coloration immédiate avec des colorants spécifiques cible (section 2 et Figure 4 b-D) ou Time-lapse microscopie (section 3.2 et Figure 4 a) . Alternativement, pré appauvrissent les cellules pendant le temps désiré par les cellules dans les médias sans inducteur avant la coloration et l’imagerie des cellules en croissance.
2. objectif spécifique coloration des cellules d’a. tumefaciens
-
Marquage fluorescent d-amino acide pariétaux
Remarque : FDAAs sont non toxique des acides aminés d fluorescents qui sont facilement intégrés dans le peptidoglycane d’a. tumefaciens . Cette procédure simple étiquetage sondes croissance patterning dans des cellules vivantes6. Protocoles pour la synthèse des quatre FDAAs sont disponible38.- Pellets 500 µL de culture exponentielle (OD600 = ~0.4-0.6) par centrifugation à 7 000 x g pendant 5 min dans une centrifugeuse de bureau. Resuspendre le culot dans 100 µL de supports neufs.
- Ajouter 5 µL de 5 mM FDAA à lavé et concentrée de cellules et incuber pendant 2 min dans l’obscurité.
NOTE : Limiter l’exposition à la lumière FDAA. Le temps d’incubation varie selon le taux de croissance de la souche. En règle générale, les temps d’incubation devraient être 5 à 10 % du temps doublement6. - Les cellules de granule par centrifugation et laver granules cellulaires avec du sérum physiologique tamponné au phosphate (PBS) trois fois.
- Resuspendre le culot dans ~ 50 µL de PBS.
Remarque : Le volume de PBS, utilisé pour la remise en suspension peut-être varier selon le taille de granule. - Appliquer 0,8-1 µL de cellules pour un coussin de gel d’agarose et image à l’aide de la microscopie à épifluorescence immédiatement (voir les sections 3.1 et 3.2).
- Vous pouvez également arrêter davantage incorporation d’étiquette en fixant les cellules avec glacee éthanol à 70 %. Resuspendre le culot dans 1 mL d’éthanol glacé à 70 % et incuber sur la glace pendant 15 minutes.
- Prélever des cellules par centrifugation et remettre en suspension dans un petit volume de PBS pour l’imagerie à une date ultérieure. Stocker à 4 ° C et l’image des suspensions de cellules dans les 48 heures.
-
ADN coloration au DAPI ou tache Orange
Remarque : Pour observer l’ADN structure, les cellules sont marquées avec DAPI ou Orange (tache de cellules mortes). Bien que classiquement décrite comme une « tache de cellules mortes », Orange est perméable, PhotoStation et n’affecte pas la croissance des cellules bactériennes, permettant aux cellules vivantes ADN imagerie10. Dans a. tumefaciens, travaux de marquage Orange bien dans les cellules vivantes et est adapté pour la microscopie Time-lapse (Figure 3). En revanche, l’ultraviolet (UV) exposition à la lumière nécessaire pour l’imagerie de cellules colorés au DAPI est phototoxique (Figure 3) et donc la coloration DAPI est le mieux adaptée pour la coloration des cellules vivantes pour immédiate d’imagerie ou de souiller les cellules fixes.- La coloration des cellules DAPI
- 1 mL de culture exponentielle de granule (OD600 = ~0.4-0.6) par centrifugation à 7 000 x g pendant 5 min dans une centrifugeuse de bureau.
- Éventuellement, pour fixer les cellules avant la coloration, resuspendre le culot cellulaire dans 1 mL d’éthanol glacé à 70 %. Incuber sur glace pendant 10-15 min. collecter les cellules par centrifugation comme décrit au point 2.2.1.1.
- Resuspendre le culot dans 1 mL de PBS contenant 1 µL de solution mère DAPI (1 mg/mL ; Voir le Tableau des matériaux). Mélanger doucement de pipetage et incuber pendant 5 minutes dans l’obscurité.
- Cellules de granule par centrifugation à 7 000 x g pendant 5 min et remettre en suspension dans 1 mL de PBS pour enlever l’excès DAPI. Laver les cellules deux fois plus et Resuspendre le culot dans 50 µL PBS ou médias.
- Appliquer 0,8-1 µL de cellules pour un coussin de gel d’agarose et image en utilisant la microscopie à épifluorescence immédiatement. Voir les sections 3.1 et 3.2.
Remarque : Ce protocole n’est pas optimal pour la microscopie Time-lapse d’observer la dynamique des chromosomes (Figure 3). Envisagez d’utiliser une coloration Orange comme alternative (voir la section 2.2.2).
- Coloration orange de cellules vivantes
- 1 mL de culture exponentielle de granule (OD600 = ~0.4-0.6) par centrifugation à 7 000 x g pendant 5 min dans une centrifugeuse de bureau. Resuspendre le culot dans 1 mL de PBS contenant 1 µL de solution mère de Orange (voir la liste des matériaux). Mélanger doucement de pipetage et incuber pendant 5 min dans le noir.
- Cellules de granule par centrifugation et remettre en suspension dans 1 mL de PBS pour enlever excès Orange. Laver les cellules deux fois plus et Resuspendre le culot dans 50 µL de PBS.
- Appliquer 0,8-1 µL de cellules pour un coussin de gel d’agarose et image en utilisant la microscopie à épifluorescence immédiatement. Voir les sections 3.1 et 3.2.
Remarque : Ce protocole fonctionne bien avec des cellules vivantes et est adapté pour la microscopie Time-lapse d’observer la dynamique des chromosomes (Figure 3). S’il n’est pas commode d’imager immédiatement, les cellules peuvent être fixés dans glacee éthanol à 70 % (voir point 2.2.1.2).
- La coloration des cellules DAPI
-
Membrane d’étiquetage
Remarque : Le colorant fluorescent lipophiles styryl 4-64 a été largement utilisé pour l’observation de la membrane des cellules bactériennes. Contrairement à nombreuses bactéries, 4-64 marqué a. tumefaciens cellules étiqueter pas uniformément, mais plutôt fréquemment présentent un « fer à cheval » modèle14,15. Remarquablement, le pôle de croissance est presque dépourvu de taches alors que le pôle vieux est étiqueté intensément. Ainsi, 4-64 permet la visualisation des profils de croissance cellulaire et structure de la membrane dans a. tumefaciens.- Ajouter FM 4-64 à une concentration finale de 8 µg/mL à 1 mL de culture de cellules de phase exponentielle et incuber à température ambiante pendant 5 minutes dans l’obscurité.
- Granulés de cellules marquées par centrifugation à 7 000 x g pendant 5 minutes dans une centrifugeuse bureau et Resuspendre le culot dans 1 mL de PBS. Laver les cellules au total trois fois pour enlever le colorant excédentaire.
- Remettre en suspension les cellules dans un petit volume de PBS et comptant sur une garniture d’agarose pour image immédiatement. (Voir les sections 3.1 et 3.2)
ATTENTION : Les cellules doivent être copiés immédiatement afin d’observer les motifs caractéristiques d’étiquetage.
3. imagerie de cellules a. tumefaciens
-
Préparation de coussinet de gel d’agarose
Remarque : La Figure 1 contient une séquence d’images (groupe A) et le schéma (groupe B), d’une plaquette d’agarose typiques préparée pour la microscopie Time-lapse. Coussinets de gel d’agarose sont typiquement préparés selon les besoins.- 3.1.1. utiliser un lamelle couvre-objet comme guide et couper une 22 x 22 mm carré de laboratoire film (voir la liste des matériaux) en exécutant un scalpel autour des bords.
- Découper un carré dans le centre du film laboratoire laissant un ~ 2-5 bordure mm pour servir un joint pour le coussin de gel d’agarose. Jeter la découpe du centre.
- Placez le joint de film sur une lame de verre (75 x 25 mm) nettoyé avec l’ammoniac et nettoyant sans alcool (voir la liste des matériaux) et chauffer jusqu'à ce que le film est un peu fondu sur verre (image du haut dans la Figure 1 a). Pour faire fondre un film de laboratoire, utilisez le tranchant d’un ensemble de bloc thermique à 70 ° C ou faire fondre légèrement au-dessus d’une flamme.
- Préparer la solution d’agarose en mélangeant ~0.075 g d’agarose (voir la liste des matériaux) dans 5 mL de médias dans un petit ballon. Chauffer la solution dans un four à micro-ondes avec périodique remuant pour bien mélanger jusqu'à ce que l’agarose est dissoute et la solution est claire. Conserver la solution de gel d’agarose à 55-70 ° C et l’utiliser pour la construction de plusieurs coussins de gel d’agarose dans les 48 heures.
- Pipetter médias contenant de 1,2 à 1,5 % d’agarose dans le centre de la garniture.
Remarque : Le volume des médias est généralement 50-60 µL mais variera selon taille du joint. Ajout de média à une diapositive froide peut causer l’agarose à solidifier trop rapidement, donc la diapositive doit être maintenue sur une surface chaude. Au bord d’un bloc chauffant mis à 70 ° C fonctionne bien. Eau d’agarose ou d’agarose dans la mémoire tampon des solutions comme en solution saline tamponnée au phosphate (PBS) peuvent être utilisées au lieu de milieux contenant du gel d’agarose pour les applications dans lesquelles la croissance cellulaire continue n’est pas nécessaire. Pour les souches de l’appauvrissement de l’imagerie, inducteur peut être présents ou absents dans les médias. Présence d’inducteur peut être utilisée comme un contrôle alors que l’absence d’inducteur révélera le phénotype de l’appauvrissement de la couche. - Placez une lamelle sur le joint pour répartir l’agarose.
- Placez sur une surface plane, cool pour solidifier pour environ 2 min. Evitez surséchage du pad d’agarose comme cela se traduira par une surface ridée sur le coussinet de gel d’agarose et de mise en commun de la suspension cellulaire lorsqu’il est appliqué à la surface.
- Glisser délicatement la lamelle sur le coussinet de gel d’agarose.
ATTENTION : Ne vous précipitez pas à cette étape. Le coussin de gel d’agarose peut facilement déchirer et un tampon d’agarose inégale risquent de compliquer l’imagerie. - Laisser le tampon de gel d’agarose à l’air sec pendant 1 à 2 min à température ambiante jusqu'à ce que la surface du pavé tactile semble sec. À l’aide d’un scalpel, enlever une petite bande d’agarose pour créer une poche d’air (image du milieu, la Figure 1 a) ; la poche d’air est généralement ~ 2 x 7-10 mm et les cellules a. tumefaciens ont tendance à croître plus près de la poche d’air (Figure 1).
Remarque : Pour l’imagerie de cellules fixes ou dans des conditions où la croissance n’est pas surveillée, une poche d’air n’est pas nécessaire. - Spot 0,8-1 µL de cellules sur le coussinet de gel d’agarose et couvrir avec une lamelle de nouveau. Placez délicatement la lamelle sur le dessus du coussin d’agarose pour distribuer les cellules sur la surface du coussin d’agarose.
- Sceller les bords de la lamelle à l’aide de VALAP fondu (voir Table des matières pour la recette ; Figure 1 a, image du bas). Si pour sceller le long de tous les bords et les coins de la lamelle risque de dessèchement de la garniture de l’agarose, qui conduira à la dérive des cellules au cours de l’imagerie. Remarque : L’étanchéité de la lamelle n’est nécessaire pour l’imagerie de Time-lapse à long terme.
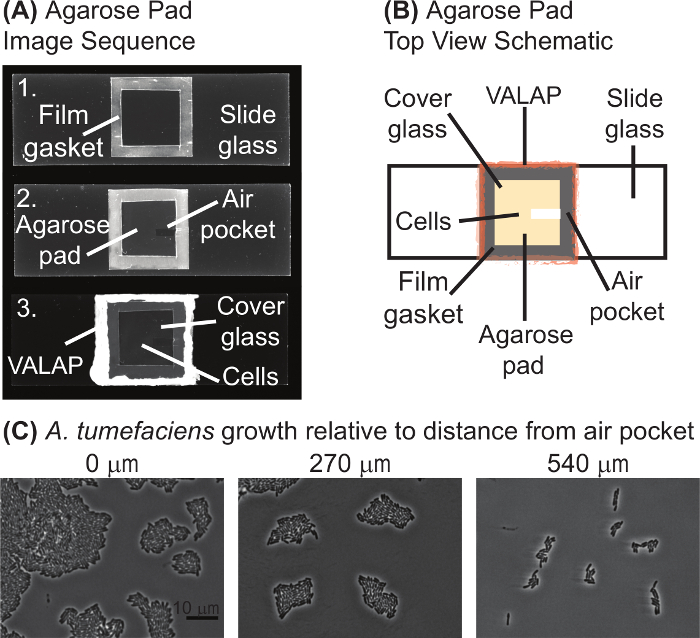
Figure 1 : préparation de coussinet de gel d’Agarose. (A) Image de séquence du protocole d’agarose pad préparation. Image 1 est une diapositive avec le joint d’étanchéité de laboratoire pour le film. Dans l’image 2, le coussin de gel d’agarose et la poche d’air sont visualisées. Enfin, image 3 montre le tampon d’agarose complète avec les cellules sous une lamelle et collés avec VALAP. (B), un schéma d’un coussinet de gel d’agarose pour la microscopie Time-lapse est fourni. Principales caractéristiques du coussin d’agarose sont indiquées sur le schéma. (C), la poche d’air a favorisé la croissance d’a. tumefaciens sur coussinets de gel d’agarose. Image de type sauvage a. tumefaciens cellules cultivées sur un coussin de gel d’agarose pour 20 heures sont affichées. Des images ont été prises à des postes de plus en plus éloignés de la poche d’air. La distance entre le bord le plus proche de l’image et la poche d’air est montrée au-dessus de chaque image. Echelle = 10 µm. s’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
-
Imagerie
NOTE : Le contraste interférentiel différentiel, contraste de phase et l’imagerie de fluorescence incidente est réalisée avec un microscope inversé équipé d’un stade automatisé, filtres standard, une source de lumière LED, mise au point automatique basée sur le matériel, immersion dans l’huile 60 X objectifs (1,4 NA) pour contraste de phase ou de contraste interférentiel différentiel (DIC) et une caméra de charge-coupled-device (EMCCD) électron-multipliant 1K. Le microscope doit être placé dans une pièce à température contrôlée. Alternativement, un stade plus chaud ou une chambre peut servir à maintenir des températures constantes au cours de l’imagerie.- Microscopie à épifluorescence
Remarque : Pour l’imagerie de fluorescence des cellules vivantes, il est nécessaire de limiter le nombre d’acquisitions d’image et d’optimiser le temps d’exposition afin de minimiser le photoblanchiment et phototoxicité. Pour l’imagerie de chaque colorant, il est recommandé de trouver un temps de pose optimale qui assure la détection par fluorescence suffisante mais n’entraîne pas de photoblanchiment ou phototoxicité. Dans les Figures 2-4, temps d’exposition de 200 ms ont été utilisés pour toutes les images de fluorescence. Pour les séquences de Time-lapse illustrés à la Figure 3, les images ont été acquises toutes les 10 min pendant 3 h.- Placer l’huile à immersion sur l’objectif recherché et placez le chariot inversé dans le support de diapositive sur la scène. Utilisez les boutons de mise au point pour intégrer les cellules de mise au point.
- Acquérir des images en phase (ou DIC) et le filtre de fluorescence désirée.
Remarque : Les maximales longueurs d’onde d’excitation et d’émission pour les taches utilisées dans la Figure 2, Figure 3et Figure 4 sont les suivants : HADA (405/460), NADA (450/555), TADA (555/570), 4-64 (515/640), DAPI (360/460), Orange (547/570).
- Time-lapse microscopie
Remarque : Pendant le Time-lapse microscopie les fonctionnalités suivantes sont contrôlés par l’ordinateur : x, y et z filtres de position, les volets et fluorescence. Un système autofocus matérielle est optimal pour maintenir l’accent au cours de l’imagerie Time-lapse. Par ailleurs, une boucle d’autofocus logicielle peut être utilisée.- Placer l’huile à immersion sur l’objectif recherché et placez le chariot inversé dans le support de diapositive sur la scène. Utilisez les boutons de mise au point pour intégrer les cellules de mise au point.
- En option : Acquérir multiples (x, y) postes. Choisir au hasard 10 champs de cellules à proximité de la poche d’air des plaquettes d’agarose.
- Mise en place l’acquisition de séquence de temps à l’image en phase ou DIC à l’intervalle de temps souhaité.
Remarque : Pour la séquence de Time-lapse illustrée à la Figure 4, DIC images ont été acquises avec une exposition de 30 ms toutes les 10 min pendant 14 h. Lors de l’utilisation d’imagerie de fluorescence au cours de la microscopie Time-lapse, régler le temps d’intervalle et l’exposition acquisition afin de minimiser le photoblanchiment et phototoxicité.
- Microscopie à épifluorescence
Résultats
Étiquetage du type sauvage spécifique à la cible A. tumefaciens cellules
Afin d’illustrer que la morphologie cellulaire n’est pas affecté par les étapes de lavage ou un traitement avec 1 % DMSO (qui sert à diluer les colorants fluorescents), les cellules ont été imagées directement à partir de culture (Figure 2 a, panneau de l’extrême gauche), après avoir lavé les cellules par ...
Discussion
Ce protocole contient une série de procédures d’enquête sur les souches de type sauvage, mutant et appauvrissement de la couche a. tumefaciens . Il est à noter que toutes les procédures répertoriées dans la section protocole peuvent être facilement adapté pour d’autres souches bactériennes avec modifications pour tenir compte des milieux de croissance, des températures et des taux de croissance.
L’utilisation des colorants spécifiques à la cible est un outil précie...
Déclarations de divulgation
Les auteurs n’ont rien à divulguer.
Remerciements
Nous remercions Michael VanNieuwenhze (Université de l’Indiana) pour le don des FDAAs utilisée dans la Figure 2 et Figure 4. Nous remercions les membres du laboratoire brun pour vos commentaires lors de la préparation de ce manuscrit. La recherche en laboratoire brun sur la division et la croissance des cellules a. tumefaciens est pris en charge par la National Science Foundation (IOS1557806).
matériels
| Name | Company | Catalog Number | Comments |
| Bacterial Strains | |||
| Agrobacterium tumefaciens C58 | ATCC | 33970 | Watson B, Currier TC, Gordon MP, Chilton MD, Nester EW. 1975. Plasmid required for virulence of Agrobacterium tumefaciens. J Bacteriol 123:255-264. |
| Agrobacterium tumefaciens C58ΔtetRA::mini-Tn7T-GM-Ptac-ctrA ΔctrA | Figueroa-Cuilan W, Daniel JJ, Howell M, Sulaiman A, Brown PJB. 2016. Mini-Tn7 insertion in an artificial attTn7 site enables depeltion of the essentail master regulator CtrA in the phytopathogen Agrobacterium tumefaciens. Appl Environ Microbiol. 82:5015-5025. | ||
| Name | Company | Catalog Number | Comments |
| Media Components | |||
| ATGN Minimal Medium | To 1 L of sterilized water add 50 ml 20X Buffer, 50 ml 20X Salts, 12.5 ml 40% glucose. For plates, add 15 g Bacto Agar to 1 L of water and autoclave. Cool to 55 °C and add 50 ml 20X Buffer, 50 ml 20X Salts, 12.5 ml 40% glucose. | ||
| 20X AT Buffer | Add 214 g/L KH2PO4 to water and adjust pH to 7.0 with sodium hydroxide. Autoclave. | ||
| NaOH | Fisher BioReagents | BP359 | |
| KH2PO4 | Fisher Chemical | P288 | |
| 20X AT Salts | Add 40 g/L (NH4)2SO4, 3.2 g/L MgSO4•7H2O, 0.2 g/L CaCl2•2H2O, and 0.024 g/L MnSO4•H2O to water. Autoclave. | ||
| (NH4)2SO4 | Fisher Chemical | A701 | |
| MgSO4•7H2O | Fisher BioReagents | BP213 | |
| CaCl2•2H2O | Fisher BioReagents | BP510 | |
| MnSO4•H2O | Fisher Chemical | M114 | |
| Glucose | Fisher Chemical | D16 | Prepare 40% stock in water. Filter sterilize. |
| Bacto Agar | Fisher BioReagents | BP1423 | Add 15 g to 1 L of water when preparing plates. |
| Name | Company | Catalog Number | Comments |
| Optional Media Additives | |||
| Kanamycin | GoldBio | K-120 | Prepare as a 100 mg/ml stock solution in water and filter sterilize. Use at final concentration of 200 µg/ml. |
| IPTG | GoldBio | I2481C5 | Prepare as a 1 M stock solution in water and filter sterilize. Use at final concentration of 1 mM as needed for induction. |
| Name | Company | Catalog Number | Comments |
| Microscopy Materials | |||
| Microscope Slides | Fisherbrand | 12-550D | 25 X 75 X 1.0 mm. Clean with Sparkle glass cleaner. |
| Microscope Cover Glass | Fisherbrand | 12-541-B | 22 X 22 mm. No. 1.5. Clean with Sparkle glass cleaner. |
| Sparkle Glass Cleaner | Home Depot | 203261385 | Ammonia and alcohol free. |
| Ultra Pure Agarose | Invitrogen | 16500-100 | Add to water, PBS, or media to a final concentration of 1 - 1.5%. Melt in microwave and place on 70 C |
| PBS | Fisher BioReagents | BP399500 | 10X solution to be diluted to 1X with sterile water. |
| Parafilm | Bemis | PM-999 | Laboratory film used as gasket in agarose pad preparation. |
| VALAP | Add equal weights of lanolin, parafin wax, and petroleum jelly to a conical tube. Heat tube in 70 °C dry, bead or water bath to melt and mix. Apply VALAP while still molten. | ||
| Lanolin Butter | SAAQIN | SQ-LAB-R1 | |
| Petroleum Jelly | Target Corp. | 06-17644 | |
| Paraffin Wax | Crafty Candles | 263012 | |
| Name | Company | Catalog Number | Comments |
| Target-specific dyes | |||
| DMSO | Fisher BioReagents | BP231-1 | Use to dilute stock solutions of dyes as needed. |
| FDAAs (NADA, HADA,TADA) | FDAAs can be synthesized or acquired through agreement with Mike VanNieuwenhze (Indiana University). Prepare 100 mM stock solution in DMSO. Use at a final concentration of 5 mM. | ||
| DAPI | ThermoFisher Scientific | 62247 | Prepare 1 mg/ml stock solution in DMSO. Use at final concentration of 1 µg/ml. |
| SYTOX Orange Nucleic Acid Stain | Invitrogen | S11368 | Stock concentration is 5 mM in DMSO. Use at final concentration of 5 µM. |
| FM4-64 | Invitrogen | T3166 | Prepare 8 mg/ml stock solution in DMSO. Use at final concentration of 8 µg/ml. |
| Name | Company | Catalog Number | Comments |
| Equipment | |||
| Dry bath | Sheldon Manufacturing, Inc. | 52120-200 | |
| Metallic thermal beads | Lab Armor | 42370-002 | |
| Epifluorescence microscope equipped with an EMCDD camera | Nikon Eclipse TiE equipped with a QImaging Rolera em-c2 1K electron-multiplying charge-coupled-device (EMCCD) camera is used in this work. |
Références
- Daniel, R. A., Errington, J. Control of cell morphogenesis in bacteria: two distinct ways to make a rod-shaped cell. Cell. 113 (6), 767-776 (2003).
- Tiyanont, K., et al. Imaging peptidoglycan biosynthesis in Bacillus subtilis with fluorescent antibiotics. Proc Natl Acad Sci U S A. 103 (29), 11033-11038 (2006).
- Turner, R. D., et al. Peptidoglycan architecture can specify division planes in Staphylococcus aureus. Nat Commun. 1, 26 (2010).
- Wheeler, R., Mesnage, S., Boneca, I. G., Hobbs, J. K., Foster, S. J. Super-resolution microscopy reveals cell wall dynamics and peptidoglycan architecture in ovococcal bacteria. Mol Microbiol. 82 (5), 1096-1109 (2011).
- Turner, R. D., Hurd, A. F., Cadby, A., Hobbs, J. K., Foster, S. J. Cell wall elongation mode in Gram-negative bacteria is determined by peptidoglycan architecture. Nat Commun. 4, 1496 (2013).
- Kuru, E., et al. In Situ probing of newly synthesized peptidoglycan in live bacteria with fluorescent D-amino acids. Angew Chem Int Ed Engl. 51 (50), 12519-12523 (2012).
- Siegrist, M. S., et al. (D)-Amino acid chemical reporters reveal peptidoglycan dynamics of an intracellular pathogen. ACS Chem Biol. 8 (3), 500-505 (2013).
- Kepner, R. L., Pratt, J. R. Use of fluorochromes for direct enumeration of total bacteria in environmental samples: past and present. Microbiol Rev. 58 (4), 603-615 (1994).
- Johnson, M. B., Criss, A. K. Fluorescence microscopy methods for determining the viability of bacteria in association with mammalian cells. J Vis Exp. (79), (2013).
- Bakshi, S., et al. Nonperturbative imaging of nucleoid morphology in live bacterial cells during an antimicrobial peptide attack. Appl Environ Microbiol. 80 (16), 4977-4986 (2014).
- Barak, I., Muchova, K. The role of lipid domains in bacterial cell processes. Int J Mol Sci. 14 (2), 4050-4065 (2013).
- Fishov, I., Woldringh, C. L. Visualization of membrane domains in Escherichia coli. Mol Microbiol. 32 (6), 1166-1172 (1999).
- Barak, I., Muchova, K., Wilkinson, A. J., O'Toole, P. J., Pavlendova, N. Lipid spirals in Bacillus subtilis and their role in cell division. Mol Microbiol. 68 (5), 1315-1327 (2008).
- Cameron, T. A., Anderson-Furgeson, J., Zupan, J. R., Zik, J. J., Zambryski, P. C. Peptidoglycan synthesis machinery in Agrobacterium tumefaciens during unipolar growth and cell division. MBio. 5 (3), e01219 (2014).
- Zupan, J. R., Cameron, T. A., Anderson-Furgeson, J., Zambryski, P. C. Dynamic FtsA and FtsZ localization and outer membrane alterations during polar growth and cell division in Agrobacterium tumefaciens. Proc Natl Acad Sci U S A. 110 (22), 9060-9065 (2013).
- Eberhardt, A., Wu, L. J., Errington, J., Vollmer, W., Veening, J. W. Cellular localization of choline-utilization proteins in Streptococcus pneumoniae using novel fluorescent reporter systems. Mol Microbiol. 74 (2), 395-408 (2009).
- Iniesta, A. A., Garcia-Heras, F., Abellon-Ruiz, J., Gallego-Garcia, A., Elias-Arnanz, M. Two systems for conditional gene expression in Myxococcus xanthus inducible by isopropyl-beta-D-thiogalactopyranoside or vanillate. J Bacteriol. 194 (21), 5875-5885 (2012).
- Figueroa-Cuilan, W., Daniel, J. J., Howell, M., Sulaiman, A., Brown, P. J. Mini-Tn7 Insertion in an Artificial attTn7 Site Enables Depletion of the Essential Master Regulator CtrA in the Phytopathogen Agrobacterium tumefaciens. Appl Environ Microbiol. 82 (16), 5015-5025 (2016).
- Jacob, F., Monod, J. Genetic regulatory mechanisms in the synthesis of proteins. J Mol Biol. 3, 318-356 (1961).
- Khan, S. R., Gaines, J., Roop, R. M., Farrand, S. K. Broad-host-range expression vectors with tightly regulated promoters and their use to examine the influence of TraR and TraM expression on Ti plasmid quorum sensing. Appl Environ Microbiol. 74 (16), 5053-5062 (2008).
- Yansura, D. G., Henner, D. J. Use of the Escherichia coli lac repressor and operator to control gene expression in Bacillus subtilis. Proc Natl Acad Sci U S A. 81 (2), 439-443 (1984).
- Guzman, L. M., Belin, D., Carson, M. J., Beckwith, J. Tight regulation, modulation, and high-level expression by vectors containing the arabinose PBAD promoter. J Bacteriol. 177 (14), 4121-4130 (1995).
- Thanbichler, M., Iniesta, A. A., Shapiro, L. A comprehensive set of plasmids for vanillate- and xylose-inducible gene expression in Caulobacter crescentus. Nucleic Acids Res. 35 (20), e137 (2007).
- Topp, S., et al. Synthetic riboswitches that induce gene expression in diverse bacterial species. Appl Environ Microbiol. 76 (23), 7881-7884 (2010).
- Peters, J. M., et al. A Comprehensive, CRISPR-based Functional Analysis of Essential Genes in Bacteria. Cell. 165 (6), 1493-1506 (2016).
- Qi, L. S., et al. Repurposing CRISPR as an RNA-guided platform for sequence-specific control of gene expression. Cell. 152 (5), 1173-1183 (2013).
- Griffith, K. L., Grossman, A. D. Inducible protein degradation in Bacillus subtilis using heterologous peptide tags and adaptor proteins to target substrates to the protease ClpXP. Mol Microbiol. 70 (4), 1012-1025 (2008).
- McGinness, K. E., Baker, T. A., Sauer, R. T. Engineering controllable protein degradation. Mol Cell. 22 (5), 701-707 (2006).
- Schneider, J. P., Basler, M. Shedding light on biology of bacterial cells. Philos Trans R Soc Lond B Biol Sci. 371 (1707), (2016).
- Escobar, M. A., Dandekar, A. M. Agrobacterium tumefaciens as an agent of disease. Trends Plant Sci. 8 (8), 380-386 (2003).
- Gelvin, S. B. Agrobacterium-mediated plant transformation: the biology behind the "gene-jockeying" tool. Microbiol Mol Biol Rev. 67 (1), 16-37 (2003).
- Nester, E. W. Agrobacterium: nature's genetic engineer. Front Plant Sci. 5, 730 (2014).
- Imam, J., Singh, P. K., Shukla, P. Plant Microbe Interactions in Post Genomic Era: Perspectives and Applications. Front Microbiol. 7, 1488 (2016).
- Subramoni, S., Nathoo, N., Klimov, E., Yuan, Z. C. Agrobacterium tumefaciens responses to plant-derived signaling molecules. Front Plant Sci. 5, 322 (2014).
- Yuan, Z. C., Williams, M. A really useful pathogen, Agrobacterium tumefaciens. Plant Cell. 24 (10), (2012).
- Alvarez-Martinez, C. E., Christie, P. J. Biological diversity of prokaryotic type IV secretion systems. Microbiol Mol Biol Rev. 73 (4), 775-808 (2009).
- Pitzschke, A. Agrobacterium infection and plant defense-transformation success hangs by a thread. Front Plant Sci. 4, 519 (2013).
- Kuru, E., Tekkam, S., Hall, E., Brun, Y. V., Van Nieuwenhze, M. S. Synthesis of fluorescent D-amino acids and their use for probing peptidoglycan synthesis and bacterial growth in situ. Nat Protoc. 10 (1), 33-52 (2015).
- Curtis, P. D., Brun, Y. V. Identification of essential alphaproteobacterial genes reveals operational variability in conserved developmental and cell cycle systems. Mol Microbiol. 93 (4), 713-735 (2014).
- Kim, J., Heindl, J. E., Fuqua, C. Coordination of division and development influences complex multicellular behavior in Agrobacterium tumefaciens. PLoS One. 8 (2), e56682 (2013).
- Jong, I. G., Beilharz, K., Kuipers, O. P., Veening, J. W. Live Cell Imaging of Bacillus subtilis and Streptococcus pneumoniae using Automated Time-lapse Microscopy. J Vis Exp. (53), (2011).
- Turnbull, L., et al. Super-resolution imaging of the cytokinetic Z ring in live bacteria using fast 3D-structured illumination microscopy (f3D-SIM). J Vis Exp. (91), e51469 (2014).
- Zeng, L., Golding, I. Following cell-fate in E. coli after infection by phage lambda. J Vis Exp. (56), e3363 (2011).
- Brown, P. J., et al. Polar growth in the Alphaproteobacterial order Rhizobiales. Proc Natl Acad Sci U S A. 109 (5), 1697-1701 (2012).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon