Method Article
タンパク質とリボ核蛋白質複合体の 3 D ページのナタネから師部 Sap サンプリング
要約
大規模なネイティブ蛋白質: 蛋白質および蛋白質の蛋白質組成を分析するためのプロトコルの紹介: ナタネから核酸複合体 (ナタネ)ブルーを組み合わせて 3 D ポリアクリルアミドゲル電気泳動 (ページ) アプローチを使用して篩部の滲出液ネイティブ (BN) 質量分析による同定が続く変化の 2 つのページを持つ。
要約
高等植物の篩管液をサンプリング、しばしば骨の折れると植物種に大きく依存。ただし、別の植物種の変性条件下のプロテオーム研究を達成できます。ネイティブ蛋白質: 蛋白質および蛋白質: 師部のサンプルから核酸複合体、まだやっとのことで分析されているこの特殊な区画の維持または長距離シグナル伝達で重要な役割を果たしている可能性が。大きな分子は、青ネイティブ ゲル電気泳動法 (BN] ページ) を使用して分離できます。その後ナトリウム ドデシル硫酸ページ (SDS-PAGE) による、タンパク質成分を分離できます。ただし、同じような分子量が付いている蛋白質共同移行、質量分析法によるタンパク質の同定を妨げることができるか。BN-PAGE-を組み合わせて 2 つ異なる変性ゲル電気泳動手順、すなわちトリス トリシン尿素と SDS ページの親水性/疎水性に従ってタンパク質を追加分離できます、解像度と成功を増やしますタンパク質同定。それも違いだけで、翻訳後修飾タンパク質を区別することができます。さらに、高分子複合体の RNA コンポーネントを識別するには、青いネイティブ北しみが付くことを適用できます。我々 のプロトコルが蛋白質およびネイティブ蛋白質: 蛋白質および師部のサンプルで発生するリボ核タンパク質 (RNP) 複合体の RNA 成分の解明に適していることを示します。青を組み合わせること 2 つの異なる変化ページ手順でネイティブ ページは異なった種類の大きい蛋白質の複合体を分離することができ、質量分析法による部品の高められた識別率ができます。さらに、プロトコルは同時に 1 つの蛋白質の複合体の内で潜在的にバインドされている核酸を検出する十分な堅牢です。
概要
師部の長距離導管高等植物の有機性栄養素の配分に欠かせないが成長と発展、病原体の防御と悪影響への適応の重要な多くの異なるプロセスに関与するも環境条件。イオン、植物ホルモン、または自分自身の代謝物のような小さなエフェクタは、ほかのタンパク質と Rna のような高分子最近潜在的な信号として浮上しています。
研究は、蛋白質の師部読み込みがサイズ依存と伴細胞 (CC) の接続孔 plasmodesmal 単位のサイズ排除限界 (SEL) で制御したことを示されているふるいの要素 (SE) に 10 の範囲に通常ある、> 67 kDa の1,2,3しかし、にもかかわらずこれらのサイズ制限より大きい蛋白質および蛋白質の複合体は、サイズ排除4のアイデアに挑戦師部システム内発見されている、推奨5 、CC からタンパク質が SE にも非特異的失われて。.
物質として大きなリボ核蛋白質複合体は、生合成・ セル6の構造の整合性の維持に極めて重要な役割を再生します。4、7を師部携帯タンパク質とリボ核蛋白質複合体に存在する証拠があるが、その機能についてはほとんど知られている日付に。篩ふるいの要素: タンパク質と RNP 複合体が関与している長距離輸送および/または信号8,9。その組成変動を受ける環境を勉強して転体の機能の解明や、ストレス条件が必要です。そのためには、ネイティブ複合体を分離する必要があり、そのコンポーネントは、十分な解像度と分離しなければなりません。
師部から高分子量複合体を分離、十分な質と量の sap サンプルを取得するの先決です。師部をサンプリングするために使用できる手法は、興味の植物種に依存します。師管液を取得する 3 つの主な方法存在10: 昆虫 stylectomy11、EDTA 促進溶出12、および自発的な滲出物13。
シロイヌナズナ(a)、研究所、世界中で主要なモデル プラントからの師部のサンプルは、EDTA 促進溶出やアブラムシ stylectomy14,15でのみ取得できます。両方のメソッドは、非常に希釈師部 sap または非常に低いサンプル量に します。EDTA 促進溶出も汚染しやすい、実際師部組成16を表さないことがあります。自発師部滲出物は、植物の部分を切断は簡単に収集した13,17,18をすることができます師部樹液の自然放出につながる、ウリ、ルピナス、ユッカなど植物に限定 19。我々 は最近篩分析のための適切なモデル システムとしてナタネ (ナタネ) を設立、シロイヌナズナの近親と師管液のいくつかマイクロリットルを示されている小切開から取得できますので高純度4,8,20であります。さらに、ナタネのゲノム塩基配列は 2014年21にリリースされました。
アブラムシの stylectomy を除いて説明すべての師部コレクション方法は周囲のサンプリング、現場組織の損傷を含めるし、師管液の組成は、損傷に応答変更可能性があります。したがってサンプリング法に関係なく、師部サンプルの品質をチェックする不可欠です。例えばRNase 活動22,23Rubisco8,24プライマーと PCR または砂糖の分析による決定により収集された師部 sap の品質管理のさまざまな方法があります。組成8。
サイズ排除クロマトグラフィー、ショ糖密度遠心明確な分子量カット膜によるサンプルろ過などを分離し、タンパク質複合体を分離するさまざまな方法を適用できるトレードオフです。ただし、これらのアプローチは高解像度を許さない。最初 Schäggerらによって記述された青ネイティブ ページ (BN-PAGE) と呼ばれる手法。25日は解像度の面で優れています。それは正常に様々 な細胞小器官や細胞可溶化物の相対的な存在量26,27から大規模なネイティブ タンパク質の分子量を決定するため使用されました。
さらにタンパク質の複雑な組成の解明、複雑な部品の完全な分解につながる条件の変化の下で個々 のコンポーネントを分離する必要は。これは SDS ページ28,29またはより高い解像度を実現、等電集中 (IEF) の 2 番目の次元で続いて SDS ページ30の third dimension との識別をその後に 2 番目の次元によって達成することができます。質量分析法を用いたタンパク質。
このプロトコルでは、ナタネ植物から純粋な師部樹液の採取・ BN-PAGE-トリス-トリシン-尿素-ページおよび SDS のページを組み合わせて 3 D 電気泳動のアプローチを使用してこのような師部サンプルからタンパク質複合体の分析について説明します。複雑なコンポーネントは、質量分析法による識別されますその後分離されたタンパク質。さらに、紹介するブルー ネイティブ北しみが付くこと大きい RNP 複合体4Rna の検出を許可する方法としてアプローチの適用性を拡張します。
プロトコル
1. サンプリングやナタネから師管液の準備植物4,8,20
- サンプリング、師部樹液植えた 8 週齢を使用してナタネ植物花粉の汚染を防ぐためにそのショーの唯一の初期開花。
- 注射針を使用して (Ø 0.8 mm) および点状花房茎数回。いくつかの他の花房の茎と十分な師部 sap (図 2 a) を取得する他の植物を繰り返します。
メモ: 複素解析の勧め師部樹液の少なくとも 600 μ L で開始します。4 に 5 の植物師管液の 200 と 700 μ L の間になります。 - ろ紙を負傷したサイトから最初のドロップをふき取りなさい。これらの低下は主に負傷した組織の周囲から高汚染物質を含むし、破棄する必要があります。
- ピペッティングで次の滴を収集し、凍らない冷却システムを用いた-20 ° C であらかじめ冷やして円錐 1.5 mL の反応管内の収集した滲出液を格納します。
- さらにドロップ形成される観察可能なしかし、もはや 1 時間以上まで収集を継続します。収集した師管液の重要な損失することがなく 2 日後、この手順を繰り返すことができます。
注: 収集した師管液することができますすぐに使用または将来の分析のための-80 ° C で保存します。-80 ° C のストレージの衝撃-凍結反応チューブ液体窒素で。 - 分子量 10,000 ダルトン (Da) のカット (MWCO) を用いた遠心コンセントレーター最初のボリュームの約 1/6thに師部サンプルを集中します。4 ° C とダスト粒子を除去する、少なくとも 15 分間 20,000 × g の集中のサンプルを遠心します。
注: 師部 sap 濃度は蛋白質の低下にはつながらない。 - その後の複雑な分析手順 3.1 に進みます。
2. 師部 sap 純度制御逆転写 PCR (RT-PCR) 4,8,20を使用して
- 師管液液体材料の標準的な RNA 抽出法を使用してからの総 RNA の隔離 (例えばTRIzol またはフェノール-クロロホルム抽出に続いた水相洗浄手順によるとエタノールからイソプロパノールの沈殿物によって、製造元のプロトコル)。
注意: フェノール クロロホルムは有毒です。適切な個人的な保護装置のフードの下で動作します。6 ヶ月間-80 ° c のエタノールで抽出した RNA を保存できます。 - DNA の DNase と消化による汚染を削除私は 37 ° C で 45 分間 (1 u/μ g) EDTA を最終濃度 5 mM に追加することによって、酵素を不活性化し、75 ° C 10 分でサンプルをインキュベートします。
- 純粋な RNA 浄化し、もう一歩浄化 (例えばRNA のクリーンアップ キット、製造元の指示に従ってを使用して) このサンプルを集中します。
- 逆転写 PCR (RT-PCR) を共演 150 を使用して cDNA 合成キット cDNA の合成純粋な RNA の ng はメーカーで指定されているプロトコル 's。
注: cDNA は-20 ° C でさらに使用するまで格納できます。 - コンパートメント特定の成績証明書を増幅し、アガロース電気泳動でチェックにテンプレートとしての製造元によって指定された標準 PCR の反作用のための cDNA の 5 μ L を使用します。例えば rubisco 小サブユニット、チオレドキシン h、および花粉外被蛋白質 (表 1図 1 b) のプライマーを使用して篩部試料の純度を確認してください。
3. 青ネイティブ ページ4,25,26,31
- 27または商業 Bis トリス グラデーション ジェルに従って別の場所でどちらかの自己注がれたネイティブ勾配ゲルを使用します。
- 負荷のゲルのウェルあたり高濃度タンパク質サンプルの 20 に 30 μ g を少なくともトリプリケートします。
- 4 ° C および 150 V でゲルを実行サンプル (約20 〜 30 分、図 3 a) ゲルの 1/3rdを渡されるまで、暗い青カソード バッファー B と陽極バッファー (表 2) を使用しています。
注: 北しみが付くこと、単一ゲル レーンで十分中、3 D ページおよびより少なく豊富な蛋白質を集中する最大 10 個のゲル レーンから同じバンドを組み合わせることをお勧めは質量分析によるタンパク質組成分析のためです。 - 光青カソードと交換暗い青陰極バッファー B B/10 (表 2) をバッファーの実行や、実行を一時停止します。ゲル (ca. 120 に 180 分、図 3 a) ゲルから溶出するブルーのフロントの起動時に実行を終了します。
注: 完成した青ネイティブ ゲルは 4 ° C で少なくとも 1 週間保存できます。長期の保管はバンドをびまん性につながることができ、避けるべきであります。暗い青バッファー B は再利用できます。
4. ブルー ネイティブ北しみが付く4を使用して RNA を検出するオプション:
- 明瞭なバンドをカット、ネイティブ ブルージェルから全体のゲルのレーンを使用または半乾燥する 60 分の 3.5 mA/cm2でしみが付くことによってナイロン膜にそれらを転送し、膜 (図 4 a) に鉛筆で上限と下限のゲルの境界線をマークします。
- ブロッティング後 DEPC 処理水の膜を洗浄し、2 つのフィルター ペーパーの乾燥します。
- 120,000 μ J/cm2でブロッティング膜の紫外架橋を実行 (85 sテーブルの材料で、架橋剤を使用している場合)。
- 前ハイブリダイゼーション オーブン (図 4 a) で 68 ° C で 60 分間交配バッファー (市販または自作) の 6 mL で膜を交配させます。
- 3'-ビオチン化プローブの 4 μ L を含む交配バッファーの追加 1 mL を追加 (100 nM)。
- 37 ° c. に 68 の ° C からオーブンを冷却しながら一晩、プローブと膜を孵化します。
- 不特定にバインドされているプローブを削除する部屋の温度で 5 分間、2 x SSC と 0.1 %sds を格納するバッファーで膜を洗浄します。
注意: SDS は、炎症を引き起こす可能性が。SDS とソリューションを含む SDS を使用するときは、手袋、白衣を着用します。 - 西洋わさびペルオキシダーゼ (HRP) 基板、高感度化学発光反応 (図 4 a) の結果として、膜などストレプトアビジン-HRP-共役とルミノールの 3'-ビオチン化プローブの検出を実行します。
5. 2 番目の寸法: トリス トリシン尿素ページ4,28
- 青ネイティブ ゲルから単一のバンドを切除します。さらに複雑なタンパク質 (図 5) 濃度を達成するために車線を並列で実行サンプルからバンドを結合します。
注: 摘出バンドに格納できる単一反応管 4 ° C でさらに使用するまで - 12% トリス トリシン尿素ゲルを注ぎなさい (厚さ 1 mm、6.8 × 8.6 cm (幅 × 長さ))。(表 2)、分離ゲルのゲル溶液の 10 mL を準備 2 つのガラス板とイソプロパノールとオーバーレイの間注ぐ。重合終了後、イソプロパノールを取り外し、ゲルと、10 もくし挿入にスタッキングのゲルのゲル液 B (表 2) の 4 つの mL。
注意: 未重合アクリルアミドは神経毒性することができます、TEMED 吸入すると有害。常に個人保護具を着用し、フードの下で働きます。 - 1.5 mL の反応管に摘出したゲルのバンドを転送し、ゲル部分 (図 5表 2) 平衡の SDS サンプルバッファー x 2 の 1 mL を追加します。
- 暖房のブロック (95 ° C) または電子レンジで沸騰する前に、室温で 10 分間のサンプルをインキュベートし、さらに 15 分の室温で再度サンプルをインキュベートします。
- 1 つの単一のゲルのポケットに同じ建物を表すいくつかの平衡ゲル部分をスタックします。
- アノードとカソード 45 ~ 60 分の 150 V でバッファーを実行するを使用して電気泳動を行い、全体のゲル レーンを消費税します。
- 酸性バッファー (100 mM 100 mM 酢酸トリス) で 20 分間カット レーンをインキュベートし、125 mm トリス-HCl、別の 20 分 (図 5) の 6.8 pH 平衡します。
6. 3 次元: SDS ページ4,32
- 塩化物イオンと32によるとゲルの分離 15 %sds を注ぐ (厚さ: 1.5 ミリメートル、6.8 × 8.6 cm (幅 × 長さ)) (表 2)、ゲル上のスタック 4% の薄い層を追加。
注意: SDS、アクリルアミド、TEMED が有毒、有害であります。フードの下で働くし、個人用保護具を着用します。 - SDS ゲルの平衡ゲル レーンを転送し、ブロモフェノール ブルー (図 5) の微量を添加したバッファーを実行する 1 x SDS の 0.5% (w/w) agarose のゲルをカバーします。
- 電子レンジでの agarose が付いてのバッファーをゲルにロード前に実行中の SDS を沸騰させます。
- 単一成分の分子量を推定するためのタンパク質マーカーを実行、agarose が固化するまで一枚のゲルの 1 つの端にフィルター紙を挿入または通常 IPG ゲルに使用されます特殊な櫛を使用します。
- 150 V 60 分と汚れでゲルを実行コロイド Coomassie、銀染色のいずれか、またはその他のゲル染色法以降の質量分析に適した。
- 分析のようなマトリックス支援レーザー脱離イオン化質量分析法によるアプローチを使用して目に見える蛋白スポット飛行時間 (MALDI-TOF) やクロマトグラフィー-タンデム質量質量分析法。
注: 染色としてすでに記載されている33コロイド Coomassie を使用する勧めします。私たちの手でより豊富な蛋白質十分に染色することができますを使用することができ、合理的なデータ品質の質量分析。
結果
ここで提案する蛋白質: 蛋白質の解析を可能にするプロトコルナタネから師部サンプルの蛋白質: RNA の複合体と同様。ワークフローを図 1に示します。他のモデル生物でナタネの主要な利点の 1 つは、花房内の茎に開けた小さな穴から比較的簡単に師部サンプル コレクションの可能性です。これは比較的大量に篩部の純粋なサンプルを得ることができます。師部をサンプリングするときは常に、RT-PCR 法8たとえば、汚染をチェックすることをお勧めです。純粋な師部サンプルから得られる代表的な結果は、図 2で描かれています。Rubisco と花粉外被蛋白質のための信号が表示されないように、非汚染師部サンプル チオレドキシン h の表示バンドが表示されます。Rubisco の小サブユニットは、葉からのサンプル内のコントロールとして使用できる花房茎や花粉。チオレドキシン h は、花粉のコート蛋白質のトラン スクリプトが花芽のサンプルに表示されますのみ、その mRNA は異なる種の篩部で発見されているのですべてのサンプルで検出する必要があります。汚染は、完全に開花植物 (花粉外被蛋白質)、または穿刺のサンプリング師部から最初の滴が正しく破棄されないときに師部サンプリングが実行されるときに発生する可能性が (rubisco)。
BN-PAGE - 前師部樹液を集中するために必須です。蛋白質の 20 に 30 μ g を使用すると、よくあたり、低すぎる、ナタネ師部樹液の BN ページの後、少なくとも 4 つの主要な蛋白質複雑なバンドを見えるようになるまたは高すぎる濃度の錯体 (図 3) を明確に分離できないように。さらに下流の処理と後続の質量分析による同定の各単一の複合体から蛋白質を集中する 2 番目の次元ゲルの 1 つのシングル ポケットに同じ建物を表すいくつかのゲル バンドをロードすることをお勧めします。
篩部高分子複合体の個々 のコンポーネントを識別するためには、2 番目の次元トリス トリシン尿素の初期 BN-PAGE の併用し、3 次元 SDS-PAGE 単一蛋白質の分離を達成するために。ここでは、高解像度と分離は尿素と SDS ページのゲルの異なった蛋白質の移行挙動により観察できます。通常、単一の複合体に属する蛋白質はゲルの上で斜めの線として表示されます。蛋白質この行の上の行の下の蛋白質を表すより多くの親水性のもの28 (図 5図 6) に対し、増加の疎水性があります。
RNP 複合体のキャラクタリゼーション、BN 北しみが付くことを実行するのに BN-PAGE - ゲルの一部を使用しました。紫外線によるナイロン膜および架橋に全レーンのしみが付くこと後は、図 4に示すように特定の RNA プローブを用いた RNA を視覚化できます。ここで特定の tRNA がのみ 1 つの特定の複合体に存在することを示します。この tRNA 結合複合体のタンパク質成分は、上記 3 D ゲルのアプローチを使用してさらに調べることができます。質量分析は、BN 北しみが付くことによって検出された tRNA の結合活性を確認どのような主に tRNA のリガーゼをこの複合体で含まれていることを示した。
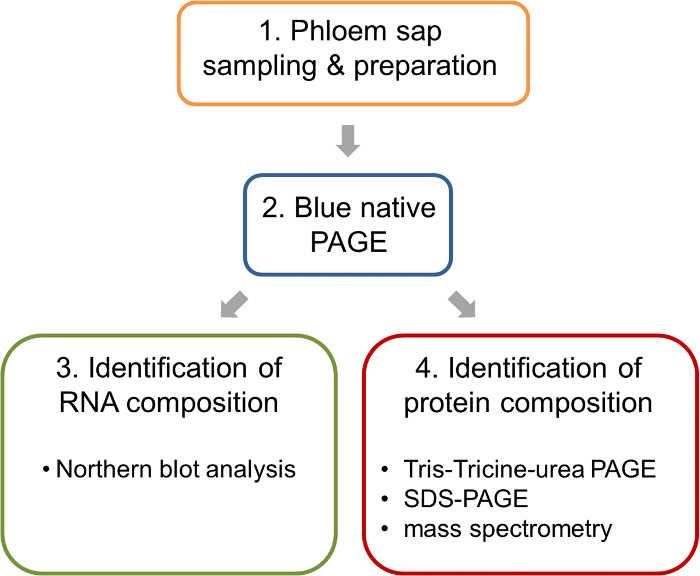
図 1: ナタネの師部樹液でリボ核蛋白質複合体の解析の流れを作業します。サンプリングと篩部試料の調製、ネイティブの複合体を分離する BN ページが実行されます。このような BN-PAGE - ゲルは、BN 北しみが付くことによる核酸の平行分析質量分析法による複合体のタンパク質成分の同定を許可します。この図の拡大版を表示するのにはここをクリックしてください。

図 2:ナタネの師部樹液のサンプリングと純度制御します。滅菌注射針と 8-10 週古い油糧種子植物の花茎を穿刺後滲出液をピペットで収集され、冷たい反応管 (a) RT-PCR 法を実行すると、篩部試料の純度を確認するために転送チオレドキシン h、rubisco の小サブユニット、および組織に固有のサンプル (b) RT-PCR 逆転写酵素なしで花粉外被蛋白質の転写産物を増幅制御 (-) として行われました。この図の拡大版を表示するのにはここをクリックしてください。
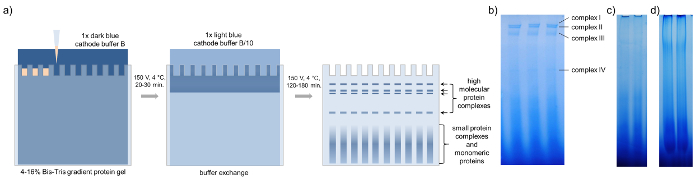
図 3: BN ページ。青ネイティブ ページの模式図の集中師部サンプルとナタネ。(a) 集中師部樹液の 15-20 μ L でグラデーション蛋白質ゲルをロードした後 BN ページは冷蔵室で 20-30 分の 150 V で 1 x 暗い青陰極バッファー B で実行されます。1 x 光青陰極バッファー B/10 に対してバッファーを交換し、ページはゲルからフロントの実行を実行している濃い青まで 120-180 分 150 V で終了。BN-PAGE - ゲルは、4 つの異なる高分子量複合体を表す 4 つの明瞭なバンドを示します。(b) ロードも少し (c) またはあまりにも多く (d) 師部蛋白質個々 の複合体の可視性を低減します。この図の拡大版を表示するのにはここをクリックしてください。

図 4: BN 北しみが付くこと.北部メソッドをしみが付くことの BN の模式図。(a) 億ページのレーンは、半乾燥しみが付くことによってナイロン膜に転送されます。UV 架橋前の交配後、膜は一晩ハイブリダイゼーション オーブンの 3' 端にビオチン標識である RNA 特定プローブ (ここメチオニン tRNA 固有プローブ) と培養です。無料プローブが手順を洗浄することにより削除され、ストレプトアビジン-HRP 共役とルミノールによる RNA の検出を行います。このアプローチを使用すると、BN ページから複雑な IV にメチオニンが含まれていることがわかった tRNA。(b) Coomassie 染色ゲルと tRNA のしみ 2 別のゲル車線移行の違いを避けるために並列で実行に似ています。この図の拡大版を表示するのにはここをクリックしてください。
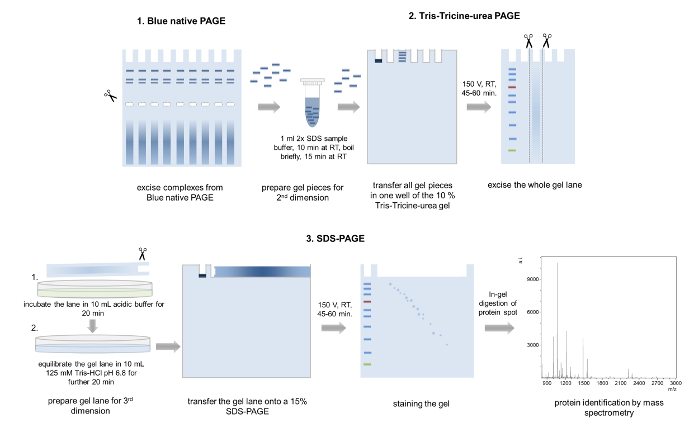
図 5: 3 D ページ。3 D ページ アプローチの模式図。同じコンプレックスからいくつかのバンドは、BN-PAGE - ゲルから摘出した、バッファーの読み込みに、トリス トリシン尿素ページの 2 番目の次元の一つのウェルに転送。電気泳動後全体のゲルのレーンは、酸性酸性バッファーで洗浄、平衡、切り取って 15 %sds ページのゲルの上に配置。第 3 の次元ページ分けられた蛋白質が Coomassie ステンド グラスし、質量分析法による分析です。この図の拡大版を表示するのにはここをクリックしてください。
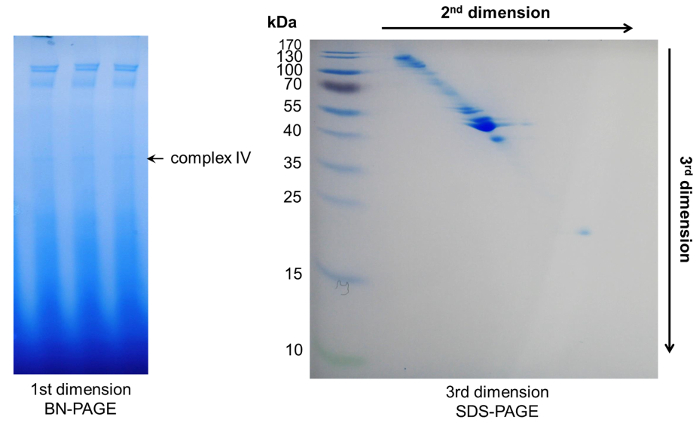
図 6: 3 D ページの後結果を代表します。BN-PAGE-の最初のディメンション内でいくつかの複合体が登場しました。彼らのタンパク質成分さらにスポットの対角に 2 つ以降変化のページに分割されます。この図の拡大版を表示するのにはここをクリックしてください。
| プローブ | シーケンス |
| チオレドキシン h |
フォワード: CTCAAGGCAGCCAAAGAATC 逆: ATGGCCTGAACATCGAACTC |
| ルビスコ小鎖 |
フォワード: TTCACCGGCTTGAAGTCATC 逆: CCGGGTACTCCTTCTTGCAT |
| 花粉外被蛋白質 BRU77666 |
フォワード: TCAGAACTGGAGCTTCAACG 逆: TTCCTTAATGGCCTCAGTGG |
表 1: プライマー師部サンプル純度制御に使用します。師部サンプル純度を確認するには、cDNA 増幅のルビスコ小鎖、チオレドキシン h、および花粉外被蛋白質のために特定のプライマーを使用できます。
| バッファーおよび解決 | コンテンツ | コメント | |
| 暗い青陰極バッファー B | 15 mM ビス トリス pH 7.0 50 mM トリシン 0.02% (w/v) Coomassie 青い G250 | Fiala ら17からレシピを適応します。 7.0 に pH を調整する必要があります。 バッファーは、10 x の在庫として作ることができます。 | |
| 光青陰極バッファー B/10 | 15 mM ビス トリス pH 7.0 50 mM トリシン 0.002% (w/v) Coomassie 青い G250 | バッファーは陰極バッファー B Coomassie なし陰極バッファーで希釈することによって準備することができます。 | |
| 陽極バッファー |
50 mM ビス トリス pH 7.0 | X 貯蔵液 10 としてバッファーを準備すること | |
| SDS のサンプルバッファー x 2 |
125 mM トリス塩酸 pH 6.8 4% SDS 20% グリセロール 0.2 M の DTT 0.02% (w/v) ブロモフェノール ブルー | ||
| 転送バッファー 1 x TBE |
89 mM Tris pH 7.6 89 mM ホウ酸 2 ミリメートルの EDTA | TBE バッファーを作った、5 倍または 10 倍の株として格納できます。RNase フリー脱イオン水することが重要です。 | |
| SSC バッファー x 2 |
300 mM の NaCl 30 mM Na3クエン酸 pH 7.0 | ||
| ゲルのバッファー x 3 |
3 M トリス 1 M HCl 0.3% SDS pH 8.45 | レシピ Schägger の19からの適応 | |
| トリス トリシン ゲル液 A |
10 ml: 30% アクリルアミド-bisacrylamide (29:1) 3.34 mL 6 M 尿素 3 x ゲル バッファー 3.34 mL 70% のグリセロール 1.4 mL ddH2O 10 mL を追加します。 10% APS 40 Μ L TEMED 4 Μ L | 6 M 尿素を減量し、ゲル バッファー、アクリルアミド bisacrylamide ソリューションおよび 70 %gycerol × 3 を追加します。尿素を可溶化するために湯せんで 60 ° C に熱する。DdH2O 10 mL, 10% を追加 AP そして重合 TEMED。 | |
| トリス トリシン ゲル溶液 B |
6 ml: 30% アクリルアミド-bisacrylamide (29:1) 0.8 mL 6 M 尿素 3 x ゲル バッファー 1.5 mL ddH2O 6 mL を追加します。 10% APS 45 Μ L TEMED 4.5 Μ L | 6 M 尿素を減量し、ゲル バッファーとアクリルアミド bisacrylamide 溶液 × 3 を追加します。尿素を可溶化するために湯せんで 60 ° C に熱する。DdH2O 10 mL, 10% を追加 AP そして重合 TEMED。 | |
| トリス トリシン実行バッファー (アノード) x 1 |
100 mM トリス 22.5 mM 塩酸 pH 8.9 | レシピ Schägger の19からの適応 | |
| トリス トリシン実行バッファー (カソード) x 1 |
100 mM トリス 100 mM トリシン 1% SDS pH 8.25 | レシピ Schägger の19からの適応 | |
| 酸性バッファー |
100 mM トリス 100 mM 酢酸 | ||
| 4 %sds スタッキングのゲル |
2 ml: ddH2O 1.4 mL 30% アクリルアミド-bisacrylamide (37.5:1) 0.33 mL 1 M トリス pH 6.8 0.25 mL 10% SDS 20 Μ L 10% APS 20 Μ L TEMED 2 Μ L | ||
| 15 %sds のゲルの分離 |
10 ml: ddH2O 2.3 mL 30% アクリルアミド-bisacrylamide (37.5:1) 5 mL 1.5 M トリス pH 8.8 2.5 mL 10% SDS 100 Μ L 10% APS 100 Μ L TEMED 4 Μ L | ||
| SDS 実行バッファー x 1 |
25 mM トリス 192 mM グリシン 0.1% SDS | ||
| Coomassie 染色液 |
5% (w/v) 硫酸アルミニウム-(14-18) 水和物 10% (v/v) エタノール (96%) オルトリン酸 2% (v/v) (85%) 0.02% (w/v) Coomassie ブリリアント ブルー G-250 | ||
| ULTRAhyb 超高感度交配バッファー | Ambion、生命技術 | ||
表 2: ソリューションおよびバッファー調理法。すべてのバッファーと 3 D ページの分析に必要なソリューションのリストです。
| スポットはありません。 | MW 観測 [kDa] | 識別 | 生物 | いいえを受入します。 | MW [kDa] | マスコットのスコア | |
| CIV_1 | 130 | 推定上病気耐性蛋白質 At4g19050 | ナタネ | CDX78917 | 131.1 | 110 | |
| CIV_2 | 130 | 推定上病気耐性蛋白質 At4g19050 | ナタネ | CDX78917 | 131.1 | 89 | |
| CIV_3 | 100 | 熱衝撃 70 kDa タンパク質 14 のような | ナタネ | XP_013748865 | 89.9 | 113 | |
| CIV_4 | 90 | 真核伸長因子 2 細胞分裂制御タンパク質 48 相同物 A | ナタネ ナタネ | CDX90241 CDY41316.1 | 89.5 89.5 | 111 197 | |
| CIV_5 | 85 | 熱ショック蛋白質 90 2 様 | B. rapa | XP_009132342 | 79.8 | 172 | |
| CIV_6 | 80 | スレオニン - tRNA のリガーゼ グリシン - tRNA のリガーゼ | B. oleracea ナタネ | XP_013599115 CDY43195 | 81.5 80.8 | 110 88 | |
| CIV_7 | 70 | 熱衝撃蛋白質 70 kDa リジン - tRNA のリガーゼのような | B. oleracea ナタネ | XP_013685267 XP_013747530 | 67.8 69.9 | 230 150 | |
| CIV_8 | 65 | ミロシナーゼ アスパラギン酸 - tRNA のリガーゼ 2 アスパラギン - tRNA のリガーゼ 1 | ナタネ ナタネ ナタネ | ABQ42337 XP_013670803 XP_013729126 | 60.6 61.6 63.5 | 183 141 153 | |
| CIV_9 | 60 | ミロシナーゼ | ナタネ | ABQ42337 | 60.6 | 164 | |
| CIV_10 | 55 | Adenosylhomocysteinase 2 のような | B. rapa | XP_009135865 | 53.1 | 139 | |
| CIV_11 | 55 | 伸長因子 1 α 1 のような | B. oleracea | XP_013586115 | 51.9 | 161 | |
| CIV_12 | 50 | シスチン リアーゼ CORI3 のような | ナタネ | XP_013653143 | 48.1 | 237 | |
| CIV_13 | 44 | シスチン リアーゼ CORI3 のような | B. rapa | XP_009108611 | 48.3 | 213 | |
| CIV_14 | 38 | フルクトース-ビスリン酸アルドラーゼ | B. rapa | XP_009115993 | 38.4 | 226 | |
| CIV_15 | 45 | シスチン リアーゼ CORI3 のような | B. rapa | XP_009108611 | 48.3 | 219 | |
| CIV_16 | 45 | シスチン リアーゼ CORI3 | B. rapa | CDY69765 | 46.9 | 209 | |
| CIV_17 | 38 | フルクトース-ビスリン酸アルドラーゼ | B. rapa | XP_009115993 | 38.4 | 124 | |
| CIV_18 | 18 | プロリン シス-トランス異性化酵素 CYP18 の 3 のような | ナタネ | XP_009101932 | 18.3 | 128 | |
| CIV_19 | 45 | シスチン リアーゼ CORI3 のような | B. rapa | XP_009108611 | 48.3 | 177 | |
テーブル 3: 複雑な IV からのタンパク質成分を識別します。TRNA 結合複合体で発見されたいくつかの tRNA のリガーゼとさらに蛋白質、ドーパミンを使用して大量の分析 3 D ページの後。
ディスカッション
師部は高利のコンパートメント、深水の主要交通ルートを構成する、別の植物の部品9,20,34、間長距離シグナル伝達に必須でも 35。小さな分子に加えてタンパク質と Rna のような高分子は師部メンテナンスとシグナリング4,7,8関与しています。師部組成の分析は、ほとんど植物の師部サンプルに限られたアクセシビリティによって制限されます。昆虫スタイレット手法は広く適用が実験的に厳しいと師部サンプル11の非常に少量の結果します。師部をサンプリングするために使用される 1 つの簡単なテクニックは、EDTA 促進溶出12です。EDTA はカルシウムのような二価イオンをキレートおよび P 蛋白質およびカロースによる篩板の閉鎖を防止すること。それは使いやすいが、損傷した細胞による汚染しやすい、サンプルを希釈します。EDTA による二価イオンの破壊も、既存の複合体の破壊に 。ただし、それはモデル植物シロイヌナズナ15を含む種の広い範囲からのサンプリングができます。師部樹液の自発的な滲出物は、植物種の小さい数でのみ発生します。植物組織10の重大な傷害を伴う侵襲的な方法です。我々 は最近の長距離輸送4,8,20を研究モデル種としてナタネを設立しました。ここで、師管液楽しめます小さな切開から負傷の影響と汚染源を軽減できます。
別のサンプリング技術を使用して、別の植物種から採取された師部のプロテオームを近年7,8,17,36,37,で検討されている38,39。これらのプロテオーム研究蛋白質で抽出、条件の変化の下で沈殿蛋白質: 蛋白質および蛋白質についての結論を許可しないため、: 核酸相互作用が原産の条件の下に存在します。Co-免疫沈降 (CoIP) とオーバーレイの試金示されるカボチャ40、師部樹液の主要なリボ核蛋白質の複雑ながありますが、内のネイティブの条件の下で任意の高分子複合体が存在することを認められなかった、師部システム。
Schäggerらによって導入された、青ネイティブ ページ26日以降高解像度ゲル電気泳動手順との組み合わせでは、大規模なタンパク質複合体の個々 のコンポーネントの分離を有効にします。ネイティブナタネの 3 D ページ解析を使用して, 篩部の滲出液が最近ことを示すいくつかの高分子量蛋白質: 蛋白質と RNP 複合体師部サンプルに存在が酵素によって実行中4。サイズ排除クロマトグラフィーのような蛋白質の複合体として結合を分離する方法は存在するが、BN ページと比較した場合は解像度の面で失敗する可能性があります。さらに、複雑な分離の合理的な品質、サイズ排除列行列は調査されている全体的な複雑な分子量に応じて対応しなければなりません。異なるサイズの複合体を分析、したがって集中的なコストは、BN ページとして高い焦点を力として可能にしない列の種類を要求します。
ナタネから複合体を分析するための重要なステップは、師管液の開始材料として使用を含めます。師部サンプルの少なくとも 600 μ L 必要し、5 つ植えた植物から得られます。3 D ページおよび BN 北しみが付くことの師部サンプルの十分な量の初期の BN ゲルを開始する必要が。これを達成するには、師部サンプル集中している蛋白質の 20 に 30 μ g は、よく、少なくともそれぞれに読み込まれることができますまで並列で実行される同じサンプルのトリプリケートします。低すぎるか高すぎる濃度を減らす複合体 (図 3) の検出。師部のサンプルは、氷の上の反応管に収集する必要があります、タンパク質分解と売り上げ高を避けるために後で使用できるフリーズされて保存されるべきであります。プロテアーゼ阻害剤は、すべて師部プロテオーム解析で発見されているが、ナタネ4の師部の完全にアクティブなプロテアソームがまた記述されていた。さらに、ボリュームと師部サンプル組成依存サンプリング時点で環境条件10。したがって同じような条件の下で一日の同じ時間で収集するためには注意が必要があります。ストレス治療 (干ばつなど) は収集できる師部の量を減らすことができます。質量分析法による単一蛋白質を識別するために、すべての時間の手袋を装着して可能なケラチン汚染を制限する必須です。ケラチンは質量スペック解析時に追加の信号につながるし、タンパク質の同定を妨げません。高電圧や周囲温度で BN ページで実行すると、壊れやすい複合体の分解が生じるゲルの加熱につながります。
ここで提示されたプロトコルは蛋白質: 蛋白質複合体の解析に適しています。また、使用と並列の複合体に含まれている特定の Rna を検出できますが.これを達成するには、最近 BN 北部4しみが付くことのためのプロトコルを導入しました。Rna は、RNAse 汚染による劣化になりやすいです。このような劣化を抑える DEPC 処理水使用してください。
本手法の主な制限は、移行動作サイズ、形状とも複合体31、翻訳後修飾によって異なりますので BN-PAGE - で区切られたタンパク質の分子量の推定 41。RNP 複合体のサイズ推定は移行挙動に及ぼす核酸が原因も複雑です。それも防止できない同じサイズの複合体は共同 1 つの単一バンドに移行。これは複雑な組成の誤った予測につながることができます。例えば機能アッセイ4、したがって、相補的な手法で観測された相互作用を検証するは必要で 。蛋白質だけ弱いか一過性高分子複合体に関連付けられている使用される BN バッファー失われる可能性があります。これを避けるためには、サンプルが架橋、成果物につながることができますか、タンパク質同定を防ぐことができるまたはバッファー条件を最適化することができます。
古典的な 2 D のページとは対照的組み合わせ尿素-SDS-PAGE の疎水性と等電点 (pI) と分子量28ではなく分子量に従って分離し、蛋白質の分離は制限されません、特定の pI の範囲です。古典に類似した 2 D ページ、分離タンパク質シングル スポットとして表示されますが、対角線上線4,28 (図 5, 図 6)。ライン28下より親水性タンパク質が見つかったに対し、この対角線上のスポットで多くの疎水性タンパク質が表示されます。このプロトコルで使用されるポリアクリルアミド濃度は 25 に 80 kDa のタンパク質をよく分離されます。小さいまたは大きいタンパク質の分離、ポリアクリルアミドを許可するように濃度を変えることができるまたは勾配ゲルを 3 番目のディメンションで使用できます。3 D ページ (図 6) で区切られた複雑な IV (図 3) のコンポーネントは、トリプシン消化、および質量分析法による分析をカットされました。これは、いくつかの tRNA のリガーゼの特定につながった。他の複合体のコンポーネントをされている最近、 4を公開します。当社の 3 D ページには、異なるリガーゼ、すなわちアスパラギン酸、アスパラギン、スレオニン、リジン、グリシンのリガーゼ、同じような分子量 (表 3) との分離が有効になります。メチオニン tRNA 特定プローブを使用して、我々 は複雑な IV 内可能性がありますバインドされた tRNA を検出することができたが、フルレングス tRNA または tRNA 断片が、複雑な場合は使用プローブと差別されることはできません。プロテアーゼ消化師部 sap である核酸は本当にコンプレックス内連結および共同移行ではない場合、チェックするには、サンプルは適切なマイグレーション制御として役立つかもしれない。
それは師部篩管内でタンパク質翻訳することができます行われる師部サンプル4,7で伸長因子と同様、全体のリボソームのほとんどのコンポーネントが検出されましたので以前推測されています。TRNA と tRNA のリガーゼの私達の見つけることはこの考えを支持するようです。しかし、カボチャから篩部のサンプルで現在の tRNA 断片が翻訳42の強力な阻害剤に示されている、機能的なリボゾーム欠席4翻訳が場所を取ることができないことを示唆するように見えます。
一緒に取られて、ここで提示されたプロトコルは、高解像度の個々 のコンポーネントのネイティブ蛋白質: 蛋白質および師部のサンプルと分離からも RNP 複合体の分離と同定をことができます。このメソッドのアプリケーションは新規タンパク質と師部サンプル RNP 複合体の検出を有効し、したがってこの専門性の高い工場区画の新機能を解明することができます。
開示事項
著者が明らかに何もありません。
謝辞
彼らの科学的なサポート、ジェニファー フェイントをかけるとウルシュラ Zarzenska に感謝します。この仕事はキャリア統合許可 (CIG、PCIG14-ジョージア州-2013-63 0734) 第 7 フレームワーク プログラム、LFF GK06 'DELIGRAH' (Landesforschungsförderung ハンブルク)、グラントと DFG グラント (DFG KE 856_6-1) 内にある欧州委員会によって財政上支えられるJK に授与。
資料
| Name | Company | Catalog Number | Comments |
| 1.5 mL reaction tubes | Eppendorf | 30120086 | |
| 10 well comb for SDS PAGE, thickness 1 mm | BioRad | 1653359 | |
| 2-Propanol | AppliChem | 131090 | |
| 30 % Acrylamide-bisacrylamide solution (29:1) solution | BioRad | 1610156 | |
| 30 % Acrylamide-bisacrylamide solution (37.5:1) solution | BioRad | 1610158 | |
| 96 % Ethanol | Carl Roth | P075 | |
| Acetic acid | Carl Roth | 6755 | |
| Agarose | Lonza | 50004 | |
| Aluminum sulfate-(14-18)-hydrate | AppliChem | 141101 | |
| Ammonium peroxodisulfate (APS) | Carl Roth | 9592.3 | |
| Bis-Tris | AppliChem | A3992 | |
| Boric acid | AppliChem | A2940 | |
| Bromophenol blue | AppliChem | A2331 | |
| Centrifugal concentrators e.g. Vivaspin 500 (Mwco 10 kDa) | Sartorius | VS0102 | |
| Chemiluminescent Nucleic Acid Detection Module Kit | Thermo Fisher Scientific | 89880 | |
| Chromatography papers e.g. 3 mm CHR | Whatman | 3030-153 | |
| CoolBox M30 System | Biocision | BCS-133 | |
| Coomassie brilliant Blue G-250 | AppliChem | A3480 | |
| Diethyl pyrocarbonate (DEPC) | AppliChem | A0881 | |
| Dithiothreitol (DTT) | AppliChem | A1101 | |
| DNase I | AppliChem | A3778 | |
| Ethanol abs. | AppliChem | A3693 | |
| Ethylenediaminetetraacetic acid (EDTA) | AppliChem | A1103 | |
| Gel Imaging system e.g. Chemidoc Touch | BioRad | 1708370 | |
| GeneRuler 1kb plus | Thermo Fisher Scientific | SM1331 | |
| Glycerol | AppliChem | 141339 | |
| Glycine | AppliChem | 131340 | |
| Heating block TS1 | Biometra | 846-051-500 | |
| Hybridization oven e.g. GFL Hybridization Incubator 7601 | GFL | 7601 | |
| Hypodermic needle (0.8 mm) | B Braun | 4658309 | |
| IPG gel comb, thickness 1 mm | BioRad | 1653367 | |
| Labeling of RNA e.g.Biotin 3'end DNA Labeling Kit | Thermo Fisher Scientific | 89818 | |
| Native gradient gel e.g. Novex NativePAGE 4–16% Bis-Tris protein gel | Invitrogen | BN1002BOX | |
| Nylon membrane e.g. Hybond-N+ | Amersham Pharmacia | RPN203B | |
| Ortho-phosphoric acid | Carl Roth | 6366.1 | |
| PageRuler prestained protein ladder | Thermo Fisher Scientific | 26616 | |
| Power supply for Gel electrophoresis EPS | Amersham Pharmacia | 18-1130-01 | available from GE Healthcare |
| RNA Cleanup Kit | Qiagen | 74204 | |
| Semi dry blot e.g. Fastblot 43B semi dry Blot | Biometra | 846-015-100 | |
| Sodium chloride (NaCl) | AppliChem | A1149 | |
| Sodium dodecyl sulfate (SDS) | AppliChem | 142363 | |
| Synthesis of cDNA e.g. RevertAid First Strand cDNA Synthesis Kit | Thermo Fisher Scientific | K1621 | |
| Tetramethylethylenediamine (TEMED) | AppliChem | A1148 | |
| Tricine | AppliChem | A3954 | |
| Tris | AppliChem | A1086 | |
| Tri-sodium citrate dihydrate | AppliChem | A3901 | |
| TRIzol Reagent | Thermo Fisher Scientific | 15596026 | |
| ULTRAhyb Ultrasensitive Hybridization Buffer | Thermo Fisher Scientific | AM8670 | |
| Urea | AppliChem | A1360 | |
| UV Crosslinker e.g. Stratalinker 2400 | Agilent | NC0477671 | from Fisher Scientific |
| Vertical electrophoresis apparatus e.g.The XCell SureLock Mini-Cell or Mini-Protean III System | Thermo Fisher Scientific or BioRad | EI0001 or 1658004 | |
| Wipers e.g. KIMWIPES Delicate Task Wipers | Kimberly-Clark | 34120 |
参考文献
- Kempers, R., van Bel, A. J. E. Symplasmic connections between sieve element and companion cell in the stem phloem ofVicia faba L. have a molecular exclusion limit of at least 10 kDa. Planta. 201 (2), 195-201 (1997).
- Stadler, R., et al. Expression of GFP-fusions in Arabidopsis companion cells reveals non-specific protein trafficking into sieve elements and identifies a novel post-phloem domain in roots. Plant J. 41 (2), 319-331 (2005).
- Imlau, A., Truernit, E., Sauer, N. Cell-to-cell and long-distance trafficking of the green fluorescent protein in the phloem and symplastic unloading of the protein into sink tissues. Plant Cell. 11 (3), 309-322 (1999).
- Ostendorp, A., et al. Functional analysis of Brassica napus phloem protein and ribonucleoprotein complexes. New Phytol. 214 (3), 1188-1197 (2017).
- Paultre, D. S. G., Gustin, M. -. P., Molnar, A., Oparka, K. J. Lost in Transit: Long-Distance Trafficking and Phloem Unloading of Protein Signals in Arabidopsis Homografts. Plant Cell. 28 (9), 2016-2025 (2016).
- Sali, A., Glaeser, R., Earnest, T., Baumeister, W. From words to literature in structural proteomics. Nature. 422 (6928), 216-225 (2003).
- Lin, M. -. K., Lee, Y. -. J., Lough, T. J., Phinney, B. S., Lucas, W. J. Analysis of the Pumpkin Phloem Proteome Provides Insights into Angiosperm Sieve Tube Function. Mol. Cell. Proteomics. 8 (2), 343-356 (2008).
- Giavalisco, P., Kapitza, K., Kolasa, A., Buhtz, A., Kehr, J. Towards the proteome of Brassica napus phloem sap. Proteomics. 6 (3), 896-909 (2006).
- Lucas, W. J., Yoo, B. C., Kragler, F. RNA as a long-distance information macromolecule in plants. Nat. Rev. Mol. Cell. Bio. 2 (11), 849-857 (2001).
- Dinant, S., Kehr, J. Sampling and Analysis of Phloem Sap. Plant Mineral Nutrients. 953, 185-194 (2013).
- Kennedy, J. S., Mittler, T. E. A Method of obtaining Phloem Sap via the Mouth-parts of Aphids. Nature. 171 (4351), 528 (1953).
- King, R. W., Zeevaart, J. A. Enhancement of Phloem exudation from cut petioles by chelating agents. Plant Physiol. 53 (1), 96-103 (1974).
- Alosi, M. C., Melroy, D. L., Park, R. B. The regulation of gelation of Phloem exudate from cucurbita fruit by dilution, glutathione, and glutathione reductase. Plant Physiol. 86 (4), 1089-1094 (1988).
- Dinant, S., Bonnemain, J. -. L., Girousse, C., Kehr, J. Phloem sap intricacy and interplay with aphid feeding. Comptes rendus biologies. 333 (6-7), 504-515 (2010).
- Tetyuk, O., Benning, U. F., Hoffmann-Benning, S. Collection and analysis of Arabidopsis phloem exudates using the EDTA-facilitated Method. J Vis Exp. (80), e51111 (2013).
- Kovalskaya, N., Owens, R., Baker, C. J., Deahl, K., Hammond, R. W. Application of a modified EDTA-mediated exudation technique and guttation fluid analysis for Potato spindle tuber viroid RNA detection in tomato plants (Solanum lycopersicum). J. Virol. Methods. 198, 75-81 (2014).
- Rodriguez-Medina, C., Atkins, C. A., Mann, A. J., Jordan, M. E., Smith, P. M. Macromolecular composition of phloem exudate from white lupin (Lupinus albus L). BMC Plant Biol. 11 (1), 36 (2011).
- Taylor, J. S., Thompson, B., Pate, J. S., Atkins, C. A., Pharis, R. P. Cytokinins in the Phloem Sap of White Lupin (Lupinus albus L.). Plant Physiol. 94 (4), 1714-1720 (1990).
- Yoo, B., et al. A Systemic Small RNA Signaling System in Plants. Plant Cell. 16 (8), 1979-2000 (2004).
- Buhtz, A., Springer, F., Chappell, L., Baulcombe, D. C., Kehr, J. Identification and characterization of small RNAs from the phloem of Brassica napus. Plant J. 53 (5), 739-749 (2008).
- Chalhoub, B., et al. Plant genetics. Early allopolyploid evolution in the post-Neolithic Brassica napus oilseed genome. Science. 345 (6199), 950-953 (2014).
- Sasaki, T., Chino, M., Hayashi, H., Fujiwara, T. Detection of several mRNA species in rice phloem sap. Plant Cell Phys. 39 (8), 895-897 (1998).
- Doering-Saad, C., Newbury, H. J., Bale, J. S., Pritchard, J. Use of aphid stylectomy and RT-PCR for the detection of transporter mRNAs in sieve elements. J Exp. Bot. 53 (369), 631-637 (2002).
- Ruiz-Medrano, R., Xoconostle-Cázares, B., Lucas, W. J. Phloem long-distance transport of CmNACP mRNA: implications for supracellular regulation in plants. Dev. 126 (20), 4405-4419 (1999).
- Schägger, H., Cramer, W. A., von Jagow, G. Analysis of molecular masses and oligomeric states of protein complexes by blue native electrophoresis and isolation of membrane protein complexes by two-dimensional native electrophoresis. Anal. Biochem. 217 (2), 220-230 (1994).
- Schägger, H., von Jagow, G. Blue native electrophoresis for isolation of membrane protein complexes in enzymatically active form. Anal. Biochem. 199 (2), 223-231 (1991).
- Fiala, G. J., Schamel, W. W. A., Blumenthal, B. Blue native polyacrylamide gel electrophoresis (BN-PAGE) for analysis of multiprotein complexes from cellular lysates. J Vis Exp. (48), e2164 (2011).
- Rais, I., Karas, M., Schägger, H. Two-dimensional electrophoresis for the isolation of integral membrane proteins and mass spectrometric identification. Proteomics. 4 (9), 2567-2571 (2004).
- Schägger, H. Tricine-SDS-PAGE. Nature protocols. 1 (1), 16-22 (2006).
- Wittig, I., Braun, H. -. P., Schägger, H. Blue native PAGE. Nat Protoc. 1 (1), 418-428 (2006).
- Eubel, H., Braun, H. -. P., Millar, A. H. Blue-native PAGE in plants: a tool in analysis of protein-protein interactions. Plant Methods. 1 (1), 11 (2005).
- Laemmli, U. K. Cleavage of structural proteins during the assembly of the head of bacteriophage T4. Nature. 227 (5259), 680-685 (1970).
- Dyballa, N., Metzger, S. Fast and sensitive colloidal coomassie G-250 staining for proteins in polyacrylamide gels. J Vis Exp. (30), (2009).
- Turnbull, C. G. N., Lopez-Cobollo, R. M. Heavy traffic in the fast lane: long-distance signalling by macromolecules. New Phytol. 198 (1), 33-51 (2013).
- Hall, S. M., Baker, D. A. The chemical composition of Ricinus phloem exudate. Planta. 106 (2), 131-140 (1972).
- Barnes, A., Bale, J., Constantinidou, C., Ashton, P., Jones, A., Pritchard, J. Determining protein identity from sieve element sap in Ricinus communis L. by quadrupole time of flight (Q-TOF) mass spectrometry. J. Exp. Bot. 55 (402), 1473-1481 (2004).
- Walz, C., Giavalisco, P., Schad, M., Juenger, M., Klose, J., Kehr, J. Proteomics of curcurbit phloem exudate reveals a network of defence proteins. Phytochemistry. 65 (12), 1795-1804 (2004).
- Walz, C., Juenger, M., Schad, M., Kehr, J. Evidence for the presence and activity of a complete antioxidant defence system in mature sieve tubes. Plant J. 31 (2), 189-197 (2002).
- Kehr, J. Phloem sap proteins: their identities and potential roles in the interaction between plants and phloem-feeding insects. J Exp. Bot. 57 (4), 767-774 (2006).
- Ham, B. -. K., Brandom, J. L., Xoconostle-Cázares, B., Ringgold, V., Lough, T. J., Lucas, W. J. A polypyrimidine tract binding protein, pumpkin RBP50, forms the basis of a phloem-mobile ribonucleoprotein complex. Plant Cell. 21 (1), 197-215 (2009).
- Schamel, W. W. Biotinylation of protein complexes may lead to aggregation as well as to loss of subunits as revealed by Blue Native PAGE. J. Immunol. Methods. 252 (1-2), 171-174 (2001).
- Zhang, S., Sun, L., Kragler, F. The phloem-delivered RNA pool contains small noncoding RNAs and interferes with translation. Plant Physiol. 150 (1), 378-387 (2009).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved