Method Article
大規模エピジェネティック研究のための半自動化されたChIP-Seq法
Erratum Notice
要約
本論文では、ChIP液体ハンドラプラットフォームを用いた特定のヒストン修飾(H3K27ac)に関連するDNA領域の半自動マイクロスケールのChIP-Seqプロトコルについて説明し、続いてタグメンテーションを用いたライブラリ調製について述べている。この手順には、品質と量の目的に対する制御評価が含まれており、他のヒストン修飾または転写因子に採用することができます。
要約
クロマチン免疫沈降に続いてシーケンシング(ChIP-Seq)は、CIS調節性DNA要素の同定に役立つH3K27acなどの特定のヒストン修飾に関連するクロマチンDNAをプロファイルするための強力で広く使用されているアプローチです。ChIP-Seqを完了するための手動プロセスは、労働集約的で技術的に困難であり、多くの場合、大きなセル数(>100,000細胞)を必要とします。ここで説明する方法は、これらの課題を克服するのに役立ちます。細胞固定、クロマチンせん断、免疫沈降、およびシーケンシングライブラリ調製を含む完全な半自動のマイクロスケーリングされたH3K27ac ChIP-Seq手順は、100,000未満の細胞数入力のための48サンプルのバッチ用に詳細に説明されています。半自律型プラットフォームは、技術的な変動性を低減し、信号対雑音比を改善し、労働力を大幅に削減します。これにより、反応量の削減によりコストを削減でき、酵素、磁気ビーズ、抗体、ハンズオン時間などの高価な試薬の数を制限できます。ChIP-Seq法のこれらの改良は、非常に再現性の高い方法で限られた細胞数を有する臨床サンプルの大規模なエピジェネティック研究に完全に適している。
概要
特定のヒストン修飾に関連するDNAの断片を決定するためのChIP-Seqアッセイの広い使用は、活性エンハンサー、プロモーター、サイレンサー、ヘテロクロマチン、および他の1、2、3、4を含むシス調節性DNA要素を同定する能力の一部である。ゲノム全体にわたる非コード調節領域の同定は、健康および疾患における遺伝子調節をよりよく理解するための貴重な洞察を示した4.研究室からの以前の研究は、シス- 調節要素が異なる細胞タイプ5で重要な役割を果たすことができることを示すためにChIP-Seqを使用してきました。転写因子(TF)のChIPアッセイは、疾患関連リスク一塩基多型を示すために利用されている。
ヒト臨床サンプルでのChIP-Seqの使用は、主に細胞数または所望の組織サンプルの制限により、困難です。その結果、これらの技術を改善し、マイクロスケールする分野で協調的な努力があった、その結果、いくつかのアッセイ、例えば、CUT&TAG5、7、8、9、10、11、12。本アッセイは、特定の抗体9に結合したゲノム領域をタグ付けおよび単離するトランスポザーゼを利用する。この技術は、細胞数を1,000sに減らし、場合によっては単一細胞にまで減少させることができましたが、翻訳研究および臨床セットアップにおけるこの技術の使用は、この方法9,12に対して生細胞を使用する要件による限界を示しています。ライブセルの要件により、臨床サンプルの処理がロジスティックに困難になり、同時に処理されない場合はバッチ効果を導入できます。また、高スループットでここで適応されるChIPmentation11の開発を含む、ホルムアルデヒド固定細胞のマイクロスケーリング技術を最適化しているものもあります。固定セルを使用すると、バッチ効果を最小限に抑えるために、すべてのサンプルを収集し、その後の処理を行うまで、サンプルを保存できます。
ここでは、ヒストン修飾10をプロファイルする実験的な実務時間を短縮する半自動マイクロスケーリングされたChIP-Seqアッセイが記載されている。半自動化方式により、高スループットのChIP-Seqアッセイが可能になり、最大48個のサンプルを完全に処理し、わずか5日間で、ChIP液体ハンドラを使用してサンプルあたりわずか10,000個のセルでシーケンシングを行うことができます。このハンドラーは、自律的な方法で免疫沈降(IP)とその後の打ち込み処理を完了し、サンプル間の変動性を低減するのに役立ちます。半自動化された方法は、48サンプルのハンズオン時間と技術的変動の両方を15時間以上短縮し、一次細胞または培養細胞に対して再現可能かつ迅速な方法で大規模なエピジェネティック研究を行うことを可能にする。プロトコルは、高品質のChIP-Seqのための最初から最後までプロセスを説明します。特定のマシンが使用できない場合でも、このプロトコルは ChIP-Seq 実験を手動でセットアップして問題を解決するのに役立つリソースとなります。
アッセイは、3つの異なるヒト免疫細胞型および1つの培養細胞株(HUT78 –ATCC:TIB-161)で行った。明確にするために、プロトコルは7つのセクションに分かれています:細胞固定、超音波処理によるクロマチン剪断、自動クロマチン免疫沈降、DNA断片タグ化によるライブラリ調製、ライブラリ増幅、ライブラリ精製、続いてDNA定量化。バッファレシピについては、補足表1を参照してください。
プロトコル
ラホヤアレルギー免疫研究所(LJI;)IRB プロトコルいいえ。SGE-121-0714)は、研究を承認しました。健康なボランティアが募集され、書面によるインフォームド・コンセントの後に白血病サンプルを提供しました。
1. 細胞固定
- 細胞懸濁液濃度を15mLチューブ(<10mLの懸濁液)または50mLチューブ(10〜30mLの細胞)に完全な細胞培養培地を用いて1〜2 x 106 細胞/mLに持ち込みます。<1 x 106 細胞の場合、1.5mLチューブ内の培地0.5mLを使用してください。
- セルサスペンションを穏やかに渦巻き、10x細胞固定バッファーを滴下(1:10;vol:vol)に加え、室温(RT)で10分間低速で回転させます。
- 穏やかに渦巻きで反応を停止し、1:20(vol:vol)の比で2.5 Mグリシンを加えます。チューブを数回反転し、氷の上で少なくとも5分間インキュベートします。
- 残りのステップは4°Cまたは氷上で行います。4°Cで5分間800 x g でチューブを回転させ、上清を捨てます。
- 5 mLの氷冷1x PBSでペレットを緩やかに再懸濁し、氷の上で2分間インキュベートします。
- 1 mLの氷冷1x PBSで1.4と1.5を繰り返し、サンプルを予冷した1.5 mLチューブ(長期保存用にラベル付け)に移します。該当する場合は、アリコートの準備をお勧めします。
- 4°Cで1,200 x gでチューブを回転させ、細胞ペレットを邪魔することなく、できるだけ多くの上清を取り除きます。液体窒素中のペレットを凍結スナップします。-80°Cで保管してください。
注意: 液体窒素を取り扱う際には適切な保護を行ってください。
2. クロマチンせん断
注:このプロトコルは0.65 mL低結合管の0.3から3 x 106 細胞のクロマチンの剪断のために最適化されています。
- サンプルチューブを-80°Cから凍結した細胞ペレットで取り出し、乾燥した氷の上に保管して、リシスバッファーを追加する前にペレットの解凍を避けます。この手順は非常に重要です。
- ペレットに新鮮なRT完全なリシスバッファーの70 μLを追加し、1分間RTで保ちます。
- ペレットを気泡を導入せずに1分間再懸濁し、サンプルを氷の上に置く前にRTで1分間培養します。
- 再懸濁されたペレットを0.65 mLの低結合チューブに移し、氷の上に置いてください。
注:再現性の超音波処理を得るために、サンプルを超音波処理する前に3〜6サイクルのブランクチューブだけでそれを実行することによって、ソシレータを事前に暖めます。 - サンプルを超音波処理器のチューブホルダーに入れ、70 μL の水で満たされたバランスチューブで隙間を埋めます。超音波処理を開始する前に約1分間、水浴中にサンプルを残します。
- xサイクル(セルタイプに応じて)の超音波処理を実行します(セルの種類に応じて)。
注: このステップでは、効率的な超音波処理のためのサイクルの最適な数を決定する検証実験が必要になります。 - 3サイクルごとに、超音波処理器からサンプルを取り出し、穏やかに渦を取り出し、パルスはホルダーに戻す前にチューブを回転させます。気泡形成を引き起こす可能性があるので、チューブの外側に小さな液滴がないことを確認してください。
- 必要なサイクルを完了した後、4°Cで15分間>14,000 x gでサンプルを回転させます。 上清を新しい、事前に冷却された低結合の0.65 mL低結合の管に移し、氷の上に保つ。
- 超音波処理効率をチェックするために超音波処理されたサンプルの一部をリンク解除します。
- 上清の1~7μL(約250ngのシアドクロマチンに相当)を0.2 mLのPCRチューブに移し、RTで短期溶解バッファを使用して最大10μLの体積を作ります。
- 1 μL RNase Aを加え、800 rpmで37°Cで30分間インキュベートし、1 μLプロテイナーゼKを加えます。
- 55°Cで2時間インキュベートし、1,000rpmで振る。
- 蛍光定量アッセイ10 を用いたDNA定量用の非架化サンプル2μLを除去する(分光光度計は石鹸と分解タンパク質が結果に偏りを生じさせる可能性があるとして推奨されない)。
- 残りのサンプルを70 Vのステインで1.2%のアガロースゲルで1時間実行し、UVトランスイルミエーターを使用してゲルを読み取ります。
- クロマチンストックアリコートを貯蔵用に準備します(完全な溶解バッファーで20 μLでサンプルを25 ng/μLに希釈します)。すべてのシアクロマチンを-80°Cで保管してください。
ヒストン修正のための自動化されたChIP-Seq
注: このプロトコルは、ChIP 液体ハンドラ上で動作するように設計されています。システムはカスタマイズされたバッファを使用できますが、すべてのバッファは ChIP キットに付属しています。このセクションで使用される8本の管が付いているChIPストリップはChIPの液体ハンドラに特有である。
- -80°Cから20μLに500ngのシアドクロマチンを含む16サンプルのアリコートを移し、氷の上に置いてクロマチンをゆっくりと解凍します。一度完全に解凍し、渦を短時間とパルススピン。
- クロマチンの調製
- 1xプロテアーゼ阻害剤と20 mMナトリウムブチレート(完全なtC1バッファー)を2つのChIP 8チューブストリップに補ったtC1バッファーのピペット100 μL。
- 完全なtC1バッファーの100 μLを含むChIP 8管ストリップの適切なチューブに、各クロマチンサンプルの20 μLを移します。クロマチンチューブに80 μL完全tC1バッファーを加えてクロマチンチューブを洗浄し、ChIP 8チューブストリップの適切なチューブに戻して200 μLの最終容積に戻します。
- 抗体の調製
- 各チューブに0.5μgの抗体が入っている様に、抗体の体積を計算します。
抗体の容量 = (反応あたりのサンプル x 抗体数) / 抗体濃度 - tBW1バッファーの500 μLに、算出された量の抗体を追加します。素早く渦とパルススピン。
- tBW1のピペット70 μLを2つのChIP 8チューブストリップのそれぞれに加え、各チューブに30 μLの抗体+tBW1を加えます。これにより、各チューブの総容積は100 μLになります。
- 各チューブに0.5μgの抗体が入っている様に、抗体の体積を計算します。
- 磁気ビーズの調製
- タンパク質Aビーズ溶液を徹底的にボルテックスする。0.5 μgの抗体の場合、ピペット 5 μL のビーズを新しいセットの ChIP 8 チューブストリップとパルススピンに組み込みます。
- ChIP 液体ハンドラの最後の行にラベル付きの空の ChIP 8 チューブ ストリップを記入します。
- ChIP-16-IPure-200Dプログラム仕様に従って、ChIP 液体処理機内のすべてのストリップを配置します。バッファーを正しい位置に追加しますが、tE1 バッファーの代わりに tW4 を使用します。
メモ: ChIP 液体ハンドラが ChIP を一晩実行する日を整理します。プログラムは16サンプルで約16時間実行されます。これは 1 日目の終わりを示します。
4. ライブラリの準備のためのライブラリアダプタのトランスポザーゼ統合
- サーモミキサーを37°Cおよび500rpmに事前に設定します。氷の上に0.2 mLチューブストリップのための磁石を冷却します。
- 16個のサンプルの場合、440 μLのタグメントバッファーを氷上に用意します。ピペット53 μLを1つの新しい8チューブストリップに入れ、氷の上に保管します。
- 新しい0.2 mL 8チューブストリップに、220 μLのコールドtC1バッファを追加し、氷の上に置きます。8つのストリップ管はこの容積を保持し、それでも上限を設けることができる。
- ChIP液体ハンドラマシン(行12)から「IPサンプル」ストリップチューブを取り外し、パルス回転前にチューブをキャップします。8管ストリップ用のマグネットを2分間使用してビーズを取り込み、上清を慎重に取り除きます。
- タグメント化バッファーの 25 μL をマルチチャネルでビーズに移し、磁石から取り出し、ビーズが均質になるまで穏やかに混ぜます(ピペットを20 μLに設定して約5倍上下に)。
- チューブをキャップし、予熱熱したサーモミキサーに入れ、3分間インキュベートします。時間を増やすと、ライブラリの準備の効率が低下します。
- チューブを冷やした金属ラックに移し、各サンプルに100 μLのチルドtC1バッファを追加します。マルチチャンネルピペットを80μLにセットし、ビーズが均質になるまでサンプルを混ぜて、タグメント反応を止めます。
- サンプルを ChIP 液体ハンドラに戻し、洗浄手順 Washing_for_IP-reacts_16_Ipureに進みます。洗浄はtC1バッファで2回、tW4で2回行われます。溶出は、バッファ tE1 で、プログラムレイアウトによってマークされるように完了する必要があります。
- DNAの逆リンク
- ChIP液体処理器の最後の行のChIP 8管ストリップを取り外し、各サンプルに2μL RNase Aを加えます。
- チューブをキャップし、パルススピンし、混合物が均質になるまでマルチチャネルピペットでビーズを穏やかに混ぜ、チューブを再キャップします。
- 37°Cおよび900 rpmで30分間、サーモミキサーでサンプルをインキュベートします。
- サーモミキサーからサンプルを取り出し、2 μLのプロテインナーゼKを加え、添加後の4.9.2と同じ手順に従います。
- サンプルを55°Cおよび1,250 rpmで4時間熱ミキサーにインキュベートし、続いて1,000rpmで65°Cを一晩でインキュベートします。
注:これは2日目の終わりです。
5. タグ付きDNA断片精製
- 適切なサンプル数で16個の1.5mLチューブにラベルを付け、DNAクリーンアップキットから400 μLのDNA結合バッファーをそれぞれに追加します。
- サーモミキサーから8チューブストリップを取り外し、ストリップをパルス回転させて、蒸発した製品が保持されていることを確認します。8ストリップ磁石にストリップを置き、ビーズを取り込みます。
- 1.5 mLチューブのそれぞれに、100 μLの架橋解除DNAを移します。8チューブストリップに100μLのDNA結合バッファーを加え、ビーズを洗浄し、適切な1.5mLチューブに移します。
- 約10 sの渦とパルススピン1.5 mLチューブ。
- DNA結合バッファーと ChIP サンプルを含む 600 μL でカラムをロードします。
- 10,000 x gで 20 s のスピン サンプルを実行し、フロースルーで列をリロードします。同じ条件で再びスピンし、フロースルーを破棄します。
- 200 μLの洗浄バッファーで2回洗浄し(前のステップと同じ遠心分離)、フロースルーを廃棄します。
- 12,000 x g で 2 分間遠心分離して柱 を乾燥 させます。
- カラムを新しい1.5 mLコレクションチューブに移し、9 μLのウォームTEバッファ(55°Cに予熱)をカラムマトリックスに直接追加します。10,000 x gで 1 分間遠心分離する前に 1 分間インキュベートするカラムを許可します。
- 9 μLのエルテを8チューブストリップの適切な新しいセットに移します。
- 8 μL TE バッファを使用して溶出を再度完了します。エルチを適切な8チューブストリップ(最終体積17μL/サンプル)に移し、氷の上に置きます。
精製サンプルの増幅とサイズ選択
- 次の手順では、qPCR を使用して、最適な増幅に必要なサイクル数を決定します (CtD – Ct の決定)
- CtD ミックス バッファの内容にサンプル数を掛けて、すべてのサンプルに対して CtD ミックスを準備します。
- 3.6 μLのCtDをqPCRプレートに混ぜて、タグ付きDNAサンプルを1.4 μL加えます(総体積の10%)。qPCR は、3 分間 98 °C、5 分間 72 °C、30 s の場合は 98 °C、10 s の 98 °C の 26 サイクル、30 s の場合は 63 °C、30 s の場合は 72 °C です。
- AMP ミックス バッファの内容にサンプル数を掛けて、すべてのサンプルに対してアンプ ミックスを準備します。タグ付けされたDNAの14 μLをPCRプレートの別々のウェルに分配し、各サンプルに2.5 μLの2つのシーケンシングインデックスプライマー(25 μM)を加えます(最終反応容積は50μL)。
- マルチチャンネルピペットでサンプルを混合し、適切なサイクル数でCtDで使用される増幅プログラムを実行します。
注: 増幅されたサンプルは-20 °Cで数週間保存できるため、これは良い停止点です。しかし、精製は同じ日に完了することができます。48個のサンプルの場合、ステップ3~6.5は、他の2つの別々のバッチで完了し、次に、以下に説明する1つのバッチで増幅した。 - 後述の方法で、後の増幅、サイズ選択、およびタグ付けされたDNAの定量化を行います。これは通常、48個のサンプルで完了できます(必要に応じて少ないサンプルで完了できます)。
- 各ウェルに90 μL (1:1.8比) を加えて混合し、RTで2分間インキュベートします。
- プレート磁石を使用してビーズを取り込み、上清を捨てます。ビーズペレットを破壊することなく、新鮮な80%エタノールを200 μLで3回洗浄します。
- 最終洗浄後に20μLチップを含む余分なエタノールを取り除き、ビーズを10分間乾燥させるか、ビーズペレットに亀裂が入るまで乾燥させます。
- プレートを磁石の上に置いたまま、各ウェルに40μLの事前温水を加えます。プレートを密封し、渦を徹底的に密封し、プレートを短時間パルス回転させます。
- プレートを磁石に戻してビーズを取り込み、40 μLのエルテを新しい「サンプル」プレートに移します。サンプルは精製され、次のステップは200〜1,000 bpの範囲の断片を濃縮する。
- オプションのQCステップ:サンプルから4μLを取り出し、QCプレートに移します。サンプルに4μLの水を加えます。これは、大きなフラグメントの割合を決定します。
- 22 μL の常磁性ビーズ(1:0.55比)をサンプルに加え、慎重に混合し、RT で 2 分間インキュベートします。
- 磁石の上に置き、ビーズを5分間捕獲し、上澄み物を「サンプル」プレートの7〜12列に移します。磁石からプレートを取り出し、ビーズを30 μL(最終比1:1.3)を加えます。慎重に混ぜて、RTに2分間座るようにします。
- 5分間ビーズを取り込み、上清を捨てます。
- 前述のように、200 μLの新鮮な80%エタノールで3回すべてのビーズを洗浄します(ステップ6.6.2 - 6.6.3)。
- ペレットが乾燥したら、8 μL の事前温め TE バッファーを持つ DNA を各ウェルに溶出し、磁石の上に置きます。
- マグネット、シール、渦からプレートを徹底的に取り外します。プレートをRTで2分間インキュベートし、パルススピンし、プレートを磁石に戻して2分間置きます。上清を新しいプレート(プレート2)に移します。
- 最大の回復のために、予め温めたTEバッファの余分な8 μLの溶出を繰り返します。各サンプルが最終ライブラリの16 μLを持つような適切なウェルにサンプルを入れます。
注: このステップの最後には、2 つのプレートが存在する必要があります(QC プレートが完成していない場合は 1 つ)。QCプレートには事前サイズ選択の断片があり、2番目のプレートには48個の最終ライブラリ(合計16 μL)のウェルが必要です。
7. 蛍光アッセイを用いて最終ライブラリとQCサンプルを定量化する
- 蛍光定量アッセイまたは同様の方法を用いた完全なDNA定量。
- QC 定量が完了した場合、1,000 bp <されたサンプルの損失の割合を決定します。約 20% 以下の損失があるはずです – それ以上の場合は、適用されたビーズの比率に問題がある可能性があります。
- 各試料の断片の大きさを決定し、好ましくは毛細管電気泳動機を用いる。モル濃度を計算するには、[ライブラリ濃度(ng/μL)* 10 6]/[660* 中央値フラグメントサイズ(bp)]の式を使用します。
注: ライブラリは、プール可能で (等量) し、標準の次世代シーケンス手順に従って順序付けされます。
結果
概念実証として、ChIP-Seqは、ナイーブCD4 T細胞(CD4)、古典的な単球(MO)およびナチュラルキラー細胞(NK)の3セットの免疫細胞を有する6人のヒトドナーに対して完成し、13以前に説明したようにFACSソートによって濃縮された。下線付きの手順は 、図 1に示すように 9 つの異なる手順で構成されます。
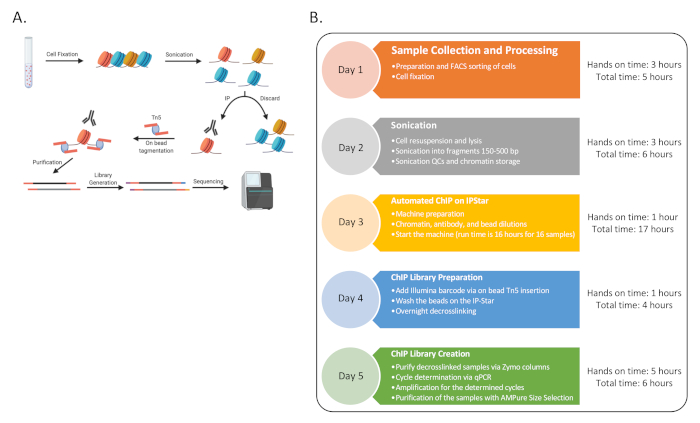
図1: 手順の一般的なフローチャート. ( A) 全体の手順の漫画 (BioRender で生成)(B) プロトコルのすべての主要なステップのフローチャートと、各日に関連付けられた推定ハンズオン時間と合計時間。シーケンスは、5 日目の終わりに、または複数のラウンドで発生する可能性があります。タイムラインは週を通してずらされ、連続する3-4を1週間に複数回完了して48のChIPサンプルを生成することができます。 この図の大きなバージョンを表示するには、ここをクリックしてください。
フローサイトメトリー13による細胞分離後、並び替えられた細胞を遠心分離し、細胞を固定し、上記のように保存した。すべてのサンプルを収集したら、サンプルを分解し、上記のように12のバッチで色彩のせん断のために調製した。各サンプルについて、最適な超音波処理に到達するためのサイクル数は10を完了しました。定量的測定は、染色クロマチンフラグメントサイズ測定と同様に、3組の免疫細胞に対する我々の方法の大きな再現性を示した(図2A)。異なるヒト免疫細胞は、別々のバッチで超音波処理され、14サイクル(16秒ON、サイクルあたり32 s OFF)の間のサンプルの>70%で非常に一貫して得た。この時点で、超音波処理後の大きな断片を有するサンプル(<100〜500 bpの間のサンプルの70%)が失敗したと考えられた。これらのサンプルは、1〜2サイクル超音波処理されるか、廃棄され、後で別のペレットの細胞に置き換えられた。我々の方法は、より多くの超音波処理を必要とするサンプルのいずれも示さなかったか、または排除され、手順の絶対的な堅牢性を示唆した。
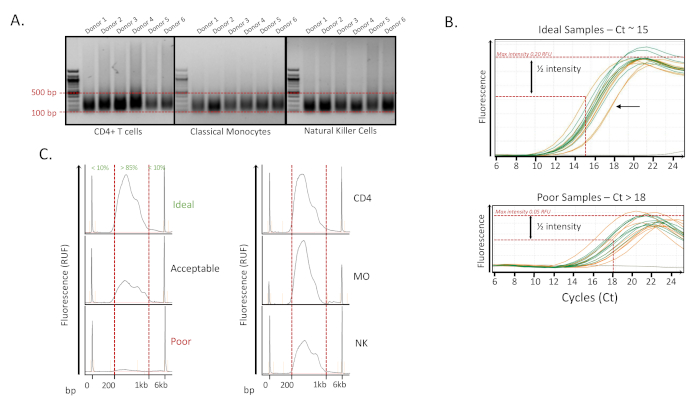
図2:予一シーケンシングQCの例.(A)1.2%アガロースゲルは超音波処理の再現性を示す。ナイーブ CD4 T 細胞 (CD4)、古典的な単球 (MO)、およびナチュラルキラー細胞 (NK) の 3 つの細胞の 6 つのドナーのための超音波処理サンプル。サンプルは14サイクル(16秒ON、32秒オフ/サイクル)で超音波処理された。各サンプルについて約200ngの脱架結クロマチンを1%アガロースゲルにロードした。サンプルは、断片の70%以上が100-500 bp.(B)上 - 増幅のためのサイクルの最適数を決定するためにqPCR増幅曲線を分析する場合に良好であると考えられた(最大強度は1/2あるCt)。理想的なサンプルは約15のCtを有し、増幅は測定されたCtの2サイクルまで完了することができる。矢印はCtが18より大きい悪い例の例である。下 - 18 を超える Ct を持つサンプルの貧弱なセットの例を示します。これらのサンプルはまた、より低い蛍光強度を示した。(C)左 - フラグメント分析電気泳動トレースは、増幅およびサイズ選択後の最終的なタグ付きライブラリの分布を示した。フラグメントライブラリの85%以上のサンプルは200-1,000 bp以内にあるが良いサンプルとして考えられた。蛍光のピーク強度の測定も重要なQCパラメータとして考えられ、実際に信号が低い場合、試料がうまく配列する可能性は低い。右 - CD4、MO、およびNKの陽性サンプルの例を示します。この図の大きなバージョンを表示するには、ここをクリックしてください。
定量後、サンプルをH3K27ac抗体を用いたChIP液剤処理器上で実行し、続いてTn5トランスポザーゼ酵素によるタグメンテーションを行った。qPCRによる適切な増幅サイクル数を決定するために、タグ付けされたサンプルの10%を使用した。サンプルの増幅のためのサイクル数の決定のために、サンプルの強度がサイクル決定のための平均最大値の半分であるサイクルを見つける(図2B)。Ct 値が 18 を超えるサンプルは、シーケンス後の動作が適切ではなく、その Ct 値が失敗した ChIP サンプルを示すものでした。これらのサンプルは一般に、増幅後のDNAの量を少ない量で得た。Ct 値が 15 以下のサンプル (100,000 個のセル入力) が理想的であり、15 ~ 18 の間のサンプルは許容可能であったが、一貫性のないポスト シーケンスです。入力セルが 100,000 未満のサンプルの場合、Ct 値は通常 15 ~ 18 の間で検出されましたが、シーケンス処理に十分な積を生成するのに 18 サイクルを超える必要はありません。
DNAタグ付き増幅後、ライブラリを精製し、サイズ選択して、NextGenシーケンシング用に200~1,000 bpの範囲の理想的なサイズ分布を得ました。DNA断片の85%以上が200~1,000bpの範囲であったときに最良のシーケンシングデータが得られたため、各ライブラリのサイズ分布評価が完了した(図2C)。特に、同量のDNA(蛍光定量法で測定)がロードされたように、一般的に低い蛍光強度のサンプルが一般にあまり配列不良に見舞われた(図2C)。
エンコード ChIP-Seq ガイドラインに基づくシーケンス処理後の標準の品質管理が5、14、15に適用されました。
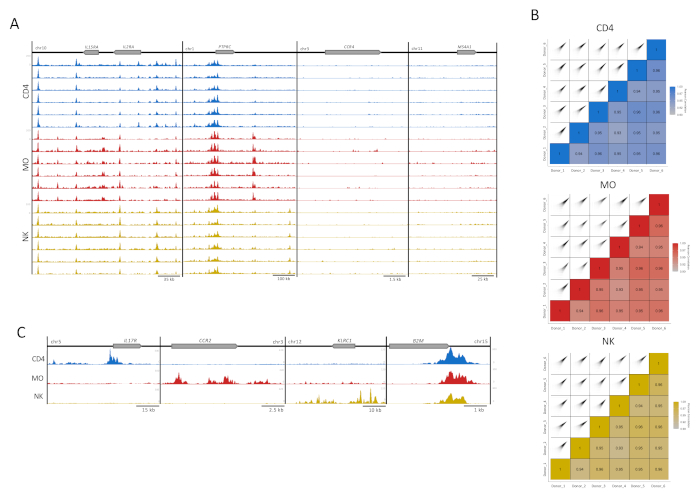
図3:免疫細胞サンプルの再現性。(A)H3K27acトラック(UCSCゲノムブラウザ、最大強度、4の平滑化機能、すべて均等にスケーリングされたY軸)を各細胞タイプ(CD4、MO、およびNK)の6ドナー(複製あたり100,000細胞)4つの例示的な遺伝子座が示されているが、2つは(IL2RA遺伝子座およびPTPRC)を有し、2つはH3K27ac(CCR4およびMS4A1)に対して濃縮されていない。(B)細胞タイプごとにMEDIPSパッケージ内で300bp拡張と500bpウィンドウを用いて生成されたドナーと対応する相関プロットとの間のピアソン相関は16を複製する。(C)細胞タイプ特異的領域におけるH3K27acトラック(UCSCゲノムブラウザ最大強度、4の平滑化機能)を示す各細胞タイプのドナーファイルをマージし、全ての細胞タイプに存在するハウスキーピング遺伝子B2M(CD4用IL17R、MO用CCR2、およびKLRC1)この図の大きなバージョンを表示するには、ここをクリックしてください。
視覚品質管理のために、UCSCゲノムブラウザに表示するH3K27ac濃縮トラックを用意した。4つの遺伝子遺伝子座について、各サンプルの個々のトラックは、我々のアッセイの高い一貫性と堅牢性を反映した高いマッピング品質と信号対雑音比を示した(図3A)。左に2つの座座は、これらの細胞型においてよく発現した遺伝子を収容し、一方、右に2つの遺伝子は発現せず、バックグラウンドコントロール13として機能する(図3A)。また、MEDIPS分析パッケージを、技術複製(図3B)5、16、17の間の相関指数を評価するために、500bpビン16に対する読み込み濃縮レベルの相関度を確立するために、シーケンス後変数として使用した。ペアワイズ比較の大部分について、ピアソン相関指数は、生物学的複製間の高い一定性を示唆する90%以上の相関を示した(図3B)。許容可能な相関を持つ反復が結合され、信号対雑音比が増加した。細胞型特異的な遺伝子は適切な細胞に高い濃縮を示したが、家事遺伝子(B2M)は非常に一貫したヒストン修飾を示した(図3C)。解析の場合、反復からトラックをマージすると、濃縮が増加し、重要な細胞型特異的エンハンサーを含む特定の信号を補強し、ヒトサンプル5に固有の個人間変動性を低減する。
この研究には10万個の細胞が使用されたが、ヒト培養T細胞株(HUT78)では1万個の細胞に対する再現性が高かった。100,000未満の細胞を有するサンプルから行われたChIP-Seqデータセット間の相関分析は、10,000細胞まで高い再現性と相関を示した(図4A)。
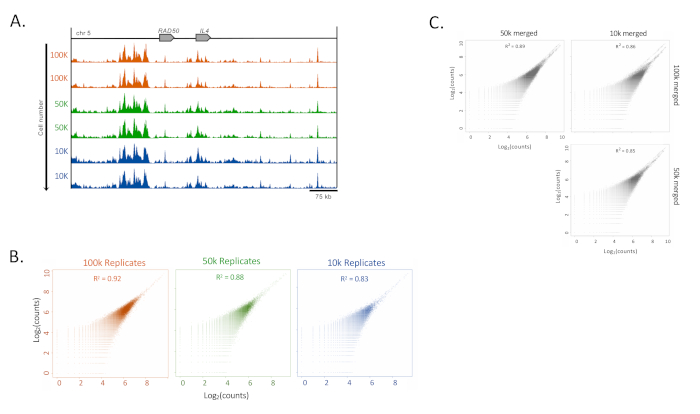
図4: 低入力サンプルの再現性. (A) 細胞100,000個のH3K27ac ChIP-Seqの一貫性を、HUT-78細胞(T細胞リンパ腫細胞株)において100,000個まで下げた例。トラック(UCSCゲノムブラウザ、最大強度、4の平滑化機能、すべて均等にスケーリングされたY軸)は、IL4軌跡を示しています。(B) MEDIPS パッケージ16内で 300 bp 拡張および 500 bp ウィンドウを使用した複製のピアソン相関 (C)(B)16と同じMEDIPSパラメータを用いて、異なる細胞数群(100,000、50,000、および10,000細胞)間のピアソン相関。この図の大きなバージョンを表示するには、ここをクリックしてください。
ピアソン相関分析は高相関指数(83%~92%)を示し、低細胞数サンプルにおけるシグナルの維持を示唆した。しかし、相関係数の低下と同様に細胞数が減少するにつれて背景が増加した(図4B)。低いバックグラウンド信号を維持するために、技術的な重複がマージされ、グループ間の相関がテストされました(図4C)。
| 10X細胞固定バッファー | |
| 化合物 | 最終濃度 |
| ホルムアルデヒド溶液 | 11% |
| ナクル | 100mM |
| EDTA, pH 8.0 | 1 mM |
| EGTA, pH 8.0 | 0.5 mM |
| ヘペス、pH 7.5 | 50mM |
| 完全なリシスバッファー | |
| 化合物 | 最終濃度 |
| トリス HCI, pH 8.0 | 50mM |
| EDTA, pH 8.0 | 10mM |
| SDS | 0.25% |
| ブティレートナトリウム | 20mM |
| プロテアーゼ阻害剤カクテル | 1X |
| 短期のリシスバッファー | |
| 化合物 | 最終濃度 |
| トリス HCI, pH 8.0 | 50mM |
| EDTA, pH 8.0 | 10mM |
| SDS | 0.25% |
| タグメンテーションミックス | |
| 化合物 | 最終濃度 |
| トリス HCI, pH 8.0 | 10mM |
| MgCl2 | 5 mM |
| N,N-ジメチルホルムアミド | 10% |
| イルミナタグメンテーション酵素 | 1:24 vol:vol |
| CtDミックス | |
| 化合物 | サンプル当たり (μL) |
| ネクストエラインデックスプライマーA(25μM) | 0.275 |
| ネクストエラインデックスプライマーB(25 μM) | 0.275 |
| 2X KAPA ハイファイ ホットスタート レディ ミックス | 2.75 |
| 1:1000 SYBRグリーン染料 | 0.11 |
| ROXパッシブ染料 | 0.11 |
| 水 | 4 μLまで充填 |
| AMPミックス | |
| 化合物 | サンプル当たり (μL) |
| 2X KAPA ハイファイ ホットスタート レディ ミックス | 27.5 |
| 水 | 31 μLまで充填 |
補足表 1: バッファーレシピ。
補足表2:6ドナーおよび各細胞タイプのスピアマンおよびピアソンのサンプル相関こちらの表をダウンロードしてください。
ディスカッション
ここで説明する方法は、プロトコルを自動化およびマイクロスケーリングすることにより、DNA精製の前にタグメンテーションライブラリ調製プロトコルを実施するChIPmentationation手順11に拡大する。ChIP-Seqの発症以来、必要な細胞数は、ヒストンの約2,000万個の細胞から数百個、さらには単一細胞1、7、10、12、18、19、20、21まで大幅に減少しました。これらの新しく開発された方法は、感受性を高め、稀な臨床細胞集団を5、6、12、17にテストできるようにすることで、シス-調節機構が細胞でどのように機能しているかをより深く理解することを可能にした。たとえば、最近の一般的な手順の 1 つである CUT&TAG は、堅牢で敏感な ChIP-Seq 代替9として呼び出されます。Tn5酵素がAタンパク質に共有結合し、高特異性9のChIP抗体のFc鎖を認識するので、優れたシグナル対雑音比を生み出す。Tn5酵素のバックグラウンド活性は、標的抗体9に結合する前に酵素が機能しなくなるので低下する。しかし、臨床文脈におけるこの方法の実施は、非固定の生細胞を必要とするため、限定的である。また、低張核からのDNA断片の除去は、アッセイ中に除去されるクロマチンに悪影響を及ぼす可能性がある。新生細胞と連携するために必要な要件は、5を収集するために大きなコホートが何年もかかることがあるので、希少な臨床サンプルとサンプルの大規模なコホートのための問題の源です。別のタイプの方法は、ドロップ-ChIP、マイクロ流体デバイスを使用して、ChIP19を処理する前に液滴ベースのタグメンションを生成する。しかし、高度に特殊化されたマイクロ流体装置を使用し、単一細胞のChIP-Seqを完成させることは可能である一方で、生細胞7、8、9、18、19の使用にも限定される。PLAC-SeqやHiChIPなどのChIP-Seqに依存する新しい方法は、ChIP-Seqピーク22,23間の3次元(3D)相互作用を理解しようとする。これらの3D法は、ゲノム全体にわたるシス-調節またはTF媒介相互作用を同定し、細胞の種類、健康な組織および疾患の文脈における遺伝子発現の調節をよりよく理解しているので、エキサイティングです。
超音波クロマチンの品質や抗体の品質など、プロトコルが成功するために考慮すべき重要なステップがいくつかあります。剪断効率が重要であり、クロマチンがうまく超音波処理されない場合、アッセイの効率は24を大幅に低下させる。超音波処理は、必要なセル番号に起因するChIP-Seqの挑戦的な側面です。プロトコルで使用される超音波処理器では、効率が30万細胞の下で大幅に減少しました。これは、そのレベルで超音波処理するChIP-Seqの挑戦的な側面であり、多くの場合、あまり公平ではない酵素的断片化を必要とします。その結果、超音波処理は真のマイクロスケールのChIP-Seqの主要な限られた要因です。他の超音波処理プラットフォームと市販のキットは、クロマチンを超音波処理するためにテストされましたが、ここで使用される超音波処理器は、最も堅牢で再現可能な結果を持っていました。超音波処理器のもう一つの利点は、大量のサンプルを扱うときにコストを削減する超音波処理を実行するために特殊なチューブを購入する必要はありません。最適な超音波処理のために、まず、上記のように超音波処理器を事前に温めておく事が重要です。第二に、ペレットを分解するには、ピペットチップがチューブの底部に触れ、より物理的な制約を持つ細胞を分解するようにlysingすることが推奨されます。第三に、超音波処理の前に任意のバブル形成は、均一に超音波処理されるサンプルの能力を妨げる。ライシス中に泡が形成されている場合は、ピペットで除去することが重要です。これは、サンプルを多く取り除くことなく難しい場合がありますが、先端が軽くバブルに押し付けられると、サンプルを失うことなくゆっくりと描くことができます。最後に、サイクル数を決定する際に、3サイクルごとに、サンプルを除去し、精製し、アガロースゲル上で実行するタイムコースを完了する。これは、ChIP効率を低下させるので、サンプルの超音波処理のオーバー/アンダーを避けます。サンプルが超音波処理されている場合、大きな断片はChIP-Seq品質24に悪影響を及ぼす可能性があります。一方、サンプルが超音波処理を超えた場合、その過程で標的エピトープが失われる危険性があります。
ChIP-Seqのもう一つの重要な部分は、抗体の品質です。大規模な試験を行う前に、使用する抗体を最適化する必要があります。目標は、ゲノムの既知の領域のノイズ比に対して有意に高い信号を得るものであり、もう一つは再現性である。抗体が多くのバックグラウンドシグナルを引っ張っている場合は、より大きな入力を使用するか、別のロット/サプライヤーを試してみるのが推奨される場合があります。これは大規模な実験を開始する前に時間を追加しますが、それは不可欠なステップです。シグナル・ツー・ノイズをテストするには、抗体の標的であることが知られている領域と、存在しない別の領域でqPCRを使用することをお勧めします。ヒストンの変更は、TFよりも堅牢で最適化が容易であることがわかります。
上述のプロトコルは、半自律的なマイクロスケール方式でハイスループットヒストン修飾ChIP-Seqに対する堅牢な方法を提供する。この方法は、ハンズオン時間の量を制限し、手動ChIP-Seqよりも再現性を高める。研究室で完了した以前の研究は、技術的な複製に関する手動ChIPを使用し、スピアマン相関平均0.505を得たが、しかし、半自動化システムでは、NK細胞平均0.66を有する異なるドナー間のスピアマン相関(補足表2)。これはまた、約40%少ない実践的な時間で完了しました。ここで説明する方法はヒストン修飾(H3K27acはこちらに示されていますが、プロトコルは他の人には変更を必要としません)、TF ChIP-Seqに実装するためにわずかな変更しか必要としません。抗体の品質にもかかわらず、主な変更は超音波処理時間のためであり、潜在的にIPの間に使用されるバッファです。通常、TF ChIPアッセイの場合、TF:DNA複合体は厳密な超音波処理6を通じて維持されにくいように、この方法はクロマチンのわずかに長い断片(約350〜800 bpの範囲)でより良く働く可能性がある。また、TFs は histone の変更とは異なる動作をする可能性があるため、バッファーをカスタム ミックスや他の業界で利用可能なキットに変更する必要がある場合もあります。
自動化されたChIP液剤は10,000個の細胞に対してテストされたが、クロマチン濃度の低下で再現性が著しい低下した。このため、このプロトコルは10,000未満のセルに推奨されておらず、100,000個のセルが最適な条件でした。このプロトコルは業界のChIPバッファを使用して完成しましたが、これは追加費用でしたが、より高品質なデータを提供しました。プロトコルは、超音波処理条件に関して変更することができ(シアレクロマチンが同じ範囲内に保たれている限り)、バッファーは免疫沈降(IP;最適化が必要である可能性がある)のためにカスタマイズすることができ、ChIP液体ハンドラは使用されない可能性があります。プロトコルの制限は、ChIP 液体ハンドラの使用であり、これは高価な投資であり、一度に 16 サンプルしか実行できません。ChIP液剤は小規模な反応に限定され、100万個を超える細胞数はお勧めできません。しかし、プロトコルは、IPと洗浄の手順を手動で完了することによって、それがなくても完了することができます。IPとワッシュを手作業で完了すると、アッセイの完了時間が長くなり、再現性が低下する可能性がありますが、このガイドは高品質のChIP-Seq実験を実行するのに役立ちます。注意すべきは、他の液体ハンドラは、半自動化されたChIP反応を実行するように適応することができる。
要約すると、IPと洗浄のステップが自律的に完了するため、このシステムの主な利点は、高スループットの性質です。そのため、ChIP実験の順次ラウンドを完了し、5日間で最大48個のサンプルを完全に処理してシーケンシングの準備を整え、手動のChIP-Seq実験と比較してハンズオン時間が制限されます。もう1つの利点は、ChIP-Seqが再現性の高い結果を得ることが困難な場合があるため、再現性の向上です。他の方法は、生細胞、複雑なマイクロピペットシステム、または作業をすべて手作業で完了する必要があります。このシステムは、低入力サンプル(<10,000細胞)に最適化する必要があり、最終的には単一細胞のChIP反応を可能にします。システムはまた、PLAC-SeqおよびHiChIP22、23のような新しいChIP方法のために適応することができる。
開示事項
著者らは開示するものは何もない。
謝辞
ヴィジャヤナンド研究所のメンバーに技術的な助けと建設的な議論、ディアーゲノードのシャロン・スクワッツォ博士とジェフリー・ベルゲ氏に、超音波処理機とChIP液体ハンドラマシンとプロトコルの技術支援を感謝します。この研究は、NIH助成金AI108564、R01HL114093、S10RR027366(BDファカリアII)、およびS10OD016262(イルミナHiSeq 2500)によってサポートされました。
資料
| Name | Company | Catalog Number | Comments |
| 200 µl tube strips (8 tubes/strip) + cap strips | Diagenode | C30020002 | Strip tubes for use on the IP Star; ChIP 8-tube strip |
| AMPure XP for PCR Purification | Beckman Coulter | A63880 | SPRI beads |
| Axygen 0.6 mL MaxyClear Snaplock Microcentrifuge Tube | Corning | MCT-060-C | 0.65 mL low binding tube |
| Bioruptor Pico Sonicator | Diagenode | B01060010 | Sonicator used in the lab but others can be used |
| ChIP DNA Clean & Concentrator (Capped Columns) | Zymo Research | D5205 | DNA clean-up kit |
| Dynabeads Protein A for Immunoprecipitation | ThermoFisher | 10001D | |
| EDTA (0.5 M), pH 8.0, RNase-free | ThermoFisher | AM9260G | |
| EGTA pH 8.0 | Millipore Sigma | E3889-25G | |
| Eppendorf ThermoMixer C | Eppendorf | 2231000667 | |
| Formaldehyde solution | Millipore Sigma | 252549-1L | |
| Glycine | Millipore Sigma | 50046-250G | |
| H3K27ac polyclonal antibody - Premium | Diagenode | C15410196 | |
| HEPES (1 M) pH 7.5 | ThermoFisher | 15630080 | |
| IDT for Illumina Nextera DNA Unique Dual Indexes | Illumina | 20027213 | |
| Illumina Tagment DNA Enzyme and Buffer Small Kit | Illumina | 20034197 | |
| IP-Star Compact Automated System | Diagenode | B03000002 | Automated system for ChIP-Seq studies; ChIP liquid handler |
| KAPA HiFi HotStart ReadyMix | Roche | KK2601 | PCR mix |
| Medium reagent container for SX-8G IP-Star Compact | Diagenode | C30020003 | |
| MgCl2 (magnesium chloride) (25 mM) | ThermoFisher | R0971 | |
| N,N-Dimethylformamide | Millipore Sigma | D4551-250ML | CAUTION - low flash point |
| NaCl (5 M), RNase-free | ThermoFisher | AM9760G | |
| PBS (10X), pH 7.4 | ThermoFisher | 70011044 | |
| PCR Flex-free 8-tube stripes, attached individual optical caps | USA Scientific | 1402-4700 | 8 strip tubes, 0.2 mL 8-tube strip |
| Proteinase Inhibitor Cocktail | Millipore Sigma | P8340 | |
| Proteinase K Solution (20 mg/mL), RNA grade | ThermoFisher | 25530049 | |
| PureLink RNase A (20 mg/mL) | ThermoFisher | 12091021 | |
| Quant-iT PicoGreen dsDNA Reagent | ThermoFisher | P7581 | Used in the flourescence quantification |
| QuantStudio 6 Flex Real-Time PCR System | ThermoFisher | 4485699 | qPCR |
| ROX Reference Dye | ThermoFisher | 12223012 | |
| Sodium butyrate | Millipore Sigma | 303410-100G | |
| SYBR Gold Nucleic Acid Gel Stain (10,000X Concentrate in DMSO) | ThermoFisher | S11494 | nucleic acid dye |
| SYBR Green I Nucleic Acid Gel Stain - 10,000X concentrate in DMSO | ThermoFisher | S7563 | |
| TE Buffer | ThermoFisher | 12090015 | |
| Tips (bulk) | Diagenode | C30040020 | Tips for the IP Star |
| True MicroChIP Kit | Diagenode | C01010130 | Contains all the buffers for the IP; ChIP kit |
| UltraPure 1M Tris-HCI, pH 8.0 | ThermoFisher | 15568025 | |
| UltraPure SDS Solution, 10% | ThermoFisher | 24730020 |
参考文献
- Barski, A., et al. High-resolution profiling of histone methylations in the human genome. Cell. 129 (4), 823-837 (2007).
- Johnson, D., Mortazavi, A., Myers, R., Wold, B. Genome-Wide Mapping of in Vivo Protein-DNA Interactions. Science. 316, 1497-1502 (2007).
- Mikkelsen, T. S., et al. Genome-wide maps of chromatin state in pluripotent and lineage-committed cells. Nature. 448 (7153), 553-560 (2007).
- Furey, T. S. ChIP-seq and beyond: new and improved methodologies to detect and characterize protein-DNA interactions. Nature Reviews Genetics. 13 (12), 840-852 (2012).
- Seumois, G., et al. Epigenomic analysis of primary human T cells reveals enhancers associated with TH2 memory cell differentiation and asthma susceptibility. Nature Immunology. 15 (8), 777-788 (2014).
- Schmiedel, B. J., et al. 17q21 asthma-risk variants switch CTCF binding and regulate IL-2 production by T cells. Nature Communication. 7, 13426(2016).
- Ai, S., et al. Profiling chromatin states using single-cell itChIP-seq. Nature Cell Biology. 21 (9), 1164-1172 (2019).
- Brind'Amour, J., et al. An ultra-low-input native ChIP-seq protocol for genome-wide profiling of rare cell populations. Nature Communication. 6, 6033(2015).
- Kaya-Okur, H. S., et al. CUT&Tag for efficient epigenomic profiling of small samples and single cells. Nature Communication. 10 (1), 1930(1930).
- Youhanna Jankeel, D., Cayford, J., Schmiedel, B. J., Vijayanand, P., Seumois, G. An Integrated and Semiautomated Microscaled Approach to Profile Cis-Regulatory Elements by Histone Modification ChIP-Seq for Large-Scale Epigenetic Studies. Methods Molecular Biology. 1799, 303-326 (2018).
- Schmidl, C., Rendeiro, A. F., Sheffield, N. C., Bock, C. ChIPmentation: fast, robust, low-input ChIP-seq for histones and transcription factors. Nature Methods. 12 (10), 963-965 (2015).
- Skene, P. J., Henikoff, S. An efficient targeted nuclease strategy for high-resolution mapping of DNA binding sites. Elife. 6, 21856(2017).
- Schmiedel, B. J., et al. Impact of Genetic Polymorphisms on Human Immune Cell Gene Expression. Cell. 175 (6), 1701-1715 (2018).
- Landt, S. G., et al. ChIP-seq guidelines and practices of the ENCODE and modENCODE consortia. Genome Research. 22 (9), 1813-1831 (2012).
- Chen, L., et al. Genetic Drivers of Epigenetic and Transcriptional Variation in Human Immune Cells. Cell. 167 (5), 1398-1414 (2016).
- Lienhard, M., Grimm, C., Morkel, M., Herwig, R., Chavez, L. MEDIPS: genome-wide differential coverage analysis of sequencing data derived from DNA enrichment experiments. Bioinformatics. 30 (2), 284-286 (2014).
- Engel, I., et al. Innate-like functions of natural killer T cell subsets result from highly divergent gene programs. Nature Immunology. 17 (6), 728-739 (2016).
- Grosselin, K., et al. High-throughput single-cell ChIP-seq identifies heterogeneity of chromatin states in breast cancer. Nature Genetics. 51 (6), 1060-1066 (2019).
- Rotem, A., et al. Single-cell ChIP-seq reveals cell subpopulations defined by chromatin state. Nature Biotechnology. 33 (11), 1165-1172 (2015).
- Wang, Q., et al. CoBATCH for High-Throughput Single-Cell Epigenomic Profiling. Molecular Cell. 76 (1), 206-216 (2019).
- Consortium, E. P. An integrated encyclopedia of DNA elements in the human genome. Nature. 489 (7414), 57-74 (2012).
- Fang, R., et al. Mapping of long-range chromatin interactions by proximity ligation-assisted ChIP-seq. Cell Research. 26 (12), 1345-1348 (2016).
- Mumbach, M. R., et al. HiChIP: efficient and sensitive analysis of protein-directed genome architecture. Nature Methods. 13 (11), 919-922 (2016).
- Diagenode. The Ultimate Guide for Chromatin Shearing Optimization with Bioruptor Standard and Plus. Diagenode. , (2012).
Erratum
Formal Correction: Erratum: A Semiautomated ChIP-Seq Procedure for Large-scale Epigenetic Studies
Posted by JoVE Editors on 9/14/2020. Citeable Link.
An erratum was issued for: A Semiautomated ChIP-Seq Procedure for Large-scale Epigenetic Studies. An author's name was updated.
The name was corrected from:
Pandurangan Vijayanad
to:
Pandurangan Vijayanand
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved