Method Article
Büyük Ölçekli Epigenetik Çalışmalar için Yarı Otomatik ChIP-Seq Prosedürü
Bu Makalede
Erratum Notice
Özet
Bu makalede, ChIP sıvı işleyici platformu kullanılarak belirli bir histone modifikasyonu (H3K27ac) ile ilişkili DNA bölgelerinin yarı otomatik, mikro ölçekli ChIP-Seq protokolü ve ardından etiketleme kullanılarak kütüphane hazırlığı açıklanmaktadır. Prosedür, kalite ve nicelik amaçları için kontrol değerlendirmesini içerir ve diğer histone modifikasyonlarına veya transkripsiyon faktörlerine benimsenebilir.
Özet
Kromatin immünorepitasyonu ve ardından dizileme (ChIP-Seq), CIS düzenleyici DNA elementlerinin tanımlanmasına yardımcı olmak için H3K27ac gibi spesifik histone modifikasyonları ile ilişkili kromatin DNA'sını profillemek için güçlü ve yaygın olarak kullanılan bir yaklaşımdır. ChIP-Seq'i tamamlamak için manuel işlem emek yoğundur, teknik olarak zordur ve genellikle büyük hücreli sayılar (>100.000 hücre) gerektirir. Burada açıklanan yöntem, bu zorlukların üstesinden gelmeye yardımcı olur. 100.000'den az hücre sayısı girişi için 48 örnekten oluşan bir grup için hücre fiksasyonu, kromatin kesme, immünorepitasyon ve sıralama kütüphanesi hazırlama dahil olmak üzere tam yarı otomatik, mikro ölçekli H3K27ac ChIP-Seq prosedürü ayrıntılı olarak açıklanmıştır. Yarı otomatik platform teknik değişkenliği azaltır, sinyal-gürültü oranlarını iyileştirir ve işçiliği büyük ölçüde azaltır. Sistem böylece reaksiyon hacimlerinin azalmasına izin vererek, enzimler, manyetik boncuklar, antikorlar ve uygulamalı süre gibi pahalı reaktiflerin sayısını sınırlayarak maliyetleri azaltabilir. ChIP-Seq yöntemindeki bu iyileştirmeler, sınırlı hücre sayısına sahip klinik örneklerin yüksek oranda tekrarlanabilir bir şekilde büyük ölçekli epigenetik çalışmaları için mükemmel bir şekilde uygundur.
Giriş
ChIP-Seq tahlillerinin belirli histone modifikasyonlarıyla ilişkili DNA parçalarını belirlemek için geniş kullanımı, kısmen aktif arttırıcılar, promotörler, susturucular, heterokromatin ve diğerleri de dahil olmak üzere cis düzenleyici DNA elementlerini tanımlama yeteneğinden kaynaklanmaktadır1,2,3,4. Genom genelinde kodlamayan düzenleyici bölgelerin tanımlanması, sağlık ve hastalıklarda gen düzenlemesini daha iyi anlamak için değerli içgörüler göstermiştir4. Laboratuvardan önceki çalışmalar, cis-düzenleyici unsurların farklı hücre tiplerinde önemli roller oynayabileceğini göstermek için ChIP-Seq'i kullanmıştır5. Transkripsiyon faktörü (TF) ChIP tahlilleri hastalık ilişkili risk tek nükleotid polimorfizmlerini göstermek için kullanılmıştır6.
ChIP-Seq'in insan klinik örnekleriyle kullanımı, esas olarak hücre sayılarının veya istenen doku örneğinin sınırlandırılması nedeniyle zordur. Sonuç olarak, bu teknikleri geliştirmek ve mikro ölçekli hale getirmek için sahada birlikte bir çaba gösterilmiştir ve sonuç olarak CUT&TAG 5 , 7 , 8 ,9,10,11,12gibi çeşitli tahliller ortaya çıkmıştır. Bu tahlil, belirli bir antikor9ile bağlanmış genomik bölgeleri etiketlemek ve izole etmek için bir transposaz kullanır. Bu teknik hücre sayılarını 1.000'lere ve bazı durumlarda tek hücreye düşürmeyi başarmıştır, ancak bu tekniğin çevirisel araştırma ve klinik kurulumda kullanılması, bu yöntem için canlı hücre kullanma gereksinimleri nedeniyle sınırlamalar göstermiştir9,12. Canlı hücre gereksinimi, klinik örneklerin işlenmesini lojistik olarak zorlaştırır ve numuneler aynı anda işlenmezse toplu iş efektlerini ortaya getirebilir. Diğerleri formaldehit sabit hücreler için mikro ölçekli teknikleri optimize etti, ChIPmentation11'in geliştirilmesi de dahil olmak üzere , burada yüksek verimli bir şekilde uyarlanmıştır. Sabit hücrelerin kullanımı, toplu iş etkilerini en aza indirmek için tüm örneklerin toplanmasına ve daha sonra işlenmesine kadar örneklerin depolanmasını sağlar.
Burada, histone modifikasyonlarının profilini çıkarmak için deneysel uygulamalı zamanı azaltan yarı otomatik mikro ölçekli chIP-Seq tahlili açıklanmaktadır10. Yarı otomatik yöntem, yüksek verimli ChIP-Seq testlerine izin vererek, chip sıvı işleyicisi kullanarak numune başına 10.000 hücre kadar kısa bir süre için 48 numunenin 5 gün gibi kısa bir sürede tamamen işlenmesini ve sıralamaya hazır olmasını sağlar. İşleyici immün özürlülük (IP) tamamlar ve daha sonra numuneler arasındaki değişkenliği azaltmaya yardımcı olan otonom bir şekilde yıkanır. Yarı otomatik yöntem, 48 örnek için hem uygulamalı zamanı 15 saatin üzerinde hem de teknik değişkenliği azaltarak, büyük ölçekli epigenetik çalışmaların birincil veya kültürlü hücreler için tekrarlanabilir ve hızlı bir şekilde yürütülmesini sağlar. Protokol, yüksek kaliteli ChIP-Seq için baştan sona süreci açıklar. Belirli makineler kullanılamıyorsa, protokol ChIP-Seq denemelerini el ile ayarlamak ve sorun çekmek için yararlı bir kaynak olmaya devam eder.
Test üç farklı primer insan immün hücre tipi ve bir kültürlü hücre hattı ile gerçekleştirildi (HUT78 – ATCC: TIB-161). Netlik için, protokol yedi bölüme ayrılmıştır: hücre fiksasyonu, sonikasyon yoluyla kromatin kesme, otomatik kromatin immün önksepitasyon, DNA parça etiketlemesi ile kütüphane hazırlama, kütüphane amplifikasyonu, kütüphane saflaştırma, ardından DNA nicelemesi. Tampon tarifleri için lütfen Ek Tablo 1'e bakın.
Protokol
La Jolla Alerji ve İmmünoloji Enstitüsü (LJI) Kurumsal İnceleme Kurulu (IRB; IRB protokol no. SGE-121-0714) çalışmayı onayladı. Sağlıklı gönüllüler işe alındı ve yazılı bilgilendirilmiş onaydan sonra lökerez örnekleri sağlandı.
1. Hücre fiksasyonu
- Hücre süspansiyon konsantrasyonu 15 mL tüpte (<10 mL süspansiyon) veya 50 mL tüpte (10-30 mL hücre) tam hücre kültürü ortamı ile 1 ila 2 x 106 hücre /mL'ye getirin. <1 x 106 hücre, 1,5 mL'lik bir tüpte ortamın 0,5 mL'lik kısmını kullanın.
- Hücre süspansiyonunu hafifçe girdaplayın, damla yönünde 10x hücre sabitleme tamponu ekleyin (1:10; vol:vol) ve oda sıcaklığında (RT) 10 dakika boyunca düşük hızda döndürün.
- Reaksiyonu hafifçe girdaplayarak durdurun ve 1:20 (vol:vol) oranında 2,5 M glisin ekleyin. Tüpleri birkaç kez ters çevirin ve en az 5 dakika buzda kuluçkaya yatırın.
- Kalan adımları 4 °C'de veya buzda gerçekleştirin. Tüpleri 4 °C'de 5 dakika boyunca 800 x g'da döndürün ve üst düğmeyi atın.
- Peleti 5 mL buz gibi 1x PBS ile hafifçe yeniden depolayın ve buzda 2 dakika kuluçkaya yatırın.
- 1,4 ve 1,5'i 1 mL buz gibi 1x PBS ile tekrarlayın ve numuneyi önceden soğmuş 1,5 mL'lik bir tüpe aktarın (uzun süreli depolama için etiketlenmiştir). Varsa, aliquots hazırlanması önerilir.
- Tüpleri 4 °C'de 1.200 x g'da döndürün ve hücre peletini bozmadan süpernatanttan mümkün olduğunca çok çıkarın. Peletin sıvı nitrojenle dondurulmasının ardından. -80 °C'de saklayın.
DİkKAT: Sıvı nitrojeni kullanırken uygun korumayı alın.
2. Kromatin kesme
NOT: Bu protokol, 0,65 mL düşük bağlayıcı tüplerde 0,3 ila 3 x 106 hücreli peletlerin kromatin kesmesi için optimize edilmiştir.
- Numune tüpünü -80 °C'den donmuş hücre peletleri ile çıkarın ve lizis tamponu eklemeden önce peletin çözülmesini önlemek için kuru buzda saklayın. Bu adım kritiktir.
- Pelete 70 μL taze, RT komple liziz tamponu ekleyin ve 1 dakika RT'de tutun.
- Herhangi bir kabarcık sokmadan peletin 1 dakika boyunca yeniden kullanılabilir ve ardından numuneyi buza koymadan önce hücre süspansiyonunu RT'de 1 dakika kuluçkaya yatırın.
- Yeniden dirilen peletı 0,65 mL düşük bağlayıcı bir tüpe aktarın ve buzda tutun.
NOT: Tekrarlanabilir sonikasyon elde etmek için, sonicator'u numuneleri sonicating önce 3-6 döngü boyunca sadece boş tüplerle çalıştırarak önceden ısıtın. - Numuneleri sonicator'un tüp tutucusuna yerleştirin ve boşlukları 70 μL su ile doldurulmuş denge tüpleriyle doldurun. Sonikasyona başlamadan önce numuneleri yaklaşık 1 dakika su banyosunda bırakın.
- Döngü başına 16 s ON / 32 s OFF ile x döngüleri için sonication (hücre tipine bağlı olarak) gerçekleştirin.
NOT: Bu adım, verimli sonication için en uygun döngü sayısını belirlemek için doğrulama denemeleri gerektirecektir. - Her 3 döngüden sonra, numuneleri sonicator'dan çıkarın, yavaşça girdap ve nabız, boruları tutucuya geri koymadan önce döndürün. Kabarcık oluşumuna neden olabileceği için tüpün dışında küçük damlacıklar olmadığından emin olun.
- Gerekli döngüleri tamamladıktan sonra, numuneleri 4 °C'de 15 dakika boyunca 14.000 x g > döndürün. Süpernatantı yeni, önceden soğutulmuş, düşük bağlayıcı 0,65 mL düşük bağlayıcı bir tüpe aktarın ve buzda tutun.
- Sonikasyon verimliliğini kontrol etmek için sonikated örneklerin bir kısmını decrosslink.
- Süpernatantın 1-7 μL'lik kısmını (yaklaşık 250 ng yamlu kromatin)'i 0,2 mL PCR tüpüne aktarın ve RT'de kısa süreli lizis tamponu ile hacmi 10 μL'ye kadar yapın.
- 1 μL RNase A ekleyin ve 800 rpm'de 37 °C'de 30 dakika kuluçkaya yaslanın, ardından 1 μL proteinaz K ekleyin.
- 55 °C'de 2 saat kuluçkaya yaslanın ve 1.000 rpm'de sallanın.
- Floresan nicelik testi 10 kullanarak DNA nicelemesi için decrosslinked örneğinin2 μL'sini çıkarın (sabun olarak spektrofotometre önerilmez ve bozulmuş protein sonuçlarda önyargı üretebilir).
- Kalan numuneyi 70 V'ta 1 saat boyunca %1,2 agarose jel üzerinde çalıştırın, nükleik asit boyası (1:20.000) lekesi ve UV transilluminator kullanarak jeli okuyun.
- Kromatin stok aliquots depolama için hazırlayın (tam lizis tampon ile 20 μL'de 25 ng / μL için numuneyi seyreltin). Tüm yamlu kromatinleri -80 °C'de saklayın.
3. Histone modifikasyonu için otomatik ChIP-Seq
NOT: Bu iletişim kuralı chip sıvı işleyicisi üzerinde çalışacak şekilde tasarlanmıştır. Sistem özelleştirilmiş arabellekler kullanabilse de, tüm arabellekler ChIP kiti ile sağlanır. Bu bölümde kullanılan 8 tüplü ChIP şeritleri ChIP sıvı işleyicisine özgüdir.
- 500 ng'lik kesme kromatinli 16 örnek aliquot'u -80 °C'den 20 μL'ye aktarın ve kromatinleri yavaşça eritmek için buza yerleştirin. Tamamen çözüldükten sonra, kısa bir süre girdap ve nabız döndürücü.
- Kromatin hazırlanması
- Pipet 100 μL tC1 tamponu, iki ChIP 8 tüplü şerit halinde 1x proteaz inhibitörü ve 20 mM sodyum bütirat (komple tC1 tamponu) ile desteklenmiştir.
- Her kromatin örneğinin 20 μL'sini, 100 μL komple tC1 tamponu içeren ChIP 8 tüplü şeritlerin uygun bir tüpüne aktarın. Kromatin tüplerine 80 μL komple tC1 tampon ekleyerek kromatin tüplerini yıkayın ve ardından 200 μL'lik son hacim için ChIP 8 tüplü şeritlerin uygun tüpüne geri aktarın.
- Antikorun hazırlanması
- Antikor hacmini, her tüpte 0,5 μg antikor olacak şekilde hesaplayın.
Antikor hacmi = (numune sayısı x reaksiyon başına antikor) / antikor konsantrasyonu - Hesaplanan antikor miktarını 500 μL tBW1 tamponuna ekleyin. Hızlı bir şekilde girdap ve nabız döndürme.
- Pipet 70 μL tBW1 iki ChIP 8 tüplü şeritlerin her birine ve her tüpe 30 μL antikor + tBW1 ekleyin. Bu, tüplerin her birinde toplam hacmi 100 μL'ye getirecektir.
- Antikor hacmini, her tüpte 0,5 μg antikor olacak şekilde hesaplayın.
- Manyetik boncuk hazırlanması
- Protein A boncuk çözeltisini iyice girdap. 0,5 μg antikor için pipet 5 μL boncuk yeni bir ChIP 8 tüplü şeritler ve darbe dönüşü kümesine.
- ChIP sıvı işleyicisinin son satırını etiketli, boş ChIP 8 tüplü şeritlerle doldurun.
- ChIP sıvı işleyici makinesindeki tüm şeritlerin yerleştirilmesi için ChIP-16-IPure-200D program teknik özelliklerini izleyin. Arabellekleri doğru konuma ekleyin, ancak tE1 arabelleği yerine tW4 kullanın.
NOT: Günü ChIP sıvı işleyicisinin ChIP'i bir gecede gerçekleştireceği şekilde düzenleyin. Program 16 örnek için yaklaşık 16 saat çalışacaktır. Bu, 1.
4. Kütüphane hazırlığı için kütüphane adaptörlerinin transposaz entegrasyonu
- Bir termokseri 37 °C ve 500 rpm'ye önceden ayarlayın. Buz üzerinde 0,2 mL tüp şeritleri için bir mıknatısı soğutun.
- 16 numune için buz üzerinde 440 μL etiketleme tamponu hazırlayın. Pipet 53 μL tek bir yeni 8 tüplü şerit içine ve buz üzerinde tutun.
- Yeni bir 0,2 mL 8 tüplü şeritte, 220 μL soğuk tC1 tampon ekleyin ve buz üzerinde tutun. 8 şeritli tüpler bu hacmi tutabilir ve hala kaplanabilir.
- ChIP sıvı işleyici makinesinden (satır 12) "IP örnekleri" şerit tüpünü çıkarın ve darbe eğirme öncesinde tüpleri kaplayın. 2 dakika boyunca 8 tüplü şeritler için mıknatısı kullanarak boncukları yakalayın ve süpernatant'ı dikkatlice çıkarın.
- Etiketleme tamponunun 25 μL'lik kısmını çok kanallı boncuklara aktarın, mıknatıstan çıkarın ve boncuklar homojen olana kadar hafifçe karıştırın (pipet 20 μL'ye ayarlanmış pipetle yaklaşık 5 kat yukarı ve aşağı).
- Tüpleri kaplayın ve önceden ısıtılmış termomikserin içine yerleştirin ve 3 dakika kuluçkaya yatırın. Sürenin artırılması kütüphane hazırlığının verimliliğini azaltacaktır.
- Tüpleri soğutulmuş bir metal rafa aktarın ve her numuneye 100 μL soğutulmuş tC1 tampon ekleyin. Çok kanallı bir pipeti 80 μL'ye ayarlayın ve boncuklar homojen olana kadar numuneyi karıştırın ve etiketleme reaksiyonunu durdurun.
- Numuneleri ChIP sıvı işleyicisine geri yerleştirin ve yıkama prosedürüne Washing_for_IP reacts_16_Ipuredevam edin. Yıkamanın tC1 tampon ile iki kez ve tW4 ile iki kez gerçekleştirilmesini sağlayın. Elution, arabellek tE1 ile program düzeni tarafından işaretlenmiş olarak tamamlanmalıdır.
- DNA'nın decrosslinking
- ChIP sıvı işleyicisinin son satırındaki ChIP 8 tüplü şeritleri çıkarın ve her numuneye 2 μL RNase A ekleyin.
- Tüpleri kaplayın, nabız döndürün, boncukları karışım homojen olana kadar çok kanallı bir pipetle hafifçe karıştırın ve tüpleri yeniden kaplayın.
- Numuneleri 37 °C ve 900 rpm'de 30 dakika boyunca bir termomikserde kuluçkaya yatırın.
- Numuneleri termomikserden çıkarın, 2 μL Proteinaz K ekleyin.
- Numuneleri bir termomikserde 55 °C ve 1.250 rpm'de 4 saat, ardından bir gecede 1.000 rpm'de 65 °C'ye inkübte edin.
NOT: Bu 2.
5. Etiketli DNA parçaları saflaştırma
- Uygun örnek numarasına sahip on altı adet 1,5 mL tüpü etiketleyin ve DNA temizleme kitinden her birine 400 μL DNA bağlama tamponu ekleyin.
- Buharlaştırılmış herhangi bir ürünün muhafazasını sağlamak için 8 tüplü şeritleri termokserden çıkarın ve şeritleri nabız döndürün. Boncukları yakalamak için 8 şeritli bir mıknatısa şeritler yerleştirin.
- 1,5 mL'lik tüplerin her birine 100 μL decrosslinked DNA aktarın. Boncukları yıkamak için 8 tüplü şeritlere 100 μL DNA bağlama tamponu ekleyin ve ardından uygun 1,5 mL tüpe aktarın.
- Yaklaşık 10 sn için girdap ve 1,5 mL tüpleri nabız döndürün.
- Sütunları DNA bağlama tamponu ve ChIP örneğini içeren 600 μL ile yükleyin.
- Örnekleri 10.000 x g'da 20 sn döndürün ve sütunu akışla yeniden yükleyin. Aynı koşullarla tekrar döndürün ve akışı atın.
- Kolonları 200 μL yıkama tamponu (önceki adımla aynı santrifüj) ile iki kez yıkayın ve akışı atın.
- Sütunları 12.000 x g'da 2 dakika santrifüjleyarak kurutun.
- Sütunu yeni bir 1,5 mL toplama tüpüne aktarın ve doğrudan sütun matrisine 9 μL sıcak TE Tamponu (önceden 55 °C'ye ısıtılmış) ekleyin. Sütunun santrifüjlemeden önce 1 dakika boyunca 10.000 x g'da1 dakika kuluçkaya yatmasına izin verin.
- 9 μL'lik eluteyi uygun yeni bir 8 tüplü şerit setine aktarın.
- Daha önce olduğu gibi 8 μL TE Tampon ile işlemi tekrar tamamlayın. Elute'yi uygun 8 tüplü şeritlere (numune başına son hacim 17 μL) aktarın ve buz üzerinde tutun.
6. Saflaştırılmış numunelerin amplifikasyonu ve boyut seçimi
- Aşağıdaki adımlar, optimum amplifikasyon için gereken döngü sayısını belirlemek için qPCR kullanır (CtD – Ct tayini)
- CtD Mix Buffer içeriğini örnek sayısıyla çarparak ctd karışımını tüm numuneler için hazırlayın.
- 3.6 μL CtD karışımını bir qPCR plakasına dağıtın ve 1.4 μL etiketli DNA örnekleri ekleyin (toplam hacmin ~ % 10'u). Aşağıdaki qPCR'yi gerçekleştirin: 3 dakika için 98 °C, 5 dakika için 72 °C, 30 s için 98 °C, 10 s için 98 °C 26 döngü, 30 s için 63 °C ve 30 s için 72 °C.
- AMP Mix Buffer içeriğini örnek sayısıyla çarparak Amp Mix'i tüm numuneler için hazırlayın. 14 μL etiketli DNA'yı bir PCR plakasının ayrı kuyularına dağıtın, ardından her numuneye 2,5 μL iki dizin astarı (25 μM) ekleyin (son reaksiyon hacmi 50 μL'dir).
- Numuneleri çok kanallı pipet ile karıştırın ve CTD'de kullanılan amplifikasyon programını uygun sayıda döngü ile gerçekleştirin.
NOT: Güçlendirilmiş numuneler birkaç hafta boyunca -20 °C'de saklanabildiği için bu iyi bir durma noktasıdır. Ancak, saflaştırma aynı gün tamamlanabilir. 48 örnek için, 3 - 6.5 adımları diğer iki ayrı grupla tamamlandı ve daha sonra aşağıda açıklandığı gibi tek bir partide yükseltildi. - Aşağıda açıklandığı gibi etiketlenmiş DNA'nın yükseltilme sonrası, boyut seçimi ve nicelemesini gerçekleştirin. Bu genellikle 48 örnekle tamamlanabilir (istenildiği gibi daha az örnekle tamamlanabilir).
- Her kuyuya 90 μL paramanyetik boncuk (1:1,8 oranı) ekleyin, karıştırın ve RT'de 2 dakika kuluçkaya yatmasına izin verin.
- Bir plaka mıknatısı kullanarak boncukları yakalayın ve süpernatant atın. Boncuk peletini bozmadan boncukları 200 μL taze% 80 etanol ile 3 kez yıkayın.
- Son yıkamadan sonra 20 μL uçlu fazla etanolleri çıkarın ve boncukları 10 dakika boyunca veya boncuk peletlerinde çatlaklar görünene kadar kurumaya bırakın.
- Plaka hala mıknatıstayken, her kuyuya önceden ısıtılmış suyun 40 μL'sini ekleyin. Plakayı, girdabı iyice kapatın ve plakayı kısa bir süre nabız döndürün.
- Plakayı mıknatısın üzerine geri yerleştirerek boncukları yakalayın ve 40 μL elute'yi yeni bir "örnek" Plakaya aktarın. Numuneler şimdi saflaştırılmıştır ve sonraki adımlar 200-1.000 bp arasında değişen parçaları zenginleştirir.
- İsteğe bağlı QC adımı: Numunelerden 4 μL çıkarın ve bir QC plakasına aktarın. Numunelere 4 μL su ekleyin. Bu, büyük parçaların yüzdesini belirler.
- Örneklere 22 μL paramanyetik boncuk (1:0,55 oranı) ekleyin, dikkatlice karıştırın ve RT'de 2 dakika kuluçkaya yatırın.
- Boncukları 5 dakika boyunca yakalamak için mıknatıs üzerine yerleştirin ve süpernatantları "örnek" plakanın 7-12 kolonlarına aktarın. Plakayı mıknatıstan çıkarın ve 30 μL boncuk ekleyin (son oran 1:1.3). Dikkatlice karıştırın ve RT'de 2 dakika oturmasına izin verin.
- Boncukları 5 dakika boyunca yakalayın ve ardından süpernatant atın.
- Tüm boncukları daha önce açıklandığı gibi 200 μL taze % 80 etanol ile 3 kez yıkayın (adım 6.6.2 – 6.6.3).
- Peletler kurudıktan sonra, mıknatıs üzerindeyken her kuyuya 8 μL önceden ısıtılmış TE tamponu ile DNA elute edin.
- Plakayı mıknatıstan, mühürden ve girdaptan iyice çıkarın. Plakanın RT'de 2 dakika kuluçkaya yatmasına izin verin, nabız döndürün ve plakayı 2 dakika boyunca mıknatısın üzerine geri yerleştirin. Süpernatant'ı yeni bir tabağa aktarın (Plaka 2).
- Maksimum geri kazanım için, ekstra 8 μL önceden ısıtılmış TE tamponu ile tekrarlayın. Örnekleri, her numunenin 16 μL son kütüphaneye sahip olacak şekilde uygun kuyulara yerleştirin.
NOT: Bu adımın sonunda iki plaka olmalıdır (QC plakası tamamlanmışsa bir tane). QC plakası önceden seçilmiş parçalara sahip olacak ve ikinci plaka 48 kuyu final kütüphanesine sahip olmalıdır (toplam 16 μL).
7. Floresan tahlili kullanarak nihai kütüphaneleri ve QC örneklerini ölçün
- Floresan nicel test veya benzer bir yöntem kullanarak tam DNA nicelemesi.
- QC nicelemesi tamamlandıysa, 1.000 bp'< olan numune kaybının yüzdesini belirleyin. Yaklaşık% 20'den fazla kayıp olmamalıdır - daha fazlası varsa, uygulanan boncuk oranlarıyla ilgili bir sorun olabilir.
- Tercihen bir kılcal elektroforez makinesi kullanarak her numunenin parçalarının boyutunu belirleyin. Molar konsantrasyonu hesaplamak için aşağıdaki denklemi kullanın: [Kütüphane konsantrasyonu (ng/μL) * 106]/[660 * Ortanca parça boyutu (bp)].
NOT: Kitaplıklar, standart yeni nesil sıralama yordamlarını izleyerek birikmeye (eşitlikçi tutarlar) ve sıraya konmaya hazırdır.
Sonuçlar
Kavramın kanıtı olarak, ChIP-Seq üç immün hücre tipine sahip altı insan bağışçısı için tamamlandı: naif CD4 T hücreleri (CD4), klasik monositler (MO) ve doğal öldürücü hücreler (NK),13'denönce açıklandığı gibi FACS sıralama ile zenginleştirilmiş . Altı çizili yordam Şekil 1'detemsil edildiği gibi dokuz ayrı yordamdan oluşur.
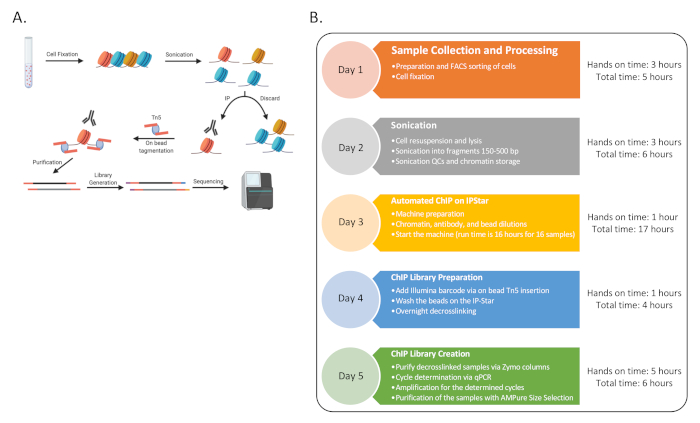
Şekil 1: Prosedür için Genel Akış Çizelgesi. (A) Genel prosedürün bir karikatürü (BioRender'da oluşturulur). (B) Protokol için tüm ana adımlar ve her günle ilişkili tahmini uygulamalı ve toplam süre için akış şeması. Sıralama, 5. Zaman çizelgesi, sıralı Gün 3-4'ün 48 ChIP örneği oluşturmak için bir haftada birden fazla kez tamamlanabileceği hafta boyunca da kademelendirilebilir. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
Akış sitometrisi13ile hücre izolasyonundan sonra, sıralanmış hücreler santrifüjlendi ve hücreler yukarıda açıklandığı gibi sabitlendi ve depolandı. Tüm numuneler toplandıktan sonra, numuneler yutuldu ve yukarıda açıklandığı gibi 12'şer parti halinde kromatik kesme için hazırlandı. Her örnek için, optimum sonication ulaşmak için döngü sayısıtamamlandı 10. Nicel ölçüm ve yamlu kromatin parça boyutu ölçümleri, yöntemimizin üç bağışıklık hücresi kümesi üzerinde büyük tekrarlanabilirliğini göstermiştir (Şekil 2A). Farklı insan bağışıklık hücreleri ayrı gruplar halinde sonikated ve 14 döngüler için 100 - 500 bp arasında numunenin >% 70 ile çok tutarlı bir şekilde verim (16 s ON, 32 s OFF döngü başına). Bu noktada sonication sonrası büyük parçalara sahip örnekler (< 100 - 500 bp arasında numunenin % 70'i) başarısız olarak kabul edildi. Bu örnekler ya 1-2 ek döngü için sonicated ya da atılmış ve daha sonra başka bir pelet hücreleri ile değiştirildi. Yöntemimiz, numunelerin hiçbirinin daha fazla sonikasyon gerektirmediğini veya ortadan kaldırıldığını gösterdi, bu da prosedürün mutlak sağlamlığını düşündürmektedir.
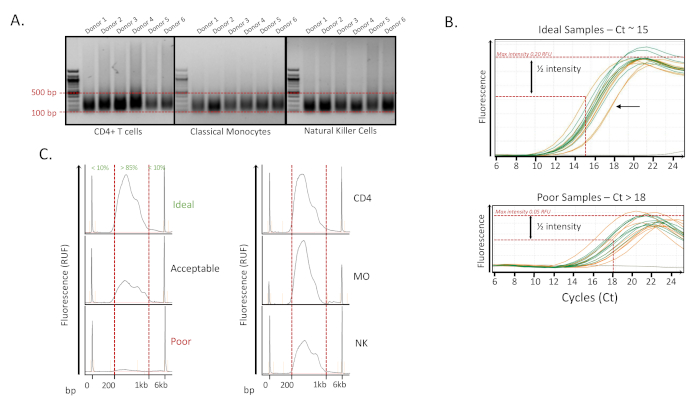
Şekil 2: QC örneklerinin önceden sıralanması. (A) % 1.2 agarose jelleri sonication tekrarlanabilirliğini gösterir. Naif CD4 T hücreleri (CD4), Klasik monositler (MO) ve Doğal öldürücü hücreler (NK) olmak üzere üç hücre tipinde 6 donör için sonication örnekleri. Örnekler 14 döngü boyunca sonikated edildi (16 s ON, 32 s OFF döngü başına). Her örnek için yaklaşık 200 ng decrosslinked kromatin% 1 agarose jel üzerine yüklendi. Parçaların % 70'inden fazlası 100-500 bp içindeyse numuneler iyi kabul edildi. (B) Üst - Amplifikasyon için en uygun döngü sayısını belirlemek için analiz qPCR amplifikasyon eğrileri (Maksimum yoğunluğun 1/2 olduğu Ct). İdeal örneklerin ct'si yaklaşık 15'tir ve amplifikasyon ölçülen Ct'nin 2 döngüsüne kadar tamamlanabilir. Alt - Ct'si 18'den büyük olan zayıf bir örnek kümesi örneği gösterilmiştir. Bu örnekler ayrıca daha düşük floresan yoğunluğu gösterdi. (C) Sol - Parça analizörü elektroforez izleri, amplifikasyon ve boyut seçiminden sonra son etiketli kütüphanelerin dağılımını gösterdi. 200-1.000 bp'de parça kütüphanesinin %85'inden fazla olan örnekler iyi örnekler olarak kabul edildi. Floresan pik yoğunluğunun ölçümü de önemli bir QC parametresi olarak kabul edilir, gerçekten de sinyal düşükse, numunenin iyi sıralanma olasılığı düşüktür. Sağ - CD4, MO ve NK'deki pozitif örnekler gösterilmiştir. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
Nicelemeden sonra, numuneler H3K27ac antikorları ile bir ChIP sıvı işleyicisi üzerinde çalıştırıldı ve ardından Tn5 transposaz enzimi ile etiketleme yapıldı. qPCR ile uygun sayıda amplifikasyon döngüsünü belirlemek için etiketli örneklerin% 10'u kullanılmıştır. Numunelerin amplifikasyonu için döngü sayısının belirlenmesi için, numunenin yoğunluğunun döngü belirleme için ortalama maksimumun yarısı olduğu döngüyü buluyoruz (Şekil 2B). Ct değerleri 18'den fazla olan örnekler sıralama sonrası iyi performans göstermemiştir ve ct değerleri bu nedenle başarısız bir ChIP örneğinin göstergesiydi. Bu örnekler genellikle amplifikasyondan sonra daha düşük miktarda DNA verdi. Ct değeri 15'e eşit veya daha az olan örnekler (100.000 hücre girişi) idealdi ve 15 ile 18 arasındaki örnekler kabul edilebilir ancak daha az tutarlı post dizilemeydi. 100.000'den az giriş hücresine sahip numuneler için Ct değerleri genellikle 15 ila 18 arasında bulunurdu, ancak sıralama için yeterli ürün elde etmek için 18 döngüden fazlasına ihtiyaç duymazlardı.
DNA etiketli amplifikasyondan sonra, NextGen dizilimi için 200 ila 1.000 bp arasında ideal bir boyut dağılımı elde etmek için kütüphaneler saflaştırılmış ve boyut olarak seçilmiştir. Dna parçalarının %85'inden fazlası 200 ila 1.000 bp arasında değiştiğinde en iyi sıralama verileri elde edildiğinden, kütüphanelerin her birinde boyut dağılımı değerlendirmesi tamamlandı (Şekil 2C). Özellikle, aynı miktarda DNA (floresan niceliği ile ölçülür) yüklendiğinden, daha düşük floresan yoğunluğuna sahip örneklerin genellikle kötü sıralanmış olduğu fark edildi (Şekil 2C).
Sıralama sonrası, ENCODE ChIP-Seq yönergelerine dayalı standart kalite kontrolleri5, 14,15uygulandı.
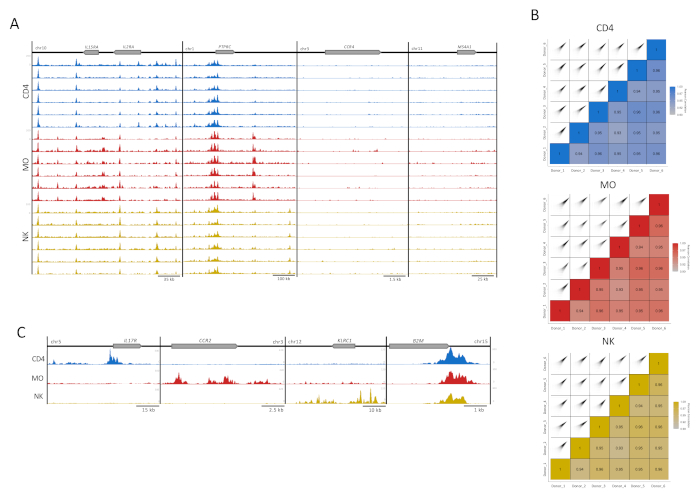
Şekil 3: Bağışıklık hücresi örneklerinin tekrarlanabilirliği. (A) H3K27ac, her hücre tipinde (CD4, MO ve NK) 6 donör (çoğaltma başına 100.000 hücre) için 4'lük maksimum yoğunluk, yumuşatma işlevi 4' tir. İkisi (IL2RA lokus ve PTPRC) ve ikisi H3K27ac (CCR4 ve MS4A1) için zenginleştirmesiz olmak üzere dört örnek loci gösterilmiştir. (B) Bağışçılar arasındaki pearson korelasyonu ve hücre türünün her biri için MEDIPS paketi içinde 300 bp uzantısı ve 500 bp penceresi kullanılarak oluşturulan karşılık gelen korelasyon çizimleri16'yı çoğaltır. (C) Hücre tipine özgü bölgelerde (CD4 için IL17R, MO için CCR2 ve NK için KLRC1) H3K27ac izlerini (UCSC Genom Tarayıcısı maksimum yoğunluğu, 4'ü yumuşatma işlevi) ve tüm hücre tiplerinde bulunan ev tutma geni B2M'yi gösteren her hücre tipi için birleştirilmiş donör dosyaları. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
Görsel kalite kontrolü için UCSC genom tarayıcısında görüntülenmek üzere H3K27ac zenginleştirme parçaları hazırlandı. Dört gen loci için, her örnek için ayrı parçalar, testimizin yüksek tutarlılığını ve sağlamlığını yansıtan yüksek haritalama kalitesi ve sinyal-gürültü oranı gösterdi (Şekil 3A). Sol limandaki iki loci bu hücre tiplerinde genleri iyi ifade ederken, sağdaki iki locideki genler ifade edilmez ve arka plan kontrolleri olarak servis edilir13 (Şekil 3A). Ayrıca, MEDIPS analiz paketi, teknik yinelemeler arasındaki korelasyon indeksini değerlendirmek için sıralama sonrası değişken olarak kullanılmıştır (Şekil 3B)5,16,17, 500 bp depo gözleri16için okuma zenginleştirme düzeyi için korelasyon derecesini belirlemek. Çift yönlü karşılaştırmaların çoğunda, Pearson korelasyon indeksleri biyolojik yinelemeler arasında yüksek tutarlılık seviyesi öneren% 90'dan fazla korelasyon göstermiştir (Şekil 3B). Kabul edilebilir korelasyona sahip çoğaltmalar, sinyal-gürültü oranını artırmak için birleştirildi. Hücre tipine özgü loci uygun hücrelerde yüksek zenginleşme gösterirken, bir ev tutma geni (B2M) çok tutarlı histone modifikasyonu gösterdi (Şekil 3C). Analiz için, parçaların çoğaltılmasından birleştirilmesi zenginleştirmeyi artıracak, önemli hücre tipine özgü arttırıcılar da dahil olmak üzere belirli sinyali güçlendirecek ve insan örneklerinin doğasında bulunan bireyler arası değişkenliğiazaltacaktır 5.
Bu çalışma için 100.000 hücre kullanılmasına rağmen, insan kültürlü bir T hücre hattında (HUT78) 10.000 kadar hücre için yüksek tekrarlanabilirlik vardı. 100.000'den az hücreli örneklerden gerçekleştirilen ChIP-Seq veri kümesi arasındaki korelasyon analizi, 10.000 hücreye kadar yüksek tekrarlanabilirlik ve korelasyon göstermiştir (Şekil 4A).
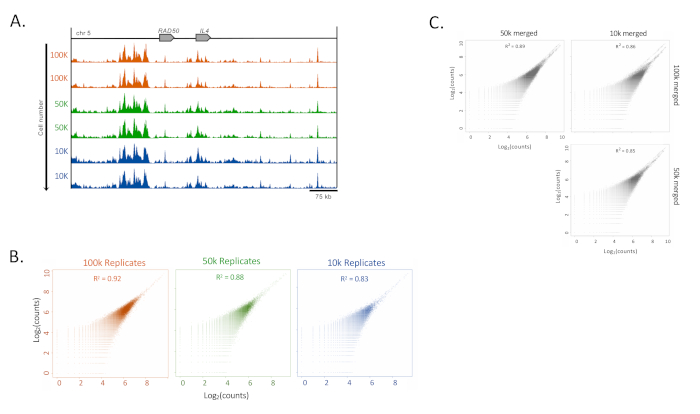
Şekil 4: Düşük giriş örneklerinin tekrarlanabilirliği. (A) HUT-78 hücrelerinde (T hücreli lenfoma hücre hattı) 100.000'den 10.000'e kadar olan hücreler için H3K27ac ChIP-Seq tutarlılığının örnekleri. Parçalar (UCSC Genom Tarayıcısı, maksimum yoğunluk, 4 yumuşatma işlevi, hepsi eşit ölçeklenmiş Y ekseni ile) IL4 locus'u gösterir. (B) MEDIPS paketi16içinde 300 bp uzantı ve 500 bp pencere kullanarak çoğaltmaların Pearson korelasyonları . (C) (B) 16 ile aynı MEDIPS parametrelerini kullanarak farklı hücre sayısı grupları (100.000, 50.000 ve10.000hücre) arasındaki pearson korelasyonları. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
Pearson korelasyon analizi yüksek korelasyon indeksi (%83 ila %92) göstererek düşük hücre sayısı örneklerinde sinyalin korunmasını düşündürmektedir. Bununla birlikte, hücre sayıları azaldığı için arka plan arttı ve korelasyon katsayılarının düşmesi (Şekil 4B). Düşük arka plan sinyallerini korumak için teknik yinelemeler birleştirildi ve gruplar arasındaki korelasyon test edildi (Şekil 4C).
| 10X Hücre Sabitleme Arabelleği | |
| Bileşik | Son Konsantrasyon |
| Formaldehit çözümü | 11% |
| NaCl | 100 mM |
| EDTA, pH 8.0 | 1 mM |
| EGTA, pH 8.0 | 0,5 mM |
| HEPES, pH 7.5 | 50 mM |
| Tam Lizis Arabelleği | |
| Bileşik | Son Konsantrasyon |
| Tris-HCI, pH 8.0 | 50 mM |
| EDTA, pH 8.0 | 10 mM |
| SDS | 0.25% |
| Sodyum Bütıraz | 20 mM |
| Proteaz Inhibitörü Kokteyli | 1 Kat |
| Kısa Süreli Lizis Tamponu | |
| Bileşik | Son Konsantrasyon |
| Tris-HCI, pH 8.0 | 50 mM |
| EDTA, pH 8.0 | 10 mM |
| SDS | 0.25% |
| Etiketleme Karışımı | |
| Bileşik | Son Konsantrasyon |
| Tris-HCI, pH 8.0 | 10 mM |
| MgCl2 | 5 mM |
| N,N-dimetilformamid | 10% |
| Illumina tagmentasyon enzimi | 1:24 cilt:vol |
| CtD Karışımı | |
| Bileşik | Numune başına (μL) |
| NextEra İndeks A (25 μM) | 0.275 |
| NextEra İndeks Astarı B (25 μM) | 0.275 |
| 2X KAPA HiFi HotStart Hazır Karışım | 2.75 |
| 1:1000 SYBR Yeşil boya | 0.11 |
| ROX pasif boya | 0.11 |
| Su | 4 μL'ye doldurun |
| AMP Karışımı | |
| Bileşik | Numune başına (μL) |
| 2X KAPA HiFi HotStart Hazır Karışım | 27.5 |
| Su | 31 μL'ye doldurun |
Ek Tablo 1: Tampon tarifleri.
Ek Tablo 2: Spearman ve Pearson 6 donör ve her hücre tipi için örnek korelasyonlar. Bu tabloyu indirmek için lütfen tıklayınız.
Tartışmalar
Burada açıklanan yöntem, protokolü otomatikleştirerek ve mikro ölçeklendirerek DNA saflaştırmadan önce bir etiketleme kitaplığı hazırlama protokolü uygulayan ChIPmentation prosedürü11'de genişler. ChIP-Seq'in başlangıcından bu yana, gerekli hücre sayıları büyük ölçüde azaltıldı, histonlar için yaklaşık 20 milyon hücreden yüzlerce ve hatta tek hücreli 1,7 ,10,12,18,19,20,21. Yeni geliştirilen bu yöntemler, duyarlılığı artırarak ve nadir klinik hücre popülasyonlarının test edilmesine izin vererek hücrelerde cis-düzenleyici mekanizmaların nasıl çalıştığının daha derin bir şekilde anlaşılmasını sağlamıştır5,6,12,17. Örneğin, cut&tag olarak adlandırılan daha yeni ve popüler prosedürlerden biri, sağlam ve hassas ChIP-Seqalternatifi 9. Tn5 enzimi A proteinine yaygın olarak bağlı olduğu ve chip antikorunun Fc zincirini yüksek özgüllükle tanıdığı için mükemmel bir sinyal-gürültü oranı üretir9. Hedef antikora bağlanmadan önce enzim işlevsel olmadığı için Tn5 enziminin arka plan aktivitesi azalır9. Bununla birlikte, bu yöntemin klinik bağlamda uygulanması sınırlıdır, çünkü sabit olmayan canlı hücreler gerektirir. Ayrıca, DNA parçalarının hipotonik çekirdekten çıkarılması, test sırasında çıkarıldığı için kromatin üzerinde olumsuz etkilere neden olabilir. Taze ve canlı hücrelerle çalışmak için gerekli gereklilik, nadir klinik örnekler ve büyük numune kohortları için bir sorun kaynağıdır, çünkü büyük kohortların toplanması çok sayıda yılsürebilir. Başka bir yöntem türü olan drop-ChIP, ChIP19'uişlemeden önce damlacık bazlı etiketleme oluşturmak için zarif bir mikroakışkanlık cihazı kullanır. Bununla birlikte, son derece özel bir mikroakışkan cihaz kullanır ve tek hücreli ChIP-Seq'i tamamlamak mümkün olsa da, canlı hücrelerin kullanımıyla da sınırlıdır 7 ,8,9,18,19. PLAC-Seq veya HiChIP gibi ChIP-Seq'e dayanan daha yeni yöntemler, ChIP-Seq tepeleri arasındaki 3 boyutlu (3D) etkileşimleri anlamaya çalışır22,23. Bu 3D yöntemler, genom genelinde cis-düzenleyici veya TF aracılı etkileşimleri tanımladıkları ve hücre içi ilgi türlerinde, sağlıklı dokularda ve hastalık bağlamında gen ekspresyonunun düzenlenmesinin daha iyi anlaşılması için heyecan vericidir.
Protokolün başarılı olması için sonikat kromatin kalitesi ve antikorun kalitesi gibi dikkate alınması gereken birkaç kritik adım vardır. Kesme verimliliği kritiktir, kromatin iyi sonicated değilse, test verimliliği büyük ölçüde azalır24. Sonication, gerekli hücre sayıları nedeniyle ChIP-Seq'in zorlu bir yönüdür. Protokolde kullanılan sonicator'da, verimlilik 300.000 hücrenin altında büyük ölçüde azaltıldı. Bu, ChIP-Seq'te bu seviyenin altında sonicate etmek için genellikle daha az tarafsız olan enzymatic parçalanmayı gerektirecek zorlu bir husustur. Sonuç olarak, sonication gerçek mikro ölçekli ChIP-Seq için önemli bir sınırlı faktördür. Diğer sonication platformları ve ticari olarak mevcut kitler kromatin sonicating için test edildi, ancak burada kullanılan sonicator en sağlam ve tekrarlanabilir sonuçlara sahipti. Sonicator'un bir başka avantajı, çok sayıda örnekle uğraşırken maliyetleri azaltan sonication'ı çalıştırmak için özel tüpler satın almak zorunda kalmamaktır. Optimal sonication için, öncelikle, yukarıda açıklandığı gibi sonicator önceden ısıtmak önemlidir. İkincisi, peletin izdimarı için, pipet ucunun daha fiziksel kısıtlı hücrelere parçalamak için yutel yaparken tüpün altına dokunması önerilir. Üçüncüsü, sonication önce herhangi bir kabarcık oluşumu örnek eşit olarak sonicated yeteneğini engeller. Lizis sırasında herhangi bir kabarcık oluşuyorsa, bunları bir pipetle çıkarmak önemlidir. Bu, çok fazla örnek çıkarmadan zor olabilir, ancak uç kabarcıka hafifçe bastırılırsa, çok fazla örnek kaybetmeden yavaşça çekilebilir. Son olarak, döngü sayısını belirlerken, her üç döngüde bir numunenin çıkarıldığı, saflaştığı ve bir agarose jeli üzerinde çalıştırıldığı bir zaman dilimini tamamlayın. ChIP verimliliğini azalttıklarından numunelerin aşırı/az sonication kaçının. Örnek sonikated altında ise, büyük parçalar ChIP-Seq kalitesi24üzerinde olumsuz bir etkiye sahip olabilir. Öte yandan, örnek aşırı sonicated ise, hedef epitopun işlemde kaybolma riski vardır.
ChIP-Seq'in bir diğer önemli parçası antikorun kalitesidir. Herhangi bir büyük ölçekli çalışma yapmadan önce, kullanılacak antikorun optimize edilmesi gerekir. Amaç, genomun bilinen bölgelerinin gürültü oranına önemli ölçüde yüksek bir sinyal elde etmektir ve bir diğeri tekrarlanabilirliktir. Antikor çok fazla arka plan sinyali çekiyorsa, daha büyük bir giriş kullanmanız veya farklı bir lot / tedarikçi denemeniz önerilebilir. Bu, büyük ölçekli bir deneye başlamadan önce zaman ekler, ancak bu önemli bir adımdır. Sinyalden gürültüye test etmek için qPCR'nin antikorunuzun hedefi olduğu bilinen bölgeler ve bulunmadığı bilinen başka bir bölge ile kullanılması önerilir. Histone modifikasyonlarının TF'lere göre daha sağlam ve optimize edilmesinin daha kolay olduğu fark edilmiştir.
Yukarıda açıklanan protokol, yarı otomatik, mikro ölçekli bir şekilde yüksek verimli histone modifikasyon ChIP-Seq için sağlam bir yöntem sağlar. Yöntem, uygulamalı süre miktarını sınırlar ve manuel ChIP-Seq üzerinden tekrarlanabilirliği artırır. Laboratuvarda tamamlanan önceki çalışmalar teknik çoğaltmalar üzerinde manuel ChIP kullandı ve 0.505Spearman korelasyon ortalaması elde etti, ancak yarı otomatik sistemle, NK hücreleri ortalaması 0.66 olan farklı donörler arasındaki Spearman korelasyonu(Ek Tablo 2). Bu da yaklaşık% 40 daha az uygulamalı zaman ile tamamlandı. Burada açıklanan yöntem histone modifikasyonları için optimize edilmiştir (burada gösterilen H3K27ac, ancak protokol başkaları için herhangi bir değişikliğe ihtiyaç duymamalıdır) ve TF ChIP-Seq için uygulanması için yalnızca küçük değişiklikler gerektirecektir. Antikorun kalitesine rağmen, ana modifikasyon sonikasyon süresi ve potansiyel olarak IP sırasında kullanılan tamponlar için olacaktır. Genellikle, TF ChIP tahlilleri için, yöntem biraz daha uzun kromatin parçalarıyla (yaklaşık 350-800 bp aralığı ile) daha iyi çalışabilir, çünkü TF:DNA kompleksleri titiz sonication6ile daha az korunabilir. ARABELLEKLER, TF'ler histone değişikliklerinden farklı davranabileceğinden, özel bir karışıma veya diğer endüstri mevcut kitlerine de dönüşebilir.
Otomatik ChIP sıvı işleyicisi 10.000 kadar az hücre için test edilmiş olsa da, daha düşük kromatin konsantrasyonlarında tekrarlanabilirlikte gözle görülür bir azalma oldu. Bu nedenle, protokol 10.000'den az hücreye önerilmedi ve 100.000 hücre en uygun koşullardı. Protokol ayrıca, ek bir masraf olan ancak daha yüksek kaliteli veriler sağlayan endüstri ChIP tamponları kullanılarak tamamlandı. Protokol sonikasyon koşullarına göre değiştirilebilir (yamulmuş kromatin aynı aralıkta tutulduğu sürece), tamponlar immün önkörükleme için özelleştirilebilir (IP; optimizasyon gerekebilir) veya ChIP sıvı işleyicisi kullanılamaz. Protokolün bir sınırlaması, pahalı bir yatırım olabilen ve aynı anda yalnızca 16 örnek çalıştırabilen ChIP sıvı işleyicisinin kullanılmasıdır. ChIP sıvı işleyicisi küçük ölçekli reaksiyonlarla sınırlıdır ve bir milyondan büyük hücre numaraları önerilmez. Ancak, protokol IP ve yıkama adımlarını el ile tamamlayarak onsuz tamamlanabilir. IP ve yıkamalar elle tamamlandıysa, tahlilleri tamamlama süresi artacak ve tekrarlanabilirlik azalabilir, ancak bu kılavuz yine de yüksek kaliteli bir ChIP-Seq deneyi çalıştırarak yararlı olacaktır. Not olarak, diğer sıvı işleyiciler yarı otomatik ChIP reaksiyonlarını çalıştırmak için uyarlanabilir.
Özetlemek gerekirse, IP ve yıkama adımları otonom olarak tamamlandığından, bu sistemin en büyük faydaları yüksek verimli yapıdır. Bu nedenle, chip deneylerinin sıralı turları tamamlanabilir ve manuel ChIP-Seq deneylerine kıyasla sınırlı uygulamalı süre ile 48 numunenin 5 gün içinde tamamen işlenmesine ve sıralamaya hazır olmasına izin verebilir. ChIP-Seq'in yüksek oranda tekrarlanabilir sonuçlar elde etmesi zor olabileceği için bir diğer avantaj da tekrarlanabilirliğin artmasıdır. Diğer yöntemler ya canlı hücreler, karmaşık mikro pipetleme sistemleri ya da işin hepsinin elle tamamlanmasını gerektirir. Bu sistemin düşük girişli numuneler (<10.000 hücre) için optimize edilmesi ve sonuçta tek hücreli ChIP reaksiyonlarına izin vermesi gerekecektir. Sistem ayrıca PLAC-Seq ve HiChIP22,23gibi daha yeni ChIP yöntemleri için uyarlanabilir.
Açıklamalar
Yazarların açıklayacak bir şeyi yok.
Teşekkürler
Vijayanand laboratuvar üyelerine teknik yardım ve yapıcı tartışmalar için ve Diagenode'den Dr. Sharron Squazzo ve Bay Geoffrey Berguet'e sonicator ve ChIP sıvı işleyici makinesi ve protokolleri ile ilgili teknik yardım için teşekkür ederiz. Bu çalışma NIH hibeleri AI108564, R01HL114093, S10RR027366 (BD FACSAria II) ve S10OD016262 (Illumina HiSeq 2500) tarafından desteklenmiştir.
Malzemeler
| Name | Company | Catalog Number | Comments |
| 200 µl tube strips (8 tubes/strip) + cap strips | Diagenode | C30020002 | Strip tubes for use on the IP Star; ChIP 8-tube strip |
| AMPure XP for PCR Purification | Beckman Coulter | A63880 | SPRI beads |
| Axygen 0.6 mL MaxyClear Snaplock Microcentrifuge Tube | Corning | MCT-060-C | 0.65 mL low binding tube |
| Bioruptor Pico Sonicator | Diagenode | B01060010 | Sonicator used in the lab but others can be used |
| ChIP DNA Clean & Concentrator (Capped Columns) | Zymo Research | D5205 | DNA clean-up kit |
| Dynabeads Protein A for Immunoprecipitation | ThermoFisher | 10001D | |
| EDTA (0.5 M), pH 8.0, RNase-free | ThermoFisher | AM9260G | |
| EGTA pH 8.0 | Millipore Sigma | E3889-25G | |
| Eppendorf ThermoMixer C | Eppendorf | 2231000667 | |
| Formaldehyde solution | Millipore Sigma | 252549-1L | |
| Glycine | Millipore Sigma | 50046-250G | |
| H3K27ac polyclonal antibody - Premium | Diagenode | C15410196 | |
| HEPES (1 M) pH 7.5 | ThermoFisher | 15630080 | |
| IDT for Illumina Nextera DNA Unique Dual Indexes | Illumina | 20027213 | |
| Illumina Tagment DNA Enzyme and Buffer Small Kit | Illumina | 20034197 | |
| IP-Star Compact Automated System | Diagenode | B03000002 | Automated system for ChIP-Seq studies; ChIP liquid handler |
| KAPA HiFi HotStart ReadyMix | Roche | KK2601 | PCR mix |
| Medium reagent container for SX-8G IP-Star Compact | Diagenode | C30020003 | |
| MgCl2 (magnesium chloride) (25 mM) | ThermoFisher | R0971 | |
| N,N-Dimethylformamide | Millipore Sigma | D4551-250ML | CAUTION - low flash point |
| NaCl (5 M), RNase-free | ThermoFisher | AM9760G | |
| PBS (10X), pH 7.4 | ThermoFisher | 70011044 | |
| PCR Flex-free 8-tube stripes, attached individual optical caps | USA Scientific | 1402-4700 | 8 strip tubes, 0.2 mL 8-tube strip |
| Proteinase Inhibitor Cocktail | Millipore Sigma | P8340 | |
| Proteinase K Solution (20 mg/mL), RNA grade | ThermoFisher | 25530049 | |
| PureLink RNase A (20 mg/mL) | ThermoFisher | 12091021 | |
| Quant-iT PicoGreen dsDNA Reagent | ThermoFisher | P7581 | Used in the flourescence quantification |
| QuantStudio 6 Flex Real-Time PCR System | ThermoFisher | 4485699 | qPCR |
| ROX Reference Dye | ThermoFisher | 12223012 | |
| Sodium butyrate | Millipore Sigma | 303410-100G | |
| SYBR Gold Nucleic Acid Gel Stain (10,000X Concentrate in DMSO) | ThermoFisher | S11494 | nucleic acid dye |
| SYBR Green I Nucleic Acid Gel Stain - 10,000X concentrate in DMSO | ThermoFisher | S7563 | |
| TE Buffer | ThermoFisher | 12090015 | |
| Tips (bulk) | Diagenode | C30040020 | Tips for the IP Star |
| True MicroChIP Kit | Diagenode | C01010130 | Contains all the buffers for the IP; ChIP kit |
| UltraPure 1M Tris-HCI, pH 8.0 | ThermoFisher | 15568025 | |
| UltraPure SDS Solution, 10% | ThermoFisher | 24730020 |
Referanslar
- Barski, A., et al. High-resolution profiling of histone methylations in the human genome. Cell. 129 (4), 823-837 (2007).
- Johnson, D., Mortazavi, A., Myers, R., Wold, B. Genome-Wide Mapping of in Vivo Protein-DNA Interactions. Science. 316, 1497-1502 (2007).
- Mikkelsen, T. S., et al. Genome-wide maps of chromatin state in pluripotent and lineage-committed cells. Nature. 448 (7153), 553-560 (2007).
- Furey, T. S. ChIP-seq and beyond: new and improved methodologies to detect and characterize protein-DNA interactions. Nature Reviews Genetics. 13 (12), 840-852 (2012).
- Seumois, G., et al. Epigenomic analysis of primary human T cells reveals enhancers associated with TH2 memory cell differentiation and asthma susceptibility. Nature Immunology. 15 (8), 777-788 (2014).
- Schmiedel, B. J., et al. 17q21 asthma-risk variants switch CTCF binding and regulate IL-2 production by T cells. Nature Communication. 7, 13426(2016).
- Ai, S., et al. Profiling chromatin states using single-cell itChIP-seq. Nature Cell Biology. 21 (9), 1164-1172 (2019).
- Brind'Amour, J., et al. An ultra-low-input native ChIP-seq protocol for genome-wide profiling of rare cell populations. Nature Communication. 6, 6033(2015).
- Kaya-Okur, H. S., et al. CUT&Tag for efficient epigenomic profiling of small samples and single cells. Nature Communication. 10 (1), 1930(1930).
- Youhanna Jankeel, D., Cayford, J., Schmiedel, B. J., Vijayanand, P., Seumois, G. An Integrated and Semiautomated Microscaled Approach to Profile Cis-Regulatory Elements by Histone Modification ChIP-Seq for Large-Scale Epigenetic Studies. Methods Molecular Biology. 1799, 303-326 (2018).
- Schmidl, C., Rendeiro, A. F., Sheffield, N. C., Bock, C. ChIPmentation: fast, robust, low-input ChIP-seq for histones and transcription factors. Nature Methods. 12 (10), 963-965 (2015).
- Skene, P. J., Henikoff, S. An efficient targeted nuclease strategy for high-resolution mapping of DNA binding sites. Elife. 6, 21856(2017).
- Schmiedel, B. J., et al. Impact of Genetic Polymorphisms on Human Immune Cell Gene Expression. Cell. 175 (6), 1701-1715 (2018).
- Landt, S. G., et al. ChIP-seq guidelines and practices of the ENCODE and modENCODE consortia. Genome Research. 22 (9), 1813-1831 (2012).
- Chen, L., et al. Genetic Drivers of Epigenetic and Transcriptional Variation in Human Immune Cells. Cell. 167 (5), 1398-1414 (2016).
- Lienhard, M., Grimm, C., Morkel, M., Herwig, R., Chavez, L. MEDIPS: genome-wide differential coverage analysis of sequencing data derived from DNA enrichment experiments. Bioinformatics. 30 (2), 284-286 (2014).
- Engel, I., et al. Innate-like functions of natural killer T cell subsets result from highly divergent gene programs. Nature Immunology. 17 (6), 728-739 (2016).
- Grosselin, K., et al. High-throughput single-cell ChIP-seq identifies heterogeneity of chromatin states in breast cancer. Nature Genetics. 51 (6), 1060-1066 (2019).
- Rotem, A., et al. Single-cell ChIP-seq reveals cell subpopulations defined by chromatin state. Nature Biotechnology. 33 (11), 1165-1172 (2015).
- Wang, Q., et al. CoBATCH for High-Throughput Single-Cell Epigenomic Profiling. Molecular Cell. 76 (1), 206-216 (2019).
- Consortium, E. P. An integrated encyclopedia of DNA elements in the human genome. Nature. 489 (7414), 57-74 (2012).
- Fang, R., et al. Mapping of long-range chromatin interactions by proximity ligation-assisted ChIP-seq. Cell Research. 26 (12), 1345-1348 (2016).
- Mumbach, M. R., et al. HiChIP: efficient and sensitive analysis of protein-directed genome architecture. Nature Methods. 13 (11), 919-922 (2016).
- Diagenode. The Ultimate Guide for Chromatin Shearing Optimization with Bioruptor Standard and Plus. Diagenode. , (2012).
Erratum
Formal Correction: Erratum: A Semiautomated ChIP-Seq Procedure for Large-scale Epigenetic Studies
Posted by JoVE Editors on 9/14/2020. Citeable Link.
An erratum was issued for: A Semiautomated ChIP-Seq Procedure for Large-scale Epigenetic Studies. An author's name was updated.
The name was corrected from:
Pandurangan Vijayanad
to:
Pandurangan Vijayanand
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiDaha Fazla Makale Keşfet
This article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır