このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
細菌におけるRIBO-seq:NGSシーケンシングのためのサンプル収集とライブラリ調製プロトコル
要約
ここでは、細菌におけるRIBO-seqのサンプル採取および調製の段階について説明する。これらのガイドラインに従って作成されたライブラリのシーケンシングは、包括的なバイオインフォマティクス分析のための十分なデータをもたらす。私たちが提示するプロトコルはシンプルで、標準的な実験装置を使用し、リシスから図書館の取得まで7日かかります。
要約
リボソームプロファイリング技術(RIBO-seq)は、現在 、生体内でのタンパク質合成のプロセスを研究するための最も効果的なツールです。この方法の利点は、他のアプローチと比較して、mRNA転写物上のリボソームの位置と数を正確にマッピングすることによって翻訳を監視する能力である。
本稿では、細菌におけるRIBO-seq法のサンプル採取と準備の連続した段階について述べ、実験の計画と実行に関連する詳細を強調する。
RIBO-seqは、インタクトリボソームおよび関連するmRNAに依存しているため、重要なステップは、翻訳の迅速な阻害および細胞の適切な崩壊である。したがって、クロラムフェニコールによる任意の前処理で細胞収穫のための液体窒素中のろ過とフラッシュ凍結を提案し、細菌の翻訳を停止する。崩壊のために、我々は、機械的に細胞壁を破壊する酸化アルミニウムの存在下でモルタルおよび害虫で凍結細胞を粉砕することを提案する。このプロトコルでは、単数精製のためのスクロースクッションまたはショ糖勾配超遠心分離は必要ない。代わりに、ポリアクリルアミドゲル電気泳動(PAGE)を用いたmRNA分離に続いてリボソームフットプリント切除(28-30ntバンド)が適用され、満足のいく結果を提供する。これにより、この方法が大幅に簡素化され、手順の時間と設備の要件が削減されます。ライブラリの準備のために、我々はある程度の最適化とメーカーのガイドラインに従って、ニューイングランドバイオラボからのイルミナシーケンシングのための市販の小さなRNAキットを使用することをお勧めします。
結果として得られる cDNA ライブラリは、次世代シーケンシング(NGS)に必要な適切な量と品質を示します。記載されたプロトコルに従って調製されたライブラリのシーケンス化は、サンプル当たり2〜10mlnの一意にマッピングされた読み取りで、包括的なバイオインフォマティクス分析に十分なデータを提供する。私たちが提示するプロトコルは、迅速かつ比較的簡単であり、標準的な実験装置で実行することができます。
概要
リボソームプロファイリング技術(RIBO-seq)は、カリフォルニア大学サンフランシスコ校のジョナサン・ワイスマンの研究室で開発されました。RIBO-seqは、トランスレーショナルレベルで遺伝子発現を研究するために使用される他の方法と比較して、mRNAに結合する各リボソームに焦点を当て、その位置およびトランスクリプト上のリボソームの相対的な数に関する情報を提供します。それは、生体内でのタンパク質合成のプロセスを監視することを可能にし、細胞内の個々のmRNAおよび全転写体に沿って両方のリボソーム密度の測定を可能にする単一のコドンの分解能および正確さを提供することができる。RIBO-seq技術の基礎には、翻訳中にリボソームがmRNA分子に結合し、リボヌクレアーゼ消化から転写物の埋もれた断片を保護するという事実があります。リボヌクレアーゼを添加すると、保護されていないmRNAが消化され、リボソーム(通常〜28〜30 ntの長さ)で囲まれた断片はそのまま残ります。リボソームフットプリント(RF)と呼ばれるこれらの断片は、リボソームの正確な位置を検出することから生まれたトランスクリプトに分離、配列決定、マッピングすることができます。実際、mRNA断片を保護するリボソーム能力は、1960年代からリボソーム結合および翻訳開始部位(TIS)2、3、4を研究するために使用されてきた。しかし、深いシーケンシング技術の進歩に伴い、RIBO-seqは、リボソーム関与を通じて、タンパク質合成に関するゲノム全体の情報を提供することができる翻訳モニタリング5のゴールドスタンダードとなっている。リボソームプロファイリングは、トランスクリプトームとプロテオーム6を定量化する間に存在する技術的なギャップを埋めた。
リボソームプロファイリングを行うには、調査対象条件下で増殖した生物の細胞リセートを取得する必要があります。細胞採取およびリシス中にこれらの条件を中断すると、信頼性の低いデータが得られ得る。これを防ぐために、翻訳阻害剤、液体窒素中の急速な収穫およびフラッシュ凍結が一般的に使用されている。細胞は、ミキサーミル7、8またはビーズビーター9のような機械的ホモジナイザーでの極低温粉砕によって、ピペット10または針11でトリチュレーションすることによって、凍結粉砕することができる。細胞の粉砕の直前または直後に、分解バッファーを追加できます。私たちのプロトコルでは、乳鉢や害虫を予冷するために液体窒素を使用し、細菌細胞壁の破壊に対する優しいアプローチとして酸化アルミニウムを使用し、ソニフィケーションなどの方法が適用されたときにしばしば遭遇するRNAせん断を防ぎます。粉砕後、乳鉢の冷却された内容物に氷冷のライシスバッファーを追加します。リボソームフットプリントの最適な解像度を得るためには、適切なリシスバッファーの選択が重要です。イオン強度はRFサイズと読み取りフレーム精度の両方に影響を与えるので、現在では、バッファー組成がmRNA1,12のリボソーム占有率に影響を与えないと思われるとしても、イオン強度と緩衝能の低いリシスバッファを使用することが推奨されている。このリシスバッファーの重要な成分はマグネシウムイオンであり、その存在はリボソームサブユニットの解離を防止し、細菌リボソーム11,13における自然な立体構造変化を阻害する。カルシウムイオンもまた重要な役割を果たし、細菌性リボソームプロファイリング法14で用いられる微小球核化(MNase)の活性に必須である。グアノシン5′の添加-[β,γ-imido]三リン酸(GMP-PNP)は、GTPの非加水分解可能なアナログであり、クロラムフェニコールと共に、リシス15の間の翻訳を阻害する。
ライセートが得られると、遠心分離によって明確化され、RIBO-seqと高スループットの総mRNAシーケンシング(RNA-seq)が同時に行われるため、それぞれ2つの部分に分けられる(図1)。RNA-seqは、データ分析中にRIBO-seqとRNA-seqの両方からのデータを比較できる基準点を提供します。調査した翻訳は、リボソームフットプリントをmRNAの存在量16に正規化することによって定義される。RNA-seq からのデータは、クローニングまたはシーケンシングアーティファクト17を識別するのにも役立ちます。
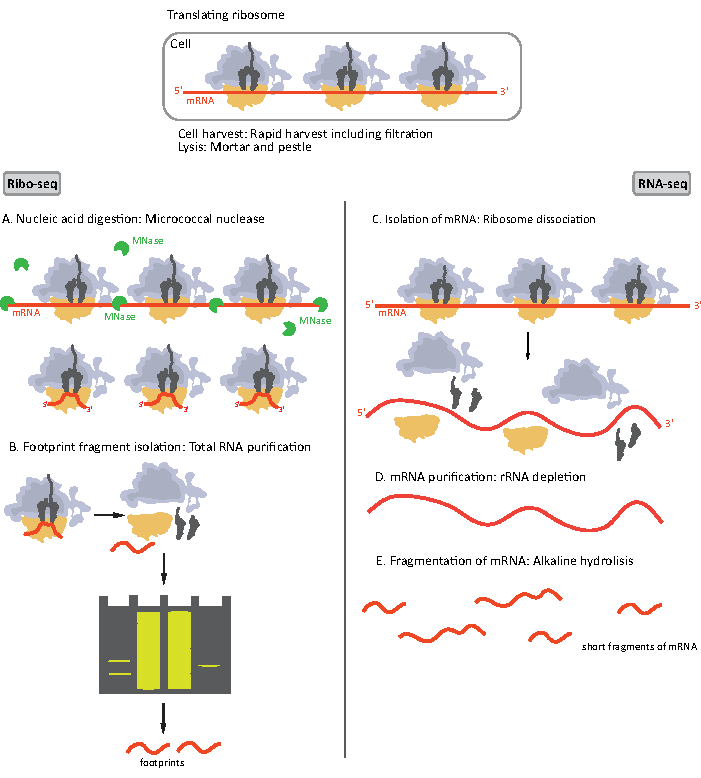
図 1.RIBO-セクおよびRNA-セクに対するmRNAサンプル調製物の概略図。 RIBO-seqライブラリ調製の場合、RNAはMNase(A)で消化され、続いて〜28〜30 ntの長さ(B)のRFのサイズ選択が続きます。RNA-セクRNAが単離(C)であるため、rRNA(D)の枯渇、および得られたmRNAは、様々な長さの断片(E)にランダムに断片化される。 この図の大きなバージョンを表示するには、ここをクリックしてください。
RIBO-seqとRNA-seqのサンプル調製手順の初期ステップは若干異なる(図1)。リボソームプロファイリングの場合、リソソームによって保護されていないmRNA分子を分解するために、特定のエンドヌクレアーゼによってリセートを消化する必要があります。標準的なプロトコルにおいて、得られたモノソームは、ショ糖クッション超遠心分離またはショ糖勾配超遠心8、14によって回収される。本稿では、このステップは、真核生物細胞18に対しても同様に、細菌中のRIBO-seqに必要なRFを分離する必要はないことを示し、ポリアクリルアミドゲルからの適切な長さのmRNA断片のサイズ選択が十分であることを示す。
RNA-seqの場合、mRNAは、全RNA-rRNA分子からストレプトアビジンコーティングされた磁気ビーズに結合するビオチン化オリゴヌクレオチドプローブにハイブリダイズする全RNAからrRNAの枯渇によって得られる。次いで、rRNAオリゴヌクレオチドビーズ複合体を磁石でサンプルから除去し、rRNAが枯渇したサンプル19,20を得る。精製されたmRNA分子は、次いでアルカリ加水分解によってランダムに断片化される。得られたmRNAの断片とリボソーム足跡はcDNAライブラリに変換され、深いシーケンシングのために準備される(図2)。これは、アルカリ加水分解(mRNAの場合)と酵素消化(RF用)の後に必要な修復を終了することを含む:3'末端の脱リン酸化後に5'末端のリン酸化が続く。次のステップは、アダプタライゲーションと逆転写を行い、Illuminaプラットフォームを使用して次世代シーケンシング(NGS)に必要な配列でフレーム化されたcDNAインサートを作成します。ライブラリー調製の最後の段階は、構成体を増幅し、サンプル固有のバーコードでラベル付けし、1つのチャネルでさまざまなサンプルを多重化およびシーケンス処理できるようにする PCR 反応です。シーケンシングの前に、ライブラリの質と量は高感度DNAオンチップ電気泳動によって評価されます。適切なパラメータを持つcDNAライブラリをプールし、シーケンスを作成できます。シーケンスは、ライブラリの数、必要な読み取り長さ、およびシーケンス深度に応じて、MiSeq、NextSeq、HighSeqなどのさまざまなイルミナプラットフォームで実行できます。シーケンシング後、バイオインフォマティクス解析が行われる。
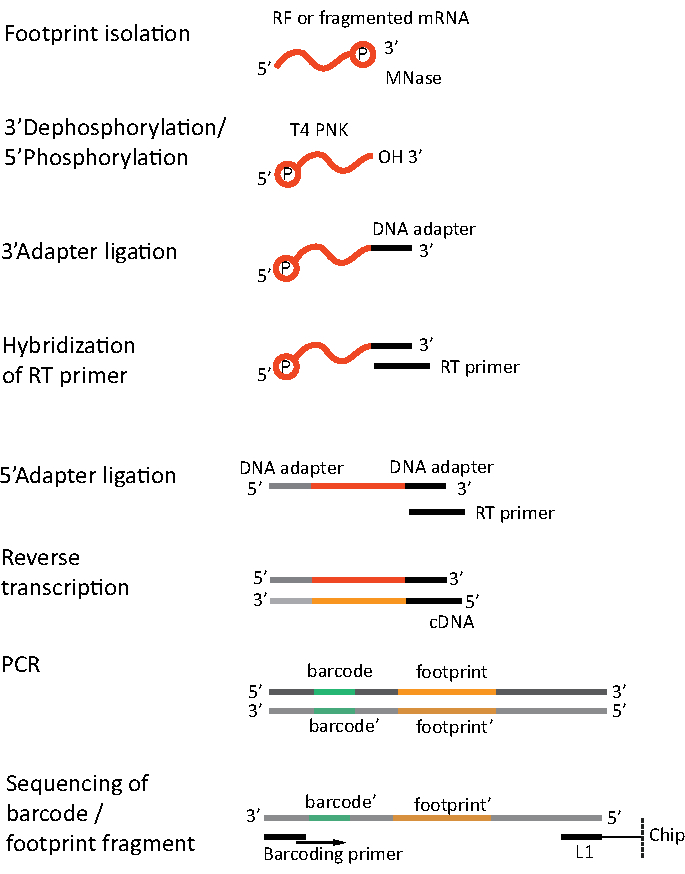
図 2.図書館の準備。 ライブラリの調製は、エンド修復、アダプターライゲーション、逆転写およびバーコードによる増幅を含む。 この図の大きなバージョンを表示するには、ここをクリックしてください。
リボソームプロファイリングは、科学的な質問に従って簡単に修正および調整することができる普遍的な方法です。もともとは酵母1で使用されていたが、マウス10、ゼブラフィッシュ22、フルーツフライ23及びシロイヌナズナ24を含む真核生物と同様に細菌細胞21に適用された直後に。また、細胞質、ミトコンドリア25、26、葉葉芽細胞27、28の異なるリボソームタイプの研究にも使用されました。真核生物では、RIBO-seqは、一般的に、開始10、11、29、30、31、32、伸び1、10、11、31、33、リボソーム失速33およびコンフォメーション変化33を含む翻訳の特定の側面を調査するために適応および精製される。ほとんどの修飾は、異なる翻訳阻害剤の使用を含む。しかし、細菌では、作用機序34を有する阻害剤の貧弱性のために類似の研究が行いにくい。細菌の中で最も一般的に使用される翻訳阻害剤は、ペプチジルトランスファーゼセンター(PTC)に結合し、Aサイトにおけるアミノアシル-tRNAの正しい位置を防ぐクロラムフェニコール(CAM)です。その結果、CAMは、伸びるリボソーム35を阻止することにつながるペプチド結合の形成を防止する。細菌における翻訳阻害剤の他の例としては、テトラサイクリン(TET)36、リタパミュリン(RET)34およびOnc11237があり、翻訳開始部位の調査に使用されている。TETは、A-サイトでのtRNAの抗コドンステムループと直接重なることによってリボソームへのtRNA送達を防止し、これらは共に抗生物質が翻訳伸長38を阻害するのでCAM治療から得られた結果を検証するために適用された。TET はプライマリ TIS を検出することが判明したが、内部 TIS36を明らかにすることができなかった。RETは細菌リボソームのPTCに結合し、A部位におけるアミノアシル-tRNAを細長く干渉させることにより第1のペプチド結合の形成を防止する。RETを適用すると、プライマリと内部のTIS34の両方でリボソームが逮捕されます。Onc112は、プロリンが豊富な抗菌ペプチドであり、出口トンネル内で結合し、リボソームA部位におけるアミノアシル-tRNA結合をブロックする。その結果、Onc112は開始複合体が伸びフェーズ37に入るのを防ぎます。
リボソームプロファイリングが提供する主な情報は、リボソーム密度およびmRNA上でのそれらの位置である。様々な成長条件1,6の翻訳レベルでの遺伝子発現の差動を調べ、翻訳効率1、38、39を測定し、リボソーム休止などの翻訳調節事象を検出するために応用に成功した。RIBO-seqはまた、新しいおよび/または非常に短いタンパク質コード遺伝子10、12、22、30、37の同定につながる、アノト化ncRNA、擬似遺伝子および無公認の小さなオープンリーディングフレーム(ORF)の翻訳を明らかにすることを可能にする。このような場合、RIBO-seqはゲノムアノテーションを微調整し、改善することができます。翻訳されたORFの同定とその定量的性質に対する高感度を有するリボソームプロファイリングは、プロテオームの決定の代理としても、またはプロテオミクス研究31、34、39を補助する役割を果たすことができる。TISをマッピングすることにより、リボソームプロファイリングは、既知のタンパク質10,32のN末端拡張および切り捨てられたアイソフォームを明らかにする。RIBO-seqはまた、タンパク質14、21、24の共翻訳折りたたみを研究するために適応された。この方法は、個々のコドン6の伸び速度1、10、39またはデコード速度の測定を可能にし、翻訳17の定量モデルの開発に役立ちます。リボソームプロファイリング法は、細菌7、15、17、フレームシフト40、ストップコドン読み出し21、終了/リサイクル欠陥41、42およびリボソーム立体構造変化33の細菌におけるリボソーム休止に対する機械学的洞察を提供することもできる。RIBO-seqはまた、真核生物におけるmiRNA6およびRNA結合タンパク質などの翻訳に対する特異的トランス作用因子の影響を調べるためにも適応された。しかしながら、RIBO-seqの実験計画および得られた解像度が、得られたシーケンシングデータ12から抽出できる情報量を決定することを認めることが重要である。
プロトコル
1. サンプルコレクション
- 細菌培養を準備します。1サンプルあたり100mLの培養量をお勧めします。
- サンプル採取用の装置および試薬を準備する:サンプルあたり2つのスコップラ、無菌0.45 μm混合セルロースエステル膜(MCE)フィルター、50 mL滅菌チューブ、液体窒素、50mg/mLクロラムフェニコール70%(vol/vol)エタノール(オプション)。液体窒素蒸発を可能にし、閉じた管の爆発を防ぐために50 mLの管のふたを穿刺する。実験室用洗剤と70%エタノールでスコップラを除染します。
- 濾過装置とスコップラの1つを細菌培養の成長温度に予熱する。
注:漏斗、フリットベース、グランドジョイントフラスコ、47 mm Øスプリングクランプを備えたオールガラス濾過装置をお勧めします。 - サンプル収穫前に、菌培養物にクロラムフェニコールを加えて、最終濃度の100 μg/mLにし、1分間インキュベートします(オプション)。
- 液体窒素を50 mLの無菌チューブに注ぎ、2番目のすくい管をチューブの中に入れて冷却します。
注意!液体窒素は、蒸発時の圧力変化により、閉じた容器が爆発する可能性があります。これを防ぐためには、液体窒素の蒸発を可能にするために50 mLチューブの蓋にいくつかの穴を作成する必要があります。 - ろ過によって細胞を集める。培地が膜を通過した場合はフィルタリングを停止しますが、フィルタを完全に乾燥させないでください。
- プリウォームドスクープラを使用してフィルターディスクから細胞を迅速に掻き取ることによって細菌ペレットを収集します。収穫した細胞を液体窒素で満たした50 mLチューブにすぐにすくい全体を入れます。収穫されたペレットは、完全に液体窒素で覆われるべきです。
- ペレットを完全に凍結させ、以前に冷却した2番目のスクープラを使用して凍結細胞を取り除きます。穿刺された蓋を閉じ、チューブを-80°Cのストップポイントに移します
注:各サンプル採取後にスコップラを消毒してください。
2. 細胞の起水
- 10 mM トリス pH 8、DNase I、1.5 mL 反応チューブで 0.1 M GMP-PNP を準備し、氷の上に保管します。遠心分離機を4°Cに冷却します。
- リシスバッファー(20 mM TRIS pH 7.6、10 mM MgAcet、150 mM KAcet、0.4%トリトンX-100、6 mM βメルカプトエタノール、5 mM CaCl2 およびオプションの1 mMクロラムフェニコール)サンプル当たりのリシスバッファーのアリコート500 μLを1.5 mL反応チューブに入れ、氷の上に置きます。
- モルタルと害虫を実験室用洗剤と70%エタノールで除染し、乾燥させます。乳鉢と害虫を冷却し、液体窒素をモルタルに注ぎます。
- 冷凍ペレットを冷やしたモルタルに移し、粉末状に粉砕します。へらで、酸化アルミニウムの約1体積を加え、粉砕を続けます。必要に応じて液体窒素を注ぎ込んでモルタル、害虫、細胞を冷やし、モルタルの内容物を解凍させないでください。
- ライシスバッファーを使用する直前に、GMP-PNP と DNase I を、それぞれ 2 mM と 100 U/mL の最終濃度にリシス バッファー アリコートに加えます。セルと酸化アルミニウムで溶液をモルタルに移し、粉砕を続けます。ライセートは、粉砕しながらゆっくりと解凍し、予め冷却された1.5 mL反応チューブに混合物を転送し、すぐに氷の上に置きます。
- 遠心分離機は4°Cで5分間20 000xgでリセートする。 上清を新しい、予冷した1,5 mLの反応管に移し、氷の上に置いてください。
- ナノドロップを用いて各サンプルのRNA濃度を測定します。1:10希釈サンプルと1:10の溶解バッファーをヌクレアーゼフリー水にブランクとして使用してください。
注: クロラムフェニコールは260 nmで有意な吸収を示していることに注意してください。 - 各ライセートを2つの部分に分けます:1つはRIBO-seq(0.5-1mgのRNA)とRNA-セク(残り)の2つ目です。
- メーカーのプロトコルに従って市販のRNAクリーンアップキットを使用して、RNA-seqのサンプルを洗浄します。
注意:Zymo RNA クリーン&コンセントレート -25 キットを使用することをお勧めします( 材料表を参照)。また、短いRNA断片の精製効率を高めるために、リボソームフットプリントと断片化したmRNAのサンプルと結合バッファーのミックスに4.5量のエタノール(サンプル量と比較して)を追加することをお勧めします。 - RNA-seqを対象としたサンプルを-80 °Cに保存してください。 RNA-seqの手順は、ステップ5から継続されます。
3. リボ-セクのためのサンプルのMNase消化
- RNAの1mgは10 mM Tris pH 8に187.5 U/μL MNaseの3.8 μLを加え、500 μLの総体積にリシスバッファーで上げる。
- 25°C、300 RPMでインキュベートし、サーモミキサーで45分間インキュベートします。
- 市販のRNAクリーンアップキットでサンプルをクリーニングします(ステップ2.9のように)。
- RIBO-seqのサンプルを-80 °C(ストップポイント)に保存するか、サイズ選択に進みます。
4. ポリアクリルアミドゲル電気泳動(PAGE)およびRIBO-セク用サンプルのサイズ選択
- 8 M尿素で15%ポリアクリルアミド-TBEゲルを調製し、TBEバッファー付きタンクに入れます。200 Vの一定電圧で最低10分間のプレラン。
- TBE-尿素サンプルバッファーと混合し、95 °Cで1分間変性し、すぐに氷の上に置きます。
- 注射器を使用してTBEバッファーをゲルウェルに注入して尿素を洗い流します。各サンプルを分離し、クロス汚染を防ぐために、それらの間に1つの井戸スペースを残してサンプルをロードします。マーカーとして29ntオリゴヌクレオチドと26ntおよび32 ntオリゴヌクレオチドの混合物を使用してください。電気泳動は180Vの一定電圧で実行します。
- 一晩のインキュベーション(5 mM EDTA、10 mM NaOAc pH 5)のための無菌バッファーを準備する。
- 電気泳動後、SYBRゴールドバスでゲルを約2〜3分間染色し、ヌクレアーゼを含まない水でゲルをすすます。
- 滅菌針またはカミソリの刃で26と32ntの間のゲルの切物片を、各サンプルのための別々の1.5 mLの管にゲルの断片を置く。サンプル間の針またはカミソリの刃を変更します。
- 各反応管に一晩インキュベーションバッファーの 200 μL を加えます。
- サーモミキサーで10°C、1000RPMで一晩インキュベートします。
- 翌日、ステップ 2.9 のようにサンプルをクリーニングします。80 μL のヌクレアーゼを含まない水でフットプリントを溶かします。
- サンプルは-80°Cのストップポイントで保管してください。RIBO-seq の手順は、ステップ 7 から続行されます。
5. RNA-セク用サンプルからのrRNA枯渇による細菌mRNAの精製
- 細菌性mRNA精製キット(例えば、インビトロジェンMICROBExpress)を用いてサンプルからrRNAを除去する。製造元のプロトコルに従います。
- ステップ 2.9 のようにサンプルをクリーニングします。mRNAを50μLのヌクレアーゼフリー水で溶出します。
- RNA-seqのサンプルを-80°C(STOPポイント)に保存するか、アルカリ性の断片化を継続します。
6. RNA-seqのサンプルのアルカリ性断片化
- アルカリ加水分解バッファー(0.1 M NaHCO 3の220 μL、0.1 M Na2CO3の30 μL、0.5 M EDTAの1 μLを混合する)アルカリバッファーの1ボリューム(50μL)を1ボリューム(50μL)のサンプルと混合し、95°Cで25分間インキュベートします。
- 反応を止めるために3 M NaOAc pH 5.5の5μLを加える。
- ステップ2.9のようにサンプルを洗浄し、ヌクレアーゼを含まない80 μLの水でサンプルを溶出します。
- 可能な停止点: -80 °CでRNA-seqサンプルを保存します。 ただし、不要な凍結や解凍を避けるために、ステップ7に直接行くことをお勧めします。
7. RNAとRIBO-seqの両方のサンプルの脱リン酸化とリン酸化
- 各サンプルに10倍反応バッファーPNKと5μL T4 PNKを10μL加えます。3'末端を脱リン酸化するために、37°Cで1.5時間インキュベートします。
- 1 mM ATPの3 μLを加え、5'の端をリン酸化するために37°Cで1時間インキュベートします。
- ステップ2.9のようにサンプルを洗浄し、30 μLヌクレアーゼを含まない水で溶出します。
- サンプルは-80°Cのストップポイントで保管してください。
8. NEBNext マルチプレックス スモール RNA ライブラリ準備セットを用いたライブラリの準備
- ヌクレアーゼを含まない水を6μLで100-1000ngの入力RNA(得られたmRNA断片およびリボソーム足跡)を調製し、3'SRアダプターの1μLを加えます。メーカーのプロトコルに従ってインキュベートします。
- 3'ライゲーション反応バッファー(2X)の10 μLと3'ライゲーション酵素ミックスの3 μLを加え、メーカーのプロトコルが定める25°Cで1時間ではなく、37°Cで2.5時間インキュベートします。
- 改変プログラムを使用して、製造者のプロトコルに従って逆転写プライマーをハイブリダイズします:75°Cで5分、37°Cで30分、4°Cで保持します。
- 5' SR アダプターをリゲートします。1つの変更でメーカーのプロトコルに従ってください:25°Cで1時間ではなく、2.5時間37°Cでインキュベーションを行います。
- 製造元のプロトコルに従って逆転写を実行します。
- 可能なSTOPポイント:70°Cで合成されたcDNAを含むサンプルを15分間インキュベートし、逆転写酵素を不活性化し、-80°Cで保存します。 ただし、サンプルの不要な凍結や解凍を避けるために、次の手順に直接進むのがおすすめです。
- 各cDNAライブラリの半分を-80°Cにバックアップとして保存します。
- メーカーのプロトコルに従って PCR 増幅を行います。反応ミックスの半分を使用してください。得られたライブラリを-80°Cの停止点に保管してください。
9. PAGEを使用したcDNAライブラリのサイズ選択
- 6%ポリアクリルアミド-TBEゲルを調製し、TBEバッファー付きタンクに入れます。200 Vの一定電圧で最低10分間のプレラン。
- サンプルをゲルローディング染料、ブルー(6X)と混ぜ、ゲルにセットします。クイックロードpBR322 DNA-MspIダイジェストをはしごとして使用します。最初は、電気泳動を120Vの定電圧で実行し、DNAがゲルに入るようにしてから電圧を180Vに変更します。
- 電気泳動が終了したら、SYBRゴールドバスでゲルを約2〜3分間染色し、ヌクレアーゼを含まない水でゲルをすすます。
- ライブラリを含む物品物ゲル断片。RNA-seq サンプルの 135-180 nt の間および 135-170 nt の間の RIBO-seq の間の物品切り。滅菌針またはカミソリの刃を使用し、切除されたゲル断片を別々の1.5 mL反応チューブに入れる。サンプル間の針またはカミソリの刃を変更することを忘れないでください。
注:NEBNextマルチプレックススモールRNAライブラリ準備セットのアダプタとバーコードは、合計で119ntで、切除カットオフの下限を決定します。 - 100 μL のヌクレアーゼを含まない水を各摘出されたゲルフラグメントに加えます。
- ゲル断片を10°C、450RPMで一晩サーモミキサーでインキュベートする。
- 製造者のプロトコルに従って、商用のDNAクリーンアップキットでcDNAライブラリを清掃し、濃縮します。
注意:Zymo DNAクリーン&コンセントレート-5キット(資料表を参照)を使用することをお勧めします。 - 精製したcDNAライブラリを-80 °Cのストップポイントに保存します。
結果
ここで提示された例示的な結果は、WT バチルス・サステイリス 細胞の翻訳調節を調べる研究で得られた。一晩培養物を、100mLの濃厚培地で0.1に等しいOD600に希釈し、OD600 が0.5-0.6に達するまで激しく揺れで37°Cでインキュベートした。 次いで、培養を最小限の培地に置換して胞子化を誘導し、最大4時間培養を続けた。細胞は、T0で始まる1時間ごとに採取された - 胞子誘?...
ディスカッション
リボソームプロファイリングの重要な技術的課題は、特定の生理学的状態でmRNA上のリボソームのスナップショットをキャプチャするために、翻訳を迅速に阻害する必要性である。これを達成するために、翻訳阻害剤、液体窒素中の急速な収穫およびフラッシュ凍結が一般的に使用されている。抗生物質の適用は、アーティファクトを引き起こす可能性があるため、オプションです。クロラム...
開示事項
著者らは開示するものは何もない。
謝辞
ALSは、EMBOインストール補助金IG 3914およびPOIRの財政支援を認めたいと考えています。04.04.00-00-3E9C/17-00 欧州連合(EU)が欧州地域開発基金に共同出資するポーランド科学財団のファーストTEAMプログラム内で実施。
資料
| Name | Company | Catalog Number | Comments |
| 10X TBE (powder) | Invitrogen | AM9864 | |
| 2-Mercaptoethanol, 99%, pure | Acros Organics | 125472500 | |
| Adenosine 5'-Triphosphate (ATP) | New England Biolabs | P0756S | |
| Aluminium oxide calcinated pure p.a. | Chempur | 114560600 | |
| Calcium chloride dihydrate | Sigma-Aldrich | C3881-500G | |
| Chloramphenicol | MP Biomedicals | 190321 | |
| DNA Clean & Concentrator -5 | Zymo Research | D4004 | |
| Dnase I recombinant, Rnase-free | Roche | 4716728001 | |
| EDTA disodium salt | Fisher Scientific | E/P140/48 | |
| Ethyl Alcohol Absolut 99,8% Pure-P.A.-Basic | POCH Avantor Performance Materials Poland S.A | BA6480111 | |
| Filtration apparatus | VWR Collection | 511-0265 | all-glass filtration apparatus, with funnel, fritted base, cap, 47 mm Ø spring clamp and ground joint flask |
| Gel 40 (19:1) | Rotiphorese | 3030.1 | |
| Gel Loading Dye, Blue, 6X | New England Biolabs | E6138G | |
| Guanosine 5′-[β,γ-imido]triphosphate trisodium salt hydrate | Sigma-Aldrich | G0635-25MG | |
| labZAP | A&A Biotechnology | 040-500 | |
| Magnesium acetate tetrahydrate | Sigma-Aldrich | M5661-250G | |
| MCE membrane fiter | Alfatec Technology | M47MCE45GWS | pore size: 0.45um |
| MICROBExpress Bacterial mRNA Purification | Invitrogen | AM1905 | |
| Multiplex Small RNA Library Prep Set for Illumina | New England Biolabs | E7300S | |
| Nuclease-Free Water | Ambion | AM9937 | |
| Potassium Acetate Anhydrous Pure P.A. | POCH Avantor Performance Materials Poland S.A | 744330113 | |
| Quick-Load pBR322 DNA-MspI Digest | New England Biolabs | E7323A | |
| RNA Clean & Concentrator -25 | Zymo Research | R1018 | |
| Sodium acetate | Sigma-Aldrich | S2889-250G | |
| Sodium carbonate | Sigma-Aldrich | 223530-500G | |
| Sodium hydrogen carbonate pure p.a. | POCH Avantor Performance Materials Poland S.A | 810530115 | |
| SYBR Gold nucleic acid gel stain | Life Technologies | S11494 | |
| T4 Polynucleotide Kinase | New England Biolabs | M0201L | |
| T4 Polynucleotide Kinase Reaction Buffer | New England Biolabs | B0201S | |
| TBE-Urea Sample Buffer (2x) | Invitrogen | LC6876 | |
| Tris(hydroxymethyl)amino-methane, ultrapure, 99,9% | AlfaAesar | J65594 | |
| Triton X-100, 98% | Acros Organics | 327371000 | |
| Urea G.R. | lach:ner | 40096-AP0 |
参考文献
- Ingolia, N. T., Ghaemmaghami, S., Newman, J. R., Weissman, J. S. Genome-wide analysis in vivo of translation with nucleotide resolution using ribosome profiling. Science. 324 (5924), 218-223 (2009).
- Takanami, M., Yan, Y., Jukes, T. H. Studies on the site of ribosomal binding of f2 bacteriophage RNA. Journal of Molecular Biology. 12 (3), 761-773 (1965).
- Steitz, J. A. Polypeptide Chain Initiation: Nucleotide Sequences of the Three Ribosomal Binding Sites in Bacteriophage R17 RNA. Nature. 224 (5223), 957-964 (1969).
- Wolin, S. L., Walter, P. Ribosome pausing and stacking during translation of a eukaryotic mRNA. The EMBO journal. 7 (11), 3559-3569 (1988).
- Argüello, R. J., et al. SunRiSE - measuring translation elongation at single-cell resolution by means of flow cytometry. Journal of Cell Science. 131 (10), 214346 (2018).
- Michel, A. M., Baranov, P. V. Ribosome profiling: a Hi-Def monitor for protein synthesis at the genome-wide scale. Wiley Interdisciplinary Reviews: RNA. 4 (5), 473-490 (2013).
- Woolstenhulme, C. J., Guydosh, N. R., Green, R., Buskirk, A. R. High-precision analysis of translational pausing by ribosome profiling in bacteria lacking EFP. Cell reports. 11 (1), 13-21 (2015).
- McGlincy, N. J., Ingolia, N. T. Transcriptome-wide measurement of translation by ribosome profiling. Methods (San Diego, Calif). 126, 112-129 (2017).
- Gerashchenko, M. V., Gladyshev, V. N. Translation inhibitors cause abnormalities in ribosome profiling experiments. Nucleic acids research. 42 (17), 134 (2014).
- Ingolia, N. T., Lareau, L. F., Weissman, J. S. Ribosome Profiling of Mouse Embryonic Stem Cells Reveals the Complexity of Mammalian Proteomes. Cell. 147 (4), 789-802 (2011).
- Ingolia, N. T., Brar, G. A., Rouskin, S., McGeachy, A. M., Weissman, J. S. The ribosome profiling strategy for monitoring translation in vivo by deep sequencing of ribosome-protected mRNA fragments. Nature protocols. 7 (8), 1534-1550 (2012).
- Hsu, P. Y., et al. Super-resolution ribosome profiling reveals unannotated translation events in Arabidopsis. Proceedings of the National Academy of Sciences of the United States of America. 113 (45), 7126-7135 (2016).
- Blanchard, S. C., Kim, H. D., Gonzalez, R. L., Puglisi, J. D., Chu, S. tRNA dynamics on the ribosome during translation. Proceedings of the National Academy of Sciences of the United States of America. 101 (35), 12893-12898 (2004).
- Becker, A. H., Oh, E., Weissman, J. S., Kramer, G., Bukau, B. Selective ribosome profiling as a tool for studying the interaction of chaperones and targeting factors with nascent polypeptide chains and ribosomes. Nature protocols. 8 (11), 2212-2239 (2013).
- Li, G. W., Oh, E., Weissman, J. S. The anti-Shine-Dalgarno sequence drives translational pausing and codon choice in bacteria. Nature. 484 (7395), 538-541 (2012).
- King, H. A., Gerber, A. P. Translatome profiling: methods for genome-scale analysis of mRNA translation. Briefings in functional genomics. 15 (1), 22-31 (2014).
- Mohammad, F., Woolstenhulme, C. J., Green, R., Buskirk, A. R. Clarifying the Translational Pausing Landscape in Bacteria by Ribosome Profiling. Cell reports. 14 (4), 686-694 (2016).
- Reid, D. W., Shenolikar, S., Nicchitta, C. V. Simple and inexpensive ribosome profiling analysis of mRNA translation. Methods (San Diego, Calif). 91, 69-74 (2015).
- Sanz, E., Yang, L., Su, T., Morris, D. R., McKnight, G. S., Amieux, P. S. Cell-type-specific isolation of ribosome-associated mRNA from complex tissues. Proceedings of the National Academy of Sciences of the United States of America. 106 (33), 13939-13944 (2009).
- Petrova, O. E., Garcia-Alcalde, F., Zampaloni, C., Sauer, K. Comparative evaluation of rRNA depletion procedures for the improved analysis of bacterialbiofilm and mixed pathogen culture transcriptomes. Scientific Reports. 7, 41114 (2017).
- Oh, E. Selective ribosome profiling reveals the cotranslational chaperone action of trigger factor in vivo. Cell. 147 (6), 1295-1308 (2011).
- Chew, G. L. Ribosome profiling reveals resemblance between long non-coding RNAs and 5' leaders of coding RNAs. Development. 140 (13), 2828-2834 (2013).
- Dunn, J. G., Foo, C. K., Belletier, N. G., Gavis, E. R., Weissman, J. S. Ribosome profiling reveals pervasive and regulated stop codon readthrough in Drosophila melanogaster. eLife. 2, 01179 (2013).
- Liu, M. J., et al. Translational landscape of photomorphogenic Arabidopsis. The Plant cell. 25 (10), 3699-3710 (2013).
- Rooijers, K., Loayza-Puch, F., Nijtmans, L. G., Agami, R. Ribosome profiling reveals features of normal and disease-associated mitochondrial translation. Nature Communications. 4, 2886 (2013).
- Danielle, L., et al. Fidelity of translation initiation is required for coordinated respiratory complex assembly. Science Advances. 5 (12), 2118 (2019).
- Zoschke, R., Watkins, K. P., Barkan, A. A rapid ribosome profiling method elucidates chloroplast ribosome behavior in vivo. The Plant cell. 25 (6), 2265-2275 (2013).
- Chotewutmontri, P., Barkan, A. Dynamics of chloroplast translation during chloroplast differentiation in maize. PLoS genetics. 12 (7), 1006106 (2016).
- Lee, S., Liu, B., Huang, S. X., Shen, B., Qian, S. B. Global mapping of translation initiation sites in mammalian cells at single-nucleotide resolution. Proceedings of the National Academy of Sciences of the United States of America. 109 (37), 2424-2432 (2012).
- Martinez, T. F., Chu, Q., Donaldson, C., Tan, D., Shokhirev, M. N., Saghatelian, A. Accurate annotation of human protein-coding small open reading frames. Nature chemical biology. 16 (4), 458-468 (2020).
- Simsek, D., et al. The Mammalian Ribo-interactome Reveals Ribosome Functional Diversity and Heterogeneity. Cell. 169 (6), 1051-1065 (2017).
- Fritsch, C., et al. Genome-wide search for novel human uORFs and N-terminal protein extensions using ribosomal footprinting. Genome research. 22 (11), 2208-2218 (2012).
- Lareau, L. F., Hite, D. H., Hogan, G. J., Brown, P. O. Distinct stages of the translation elongation cycle revealed by sequencing ribosome-protected mRNA fragments. eLife. 3, 01257 (2014).
- Meydan, S., et al. Retapamulin-Assisted Ribosome Profiling Reveals the Alternative Bacterial Proteome. Molecular Cell. 74 (3), 481-493 (2019).
- Wilson, D. N. The A-Z of bacterial translation inhibitors. Critical Reviews in Biochemistry and Molecular Biology. 44, 393-433 (2009).
- Nakahigashi, K. Comprehensive identification of translation start sites by tetracycline-inhibited ribosome profiling. DNA research : an international journal for rapid publication of reports on genes and genomes. 23 (3), 193-201 (2016).
- Weaver, J., Mohammad, F., Buskirk, A. R., Storz, G. Identifying Small Proteins by Ribosome Profiling with Stalled Initiation Complexes. mBio. 10 (2), 02819 (2019).
- Nakahigashi, K. Effect of codon adaptation on codon-level and gene-level translation efficiency in vivo. BMC genomics. 15 (1), 1115 (2014).
- Li, G. W., Burkhardt, D., Gross, C., Weissman, J. S. Quantifying absolute protein synthesis rates reveals principles underlying allocation of cellular resources. Cell. 157 (3), 624-635 (2014).
- Michel, A. M., et al. Observation of dually decoded regions of the human genome using ribosome profiling data. Genome research. 22 (11), 2219-2229 (2012).
- Guydosh, N. R., Green, R. Dom34 rescues ribosomes in 3' untranslated regions. Cell. 156 (5), 950-962 (2014).
- Young, D. J., Guydosh, N. R., Zhang, F., Hinnebusch, A. G., Green, R. Rli1/ABCE1 Recycles Terminating Ribosomes and Controls Translation Reinitiation in 3'UTRs In Vivo. Cell. 162 (4), 872-884 (2015).
- Ingolia, N. T. Ribosome profiling: new views of translation, from single codons to genome scale. Nature Reviews Genetics. 15 (3), 205-213 (2014).
- Dobin, A., et al. ultrafast universal RNA seq aligner. Bioinformatics. 29 (1), 15-21 (2013).
- . . Babraham Bioinformatics. Fast QC. , (2021).
- Gerashchenko, M. V., Gladyshev, V. N. Ribonuclease selection for ribosome profiling. Nucleic acids research. 45 (2), 6 (2017).
- Verbruggen, S., Menschaert, G. mQC: A post-mapping data exploration tool for ribosome profiling. Computer Methods and Programs in Biomedicine. 181, 104806 (2019).
- Cui, H., Hu, H., Zeng, J., Chen, T. DeepShape: estimating isoform-level ribosome abundance and distribution with Ribo-seq data. BMC bioinformatics. 20, 678 (2019).
- Choi, J. . RiboToolkit: an integrated platform for analysis and annotation of ribosome profiling data to decode RNA translation at codon resolution. , (2021).
- Choi, J. Dynamics of the context-specific translation arrest by chloramphenicol and linezolid. Nature chemical biology. 16 (3), 310-317 (2020).
- Tompson, J., O'Connor, M., Mills, J. A., Dahlberg, A. E. The protein synthesis inhibitors, oxazolidinones and chloramphenicol, cause extensive translational inaccuracy in vivo. Journal of Molecular Biology. 322 (2), 273-279 (2002).
- Brar, G. A., Weissman, J. S. Ribosome profiling reveals the what, when, where and how of protein synthesis. Nature reviews. Molecular cell biology. 16 (11), 651-664 (2015).
- Glaub, A., Huptas, C., Neuhaus, K., Ardern, Z. Recommendations for bacterial ribosome profiling experiments based on bioinformatic evaluation of published data. Journal of Biological Chemistry. 295, 8999-9011 (2020).
- Sorour, M. H., Hani, H. A., Shaalan, H. F., El-Sayed, M. M. H. Experimental screening of some chelating agents for calcium and magnesium removal from saline solutions. Desalination and Water Treatment. 57 (48-49), 22799-22808 (2015).
- Mohammad, F., Green, R., Buskirk, A. R. A systematically-revised ribosome profiling method for bacteria reveals pauses at single-codon resolution. Elife. 8, 42591 (2019).
- O'Connor, P. B., Li, G. W., Weissman, J. S., Atkins, J. F., Baranov, P. V. rRNA:mRNA pairing alters the length and the symmetry of mRNA-protected fragments in ribosome profiling experiments. Bioinformatics. 29 (12), 1488-1491 (2013).
- Protocol for RNA Clean & Concentrator -25. Zymo Research Available from: https://files.zymoresearch.com/protocols/_r1017_r1018_rna_clean_concentrator-25.pdf (2021)
- Protocol for use with NEBNext Small RNA Library Prep Set for Illumina (E7300, E7580, E7560, E7330). New England Biolabs Available from: https://www.international.neb.com/protocols/2018/03/27/protocol-for-use-with-nebnext-small-rna-library-prep-set-for-illumina-e7300-e7580-e7560-e7330 (2021)
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved