Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
RIBO-seq in Bacteria: a Sample Collection and Library Preparation Protocol for NGS Sequencing
Dans cet article
Résumé
Nous décrivons ici les étapes de la collecte d’échantillons et de la préparation pour RIBO-seq chez les bactéries. Le séquençage des bibliothèques préparées conformément à ces lignes directrices donne suffisamment de données pour une analyse bioinformatique complète. Le protocole que nous présentons est simple, utilise un équipement de laboratoire standard et prend sept jours de la lyse à l’obtention des bibliothèques.
Résumé
La technique de profilage des ribosomes (RIBO-seq) est actuellement l’outil le plus efficace pour étudier le processus de synthèse des protéines in vivo. L’avantage de cette méthode, par rapport à d’autres approches, est sa capacité à surveiller la traduction en cartographiant précisément la position et le nombre de ribosomes sur une transcription d’ARNm.
Dans cet article, nous décrivons les étapes consécutives de la collecte d’échantillons et de la préparation de la méthode RIBO-seq chez les bactéries, en soulignant les détails pertinents à la planification et à l’exécution de l’expérience.
Puisque le RIBO-seq repose sur des ribosomes intacts et des ARNm connexes, l’étape clé est l’inhibition rapide de la traduction et la désintégration adéquate des cellules. Ainsi, nous suggérons la filtration et le flash-congélation dans l’azote liquide pour la récolte cellulaire avec un prétraitement facultatif avec le chloramphénicol pour arrêter la traduction dans les bactéries. Pour la désintégration, nous proposons de broyer les cellules congelées avec du mortier et du pilon en présence d’oxyde d’aluminium pour perturber mécaniquement la paroi cellulaire. Dans ce protocole, un coussin de saccharose ou une ultracentrifugation à gradient de saccharose pour la purification par monosomes n’est pas nécessaire. Au lieu de cela, la séparation d’ADN messagère utilisant l’électrophorèse de gel de polyacrylamide (PAGE) suivie de l’excision ribosomique d’empreinte (bande 28-30 nt) est appliquée et fournit des résultats satisfaisants. Cela simplifie en grande partie la méthode et réduit le temps et les exigences d’équipement pour la procédure. Pour la préparation de la bibliothèque, nous vous recommandons d’utiliser le petit kit d’ARN disponible dans le commerce pour le séquençage Illumina à partir de New England Biolabs, en suivant les directives du fabricant avec un certain degré d’optimisation.
Les bibliothèques d’ADNc qui en résultent présentent la quantité et la qualité appropriées requises pour le séquençage de nouvelle génération (NGS). Le séquençage des bibliothèques préparées selon le protocole décrit donne 2 à 10 mln lectures cartographiées de manière unique par échantillon fournissant des données suffisantes pour une analyse bioinformatique complète. Le protocole que nous présentons est rapide et relativement facile et peut être réalisé avec un équipement de laboratoire standard.
Introduction
La technique de profilage des ribosomes (RIBO-seq) a été développée dans le laboratoire de Jonathan Weissman à l’Université de Californie à San Francisco1. En comparaison avec d’autres méthodes utilisées pour étudier l’expression des gènes au niveau translationnel, RIBO-seq se concentre sur chaque ribosome se liant à l’ARNm et fournit des informations sur son emplacement et le nombre relatif de ribosomes sur une transcription. Il permet de surveiller le processus de synthèse des protéines in vivo et peut fournir une résolution et une précision de codon unique permettant la mesure de la densité du ribosome sur les deux, l’ARNm individuel et le long de l’ensemble du transcriptome dans la cellule. A la base de la technique RIBO-seq se trouve le fait que lors de la traduction le ribosome lie la molécule d’ARNm et protège ainsi le fragment enfoui de la transcription d’une digestion ribonucléase. Lors de l’ajout de la ribonucléase, l’ARNm non protégé est digéré et les fragments enfermés par des ribosomes - généralement de ~ 28-30 nt de long - restent intacts. Ces fragments, appelés empreintes ribosomiques (RF), peuvent ensuite être isolés, séquencés et cartographiés sur la transcription dont ils sont issus, ce qui a permis de détecter la position exacte des ribosomes. En fait, la capacité du ribosome à protéger les fragments d’ARNm est utilisée depuis les années 1960 pour étudier les sites ribosomiques de liaison et d’initiation de traduction (TIS)2,3,4. Cependant, avec les progrès de la technologie de séquençage en profondeur, RIBO-seq est devenu une référence pour la surveillance de la traduction5 qui, grâce à l’engagement du ribosome, peut fournir une information à l’échelle du génome sur la synthèse des protéines6. Le profilage du ribosome a comblé le fossé technologique qui existait entre la quantification du transcriptome et le protéome6.
Pour effectuer le profilage du ribosome, nous devons obtenir le lysat cellulaire de l’organisme qui s’était développé dans les conditions étudiées. La perturbation de ces conditions pendant la collecte et la lyse cellulaires peut fournir des données peu fiables. Pour éviter cela, les inhibiteurs de traduction, la récolte rapide et la congélation instantanée dans l’azote liquide sont couramment utilisés. Les cellules peuvent être lysées par broyage cryogénique dans un homogénéisateur mécanique tel qu’un mélangeur7,8 ou un batteur de perles9,et par trituration à travers une pipette10 ou avec une aiguille11. Le tampon de lyse peut être ajouté juste avant ou peu de temps après la pulvérisation des cellules. Dans notre protocole, nous utilisons de l’azote liquide pour pré-refroidir le mortier et le pilon, ainsi que de l’oxyde d’aluminium comme une approche plus douce de la perturbation de la paroi cellulaire bactérienne, ce qui empêche le cisaillement de l’ARN souvent rencontré lorsque des méthodes telles que la sonification sont appliquées. Après pulvérisation, nous ajoutons un tampon de lyse glacé dans le contenu refroidi du mortier. La sélection d’un tampon de lyse approprié est importante pour obtenir la meilleure résolution des empreintes ribosomiques. Étant donné que la force ionique affecte à la fois la taille rf et la précision du cadre de lecture, il est actuellement recommandé d’utiliser des tampons de lyse à faible force ionique et capacité tampon, même s’il apparaît que la composition tampon n’affecte pas l’occupation ribosomique sur les ARNm11,12. Les composants importants du tampon de lyse sont les ions magnésium, dont la présence empêche la dissociation des sous-unités ribosomiques et inhibe les changements conformationnels spontanés dans les ribosomes bactériens11,13. Les ions calcium jouent également un rôle important et sont essentiels pour l’activité de la nucléase micrococcique (MNase) utilisée dans la méthode de profilage des ribosomes bactériens14. L’addition de guanosine 5′-[β,γ-imido]triphosphate (GMP-PNP), un analogue non hydrolysable du GTP, avec le chloramphénicol inhibe la traduction pendant la lyse15.
Lorsque le lysat est obtenu, il est clarifié par centrifugation et divisé en deux parties, chacune pour un RIBO-seq et un séquençage total à haut débit de l’ARNm (RNA-seq) puisqu’ils sont effectués simultanément(figure 1). RNA-seq fournit un point de référence qui permet la comparaison des données de RIBO-seq et RNA-seq lors de l’analyse des données. Le translatome étudié est défini par la normalisation des empreintes ribosomiques à l’abondance de l’ARNm16. Les données de RNA-seq peuvent également aider à identifier le clonage ou le séquençage d’artefacts17.
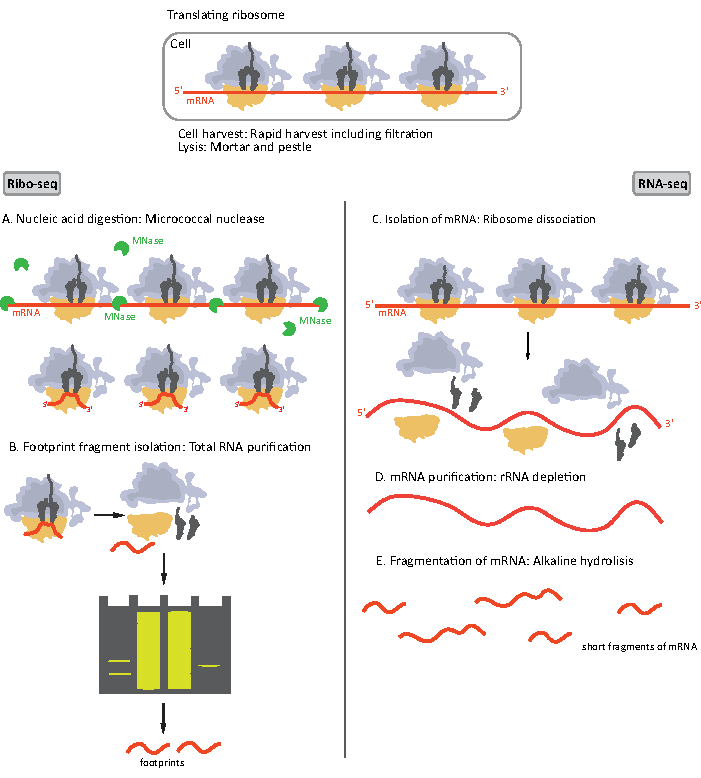
Figure 1. Schémas de préparation d’échantillons d’ARNm pour RIBO-seq et RNA-seq. Pour la préparation de la bibliothèque RIBO-seq, l’ARN est digéré avec de la MNase (A), suivie de la sélection de la taille de RF d’une longueur d’environ 28-30 nt (B); pour l’ARN-seq, l’ARN est isolé (C), appauvri en ARNr (D), et l’ARNm résultant est fragmenté aléatoirement en fragments de longueurs variables (E). Veuillez cliquer ici pour voir une version plus grande de cette figure.
Les étapes initiales de la procédure de préparation de l’échantillon pour RIBO-seq et RNA-seq diffèrent légèrement(Figure 1). Pour le profilage ribosomique, le lysat doit être digéré par une endonucléase spécifique pour dégrader les molécules d’ARNm non protégées par les ribosomes. Dans les protocoles standards, les monosomes obtenus sont récupérés par un coussin de saccharose ultracentrifugation ou un gradient de saccharose d’ultracentrifugation8,14. Dans cet article, nous montrons que cette étape n’est pas nécessaire pour isoler le RF requis pour le RIBO-seq dans les bactéries, de même pour les cellules eucaryotes18,et que la sélection de taille des fragments d’ARNm de longueur appropriée à partir du gel de polyacrylamide est suffisante.
Pour l’ARN-seq, l’ARNm est obtenu par épuisement de l’ARNr à partir de l’ARN total - les molécules d’ARNr s’hybrident aux sondes oligonucléotidiques biotinylées qui se lient aux billes magnétiques recouvertes de streptavidine. Les complexes ARNr-oligonucléotide-billes sont ensuite retirés de l’échantillon avec un aimant aboutissant à un arnr appauvri échantillon19,20. Les molécules d’ARNm purifiées sont ensuite fragmentées aléatoirement par hydrolyse alcaline. Les fragments d’ARNm obtenus ainsi que les empreintes ribosomiques sont convertis en bibliothèques d’ADNc et préparés pour le séquençage en profondeur (Figure 2). Cela implique la réparation des extrémités nécessaires après hydrolyse alcaline (pour l’ARNm) et la digestion enzymatique (pour RF): déphosphorylation des extrémités 3' suivie d’une phosphorylation des extrémités 5'. Les étapes suivantes sont la ligature des adaptateurs et la transcription inverse pour créer des inserts d’ADNc encadrés par des séquences requises pour le séquençage de nouvelle génération (NGS) à l’aide de la plate-forme Illumina. La dernière phase de la préparation de la bibliothèque est une réaction pcr dans laquelle les constructions sont amplifiées et étiquetées avec des codes à barres spécifiques à l’échantillon pour permettre le multiplexage et le séquençage de divers échantillons sur un canal. Avant le séquençage, la qualité et la quantité des bibliothèques sont évaluées par électrophorèse sur puce à haute sensibilité de l’ADN. Les bibliothèques d’ADNc avec les paramètres appropriés peuvent ensuite être regroupées et séquencées. Le séquençage peut être effectué sur différentes plates-formes Illumina, telles que MiSeq, NextSeq ou HighSeq, en fonction du nombre de bibliothèques, de la longueur de lecture requise et de la profondeur de séquençage. Après le séquençage, l’analyse bioinformatique est effectuée.
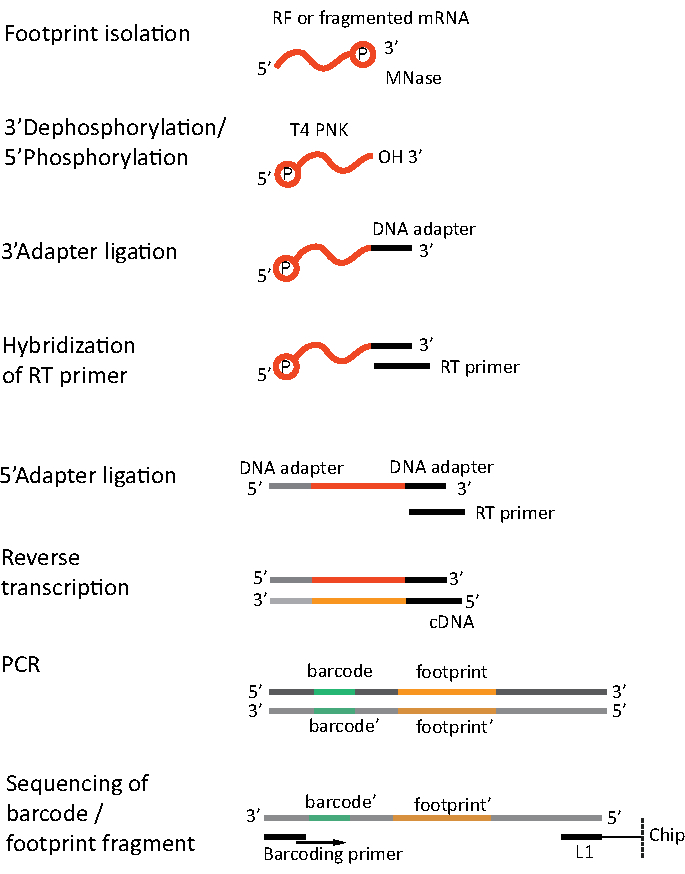
Figure 2. Préparation de la bibliothèque. La préparation de la bibliothèque comprend la réparation des extrémités, la ligature des adaptateurs, la transcription inverse et l’amplification avec codage à barres. Veuillez cliquer ici pour voir une version plus grande de cette figure.
Le profilage des ribosomes est une méthode universelle qui peut être facilement modifiée et ajustée en fonction de la question scientifique. À l’origine, il a été utilisé dans la levure1, mais peu de temps après, il a été appliqué sur les cellules bactériennes21 ainsi que sur des organismes modèles eucaryotes, y compris la souris10,le poisson zèbre22,la mouche des fruits23 et Arabidopsis thaliana24. Il a également été utilisé pour étudier différents types de ribosomes: cytoplasmique, mitochondrial25,26 et chloroplaste27,28. Chez les eucaryotes, ribo-seq est couramment adapté et affiné pour étudier des aspects spécifiques de la traduction, y compris l’initiation10,11,29,30,31,32,l’allongement1,10,11,31,33,le ribosome calant33 et le changement de conformation33. La plupart des modifications impliquent l’utilisation de différents inhibiteurs de traduction. Chez les bactéries cependant, des études analogues ont été difficiles à mener en raison de la rareté des inhibiteurs avec le mécanisme d’action requis34. L’inhibiteur de traduction le plus couramment utilisé chez les bactéries est le chloramphénicol (CAM) qui se lie au centre de la peptidyl transférase (PTC) et empêche le positionnement correct de l’aminoacyl-ARNt dans le site A. En conséquence, cam empêche la formation d’une liaison peptidique qui conduit à arrêter les ribosomes allongés35. D’autres exemples d’inhibiteurs de traduction chez les bactéries sont la tétracycline (TET)36,la retapamuline (RET)34 et l’Onc11237 qui ont été utilisés pour étudier les sites d’initiation de la traduction. Le TET, qui empêche la livraison de tRNA au ribosome en chevauchant directement avec l’anticodon tige-boucle du tRNA au A-site, a été à l’origine appliqué pour vérifier les résultats obtenus du traitement de CAM puisqu’ils sont les deux antibiotiques inhibant l’élongation de traduction38. TET s’est avéré pour détecter tis primaire, cependant n’a pas pu indiquer TISinterne 36. RET se lie au PTC du ribosome bactérien, et empêche la formation de la première liaison peptidique en interférant avec un aminoacyl-ARNt allongé dans le site A. L’application de RET entraîne l’arrêt des ribosomes à la fois primaires et internesTIS 34. Onc112, un peptide antimicrobien riche en proline, se lie dans le tunnel de sortie et bloque la liaison aminoacyl-ARNt dans le site A ribosomique. En conséquence, Onc112 empêche les complexes d’initiation d’entrer dans la phase d’allongement37.
La principale information que fournit le profilage des ribosomes est la densité des ribosomes et leur position sur l’ARNm. Il a été appliqué avec succès pour étudier l’expression différentielle des gènes au niveau de la traduction dans diverses conditions de croissance1,6,mesurer l’efficacité translationnelle1,38,39 et détecter les événements de régulation de la traduction tels que la suspension ribosomique10. Ribo-seq permet également de découvrir la traduction d’ARNc annotés, de pseudogènes et de petits cadres de lecture ouverts non annotés (ORF) conduisant à l’identification de gènes codant des protéines nouvelles et/ou très courtes10,12,22,30,37. Dans de tels cas, RIBO-seq peut affiner et améliorer l’annotation du génome. Avec sa grande sensibilité pour l’identification des ORF traduits et sa nature quantitative, le profilage des ribosomes peut également servir d’indicateur pour la détermination du protéome ou pour faciliter les études protéomiques31,34,39. En cartographiant le TIS, le profilage des ribosomes révèle des isoformes N-terminalement étendues et tronquées de protéines connues10,32. Ribo-seq a également été adapté pour étudier le repliement co-traductionnel des protéines14,21,24. Cette méthode permet de mesurer des vitesses d’allongement1,10,39 ou de décoder des codons individuels6 et aide à développer des modèles quantitatifs de translation17. La méthode de profilage du ribosome est également capable de fournir des informations mécanistes sur le ribosome en pause chez les bactéries7,15,17,le déphasage40,la lecture stop-codon21,les défauts de terminaison/recyclage41,42 et les changements de conformation ribosomique33 chez les eucaryotes. Ribo-seq a également été adapté pour examiner l’impact de facteurs trans-agissants spécifiques sur la traduction tels que les miARN6 et les protéines de liaison à l’ARN chez les eucaryotes16,43. Cependant, il est important de reconnaître que le plan expérimental et la résolution obtenue de RIBO-seq déterminent la quantité d’informations qui peuvent être extraites des données de séquençage résultantes12.
Protocole
1. Prélèvement d’échantillons
- Préparer une culture bactérienne. Nous recommandons un volume de culture de 100 mL par échantillon.
- Préparer l’équipement et les réactifs pour le prélèvement des échantillons : deux scoopulas par échantillon, filtres à membrane d’esters de cellulose (ECM) mélangés stériles de 0,45 μm, tubes stériles de 50 mL, azote liquide, chloramphénicol à 50 mg/mL dans de l’éthanol à 70 % (vol/vol) (facultatif). Percer le couvercle des tubes de 50 mL pour permettre l’évaporation de l’azote liquide et empêcher l’explosion des tubes fermés. Décontaminer les scoopulas avec du détergent de laboratoire et de l’éthanol à 70%.
- Pré-warm l’équipement de filtrage et l’une des scoopulas à la température de croissance de la culture bactérienne.
REMARQUE: Nous recommandons un appareil de filtration entièrement en verre avec un entonnoir, une base fritte, une fiole de joint broyé et une pince à ressort Ø de 47 mm. - Avant la récolte de l’échantillon, ajouter le chloramphénicol dans la culture bactérienne à la concentration finale de 100 μg/mL et incuber 1 minute (facultatif).
- Verser de l’azote liquide dans un tube stérile de 50 mL et placer la deuxième cuillère à l’intérieur du tube pour refroidir.
prudence! L’azote liquide peut faire exploser les récipients fermés en raison du changement de pression lors de l’évaporation. Pour éviter cela, il est nécessaire de créer quelques trous dans le couvercle des tubes de 50 mL pour permettre l’évaporation de l’azote liquide. - Recueillir les cellules par filtration. Arrêtez le filtrage lorsque le milieu a traversé la membrane, mais ne laissez pas le filtre sécher complètement.
- Recueillir les pastilles bactériennes en grattant rapidement les cellules du disque filtrant à l’aide d’une scoopula pré-contre-laguer. Placez immédiatement la scoopula entière avec les cellules récoltées dans le tube de 50 mL rempli d’azote liquide. Le granulé récolté doit être complètement recouvert d’azote liquide.
- Laissez le granulé geler à fond et déloger les cellules congelées à l’aide d’une deuxième cuillère préalablement refroidie. Fermez le couvercle perforé et transférez le tube à -80 °C. Point d’arrêt
NOTA : Désinfecter les scoopulas après chaque récolte d’échantillon.
2. Lyse cellulaire
- Préparer 0,1 M GMP-PNP dans des tubes de réaction Tris pH 8, DNase I et 1,5 mL de 10 mM pour les lysats et les garder sur la glace. Refroidir la centrifugeuse à 4 °C.
- Préparer le tampon de lyse (20 mM TRIS pH 7,6, 10 mM MgAcet, 150 mM KAcet, 0,4% TRITON X-100, 6 mM β-mercaptoéthanol, 5 mM CaCl2 et 1 mM de chloramphénicol en option). Aliquote 500 μL du tampon de lyse par échantillon dans des tubes de réaction de 1,5 mL et les placer sur de la glace.
- Décontaminez le mortier et le pilon avec un détergent de laboratoire et de l’éthanol à 70%, et séchez-les. Refroidir le mortier et le pilon en versant de l’azote liquide dans le mortier.
- Transférer les granulés congelés dans le mortier pré-réfrigéré et les broyer en poudre. Avec une spatule, ajoutez environ 1 volume d’oxyde d’aluminium et continuez à broyer. Gardez le mortier, le pilon et les cellules au frais en versant de l’azote liquide au besoin, ne laissez pas le contenu du mortier décongeler.
- Juste avant d’utiliser le tampon de lyse, ajoutez GMP-PNP et DNase I dans la partie aliquote du tampon de lyse à la concentration finale de 2 mM et 100 U/mL respectivement. Transférer la solution dans le mortier avec des cellules et de l’oxyde d’aluminium et continuer le broyage. Laisser le lysat décongeler lentement pendant le broyage et transférer le mélange dans le tube de réaction pré-refroidi de 1,5 mL et le placer immédiatement sur de la glace.
- Lysats de centrifugeuses à 20 000 x g pendant 5 minutes à 4 °C. Transférer les surnageants dans de nouveaux tubes de réaction pré-refroidis de 1,5 mL et les garder sur la glace.
- Mesurer la concentration d’ARN dans chaque échantillon avec un NanoDrop. Utiliser des dilutions 1:10 des échantillons et une dilution 1:10 du tampon de lyse dans de l’eau sans nucléase comme blanc.
NOTA : Sachez que le chloramphénicol présente une absorption importante à 260 nm. - Divisez chaque lysat en deux parties: une pour RIBO-seq (0,5 - 1 mg d’ARN) et la seconde pour RNA-seq (le reste).
- Nettoyez les échantillons pour l’ARN-seq à l’aide d’un kit de nettoyage de l’ARN disponible dans le commerce conformément au protocole du fabricant.
REMARQUE: Nous vous recommandons d’utiliser le kit Zymo RNA Clean &Concentrate -25 (voir la table des matériaux). Nous recommandons également d’ajouter 4,5 volumes d’éthanol (par rapport au volume de l’échantillon) dans le mélange d’échantillon et de tampon de liaison pour les empreintes ribosomiques et l’ARNm fragmenté, afin d’augmenter l’efficacité de purification des fragments d’ARN courts. - Stocker les échantillons destinés à l’ARN-seq à -80 °C. La procédure pour RNA-seq se poursuivra à partir de l’étape 5.
3. Digestion MNase des échantillons pour RIBO-seq
- À 1 mg d’ARN, ajoutez 3,8 μL de 187,5 U/μL de MNase dans 10 mM tris pH 8 et rechargez-le avec un tampon de lyse jusqu’au volume total de 500 μL.
- Incuber à 25 °C, 300 tr/min, pendant 45 minutes dans un thermomixeur.
- Nettoyez les échantillons avec un kit de nettoyage de l’ARN disponible dans le commerce (comme à l’étape 2.9).
- Conservez les échantillons pour RIBO-seq à -80 °C (point STOP) ou passez à la sélection de la taille.
4. Électrophorèse sur gel de polyacrylamide (PAGE) et sélection de la taille des échantillons pour RIBO-seq
- Préparez un gel polyacrylamide-TBE à 15% avec de l’urée 8 M et placez-le dans un réservoir avec tampon TBE. Pré-exécution pendant au moins 10 minutes à une tension constante de 200 V.
- Mélanger les échantillons avec le tampon d’échantillon de TBE-Urée, les dénaturer à 95 °C pendant 1 minute et les placer immédiatement sur de la glace.
- Lavez l’urée en injectant du tampon TBE dans des puits de gel à l’aide d’une seringue. Chargez les échantillons en laissant un espace de puits entre eux pour séparer chaque échantillon et éviter la contamination croisée. Utilisez 29 nt oligonucléotides et un mélange de 26 nt et 32 nt oligonucléotides comme marqueurs. Exécutez l’électrophorèse à une tension constante de 180 V.
- Préparer un tampon stérile pour l’incubation pendant la nuit (EDTA de 5 mM, 10 mM de NaOAc pH 5).
- Après électrophorèse, colorer le gel dans un bain SYBR Gold pendant environ 2-3 minutes et rincer le gel avec de l’eau sans nucléase.
- Exciser les fragments de gel entre 26 et 32 nt avec les aiguilles stériles ou les lames de rasoir et placer les fragments de gel dans des tubes séparés de 1,5 mL pour chaque échantillon. Changez l’aiguille ou la lame de rasoir entre les échantillons.
- Ajouter 200 μL du tampon d’incubation pendant la nuit à chaque tube de réaction.
- Incuber les échantillons à 10 °C, 1000 rpm pendant la nuit dans un thermomixeur.
- Le lendemain, nettoyez les échantillons comme à l’étape 2.9. Éluer les empreintes dans 80 μL d’eau sans nucléase.
- Conservez les échantillons à -80 °C. Point d’arrêt. La procédure pour RIBO-seq se poursuivra à partir de l’étape 7.
5. Purification de l’ARNm bactérien par épuisement de l’ARNr à partir d’échantillons pour l’ARN-seq
- Retirer l’ARNr des échantillons à l’moyen d’une trousse de purification de l’ARNm bactérien (p. ex. Invitrogen MICROBExpress). Suivez le protocole du fabricant.
- Nettoyez les échantillons comme à l’étape 2.9. Éluer l’ARNm dans 50 μL d’eau sans nucléase.
- Stocker les échantillons pour RNA-seq à -80 °C (point STOP) ou continuer à la fragmentation alcaline.
6. Fragmentation alcaline des échantillons pour l’ARN-seq
- Préparer un tampon d’hydrolyse alcaline (mélanger 220 μL de NaHCO30,1 M, 30 μL de 0,1 MNa2CO3 et 1 μL d’EDTA 0,5 M). Mélanger 1 volume (50 μL) de tampon alcalin avec 1 volume (50 μL) de l’échantillon et incuber à 95 °C pendant 25 minutes.
- Ajouter 5 μL de 3 M De NaOAc pH 5,5 pour arrêter la réaction.
- Nettoyer les échantillons comme à l’étape 2.9 et éluer les échantillons dans 80 μL d’eau sans nucléase.
- Point d’arrêt possible : stocker les échantillons d’ARN-seq à -80 °C. Cependant, nous vous recommandons d’aller directement à l’étape 7 afin d’éviter la congélation et la décongélation inutiles.
7. Déphosphorylation et phosphorylation des échantillons pour l’ARN et le RIBO-seq
- Ajouter 10 μL de PNK de tampon de réaction 10x et 5 μL de PNK T4 à chaque échantillon. Incuber à 37 °C pendant 1,5 heure afin de déphosphoryler les extrémités 3'.
- Ajouter 3 μL d’ATP à 1 mM et incuber à 37 °C pendant 1 heure afin de phosphoryler les extrémités de 5'.
- Nettoyer les échantillons comme à l’étape 2.9, éluer dans de l’eau sans nucléase de 30 μL.
- Conservez les échantillons à -80 °C. Point d’arrêt.
8. Préparation de la bibliothèque à l’aide de NEBNext Multiplex Small RNA Library Prep Set pour Illumina
- Préparer 100-1000 ng d’ARN d’entrée (fragments d’ARNm obtenus et empreintes ribosomiques) dans 6 μL d’eau sans nucléase et ajouter 1 μL d’adaptateur SR 3'. Incuber selon le protocole du fabricant.
- Ajouter 10 μL de tampon de réaction de ligature de 3' (2X) et 3 μL de mélange d’enzymes de ligature de 3' et incuber à 37 °C pendant 2,5 heures, au lieu de 1 heure à 25 °C comme le précise le protocole du fabricant.
- Hybrider l’amorce de transcription inverse selon le protocole du fabricant avec un programme modifié : 5 minutes à 75 °C, 30 minutes à 37 °C et maintenir à 4 °C.
- Ligate l’adaptateur SR 5'. Suivez le protocole du fabricant avec un changement : effectuer l’incubation à 37 °C pendant 2,5 heures, au lieu de 1 heure à 25 °C.
- Effectuez la transcription inverse selon le protocole du fabricant.
- Point STOP possible : incuber les échantillons contenant des ADNc synthétisés à 70 °C pendant 15 minutes pour inactiver la transcriptase inverse et les stocker à -80 °C. Cependant, nous vous recommandons de passer directement aux étapes suivantes pour éviter le gel et le dégel inutiles des échantillons.
- Stockez la moitié de chaque bibliothèque d’ADNc à -80 °C en tant que sauvegarde.
- Effectuez une amplification PCR selon le protocole du fabricant. Utilisez la moitié du mélange de réaction. Stockez les bibliothèques obtenues à -80 °C. Point STOP.
9. Sélection de taille des bibliothèques d’ADNc à l’aide de PAGE
- Préparez le gel polyacrylamide-TBE à 6% et placez-le dans un réservoir avec tampon TBE. Pré-exécution pendant au moins 10 minutes à une tension constante de 200 V.
- Mélanger les échantillons avec gel loading dye, bleu (6X) et les charger sur le gel. Utilisez le résumé d’ADN-MspI pBR322 à chargement rapide comme échelle. Au début, exécutez l’électrophorèse à une tension constante de 120 V pour permettre à l’ADN d’entrer dans le gel, puis modifiez la tension à 180 V.
- Lorsque l’électrophorèse est terminée, colorez le gel dans un bain SYBR Gold pendant environ 2-3 minutes et rincez le gel avec de l’eau sans nucléase.
- Fragments de gel d’accise contenant les bibliothèques. Pour les échantillons d’ARN-seq excise entre 135-180 nt et pour RIBO-seq entre 135-170 nt. Utilisez une aiguille stérile ou une lame de rasoir et placez les fragments de gel excisés dans des tubes de réaction séparés de 1,5 mL. N’oubliez pas de changer l’aiguille ou la lame de rasoir entre les échantillons.
REMARQUE: Les adaptateurs et le code à barres de NEBNext Multiplex Small RNA Library Prep Set pour Illumina ont 119 nt au total, ce qui détermine le seuil d’excision inférieur. - Ajouter 100 μL d’eau sans nucléase à chaque fragment de gel excisé.
- Incuber les fragments de gel à 10 °C, 450 rpm pendant la nuit dans un thermomixeur.
- Nettoyez et concentrez les bibliothèques d’ADNc avec un kit commercial de nettoyage de l’ADN selon le protocole du fabricant.
REMARQUE: Nous vous recommandons d’utiliser le kit Zymo DNA Clean &Concentrate -5 (voir la table des matériaux). - Conservez les bibliothèques d’ADNc purifiées à -80 °C. Point d’arrêt.
Résultats
Les résultats exemplaires présentés ici ont été obtenus dans une étude examinant la régulation de traduction dans les cellules sporulantes de Bacillus subtilis de WT. Pendant la nuit, les cultures ont été diluées à600 DO égales à 0,1 dans 100 mL de milieu riche et incubées à 37 °C avec des secousses vigoureuses jusqu’à ce que la DO600 atteigne 0,5-0,6. Le milieu riche a ensuite été remplacé par un milieu minimal pour induire le processus de sporulation et l’incubatio...
Discussion
Le principal défi technique du profilage des ribosomes est la nécessité d’inhiber rapidement la traduction afin de capturer un instantané des ribosomes sur les ARNm à un état physiologique d’intérêt particulier. Pour ce faire, les inhibiteurs de traduction, la récolte rapide et la congélation instantanée dans l’azote liquide sont couramment utilisés. L’application d’antibiotiques est facultative car ils peuvent causer des artefacts. Le chloramphénicol est un médicament couramment utilisé pour arr...
Déclarations de divulgation
Les auteurs n’ont rien à divulguer.
Remerciements
La SLA tient à souligner le soutien financier des subventions d’installation DE L’EMBO IG 3914 et du POIR. 04.04.00-00-3E9C/17-00 réalisé dans le cadre du programme First TEAM de la Fondation pour la science polonaise cofinancé par l’Union européenne au titre du Fonds européen de développement régional.
matériels
| Name | Company | Catalog Number | Comments |
| 10X TBE (powder) | Invitrogen | AM9864 | |
| 2-Mercaptoethanol, 99%, pure | Acros Organics | 125472500 | |
| Adenosine 5'-Triphosphate (ATP) | New England Biolabs | P0756S | |
| Aluminium oxide calcinated pure p.a. | Chempur | 114560600 | |
| Calcium chloride dihydrate | Sigma-Aldrich | C3881-500G | |
| Chloramphenicol | MP Biomedicals | 190321 | |
| DNA Clean & Concentrator -5 | Zymo Research | D4004 | |
| Dnase I recombinant, Rnase-free | Roche | 4716728001 | |
| EDTA disodium salt | Fisher Scientific | E/P140/48 | |
| Ethyl Alcohol Absolut 99,8% Pure-P.A.-Basic | POCH Avantor Performance Materials Poland S.A | BA6480111 | |
| Filtration apparatus | VWR Collection | 511-0265 | all-glass filtration apparatus, with funnel, fritted base, cap, 47 mm Ø spring clamp and ground joint flask |
| Gel 40 (19:1) | Rotiphorese | 3030.1 | |
| Gel Loading Dye, Blue, 6X | New England Biolabs | E6138G | |
| Guanosine 5′-[β,γ-imido]triphosphate trisodium salt hydrate | Sigma-Aldrich | G0635-25MG | |
| labZAP | A&A Biotechnology | 040-500 | |
| Magnesium acetate tetrahydrate | Sigma-Aldrich | M5661-250G | |
| MCE membrane fiter | Alfatec Technology | M47MCE45GWS | pore size: 0.45um |
| MICROBExpress Bacterial mRNA Purification | Invitrogen | AM1905 | |
| Multiplex Small RNA Library Prep Set for Illumina | New England Biolabs | E7300S | |
| Nuclease-Free Water | Ambion | AM9937 | |
| Potassium Acetate Anhydrous Pure P.A. | POCH Avantor Performance Materials Poland S.A | 744330113 | |
| Quick-Load pBR322 DNA-MspI Digest | New England Biolabs | E7323A | |
| RNA Clean & Concentrator -25 | Zymo Research | R1018 | |
| Sodium acetate | Sigma-Aldrich | S2889-250G | |
| Sodium carbonate | Sigma-Aldrich | 223530-500G | |
| Sodium hydrogen carbonate pure p.a. | POCH Avantor Performance Materials Poland S.A | 810530115 | |
| SYBR Gold nucleic acid gel stain | Life Technologies | S11494 | |
| T4 Polynucleotide Kinase | New England Biolabs | M0201L | |
| T4 Polynucleotide Kinase Reaction Buffer | New England Biolabs | B0201S | |
| TBE-Urea Sample Buffer (2x) | Invitrogen | LC6876 | |
| Tris(hydroxymethyl)amino-methane, ultrapure, 99,9% | AlfaAesar | J65594 | |
| Triton X-100, 98% | Acros Organics | 327371000 | |
| Urea G.R. | lach:ner | 40096-AP0 |
Références
- Ingolia, N. T., Ghaemmaghami, S., Newman, J. R., Weissman, J. S. Genome-wide analysis in vivo of translation with nucleotide resolution using ribosome profiling. Science. 324 (5924), 218-223 (2009).
- Takanami, M., Yan, Y., Jukes, T. H. Studies on the site of ribosomal binding of f2 bacteriophage RNA. Journal of Molecular Biology. 12 (3), 761-773 (1965).
- Steitz, J. A. Polypeptide Chain Initiation: Nucleotide Sequences of the Three Ribosomal Binding Sites in Bacteriophage R17 RNA. Nature. 224 (5223), 957-964 (1969).
- Wolin, S. L., Walter, P. Ribosome pausing and stacking during translation of a eukaryotic mRNA. The EMBO journal. 7 (11), 3559-3569 (1988).
- Argüello, R. J., et al. SunRiSE - measuring translation elongation at single-cell resolution by means of flow cytometry. Journal of Cell Science. 131 (10), 214346 (2018).
- Michel, A. M., Baranov, P. V. Ribosome profiling: a Hi-Def monitor for protein synthesis at the genome-wide scale. Wiley Interdisciplinary Reviews: RNA. 4 (5), 473-490 (2013).
- Woolstenhulme, C. J., Guydosh, N. R., Green, R., Buskirk, A. R. High-precision analysis of translational pausing by ribosome profiling in bacteria lacking EFP. Cell reports. 11 (1), 13-21 (2015).
- McGlincy, N. J., Ingolia, N. T. Transcriptome-wide measurement of translation by ribosome profiling. Methods (San Diego, Calif). 126, 112-129 (2017).
- Gerashchenko, M. V., Gladyshev, V. N. Translation inhibitors cause abnormalities in ribosome profiling experiments. Nucleic acids research. 42 (17), 134 (2014).
- Ingolia, N. T., Lareau, L. F., Weissman, J. S. Ribosome Profiling of Mouse Embryonic Stem Cells Reveals the Complexity of Mammalian Proteomes. Cell. 147 (4), 789-802 (2011).
- Ingolia, N. T., Brar, G. A., Rouskin, S., McGeachy, A. M., Weissman, J. S. The ribosome profiling strategy for monitoring translation in vivo by deep sequencing of ribosome-protected mRNA fragments. Nature protocols. 7 (8), 1534-1550 (2012).
- Hsu, P. Y., et al. Super-resolution ribosome profiling reveals unannotated translation events in Arabidopsis. Proceedings of the National Academy of Sciences of the United States of America. 113 (45), 7126-7135 (2016).
- Blanchard, S. C., Kim, H. D., Gonzalez, R. L., Puglisi, J. D., Chu, S. tRNA dynamics on the ribosome during translation. Proceedings of the National Academy of Sciences of the United States of America. 101 (35), 12893-12898 (2004).
- Becker, A. H., Oh, E., Weissman, J. S., Kramer, G., Bukau, B. Selective ribosome profiling as a tool for studying the interaction of chaperones and targeting factors with nascent polypeptide chains and ribosomes. Nature protocols. 8 (11), 2212-2239 (2013).
- Li, G. W., Oh, E., Weissman, J. S. The anti-Shine-Dalgarno sequence drives translational pausing and codon choice in bacteria. Nature. 484 (7395), 538-541 (2012).
- King, H. A., Gerber, A. P. Translatome profiling: methods for genome-scale analysis of mRNA translation. Briefings in functional genomics. 15 (1), 22-31 (2014).
- Mohammad, F., Woolstenhulme, C. J., Green, R., Buskirk, A. R. Clarifying the Translational Pausing Landscape in Bacteria by Ribosome Profiling. Cell reports. 14 (4), 686-694 (2016).
- Reid, D. W., Shenolikar, S., Nicchitta, C. V. Simple and inexpensive ribosome profiling analysis of mRNA translation. Methods (San Diego, Calif). 91, 69-74 (2015).
- Sanz, E., Yang, L., Su, T., Morris, D. R., McKnight, G. S., Amieux, P. S. Cell-type-specific isolation of ribosome-associated mRNA from complex tissues. Proceedings of the National Academy of Sciences of the United States of America. 106 (33), 13939-13944 (2009).
- Petrova, O. E., Garcia-Alcalde, F., Zampaloni, C., Sauer, K. Comparative evaluation of rRNA depletion procedures for the improved analysis of bacterialbiofilm and mixed pathogen culture transcriptomes. Scientific Reports. 7, 41114 (2017).
- Oh, E. Selective ribosome profiling reveals the cotranslational chaperone action of trigger factor in vivo. Cell. 147 (6), 1295-1308 (2011).
- Chew, G. L. Ribosome profiling reveals resemblance between long non-coding RNAs and 5' leaders of coding RNAs. Development. 140 (13), 2828-2834 (2013).
- Dunn, J. G., Foo, C. K., Belletier, N. G., Gavis, E. R., Weissman, J. S. Ribosome profiling reveals pervasive and regulated stop codon readthrough in Drosophila melanogaster. eLife. 2, 01179 (2013).
- Liu, M. J., et al. Translational landscape of photomorphogenic Arabidopsis. The Plant cell. 25 (10), 3699-3710 (2013).
- Rooijers, K., Loayza-Puch, F., Nijtmans, L. G., Agami, R. Ribosome profiling reveals features of normal and disease-associated mitochondrial translation. Nature Communications. 4, 2886 (2013).
- Danielle, L., et al. Fidelity of translation initiation is required for coordinated respiratory complex assembly. Science Advances. 5 (12), 2118 (2019).
- Zoschke, R., Watkins, K. P., Barkan, A. A rapid ribosome profiling method elucidates chloroplast ribosome behavior in vivo. The Plant cell. 25 (6), 2265-2275 (2013).
- Chotewutmontri, P., Barkan, A. Dynamics of chloroplast translation during chloroplast differentiation in maize. PLoS genetics. 12 (7), 1006106 (2016).
- Lee, S., Liu, B., Huang, S. X., Shen, B., Qian, S. B. Global mapping of translation initiation sites in mammalian cells at single-nucleotide resolution. Proceedings of the National Academy of Sciences of the United States of America. 109 (37), 2424-2432 (2012).
- Martinez, T. F., Chu, Q., Donaldson, C., Tan, D., Shokhirev, M. N., Saghatelian, A. Accurate annotation of human protein-coding small open reading frames. Nature chemical biology. 16 (4), 458-468 (2020).
- Simsek, D., et al. The Mammalian Ribo-interactome Reveals Ribosome Functional Diversity and Heterogeneity. Cell. 169 (6), 1051-1065 (2017).
- Fritsch, C., et al. Genome-wide search for novel human uORFs and N-terminal protein extensions using ribosomal footprinting. Genome research. 22 (11), 2208-2218 (2012).
- Lareau, L. F., Hite, D. H., Hogan, G. J., Brown, P. O. Distinct stages of the translation elongation cycle revealed by sequencing ribosome-protected mRNA fragments. eLife. 3, 01257 (2014).
- Meydan, S., et al. Retapamulin-Assisted Ribosome Profiling Reveals the Alternative Bacterial Proteome. Molecular Cell. 74 (3), 481-493 (2019).
- Wilson, D. N. The A-Z of bacterial translation inhibitors. Critical Reviews in Biochemistry and Molecular Biology. 44, 393-433 (2009).
- Nakahigashi, K. Comprehensive identification of translation start sites by tetracycline-inhibited ribosome profiling. DNA research : an international journal for rapid publication of reports on genes and genomes. 23 (3), 193-201 (2016).
- Weaver, J., Mohammad, F., Buskirk, A. R., Storz, G. Identifying Small Proteins by Ribosome Profiling with Stalled Initiation Complexes. mBio. 10 (2), 02819 (2019).
- Nakahigashi, K. Effect of codon adaptation on codon-level and gene-level translation efficiency in vivo. BMC genomics. 15 (1), 1115 (2014).
- Li, G. W., Burkhardt, D., Gross, C., Weissman, J. S. Quantifying absolute protein synthesis rates reveals principles underlying allocation of cellular resources. Cell. 157 (3), 624-635 (2014).
- Michel, A. M., et al. Observation of dually decoded regions of the human genome using ribosome profiling data. Genome research. 22 (11), 2219-2229 (2012).
- Guydosh, N. R., Green, R. Dom34 rescues ribosomes in 3' untranslated regions. Cell. 156 (5), 950-962 (2014).
- Young, D. J., Guydosh, N. R., Zhang, F., Hinnebusch, A. G., Green, R. Rli1/ABCE1 Recycles Terminating Ribosomes and Controls Translation Reinitiation in 3'UTRs In Vivo. Cell. 162 (4), 872-884 (2015).
- Ingolia, N. T. Ribosome profiling: new views of translation, from single codons to genome scale. Nature Reviews Genetics. 15 (3), 205-213 (2014).
- Dobin, A., et al. ultrafast universal RNA seq aligner. Bioinformatics. 29 (1), 15-21 (2013).
- . . Babraham Bioinformatics. Fast QC. , (2021).
- Gerashchenko, M. V., Gladyshev, V. N. Ribonuclease selection for ribosome profiling. Nucleic acids research. 45 (2), 6 (2017).
- Verbruggen, S., Menschaert, G. mQC: A post-mapping data exploration tool for ribosome profiling. Computer Methods and Programs in Biomedicine. 181, 104806 (2019).
- Cui, H., Hu, H., Zeng, J., Chen, T. DeepShape: estimating isoform-level ribosome abundance and distribution with Ribo-seq data. BMC bioinformatics. 20, 678 (2019).
- Choi, J. . RiboToolkit: an integrated platform for analysis and annotation of ribosome profiling data to decode RNA translation at codon resolution. , (2021).
- Choi, J. Dynamics of the context-specific translation arrest by chloramphenicol and linezolid. Nature chemical biology. 16 (3), 310-317 (2020).
- Tompson, J., O'Connor, M., Mills, J. A., Dahlberg, A. E. The protein synthesis inhibitors, oxazolidinones and chloramphenicol, cause extensive translational inaccuracy in vivo. Journal of Molecular Biology. 322 (2), 273-279 (2002).
- Brar, G. A., Weissman, J. S. Ribosome profiling reveals the what, when, where and how of protein synthesis. Nature reviews. Molecular cell biology. 16 (11), 651-664 (2015).
- Glaub, A., Huptas, C., Neuhaus, K., Ardern, Z. Recommendations for bacterial ribosome profiling experiments based on bioinformatic evaluation of published data. Journal of Biological Chemistry. 295, 8999-9011 (2020).
- Sorour, M. H., Hani, H. A., Shaalan, H. F., El-Sayed, M. M. H. Experimental screening of some chelating agents for calcium and magnesium removal from saline solutions. Desalination and Water Treatment. 57 (48-49), 22799-22808 (2015).
- Mohammad, F., Green, R., Buskirk, A. R. A systematically-revised ribosome profiling method for bacteria reveals pauses at single-codon resolution. Elife. 8, 42591 (2019).
- O'Connor, P. B., Li, G. W., Weissman, J. S., Atkins, J. F., Baranov, P. V. rRNA:mRNA pairing alters the length and the symmetry of mRNA-protected fragments in ribosome profiling experiments. Bioinformatics. 29 (12), 1488-1491 (2013).
- Protocol for RNA Clean & Concentrator -25. Zymo Research Available from: https://files.zymoresearch.com/protocols/_r1017_r1018_rna_clean_concentrator-25.pdf (2021)
- Protocol for use with NEBNext Small RNA Library Prep Set for Illumina (E7300, E7580, E7560, E7330). New England Biolabs Available from: https://www.international.neb.com/protocols/2018/03/27/protocol-for-use-with-nebnext-small-rna-library-prep-set-for-illumina-e7300-e7580-e7560-e7330 (2021)
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon