JoVE 비디오를 활용하시려면 도서관을 통한 기관 구독이 필요합니다. 전체 비디오를 보시려면 로그인하거나 무료 트라이얼을 시작하세요.
Method Article
핵산 프로브를 이용한 뉴클레아제 활성의 운동 스크리닝
* 이 저자들은 동등하게 기여했습니다
요약
변경된 뉴클레아제 활성은 바이오마커로서의 잠재력을 기점으로 다른 인간 조건과 연관되어 있다. 이 논문에 제시된 모듈식 및 구현 방법론은 뉴클레아제 활성을 질병의 바이오마커로 활용하기 위한 특정 핵산 프로브를 선택할 수 있게 한다.
초록
뉴클레아제는 리보오스 설탕을 연결하는 인산 분해 결합의 가수분해를 촉매하여 핵산을 분해하는 효소의 한 부입니다. 핵약은 원핵 생물과 진핵 생물에서 다양한 중요한 생리적 역할을 나타내며, 게놈 안정성 유지에서 병원균에 대한 보호 제공에 이르기까지 다양합니다. 변경된 뉴클레아제 활동은 세균성 감염 및 암을 포함하여 몇몇 병리학적인 조건과 연관되었습니다. 이를 위해, 뉴클레아제 활성은 특정 바이오마커로서 악용될 수 있는 큰 잠재력을 보여주었다. 그러나, 이 활성에 기초한 견고하고 재현 가능한 스크리닝 방법은 매우 바람직하다.
본 명세서에서, 우리는 병리학적 및 건강한 상태 사이의 분화의 범위와 함께, 기질로서 핵산 프로브를 사용하여 뉴클레아제 활성을 스크리닝가능하게 하는 방법을 소개한다. 이 방법을 사용하면 반복적인 방식으로 특이성이 증가하면서 새로운 프로브 라이브러리를 설계할 수 있습니다. 따라서 화학적으로 변형된 핵산의 가용성을 활용하여 향상된 기능으로 프로브의 설계를 구체화하기 위해 여러 라운드의 스크리닝이 필요합니다. 제안된 기술의 상당한 잠재력은 질병 상태와 관련된 핵활성의 스크리닝을 위한 유연성, 높은 재현성 및 다기능성에 있다. 이 기술은 클리닉에서 큰 잠재력을 가진 유망한 진단 도구의 개발을 허용 할 것으로 예상된다.
서문
뉴클레아제는 핵산 분자의 백본 구조를 형성하는 인산염 결합을 분리할 수 있는 효소의 부류입니다. 뉴클레아제의 광대 한 다양성은 그들의 분류를 오히려 어렵게 만듭니다. 그러나, 기질 선호도(deoxyribonucleic acid (DNA) 또는 리보핵산 (RNA)), 절단 부위 (엔도누클레스 또는 외핵약), 또는 금속 이온 의존성, 다른 사람의 사이에서 뉴클레아제를 기술하는 데 사용되는 몇 가지 일반적인 기준이있다 1. 핵활성제는 원핵생물및 진핵생물 모두에서 근본적인 역할을 가지며, 유전자 편집도구로서계속 사용되고 있는 고도로 보존된 촉매 효소이다 2. 그(것)들은 또한 DNA 정비 및 복제에 있는 기본적인 배우입니다, 게놈 안정성을 지키는 것을 돕고 증명 독서 프로세스에참여3. 박테리아에서, 예를 들어, 뉴클레아제는 숙주의 면역 계통의 효능을 감소시킴으로써 세균 생존을 촉진할 수 있는 중요한 독성 인자로 확인되었다4,5,6,7 ,8. 포유동물에서, 뉴클레아제는 세포자멸9,미토콘드리아 생물발생 및유지보수(10) 및 항균 및 항바이러스 선천적 면역 반응의 중재에 연루되는 것으로 제안되었다11. 당연히, 뉴클레아제 활성 변경, 향상 또는 부족 여부, 인간의 질병의 넓은 배열에 연루 되었습니다. 이들 질환은 다양한 암12,13, 심장 비대10 또는 자가면역질환(14)에이르기까지 다양합니다. 따라서, 뉴클레아제는 인간 조건의 이기종 그룹에 대한 바이오마커로서 흥미로운 후보가 되었다. 사실, 뉴클레아제는 이미 황색포도상구균 또는 대장균15와같은 특정 세균성 에이전트에 기인한 감염의 검출을 위한 성공적인 진단 공구로 그들의 잠재력을 보여주었습니다15, 16.많은 암 유형에서, 포도상 구균 뉴클레아제 도메인-함유 단백질 1(SND1) 리보뉴클레아제의 발현은 예후불량을 나타낸다17. 췌장암 환자에서, 높은 리보누클러스 I(RNase I) 혈청 수준은18로 보고되고 암세포 표현형19와연관될 것으로 제안되었다. 허혈성 심장 조건에서, 예컨대 심근경색 또는 불안정한 협심증, 디옥시리보누클리스 I(DNase I) 혈청 수준은 유효한 진단마커(20,21)로나타났다.
뉴클레아제 활성의 글로벌 청사진이 건강하고 질병 상태에서 다를 수 있다는 가설이 있습니다. 실제로, 최근 보고는 뉴클레아제 활성의 차이를 사용하여 건강한표현형(22)을 구별하거나 종별 방식으로 병원성 세균 감염을 식별하기 위해15,23을사용하였다. 이 사실 인정은 질병의 biomarkers로 뉴클레아제의 사용을 위한 새로운 길을 열었습니다. 따라서, 뉴클레아제 활성의 질병 관련 차이를 체계적으로 식별할 수 있는 포괄적인 스크리닝 방법의 개발이 필요하게 되는데, 이는 새로운 진단 도구의 개발에 매우 중요할 수 있다.
본 명세서에서, 우리는 건강하고 건강에 해로운, 또는 세포 또는 박테리아의 종류에 특이적인 활성에서 뉴클레아제 활성 을 구별할 수 있는 민감하고 특이적인 프로브를 식별하기 위해 새로운 시험관내 스크리닝접근법(도 1)을소개하고 기술한다. 핵산의 모듈성을 활용하여, 우리는 서로 다른 서열과 화학의 포괄적인 세트로 구성된 담금질 형광 올리고뉴클레오티드 프로브의 초기 라이브러리를 설계, 둘 다 라이브러리 설계를위한 중요한 매개 변수인. 이들 올리고뉴클레오티드 프로브는 각각 5' 및 3' 말단에서 플루오로포르(플루오레세인 아미드, FAM) 및 퀘이커(tide quencher 2, TQ2)에 의해 측면에있다(표 1). 이 형광 공명 에너지 전달 (FRET) 기반형광 분석법을 사용하여 효소 분해의 역학을 측정함으로써, 우리는 뉴클레아제 활성의 차동 패턴을 구별 할 수있는 후보 프로브를 식별 할 수있었습니다. 건강 또는 질병 상태와 관련이 있습니다. 우리는 후속 선별 단계에서 더욱 구체적인 후보 프로브를 식별할 수 있는 최상의 후보 프로브를 기반으로 새로운 라이브러리를 만드는 반복 프로세스를 설계했습니다. 더욱이, 이 접근은 감도를 증가시키기 위하여 뉴클레아제의 촉매 특성을 이용합니다. 이는 리포터 프로브의 활성 특성과 뉴클레아제의 기질 분자를 지속적으로 처리하는 능력을 활용함으로써 달성되며, 대체 항체 또는 소분자 기반 스크리닝 방법에 비해 주요 이점을 나타냅니다.
이 접근법은 건강 상태와 질병 상태를 구별할 수 있는 특정 핵산 프로브의 식별을 위한 고도로 모듈화되고 유연하며 구현하기 쉬운 스크리닝 도구를 제공하며, 새로운 개발을 위한 훌륭한 플랫폼을 제공합니다. 향후 임상 응용 분야에 맞게 조정할 수 있는 진단 도구입니다. 이와 같이, 이러한 접근법은 살모넬라 티피무리움(이하 살모넬라균)으로부터유래된 뉴클레아제 활성을 이 박테리아의 특이적 식별을 위해 식별하는데 사용되었다. 다음 프로토콜에서, 우리는 운동 분석을 사용하여 세균성 뉴클레아제 활성을 스크리는 방법에 대해 보고한다.
프로토콜
1. 올리고뉴클레오티드 라이브러리 설계 및 준비
- 도서관 디자인
- 다음 기준에 기초하여 모든 프로브에 대해 동일한 길이의 이차 구조 형성을 피하기 위해 8~12-mer 길이의 담금질 형광 올리고뉴클레오티드 프로브 라이브러리를 설계합니다.
- 아데닌(A), 구아닌(G), 시토신(C) 및 티민(T)/우라실(U)의 조합을 포함하는 적어도 하나의 DNA 및 하나의 RNA 랜덤 서열을포함한다(표 1의 DNA 및 RNA 프로브).
참고: 표 1에 기재된 DNA 및 RNA 서열은 DNase 또는 RNase 중 하나인 미지의 유형의 뉴클레아제 활성을 분류하기 위한 초기 기질로서 유용하였다. 이 2개의 순서는 박테리아와 조직 견본에 있는 뉴클레아제 활동 단면도를 검출하는 넓은 기능을 보여주었기 때문에, 어떤 검열든지를 위한 출발점으로 추천됩니다. 뉴클레아제 활성이 이러한 DNA 및 RNA 서열로 관찰되지 않는 경우, 추가적인 올리고뉴클레오티드의 설계가 요구된다. - 주어진 뉴클레아제 활성에 대한 서열 의존성을 결정하기 위해 단일 유형의 뉴클레오티드로 구성된 폴리뉴클레오티드 서열을 포함한다(DNA-폴리 A, DNA-폴리 T, DNA-폴리 C, RNA-폴리 A, RNA-폴리 U, RNA-폴리 C 및 RNA-폴리 G 표 1).
- 초기 DNA 및 RNA 서열과 동일한 뉴클레오티드 잔기 서열을 사용하여, 2'-플루오로 및 2'-O-메틸과 같은 리보오스 설탕의 2'-위치에서 화학적 변형을 포함하는 뉴클레오시드 유사체를 포함하는 서열을 엄격한 단계로 포함 뉴클레아제의 선택성을 증가시다.
- 2'-플루오로 뉴클레오시드 유사체 또는 2'-O-메틸 뉴클레오시드 유사체로 완전히 구성된 완전히 변형된 서열을 포함합니다(모든 2'-F 및 모든 2'-OMe, 표 1)
- 변형되지 않은 천연 퓨린 및 2'-플루오로 피리미딘 뉴클레오시드 유사체로 구성된 키메라 서열을 포함한다(RNA Pyr-2'F, 표 1).
- 변형되지 않은 천연 퓨린 및 2'-O-메틸 피리미딘 뉴클레오시드 유사체로 구성된 키메라 서열을 포함한다(RNA Pyr-2'OMe, 표 1).
- 변형되지 않은 천연 피리미딘 및 2'-플루오로 퓨린 뉴클레오시드 유사체로 구성된 키메라 서열을 포함한다(RNA Pur-2'F, 표 1).
- 변형되지 않은 천연 피리미딘 및 2'-O-메틸 푸린 뉴클레오시드 유사체로 구성된 키메라 서열을 포함한다(RNA Pur-2'OMe, 표 1).
- 아데닌(A), 구아닌(G), 시토신(C) 및 티민(T)/우라실(U)의 조합을 포함하는 적어도 하나의 DNA 및 하나의 RNA 랜덤 서열을포함한다(표 1의 DNA 및 RNA 프로브).
- 다음 기준에 기초하여 모든 프로브에 대해 동일한 길이의 이차 구조 형성을 피하기 위해 8~12-mer 길이의 담금질 형광 올리고뉴클레오티드 프로브 라이브러리를 설계합니다.
- 올리고뉴클레오티드 프로브 준비 및 저장
참고: 올리고뉴클레오티드는 인산포라미드 제 방법에 의해 합성됩니다. 합성 후, 올리고뉴클레오티드는 고성능 액체 크로마토그래피(HPLC)를 사용하여 정제되고 질량은 질량 분석법으로 측정된다.- 동색화된 올리고뉴클레오티드 프로브를 원심분리기를 사용하여 스핀다운하고 트리스-EDTA(TE) 버퍼에서 희석하여 뉴클레아제 분해를 방지합니다. 각 프로브의 수율에 따라 희석 량을 결정하여 500 pmol/μL의 농도로 스톡 솔루션을 렌더링합니다.
- 동우성 올리고뉴클레오티드를 4°C 또는 실온에서 단기간 저장합니다. 장기 보관(수개월에서 수년)을 위해- -20°C에서 동독화된 올리고뉴클레오티드를 저장한다. TE에서 동색 올리고뉴클레오티드를 재박탁하면 스톡 용액을 -20°C 또는 바람직하게는 -80°C에 저장합니다.
2. 세균 배양
- 멸균 조건하에서 극저온 저장 바이알에서 다공성 유리 비드 1개를 가져가십시오.
- 개별 세균 성 식민지(살모넬라와 대장균)를분리하려면, 정제 양으로 보충 된 트립틱 콩 한천 (TSA) 배지를 함유 한 페트리 접시에 직접 비드를 줄무늬 / 압연하여 사분면 방법24를 사용하십시오. 혈액.
- 24 시간 동안 37 °C에서 배양하십시오.
3. 상급 준비
- 고체 매체로부터 단일 콜로니를 50 mL 트립틱 대두 국물(TSB)으로 옮기고 200 rpm에서 24시간 동안 37°C에서 배양합니다.
- TSB(하위 배양)에서 배양(1:500)을 희석하고 200 rpm에서 24시간 동안 37°C에서 배양하여 교반인인큐베이터에서.
참고 : 24 시간 의 배양 후 세균 배양이 고정 된 단계에 109 CFU / mL보다 높은 값에 도달 할 것으로 예상됩니다. - 잠복기 후, 배양을 4,500 x g에서 30분 동안 덮인 멸균 튜브 및 원심분리기로 옮긴다.
- 상급을 수집하고 즉시 사용하십시오. 또는, 상수체를 4°C 또는 -20°C에 저장한다.
4. 뉴클레아제 활동 분석
- 작업 솔루션 준비
- 50 pmol/ μL의 최종 작업 농도에 대한 스톡 솔루션 (1:10 비율) 스톡 솔루션 (500 pmol / μL)을 희석하여 1.5 mL 뉴클레아제 무료 미세 원심 분리 튜브에서 각 프로브에 대해 20 μL 작업 솔루션을 준비하십시오. 이를 위해 MgCl2와 CaCl2를 함유한 인산완충식염수(PBS) 18μL을 2 μL의 프로브 스톡 용액과 혼합합니다.
참고 : 작동 용액에서 올리고 뉴클레오티드는 핵 분해에 더 취약하므로 반응을 설정하기 직전에 작업 용액을 준비하는 것이 좋습니다. - 준비 하는 동안 직접 빛 접촉을 피하고 어둠 속에서 유지 (호 일에 싸여), 형광 을 다룰 때.
- 50 pmol/ μL의 최종 작업 농도에 대한 스톡 솔루션 (1:10 비율) 스톡 솔루션 (500 pmol / μL)을 희석하여 1.5 mL 뉴클레아제 무료 미세 원심 분리 튜브에서 각 프로브에 대해 20 μL 작업 솔루션을 준비하십시오. 이를 위해 MgCl2와 CaCl2를 함유한 인산완충식염수(PBS) 18μL을 2 μL의 프로브 스톡 용액과 혼합합니다.
- 반응 설정
- 형광계(예: Cytation 1)를 37°C로 예온합니다.
- 각 샘플(TSB, 대장균 및 살모넬라)에대해 1.5 mL 뉴클레아제 자유 미세원원지 튜브 세트(1개의 튜브/프로브)를 준비하고 그에 따라 라벨을 붙입니다.
- TSB 멸균 배양 배지, 살모넬라 상판제 또는 대장균 상판제(3.4단계)의 96 μL을 조심스럽게 첨가합니다. 그 후, 그에 따라 프로브 작업 용액의 4 μL / 튜브를 추가합니다. 실온에서 이 단계를 수행합니다.
참고: 1차 스크리닝에는 10개의 프로브가 사용되었고, 2라운드에는 6개의 프로브가 사용되었다. - 균일 한 용액을 얻기 위해 위아래로 파이펫팅하여 철저하게 혼합하십시오. 혼합하는 동안 샘플에 기포가 들어가지 않도록 하십시오.
- 각 용액(프로브 + 상층부 또는 배양 배지)의 95 μL을 검은색 바닥, 미처리 된 96 웰 플레이트의 별도의 웰에 적재한다. 우물의 벽에 가까운 팁으로 조심스럽게 분배하여 적재 시 우물의 거품 형성을 최소화하십시오.
- 뚜껑으로 접시를 덮습니다. 뚜껑을 시각적으로 검사하고 측정 아티팩트를 유발할 수 있는 펜 표시 또는 먼지 입자 축적을 확인합니다. 뚜껑을 새 것으로 교체하십시오.
- 측정 설정
- 소프트웨어 바로 가기아이콘(그림 S1A)을클릭하여 수집 소프트웨어(Gen5 3.05)를 엽니다.
- 작업 관리자 창에서 지금 읽기를 선택하고 새로 를 선택하여 운동 측정 프로토콜(그림S1B)을만듭니다.
- 대화 상자 창에서 온도 설정을 클릭하고 37 °C를 선택합니다. 확인 (그림 S1C)를눌러 설정을 확인하고 저장합니다.
- 절차라는대화 창에서 역학 시작을 클릭합니다. 그런 다음 팝업 대화 상자 창에서 Kinetic Step이라는제목으로 런타임 입력 상자에서 2시간을 선택하고 간격 입력 상자에서 2분 동안 선택합니다. 확인 (그림 S1D)를눌러 설정을 확인하고 저장합니다.
- 대화 상자 창에서 읽기를 클릭하여 프로시저입니다. 그런 다음 팝업 대화 상자 창에서 읽기 방법이라는제목으로 형광 강도를 검색 방법으로 선택하고 끝점/역학을 읽기 유형으로 선택하고 광학 유형으로 필터링합니다. 확인 (그림 S1E)를눌러 설정을 확인하고 저장합니다.
- 팝업 대화 상자 창에서 읽기 단계(Kinetic)에서필터 집합에서 녹색을 선택합니다. 확인 (그림 S2A)를눌러 설정을 확인하고 저장합니다.
- 절차라는대화 상자 창에서 사용 뚜껑을 선택하고 유효성 검사를 클릭하여 만든 프로토콜이 유효한지 확인합니다. 이는 팝업 대화창(그림 S2B)을통해 확인할 수 있습니다.
- 메뉴 모음에서 프로토콜을 선택하고 프로시저(그림 S2C)를선택합니다.
- 절차라는대화 창에서 측정할 웰을 정의합니다(그림S2D).
- 파일 이름 입력상자(그림 S2E)에실험 이름을 입력합니다.
- 뚜껑이 있는 플레이트를 플레이트 리더에 로드합니다. 플레이트가 올바른 방향인지 확인합니다.
- 도구모음(그림 S2F)에서새 단추 읽기를 클릭하여 수집을 시작합니다.
- 데이터 분석
- 플레이트 1(그림 S3A)이라는대화 창에서 측정된 웰 중 하나를 클릭합니다.
- 웰 선택을 클릭하고 잘 선택 대화 상자에 측정된 모든 웰을 포함합니다. 확인을 눌러 설정을 확인하고 저장합니다. (그림S3B)를참조하십시오.
- 표 1이라는 대화 창에서 데이터를 선택하여 표로 된 결과를 시각화합니다(그림S3C).
- 컨텍스트메뉴(그림 S3C)에서 빠른 내보내기를 선택하여 데이터를 스프레드 시트로 내보냅니다.
- 스프레드 시트에서 각 샘플 및 프로브에 대해 그에 따라 데이터 열에 레이블을 지정합니다.
- 상대 형광 단위(RFU) 대 시간(x축: 반응 및 y축 타임라인: RCO)을 플로팅하여 마커 스타일과 라인을 사용하여 역학 그래프를 생성합니다.
참고: 스프레드시트 프로그램(예: Excel)을 사용할 때 그래프 생성에 대한 설명이 적용됩니다. 그러나 다른 프로그램을 사용하여 RFU와 시간을 플로팅하여 얻은 원시 데이터에서 그래프를 생성할 수 있습니다. - 다음 공식25에따라 측정된 각 간격에 대한 효소 반응의 비율을 계산합니다: rate=,
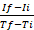 여기서 최대 간격 시간 포인트에서 RFU이고 Ii는 최소 간격 시간 지점에서 RFU인 경우, Tf와 Ti는 각각 최대 및 최소 간격 시간 포인트입니다.
여기서 최대 간격 시간 포인트에서 RFU이고 Ii는 최소 간격 시간 지점에서 RFU인 경우, Tf와 Ti는 각각 최대 및 최소 간격 시간 포인트입니다. - 각 곡선에 대해 가장 높은 값(Rmax)을가진 비율을 선택합니다. 샘플당R max가 두 개 이상인 경우 가장 빠른 측정 시간 지점에서 발생하는 값을 선택합니다.
- R 최대와R 최대가 발생하는 시간 간격의 평균 값 사이의 비율을 계산합니다: 속도 계수 =
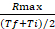
- 각 프로브에 대한 살모넬라 균과 대장균의 비율 계수 사이의 접기 차이(FD)를 계산합니다.
- 3보다 큰 배 차이 값을 유의한 것으로 간주합니다.
- 가장 높은 접기 차이 값(FD)을 가진 중요한 프로브를 후보 프로브로 간주하고 다음 선별 라운드에서 라이브러리 설계의 기초로 고려하십시오.
5. 선발 전형 기준(그림 1)
- 1차 심사
- DNA, RNA 및 폴리뉴클레오티드 프로브를 사용하여 뉴클레아제 활성 분석법(섹션 4에 기재된 대로)을 수행합니다.
- DNA 및 RNA 후보 프로브의 수와 성능(FD 값)을 비교하여 DNA 화학 또는 RNA 화학에 대한 선호도를 평가합니다.
- 그림 1에나와 있는 것처럼 가장 많은 수의 후보 프로브를 렌더링하는 핵산 화학 유형을 선택합니다.
- 2차 심사
- 제1 스크리닝 라운드에서 선택된 핵산 기질에 기초하여 설계된 완전히 변형된 프로브 및 키메라 프로브를 사용하여 뉴클레아제 활성 분석법(섹션 4에 설명된 대로)을 수행한다.
- 2'-플루오로 및 2'-O-메틸 변형 후보 프로브에 대해 얻어진 결과를 비교하여 2'-플루오로 및 2'-O-메틸 뉴클레오시드 유사체를 포함하는 서열에 대한 선호도를 평가한다.
- 그림 1에나와 있는 것처럼 가장 많은 수의 후보 프로브를 렌더링하는 뉴클레오시드 아날로그 수정 유형을 선택합니다.
결과
그림 1은 두 개의 스크리닝 라운드로 나누어진 이 방법론의 작업 흐름을 보여줍니다. 스크리닝의 첫 번째 라운드에서, 우리는 5 개의 DNA 프로브 (DNA, DNA 폴리 A, DNA 폴리 T, DNA 폴리 C 및 DNA 폴리 G)와 5 RNA 프로브 (RNA, RNA 폴리 A, RNA 폴리 U RNA 폴리 C 및 RNA 폴리 G)를 사용했습니다. 이 스크리닝 라운드의 원시 데이터는 보충 표 1에서 찾을 수 있습니다. 두 번째 라운드에?...
토론
뉴클레아제 활성의 변경은 다양한 유형의 암 및 세균 감염을 포함한 다양한 질병 표현형과 연관되어 왔다. 이러한 변화는 조건14의원인인 것으로 제안되며, 다른 경우에는 해로운 생리적사건(20) 또는 병원성제제(16,26)의결과이다. 당연히, 진단 바이오마커로서 뉴클레아제 및 뉴클레아제 활성을 사용하려는 시도는 상?...
공개
저자는 공개 할 것이 없다.
감사의 말
저자는 루이자 I. 에르난데스 (링셰핑 대학)가 원고와 귀중한 조언을 신중하게 수정한 것을 인정하고 싶습니다. 이 작품은 Knut와 앨리스 Wallenberg 재단과 링셰핑 대학에서 고급 기능성 재료에 재료 과학에 스웨덴 정부 전략 연구 영역에 의해 지원되었다 (교수 그랜트 SFO-Mat-LiU 번호 2009-00971).
자료
| Name | Company | Catalog Number | Comments |
| Black bottom, non-treated 96 well plate | Fisher Scientific | 10000631 | |
| Cytation1 | BioTek | CYT1FAV | |
| Eppendorf tubes | Thermofisher | 11926955 | |
| Escherichia coli | ATCC | 25922 | |
| Microbank cryogenic storage vial containing beads | Pro-Lab Diagnostics | 22-286-155 | |
| Nucleic acid probes | Biomers.net | # | |
| Phosphate Buffer Saline containing MgCl2 and CaCl2 | Gibco™ | 14040117 | |
| Salmonella enterica subs. Enterica | ATCC | 14028 | |
| Tris-EDTA | Fisher Scientific | 10647633 | |
| Tryptone Soya Agar with defibrinated sheep blood | Thermo Fisher Scientific | 10362223 | |
| Tryptic Soy Broth | Sigma Aldrich | 22092 |
참고문헌
- Yang, W. Nucleases: diversity of structure, function and mechanism. Quarterly Reviews of Biophysics. 44 (1), 1-93 (2011).
- Adli, M. The CRISPR tool kit for genome editing and beyond. Nature Communication. 9 (1), 1911 (2018).
- Mason, P. A., Cox, L. S. The role of DNA exonucleases in protecting genome stability and their impact on ageing. Age (Dordr). 34 (6), 1317-1340 (2012).
- Berends, E. T., et al. Nuclease expression by Staphylococcus aureus facilitates escape from neutrophil extracellular traps. Journal of Innate Immunity. 2 (6), 576-586 (2010).
- Kiedrowski, M. R., et al. Staphylococcus aureus Nuc2 is a functional, surface-attached extracellular nuclease. PLoS One. 9 (4), 95574 (2014).
- Morita, C., et al. Cell wall-anchored nuclease of Streptococcus sanguinis contributes to escape from neutrophil extracellular trap-mediated bacteriocidal activity. PLoS One. 9 (8), 103125 (2014).
- Hasegawa, T., et al. Characterization of a virulence-associated and cell-wall-located DNase of Streptococcus pyogenes. Microbiology. 156, 184-190 (2010).
- Moon, A. F., et al. Structural insights into catalytic and substrate binding mechanisms of the strategic EndA nuclease from Streptococcus pneumoniae. Nucleic Acids Research. 39 (7), 2943-2953 (2011).
- Li, L. Y., Luo, X., Wang, X. Endonuclease G is an apoptotic DNase when released from mitochondria. Nature. 412 (6842), 95-99 (2001).
- McDermott-Roe, C., et al. Endonuclease G is a novel determinant of cardiac hypertrophy and mitochondrial function. Nature. 478 (7367), 114-118 (2011).
- Wang, Y. T., et al. A link between adipogenesis and innate immunity: RNase-L promotes 3T3-L1 adipogenesis by destabilizing Pref-1 mRNA. Cell Death & Disease. 7 (11), 2458 (2016).
- Li, C. L., Yang, W. Z., Shi, Z., Yuan, H. S. Tudor staphylococcal nuclease is a structure-specific ribonuclease that degrades RNA at unstructured regions during microRNA decay. RNA. 24 (5), 739-748 (2018).
- Wan, L., et al. MTDH-SND1 interaction is crucial for expansion and activity of tumor-initiating cells in diverse oncogene- and carcinogen-induced mammary tumors. Cancer Cell. 26 (1), 92-105 (2014).
- Keyel, P. A. Dnases in health and disease. Developmental Biology. 429 (1), 1-11 (2017).
- Hernandez, F. J., et al. Noninvasive imaging of Staphylococcus aureus infections with a nuclease-activated probe. Nature Medicine. 20 (3), 301-306 (2014).
- Flenker, K. S., et al. Rapid Detection of Urinary Tract Infections via Bacterial Nuclease Activity. Molecular Therapy. 25 (6), 1353-1362 (2017).
- Kannan, N., Eaves, C. J. Tipping the balance: MTDH-SND1 curbs oncogene-induced apoptosis and promotes tumorigenesis. Cell Stem Cell. 15 (2), 118-120 (2014).
- Kemmer, T. P., Malfertheiner, P., Buchler, M., Kemmer, M. L., Ditschuneit, H. Serum ribonuclease activity in the diagnosis of pancreatic disease. International Journal of Pancreatology. 8 (1), 23-33 (1991).
- Fernandez-Salas, E., Peracaula, R., Frazier, M. L., de Llorens, R. Ribonucleases expressed by human pancreatic adenocarcinoma cell lines. European Journal of Biochemistry. 267 (5), 1484-1494 (2000).
- Fujibayashi, K., et al. Serum deoxyribonuclease I activity can be a useful diagnostic marker for the early diagnosis of unstable angina pectoris or non-ST-segment elevation myocardial infarction. Journal of Cardiology. 59 (3), 258-265 (2012).
- Kawai, Y., et al. Diagnostic use of serum deoxyribonuclease I activity as a novel early-phase marker in acute myocardial infarction. Circulation. 109 (20), 2398-2400 (2004).
- Hernandez, L. I., Ozalp, V. C., Hernandez, F. J. Nuclease activity as a specific biomarker for breast cancer. Chemical Communication (Cambridge). 52 (83), 12346-12349 (2016).
- Machado, I., et al. Rapid and specific detection of Salmonella infections using chemically modified nucleic acid probes. Analytica Chimica Acta. 1054, 157-166 (2019).
- Sanders, E. R. Aseptic laboratory techniques: plating methods. Journal of Visualized Experiment. (63), e3064 (2012).
- Worthington Biochemical Corporation. . Manual of Clinical Enzyme Measurements. , (1972).
- Burghardt, E. L., et al. Rapid, Culture-Free Detection of Staphylococcus aureus Bacteremia. PLoS One. 11 (6), 0157234 (2016).
- Bibette, J. Gaining confidence in high-throughput screening. Proceedings of the National Academy of Science U. S. A. 109 (3), 649-650 (2012).
- Hernandez, L. I., Machado, I., Schafer, T., Hernandez, F. J. Aptamers overview: selection, features and applications. Current Topics in Medicinal Chemistry. 15 (12), 1066-1081 (2015).
- Pande, J., Szewczyk, M. M., Grover, A. K. Phage display: concept, innovations, applications and future. Biotechnology Advances. 28 (6), 849-858 (2010).
- Skerra, A. Alternative binding proteins: anticalins - harnessing the structural plasticity of the lipocalin ligand pocket to engineer novel binding activities. FEBS Journal. 275 (11), 2677-2683 (2008).
- Ku, T. H., et al. Nucleic Acid Aptamers: An Emerging Tool for Biotechnology and Biomedical Sensing. Sensors (Basel). 15 (7), 16281-16313 (2015).
- Jayasena, S. D. Aptamers: an emerging class of molecules that rival antibodies in diagnostics. Clinical Chemistry. 45 (9), 1628-1650 (1999).
- Gray, B. P., Brown, K. C. Combinatorial peptide libraries: mining for cell-binding peptides. Chemical Reviews. 114 (2), 1020-1081 (2014).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유