JoVE 비디오를 활용하시려면 도서관을 통한 기관 구독이 필요합니다. 전체 비디오를 보시려면 로그인하거나 무료 트라이얼을 시작하세요.
Method Article
펩티도믹스 연구를 위한 아민의 환원 메틸화를 이용한 샘플 준비 및 상대적 수량
요약
이 문서에서는 열 불활성화를 기반으로 한 시료 준비 방법을 설명하여 내인성 펩티드를 보존하여 분해 후 모독을 피하고, 동위원소 라벨링과 LC-MS를 사용하여 상대적 수량을 유지합니다.
초록
펩티도믹스는 생물학적 샘플에서 펩타이드의 정성적 및 정량적 분석으로 정의될 수 있다. 주요 응용 분야에는 질병 또는 환경 스트레스의 펩티드 바이오마커 를 식별하고, 신경 펩티드, 호르몬 및 생리 활성 세포 내 펩티드를 식별하고, 단백질 가수분해제에서 항균 및 영양 펩티드를 발견하고, 프로테올리틱 프로세스를 이해하는 연구에 사용될 수 있습니다. 최근 시료 제제, 분리 방법, 질량 분석 기술 및 단백질 시퀀싱과 관련된 계산 도구의 사전은 확인된 펩티드 수 및 펩티돔의 증가에 기여하였다. 펩티도믹 연구는 세포에서 자연적으로 발생하는 펩티드를 자주 분석합니다. 여기서, 열불활성화에 기초한 샘플 준비 프로토콜이 기재되어 프로테아제 활성을 제거하고 온화한 조건으로 추출하므로 펩티드 결합 분열이 없다. 또한, 아민의 환원 메틸화에 의한 안정적인 동위원소 라벨링을 이용한 펩타이드의 상대적 양도 도시된다. 이 라벨링 방법은 시약이 시판되고, 다른 사람에 비해 저렴하고, 화학적으로 안정적이며, 단일 LC-MS 실행에서 최대 5개의 시료를 분석할 수 있기 때문에 몇 가지 장점이 있다.
서문
"Omics" 과학은 DNA, RNA, 단백질, 펩타이드, 대사 산물 등과 같은 분자 세트의 심층 분석을 특징으로 합니다. 이러한 생성된 대규모 데이터 세트(유전체학, 전사학, 프로테오믹스, 펩티도믹스, 메타볼로믹스 등)는 생물학에 혁명을 일으켰으며 생물학적 과정에 대한 고급 이해로 이어졌다1. 용어 펩티도믹스는 20 세기 초에 소개되기 시작하고, 몇몇 저자는 proteomics2의 분기로 그것을 언급했습니다. 그러나, 펩티도믹스는 세포 과정 동안 자연적으로 생성된 펩티드 함량뿐만 아니라 이들 분자의 생물학적 활성의 특성화를 조사하는 것이 주요 관심사인 뚜렷한 특수성을 갖는다3,4.
처음에, 생리 활성 펩티드 연구는 에드먼 분해 및 방사선 면역을 통해 신경 펩티드와 호르몬 펩티드에 제한되었다. 그러나, 이들 기술은 고농도각 펩티드의 절연에 따라, 항체의 생성을 위한 시간, 교차 반응성 가능성 외에 글로벌 분석을 허용하지 않는다5.
펩티도믹스 분석은 프로테오믹스/펩티도믹스 연구를 위한 포괄적인 데이터 풀을 전달한 액체 크로마토그래피 결합 질량 분광법(LC-MS) 및 게놈 프로젝트에서 몇 차례 발전한 후에만 가능하였다6,7. 더욱이, 펩티돔에 대한 특정 펩티드 추출 프로토콜은 뇌 샘플에서 전 세계적으로 신경펩티드를 분석한 첫 번째 연구결과에 따라 검출이 단백질의 대규모 분해에 의해 영향을 받았기 때문에 확립될 필요가 있었으며, 이는 주로 이 조직에서 발생하며, 이는 사후 1분 후에 주로 발생한다. 이러한 펩티드 단편의 존재는 신경 펩티드 신호를 마스크하고 생체 내펩티돔을 나타내지 않았다. 이 문제는 주로 전자 레인지 조사를 사용하여 프로테아제의 빠른 가열 불활성화의 적용으로 해결되었으며, 이는 이러한 유물 조각의 존재를 크게 감소시키고 신경 펩티드 조각의 식별뿐만 아니라 세포세포, 미토콘드리아 및 핵 단백질의 존재를 드러냈으며, 데그라돔6,8,9의 다른.
이러한 방법론적 절차는 잘 알려진 신경 펩티드를 넘어 펩티돔의 확장을 허용했으며, 프로테좀의 작용에 의해 주로 생성된 수백 개의 세포내 펩타이드가 효모10, 제브라피시11, 설치류 조직12 및 인간 세포13에서 확인되었다. 이러한 세포 내 펩티드의 수십 광범위하게 생물학적 및 약리학적 활동을 모두 가지고 표시되었습니다14,15. 더욱이, 이들 펩타이드는 질병 바이오마커로 사용될 수 있으며, 두개내 실동맥류16환자로부터 뇌척수액에서 입증된 바와 같이 임상적 중요성을 가질 수 있다.
현재, 펩티드 서열의 식별 외에도 질량 분석법을 통해 절대 및 상대적 수량의 데이터를 얻을 수 있다. 절대 수량에서, 생물학적 샘플의 펩티드 수준은 합성 표준과 비교되며, 상대적 수량에서 펩티드 수준은 2개 이상의 샘플 중에서 비교된다17. 상대적 수량은 다음 방법을 사용하여 수행 할 수 있습니다 : 1) "레이블 무료"18; 2) 생체 내 대사 라벨링 또는 3) 화학 라벨링. 마지막 두 펩티드19,20에 통합된 안정적인 동위원소 형태의 사용을 기반으로 한다. 라벨프리 분석에서 펩타이드 수준은 LC-MS18 동안 신호 강도(스펙트럼 수)를 고려하여 추정된다. 그러나, 동위원소 라벨링은 펩티드의 보다 정확한 상대적 수준을 얻을 수 있다.
많은 펩티도믹 연구는 트리메틸람모늄 부티레이트(TMAB)를 화학 라벨링으로 라벨링하는 시약을 사용했으며, 최근에는 포름알데히드와 시아노보하이드라이드 나트륨 시약의 유정 및 비유화 된 형태를 가진 아민 (RMA)의 환원 메틸화를 사용했습니다111,21,22. 그러나, TMAB 라벨은 상업적으로 사용할 수 없으며, 합성 과정은 매우 힘들다. 한편, RMA에서 시약은 시판되고, 다른 라벨에 비해 저렴하며, 절차는 수행하기가 간단하며, 표지된 펩타이드는 안정23,24이다.
RMA의 사용은 펩티드가 포름알데히드와 반응할 수 있도록 함으로써 쉬프 기지를 형성하는 것을 포함하고, 그 다음에 시아노보로하이드라이드를 통해 환원 반응에 선행됩니다. 이 반응은 N 단자 및 리신 사이드 체인및 단백석 N 단자 프롤라인에 무료 아미노 그룹의 디메틸화를 일으킵니다. 프롤린 잔류물이 N 단자에서 자주 드물며, N-terminus에 무료 아민이 있는 거의 모든 펩타이드는 두 개의 메틸 군23,24,25로 표시된다.
프로토콜
펩티드 추출 및 환원 메틸화를 위한 다음 절차는 이전에 발표된 절차24,25,26,27에서 조정되었다. 이 프로토콜은 국립 동물 실험 통제 위원회 (CONCEA)의 지침을 따랐으며 상파울루 주립 대학의 생물 과학 연구소에서 동물 사용 윤리위원회 (CEUA)의 승인을 받았습니다. 프로토콜 단계는 그림 1에 표시됩니다.
참고: 초순수로 모든 수성 솔루션을 준비합니다.
1. 펩타이드 추출
- 세포 배양
- 15% 태아소 혈청과 1%페니실린-연쇄절제술을 함유한 덜벡코의 수정된 이글 배지에서 37°C에서 15cm 의 접시에 SHSY5Y 세포를 재배한다.
- 각 샘플에 대해 2-3 플레이트를 사용합니다. 세포를 100% 합류로 증가시면 됩니다.
- 치료 용 후 인산염 완충 식염수로 세포를 두 번 씻으하십시오. 다음으로, 10mL의 인산염 완충식식염을 추가하고 세포를 긁어 내고 15mL 튜브로 수집합니다.
- 원심분리기 는 800 x g 에서 5 분 동안 및 상류체를 제거합니다. 80°C에서 초정화 화된 산화수의 1mL에서 펠릿을 재연한다.
- 튜브(셀룰러 리세이트)의 내용을 2mL 마이크로퍼지 튜브로 전송한다.
- 동물 조직 (주로 신경 조직):
- 야생형 남성 성인 다리오 레리오 (zebrafish)를 치명적인 MS 222(100 mg/L)로 마취하고 즉시 펩티다아제와 프로테아제의 비활성화를 위해 8s의 전자레인지 방사선을 투여합니다.
참고: 가정용 전자레인지를 사용할 수 있습니다. 900 W 전자 레인지는 전원에 8 ~ 10 s에 사용 되었습니다. 사용되는 전자 레인지는 10 s 이내에 뇌 온도를 80 °C> 올릴 수 있어야합니다. 시료 간 가열에 있는 재현성은 또한 전자레인지에 있는 동일 위치에 조직을 두어 유익할 것입니다. - 열 불활성화 후, 2mL 미세 분리 튜브에서 전체 뇌를 수집하고 분석 할 때까지 -80 °C에서 동결하십시오.
- 80°C에서 초정화 화된 탈이온수 1mL에서 조직 샘플을 재연한다. 1s의 30 펄스 (4 Hz)를 사용하여 프로브로 조직을 초음파 처리합니다.
참고: 간 및 신장 조직의 경우 20s용 10,000-30,000 rpm에서 기계적 균질화제를 사용하십시오. 근육 조직의 경우, 도자기 도가니와 유봉을 사용하여 액체 질소로 조직을 갈아. 다음 단계는 세포 용해 또는 균주 조직에 대해 동일합니다.
- 야생형 남성 성인 다리오 레리오 (zebrafish)를 치명적인 MS 222(100 mg/L)로 마취하고 즉시 펩티다아제와 프로테아제의 비활성화를 위해 8s의 전자레인지 방사선을 투여합니다.
- 세포 용액 또는 균질 조직을 80 °C에서 20 분 동안 배양합니다. 다음으로, 10-30 분 동안 얼음에 냉각.
- 10mMMM의 최종 농도를 얻기 위해 샘플 부피 1mL에 대해 1M HCl 스톡 솔루션 10μL을 추가합니다. 20s를 위해 소용돌이를 가하고 얼음에 15 분 동안 더 인큐베이션하여 섞습니다.
참고: 산성화하기 전에 높은 온도에서 산성화로 인한 펩티드 결합을 깨지 않도록 시료가 완전히 냉각되었는지 확인하십시오. - 세포 용액 또는 12,000 x g 에서 15 분 동안 4 °C에서 균질 조직을 원심 분리합니다. 낮은 결합 단백질 미세 원심 분리기 튜브에서 상체를 수집하고 -80 ° C에 저장합니다.
- 물과 원심분리기를 2300 x g 에서 3분 동안 추가하여 초여분 장치(10kDa 컷오프 필터)를 청소하십시오. 이 단계를 두 번 더 반복합니다.
- 상체는 미리 세척된 10kDa 컷오프 필터와 원심분리기를 4°C에서 2,300x g에 넣고 냉장고에 50분 간 놓습니다. 플로우루는 펩티드 추출물을 나타낸다.
- 아래 설명된 아세토닐(ACN) 및 트리플루오로아세트산(TFA) 솔루션을 사용하여 제조업체의 지침에 따라 반전 위상 정화 열에 대한 샘플을 탈염합니다.
- 1mL의 1mL로 열을 100% ACN으로 보정합니다.
- 0.1% TFA로 5% ACN의 1mL 용액으로 컬럼을 세척합니다.
- 열에 샘플의 전체 볼륨을 로드합니다.
- 0.1% TFA로 5% ACN의 1mL 용액으로 컬럼을 세척
- 단백질 저결합 마이크로센심분리기 튜브에서 0.15% TFA로 1.8mL 의 1.8mL 용액으로 컬럼에서 펩티드를 엘테.
- 진공 원심분리기에서 샘플을 완전히 건조시하십시오. 유기 용매 및 온도에 대한 농도 방법을 30 °C에서 설정합니다. 농도 시간은 디스플레이에서 모니터링됩니다.
- 다음 단계까지 -80°C에 샘플을 저장합니다.
2. 플루오스카핀을 가진 펩타이드 정량화
참고: 펩타이드의 양은 이전에 설명된 바와 같이 pH 6.8에서 플루오레스카핀을 사용하여 추정될 수 있다11,28. 이 방법은 리신(K) 잔류물 및/또는 펩티드의 N 단말에서 존재하는 1차 아민에 플루오스파민 분자의 부착으로 구성된다. 반응은 pH 6.8에서 수행되어 플루오스카민이 펩티드의 아미노산 그룹과만 반응하고 자유 아미노산이 아니라 반응한다는 것을 보장한다. 불소스카핀은 370nm의 포근파장과 480nm의 방출 파장에서 분광성계를 사용하여 측정된다.
- 표준 펩티드(0.05, 0.1, 0.15, 0.2, 0.3, 0.5 및 0.7 μg/μg/μL)의 상이한 농도를 준비하고 -20°C에 알리쿼트를 저장한다.
참고: 펩티드 5A(LTLRTKL)는 알려진 조성물 및 농도를 가지고 있기 때문에 제안된다. - 아세톤에 플루오스카핀 스톡 솔루션(0.3 mg/mL)을 준비합니다. Aliquot는 마이크로 원심 분리튜브 (1 mL)에서 신속하게 밀봉하고 파라필름을 사용하여 밀봉하고 어둠 속에서 -20 ° C에 저장합니다.
- pH 6.8에서 0.2 M 인산염 버퍼(PB)를 준비합니다.
참고: 0.1M 인산염 완충p 6.8(Na2HPO3 1M의 26.85mL) 및 0.1M 인산염 완충p 6.8(NaH2PO3 1ML 23.15mL)을 250mL의 물로 추가하여 0.2M PB를 준비하십시오. - 100-200 μL의 초정화 수에서 펩티드 샘플을 재연한다.
- 파이펫 2.5 μL 표준 펩티드 농도 및 샘플은 삼중에서 형광 성소에 대한 흰색 96 웰 플레이트에. 0.2 M 인산염 버퍼의 25 μL을 추가합니다.
- 멀티채널 파이펫으로 12.5μL의 플루오스카핀을 추가합니다. 궤도 회전기 셰이커에서 1분 동안 부드럽게 균질화합니다.
- 다음으로, 반응을 중지 멀티 채널 파이펫과 물 110 μL을 추가합니다.
참고: 플루오레스카핀 스톡 솔루션과 초순수수를 두 개의 저수지로 전송하여 멀티채널 파이펫으로 이러한 솔루션을 피펫합니다. - 분광계에서 다음 판독 매개변수를 조정합니다: 상단에서 샘플을 읽고, 370 nm에서 여기 파장, 480 nm에서 방출 파장.
- 분광로계의 판을 읽으십시오.
3. 아민 라벨링의 환원 메틸화
참고: 이 동위원소 라벨링 방법은 포름알데히드와 시아노보로하이드라이드 나트륨 시약의 유정 및 비증칭 형태를 가진 아민 군의 디메틸화에 기초한다. 이 반응의 최종 생성물은 사용 가능한 각 라벨링 부위(lysine 또는 N-terminal)에서 각 펩타이드의 최종 질량에 28Da, 30 Da, 32 Da, 34 Da 또는 36 Da를 추가합니다. 이러한 반응은 MS 스펙트럼에서 관찰된 상이한 형태로 표지된 펩티드에서 m/z 차이를 생성한다(표 1).
주의: 적절한 안전 장비를 사용하여 이러한 화합물을 처리해야 하며 노출을 최소화하기 위해 주의를 기울여야 합니다. 포름알데히드와 시아노보로하이드라이드 나트륨 시약시약이 매우 독성이 있기 때문에 연기 후드에서 수행해야 합니다(시아노보로하이드라이드 나트륨 계량 포함). 담금질 반응 및 산성화 동안, 독성 가스(시안화수소)가 생성될 수 있다.
- 초순수수로 시술 당일 주식이나 시약에서 다음과 같은 신선한 솔루션을 준비하십시오.
- 37%의 포름알데히드(CH2O)의 육수를 4%로 희석합니다.
- 20% 포름알데히드 신체(CD2O)의 주식을 4%로 희석합니다.
- 20 % 중화 된 C13 포름알데히드 (13CD2O)의 주식을 4 %로 희석합니다.
- 준비 0.6 M NaBH3CN.
- 준비 0.6 M NaBD3CN.
- 1% 암모늄 중탄산염의 용액을 준비한다.
- 용액을 5% 포믹산을 준비합니다.
참고: 펩티드의 상대적 양에 관해서는, 사용되는 라벨의 수에 따라 다른 실험 체계가 수행 될 수 있기 때문에 화학 라벨링 절차 중에주의가 필요합니다. 샘플 튜브에 잘못된 시약을 추가하는 인간의 오류 가능성을 줄이기 위해 라벨을 별도의 랙에 레이블의 작은 알리쿼트(aliquots)를 분리하는 것이 좋습니다.
- 펩티드의 최대 25 μg를 포함하는 각 샘플을 준비합니다. 샘플에는 트리스 또는 중탄산암모늄이 포함되어서는 안 됩니다.
참고: 아래에 설명된 양은 100 μL의 부피의 각 샘플에 충분합니다.
연기 후드에서 3.3-3.8 단계로 진행합니다. - 샘플에 1M TEAB의 1/10제부 피를 추가합니다(TEAB의 용액 100mMM의 최종 농도). pH 표시기 용지로 pH를 확인하십시오. 5-8 사이여야 합니다. 필요한 경우 HCl 또는 NaOH로 조정합니다.
- 확립된 라벨링 방식에 따라 유정되지 않은, 유정된 포름알데히드 또는 C13 의 4 μL을 추가합니다. 소용돌이에 의해 5 s에 대 한 혼합.
- 확립된 라벨링 방식에 따라 NaBH3CN(0.6M) 또는 NaBD3CN(0.6M)의 4μL을 추가합니다. 소용돌이에 의해 5 s에 대 한 혼합.
- 실온에서 2시간 동안 연기 후드를 배양하여 30분간격으로 섞어주세요.
- 3.4 및 3.5 단계를 반복합니다. 실온에서 밤새 연기 후드에 샘플을 배양합니다.
- 암모늄 중탄산염 (1%)의 16 μL을 추가하고 소용돌이에 의해 혼합합니다. 얼음 위에 샘플을 놓고, 포름산(5%)의 8μL을 추가하고, 5s의 소용돌이로 섞습니다.
- 샘플을 결합하고 pH를 2-4로 조정하고 이전에 설명된 1.8단계에서와 같이 반전 단계 정리 컬럼에서 결합된 샘플을 탈염한다.
- 진공 원심분리기에서 샘플을 완전히 건조시하십시오. 유기 용매 및 온도에 대한 농도 방법을 30 °C에서 설정합니다. 농도 시간은 디스플레이에서 모니터링됩니다.
- 샘플을 -20°C에 저장합니다.
4. 액체 크로마토그래피 및 질량 분광법
- 메틸 태그와 호환되는 MS 계측기와 결합된 나노HPLC 시스템을 사용하여 LC-MS 분석을 수행합니다.
참고: 다양한 MS 계측기는 RMA 라벨링과 호환됩니다. Orbitrap에 결합 된 나노 HPLC 시스템은 일반적으로 나노 전기 분무 이온 소스를 통해 LC-MS 분석을 수행하는 데 사용됩니다. 먼저, 샘플은 분석 컬럼에서 분리된 프리컬럼 및 펩타이드로 로드된다. 펩타이드의 용출은 5%-45% 아세토닐트리어의 선형 그라데이션을 사용하여 수행되며, 0.1% 포믹산, 90분 동안, 200nL/min의 흐름이 수행된다. 질량 분광계는 데이터 종속 모드에서 작동하도록 설정됩니다. 각 전체 스캔은 10-30 eV, 2.3 Kv의 강도로 획득한 다음 충돌 유발 해리(CID) 단편화를 위해 10개의 가장 높은 피크를 선택합니다. 주입 시간은 100 ms에서 이온 트랩에 설정되며, 포리에 변환 (FT)-MS 주입은 m / z 300-1800에서 1000 ms 30,000의 해상도로 고정됩니다. 최소 5,000개의 카운트와 70s의 동적 제외가 조각화 스캐닝을 수행하는 데 사용됩니다.
5. 펩타이드의 상대적 수량
참고: MS 분광은 질량 분광계 소프트웨어에서 분석됩니다. 다른 태그를 가진 표지된 펩티드의 피크 그룹은 MS 스펙트럼에서 확인됩니다. 상대적 수량은 각 단색 피크의 강도에 의해 계산됩니다. 처리된 각 그룹은 각각의 대조군과 비교된다.
- 원시 샘플 파일을 마우스 오른쪽 두 번 클릭하여 스펙트럼 분석 소프트웨어를 엽니다. 유지 시간(RT) 및 MS 스펙트럼(EM) 크로마토그램을 각각 상단과 하단의 두 탭에 로드합니다.
- 소프트웨어 도구 모음의 디스플레이 및 질량 옵션 아이콘에서 한 번 순차적으로 마우스오른쪽 단추를 클릭하고 질량 정밀도를 4개의 소수점으로 설정합니다.
- RT 탭의 마우스 커서를 어디에나 배치합니다. 분석할 해당 이온의 보존 시간을 찾아 오른쪽 마우스 버튼을 클릭합니다. 선택한 시간의 MS 스펙트럼이 자동으로 EM 탭에 표시됩니다.
- 마우스 커서를 EM 탭의 아무 곳에나 배치합니다. 분석할 이온을 찾습니다.
- 오른쪽 단추를 클릭하고 이온 근처 왼쪽에 있는 인접 한 지역에서 길게 누를 수 있습니다. 그런 다음 마우스를 원하는 범위에서 오른쪽으로 드래그하여 관심 영역을 확대합니다.
- 마우스를 EM 탭에 배치하고 오른쪽 또는 왼쪽 키보드 화살표를 클릭하여 분석할 이온 범위를 정의합니다.
- 마우스 커서를 원하는 시간 간격의 시작 부분에 RT 탭을 다시 배치합니다.
- 마우스를 마우스를 선택한 시간 값까지 마우스를 버튼을 둡니다. 이온의 누적 된 강도는 자동으로 EM 탭에 표시됩니다.
- 스프레드시트에서 m/z, z 및 이온 강도 데이터를 수집합니다.
참고: 추가된 메틸 군없이 각 펩티드의 단이국 질량은 다음 공식에서 계산됩니다.
질량 수정되지 않은 펩티드 = (m/z a x z) - (C a x T) - (1.008 x z)
m/zais는 다양한 태그 조합으로 표지된 각 펩타이드에 대한 단화 피크에 대한 값을 충전하는 관찰된 질량(샘플 번호에 대응하는 =1, 2, 3, 4 또는 5).
z 는 충전 상태입니다.
Ca는 한 쌍의 메틸 그룹의 단색 질량입니다.
a=1의 경우 Ca = 28.0313(1차 아민에 두 개의 CH3 그룹의 순 추가)
a=2의 경우 두 CHD2 그룹의 경우 Ca = 30.0439
a=3의 경우 두 CD2H 그룹에 대한 Ca = 32.0564
a=4의 경우 두 CD3 그룹에 대한 Ca= 34.0690
a=5의 경우 Ca = 13개의 CD3 그룹 2개에 대해 36.0757
T 는 펩티드에 통합된 메틸 그룹의 쌍의 수이다. T=z*(m/z5 - m/z1)/8이라는 다섯 개의 태그를 사용할 때 다음 수식에서 계산할 수 있습니다. 단일 기본 아민을 포함 하 고 따라서 두 개의 메 틸 그룹으로 표시 되는 펩 티 드에 대 한 인접한 라벨을 사용 하는 경우 MS 스펙트럼에 피크 중복 존재. 각 표지된 펩타이드의 피크 강도는 타시마와 프리커25에 의해 기술된 방정식을 사용하여 교정될 수 있다.
6. 펩타이드 식별
- 펩타이드를 식별하려면 데이터베이스 검색 엔진을 사용하여 MS/MS 데이터를 분석합니다29,30.
- 미끼 융합 방법을 사용하여 거짓 발견 속도(FDR)를 계산하려면 미끼 데이터베이스를 검색합니다.
참고: 일반적으로 사용되는 검색 매개 변수는 효소 특이성이 없습니다. 전구체 질량 공차는 15-50 ppm을 설정; 0.5 Da의 단편 이온 질량 허용 오차; 변수 수정: 펩티드 이소토성 메틸화 라벨(L1(+28), L2(+30), L3(+32), L4(+34) 및 L5(+36), 산화 메타히오닌(+15.99 Dceta)의 반응성 아민 및 펩타이드 이소토성 메틸리세이트 라벨의 N-terminus - 그런 다음 펩티드를 현지 신뢰도의 평균으로 정렬하여 FDR ≤5%에 의해 최고의 스펙트럼을 선택하여 이를 필터링합니다.
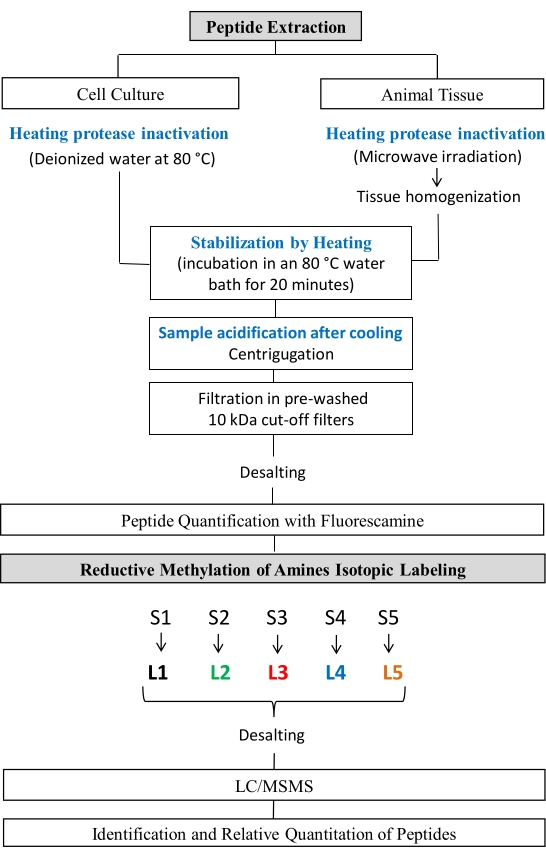
그림 1: 펩티도믹 연구 워크플로우. 아민의 펩티드 추출 및 환원 메틸화단계. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
결과
질량 분광계에서 수행된 실행에서 얻은 결과는 질량 분광계 소프트웨어에서 열 수 있는 원시 데이터 파일에 저장됩니다. MS 스펙트럼에서, 2-5 라벨에 이르기까지 사용되는 라벨링 방식에 따라 표지된 펩티드를 나타내는 피크 그룹을 관찰할 수 있다. 예를 들어 그림 2에서 크로마토그래피 시간에서 검출된 피크 쌍은 동일한 실행에서 두 개의 동위원소 레이블만 사용된 실험에...
토론
대부분의 펩티도믹스 연구에서, 중요한 단계 중 하나는 의심할 여지없이, 몇 분 후 프로테아제에 의해 생성 된 펩티드 단편의 존재를 피하기 위해 신중하게 수행해야하는 샘플 준비입니다. 비전자래드 샘플에서 제조된 뇌 추출물에 대한 초기 연구는 10kDa 마이크로필로테스에 존재하는 많은 단백질 단편을 보여주었다. 단백질 분해로부터 펩티드 스펙트럼을 피하기 위해 다른 접근법이 설명되었습?...
공개
경쟁적인 재정적 이익은 존재하지 않습니다.
감사의 말
여기에 설명 된 기술의 개발 및 사용은 브라질 국가 연구위원회 보조금 420811/2018-4 (LMC)에 의해 지원되었다; Fundação de Amparo à Pesquisa do Estado de São Paulo (www.fapesp.br) 교부금 2019/16023-6 (LMC), 2019/17433-3 (LOF) 및 21/01286-1 (MEME). 기금은 연구 설계, 데이터 수집 및 분석, 게시 결정 또는 문서 작성에 아무런 역할이 없었습니다.
자료
| Name | Company | Catalog Number | Comments |
| 10 kDa cut-off filters | Merck Millipore | UFC801024 | Amicon Ultra-4, PLGC Ultracel-PL Membrane, 10 kDa |
| Acetone | Sigma-Aldrich | 179124 | |
| Acetonitrile | Sigma-Aldrich | 1000291000 | |
| Ammonium bicarbonate | Sigma-Aldrich | 11213 | |
| analytical column (EASY-Column) | EASY-Column | (SC200) | 10 cm, ID75 µm, 3 µm, C18-A2 |
| Ethyl 3-aminobenzoate methanesulfonate | Sigma-Aldrich | E10521 | MS-222 |
| Fluorescamine | Sigma-Aldrich | F9015 | |
| Formaldehyde solution | Sigma-Aldrich | 252549 | |
| Formaldehyde-13C, d2, solution | Sigma-Aldrich | 596388 | |
| Formaldehyde-d2 solution | Sigma-Aldrich | 492620 | |
| Formic acid | Sigma-Aldrich | 33015 | |
| Fume hood | Quimis | Q216 | |
| Hydrochloric acid - HCl | Sigma-Aldrich | 258148 | |
| LoBind-Protein retention tubes | Eppendorf | EP0030108116-100EA | |
| LTQ-Orbitrap Velos | Thermo Fisher Scientific | LTQ Velos | |
| Microwave oven | Panasonic | NN-ST67HSRU | |
| n Easy-nLC II nanoHPLC | Thermo Fisher Scientific | LC140 | |
| PEAKS Studio | Bioinformatics Solutions Inc. | VERSION 8.5 | |
| Phosphate-buffered saline | Invitrogen | 3002 | tablets |
| precolumn (EASY-Column) | Thermo Fisher Scientific | (SC001) | 2 cm, ID100 µm, 5 µm, C18-A1 |
| Refrigerated centrifuge | Hermle | Z326K | for conical tubes |
| Refrigerated centrifuge | Vision | VS15000CFNII | for microtubes |
| Reversed-phase cleanup columns (Oasis HLB 1 cc Cartridge) | Waters | 186000383 | Oasis HLB 1 cc Cartridge |
| Sodium cyanoborodeuteride - NaBD3CN | Sigma-Aldrich | 190020 | |
| Sodium cyanoborohydride - NaBH3CN | Sigma-Aldrich | 156159 | |
| Sodium phosphate dibasic | Sigma-Aldrich | S9763 | NOTE: 0.2 M PB= 0.1 M phosphate buffer pH 6.8 (26.85 mL of Na2HPO3 1M) plus 0.1 M phosphate buffer pH 6.8 (23.15 mL of NaH2PO3 1M) to 250 ml of water |
| Sodium phosphate monobasic | Sigma-Aldrich | S3139 | |
| Sonicator | Qsonica | Q55-110 | |
| Standard peptide | Proteimax | amino acid sequence: LTLRTKL | |
| Triethylammonium buffer - TEAB 1 M | Sigma-Aldrich | T7408 | |
| Trifluoroacetic acid - TFA | Sigma-Aldrich | T6508 | |
| Ultra purified water | Milli-Q | Direct-Q 3UV | |
| Vacuum centrifuge | GeneVac | MiVac DNA concentrator | |
| Water bath | Cientec | 266 | |
| Xcalibur Software | ThermoFisher Scientific | OPTON-30965 |
참고문헌
- Kandpal, R., Saviola, B., Felton, J. The era of 'omics unlimited. Biotechniques. 46 (5), 354-355 (2009).
- Farrokhi, N., Whitelegge, J. P., Brusslan, J. A. Plant peptides and peptidomics. Plant Biotechnology Journal. 6 (2), 105-134 (2008).
- Schulz-Knappe, P., Schrader, M., Zucht, H. D. The peptidomics concept. Combinatorial Chemistry & High Throughput Screening. 8 (8), 697-704 (2005).
- Dallas, D. C., et al. Current peptidomics: applications, purification, identification, quantification, and functional analysis. Proteomics. 15 (5-6), 1026-1038 (2015).
- Chard, T. An introduction to radioimmunoassay and related techniques (3rd Ed). FEBS Letters. 238 (1), 223 (1988).
- Svensson, M., Sköld, K., Svenningsson, P., Andren, P. E. Peptidomics-based discovery of novel neuropeptides. Journal of Proteome Research. 2 (2), 213-219 (2003).
- Baggerman, G., et al. Peptidomics. Journal of Chromatography B. 803, 3-16 (2004).
- Theodorsson, E., Stenfors, C., Mathe, A. A. Microwave irradiation increases recovery of neuropeptides from brain tissues. Peptides. 11, 1191-1197 (1990).
- Che, F. Y., Lim, J., Pan, H., Biswas, R., Fricker, L. D. Quantitative neuropeptidomics of microwave-irradiated mouse brain and pituitary. Molecular & Cellular Proteomics. 4, 1391-1405 (2005).
- Dasgupta, S., et al. Analysis of the yeast peptidome and comparison with the human peptidome. PLoS One. 11 (9), 0163312 (2016).
- Teixeira, C. M. M., Correa, C. N., Iwai, L. K., Ferro, E. S., Castro, L. M. Characterization of Intracellular Peptides from Zebrafish (Danio rerio) Brain. Zebrafish. 16 (3), 240-251 (2019).
- Fricker, L. D. Analysis of mouse brain peptides using mass spectrometry-based peptidomics: implications for novel functions ranging from non-classical neuropeptides to microproteins. Molecular BioSystems. 6 (8), 1355-1365 (2010).
- Gelman, J. S., Sironi, J., Castro, L. M., Ferro, E. S., Fricker, L. D. Peptidomic analysis of human cell lines. Journal of Proteome Research. 10 (4), 1583-1592 (2011).
- De Araujo, C. B., et al. Intracellular peptides in cell biology and pharmacology. Biomolecules. 9, 150 (2019).
- Gewehr, M. C. F., Silverio, R., Rosa-Neto, J. C., Lira, F. S., Reckziegel, P., Ferro, E. S. Peptides from natural or rationally designed sources can be used in overweight, obesity, and type 2 diabetes therapies. Molecules. 25 (5), 1093 (2020).
- Sakaya, G. R., et al. Peptidomic profiling of cerebrospinal fluid from patients with intracranial saccular aneurysms. Journal of Proteomics. 240 (3), 104188 (2021).
- Fricker, L. Quantitative peptidomics: General considerations. Methods in Molecular Biology. 1719, 121-140 (2018).
- Southey, B. R., et al. Comparing label-free quantitative peptidomics approaches to characterize diurnal variation of peptides in the rat suprachiasmatic nucleus. Analytical Chemistry. 86 (1), 443-452 (2014).
- Chen, X., Wei, S., Ji, Y., Guo, X., Yang, F. Quantitative proteomics using SILAC: Principles, applications, and developments. Proteomics. 15 (18), 3175-3192 (2015).
- Boonen, K., et al. Quantitative peptidomics with isotopic and isobaric tags. Methods in Molecular Biology. 1719, 141-159 (2018).
- Gewehr, M. C. F., et al. The relevance of thimet oligopeptidase in the regulation of energy metabolism and diet-induced obesity. Biomolecules. 10 (2), 321 (2020).
- Fiametti, L. O., Correa, C. N., Castro, L. M. Peptide profile of zebrafish brain in a 6-OHDA-induced Parkinson model. Zebrafish. 18 (1), 55-65 (2021).
- Boersema, P. J., Raijmakers, R., Lemeer, S., Mohammed, S., Heck, A. J. Multiplex peptide stable isotope dimethyl labeling for quantitative proteomics. Nature Protocols. 4 (4), 484-494 (2009).
- Dasgupta, S., Castro, L. M., Tashima, A. K., Fricker, L. Quantitative peptidomics using reductive methylation of amines. Methods in Molecular Biology. 1719, 161-174 (2018).
- Tashima, A. K., Fricker, L. D. Quantitative peptidomics with five-plex reductive methylation labels. Journal of the American Society for Mass Spectrometry. 29 (5), 866-878 (2018).
- Che, F. Y., et al. Optimization of neuropeptide extraction from the mouse hypothalamus. Journal of Proteome Research. 6 (12), 4667-4676 (2007).
- Lyons, P. J., Fricker, L. D. Peptidomic approaches to study proteolytic activity. Current Protocols in Protein Science. , 13 (2011).
- Udenfriend, S., et al. Fluorescamine: a reagent for assay of amino acids, peptides, proteins, and primary amines in the picomole range. Science. 178 (4063), 871-872 (1972).
- Ma, B., et al. PEAKS: powerful software for peptide de novo sequencing by tandem mass spectrometry. Rapid Communications in Mass Spectrometry. 17 (20), 2337-2342 (2003).
- Zhang, J., et al. PEAKS DB: de novo sequencing assisted database search for sensitive and accurate peptide identification. Molecular & Cellular Proteomics. 11 (4), 1-8 (2012).
- Sturm, R. M., Dowell, J. A., Li, L. Rat brain neuropeptidomics: tissue collection, protease inhibition, neuropeptide extraction, and mass spectrometric analysis. Methods in Molecular Biology. 615, 217-226 (2010).
- Fricker, L. D. Limitations of mass spectrometry-based peptidomic approaches. Journal of the American Society for Mass Spectrometry. 26 (12), 1981-1991 (2015).
- Ross, , et al. Multiplexed protein quantitation in Saccharomyces cerevisiae using amine-reactive isobaric tagging reagents. Molecular & Cellular Proteomics. 3, 1154-1169 (2004).
- Thompson, A., et al. Tandem mass tags: a novel quantification strategy for comparative analysis of complex protein mixtures by MS/MS. Analytical Chemistry. 75, 1895-1904 (2003).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유