Method Article
A interferência RNA larval no Red Flour Beetle,
* Estes autores contribuíram igualmente
Neste Artigo
Erratum Notice
Resumo
Interferência de RNA (RNAi) técnicas de genes baseados em knockdown estão no centro de pesquisa Tribolium. Aqui, nós fornecemos uma visão geral de nossa técnica de RNAi larval em Tribolium castaneum. Larval RNAi é uma técnica simples, mas poderosa que fornece acesso rápido a fenótipos de perda de função, permitindo que os pesquisadores para estudar as funções dos genes em diversos contextos.
Resumo
O escaravelho vermelho da farinha, Tribolium castaneum, oferece um repertório de ferramentas experimentais para estudos genéticos e de desenvolvimento, incluindo uma seqüência totalmente anotada do genoma, transgênese com base em transposon, e RNA de interferência efetiva (RNAi). Entre essas vantagens, as técnicas genéticas knockdown baseadas em RNAi estão no centro de pesquisa Tribolium. T. castaneum mostram uma resposta sistémica RNAi robusto, tornando-se possível a realização de RNAi em qualquer fase da vida, simplesmente injectando o RNA de cadeia dupla (dsRNA) para dentro da cavidade do corpo do besouro.
Neste relatório, nós fornecemos uma visão geral de nossa técnica de RNAi larval em T. castaneum. O protocolo inclui (i) isolamento da fase apropriada de T. larvas castaneum para injecção, (ii) preparação para a configuração de injecção, e (iii) a injecção de ARNcd. Larval RNAi é uma técnica simples, mas poderosa, que nos fornece acesso rápido a phenotyp perda de funçãoes, incluindo vários genes fenótipos knockdown, bem como uma série de fenótipos hipomórfico. Uma vez que praticamente todos os T. castaneum tecidos são sensíveis a ARNdc extracelular, a técnica permite RNAi larval investigadores a estudar uma ampla variedade de tecidos em diversos contextos, incluindo a base genética de respostas organismais para o ambiente exterior. Além disso, a simplicidade dessa técnica estimula mais o envolvimento dos alunos na pesquisa, tornando T. castaneum um sistema genético ideal para uso em sala de aula.
Introdução
O escaravelho vermelho da farinha, Tribolium castaneum, está ganhando popularidade em várias áreas da biologia, em parte devido à facilidade de realização de interferência de RNA (RNAi) 1-3. Técnicas genéticas knockdown baseada em RNAi permitir que os cientistas analisam-perda de função sem a utilização de métodos genéticos complexos. T. castaneum mostram uma resposta sistémica RNAi robusto, tornando-se possível a realização de RNAi em qualquer estágio através de uma simples injecção de ARN de cadeia dupla (dsRNA) para dentro do corpo da cavidade do besouro 4-6. Knockdown simultânea de vários genes também é viável em T. castaneum por injecção de duas ou mais moléculas de dsRNA diferentes ao mesmo tempo 7,8. Além disso, uma série de fenótipos hipomórfico podem ser gerados através da redução da concentração de dsRNA injectado 8. Estas características tornam técnicas de genética reversa baseada em RNAi alternativas atraentes para a genética termo tradicional em T. castaneum. Uma vez que praticamentetudo T. tecidos castaneum são suscetíveis a moléculas de dsRNA extracelulares 9, esta técnica permite aos pesquisadores estudar uma grande variedade de tecidos em diversos contextos. Além disso, embora o presente relatório incide sobre a realização de RNAi em T. castaneum, muitos procedimentos aqui descritos são aplicáveis a outros insectos. Portanto, este protocolo é útil para aqueles que desejam realizar análises, perda de função em seus contextos de interesse em T. castaneum, bem como para pesquisadores que desejam aplicar uma técnica baseada em RNAi para outros insetos.
A injeção de dsRNA em larvas permite a análise funcional em vários estágios da vida besouro, incluindo a larva, pupa e adulto encena 4,5,10. Temos relatado anteriormente o nosso protocolo de RNAi larval total, incluindo os procedimentos de biologia molecular 11. No relatório atual, nos concentramos em descrever os procedimentos de injeção de dsRNA, que são melhor explicados com assessores visuais. Nós fornecemosprocedimentos detalhados passo-a-passo de injeção, assim como bons e maus exemplos de injeção. Este protocolo visuais complementa nosso protocolo anterior, e quando combinadas, fornecem uma visão mais abrangente dos procedimentos de larvas de RNAi em T. castaneum. Além disso, vamos discutir parâmetros para moléculas de dsRNA que podem afetar o sucesso de RNAi, aplicação de testes baseados em RNAi para a pesquisa fisiológica, bem como a aplicabilidade do protocolo de RNAi larval em um laboratório de ensino.
Protocolo
1 T. Ações castaneum e cultivando
- Decidir sobre um T. castaneum tensão a ser usado para o experimento.
NOTA: Vários T. estabelecida em laboratório cepas castaneum estão disponíveis. Ga-1 (Georgia-1) é a cepa parental de Ga-2, que foi usado para a sequenciação do genoma 3. Desde Ga-1 partes maioria dos polimorfismos de DNA (como polimorfismos de nucleotídeo único, SNPs) com Ga-2, e é mais saudável do Ga-2 devido ao menor endogamia, é ideal para experimentos de RNAi. Pu-11 é outra estirpe conveniente de usar , como o próprio amarelo proteína fluorescente (EYFP) maior expressão única no futuro primórdios ala nos permite distinguir o último estágio larval de outros estádios (veja a Figura S4 de Clark-Hachtel et al. 2.013 12). É importante documentar que tensão besouro foi utilizado no experimento, como origens genéticas parecem significativamente affect o RNAi fenótipos 13. - Prepare T. castaneum farinha cultura pela adição de 5% (em peso) de levedura de cerveja pré-peneirado para orgânica farinha de trigo integral (após mistura, armazenar a farinha de cultura à temperatura de -20 ° C). Alíquota da farinha de cultura (à temperatura ambiente) em garrafas de plástico de 6 onças banco de Drosophila (cerca de 40 g / frasco).
- Cultura T. castaneum a 30 ° C com 70% de humidade. Use uma incubadora com humidade controlada, se possível. Adicionar 30-40 adultos por frasco de cultura (relação homem: mulher 1: 1) para iniciar a cultura.
NOTA: Apesar de molde raramente é um problema a 30 ° C, temperaturas mais baixas (por exemplo, 25 ° C), com 70% de umidade pode causar o crescimento de fungos na cultura farinha. Abaixe a umidade se isso ocorrer. - Transfira os adultos para um novo frasco de cultura a cada duas semanas para a repicagem (veja o passo 5,2-5,5 para saber como isolar adultos a partir de farinha de cultura).
NOTA: Demora cerca de três semanas para obter a última larvas. Alternatvamente, T. castaneum culturas podem ser mantidas a uma temperatura mais baixa (2.025 ° C), embora com um período mais longo a cultura (tempo de desenvolvimento a 25 ° C é aproximadamente o dobro que a 30 ° C).
2: Preparação de soluções e instrumentos para larval injeção dsRNA
- Sintetizar moléculas de dsRNA homólogas ao gene alvo por transcrição in vitro. Armazenar a -80 ° C. Veja Philip e Tomoyasu 2011 11 para os procedimentos de biologia molecular detalhadas. Ver também a discussão para um número de parâmetros que devem ser considerados no projeto de moléculas de dsRNA.
- Adicione 10 mL de tampão fosfato de sódio 0,1 M (pH 7,6 a 25 ° C) por mistura de 8,5 ml de 1 M de Na 2 HPO 4 com 1,5 ml de 1 M NaH 2 PO 4. Verificar o pH com um medidor de pH e ajustar em conformidade.
- Adicione 1 ml de 10x tampão de injecção através da mistura de 10 mL de tampão de fosfato de sódio 0,1 M (pH 7,6 a 25 ° C), 100 _6, l 0,5 M de KCl, 100 ul de corante alimentar verde e 790 ul de água bidestilada (ddH2O). Diluir o tampão 10x injecção para perfazer 2x tampão de injecção (200 ul de tampão de injecção 10x + 800 ul ddH2O). Armazenar ambos 10x e injecção 2x tampões a 4 ° C até ser necessário.
- Prepare Fixo lâminas de vidro.
- Cubra toda uma lâmina de vidro com uma forma de cola reposicionável para injeções de larvas. Para adultos ou pupa injeções, fazer duas tiras finas de cola ao longo das mais longas bordas do slide.
- Deixe a cola secar por vários dias, o que garante que a cola permanece brega o suficiente para aderir besouros para o slide, ao mesmo tempo, permanecendo flexível o suficiente para facilitar a sua remoção após a injeção.
NOTA: Se a cola fica muito pegajoso, use um dedo e toque na cola, o que reduzirá a aderência. Cada slide pode conter até 40 ou 50 besouros, e pode ser reutilizado até que ele não consegue controlar com segurança os animais. Alterntivamente, fita de dupla face pode também ser usado, embora a fita adesiva, por vezes não é suficiente para manter as larvas.
- Faça uma cesta de eterização.
- Retirar o êmbolo de uma seringa descartável de 10 ml, a seringa e cortar ao meio em torno da linha 6 ml.
- Descartar a metade inferior da seringa. Resumidamente aquecer a superfície de corte da parte superior da seringa, com um queimador de gás, e empurrar rapidamente o plástico derretido sobre um pedaço de malha de nylon para colar a malha na seringa. Apare qualquer excesso de malha, uma vez que o plástico endurece.
- Prepare o frasco éter. Adicionar 30 ml de éter a uma boca estreita de 100 ou 250 ml frasco de vidro. Adicionar várias peças de papel de seda para aumentar a evaporação do éter.
NOTA: Ether apresenta um risco de incêndio e também é prejudicial. Sempre lidar com éter sob uma coifa. Feche bem a tampa quando não estiver em uso.
NOTA: O gelo pode ser utilizado como uma alternativa mais segura (tal como, num ambiente de sala de aula), embora o sedation é menos eficaz.
3 Preparação de larval Injeção Aparelho
- Coloque numa fase mecânica XY em um microscópio de dissecação.
NOTA: estágios XY mecânico estão disponíveis a partir de grandes empresas de microscópio. Em alternativa, as fases de microscópio de baixo custo pode ser utilizado para a maioria dos microscópios de dissecação com algumas modificações (ver tabela de materiais, por exemplo). - Coloque um manipulador mecânico agulha perto do palco mecânica XY.
- Montar uma seringa de injeção. Use uma torneira de quatro vias (com ligações Luer) para conectar uma seringa descartável de 30 ml para um suporte de agulha de vidro, permitindo a manipulação da seringa de injecção, sem afectar a pressão no interior da agulha de injecção (ver Philip e Tomoyasu 2011 11 para a seringa de injecção detalhada montagem).
4. Puxando as agulhas de injecção
- Puxe agulhas de vidro de borosilicato (B100-50-15, OD: 1 milímetro, ID: 0.5 mm, 15 centímetros de comprimento) por umpuxador de agulha.
NOTA: Para Sutter P-87 ou P-97, use a configuração "Heat = 70, Pull = 45, Vel = 75, Time = 90" ou seguir o Capítulo 2 da pipeta Cookbook: células aderentes, C. elegans, Drosophila, e peixe-zebra - programas recomendados. As configurações ideais variam de acordo com o modelo do puxador da agulha. A configuração para um genérico Drosophila agulha de injeção é um bom ponto de partida. - Loja puxado agulhas em uma caixa de plástico (por exemplo, uma caixa de CD de plástico) e prenda com removível massa de montagem.
5 Isolamento e Seleção de Larvas
- Coloque uma peneira # 25 em um receptor peneira.
- Inserir o conteúdo do frasco de cultura (besouros da farinha e cultura) na peneira, e peneirar o conteúdo imediatamente. Não deixe o conteúdo da peneira sem peneirar, como larvas rapidamente tentar cavar através da peneira e ficar preso. Agressivamente filtrar o conteúdo para deixar cair farinhaatravés de e deixar larvas mais velhas (normalmente 6 ° e 7 ° larvas), pupas e adultos (juntamente com suas cutículas elenco-off, exúvias) por trás em cima da peneira.
- Transferir os materiais remanescentes no peneiro (escaravelhos e Exúvia) para a panela de sementes. Continue a tocar a panela para evitar que os besouros de escapar.
- Remover o Exúvia e as restantes partículas de farinha na superfície dos besouros por soprando suavemente sobre o tabuleiro de sementes.
- Toque a panela de sementes para mover o conteúdo para o fundo da panela. Então, deixe a panela inexplorado por alguns segundos. Aguarde até que os adultos, que são mais móveis do que outras etapas, a sair da pilha. Remover adultos utilizando um pincel de arte (por exemplo, largura de 1 cm).
- Pupas separado batendo suavemente o tabuleiro de sementes, mantendo a boca da panela um pouco para baixo, como pupas tendem a rolar mais rápido em direção à boca. Utilização de um pincel para remover as pupas, deixando apenas as larvas na panela de sementes.
- Plás larvas isolado em um prato limpo Petri. Selecione um grau adequado de larvas para injecção e os coloque em um prato separado Petri.
NOTA: larvas última precoce (1-2 dias após a muda larval final) são muitas vezes adequado ao analisar o efeito RNAi em morfologias adultos, bem como sobre a metamorfose. É, no entanto, por vezes, necessário para realizar ARNi no penúltimo (ou mesmo antes) fase para avaliar a função do gene precoce (por exemplo, as Figuras 3E-3H. Também ver Clark-Hachtel et al. 2,013 12). Use pupas como referência de tamanho para identificar o estádio de larvas. A última larvas são um pouco mais longo do que pupas. Alternativamente, pu-11 pode ser usada para identificar a última larvas, bem como para determinar o período de tempo do desenvolvimento de larvas no último instar (Figura S4 de Clark-Hachtel et al. 2,013 12). - Mantenha as larvas selecionado em uma placa de Petri até eterização. Coloque o restode larvas e outros estágios de besouros (como pupas e adultos) de volta em um frasco de cultura com farinha e devolvê-lo para a incubadora.
6 Preparação de agulhas de injeção
- Coloque uma agulha de vidro previamente puxou para uma lâmina de vidro usando pegajoso ou fita dupla face. Utilizando uma pinça, testar o tirou da agulha ao olhar através do microscópio de dissecação para ver onde as curvas de ponta. Agarre a área um pouco para o lado da ponta de onde as curvas da agulha com o fórceps e torcer para quebrar.
- Manter bons agulhas e descartar outros. Considere-se uma boa agulha como tendo uma abertura que não é nem muito grande nem muito pequena (cerca de 0,05 mm de diâmetro. Figuras 1A e 1B), e é inclinada para fazer a penetração da cutícula larval mais fácil (Figuras 1B e 2A-2B). Considere-se um mau agulha como (i) muito pequena, fazendo com que a absorção de uma solução de ARNcd na agulha dificulto (etapa 7) (Figuras 1D e 2D), (ii) muito grande, causando lesão de larvas e letalidade possível após a injecção (Figuras 1C e 2F), (iii) sem corte, tornando a penetração da cutícula lesão difícil e crescente.
- Insira a agulha no porta-agulhas.
- Primeiro, desaparafusar a ponta do suporte de agulha e colocar a extremidade posterior da agulha para a ponta da titular. Em seguida, colocar a junta de borracha sob a ponta de suporte (pelo menos 1 cm a partir da extremidade posterior da agulha de vidro).
- Inserir a parte de trás da agulha no suporte, com a junta de borracha totalmente inserida na abertura do suporte de agulha. Parafuso da ponta de volta para o titular.
- Colocar o suporte da agulha montada sobre a agulha manipulador. Manter o suporte de agulha perto da horizontal. Ajustar a posição do manipulador, de modo que a ponta da agulha está localizado no centro da vista quando se olha através do microscope.
7 Solução de pré-alimentação dsRNA na agulha
- Preparar a solução de injecção de ARNcd a apenas antes da injecção, através do ajuste da concentração com ddH2O e misturando a solução com volumes iguais de ARNcd de 2x tampão de injecção (passo 2.3). Manter a solução mista no gelo. Veja a discussão sobre a concentração de dsRNA.
- Cortar a ponta de uma pipeta de 20 uL ponta descartável. Pipetar 10 ml da solução para dentro da ponta da pipeta aparado. Remova cuidadosamente a ponta da pipeta da pipeta, mantendo o líquido na ponta, fornecendo contrapressão. Alternativamente, remover cuidadosamente a ponta e, em seguida, passar a solução para a extremidade da ponta, proporcionando pressão utilizando um dedo.
- Colocar a ponta da pipeta com a solução de ARNcd na platina do microscópio usando pegajoso com a abertura virada para a ponta da agulha.
- Olhando através do microscópio, mova lentamente a ponta carregado em direção à agulha até a ponta doagulha é apenas dentro da ponta da pipeta e carregada para dentro da solução.
- Ao olhar através do microscópio, puxe o êmbolo da seringa para puxar lentamente a solução dsRNA na agulha.
NOTA: Não sobrecarregue a agulha. À medida que a extremidade de a solução se aproxima da dsRNA ponta da agulha, diminuir a taxa de puxar e parar de carregar a solução antes de a ponta da agulha atinja ar. Se o ar atinge a ponta, a solução será rapidamente arrastada para o suporte de agulha, o que resulta em graves problemas de contaminação. Procedimentos de limpeza detalhadas de suporte da agulha são descritas em Philip e Tomoyasu 11, 2011. - Retirar a pressão de injecção da seringa e suporte de agulha pela abertura da torneira. Em seguida, move a ponta da pipeta para longe da ponta da agulha. Levante a agulha para cima do palco para evitar acidentalmente danificar a agulha ao colocar as larvas no palco. Remover a ponta da pipeta a partir da fase de esvaziado.
8 injeção dsRNA
- Coloque as larvas no cesto eterização. Use menos de 10 larvas se aprender a técnica. Use 30-40 larvas em uma rodada, uma vez confortável com a técnica.
- Coloque a cesta com larvas na garrafa éter e feche a tampa.
NOTA: Ether apresenta um risco de incêndio e também é prejudicial. Execute esta etapa sob uma coifa. - Larvas Etherize durante cerca de 3 min. Confira as larvas para o movimento depois de 3 min. Colocá-los de volta na garrafa éter por 30 seg, se eles ainda estão se movendo. Não Etherize por mais de 5 minutos, pois isso pode aumentar a letalidade.
NOTA: O tempo de eterização ideal pode variar dependendo da temperatura e umidade. - Transferir as larvas anestesiado do cesto para a borda antiaderente de uma lâmina de vidro pegajoso. Utilizando uma pinça e ao olhar através do microscópio, pegar cada um larvas por um e colocá-los para o slide. Lay-los lateralmente e bata suavemente para baixo seus corpos from cabeça e cauda, com a pinça, um pouco de alongamento-los como você vai, para se certificar de que eles são garantidos para o slide.
- Coloque o slide pegajoso com larvas no palco microscópio.
- Insira a agulha carregada dsRNA suavemente para o lado dorsal das larvas para evitar danos ao sistema nervoso central. Evite também a linha média dorsal, como o coração larval está localizado aqui. Nesta e nas subsequentes etapas, usar sempre a fase de mover-se as larvas para a agulha (em vez de utilizar o manipulador de agulhas para mover a agulha).
NOTA: O local de injecção específica não importa como RNAi em T. castaneum parece ser sistêmica. No entanto, é geralmente mais fácil de injectar no lado dorsal dos segmentos torácico ou abdominal. - Após a inserção da agulha no larvas, puxar a agulha ligeiramente para trás para permitir espaço para o dsRNA de entrar na cavidade do corpo de larvas.
- Empurre a seringa de injecção até que as larvas vez verde (ou a cor do tampão de injecção) e procurar stretched e completo.
NOTA: Se aprender a técnica, tentar injetar o máximo possível para determinar a quantidade de solução que pode ser injetado em uma larva, sem letalidade significativa. Ela é geralmente de cerca de 0,5-0,7 uL. - Retirar a agulha do larvas. Ao remover a agulha, ligeiramente puxe a seringa de injeção para evitar perda de dsRNA (também tomar cuidado para não puxar o ar).
- Injetar todas as larvas no slide. Certifique-se de remover as larvas que não foram injetadas com sucesso. Injeção completa antes larvas acordar (em cerca de 10 min).
- Remover o diapositivo com larvas injectadas no microscópio de injecção e deixar a lâmina durante mais 5 min, até que todas as larvas se recuperar da eterização.
- Com fórceps e sob um microscópio de dissecação, levantar delicadamente as larvas da cabeça à cauda para liberá-los a partir do slide pegajosa. Coloque as larvas recém-lançado em um prato limpo Petri. Deixar as larvas na placa de Petri durante 10-15 minutos para permitir a injecção wound a coagular.
- Coloque as larvas para um novo frasco com farinha e limpo etiqueta adequada incluindo o nome da estirpe, o tipo de dsRNA injectado, a concentração de dsRNA, o número de larvas injectadas, e a data.
- Cultura das larvas injectadas a 30 ° C com 70% de humidade. Observe as larvas para RNAi fenótipos regularmente.
NOTA: A última empupam larvas dentro de 7 dias. Em seguida, as pupas vai EFeche em adultos após 7 dias (se não houver nenhuma anormalidade no momento causado por RNAi).
9 Confirmação de Knockdown e fenotípicas análises
- Considerar a avaliação da eficiência de RNAi derrubar por várias técnicas de biologia molecular, tais como diferentes quantitativa de transcrição reversa-PCR (qRT-PCR) e Western Blotting. Veja Miller et al. 7, 2012 e Philip e Tomoyasu 2011 11 para qPCR detalhada e Western Blot protocolo, respectivamente.
- Analisar RNAi fenótipos relacionados.
NOTA: RNAi relacionadofenótipos podem ser observadas em vários estágios diferentes e sob diferentes condições de cultura, de acordo com os genes que foram alvo. RNAi pode afectar a morfologia do besouro, metamorfose, fisiologia e comportamento. Como muitas vezes a presença do fenótipo é verificada e em que condições os besouros são criados devem ser adaptados às questões específicas a ser investigado através de RNAi.
Resultados
Um dos passos fundamentais para a injeção de sucesso é fazer uma boa agulha. Tal como descrito no passo 6, a ponta da agulha tem de ser quebrado antes da injecção de T. castaneum. Os exemplos de bons e maus agulhas são mostrados na Figura 1. Um bom agulha tem uma ponta afiada e rígida, com uma abertura de diâmetro de aproximadamente 0,05 mm (Figura 1B).
A Figura 2 apresenta uma injecção de sucesso (Figuras 2A e 2B), assim como vários casos de injecções mal sucedidas (Figuras 2C-2F). Com uma agulha de injecção de tamanho adequado, a ponta da agulha penetra a cutícula de larvas, com resistência mínima (Figura 2A), e a solução de dsRNA (verde) flui para as larvas, sem qualquer vazamento (Figura 2B). Não sobre-injeção, pois fará com que excesso da solução de dsRNA, mesmo com uma agulha de injeção de tamanho adequado (Figura2C). Se a ponta da agulha é muito fina, a ponta de agulha muitas vezes não consegue penetrar na cutícula larval e curvas, tornando difícil a injecção (Figura 2D). Se isso acontecer, corte a ponta da agulha, por vezes, ajuda. Agulhas maiores são muitas vezes ainda utilizável, embora com algumas dificuldades em penetrar na cutícula larval (Figura 2E). No entanto, uma grande quantidade de solução de dsRNA é facilmente empurrada para fora da agulha, mesmo com uma ligeira pressão sobre a seringa de injecção, resultando frequentemente em um excesso da solução de ARNdc (Figura 2F).
Ao realizar ARNi, é importante ter um controlo positivo para determinar que a RNAi está a funcionar correctamente, e um controlo negativo para verificar que o efeito observado não é um efeito não específico de ARNcd ou uma consequência de processos de injecção (tais como lesões causadas por injecção, ou anormalidade de eterização). A injecção de ARNcd de EYFP direccionamento pode servir como umbom controle positivo quando se utiliza a 5,7 tensão pu-11. Quando EYFP dsRNA é injectado na última fase de larva, uma redução significativa do sinal de EYFP nas futuras primórdios asa podem ser observadas tão cedo quanto uma injecção pós dias (Figura 3A). O knockdown de EYFP está completa, dois dias após a injecção do ARNcd EYFP (Figura 3B) e esta knockdown persiste através do (Figuras 3C e 3D) fase de pupa e ao longo da vida do besouro, se a concentração de ARNcd é suficientemente elevada (por exemplo, 1 mg / mL) 7. EYFP ARNcd pode também ser utilizado como um controlo negativo, uma vez que é uma sequência do gene não-endógenos e não deve causar perturbações morfológicas e fisiológicas (Figuras 3A-3D).
Fornecemos imagens de larvas de ARNi para vestigial (vg), um gene de asa crítica 12, como um outro exemplo de como a efcazes esta técnica baseada em RNAi injecção é em T. castaneum. Quando dsRNA para vg é injectado em larvas penúltima etapa (uma fase, antes do último estádio de larva), uma perda completa das estruturas laterais podem ser observados na fase de pupa (Figuras 3E e 3F). O adulto, resultando também carece completamente estruturas asa (Figuras 3G e 3H). O resultado RNAi para vg exemplifica tanto a robustez ea natureza sistêmica da RNAi em T. castaneum.
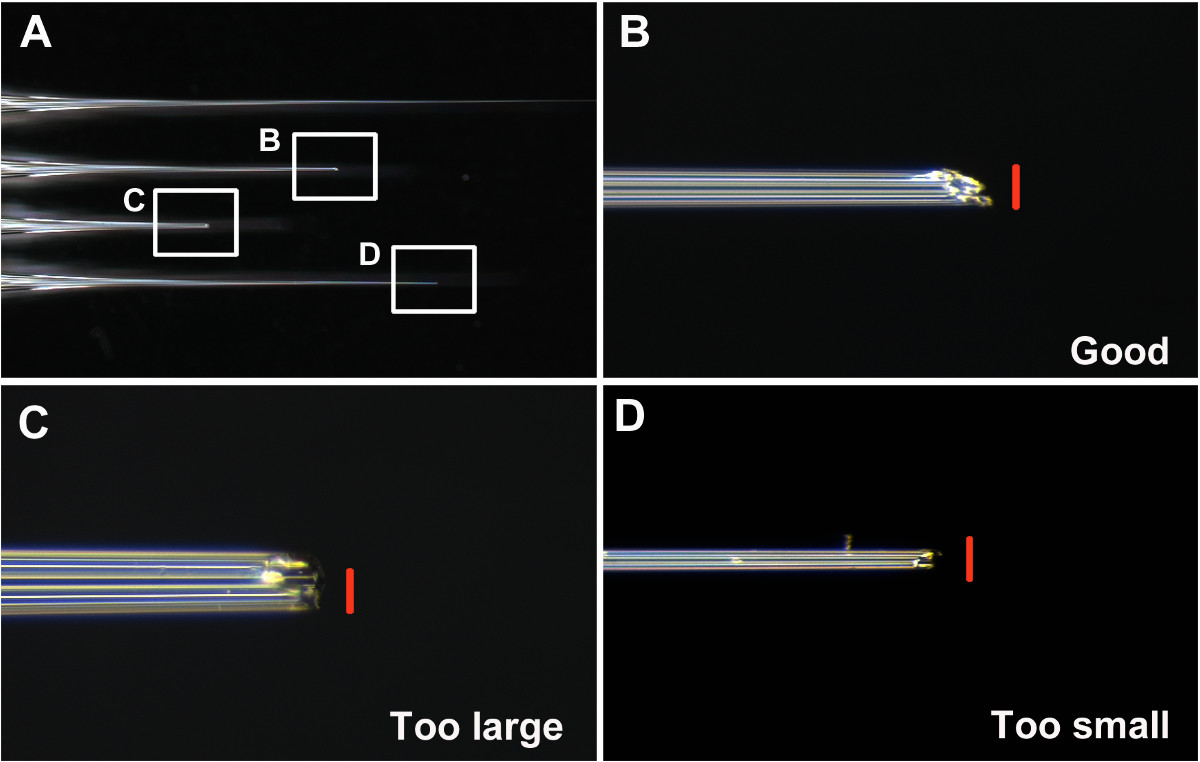
Figura 1: (A) Exemplos de agulhas não quebradas, boas e más (B) A ponta de uma agulha boa bem quebrada (C) A ponta de uma agulha quebrada que é demasiado grande e sem corte (D) A ponta... de uma falência agulha n que é muito fina. Barras de escala (vermelho) são de 0,05 mm. Clique aqui para ver uma versão maior desta figura.

Figura 2:... (A) A agulha de injeção de tamanho adequado inserido na larva de último estádio (B) Larva injetado de forma adequada com a solução de dsRNA verde (C) Estouro da solução de dsRNA no ponto de injeção causada por excesso de injeção (D ) Uma agulha fina não penetrar a cutícula larval. (E) uma grande agulha de injecção inserida na larva de último estádio. (F) Larva injectado com uma agulha, o que resulta em excesso e vazamento da solução de dsRNA. As setas indicam as agulhas."target =" ove.com/files/ftp_upload/52059/52059fig2highres.jpg _blank "> Clique aqui para ver uma versão maior desta figura.
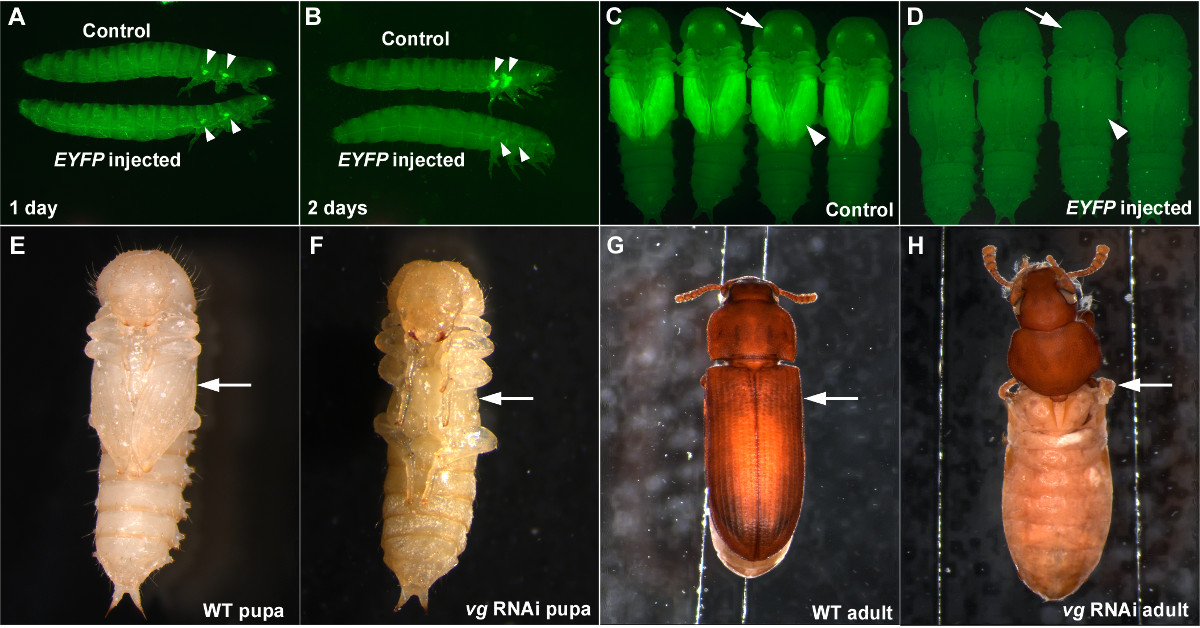
Figura 3: exemplos de sucesso de RNAi em T. castaneum. (A) Larvas um dia após a injecção. (B) Larvas dois dias após a injecção. (C) pupas de controlo (D). pupas resultante de (dC) Última injecção larvas dos resultados EYFP dsRNA numa redução da expressão EYFP. injeção larval EYFP dsRNA. Os controlos negativos são a injecção de tampão (A, B) e injecção dsRed dsRNA (C, D). Pontas de flechas e setas indicam expressão EYFP ou falta dela nos primórdios da asa e olhos, respectivamente. (EH) Penfinal larval RNAi para vg. (E) do tipo selvagem pupa. (F) vg RNAi pupa. A falta de estruturas laterais é já visível na fase de pupa (seta). G) do tipo selvagem adulto. (H) vg RNAi adulto. Estruturas Asa relacionadas são completamente ausente (seta). Clique aqui para ver uma versão maior desta figura.
Discussão
Há uma série de questões importantes que precisam ser considerados para garantir o sucesso de RNAi, incluindo o comprimento ea concentração das moléculas de dsRNA, a concorrência entre as diferentes moléculas de dsRNA (ao tentar derrubar múltipla), ea possibilidade de fora do alvo Effects (OTE).
dsRNA Comprimento
O comprimento das moléculas de dsRNA afecta a eficiência da resposta RNAi sistémica, com um ARNcd deixando de ser mais eficiente a desencadear RNAi 7,14,15 (embora o tempo limite de dsRNA é actualmente desconhecido). O comprimento do dsRNA deve ser mais longo do que 50 bp para induzir RNAi eficaz em T. castaneum 7. dsRNA entre 150 pb e 500 pb parece ser ideal para experimentos de RNAi. Embora as moléculas mais longas de dsRNA também pode ser usado, eles terão uma chance maior de OTE eo passo a clonagem de genes vai se tornar cada vez mais difícil.
dsRNA Concentração
ve_content "> diferentes graus de knockdown do gene pode ser conseguida, dependendo da concentração de ARNcd. de 1 ug / ul parece ser uma concentração de partida razoável, o que muitas vezes produz um fenótipo quase nula (pode variar dependendo do gene (s) de interesse ). RNAi pode ser realizada com uma concentração mais elevada (por exemplo, 7-8 mg / mL) para obter um forte fenótipo RNAi. ARNi com uma diluição em série de dsRNA por vezes pode ser vantajoso para a produção de uma série de fenótipos hipomórfico (Dados Complementares de Tomoyasu et al. 8, 2009, e Borràs-Castells dados não publicados).RNAi Concorrência
Knockdown múltipla do gene pode ser realizado em T. castaneum injetando várias moléculas de dsRNA diferentes simultaneamente. No entanto, sabe-se também que ter várias moléculas diferentes de dsRNA presentes dentro do organismo, muitas vezes resulta em competição entre os ARNcd de acesso aos componentes de ARNi 7,14. É importante utilizar o mesmo comprimento e a mesma concentração de todos os dsRNA ao tentar knockdown gene múltiplo para evitar um dsRNA competindo com as outras (embora, mais ajustamentos do comprimento e concentração de ARNcd pode ser necessário, quando os níveis de expressão são muito diferentes entre os genes-alvo). Nós, assim como outros, têm realizado com sucesso knockdown duplo e triplo (por exemplo, Tomoyasu et al., 2005 16, Tomoyasu et al. 17, 2009, e Yang et al., 2009 18). Embora exequível, quádruplo RNAi (ou mais) pode ser difícil, uma vez que provavelmente iria causar uma redução significativa da eficiência de ARNi para todos os quatro genes alvo.
Off-alvo
OTE é uma preocupação inerente para abordagens baseadas em RNAi. Uma maneira de minimizar OTA é identificar regiões no gene-alvo que partilham sequências similares com outros genes e evitar estas regiões ao conceber dsRNA. Uma análise simples BLAST contra o T. castaneum conjunto de genes previu pode identificar tais regiões. Várias ferramentas on-line também permite a avaliação de potencial OTE (por exemplo, E-RNAi 19). Realizando ARNi para duas regiões não se sobreponham de o gene alvo é um meio fácil e eficaz para eliminar a possibilidade de que os fenótipos observados são causadas por OTA. A possibilidade de OTE é minimizado se RNAi para duas regiões não-sobrepostas produzir os mesmos fenótipos (a não ser que as duas regiões não sobrepostas compartilham uma seqüência similar).
Avaliando gene knockdown por outras que as fenotípicas analisa meios é muitas vezes essencial para dados relacionados com RNAi efetivamente presentes. Dois grandes maneiras de avaliar knockdown gene são qRT-PCR e western blot. qRT-PCR é uma maneira conveniente de medir o nível do ARNm alvo, e tem sido utilizado em muitos estudos relacionados com o ARNi incluindo aqueles em T. castaneum (ver Miller et al. 2,012 7 por exemplo). However, o cuidado deve ser tomado, como temos visto recentemente alguns casos em que o nível de mRNA alvo é sobre-regulada por RNAi (embora o produto da proteína é regulada para baixo) (dados não publicados Borràs-Castells). Não se sabe se este mRNA RNAi induziu aumento da regulação pode ser generalizado ou única para determinados genes. A análise de transferência de Western é uma outra forma de confirmar knockdown gene. Este método é muito fiável, uma vez que mede a quantidade de proteína do produto final. A exigência de um anticorpo específico contra o produto de proteína do gene alvo é uma desvantagem para esta abordagem. Utilizando múltiplas medições independentes, além de análise fenotípica vai aumentar a confiança dos dados fenotípicos obtidos pela análise baseada em RNAi.
Desde a sua concepção em T. castaneum, principalmente RNAi foi utilizado para estudar a função de genes em desenvolvimento e formação de um padrão. Estes T. estudos de desenvolvimento castaneum foram altamente bem sucedido em caracteterizing funções evolutivamente conservadas e divergiram de genes (revistos em Denell 2008 1 e Klingler 2004 2). No entanto, estudos baseados em RNAi em T. castaneum não estão limitados a biologia do desenvolvimento. Por exemplo, o RNAi pode ser utilizado para estudar a função do gene em uma ampla gama de respostas fisiológicas e comportamentais, incluindo a tolerância ao estresse, a predação, agressão, escolha de parceiro, os padrões de atividade, e os mecanismos de defesa.
Uma dificuldade de aplicação de ARNi para estes contextos, é a probabilidade de efeitos pleiotrópicos. Frequentemente, os genes de interesse vai ter uma variedade de funções em todo o T. ciclo de vida castaneum, tornando assim a remoção de genes, sem efeitos fenotípicos indesejadas difíceis. No entanto, a capacidade de executar facilmente RNAi em uma variedade de estágios muitas vezes pode ser uma estratégia eficaz para evitar esses efeitos pleiotrópicos. Por exemplo, a realização de RNAi em adultos, em vez de larvas ou pupas pode nos permitir contornar UNINtendiam a letalidade causada por knockdown gene durante o desenvolvimento inicial. A flexibilidade da resposta de RNAi em T. castaneum torna, assim, este modelo uma opção atraente para adaptação RNAi para experimentos de função do gene em respostas fisiológicas e comportamentais.
O T. sistema castaneum também é ideal para uso em um laboratório de ensino. T. castaneum podem ser facilmente cultivadas em uma farinha / mistura de levedura, à temperatura ambiente (25 ° C), sem subcultura frequente, e técnicas de RNAi em T. castaneum são simples o suficiente para ser adaptado para um laboratório com jovens cientistas, de aprendizagem. Como RNAi está se tornando uma técnica essencial em uma variedade de áreas biológicas, é fundamental que os alunos são expostos a esta técnica. A natureza direta da técnica de RNAi larval em T. castaneum também incentiva mais os alunos a serem envolvidos na pesquisa, tornando T. castaneum um excelente candidato para uma sala de aula orientada sistema genético.
Divulgações
Os autores não têm nada a revelar.
Agradecimentos
Agradecemos ao Centro de Bioinformática e Genômica Funcional (CBFG) na Universidade de Miami para obter suporte técnico. Este trabalho foi financiado pela Miami University concessão start-up (YT), e da National Science Foundation (YT: IOS 0.950.964).
Materiais
| Name | Company | Catalog Number | Comments |
| Organic whole wheat flour | Heartland Mill Inc. Kansas | G1 | |
| Brewer’s yeast | MP Biomedicals | 2903312 | Sift yeast with #35 stainless sieve before use. Wear protective mask and cover the sieve with plastic wrap, as the sifted yeast is a fine particle and respiratory hazardus. |
| 6 oz plastic Drosophila stock bottles | Fisher Scientific | 11-888 | |
| Sieve | Fisher Scientific | 04-881N | 8 in. dia. x 2 in.D. Use #25 (Nominal opening 710 µm) for larvae, pupae, and adults. |
| Sieve | Fisher Scientific | 04 881 10P | 8 in. diameter #35 (Nominal seive opening: 600 µm) Stainless Steel Sieves, 8 in. dia. x 2 in.D. This sieve is ideal to remove clumps from yeast powder. |
| Sieve receiver | Fisher Scientific | 04-866B | 8 in. diameter |
| 1.5 Quart spouted sample pan | Seedburo Equipment Co. | Model 33 | collection pan |
| Incubator | BioCold Environmental Inc | BC26-IN | Keep it 30 °C with 70% humidity. |
| Na2HPO4 | Fisher Scientific | S374500 | |
| NaH2PO4 | Fisher Scientific | S397-500 | |
| KCl | Fisher Scientific | P217-500 | |
| Food dye | Kroger | green, blue, or red preferable | |
| Microscope glass slide | Fisher Scientific | 22-038-103 | |
| Tack-It Over&Over | Aleene's | Repositionable glue for sticky slides. Double sided tape can be used as an alternative, however, we found that the adhesiveness varies. | |
| Plastic CD case | Amazon | ||
| Boroslilicate glass capillary | Sutter Instrument | BF100-50-15 | O.D. 1 mm, I.D. 0.5 mm, 15 cm. Without filament |
| Needle puller | Sutter Instrument | P-87 or P-97 | |
| Removable mounting putty | Loctite Fun-Tak | ||
| Compressed gas duster | OfficeMax | OM96091 | |
| Forceps | Fine Science Tool | 11231-30 | Dumoxel #3 (to manipulate beetles) |
| Forceps | Fine Science Tool | 11252-20 | INOX #5 (to break needle tips) |
| Ethyl ether, anhydrous | Fisher Scientific | E138-500 | |
| Nylon mesh | Flystuff/Genessee Scientific | 57-102 | 120 µm pore size/49% open area |
| Media bottle, 100 ml | VWR | 89000-926 | |
| Stereomicroscope | Zeiss | SteREO Discovery V12. Injection microscope. | |
| Stereomicroscope | Fisher/Zeiss | 12-070-513 | Stemi2000. Use to break the needle and place larvae onto the sticky slide. |
| X-Y mechanical stage | Zeiss | 4354600000000000 | |
| X-Y mechanical stage | Microscopenet.com | A512 | Inexpensive alternative |
| Manipulator | Narishige | M-152 | |
| Magnetic stand | Narishige | GJ-1 | |
| Glass capillary holder | Narishige | IM-H1 | |
| 30 ml Disposable syringe | BD syringe | 309650 | BD Luer-Lok Tip |
| Four-way stopcock | Cole-Parmer Instrument Co. | EW-30600-03 | Stopcocks with Luer connections; 4-way; male slip |
| Art paint brush | Amazon | Art Advantage Oil and Acrylic Brush Set, 24-Piece | Any general paint brush will work. |
Referências
- Denell, R. Establishment of Tribolium as a genetic model system and its early contributions to evo-devo. Genetics. 180, 1779-1786 (2008).
- Klingler, M. Tribolium. Curr Biol. 14, R639-R640 (2004).
- Richards, S., et al. The genome of the model beetle and pest Tribolium castaneum. Nature. 452, 949-955 (2008).
- Bucher, G., Scholten, J., Klingler, M. Parental RNAi in Tribolium (Coleoptera). Curr Biol. 12, 85-86 (2002).
- Tomoyasu, Y., Denell, R. E. Larval RNAi in Tribolium (Coleoptera) for analyzing adult development. Dev Genes Evol. 214, 575-578 (2004).
- Tomoyasu, Y., et al. Exploring systemic RNA interference in insects a genome-wide survey for RNAi genes in Tribolium. Genome biology. 9, R10 (2008).
- Miller, S. C., Miyata, K., Brown, S. J., Tomoyasu, Y. Dissecting systemic RNA interference in the red flour beetle Tribolium castaneum parameters affecting the efficiency of RNAi. PLoS ONE. 7, e47431 (2012).
- Tomoyasu, Y., Arakane, Y., Kramer, K. J., Denell, R. E. Repeated co-options of exoskeleton formation during wing-to-elytron evolution in beetles. Curr Biol. 19, 2057-2065 (2009).
- Miller, S. C., Brown, S. J., Tomoyasu, Y. Larval RNAi in Drosophila. Dev Genes Evol. 218, 505-510 (2008).
- Brown, S. J., Mahaffey, J. P., Lorenzen, M. D., Denell, R. E., Mahaffey, J. W. Using RNAi to investigate orthologous homeotic gene function during development of distantly related insects. Evolutio., & development. 1, 11-15 (1999).
- Philip, B. N., Tomoyasu, Y. Gene knockdown analysis by double-stranded RNA injection. Methods Mol Biol. 772, 471-497 (2011).
- Clark-Hachtel, C. M., Linz, D. M., Tomoyasu, Y. Insights into insect wing origin provided by functional analysis of vestigial in the red flour beetle. Tribolium castaneum, Proceedings of the National Academy of Sciences of the United States of America. 110, 16951-16956 (2013).
- Kitzmann, P., Schwirz, J., Schmitt-Engel, C., Bucher, G. RNAi phenotypes are influenced by the genetic background of the injected strain. BMC genomics. 14, 5 (2013).
- Parrish, S., Fleenor, J., Xu, S., Mello, C., Fire, A. Functional anatomy of a dsRNA trigger differential requirement for the two trigger strands in RNA interference. Molecular cell. 6, 1077-1087 (2000).
- Winston, W. M., Molodowitch, C., Hunter, C. P. . Systemic RNAi in C elegans requires the putative transmembrane protein SID-1. 295, 2456-2459 (2002).
- Tomoyasu, Y., Wheeler, S. R., Denell, R. E. Ultrabithorax is required for membranous wing identity in the beetle Tribolium castaneum. Nature. 433, 643-647 (2005).
- Trauner, J., et al. Large-scale insertional mutagenesis of a coleopteran stored grain pest, the red flour beetle Tribolium castaneum, identifies embryonic lethal mutations and enhancer traps. BMC Biol. 7, 73 (2009).
- Yang, X., et al. Probing the Drosophila retinal determination gene network in Tribolium (II) The Pax6 genes eyeless and twin of eyeless. Developmental biology. 333, 215-227 (2009).
- Horn, T., Boutros, M. E-RNAi a web application for the multi-species design of RNAi reagents--2010 update. Nucleic acids research. 38, 332-339 (2010).
Erratum
Formal Correction: Erratum: Larval RNA Interference in the Red Flour Beetle, Tribolium castaneum
Posted by JoVE Editors on 10/01/2015. Citeable Link.
An erratum was issued for Larval RNA Interference in the Red Flour Beetle, Tribolium castaneum. There was a typo in the settings for Sutter P-87 in step 4.1.
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados