Method Article
Métodos para aumentar a sensibilidade de alta resolução de derretimento Single Nucleotide Polymorphism Genotipagem em Malária
Neste Artigo
Resumo
Enquanto a análise de alta resolução de fusão oferece a capacidade de diferenciar entre polimorfismos de nucleotídeo único em uma população heterogênea, viés amplificação alelo mutante pode aumentar a sua capacidade para detectar alelos presentes em percentagens relativamente baixas dentro de uma amostra. Este protocolo descreve melhorias que melhoram a sensibilidade da análise de fusão de alta resolução.
Resumo
Apesar de décadas de esforços de erradicação, a malária continua a ser um fardo global. Renovou o interesse recente na eliminação regional e erradicação global tem sido acompanhado pelo aumento da informação genômica sobre Plasmodium espécies de parasitas responsáveis pela malária, incluindo características das populações geográficas, bem como variações associadas com sensibilidade reduzida às drogas anti-malária.
Uma variação genética comum, polimorfismos de nucleotídeo único (SNPs), oferece alvos atraentes para parasita genotipagem. Estes marcadores são úteis não só para o rastreamento de marcadores de resistência a drogas, mas também para rastrear populações de parasitas por meio de marcadores que não estão sob drogas ou outras pressões seletivas.
Métodos de genotipagem SNP oferecer a capacidade de controlar a resistência aos medicamentos, bem como a impressão digital parasitas individuais para a vigilância da população, particularmente em resposta aos esforços de controlo da malária em regiões que se aproximam eliminatestado de iões.
Enquanto SNPs informativos foram identificados que são agnósticos às tecnologias de genotipagem específicas, de alta resolução derretendo análise (HRM) é particularmente adequado estudos com base em campo para. Comparado com métodos padrão fluorescente-sonda baseada que requerem SNPs individuais numa única sonda marcada e oferecem sensibilidade na melhor das hipóteses 10% para detectar SNPs em amostras que contêm múltiplos genomas (polygenomic), GRH oferece uma sensibilidade 2-5%. Modificações GRH, tais como sondas bloqueados e concentração de iniciador, assimétricos, bem como a optimização de temperaturas de recozimento para a amplificação por PCR no sentido de polarização de amplificação do alelo menor, pode ainda aumentar a sensibilidade da ARH. Embora a melhoria da sensibilidade depende do ensaio específico, que têm aumentado as sensibilidades de detecção para menos de 1% do alelo menor.
Em regiões que se aproximam de erradicação da malária, a detecção precoce de emergentes ou resistência aos medicamentos importados é essencial para a prompT resposta. Da mesma forma, a capacidade de detectar infecções polygenomic e diferenciar os tipos de parasitas importados a partir de reservatórios locais enigmáticas pode informar programas de controle.
Este manuscrito descreve modificações a tecnologia de fusão de alta resolução, que aumentam ainda mais a sua sensibilidade para identificar as infecções polygenomic em amostras de doentes.
Introdução
Apesar do renovado interesse no controle da malária e da erradicação, a malária continua a ser um fardo em todo o mundo, com quase metade da população do mundo em risco de infecção e mais de 550.000 mortes por ano, especialmente crianças na África sub-saariana 1.
Estes novos programas de controlo e erradicação têm sido apoiados pelo renascimento genômica, com grande número de parasitas da malária sequenciados e analisados para mutações associadas a sensibilidade reduzida de drogas, aumento da virulência, e para as características da população 2,3. Polimorfismos de nucleotídeo único (SNPs) estão entre as variantes genéticas mais comumente identificados 4-7.
Métodos de genotipagem SNP portáteis oferecem no local e vigilância da população em tempo real e monitoramento 8. Além fingerprinting parasitas individuais, o "código de barras molecular" também é usado para detectar deslocamentos temporais dramáticas na frequência do alelobem como variância tamanho e complexidade de infecção 9 população efetiva.
Embora este conjunto de informativo SNPs é facilmente adaptado para muitas plataformas de genotipagem, de alta resolução fusão (HRM) análise é particularmente adequado para estudos, onde a operação e detecção de mutações novas a baixo custo sensível e simples em comparação com a sequência eo outro campo baseada em abordagens são atraentes em ambientes pobres em recursos.
HRM começa com reacção em cadeia de polimerase padrão (PCR) que incorpora um corante fluorescente. Pós-PCR análise de fusão determina a temperatura de pico amplicon de fusão; uma única diferença de SNP num curto amplicão pode resultar em substancial pico de temperatura de fusão (Tm) diferenças.
Vários refinamentos a este método oferece melhor resolução genotipagem para diferenciar SNPs incluindo SNPs classe IV (AT), e detectar menores alelos mutantes em amostras com vários alelos presentes (poliGinfecções enomic). Em primeiro lugar, os ensaios de incorporar sondas curtas centradas sobre a região do SNP, além de transmitir e iniciadores que estão presentes em diferentes concentrações reversa. Estas sondas não bloqueados são amplificados durante a PCR, mas ligam-se ao fio produzido em excesso devido a concentrações de iniciadores assimétricos que aumentam a produção do produto sonda-molde. Estes produtos de amplificação de cadeia dupla separados constituídos por sonda ligada ao excesso de fita molde são ~ 20-30 pb; seu comprimento diminuiu significativamente em comparação com todo o amplicon (80-150 bp) levam a diferenças muito maiores Tm associados com a base única ou múltipla sonda-modelo não corresponde 10.
Em segundo lugar, o alelo mutante viés amplificação (Maab) reduz a temperatura de recozimento da reacção para a reacção de polarização no sentido de alelos mutantes presentes em baixas proporções em infecções polygenomic. A temperatura de recozimento é definida entre as Tms de perfeitamente compatíveis (tipo selvagem) e descasados sonda (mutante)s. A esta temperatura, a ligação do alelo de tipo selvagem é bastante estável que é impedida a amplificação em comparação com o seu equivalente incompatibilidade, por conseguinte, induzam a amplificação para o alelo mutante, quando ambos estão presentes numa única amostra de 10.
Com esses refinamentos GRH, esta tecnologia tem permitido o rastreamento de origens e identidade de parasitas associados com infecções epidêmicas na América do Sul 11 e detecção de novas mutações, a diferenciação de vários SNPs localizados imediatamente ao lado do outro em seqüência, e mutações presentes em menos de 1 % dos alelos em amostras polygenomic 10.
Aumento da sensibilidade é particularmente importante nas regiões que se aproximam status de pré-eliminação e aqueles em risco para o surgimento de resistência aos medicamentos. A capacidade de identificar rapidamente e facilmente parasitas importados e SNPs associados à redução da sensibilidade no local informa os esforços de vigilância e programas de controle sobrea eficácia das suas implementações e identifica hotspots para o aumento dos esforços de erradicação da malária e de eliminação. Este protocolo descreve os métodos para a sonda-bloqueado à base e para um aumento da sensibilidade Maab GRH genotipagem.
Protocolo
Nota: Este protocolo inclui os volumes e as concentrações para placa de 96 poços, as tiras de 8 tubos e sistemas à base de capilares (volume de reacção de 10 ul); No entanto, RH pode também ser realizada em sistemas à base de placa de 384 poços com um volume de reacção de 5 ul por escamação todos os volumes correspondentemente. A gama de detecção para a maioria dos sistemas é de 10 pg a 10 ng de ADN molde.
1. Prepare Templates
- Plasmodium falciparum obter amostras directamente a partir de sangue obtido de doentes que participaram nos estudos local de campo e armazenado como sangue completo, os glóbulos vermelhos sedimentados, ou em papel de filtro.
Nota: As amostras também podem ser a crescer in vitro adaptados-cultura; algumas cepas padrão de laboratório para análise pode ser encomendado a partir do Reagente Malaria Research and Reference Resource Center (MR4, amostras podem ser usado diretamente em glóbulos vermelhos peletizadas ou cultura, ou extraído de sangue total, glóbulos vermelhos, ou filtro de papel. - O DNA extraído a partir depapel de filtro ou cepas adaptadas à cultura
Nota: HRM é sensível à concentração de sal; diferenças de concentração devido a métodos de extração de variáveis podem afetar significativamente a temperaturas de fusão. Enquanto buffers finais específicos não são um fator significativo para o sucesso, utilizar uma eluição ou ressuspensão tampão norma comum (ou seja, água, 1x Te ['baixo Te' ou 'tampão suspensão DNA': 10 mM Tris-Cl, EDTA 0,10 mM] ou 1x TE [10 mM Tris-HCl, EDTA 1 mM]) para todas as amostras para evitar curvas de fusão anómalos ou inconsistentes.- Preparar diluições modelo de 10 pg -10 ng por mL em padrão Te, TE, ou água para cada reacção de 10 ul.
- A amplificação directa a partir de células vermelhas do sangue peletizadas
- Determinar a parasitemia de glóbulos vermelhos usando microscopia-esfregaço fina
Nota: Os métodos para determinar a parasitemia de glóbulos vermelhos infectados estão disponíveis a partir do Centro de Controle e Prevenção de Doenças (CDC): http: //www.cdc.gov/dpdx/diagnosticProcedures/blood/microexam.html - Diluir parasitemias acima de 0,5% 1: 400 em PCR-grade água, TE, ou Te. Use 3 ul para cada reação 5- 10 ul.
- Determinar a parasitemia de glóbulos vermelhos usando microscopia-esfregaço fina
- Controles
Nota: GRH pode ser utilizado para verificação do gene para detectar variantes de sequência de uma população 12; no entanto, a genotipagem preciso exige normas seqüência de verificado para conter o SNP sendo interrogado. Construções de plasmídeo ou malária isolados com genótipos conhecidos podem ser utilizados. Para a maioria das aplicações, 3D7 (MR4 ARM-151) é utilizado como um controlo de tipo selvagem. Devido à variabilidade entre-run, sempre incluir controlos positivos e negativos.- Os controlos positivos: Prepare 10 pg - 10 ng por 1 diluições ul de plasmídeo ou normas com genótipos SNP verificou-seqüência.
- Controle negativo: Use PCR água grau como um controle sem modelo (NTC) para as reações de amplificação.
- Prepare a mistura reaccional
- Uma camada de óleo (Opcional) Mineral. Nota: Alguns sistemas de gestão de recursos humanos são plataformas de fusão autônomo e não tem uma tampa aquecida para impedir a condensação e evaporação do produto durante a amplificação.
- Adicionar óleo mineral leve 20 ul a cada poço de placa antes de carregar a mistura de reacção.
- Prepare 10x stocks de trabalho de todos os ensaios
- Consulte a Tabela 1 para concentrações recomendadas 10x banco de trabalho, bem como as concentrações finais para genotipagem HRM-based-sonda bloqueada.
- Prepare misturas reaccionais
- A cada poço na placa ou tubo, adicionar 1 ul de estoque de trabalho de 10x a frente e iniciadores e sondas, 4,0 inverter - 2,5x 2,0x GRH ou 5.0 ul de mistura mestre, 1 ul de molde, e água de grau de PCR para um volume total de reacção de 10 ul .
- Cubra as placas ou tubos
- Use óptico placa seals para a amplificação baseada na placa, bonés ópticos para sistemas baseados em fita de tubo e tampas de plástico para sistemas baseados em capilares. Seja cobertura certeza é completo e placas e tampas são completamente selado e protegido para evitar a evaporação durante a amplificação.
- Amostras de spin
- Placas de Spin 3 min a 1800 xg para remover as bolhas de ar e para separar as fases aquosas de óleo e se o óleo mineral é utilizado.
- Uma camada de óleo (Opcional) Mineral. Nota: Alguns sistemas de gestão de recursos humanos são plataformas de fusão autônomo e não tem uma tampa aquecida para impedir a condensação e evaporação do produto durante a amplificação.
- Protocolo de amplificação
- Colocar as placas ou tubos de reacção em termociclador. Executar protocolos padrão-sonda bloqueada ou Maab de amplificação (Tabelas 2 e 3, respectivamente). Note-se a diferença de temperatura de recozimento entre estes métodos.
Análise 3. HRM
- Fundição
- Após amplificação por PCR, proceder à análise derretendo. Para os sistemas de fusão autônomos que não têm built-in amplificação (ex., Sistemas de BIOFIRE Defesa LightScanner), remover placa amplificado a partir termociclador e colocar em instrumento de gestão de recursos humanos. A partir do interface de software do sistema, derreter a placa da 40 - 80 ° C para produzir ambas as sondas e das amplicon picos de fusão.
Nota: Para salvar fusão e análise de tempo, a janela de fusão pode ser encurtado para cobrir faixas de temperatura para apenas sondar ou regiões amplicon de fusão. Após a fusão, as placas e os tubos podem ser re-fundida, se necessário, e armazenados a -20 ° C ou 4 ° C. Tubos de vidro loja só a 4 ° C para evitar tubos de rachar.
- Após amplificação por PCR, proceder à análise derretendo. Para os sistemas de fusão autônomos que não têm built-in amplificação (ex., Sistemas de BIOFIRE Defesa LightScanner), remover placa amplificado a partir termociclador e colocar em instrumento de gestão de recursos humanos. A partir do interface de software do sistema, derreter a placa da 40 - 80 ° C para produzir ambas as sondas e das amplicon picos de fusão.
- Análise Melt
- Retirar amostras negativas
- Dentro do software de análise, retire do mais amostras de análise que não amplificam ou que têm picos de fusão irregulares. Nota: Depois de selecionar o modo de análise para um arquivo run indivíduo, o instrumento agrupa automaticamente as amostras positivas e negativas. Antes de genotipagem de cada subconjunto, o usuário pode alterar as chamadas manualmente.
- Partindo do derretimentocurva ou pico de fusão de exibição, clique com o botão direito sobre as curvas de ser retirados para selecioná-los. Depois de selecionar as amostras classificados incorretamente, vá para "Nova chamada", selecione o grupo apropriado e clique em "Aplicar Mudança".
- Dentro do software de análise, retire do mais amostras de análise que não amplificam ou que têm picos de fusão irregulares. Nota: Depois de selecionar o modo de análise para um arquivo run indivíduo, o instrumento agrupa automaticamente as amostras positivas e negativas. Antes de genotipagem de cada subconjunto, o usuário pode alterar as chamadas manualmente.
- Normalizar
- A partir da derivada negativa de fluorescência normalizada em relação à temperatura (-dF / dT) ("Curves diferença") vista, mover barras de normalização para cercar a sonda derreta região.
Nota: A sonda derreter região é a temperatura mais baixa das duas regiões de fusão. As barras de normalização deve ser na base dos picos de fusão.
- A partir da derivada negativa de fluorescência normalizada em relação à temperatura (-dF / dT) ("Curves diferença") vista, mover barras de normalização para cercar a sonda derreta região.
- Calcule grupos
- Após a normalização, selecione "grupos Calcular 'para atribuir automaticamente amostras de grupos.
Se necessário, as amostras-re atribuir manualmente para grupos específicos.
Nota: Alguns softwares de análise tem limiares de alta sensibilidade que não podem ser alterados; Nestes casos, o Software pode atribuir amostras a diferentes grupos que visualmente pertencem a outro.
- Após a normalização, selecione "grupos Calcular 'para atribuir automaticamente amostras de grupos.
- Atribuir genótipos
- Atribuir genótipos para cada grupo com base em padrões (controles positivos).
- Depois que o usuário confirma que os grupos computados estão corretas (se não, que as mudanças corretas foram feitas sob a guia Agrupamento selecionando amostras mal classificados e selecionando o grupo desde o drop-box ao lado de "New Call"), selecione "Editar nomes de grupo "sob a guia" Agrupamento ".
- Digite os genótipos dos padrões na caixa de cor correspondente (por exemplo, se 3D7 padrão tem um conhecido Um genótipo e está no grupo vermelho, o grupo vermelho tem o genótipo A). Pressione "OK" e salvar os resultados.
- Atribuir genótipos para cada grupo com base em padrões (controles positivos).
- Resultados de exportação
- Genótipo exportação chama como uma planilha para análise posterior população da amostra.
- Retirar amostras negativas
Resultados
Os dados podem ser visualizados em várias formas diferentes, dependendo do software e instrumento de análise. Tipicamente, um gráfico da derivada negativa da fluorescência normalizada em relação à temperatura (-dF / dT) contra os resultados de temperatura é mais simples para a visualização de picos de fusão e determinação de genótipos.
Uma janela de fusão completa (40 ° C-80 ° C) irá resultar em ambas as regiões amplicon e de fusão da sonda. A Figura 1 mostra um exemplo de picos de fusão normalizados para ambas as regiões. Definir a janela de análise para apenas os resultados da região sonda em clara diferenciação dos picos correspondentes a SNPs específicas (Figura 2).
Análise baseada em Sonda mostra claramente a presença de ambos os alelos como picos individuais a temperaturas de fusão que correspondem a picos homozigóticos SNP (figura 3).
A Figura 4 mostra que progressireduzindo vamente a temperatura de recozimento durante a amplificação resultados (Maab) num viés para o alelo mutante (pico do lado esquerdo) (Figura 4A), o que resulta em um aumento da sensibilidade para o alelo mutante de uma população de alelos mistos (Figura 4B).
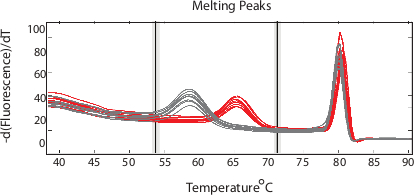
Figura 1. Sonda amplicão de fusão e as regiões. Locando a derivada negativa da fluorescência normalizada em relação à temperatura sobre uma ampla janela de resultados de fusão em ambos sonda (temperatura mais baixa) e os picos de fusão amplicão que podem ser analisados. (Adaptado de Daniels et al DOI:. 10,1128 / AAC.05737-11) Clique aqui para ver uma versão maior desta figura.
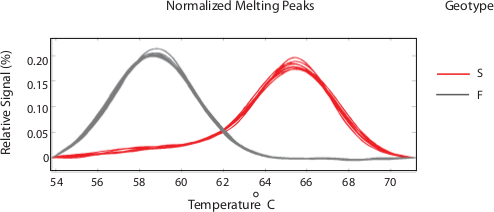
Figura 2. Sonda picos de fusão. Partidas Perfeito (geralmente do tipo selvagem alelos) resultar em picos de fusão mais elevados (vermelho), enquanto descasamentos SNP têm temperaturas de fusão mais baixas (cinza). Por favor clique aqui para ver uma versão maior desta figura.
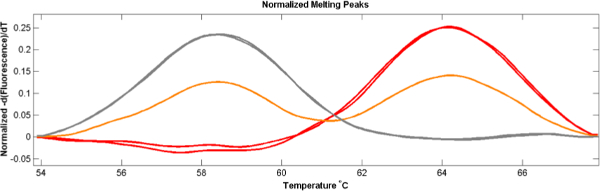
Figura 3. Polygenomic picos de fusão. Quando ambos os alelos estão presentes numa amostra, com base em análise de GRH sonda representa ambos os alelos como curvas de dois picos (laranja) com picos que correspondem a amostras de um único alelo (vermelhas e cinzentas). (Adaptado de Daniels et al DOI:. 10,1128 / AAC.05737-11) Por favor clique aqui para ver uma v maiorersion desta figura.
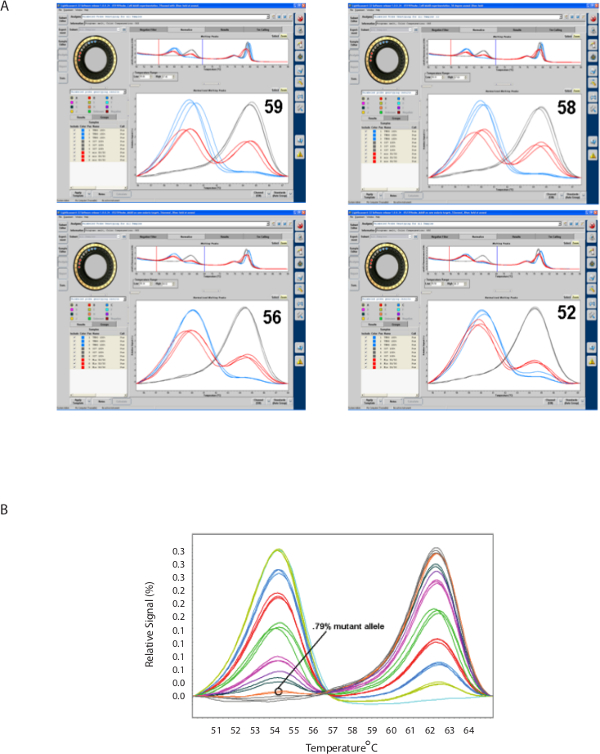
Figura 4. Mutante amplificação alelo polarização (Maab). A. Progressivamente a diminuição da temperatura de recozimento de amplificação faz o enviesamento do pico de fusão para o pico de reacção correspondente ao alelo mutante (temperatura mais baixa) numa amostra polygenomic ou polyallelic. B. Maab resulta em sensibilidade GRH para detectar alelos menores presente em menos do que 1% em amostras polygenomic ou polyallelic. (Parte B adaptado de Daniels et al DOI:. 10,1128 / AAC.05737-11) Clique aqui para ver uma versão maior desta figura.
Tabela 1. Set-up de alta resolução reacções de amplificação de fusão à base de sondagem.
| Componente | Volume por reação (placa de 96 poços e tubo capilar) | Volume por reacção (384 poços da placa) | A concentração final |
| HRM Master Mix | 4-5 ul | 2-2,5 ul | 1 x |
| 10 x iniciador em excesso | 1 ul | 0,5 ul | 0,50 mM |
| 10 x outro iniciador | 1 ul | 0,5 � | 0,1 uM |
| 10x Probe | 1 ul | 0,5 ul | 0,40 mM |
| Água PCR-grade | 1-2 ul | 0,5-1 ul | |
| ADN molde | 1 ul | 0,5 ul | 0,01-10 ng / ul |
| 10 ul | 5 ul |
Tabela 2. Standard alta resolução condições de amplificação de fusão.
| Temperatura | Tempo | |
| Aguarde | 95 ° C | 120 seg |
| 45 ciclos | 95 ° C | 30 seg |
| 68 ° C * | 30 seg | |
| 1 ciclo | 95 ° C | 30 seg |
| 28 ° C | 30 seg |
* Esta temperatura é dependente do amplicão
Tabela 3. As condições de amplificação para amplificação de polarização alelo mutante.
| Temperatura | Tempo | Cycles | Taxa de rampa | Modo de aquisição | Modo de análise | |
| Desnaturar | 95 ° C | 30 seg | 1 | 20 | Nenhum | Nenhum |
| Ciclo | 95 ° C | 2 seg | 55 | 20 | Nenhum | Quantificação |
| 56 ° C * | 15 seg | 20 | Solteira | |||
| Fundição | 40 ° C | 0 seg | 0,3 | Nenhum | Nenhum | |
| 45 ° C * | 0 seg | Continous | Fundição | |||
| 90 ° C * | 0 seg |
* Esta temperatura é dependente do amplicon e será reduzido ao realizar Maab
Discussão
Continous HRM é um passo a análise pós-PCR; por conseguinte, a amostra de ensaio e a configuração é similar com protocolos padrão de PCR, com a incorporação adicional de um corante fluorescente intercalante usado para controlar a transição da cadeia dupla de ADN de cadeia simples durante o passo de fusão de alta resolução. O fator mais importante para a análise HRM bem sucedida é um produto robusto PCR. Desenho do ensaio é a chave, e várias ferramentas otimizadas para o projeto ensaio HRM estão disponíveis comercialmente e em linha 13. Comprimentos AMPLICON de 80-150 pares de bases com acompanhamento ~ 20 pares de bases bloqueado sondas centradas sobre o SNP ou SNPs de trabalho melhor interesse para diferenciar SNPs, bem como haplótipos de múltiplos SNPs na região da sonda. As sondas podem ser bloqueadas utilizando uma 3 'espaçador C3 ou um par incompatibilidade 2 de base na extremidade 3', o que impede a extensão durante a amplificação. As sondas podem ser concebidas para combinar com a cadeia de Watson Crick ou molde; a escolhadepende de testes empíricos para determinar qual projeto sonda produz picos de fusão aceitáveis. O excesso de avanço ou retrocesso iniciadores são utilizados para a produção de produto de amplificação de cadeia simples que hibrida com as sondas bloqueados. Se a sonda contém a sequência da cadeia para a frente, em seguida, o iniciador inverso é utilizado em excesso, normalmente numa proporção de 1: 5, e vice-versa para as sondas que correspondem a cadeia inversa.
Da mesma forma, GRH funciona melhor com o produto de PCR suficientes e robusta. Reacção de PCR requer optimização de PCR e de gradiente de agarose ou géis de análise Bioanalyzer para determinar as temperaturas de recozimento óptimas. Concentração do molde pode ser aumentada usando métodos como a pré-amplificação, a amplificação multiplexada tendenciosa de todos os alvos utilizando baixa concentração do primário e o número de ciclos para aumentar a concentração de molde 13.
Apenas um punhado de instrumentos são compatíveis com Maab. As propriedades térmicas dos tubos capilares de vidro usado iN estes sistemas facilitar este método. O pequeno diâmetro interior dos capilares bem como a rápida transferência de calor através do vidro proporcionam um controlo de temperatura mais severas do que utensílios de plástico de PCR padrão, que tem mais lentas taxas de transição entre o recozimento e de desnaturação ciclos devido às propriedades isolantes térmicas dos plásticos. Com este método, as sensibilidades de detecção pode ser reduzido de 2-5% a menos de 1% de um alelo mutante em uma mistura de tipo selvagem e alelos mutantes 10.
HRM é uma ferramenta fácil, eficiente e econômica para genotipagem SNP em uma variedade de configurações e inúmeras aplicações, desde a vigilância das doenças infecciosas para a digitalização para as variantes do gene do cancro. Vários outros instrumentos, tais como o Roche Nano e sistemas LightCycler (480 e 96), bem como o de Eco em tempo real do sistema de PCR oferta GRH além de amplificação padrão, em tempo real, e a funcionalidade de número de cópias. Usando máquinas de maior rendimento, custa HRM barcoding less de US $ 0,50 por ensaio em nossas mãos, incluindo todos os reagentes e consumíveis.
No contexto descrito aqui, os aplicativos baseados em campo permitir a vigilância da população em tempo real para mudanças associadas com os esforços de controle da malária, incluindo a redução da diversidade da população, a detecção de tipos de parasitas importados, e o aparecimento ea propagação de SNPs associados à droga sensibilidade reduzida. Refinamentos ao método oferecer sensibilidade adicional útil para informar o progresso da eliminação da malária e de erradicação.
Divulgações
Os autores não têm nada a revelar.
Agradecimentos
Os autores agradecem a Fundação Bill e Melinda Gates para seu apoio de desenvolvimento de tecnologia e treinamento.
Materiais
| Name | Company | Catalog Number | Comments |
| LightScanner Master Mix | BioFire Defense | HRLS-ASY-0003 | |
| Light mineral oil | Sigma | M5904 | for BioFire Defense plate-based HRM systems |
| TE | Teknova | T0226 | |
| Te | Teknova | T0221 | TE buffer with reduced EDTA |
| PCR grade water | Teknova | W3330 | |
| Plasmodium falciparum standards | MR4 | MRA-151, 156, 205, 330 | MR4.org offers free genomic DNA |
| Light Scanner Primer Design Software | BioFire Defense | commercial sofware for HRM assay design | |
| LightCycler 480 High Resolution Melting Master Mix | Roche | 4909631001 | For use in LightCycler-480, -96, and nano. 2x concentration |
| Bioanalyzer | Agilent | G2938A | Agilent 2100 bioanalyzer |
| LightCycler 2.0 | Roche | 3531414001 | Capillary-based system for MAAB |
| LightCycler Nano | Roche | 6407773001 | Strip tube-based HRM and amplification |
| LightCycler 96 | Roche | 5815916001 | Strip-tube and plate-based HRM and amplification |
| LightCycler 480 | Roche | 5015278001 | plate-based HRM (96 and 384-well plates) and amplification |
| LightScanner-96 | BioFire Defense | LSCN-ASY-0011 | plate-based HRM (96-well) |
| LightScanner-384 | BioFire Defense | LSCN-ASY-0001 | plate-based HRM (96-well) |
| 96-well plates | Roche | 4729692001 | 96-well plates for HRM (LightCycler |
| 384-well plates | Roche | 4729749001 | 384-well plates for HRM (LightCycler) |
| 96-well plates | Bio-Rad | HSP-9665 | 96-well plates for HRM (LightScanner) |
| 384-well plates | Bio-Rad | HSP-3865 | 384-well plates for HRM (LightScanner) |
| LightCycler 8-Tube Strips (clear) | Roche | 6327672001 | strip tubes for HRM (Light Cycler 96 and Light Cycler Nano) |
| LightCycler Capillaries (20 μl) | Roche | 4929292001 | capillaries for HRM |
| Optical plate seals | Roche | 4729757001 | other brands also work |
Referências
- . . World Health Organization report 2013. , 1-286 (2013).
- Hayton, K., Su, X. -. Z. Drug resistance and genetic mapping in Plasmodium falciparum. Current genetics. 54 (5), (2008).
- Neafsey, D. E., et al. Genome-wide SNP genotyping highlights the role of natural selection in Plasmodium falciparum population divergence. Genome biology. 9 (12), R171 (2008).
- Volkman, S. K., et al. A genome-wide map of diversity in Plasmodium falciparum. Nature. 39 (1), 113-119 (2007).
- Volkman, S. K., Neafsey, D. E., Schaffner, S. F., Park, D. J., Wirth, D. F. Harnessing genomics and genome biology to understand malaria biology. Nature Reviews Genetics. 13 (5), 315-328 (2012).
- Manske, M., et al. Analysis of Plasmodium falciparum diversity in natural infections by deep sequencing. Nature. 487 (7407), 375-379 (2012).
- Auburn, S., et al. Characterization of within-host Plasmodium falciparum diversity using next-generation sequence data. PLoS ONE. 7 (2), e32891 (2012).
- Daniels, R., et al. A general SNP-based molecular barcode for Plasmodium falciparum identification and tracking. Malaria Journal. 7, 223 (2008).
- Daniels, R., et al. Genetic surveillance detects both clonal and epidemic transmission of malaria following enhanced intervention in Senegal. PLoS ONE. 8 (4), e60780 (2013).
- Daniels, R. R., et al. field-deployable method for genotyping and discovery of single-nucleotide polymorphisms associated with drug resistance in Plasmodium falciparum. Antimicrobial agents and chemotherapy. 56 (6), 2976-2986 (2012).
- Olbadia, N., et al. Clonal outbreak of Plasmodium falciparum in eastern Panama. The Journal of infectious diseases. , (2014).
- Montgomery, J., Wittwer, C. T., Palais, R., Zhou, L. Simultaneous mutation scanning and genotyping by high-resolution DNA melting analysis. Nature protocols. 2 (1), 59-66 (2007).
- Mharakurwa, S., Daniels, R., Scott, A., Wirth, D. F., Thuma, P., Volkman, S. K. Pre-amplification methods for tracking low-grade Plasmodium falciparum populations during scaled-up interventions in Southern Zambia. Malaria Journal. 13, 89 (2014).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados