Method Article
I metodi per aumentare la sensibilità di High Resolution Melting polimorfismo a singolo nucleotide genotipizzazione a malaria
In questo articolo
Riepilogo
Mentre l'analisi alta risoluzione fusione offre la capacità di distinguere tra polimorfismi a singolo nucleotide in una popolazione eterogenea, allele mutante pregiudizi amplificazione può aumentare la sua capacità di individuare alleli presenti a relativamente basse percentuali all'interno di un campione. Questo protocollo descrive miglioramenti che migliorano la sensibilità dell'analisi fusione alta risoluzione.
Abstract
Nonostante decenni di sforzi per l'eradicazione, la malaria rimane un onere globale. Recente rinnovato interesse per l'eliminazione regionale e l'eradicazione globale è stata accompagnata da una maggiore informazione genomica su Plasmodium specie parassita responsabile della malaria, comprese le caratteristiche delle popolazioni geografiche così come le variazioni associate a ridotta sensibilità ai farmaci antimalarici.
Una variazione genetica comune, polimorfismi a singolo nucleotide (SNP), offre un target interessante per parassita genotipizzazione. Questi marcatori sono utili non solo per il monitoraggio marcatori di resistenza ai farmaci, ma anche per il monitoraggio delle popolazioni di parassiti utilizzando marcatori non inferiore a droga o altre pressioni selettive.
Metodi di genotipizzazione SNP offrono la possibilità di monitorare la resistenza ai farmaci, nonché per le impronte digitali individuali parassiti per la sorveglianza della popolazione, in particolare in risposta agli sforzi di controllo della malaria nelle regioni avvicinano eliminatlo stato di ioni.
Mentre SNPs informativi sono stati identificati, che sono agnostico di specifiche tecnologie di genotipizzazione, ad alta risoluzione di fusione (HRM) analisi è particolarmente adatto studi basati-campo a. Rispetto ai metodi standard fluorescente-probe based che richiedono le singole SNPs in una singola sonda marcata e offrono sensibilità nella migliore delle ipotesi il 10% per individuare SNPs in campioni che contengono più genomi (polygenomic), gestione delle risorse umane offre sensibilità 2-5%. Modifiche HRM, quali sonde bloccate e concentrazioni dei primer asimmetrici nonché l'ottimizzazione di temperature di ricottura per polarizzare amplificazione PCR verso amplificazione allele minore, aumentano ulteriormente la sensibilità di HRM. Mentre il miglioramento sensibilità dipende dal dosaggio specifico, abbiamo aumentato sensibilità di rilevamento a meno dell'1% dell'allele minore.
Nelle regioni che si avvicinano eradicazione della malaria, la diagnosi precoce di emergere o di resistenza ai farmaci importati è essenziale per prrisposta ompt. Allo stesso modo, la capacità di rilevare infezioni polygenomic e differenziare i tipi di parassiti importati dai serbatoi locali criptici può informare i programmi di controllo.
Questo manoscritto descrive le modifiche alla tecnologia di fusione ad alta risoluzione che aumentano ulteriormente la sua sensibilità per individuare infezioni polygenomic nei campioni dei pazienti.
Introduzione
Nonostante il rinnovato interesse per il controllo della malaria e l'eradicazione, la malaria rimane un onere in tutto il mondo, con quasi la metà della popolazione mondiale a rischio di infezione e di oltre 550.000 decessi ogni anno, in particolare dei bambini in Africa sub-sahariana 1.
Questi nuovi programmi di controllo e di eradicazione sono stati sostenuti dalla rinascimento genomica, con un gran numero di parassiti della malaria sequenziato e analizzato per le mutazioni associate con la sensibilità ai farmaci ridotta, una maggiore virulenza, e per caratteristiche della popolazione 2,3. Polimorfismi a singolo nucleotide (SNP) sono tra le varianti genetiche più comunemente identificate 4-7.
Metodi di genotipizzazione SNP portatili offrono in loco e la sorveglianza della popolazione in tempo reale e il monitoraggio 8. Oltre alle impronte digitali individuali parassiti, il 'codice a barre molecolare' è anche usato per rilevare drammatici cambiamenti temporali nella frequenza dell'allelenonché varianza effettiva dimensione della popolazione e la complessità di infezione 9.
Anche se questo insieme di informativa SNP si adatta facilmente a molte piattaforme di genotipizzazione, alta risoluzione sciogliendo (HRM) analisi è particolarmente adatto agli studi, dove il funzionamento e l'individuazione di nuove mutazioni a costi contenuti sensibili e semplici rispetto al sequenziamento e l'altro campo-base approcci sono attraenti in contesti poveri di risorse.
HRM inizia con reazione a catena della polimerasi standard (PCR) che incorpora un colorante fluorescente. Post-PCR analisi di fusione determina la temperatura di picco amplicone di fusione; una sola differenza SNP in un breve amplicone può risultare in sostanziale temperatura picco di fusione (Tm) Differenze.
Diversi perfezionamenti a questo metodo offre migliore risoluzione genotipizzazione di distinguere SNPs tra cui classe IV (AT) SNPs, e rilevare minori alleli mutanti in campioni con più alleli presenti (polyginfezioni enomic). Innanzitutto, i saggi incorporano brevi sonde centrati sulla regione SNP, oltre a trasmettere e reverse primer che sono presenti a diverse concentrazioni. Queste sonde bloccate non sono amplificati durante la PCR, ma si legano al filamento prodotta in eccesso a causa di concentrazioni dei primer asimmetrici che aumentano la produzione del prodotto probe-template. Questi ampliconi doppio filamento separato, comprensivi di sonda legata al modello filo in eccesso sono ~ 20-30 bp; la loro lunghezza significativamente diminuito rispetto a tutta la amplicone (80-150 bp) portano a differenze molto più grandi Tm associati con base singola o multipla della sonda-template disallineamenti 10.
In secondo luogo, l'amplificazione allele mutato bias (MAAB) abbassa la temperatura di ricottura reazione al pregiudizio la reazione verso alleli mutanti presenti a bassi rapporti di infezioni polygenomic. La temperatura di ricottura si trova tra le Tms di perfettamente abbinati (wild-type) e non corrispondenti sonda (mutante)S. A questa temperatura, il legame del allele wild-type è abbastanza stabile che l'amplificazione è ostacolato rispetto al suo equivalente disadattamento, polarizzando in tal modo l'amplificazione allele mutante verso il quando entrambi sono presenti in un singolo campione 10.
Con questi filtri HRM, questa tecnologia ha permesso di tracciamento di origine e l'identità di parassiti associati alle infezioni epidemiche in Sud America 11 e rilevazione di nuove mutazioni, la differenziazione di più SNP situati immediatamente accanto all'altro in sequenza, e mutazioni presenti in meno di 1 % degli alleli in campioni polygenomic 10.
Aumento della sensibilità è particolarmente importante nelle regioni che si avvicinano di stato di pre-eliminazione e quelli a rischio di emergente resistenza ai farmaci. La capacità di identificare rapidamente e facilmente i parassiti importati e SNPs associati con ridotta sensibilità in loco informa gli sforzi di sorveglianza e di controllo sui programmil'efficacia delle loro implementazioni e identifica hotspot per maggiori sforzi di eradicazione della malaria e di eliminazione. Questo protocollo descrive i metodi per la sonda bloccato-based e MAAB per una maggiore sensibilità HRM genotipizzazione.
Protocollo
Nota: Questo protocollo include i volumi e le concentrazioni per 96 pozzetti, strip 8-tubo, e sistemi basati capillari-(volume di reazione 10 microlitri); tuttavia, HRM può essere eseguita anche in sistemi basati piastriformi 384 pozzetti con un volume di reazione 5 microlitri scalando tutti i volumi corrispondentemente. La gamma di rilevazione per la maggior parte dei sistemi è di 10 pg a 10 ng DNA stampo.
1. Preparare Modelli
- Ottenere campioni Plasmodium falciparum direttamente dal sangue ottenuti da pazienti che partecipano a studi sul campo del sito e memorizzati come sangue intero, globuli rossi pellet, o su carta da filtro.
Nota: I campioni possono anche essere cultura adattata a crescere in vitro; alcuni ceppi standard di laboratorio per l'analisi possono essere ordinati dalla Malaria Research and Reference reagente Resource Center (MR4, i campioni possono essere utilizzati direttamente dal pellet globuli rossi o di cultura o estratte dal sangue intero, globuli rossi, o carta da filtro. - DNA estratto dacarta da filtro o ceppi culturali adattate
Nota: HRM è sensibile alla concentrazione di sale; differenze di concentrazione a causa di metodi di estrazione variabili possono influenzare significativamente la temperatura di fusione. Mentre specifici tamponi finali non sono un fattore significativo per il successo, utilizzare un eluizione o risospensione tampone standard comune (ad esempio, acqua, 1x Te ['basso Te' o 'il DNA tampone sospensione': 10 mM Tris-Cl, 0.10 mM EDTA] o 1x TE [10 mM Tris-Cl, 1 mM EDTA]) per tutti i campioni per evitare curve melt anomale o incoerenti.- Preparare diluizioni modello di 10 pg -10 ng per ml nella norma Te, TE o acqua per ogni reazione 10 microlitri.
- Diretto amplificazione da globuli rossi pellet
- Determinare parassitemia di globuli rossi usando la microscopia sottile striscio
Nota: I metodi per la determinazione parassitemia dei globuli rossi infetti sono disponibili presso i Centers for Disease Control and Prevention (CDC): http: //www.cdc.gov/dpdx/diagnosticProcedures/blood/microexam.html - Diluire parasitemias superiore a 0,5% 1: 400 in PCR-grade acqua, TE o Te. Utilizzare 3 microlitri per ogni 5- 10 microlitri di reazione.
- Determinare parassitemia di globuli rossi usando la microscopia sottile striscio
- Controlli
Nota: HRM può essere utilizzato per la scansione gene per rilevare variazioni di sequenza in una popolazione 12; tuttavia, la genotipizzazione accurata richiede standard sequenza verificato per contenere il SNP interrogato. Costrutti plasmide o malaria isolati possono essere utilizzati con genotipi noti. Per la maggior parte delle applicazioni, 3D7 (MR4 MRA-151) è usato come controllo wild-type. A causa di conduzione tra la variabilità, includere sempre controlli positivi e negativi.- I controlli positivi: Preparare 10 pg - 10 ng per 1 ml diluizioni del plasmide o norme con sequenza verificata SNP genotipi.
- Controllo negativo: acqua pura Usa PCR come controllo no-template (NTC) per le reazioni di amplificazione.
- Preparare miscela di reazione
- Copertura di olio (opzionale) Mineral. Nota: alcuni sistemi di gestione delle risorse umane sono piattaforme di fusione standalone e non hanno un coperchio riscaldato per evitare la condensazione ed evaporazione del prodotto durante l'amplificazione.
- Aggiungere 20 ml di olio minerale leggero per ogni piastra ben prima di caricare la miscela di reazione.
- Preparare 10x scorte di lavoro di tutti i saggi
- Fare riferimento alla Tabella 1 per consigliati 10x concentrazioni azionari di lavoro, nonché le concentrazioni finali per HRM genotipizzazione basato su sonda bloccato.
- Preparare miscele di reazione
- Per ogni bene nel piatto o tubo, aggiungere 1 ml 10x lavoro stock avanti e reverse primer e sonde, 4,0-5,0 microlitri 2.5x 2.0x o HRM master mix, 1 modello ml, e di acqua grado PCR per un volume totale di reazione di 10 pl .
- Piastre di copertura o tubi
- Utilizzare lamina ottica seals per l'amplificazione basato piastra, tappi ottici per sistemi basati su nastro, tubi e tappi di plastica per sistemi basati capillari. Assicurarsi che la copertura è completa e piatti e tappi sono completamente sigillato e protetto per evitare l'evaporazione durante l'amplificazione.
- Campioni Spin
- Piastre Spin 3 min a 1.800 xg per eliminare le bolle d'aria e di separare le fasi del petrolio e acquose se si usa olio minerale.
- Copertura di olio (opzionale) Mineral. Nota: alcuni sistemi di gestione delle risorse umane sono piattaforme di fusione standalone e non hanno un coperchio riscaldato per evitare la condensazione ed evaporazione del prodotto durante l'amplificazione.
- Protocollo Amplificazione
- Posizionare piastre di reazione o tubi in termociclatore. Protocolli bloccati-sonda o MAAB amplificazione standard Run (tabelle 2 e 3 rispettivamente). Notare la differenza di temperatura di ricottura tra questi metodi.
Analisi 3. HRM
- Fusione
- Dopo l'amplificazione PCR, procedere alla fusione di analisi. Per i sistemi di fusione standalone che non sono dotati di amplificazione (ad es., Sistemi BioFire Difesa LightScanner), respostare piatto amplificato dal termociclatore e posto in strumenti HRM. Dall'interfaccia software di sistema, sciogliere il piatto 40-80 ° C per la produzione di entrambi i picchi di fusione della sonda e ampliconi.
Nota: per salvare fusione e analisi in tempo, la finestra di fusione può essere ridotto a copertura range di temperatura solo per sondare o regioni amplicone di fusione. Dopo la fusione, piastre e tubi possono essere ri-fuso se necessario e conservato a -20 ° C o 4 ° C. Conservare le provette di vetro solo a 4 ° C per evitare che i tubi di cracking.
- Dopo l'amplificazione PCR, procedere alla fusione di analisi. Per i sistemi di fusione standalone che non sono dotati di amplificazione (ad es., Sistemi BioFire Difesa LightScanner), respostare piatto amplificato dal termociclatore e posto in strumenti HRM. Dall'interfaccia software di sistema, sciogliere il piatto 40-80 ° C per la produzione di entrambi i picchi di fusione della sonda e ampliconi.
- Analisi Melt
- Rimuovere campioni negativi
- All'interno del software di analisi, togliere dal altri campioni di analisi che non amplificare o che hanno picchi di fusione frastagliate. Nota: Dopo aver selezionato la modalità di analisi di un file di esecuzione individuale, lo strumento raggruppa automaticamente i campioni positivi e negativi. Prima genotipizzazione ogni sottoinsieme, l'utente può modificare manualmente le chiamate.
- Sia da fusionecurva o picco di fusione vista, fare clic destro su curve da rimuovere per selezionarli. Dopo aver selezionato i campioni mal classificati, andare su "Nuova chiamata", selezionare il gruppo appropriato e fare clic su "Apply Change".
- All'interno del software di analisi, togliere dal altri campioni di analisi che non amplificare o che hanno picchi di fusione frastagliate. Nota: Dopo aver selezionato la modalità di analisi di un file di esecuzione individuale, lo strumento raggruppa automaticamente i campioni positivi e negativi. Prima genotipizzazione ogni sottoinsieme, l'utente può modificare manualmente le chiamate.
- Normalizzare
- Dalla derivata negativa della fluorescenza normalizzata rispetto alla temperatura (-dF / dT) ('Curve Difference') vista, spostare barre normalizzazione per circondare la sonda fondere regione.
Nota: La sonda sciogliere regione è la temperatura più bassa delle due regioni di fusione. Le barre di normalizzazione dovrebbero essere alla base dei picchi melt.
- Dalla derivata negativa della fluorescenza normalizzata rispetto alla temperatura (-dF / dT) ('Curve Difference') vista, spostare barre normalizzazione per circondare la sonda fondere regione.
- Calcolare gruppi
- Dopo la normalizzazione, selezionare 'Calcola gruppi' per assegnare automaticamente i campioni per i gruppi.
Se necessario, i campioni-assegnare nuovamente manualmente per gruppi specifici.
Nota: alcuni software di analisi ha soglie di alta sensibilità che non possono essere modificati; in questi casi, la software potrebbe assegnare i campioni a diversi gruppi che appartengono visivamente ad un altro.
- Dopo la normalizzazione, selezionare 'Calcola gruppi' per assegnare automaticamente i campioni per i gruppi.
- Assegnare genotipi
- Assegnare genotipi per ogni gruppo sulla base di standard (controlli positivi).
- Dopo che l'utente conferma che i gruppi calcolati siano corretti (se non, che i cambiamenti corrette sono state apportate nella scheda Raggruppamento selezionando campioni erroneamente classificati e selezionando il gruppo destro dal menu a casella accanto a "Nuova chiamata"), selezionare "Modifica nomi di gruppo "sotto la scheda" Raggruppamento ".
- Inserire i genotipi delle norme in area colorata corrispondente (ad esempio, se 3D7 standard ha conosciuto un genotipo ed è nel gruppo rosso, il gruppo rosso ha genotipo A). Premere il tasto "OK" e salvare i risultati.
- Assegnare genotipi per ogni gruppo sulla base di standard (controlli positivi).
- Esportazione dei risultati
- Esportare il genotipo chiama come un foglio di calcolo per ulteriori analisi campione di popolazione.
- Rimuovere campioni negativi
Risultati
I dati possono essere visualizzati in diversi modi, a seconda del software di analisi e strumento. Tipicamente, un grafico della derivata negativa della fluorescenza normalizzata rispetto alla temperatura (-dF / dT) contro Risultati temperatura è più semplice per visualizzare picchi di fusione e determinare genotipi.
Una finestra di fusione completa (40 ° C-80 ° C) si tradurrà in entrambe le regioni ampliconi e sonda di fusione. La Figura 1 mostra un esempio di picchi di fusione normalizzati di entrambe le regioni. Impostazione della finestra di analisi ai soli risultati regione sonda in chiara differenziazione di picchi corrispondenti a specifici SNPs (Figura 2).
Analisi basata-Probe mostra chiaramente la presenza di entrambi gli alleli come picchi individuali con temperature di fusione che hanno picchi SNP omozigoti (Figura 3).
Figura 4 mostra che Progressivamente riducendo la temperatura di ricottura durante amplificazione (MAAB) risultato in un orientamento verso l'allele mutante (picco lato sinistro) (Figura 4A), con conseguente aumento della sensibilità per l'allele mutante in una popolazione di alleli misti (Figura 4B).
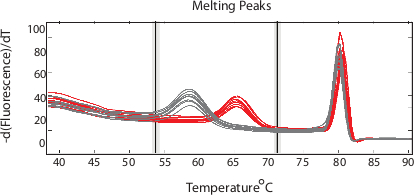
Figura 1. sonda e amplicon fusione regioni. Plot la derivata negativa della fluorescenza normalizzato rispetto alla temperatura in un ampio risultati finestra di fusione sia sonda (temperatura bassa) e picchi amplicone di fusione che possono essere analizzati. (Tratto da Daniels et al DOI:. 10,1128 / AAC.05737-11) Cliccate qui per vedere una versione più grande di questa figura.
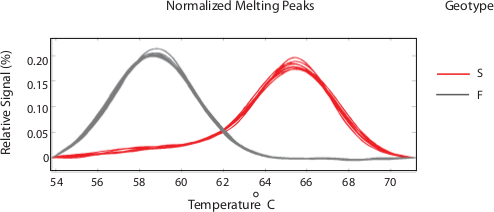
Figura 2. sonda picchi di fusione. Partite perfette (tipicamente wild type alleli) provocare picchi di fusione più elevati (rosso), mentre l'inadeguatezza SNP hanno temperature di fusione inferiori (grigio). Cliccate qui per vedere una versione più grande di questa figura.
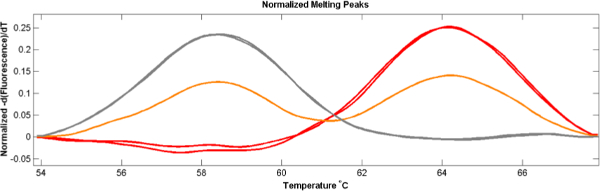
Figura 3. Polygenomic fusione picchi. Quando sono presenti in un campione entrambi gli alleli, analisi HRM sonda-based rappresenta entrambi gli alleli come le curve di due picchi (arancione), con picchi che corrispondono campioni singolo allele (rosso e grigio). (Tratto da Daniels et al DOI:. 10,1128 / AAC.05737-11) Cliccate qui per vedere una grande versione di questa figura.
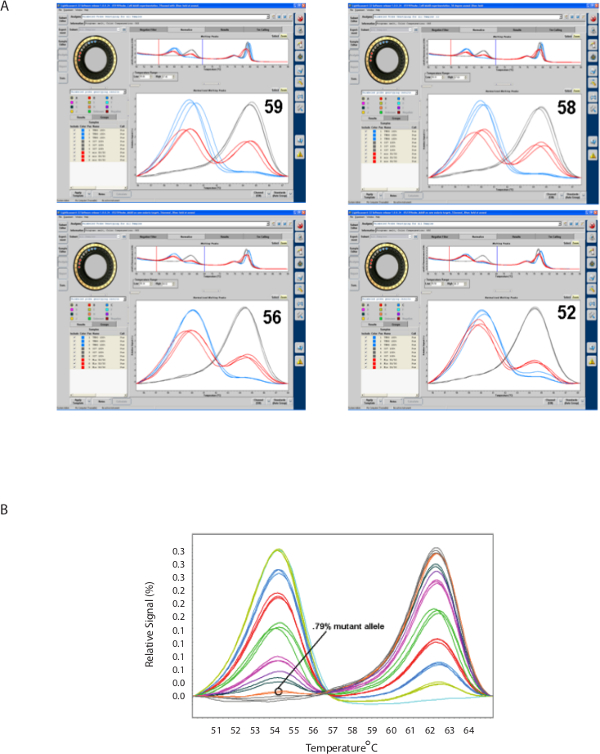
Figura 4. Mutant allele amplificazione polarizzazione (MAAB). A. Abbassando progressivamente la temperatura di ricottura di amplificazione polarizza la reazione picco di fusione verso picco corrispondente mutante (temperatura inferiore) in un campione polygenomic o polyallelic. B. MAAB traduce in sensibilità HRM per rilevare alleli minori presenti a meno dell'1% nei campioni polygenomic o polyallelic. (Parte B adattato da Daniels et al DOI:. 10,1128 / AAC.05737-11) Cliccate qui per vedere una versione più grande di questa figura.
Tabella 1. Set-up di alta risoluzione reazioni di fusione di amplificazione della sonda-based.
| Componente | Volume per reazione (96 pozzetti e capillare) | Volume per reazione (384 pozzetti) | Concentrazione finale |
| HRM Master Mix | 4-5 microlitri | 2-2,5 ml | 1 x |
| 10 x eccesso di primer | 1 ml | 0,5 microlitri | 0.50 micron |
| 10 x Altro di primer | 1 ml | 0.5μl | 0,1 micron |
| 10x Probe | 1 ml | 0,5 microlitri | 0.40 micron |
| Acqua PCR-grade | 1-2 microlitri | 0,5-1 microlitri | |
| DNA Template | 1 ml | 0,5 microlitri | 0,01-10 ng / ml |
| 10 microlitri | 5 ml |
Tabella 2. Standarad alta risoluzione delle condizioni di fusione di amplificazione d.
| Temperatura | Orario | |
| Tenere | 95 ° C | 120 sec |
| 45 cicli | 95 ° C | 30 sec |
| 68 ° C * | 30 sec | |
| 1 ciclo | 95 ° C | 30 sec |
| 28 ° C | 30 sec |
* Questa temperatura è amplicone-dipendente
Tabella 3. Condizioni di amplificazione per mutante allele pregiudizi amplificazione.
| Temperatura | Orario | Cicli | Rampa | Modalità di acquisizione | Modalità di analisi | |
| Denaturare | 95 ° C | 30 sec | 1 | 20 | Nessuna | Nessuna |
| Ciclo | 95 ° C | 2 sec | 55 | 20 | Nessuna | Quantificazione |
| 56 ° C * | 15 sec | 20 | Unico | |||
| Fusione | 40 ° C | 0 sec | 0.3 | Nessuna | Nessuna | |
| 45 ° C * | 0 sec | Continuo | Fusione | |||
| 90 ° C * | 0 sec |
* Questa temperatura è amplicone-dipendente e si riduce quando si eseguono MAAB
Discussione
Continua HRM è un'analisi fase di post-PCR; Pertanto, il campione e la configurazione del test è simile ai protocolli PCR standard, con l'inserimento aggiuntivo di un colorante intercalante fluorescente utilizzato per monitorare la transizione dal doppio filamento di DNA a singolo filamento durante la fase di fusione ad alta risoluzione. Il singolo fattore più importante per l'analisi HRM di successo è un prodotto robusto PCR. Disegno Assay è la chiave, e diversi strumenti ottimizzati per la progettazione del test HRM sono disponibili in commercio e online il 13. Lunghezze ampliconi di 80-150 coppie di basi con accompagnamento ~ 20 coppia di basi bloccate sonde centrati sul SNP o SNP di lavoro interesse migliore per differenziare SNP così come aplotipi di più SNPs nella regione della sonda. Sonde possono essere bloccati utilizzando un 'C3 distanziatore o una coppia disadattamento di base 2 al 3' 3 fine, che impediscono l'estensione durante l'amplificazione. Le sonde possono essere progettati per abbinare il filo Watson o Crick modello; la sceltadipende dalla verifica empirica per determinare quale sonda il design produce picchi di fusione accettabili. Eccesso avanti o indietro primer vengono utilizzati per la produzione del prodotto a singolo filamento di amplificazione che annealing alle sonde bloccati. Se la sonda contiene la sequenza filamento in avanti, poi invertire primer viene utilizzato in eccesso, tipicamente in un rapporto 1: 5, e viceversa per sonde corrispondenti al filamento opposto.
Allo stesso modo, HRM funziona meglio con sufficiente e robusto prodotto della PCR. Ottimizzazione reazione PCR richiede gradiente PCR e agarosio gel o analisi Bioanalyzer per determinare temperature ottimali di ricottura. Concentrazione modello può essere aumentato utilizzando metodi come la pre-amplificazione, di parte l'amplificazione multiplex di tutti gli obiettivi con bassa concentrazione di primer e numeri ciclabili per aumentare la concentrazione modello 13.
Solo una manciata di strumenti sono compatibili con MAAB. Le proprietà termiche dei tubi capillari di vetro usati in questi sistemi facilitano questo metodo. Il piccolo diametro interno dei capillari nonché il rapido trasferimento di calore attraverso il vetro offrono il controllo della temperatura più rigorose di plasticware PCR standard, che ha tassi più lenti di transizione tra ricottura e denaturazione cicli a causa delle proprietà termiche isolanti dei materiali plastici. Con questo metodo, sensibilità di rivelazione può essere ridotta da 2-5% a meno dell'1% di un allele mutante in una miscela di wild-type e mutante alleli 10.
HRM è uno strumento facile, efficiente e economica per la genotipizzazione SNP in una varietà di impostazioni e numerose applicazioni, dalla sorveglianza delle malattie infettive per la scansione di varianti del gene del cancro. Diversi altri strumenti, come il Roche Nano e sistemi LightCycler (480 e 96), nonché la Eco Real-time offre il sistema PCR HRM oltre all'amplificazione standard in tempo reale, e funzionalità di copia-numero. Utilizzando macchine alto-throughput, costa HRM barcoding less di $ 0.50 per test nelle nostre mani, compresi tutti i reagenti e materiali di consumo.
Nel contesto descritto, le applicazioni sul campo consentono in tempo reale di sorveglianza di popolazione per i cambiamenti associati con il lavoro di controllo della malaria, tra cui ridotto la diversità demografica, l'individuazione di tipi di parassiti importati, e la comparsa e la diffusione di SNPs associati alla sensibilità ai farmaci ridotta. Perfezionamenti al metodo offrono sensibilità aggiuntivo utile per informare i progressi verso l'eliminazione della malaria e l'eradicazione.
Divulgazioni
Gli autori non hanno nulla da rivelare.
Riconoscimenti
Gli autori ringraziano la Fondazione Bill e Melinda Gates per il suo sostegno dello sviluppo tecnologico e della formazione.
Materiali
| Name | Company | Catalog Number | Comments |
| LightScanner Master Mix | BioFire Defense | HRLS-ASY-0003 | |
| Light mineral oil | Sigma | M5904 | for BioFire Defense plate-based HRM systems |
| TE | Teknova | T0226 | |
| Te | Teknova | T0221 | TE buffer with reduced EDTA |
| PCR grade water | Teknova | W3330 | |
| Plasmodium falciparum standards | MR4 | MRA-151, 156, 205, 330 | MR4.org offers free genomic DNA |
| Light Scanner Primer Design Software | BioFire Defense | commercial sofware for HRM assay design | |
| LightCycler 480 High Resolution Melting Master Mix | Roche | 4909631001 | For use in LightCycler-480, -96, and nano. 2x concentration |
| Bioanalyzer | Agilent | G2938A | Agilent 2100 bioanalyzer |
| LightCycler 2.0 | Roche | 3531414001 | Capillary-based system for MAAB |
| LightCycler Nano | Roche | 6407773001 | Strip tube-based HRM and amplification |
| LightCycler 96 | Roche | 5815916001 | Strip-tube and plate-based HRM and amplification |
| LightCycler 480 | Roche | 5015278001 | plate-based HRM (96 and 384-well plates) and amplification |
| LightScanner-96 | BioFire Defense | LSCN-ASY-0011 | plate-based HRM (96-well) |
| LightScanner-384 | BioFire Defense | LSCN-ASY-0001 | plate-based HRM (96-well) |
| 96-well plates | Roche | 4729692001 | 96-well plates for HRM (LightCycler |
| 384-well plates | Roche | 4729749001 | 384-well plates for HRM (LightCycler) |
| 96-well plates | Bio-Rad | HSP-9665 | 96-well plates for HRM (LightScanner) |
| 384-well plates | Bio-Rad | HSP-3865 | 384-well plates for HRM (LightScanner) |
| LightCycler 8-Tube Strips (clear) | Roche | 6327672001 | strip tubes for HRM (Light Cycler 96 and Light Cycler Nano) |
| LightCycler Capillaries (20 μl) | Roche | 4929292001 | capillaries for HRM |
| Optical plate seals | Roche | 4729757001 | other brands also work |
Riferimenti
- . . World Health Organization report 2013. , 1-286 (2013).
- Hayton, K., Su, X. -. Z. Drug resistance and genetic mapping in Plasmodium falciparum. Current genetics. 54 (5), (2008).
- Neafsey, D. E., et al. Genome-wide SNP genotyping highlights the role of natural selection in Plasmodium falciparum population divergence. Genome biology. 9 (12), R171 (2008).
- Volkman, S. K., et al. A genome-wide map of diversity in Plasmodium falciparum. Nature. 39 (1), 113-119 (2007).
- Volkman, S. K., Neafsey, D. E., Schaffner, S. F., Park, D. J., Wirth, D. F. Harnessing genomics and genome biology to understand malaria biology. Nature Reviews Genetics. 13 (5), 315-328 (2012).
- Manske, M., et al. Analysis of Plasmodium falciparum diversity in natural infections by deep sequencing. Nature. 487 (7407), 375-379 (2012).
- Auburn, S., et al. Characterization of within-host Plasmodium falciparum diversity using next-generation sequence data. PLoS ONE. 7 (2), e32891 (2012).
- Daniels, R., et al. A general SNP-based molecular barcode for Plasmodium falciparum identification and tracking. Malaria Journal. 7, 223 (2008).
- Daniels, R., et al. Genetic surveillance detects both clonal and epidemic transmission of malaria following enhanced intervention in Senegal. PLoS ONE. 8 (4), e60780 (2013).
- Daniels, R. R., et al. field-deployable method for genotyping and discovery of single-nucleotide polymorphisms associated with drug resistance in Plasmodium falciparum. Antimicrobial agents and chemotherapy. 56 (6), 2976-2986 (2012).
- Olbadia, N., et al. Clonal outbreak of Plasmodium falciparum in eastern Panama. The Journal of infectious diseases. , (2014).
- Montgomery, J., Wittwer, C. T., Palais, R., Zhou, L. Simultaneous mutation scanning and genotyping by high-resolution DNA melting analysis. Nature protocols. 2 (1), 59-66 (2007).
- Mharakurwa, S., Daniels, R., Scott, A., Wirth, D. F., Thuma, P., Volkman, S. K. Pre-amplification methods for tracking low-grade Plasmodium falciparum populations during scaled-up interventions in Southern Zambia. Malaria Journal. 13, 89 (2014).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon