Method Article
Correlativo Confocal e Microscopia Eletrônica 3D de uma célula sensorial específica
Neste Artigo
Resumo
Here, we introduce a method, cocem3D, to unveil the ultrastructure of a specific cell in its native tissue by bridging confocal and serial block-face scanning electron microscopy.
Resumo
Delineação de ultra-estrutura de uma célula é importante para compreender a sua função. Isto pode ser difícil para um projecto de tipos de células raras difundidos em tecidos feitos de diversos tipos de células, tais como células enteroendócrinas do epitélio intestinal. Estes sensores gastrointestinais de alimentos e bactérias têm sido difíceis de estudar porque a dispersão por outras células epiteliais, na proporção de 1: 1000. Recentemente, os ratinhos transgénicos repórter foram gerados para identificar células enteroendócrinas por meio de fluorescência. Uma delas é o peptídeo YY-GFP mouse. Usando este rato, foi desenvolvido um método para correlacionar e microscopia confocal de série bloco-face eletrônica de varredura. Nós chamado o método cocem3D e aplicou-o para identificar uma célula enteroend�rina específico no tecido e desvendar ultra-estrutura das células em 3D. A resolução de cocem3D é suficiente para identificar os organelos tão pequeno como vesículas secretoras e para distinguir as membranas celulares para o volume de rendering. Cocem3D pode ser facilmente adaptado para estudar a ultraestrutura 3D de outros tipos de células específicas no tecido nativo.
Introdução
A vida dentro de uma célula tem lugar no tempo e no espaço. Mudanças ao longo do tempo são frequentemente estudadas usando microscopia de lapso de tempo combinado com técnicas de imagem de fluorescência, como a microscopia de super-resolução. Espacial, em particular, o arranjo de organelas no interior de uma célula ou interacções célula-para-célula, só pode ser obtido por um relato completo da estrutura fina da célula. Um relato abrangente da estrutura fina de uma célula pode também trazer clareza de função genómica nos casos em que o genoma está disponível, como o C. elegans nematóide 1 ou o tricoplax Placozoa plana adherens 2. Microscopia eletrônica de cortes seriados é agora um reprodutível, tempo eficiente, e de tarefas graças menos dispendioso para o desenvolvimento de tecnologias automatizadas de microscopia eletrônica 3D, como bloco de série-face microscopia eletrônica de varredura 3 (SBEM).
A necessidade de informações estruturais para elucidar a função é muito evidente em certos cell tipos em que a função depende de interações físicas célula-célula, como neurônios, glia, ou células epiteliais sensoriais. Estamos particularmente interessados em elucidar como sinais sensoriais de nutrientes no lúmen do intestino são traduzidas em um sinal elétrico que, em última análise modula comportamentos apetitivas. O circuito é complexa, mas começa na parede do intestino, onde os nutrientes entrar em contacto com as células epiteliais sensoriais, chamadas células enteroendócrinas. Ao contrário de outras células epiteliais sensoriais, tais como células de sabor, enteroendócrinas células estão dispersas por todo o epitélio intestinal numa razão de um a mil 4-7. Conseqüentemente, eles têm sido difíceis de identificar e estudar, e por muito tempo eles eram vistos apenas como uma fonte de hormônios intestinais. Mas, com o desenvolvimento de ratinhos fluorescência repórter específicos de células, a função sensorial complexo destas células está a emergir. Usando um desses ratos repórter, um YY-GFP (PyyGFP) do mouse peptídeo, descobrimos que enteroendocrine células têm um proeminente cauda citoplasmática que nós nomeamos neuropod. O aparecimento de neuropods sugeriu uma função conservada na comunicação célula-a-célula. Assim, nós concluímos que ao documentar a ultra-estrutura de uma célula enteroend�rina, a função de neuropods podia ser derivado.
A necessidade de compreender a estrutura de um apêndice numa célula dispersa que é difícil de identificar era a principal razão para o desenvolvimento de um método para combinar microscopia confocal e SBEM. A célula de interesse foi identificada usando PyyGFP enteroend�rina ratinhos repórter específicos de células. O método permitiu-nos para documentar toda a ultra-estrutura de uma célula enteroend�rina e sua neuropod. Dentro neuropods, encontramos características estruturais dos axónios neuronais, e fora neuropods, encontramos uma relação física com glia entérico 8. Com efeito, neuropods conter cerca de 70% de todas as vesículas secretoras que sugerem um papel fundamental na função de secreção dessas células. Com base nadados estruturais, mais recentemente, verificou-se que através destas neuropods, células enteroendócrinas e neurónios que enervam o intestino formar um circuito neuroepitelial, semelhante ao de células gustativas na língua 8,9.
Descobrindo tais características dos mecanismos de quimio gastrointestinais provindos de dados estruturais reunidos usando este método de microscopia correlativo. Acreditamos que este método pode ser útil em outras áreas da biologia das células, em particular em que as células estão dispersas dentro dos tecidos a uma razão muito baixa. Nós que se refere ao método confocal como correlativo e microscopia de série rosto bloco eletrônico de varredura em 3D (Cocem3D). O método é composto das seguintes etapas principais: dissecção dos tecidos, microscopia confocal, imagens SBEM, SBEM e correlação imagem confocal, e segmentação manual. Em comparação com outros métodos de correlação, o conceito é simples, porque a correlação é física em vez de química.
Protocolo
Todos os cuidados com os animais e experimentos foram feitos em conformidade com um protocolo aprovado pelo Comitê Animal Care Institucional e Uso da Universidade Duke.
Cocem3D resultou de uma combinação dos protocolos previamente publicados 10,11 e foi aplicado aqui a estudar numa célula enteroend�rina específica utilizando um repórter rato PyyGFP 12. Este método pode ser facilmente aplicada a outras células de interesse utilizando ratinhos transgénicos repórter disponíveis comercialmente. O método é descrito em três seções: microscopia confocal Correlative, bloco-face de microscopia eletrônica de varredura de série (SBEM), e dados de renderização em 3D.
Correlativo microscopia confocal
O objetivo desta seção é para colher um segmento de tecido do cólon distal de dimensões que permitem confocal correlativa e SBEM imagem. O protocolo é como se segue:
1. Colheita de Tecidos
- Preparar10 ml de solução / ml de heparina de 50 ug em PBS.
- Prepare estoque anestésico xilazina / cetamina misturando 10 ml de cetamina (100 mg / ml) e 1 ml de xilazina (20 mg / mL).
- Diluir xilazina / cetamina estoque 1: 4 em 0,1 M PBS.
- Prepare de 100 ml de solução fixadora contendo 4% de paraformaldeído e 0,1% de glutaraldeído em PBS.
- Anestesiar um rato PyyGFP, 6 a 10 semanas de idade, com uma dose letal de xilazina / cetamina anestésico. Injetar o anestésico por via intraperitoneal a 0,15 ml por 20 g de peso mouse.
- Confirme anestesia adequada rato por beliscar a cauda ou dos pés, e usar pomada nos olhos para evitar o ressecamento.
- Use um fluxo de bomba peristáltica variável para perfundir fixador intracardial 10. Usando uma tesoura cirúrgica (13 cm de comprimento) corte abrir a cavidade abdominal para expor os intestinos, coração e pulmões. Coração com fórceps estreito-padrão de curvas (comprimento 12 cm) segurar e inserir a agulha butterfly (19 g) de bomba peristáltica into do ventrículo esquerdo. Imediatamente, corte átrio direito com mola tesoura reta (comprimento 4 mm). Perfundir a uma taxa de 2 ml / min primeiro com solução de heparina durante 1 min, e, em seguida, com solução fixadora arrefecido com gelo durante 15 minutos até a cauda do ratinho é completamente rígida.
Nota: É muito importante para perfundir a uma velocidade lenta para evitar a rotura dos pequenos vasos na mucosa intestinal. Estourando de navios irá comprometer a ultra-estrutura do tecido. Se a perfusão for adequada, o fígado deve girar de cor pálida dentro de 3-5 min. - Com uma tesoura pequena abrir a cavidade abdominal e excisar todo o cólon a partir da junção com o ceco até ao recto distal. Colocar o tecido em PBS gelado. Enquanto submerso em PBS, cortado com uma tesoura pequena mola do cólon ao longo do mesentério.
2. Fixação de dissecação e segmentos de tecido
- Utilizando um bisturi, cortar cerca de 6 segmentos de tecido pequenos (2 mm 2) a partir do cólon distai. Faça isso em uma folha ocera dentária f e submerso em algumas gotas de PBS.
- Pós-fixar os segmentos de tecido em solução fixadora durante 3 horas a 4 ° C.
3. Blocos dissecando-tissue micro
- Prepare 5% de agarose de baixo ponto de fusão em PBS e mantê-lo em um banho de água a 45 ° C.
- Incorporar os segmentos de tecido em 5% de baixo ponto de fusão agarose utilizando um pequeno recipiente de plástico, como o tamanho padrão Tissue-Tek Cryomold.
- Montar as seções embutidas em uma lâmina micrótomo vibrando e encher a bandeja de tampão com PBS gelado.
- Corte 300 mm tiras de tecido em 0.8 amplitude e 0,04 mm Velocidade / seg. Nota: Neste ponto, as tiras de tecido serão desalojados da agarose.
- Re-incorpore tiras de tecido 300 uM em agarose e montá-los em micrótomo perpendicularmente à lâmina do vibratome.
- Usando um vibratome, cortar tiras de tecido com uma espessura de 50 um. Nota: Os blocos de tecido finais devem ser de cerca de 300 m de largura x 50 mm de espessura. Estes dimensions são optimizadas a partir de experiências piloto confocal que mostraram que a espessura da célula enteroend�rina é entre 15-20 um e o comprimento de neuropod 30-70 nm. Portanto, uma célula inteira pode ser contido dentro de um bloco de tecido 300 um de largura por 50 mm de espessura se a orientação da célula é paralela à face do bloco de tecido. Estas dimensões variam, dependendo do tipo de células e tecidos de interesse.
- Armazenar os blocos de tecido em PBS a 4 ° C.
4. Imagem Confocal
- Prepara-se uma solução de corante nuclear DAPI por diluição em PBS a 1: 4000.
- Incubar blocos de tecidos em corante nuclear DAPI, durante 5 min. Nota: coloração DAPI facilita células individuais de distinção pelos seus núcleos, o que é útil para correlacionar as imagens confocal e SBEM.
- Blocos de tecido montar em cobrados lâmina de vidro, adicione algumas gotas de PBS, e cobri-los com uma lamela.
- Usando um microscópio confocal, identificar blocos com vilosidades intacto e as células PyyGFP de interesse e fotografada-los para obter z-stacks secções ópticas.
- Use um Zeiss Plano Apochromat objetivo 20X / 0.8 para obter z-pilhas de 1 mícron secções ópticas.
- Para o z-pilha, usar os canais para 405 nm (DAPI) para determinar a relação a outras células, 488 nm GFP endógeno para localizar a célula de interesse, e contraste de interferência diferencial (DIC) para determinar a localização da célula em relação ao lúmen.
- Use resolução de imagem de 1024 pixels ou superior.
5. Incorporação de Blocks em Agarose
- Preparar 10 ml de fixador contendo 4% de paraformaldeído e 2,5% de glutaraldeído em PBS.
- Remover os blocos de tecido de a lâmina de vidro por adição cuidadosa de PBS nas bordas da tampa de deslizamento para deslizar a lamela de distância da lâmina de vidro, sem danificar o bloco de tecido. Em seguida, use um pincel fino arte para transferir o bloco de tecido a partir do slide para um 10,5 ml de tubo de microcentrífuga contendo 4% de paraformaldeído e 2,5% de glutaraldeído fixador.
- Blocos de tecido pós-fix O / N a 4 ° C.
- Transferir os blocos de tecido de PBS. Em seguida, inserir blocos de apartamento em 5% baixo ponto de fusão agarose por intercalando-os entre duas lâminas de vidro.
Nota: Incorporação os blocos em uma fina camada de agarose facilita a manipulação posterior durante a coloração. - Corte a agarose em uma praça e fazer um entalhe no lado superior. Nota: Este passo é necessário para manter a orientação em passos subsequentes.
- Armazenar em blocos de um tubo de microcentrífuga de 1,5 ml com PBS a 4 ° C até posterior processamento.
Serial Block-cara Microscopia Eletrônica de Varredura (SBEM)
Nesta seção, o bloco de tecido é preparado e fotografada com SBEM em baixa ampliação. A imagem levantamento da face do bloco é então correlacionado com os dados confocal para identificar a região contendo a célula de interesse. Uma vez que a regiãoé identificado, o tecido é fotografada em uma resolução de 7 nm / pixel e fatias de 70 nm. Isso foi suficiente para resolver e distinguir vesículas secretoras centrais grandes densa de outras organelas. Em células enteroendócrinas, este tipo de vesículas varia entre 100 e 150 nm de diâmetro 13. O protocolo é como se segue:
6. As secções de tecido de coloração
- Remover o tecido de PBS e lavar três vezes, 5 min cada, em tampão cacodilato 0,1 M.
- Blocos Mancha para ácido tânico 1 hora em 0,1% dissolvido em tampão cacodilato 0,1 M para aumentar o contraste das membranas celulares 14.
- Levar a cabo a coloração e a desidratação do tecido subsequente de acordo com o protocolo publicado a 11. Seguindo Deerinck et ai. protocolo, preparar as seguintes soluções: 1) 1% (w / v) thiocarbonhydrazide (THC); 2) levar solução de aspartato; 3) acetato de uranilo a 1%, e 4) de ferrocianeto de ósmio tetraoxyde / potássio (OsO4 / K Ferrocianeto). Misturar 4% OsO4 e 2xFerrocianeto de estoque K [K Ferrocianeto de 0,3 g e 0,86 g de Na em cacodilato de 10 mL de H2O] em proporção 1: 1 para fazer OsO4 / K Ferrocianeto. Nota: Tendo o tecido embebido em agarose facilita o manuseamento durante a coloração. Remova cuidadosamente agarose para posterior infiltração de resina.
- Lavar as amostras três vezes, 5 min cada, em tampão de cacodilato 0,1 M e em seguida são coradas com solução de OsO4 / K Ferrocianeto durante 2 horas a 4 ° C.
- Enxágüe em água ultra pura três vezes, 5 min cada, e mancha com TCH durante 30 minutos a 60 ° C.
- Lavar em água ultra-pura, três vezes, 5 minutos cada e mancha com 2% de OsO4 (sem K Ferrocianeto) durante 60 min à TA. Enxágüe novamente em água ultra-pura três vezes, 5 min cada.
- Mancha com acetato de uranilo 1% O / N a 4 ° C. Enxágüe em água ultra-pura três vezes, 5 min cada.
- Mancha com aspartato chumbo durante 30 min a 60 ° C. Enxágüe em água ultra-pura três vezes, 5 min cada.
- Desidratar tecidos por lavagem em solutions com concentrações crescentes de etanol. Lavar 2 vezes, 5 minutos cada, com 50%, 75%, 85%, 95%, e etanol absoluto 100%. No final, lavar com secções de óxido de propileno duas vezes, 10 min cada.
7. infiltrando e incorporação de cortes de tecido em Resina
- Infiltrar os blocos com resina utilizando o kit comercialmente disponíveis atualmente. Incorporar tecidos em resina usando um kit epon incorporação. Prepara-se uma mistura de resina de epóxi de 48%, 20% DDSA ml, 30% NMA ml, e 2% DMP30. Agita-se mistura resina-se vigorosamente durante 5 min e, em seguida, colocar mistura em vácuo durante 30 min para permitir que as bolhas de vir à superfície.
- Misture resina com óxido de propileno a proporção 1: 1 e agitar vigorosamente. Remover o enxaguamento final, a desidratação a partir de tecidos, e adicionar resina misturada com óxido de propileno. Coloque o frasco com amostras em um rotador O / N e deixar frasco destapado. No dia seguinte, adicione resina epon feitos na hora e misture por 90 min em rotador.
- Incorporar em blocos de resina mais plano possível por imprensando-los ementre lâminas de vidro que foram curados com agente de libertação de líquido para evitar que grudem uns aos outros 15.
- Uma vez que os blocos são incorporados plana, curar os blocos para uma adição de 48 h a 60 ° C.
- Puxe lâminas além de liberar os blocos.
8. Montagem e aparar o bloco de tecido for Imaging
- De acordo com um escopo de dissecação, corresponder à orientação dos blocos de tecido embebidos em resina com o das micrografias confocal para facilitar a identificação de regiões que contêm as células de interesse antes de cortar o bloco.
- Apare o bloco de resina incorporado manualmente para baixo para um bloco mm-face ~ 500 x 500.
- Montar o bloco plano sobre um pino contendo epóxi condutora e seco durante 30 min. Em seguida, coloque o bloco sobre uma superfície plana e seca O / N a 60 ° C.
- Brasão do bloco com líquido prata coloidal. Manter a cortes de tecido plano para garantir o corte do bloco em um ângulo direito para facilitar a correlating os seriais de bloco-face e confocal micrografias.
9. SBEM
- Imagem do bloco usando um Microscópio Eletrônico de Varredura equipado com um sistema SBEM (por ex., 3Ver).
- Definir secção espessura inicial no ~ 2 uM incrementos e corte até que a face do tecido emerge do bloco.
- Adquirir uma imagem Surver de toda a face do bloco.
- Usando o software de Fiji, fazer medições, tanto do bloco-face e confocal de série micrografias para gerar um denominador comum e conta para amostra deformação durante a fixação e coloração 16.
- Localizar a área de interesse sobre a face do bloco através da multiplicação do denominador para as coordenadas nas imagens confocais. Use também outras características estruturais, tais como a posição das microvilosidades, células caliciformes, ou lâmina própria, como uma referência.
- Região da imagem de juros a uma 2.25 kV e 7 nm / pixel (ou ampliação 15,147X) no modo de alto vácuo. Incrementos fatia deve ser definidoa 70 nm ou menos.
- Coletar dados SBEM em formato .dm3 16-bit cru.
10. Otimização SBEM Imagens para a superfície Segmentação
- Converter imagens de formato SBEM .dm3 bruto para 8-bit .tiff.
- Filtro .tiff imagens usando um filtro de 0,8 Gaussian Blur em Fiji.
- Reduza o conjunto de dados a 25% do tamanho original e salvá-la como uma pilha .tiff para minimizar a quantidade de memória necessária para lidar com o conjunto de RAM.
- Alinhe a pilha de imagens SBEM usando o plug-in Fiji "alinhamento pilha linear com SIFT" em modo de tradução e cortada usando o plugin "cultura 3D". Nota: O corte ajuda a reduzir ainda mais a quantidade de memória RAM necessária para segmentar e renderização de volume.
Renderização de dados em 3D
11. Microscopia Confocal
- Reconstruir z-stacks usando o modo da ferramenta "superfícies" a superar automática. Figura 1D, mostra uma reconstrução de cada canal usando a opção suave em, o detalhe área superfícies em 0,126 mm, e de limiares de intensidade absoluta.
12. Serial SEM Bloco-face
Nota: segmentação de dados manual é um processo muito demorado e, dependendo dos recursos a serem prestados, o procedimento pode levar várias semanas. Segmentação para os vídeos e números apresentados em Bohórquez et al 2014 8. Era feito manualmente e levou cerca de 500 horas de trabalho. Recomenda-se a priorizar as características essenciais que necessitam de renderização antes de iniciar o processo de segmentação. O procedimento é o seguinte:
- Use a ferramenta "superfícies" no modo de tração ea opção "contorno" no modo de 50 ms chegando ao segmento manualmente eo volume rendeu a célula de interesse.
- Traçar contornos em cada fatia da célula para renderização mais suave ou a cada 5 fatias para processamento mais rápido.
- Exportação im definitivaas idades usando a função "snapshot" com uma resolução de 300 dpi ou mais.
Resultados
O método aqui descrito foi usado para estudar a ultraestrutura de uma célula específica dentro de uma camada de epitélio. A célula de interesse, neste caso, é a célula enteroend�rina elusiva. A sua identificação in situ foi possível apenas nos últimos anos com o desenvolvimento de ratinhos fluorescência repórter específicos de células. Em 2011, foi desenvolvido um rato em que o promotor do hormônio PYY dirige a expressão da proteína verde fluorescente 17. Neste PyyGFP rato, células enteroendócrinas da parte distal do intestino delgado e cólon são facilmente distinguidos sob luz UV. Este modelo do rato foi uma base para identificar um bloco de tecido contendo uma célula enteroend�rina e processá-lo para SBEM. O método de correlação é referido aqui como cocem3D e foi construído em protocolos previamente publicados 10,11.
Optimizar as dimensões do bloco de tecido é crítica para a correlação. Em experiências preliminares,determinou-se que num bloco de tecido 300 um de comprimento x 50 mm de espessura o número de imagens SBEM é reduzida a um mínimo, enquanto que cobre todo o corpo da célula de interesse. A Figura 1A mostra uma vista do dissecção usando uma lâmina vibrante para microtrome obter o bloco de tecido com as dimensões corretas. Pelo menos 100 blocos são obtidos antes de classificar através deles para escolher aqueles com células intactas de interesse. Blocos selecionados são visualizados por microscopia confocal e de imagem z-stacks são espaçados opticamente cada 1 mm (Figura 1B e C). A Figura 1D mostra um volume vista renderizada de uma célula enteroend�rina no íleo do rato. A sua neuropod proeminente se estende por baixo de células epiteliais para ~ 60 mm.
A célula de interesse é encontrada ao correlacionar os confocal z-stacks uma imagem SBEM de toda a face do bloco com. Um exemplo é apresentado aqui de um bloco a partir do cólon distal de um PyyGFPrato. Após a obtenção confocal z pilhas (Figura 2A), o bloco é incorporado em uma fina camada de agarose de baixo ponto de fusão (Figura 2B). Isto é necessário para subsequente manipulação do bloco. É importante para manter a orientação do bloco tão plano quanto possível combinar as fatias ópticos com as fatias SBEM. Na Figura 2C, uma imagem representativa é mostrada de toda a face do bloco de tecido. Partes desta figura foram anteriormente publicados em Bohorquez et al., 2014 8. Este foi posteriormente correlacionado com a imagem confocal, tendo em conta os pontos de referência do tecido e às dimensões. A Figura 2D mostra uma sobreposição da imagem SBEM confocal e do bloco, revelando o localização precisa da nossa célula de interesse.
Uma vez que a célula tenha sido identificado, a imagem latente SBEM é feito com uma resolução suficientemente alta para identificar vesículas secretoras e outros organelos celulares. O conjunto de dados apresentados,re foi fotografada em 7 nm por pixel como imagens do bloco foram tomadas todas as 70 nm. O conjunto de dados contidos imagens 643 que se estendem por 45 um de profundidade do tecido. A poucos microns são raspadas durante o exame inicial de toda a face do bloco a baixa ampliação. O conjunto de dados SBEM-prima foi adquirido em formato .dm3 e transformado para .tiff para o tratamento subsequente. A pilha SBEM foi cortada usando o software Fiji plugin "safra (3D)" bloco de forma a conter apenas a região de interesse. Isto reduz a quantidade de memória RAM necessária para renderização de volume. A célula foi identificado pela sua posição e vesículas secretoras, e segmentado utilizando software Imaris. A Figura 3 contém uma representação em 3D da célula enteroend�rina.
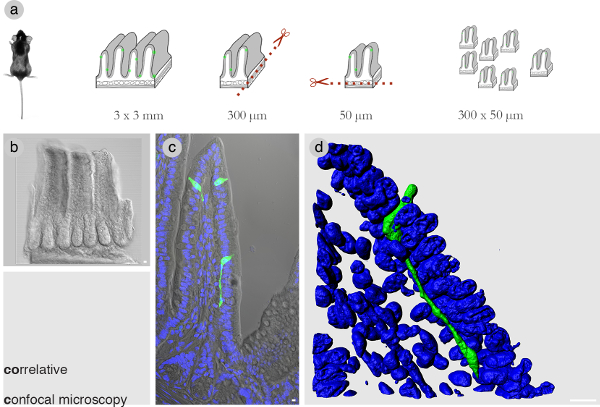
Figura 1:. Microscopia confocal Correlative (A) Fluxo de trabalho de dissecar um pedaço detecido intestinal para bloquear um tecido de 300 x 50 mm de espessura. (B) Um bloco de as dimensões corretas tecido representativo. Note-se que um bloco de tecido destas dimensões do intestino delgado do rato mede cerca de 3 vilosidades ou, no caso do cólon cerca de 8 criptas. (C) Uma das vilosidades do intestino delgado, contendo uma célula de interesse. (D) A confocal z-stack rendido em 3D usando software Imaris mostra uma célula enteroend�rina com um neuropod proeminente corrida sob o epitélio intestinal. Azul = DAPI mancha nuclear. Barras = 10 mm. Por favor clique aqui para ver uma versão maior desta figura.
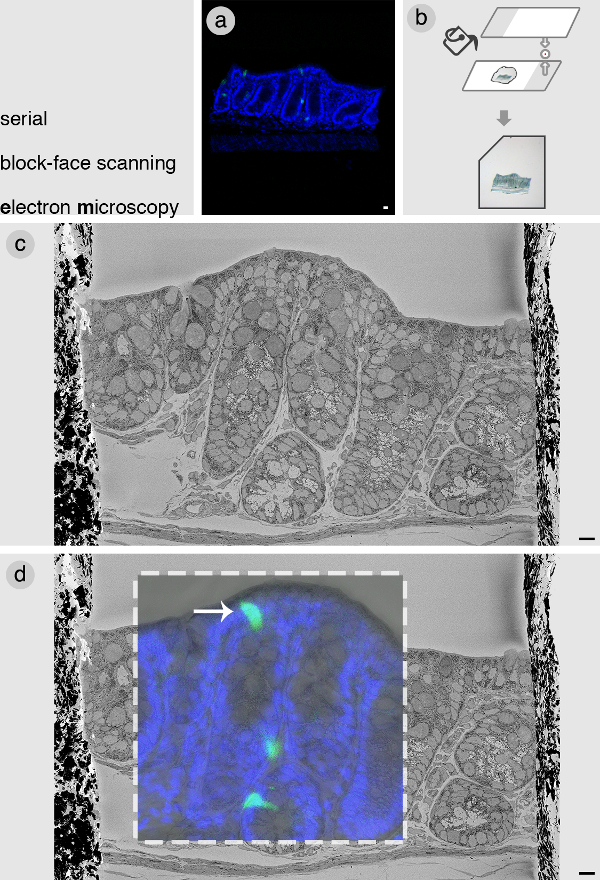
Figura 2: microscopia Serial bloco-face eletrônica de varredura (A) imagem confocal de uma sele.bloco de tecido CTED a partir do cólon contendo células PyyGFP enteroend�rina (verde) de interesse. (B) Uma vez que o bloco é fotografada por microscopia confocal, é então incorporado apartamento em baixo ponto de fusão agarose usando lâminas de vidro. Incorporando o bloco de tecido em agarose facilita a manipulação subseqüente, eo entalhe no canto esquerdo ajuda a montar o bloco dentro da SBEM na orientação correta. (C) SBEM imagem da face do bloco. (D) A correlação de dados confocal com SBEM para identificar células de interesse (seta branca). São modificados Imagens em c e d desta figura de Bohorquez et al., 2014 8. Azul = DAPI mancha nuclear. Barras = 10 mm. Por favor clique aqui para ver uma versão maior desta figura.
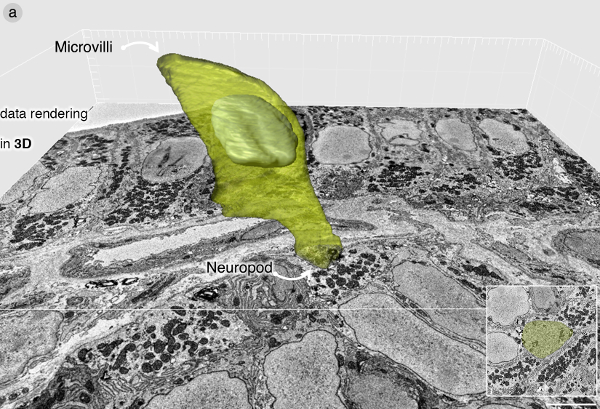
Figura 3:. Renderização de dados em 3D (A) a prestação Volume de célula enteroend�rina usando software Imaris. A célula contém um neuropod embalado com vesículas secretoras. Para uma descrição completa da ultra-estrutura de uma célula enteroend�rina, consulte Bohórquez et al., 2014 8. Por favor clique aqui para ver uma versão maior desta figura.
Discussão
Chemosensation gastrointestinal está a emergir como um novo campo emocionante na pesquisa biomédica. Isto é, em grande parte devido à descoberta de receptores gustativos funcionais em células enteroendócrinas 18. Estudos subsequentes têm mostrado que as células enteroendócrinas expressam receptores específicos para nutrientes, incluindo hidratos de carbono, lípidos, aminoácidos e 5,6,19,20. O factor catalítica para estas descobertas tem sido o desenvolvimento de ratinhos repórter, em que as células são enteroendócrinas marcado por fluorescência 20. Desenvolvemos um destes ratinhos, o rato PyyGFP, para estudar enteroendócrinas células do intestino delgado e cólon distai 17,21. Estas células são de interesse porque eles segregam PYY e glucagon-like peptide 1, ambos os quais são indutores da saciedade 22,23. Na época, uma conta de ultra-estrutural completa destas células era ausente, que acreditava que era essencial para compreender os seus mecanismos de sinalização.
ontent "> Aqui, descrevemos um visualmente um método para colmatar microscopia confocal com SBEM. O método é conhecido como Cocem3D, o que nos permitiu documentar a ultra-estrutura completa das células enteroendócrinas. Temos informou que estas células têm uma neuropod que contém três quartos de todas as vesículas secretoras 8. Dentro do neuropod, há neurofilaments e muito parecido com axônios neuronais, neuropods são alimentados por células gliais entéricas 8. Mais importante, é que estes neuropods que enteroendócrinas células conectar fisicamente aos neurônios que inervam o intestino e 9 cólon.Um dos pontos fortes do cocem3D é a sua simplicidade. A redução da amostra de tecido em um bloco que pode ser trabalhada por SBEM confocal e facilita a identificação de uma célula específica dentro de um tecido. Vesículas secretoras e outras organelas pode ser identificado com facilidade em uma resolução de 7 nm / pixel. Porque as seções em SBEM são descartados, outros procescantar de tecidos para identificar proteínas específicas não é uma opção neste momento. No entanto, o desenvolvimento de métodos, tais como ATUM 24, em que secções de tecido do bloco são preservados, são susceptíveis de permitir a identificação de proteínas específicas na célula. Determinação do local específico de receptores quimiossensoriais em células enteroendócrinas é informação essencial para o desenvolvimento de terapias com drogas para obesidade, porque as células enteroendócrinas são uma interface entre sensorial alimentos no intestino e saciedade no cérebro.
Divulgações
Dr. Satish Medicetty é um empregado de e recebe 100% do salário Renovo Neural Inc; no entanto, isso não altera a adesão do autor de políticas JoVE sobre a partilha de dados e materiais. Não há conflito de interesse é declarada para autores restantes.
Agradecimentos
Our sincere appreciation is expressed to the following people: Drs. Sam Johnson and Benjamin Carlson of the Duke Light Microscopy Core Facility for their assistance with data visualization software, and Ms. Valerie Lapham and Dr. John M. Mackenzie, Jr. of the Center for Electron Microscopy at North Carolina State University for their advice on electron microscopy. We thank Dr. Elaine B. Bohórquez for her editorial assistance. Authors contributed in the following manner: DVB, SM, and RAL designed experiments and analyzed data. DVB performed experiments and FH performed manual rendering of data. SM is director of Renovo Neural, where SBEM data was acquired. DVB wrote the manuscript and all authors reviewed and edited the final manuscript. This work was supported by NIH grants R01DK091946 and Veterans Affairs grant I01BX002230 to RAL, and F32DK094704, to DVB.
Materiais
| Name | Company | Catalog Number | Comments |
| Phosphate buffered saline | Life technologies | 10010023 | |
| Heparin sodium salt | Sigma | H4784 | |
| Paraformaldehyde | Sigma | 158127 | 4%, freshly made in PBS, final pH 7.4 |
| Glutaraldehyde | Sigma | G5882 | |
| Dental wax | Electron Microscopy Sciences | 72660 | |
| Low-melting agarose | Life technologies | 16520-100 | 5%, freshly made in PBS |
| Standard Tissue-Tek Cryomold | Electron Microscopy Sciences | 62534-25 | |
| DAPI nuclear stain | Life technologies | D1306 | |
| Postively charged glass slides and coverslips | |||
| Fine art paintbrush #1 | |||
| Cacodylate buffer | Electron Microscopy Sciences | 11650 | |
| Tannic acid | Electron Microscopy Sciences | 21700 | |
| EMbed 812 kit | Electron Microscopy Sciences | 14120 | |
| Liquid releasing agent | Electron Microscopy Sciences | 70880 | |
| Liquid silver colloidal | Electron Microscopy Sciences | 12630 | |
| CircuitWorks conductive epoxy | ITW Chemtronics | CW2400 | |
| Variable flow peristaltic pump | VWR | 70730-064 | |
| VT1200S Vibrating blade microtome | Leica | ||
| Zeiss 780i confocal microscope | Carl Zeiss | ||
| Sigma VP Scanning Electron Microscope | Carl Zeiss | ||
| 3view system | Gatan | ||
| Renovo Neural Inc (Cleveland, OH) | http://www.renovoneural.com | Renovo provides 3d EM services | |
| Fiji software | Open access software | ||
| Computer station with 16 GB of RAM or more | |||
| Data visualization software Imaris 7.5 | Bitplane |
Referências
- White, J. G., Southgate, E., Thomson, J. N., Brenner, S. The structure of the nervous system of the nematode Caenorhabditis elegans. Philosophical transactions of the Royal Society of London. Series B, Biological. 314, 1-340 (1986).
- Smith, C. L., et al. Novel cell types, neurosecretory cells, and body plan of the early-diverging metazoan Trichoplax adhaerens. Current biology : CB. 24, 1565-1572 (2014).
- Denk, W., Horstmann, H. Serial block-face scanning electron microscopy to reconstruct three-dimensional tissue nanostructure. PLoS biology. 2, e329 (2004).
- Engelstoft, M. S., et al. Seven transmembrane G protein-coupled receptor repertoire of gastric ghrelin cells. Molecular metabolism. 2, 376-392 (2013).
- Chandra, R., et al. Immunoglobulin-like domain containing receptor 1 mediates fat-stimulated cholecystokinin secretion. The Journal of clinical investigation. 123, 3343-3352 (2013).
- Wang, Y., et al. Amino acids stimulate cholecystokinin release through the Ca2+-sensing receptor. Am J Physiol Gastrointest Liver Physiol. 300, G528-G537 (2011).
- Rogers, G. J., et al. Electrical activity-triggered glucagon-like peptide-1 secretion from primary murine L-cells. The Journal of physiology. 589, 1081-1093 (2011).
- Bohórquez, D. V., et al. An enteroendocrine cell-enteric glia connection revealed by 3D electron microscopy. PloS one. 9, e89881 (2014).
- Bohórquez, D. V., et al. Neuroepithelial circuit formed by innervation of sensory enteroendocrine cells. The Journal of clinical investigation. , (2015).
- Knott, G. W., Holtmaat, A., Trachtenberg, J. T., Svoboda, K., Welker, E. A protocol for preparing GFP-labeled neurons previously imaged in vivo and in slice preparations for light and electron microscopic analysis. Nature protocols. 4, 1145-1156 (2009).
- Deerinck, T., Bushong, E., Thor, A., Ellisman, M. . NCMIR methods for 3D EM: a new protocol for preparation of biological specimens for serial block face scanning electron microscopy. , (2010).
- Bohórquez, D. V., Chandra, R., Samsa, L. A., Vigna, S. R., Liddle, R. A. Characterization of basal pseudopod-like processes in ileal and colonic PYY cells. Journal of molecular histology. 42, 3-13 (2011).
- Nilsson, O., et al. Distribution and immunocytochemical colocalization of peptide YY and enteroglucagon in endocrine cells of the rabbit colon. Endocrinology. 129, 139-148 (1991).
- Mizuhira, V., Futaesaku, Y. New fixation method for biological membranes using tannic acids. Acta Histochem Cytochem. 5, 233-236 (1972).
- Reymond, O. L., Pickett-Heaps, J. D. A routine flat embedding method for electron microscopy of microorganisms allowing selection and precisely orientated sectioning of single cells by light microscopy. Journal of microscopy. 130, 79-84 (1983).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nature. 9, 676-682 (2012).
- Bohórquez, D., Chandra, R., Samsa, L., Vigna, S., Liddle, R. Characterization of basal pseudopod-like processes in ileal and colonic PYY cells. J Mol Histol. 42, 3-13 (2011).
- Jang, H. J., et al. Gut-expressed gustducin and taste receptors regulate secretion of glucagon-like peptide-1. Proceedings of the National Academy of Sciences of the United States of America. 104, 15069-15074 (2007).
- Liou, A. P., et al. The G-protein-coupled receptor GPR40 directly mediates long-chain fatty acid-induced secretion of cholecystokinin. Gastroenterology. 140, 903-912 (2011).
- Reimann, F., et al. Glucose sensing in L cells: a primary cell study. Cell metabolism. 8, 532-539 (2008).
- Bohorquez, D. V., Liddle, R. A. Axon-like basal processes in enteroendocrine cells: characteristics and potential targets. Clinical and translational science. 4, 387-391 (2011).
- Batterham, R. L., et al. Gut hormone PYY(3-36) physiologically inhibits food intake. Nature. 418, 650-654 (2002).
- Flint, A., Raben, A., Astrup, A., Holst, J. J. Glucagon-like peptide 1 promotes satiety and suppresses energy intake in humans. The Journal of clinical investigation. 101, 515-520 (1998).
- Hayworth, K. J., et al. Imaging ATUM ultrathin section libraries with WaferMapper: a multi-scale approach to EM reconstruction of neural circuits. Frontiers in neural circuits. 8, 68 (2014).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados