Method Article
Korrelat konfokale und 3D-Elektronenmikroskopie eines spezifischen Sinneszelle
In diesem Artikel
Zusammenfassung
Here, we introduce a method, cocem3D, to unveil the ultrastructure of a specific cell in its native tissue by bridging confocal and serial block-face scanning electron microscopy.
Zusammenfassung
Abgrenzung der Ultrastruktur der Zelle ist für das Verständnis der Funktion. Dies kann eine schwierige Projekt für seltene Zelltypen im ganzen Gewebe der verschiedenen Zelltypen, wie enteroendokrinen Zellen des Darmepithels gemacht diffundieren. Diese gastrointestinalen Sensoren von Lebensmitteln und Bakterien waren schwierig zu untersuchen, da sie unter anderen epithelialen Zellen in einem Verhältnis von 1 dispergiert sind: 1,000. Kürzlich transgene Reportermäusen erzeugt wurden, um Zellen zu identifizieren enteroendokrinen mittels Fluoreszenz. Eines davon ist das Peptid YY-GFP-Maus. Mit dieser Maus, ein Verfahren zur konfokalen und Serienblock-face Rasterelektronenmikroskopie korrelieren entwickelten wir. Wir nannten das Verfahren cocem3D und wendete sie auf eine bestimmte enteroendokrinen Zelle im Gewebe erkennen und zu enthüllen, Ultrastruktur der Zelle in 3D. Die Auflösung cocem3D ist ausreichend, um Organellen für Volumenwieder identifizieren so klein wie sekretorischen Vesikeln und zu unterscheiden, Zellmembranenndering. Cocem3D leicht angepasst, um die 3D-Ultrastruktur von anderen spezifischen Zelltypen in ihrer Muttergewebe zu untersuchen.
Einleitung
Das Leben in einer Zelle stattfindet, in Zeit und Raum. Veränderungen über die Zeit oft mit Zeitraffer-Mikroskopie eine bildgebende Verfahren, wie Super-Resolution-Mikroskopie untersucht. Raum, insbesondere die Anordnung von Organellen innerhalb einer Zelle oder Zell-zu-Zell-Wechselwirkungen, kann nur durch eine vollständige Darstellung der Feinstruktur der Zelle abgeleitet werden. Eine umfassende Darstellung der Feinstruktur einer Zelle kann auch Klarheit, um genomische Funktion bringen in Fällen, in denen das Genom vorhanden ist, wie die C. elegans Nematoden-1 oder die flache Placozoa tricoplax adherens 2. Serienschnittelektronenmikroskopie ist nun eine reproduzierbare, zeiteffizient und weniger kostspielige Aufgabe dank der Entwicklung von automatischen 3D Elektronenmikroskopie Technologien wie serielle Block-face Rasterelektronenmikroskop 3 (SBEM).
Der Bedarf an Strukturinformationen zur Funktion aufzuklären ist sehr offensichtlich in bestimmten cell Typen, bei denen Funktion hängt von physikalischen Zellen-Zellen-Wechselwirkungen, wie Neuronen, Gliazellen oder sensorischen Epithelzellen. Wir sind besonders daran interessiert, bei der Aufklärung, wie sensorische Signale von Nährstoffen in das Lumen des Darms werden in ein elektrisches Signal, das letztlich moduliert appetitive Verhaltensweisen transduziert. Der Schaltkreis ist komplex, aber beginnt an der Darmwand, wo Nährstoffe in Kontakt mit sensorischen Epithelzellen, genannt enteroendokrinen Zellen kommen. Im Gegensatz zu anderen sensorischen Epithelzellen, wie Geschmackszellen werden enteroendokrinen Zellen im ganzen Darmepithel in einem Verhältnis von eins bis eintausend 4-7 dispergiert. Folglich waren sie schwer zu identifizieren und zu untersuchen, und für lange Zeit wurden sie nur als eine Quelle von Darmhormone angesehen. Aber mit der Entwicklung zellspezifischer Fluoreszenzreportermäusen, die komplexe sensorische Funktion dieser Zellen entsteht. Mit einer der folgenden Reporter Mäusen ein Peptid YY-GFP (PyyGFP) Maus, fanden wir, dass enteroendocrine Zellen eine herausragende cytoplasmatischen Schwanz, die wir mit dem Namen neuropod. Das Aussehen der neuropods vorgeschlagen eine konservierte Funktion von Zelle-zu-Zell-Kommunikation. Somit begründet, daß wir durch die Dokumentation der Ultrastruktur eine enteroendokrine Zelle, deren Funktion neuropods abgeleitet werden konnte.
Die Notwendigkeit, die Struktur von einem Fortsatz in einer Zelle verteilt, die schwer zu erkennen, ist zu verstehen war der Hauptgrund für die Entwicklung eines Verfahrens zur konfokalen Mikroskopie und SBEM kombinieren. Die Zelle von Interesse wurde mit PyyGFP enteroendokrinen zellspezifische Reporter Mäusen identifiziert. Das Verfahren erlaubt es uns, die gesamte Ultra eines enteroendokrine Zelle und ihrer neuropod dokumentieren. Innerhalb neuropods fanden wir strukturellen Merkmale neuronalen Axonen und außerhalb neuropods, fanden wir eine physikalische Beziehung zu ente Glia 8. Tatsächlich enthalten neuropods etwa 70% aller sekretorischen Vesikeln vermutlich eine wesentliche Rolle bei der sekretorischen Funktion dieser Zellen. Aufbauend auf dieStrukturdaten, in jüngerer Zeit fanden wir, dass durch diese neuropods, enteroendokrinen Zellen und Neuronen innerviert den Darm bilden eine neuroepitheliale Schaltung, ähnlich der von Geschmackszellen in der Zunge 8,9.
Aufdeckung solcher Merkmale von Magen-Darm chemosensory Mechanismen ergab sich aus Strukturdaten gesammelt mit diesen korrelative Mikroskopie-Methode. Wir glauben, dass diese Methode auch in anderen Gebieten der Zellbiologie, der besonders bei der Zellen im Gewebe zu einem sehr geringen Verhältnis verteilt werden. Wir bezogen auf die Verfahren nach Korrelat konfokalen und Serienblockfläche Rasterelektronenmikroskopie in 3D (Cocem3D). Das Verfahren besteht aus den folgenden Hauptschritte umfasst: Gewebedissektion, Konfokalmikroskopie SBEM Bildgebung SBEM und konfokaler Bildkorrelation und manuelle Segmentierung. Im Vergleich zu anderen korrelative Verfahren ist das Konzept nicht einfach, weil die Korrelation physikalischen anstatt Chemikalie.
Protokoll
Alle Tierpflege und Experimente wurden in Übereinstimmung mit einem Protokoll, das von der Duke University Institutional Animal Care und Verwenden Ausschuss genehmigt wurde.
Cocem3D stammten aus einer Kombination der zuvor veröffentlichten Protokollen 10,11 und wurde hier angewendet, um eine bestimmte enteroendokrine Zelle unter Verwendung eines Reporter PyyGFP Maus 12 zu studieren. Dieses Verfahren kann leicht auf andere Zellen von Interesse unter Verwendung von kommerziell erhältlichen transgenen Mäuse Reportergen angewendet werden. Correlative Konfokalmikroskopie serielle Block-face Rasterelektronenmikroskopie (SBEM) und Datendarstellung in 3D: Das Verfahren wird in drei Abschnitten beschrieben.
Korrelat konfokale Mikroskopie
Das Ziel dieses Abschnitts ist es, eine Gewebesegments aus dem distalen Kolon von Dimensionen, die korrelative konfokale Bildgebung SBEM ermöglichen ernten. Das Protokoll ist wie folgt:
1. Ernten der Tissue
- Bereiten10 ml von 50 ug / ml Heparinlösung in PBS.
- Vorbereitung Xylazin / Ketamin Narkose Lager durch Mischen von 10 ml Ketamin (100 mg / ml) und 1 ml Xylazin (20 mg / ml).
- Verdünnen Xylazin / Ketamin Lager 1: 4 in 0,1 M PBS.
- Bereiten Sie eine 100 ml Fixierlösung mit 4% Paraformaldehyd und 0,1% Glutaraldehyd in PBS.
- Betäuben eine PyyGFP Maus, 6 bis 10 Wochen alt, mit einer letalen Dosis von Xylazin / Ketamin-Narkose. Spritzen Sie die Narkose intraperitoneal mit 0,15 ml pro 20 g Mausgewicht.
- Bestätigen Sie die richtige Maus Betäubung durch Einklemmen des Schwanzes oder Zehen, und verwenden Salbe auf die Augen bis zur Trockenheit zu verhindern.
- Verwenden Sie eine variable Strömungs peristaltische Pumpe zu Fixiermittel intrakardial 10 durchströmen. Verwendung chirurgische Scheren (13 cm Länge) geschnitten öffnen Sie die Bauchhöhle, den Darm, Herz und Lunge aus. Halten Sie Herz mit Schmal Muster gebogenen Pinzette (12 cm Länge), und setzen Schmetterlingsnadel (19 G) aus Schlauchpumpe into linken Ventrikels. Unmittelbar, schneiden rechten Vorhof mit Feder geraden Schere (Länge 4 mm). Perfusion mit einer Geschwindigkeit von 2 ml / min zunächst mit Heparin-Lösung für 1 min, und dann mit eiskaltem Fixierungslösung für 15 Minuten, bis der Maus den Schwanz ist völlig starr.
Anmerkung: Es ist sehr wichtig, um bei einer langsamen Rate zum Bersten der kleinen Gefäße in der Darmschleimhaut verhindert perfundieren. Platzen der Schiffe wird Ultrastruktur des Gewebes beeinträchtigen. Wenn Perfusion angemessen ist, die Leber sollte blass in der Farbe innerhalb 3-5 min drehen. - Mit Hilfe einer kleinen Schere öffnen Sie die Bauchhöhle und der Verbrauch den gesamten Dickdarm von der Kreuzung mit der Blinddarm zum distalen Rektum. Legen Sie das Gewebe in eiskaltem PBS. Während in PBS eingetaucht, aufgeschnitten mit kleinen Feder Schere den Doppelpunkt entlang der Mesenterium.
2. Präparieren und Befestigungsgewebesegmente
- Mit einem Skalpell geschnitten etwa 6 kleine Gewebesegmente (2 mm 2) aus dem distalen Kolon. Tun Sie dies auf einem Blatt of Dentalwachs und in einigen Tropfen PBS eingetaucht.
- Post-fix die Gewebesegmente in Fixierungslösung für 3 h bei 4 ° C.
3. Micro-Sezieren Gewebeblöcke
- Bereiten Sie 5% niedrig schmelzenden Agarose in PBS und halten Sie sie in einem Wasserbad bei 45 ° C.
- Die Gewebesegmente einbetten in 5% niedrig schmelzende Agarose mit einem kleinen Kunststoffbehälter, wie die Standard-Größe Tissue-Tek Cryomold.
- Montieren Sie die eingebetteten Abschnitte auf einer vibrierenden Klinge Mikrotom und füllen Sie die Pufferwanne mit eiskaltem PBS.
- Cut 300 um Gewebestreifen bei 0,8 Amplitude und 0,04 mm / s Geschwindigkeit. Hinweis: Zu diesem Zeitpunkt werden die Gewebestreifen aus der Agarose verdrängt werden.
- Re-Einbettung 300 um Gewebestreifen in Agarose und montieren sie am Mikrotom senkrecht zur Vibratom Klinge.
- Mit einem Vibratom geschnitten Gewebestreifen in einer Dicke von 50 um. Hinweis: Die endgültige Gewebeblöcke sollten etwa 300 & mgr; m breit x 50 & mgr; m dick sein. Diese dbmessungen von Pilot konfokalen Experimente, die zeigten, dass die Dicke der enteroendokrinen Zelle zwischen 15-20 um und die neuropod der Länge von 30 bis 70 nm optimiert. Daher könnte eine gesamte Zelle in einem Gewebeblock 300 um Breite und 50 um Dicke enthalten sein, wenn die Ausrichtung der Zelle parallel zu der Fläche der Gewebeblock ist. Variieren diese Dimensionen je nach Gewebe und Zelltyp von Interesse.
- Die Gewebeblöcke in PBS bei 4 ° C.
4. Die konfokale Imaging
- Bereiten Sie eine Kernfärbung DAPI-Lösung durch Verdünnung in PBS bei 1: 4.000.
- Inkubieren Gewebeblöcke in DAPI Kernfärbung für 5 min. Anmerkung: DAPI-Färbung erleichtert Scheidungs einzelnen Zellen durch ihre Kerne, die hilfreich zum Korrelieren der konfokale SBEM Bildern ist.
- Berg Gewebeblöcke auf geladene Objektträger aus Glas, ein paar Tropfen von PBS, und decken Sie sie mit einem Deckglas.
- Mit einem konfokalen Mikroskop identifizieren Blocks mit intakter Villi und PyyGFP Zellen von Interesse und bebildert sie z-Stacks optische Schnitte zu erhalten.
- Verwenden Sie eine 20-fach / 0,8 Zeiss Plan Apochromat Ziel, z-Stacks von 1 um optische Schnitte zu erhalten.
- Zur Z-Stapel, verwenden die Kanäle für 405 nm (DAPI), um die Beziehung zu anderen Zellen zu bestimmen, auf 488 nm endogenen GFP die Zelle von Interesse zu lokalisieren, und die Differential-Interferenz-Kontrast (DIC) an Stelle der Zelle in Bezug auf die Bestimmung Lumen.
- Verwenden Sie Bildauflösung von 1024 Pixel oder höher.
5. Embedding Blocks in Agarose
- Herstellung von 10 ml Fixiermittel, enthaltend 4% Paraformaldehyd und 2,5% Glutaraldehyd in PBS.
- Die Gewebeblöcke vom Glas-Objektträger zu entfernen durch vorsichtige Zugabe von PBS in den Kanten des Deckglases an das Deckglas ohne Beschädigung der Gewebeblock aus dem Glasträger zu gleiten. Verwenden Sie dann eine schöne Kunstmalerpinsel, um das Gewebe Block von der Folie, um eine 1 übertragen0,5 ml-Mikrozentrifugenröhrchen, enthaltend 4% Paraformaldehyd und 2,5% Glutaraldehyd Fixativ.
- Post-fix Gewebeblöcke O / N bei 4 ° C.
- Übertragen Gewebeblöcke auf PBS. Dann einbetten Blöcke flach in 5% niedrig schmelzende Agarose durch sandwich sie zwischen zwei Glasobjektträger.
Hinweis: Einbetten der Blöcke in einer dünnen Schicht von Agarose erleichtert posterior Manipulation während der Färbung. - Schneiden Sie die Agarose in einem Quadrat und eine Kerbe auf der oberen Seite. Anmerkung: Dieser Schritt ist notwendig, um die Ausrichtung in den folgenden Schritten erhalten.
- Speicher Blöcke in einem 1,5 ml Mikrozentrifugenröhrchen mit PBS bei 4 ° C bis zur Weiterverarbeitung.
Serienblock Gesicht Rasterelektronenmikroskopie (SBEM)
In diesem Abschnitt wird die Gewebeblock hergestellt und mit SBEM abgebildet bei niedriger Vergrößerung. Das Übersichtsbild der Blockfläche wird dann mit den konfokalen Daten korreliert, um die Region, die die Zelle von Interesse zu identifizieren. Sobald die Regionidentifiziert ist, wird das Gewebe bei einer Auflösung von 7 nm / Pixel und Scheiben von 70 nm abgebildet. Dies war ausreichend, um zu beheben und zu unterscheiden groß dichten Kern sekretorischen Vesikeln aus anderen Organellen. In enteroendokrinen Zellen variiert diese Art von Vesikel zwischen 100 und 150 nm im Durchmesser, 13. Das Protokoll ist wie folgt:
6. Färbung Gewebeschnitten
- Entfernen Gewebe aus PBS und spülen dreimal jeweils 5 Minuten in 0,1 M Cacodylatpuffer.
- Fleckenblöcke für 1 h in 0,1% Gerbsäure in 0,1 M Cacodylat-Puffer gelöst, um den Kontrast von Zellmembranen 14 verbessern.
- Führen Sie nachfolgende Färbung und Austrocknung von Gewebe nach dem veröffentlichten Protokoll 11. Folgende Deerinck et al. Protokoll, bereiten diese Lösungen: 1) 1% (w / v) thiocarbonhydrazide (THC); 2) führen Aspartat-Lösung; 3) 1% Uranylacetat und 4) osmium tetraoxyde / Kaliumferrocyanid (OsO 4 / K Ferrocyanide). Mischen Sie 4% OsO 4 und 2xK Ferrocyanide stock [0,3 g K Ferrocyanide und 0,86 g Na Cacodylat in 10 ml H 2 O] im Verhältnis 1: 1 OsO 4 / K Ferrocyanide zu machen. Hinweis: Nachdem die in Agarose eingebetteten Gewebe erleichtert die Handhabung während der Färbung. Agarose für nachfolgende Harzinfiltration vorsichtig entfernen.
- Spülen Proben dreimal jeweils 5 Minuten in 0,1 M Cacodylatpuffer und dann Fleck mit OsO4 / K-Ferrocyanid Lösung für 2 Stunden bei 4 ° C.
- Spülen in Reinstwasser dreimal mit je 5 min, und Fleck mit TCH für 30 min bei 60 ° C.
- Spülen in Reinstwasser dreimal, jeweils 5 min und Fleck mit 2% OsO 4 (kein Ferrocyanid K) für 60 min bei RT. Wieder spülen in ultrareinem Wasser dreimal mit je 5 min.
- Fleck mit 1% Uranylacetat O / N bei 4 ° C. Spülen in Reinstwasser dreimal mit je 5 min.
- Fleck mit Blei Aspartat für 30 min bei 60 ° C. Spülen in Reinstwasser dreimal mit je 5 min.
- Dehydratisieren Gewebe durch Spülen in solutions mit steigenden Konzentrationen von Ethanol. Spülen 2 mal jeweils 5 Minuten mit 50%, 75%, 85%, 95% und 100% absoluten Ethanol. Am Ende gründlich Abschnitte mit Propylenoxid zweimal, jeweils 10 Minuten.
7. Als Spion and Embedding Gewebeschnitte in Resin
- Infiltrieren, die Blöcke mit Harz unter Verwendung der kommerziell avaialble kit. Betten Sie Gewebe in Harz mit einem EPON Einbettung kit. Vorbereitung einer Harzmischung aus 48% Epoxid, 20% ml DDSA, 30% NMA ml und 2% DMP30. Agitieren Harzgemisch 5 min kräftig und legen Sie dann mix im Vakuum für 30 Minuten, damit Luftblasen an die Oberfläche zu kommen.
- Mischen Sie Harz mit Propylenoxid bei 1: 1-Verhältnis und kräftig schütteln. Entfernen Sie die endgültige Austrocknung Spülen von Geweben und fügen Harz vermischt mit Propylenoxid. Platz Fläschchen mit Proben in einem Rotator O / N und lassen Fläschchen nicht begrenzten. Am nächsten Tag, fügen Sie frisch zubereitete EPON-Harz und Mischen für 90 min in Rotator.
- Betten Sie Blöcke in Harz so flach wie möglich durch Einfügen sie inzwischen Glasobjektträgern, die mit flüssigem Trennmittel ausgehärtet sind, um sie von aneinander kleben 15 verhindern.
- Sobald die Blöcke Flach eingebettet, heilen die Bausteine für eine zusätzlich 48 Stunden bei 60 ° C.
- Ziehen Sie gleitet auseinander, um die Blöcke zu lösen.
8. Montage und Trimmen des Tissue-Block für Imaging
- Unter einem Binokular, passen Sie die Ausrichtung der Gewebeblöcke in Harz mit dem der konfokalen mikroskopischen Aufnahmen eingebettet, um zu erleichtern die Identifizierung der Bereiche, die Zellen von Interesse enthält vor dem Trimmen den Block.
- Schneiden Sie das Harz eingebettet Block manuell bis zu einer ~ 500 x 500 & mgr; m-Block-face.
- Montieren Sie den Block flach auf einem Stift, die leitfähige Epoxidharz und trocken für 30 Minuten. Dann lag der Block flach auf einer Oberfläche und trocken O / N bei 60 ° C.
- Mantel der Block mit kolloidalem Silber Flüssigkeit. Pflegen Sie die Gewebeschnitte flach Schneiden des Blocks in einem rechten Winkel zu gewährleisten, um die Zusammenarbeit zu erleichtern,rrelating die Serienblock-face und konfokale Mikroaufnahmen.
9. SBEM
- Bild der Block mit einem Rasterelektronenmikroskop ausgestattet mit einem SBEM System (z .B., 3View).
- Set Ausgangsdicke Schnitt bei ~ 2 & mgr; m-Schritten und schneiden, bis das Gesicht des Gewebes ergibt sich aus dem Block.
- Erwerben Sie eine Surver Bild des gesamten Block Gesicht.
- Verwendung Fiji Software, Messungen sowohl die Serienblock-face und konfokale Mikroaufnahmen, einen gemeinsamen Nenner und machen Musterkettenschär während Fixierung und Färbung 16 zu erzeugen.
- Suchen Sie den Bereich von Interesse auf der Blockfläche durch Multiplikation der Nenner auf die Koordinaten in den konfokalen Bildern. Verwenden auch andere strukturelle Merkmale, wie die Position der Mikrovilli, Becherzellen oder der Lamina propria als Referenz.
- Bild Region von Interesse in eine 2,25 kV und 7 nm / Pixel (oder 15,147X Vergrößerung) in Hochvakuum-Modus. Slice-Schritten festgelegt werden solltenbei 70 nm oder weniger.
- Sammeln SBEM-Daten im 16-Bit-.dm3 Format.
10. Optimieren SBEM Images für Oberflächensegmentierung
- Konvertieren SBEM Bilder von rohen .dm3 Format in 8-Bit-TIFF.
- TIFF Filtern von Bildern mit einem 0,8 Gaußsche Unschärfe-Filter in Fidschi.
- Verkleinern Sie die Daten auf 25% der Originalgröße festgelegt und speichern Sie sie als TIFF-Stapel, die Menge an RAM-Speicher erforderlich, um den Satz zu behandeln minimieren.
- Richten Sie den Stapel SBEM Bilder mit der Fidschi-Plugin "linear Stapel Ausrichtung mit SIFT" im Übersetzungsmodus und beschnitten mit der "Ernte 3D" Plugin. Hinweis: Cropping hilft, die Menge an RAM-Speicher erforderlich weiter verringert für die Segmentierung und Volume-Rendering.
Daten Rendering in 3D
11. konfokale Mikroskopie
- Rekonstruieren z-Stapel mit der automatische treffen Modus des "Oberflächen" Werkzeug. 1D zeigt eine reconstruction jedes Kanals mit Hilfe der glatten Option, die Oberflächen-Bereich Detail auf 0,126 & mgr; m und Schwellen als absolute Intensität.
12. Serienblock Gesicht SEM
Hinweis: Die manuelle Datensegmentierung ist ein sehr zeitaufwändiges Verfahren und in Abhängigkeit von den Merkmalen, das Verfahren kann mehrere Wochen dauern gerendert werden. Die Segmentierung für die Videos und Figuren in Bohórquez et al. 2014 8 wurde manuell durchgeführt und dauerte etwa 500 Stunden der Arbeit. Es wird empfohlen, die wesentlichen Merkmale benötigen Rendering vor Beginn der Segmentierungsprozess zu priorisieren. Die Vorgehensweise ist wie folgt:
- Verwenden Sie die "Oberflächen" Werkzeug in Zeichenmodus und die "Kontur" -Option im Zeichenmodus von 50 ms bis Segment manuell und Volumen machte die Zelle von Interesse.
- Spuren Konturen auf jede Scheibe der Zelle für glattere Rendering oder alle 5 Scheiben für schnelleres Rendering.
- Export im FinaleAlter mit Hilfe der "Schnappschuss" Werkzeug bei einer Auflösung von 300 dpi oder mehr.
Ergebnisse
Das hier vorgestellte Verfahren wurde verwendet, um die Ultrastruktur von einer bestimmten Zelle innerhalb einer Epithelschicht studieren. Die Zelle von Interesse ist in diesem Fall das schwer fassbare enteroendokrinen Zelle. Ihre Identifikation in situ wurde erst in den letzten Jahren mit der Entwicklung von zellspezifischen Fluoreszenzreportermäusen möglich. Im Jahr 2011, haben wir eine Maus, bei der der Promotor des Hormons PYY treibt die Expression des grün fluoreszierenden Proteins 17. In diesem PyyGFP Maus werden enteroendokrinen Zellen des distalen Teils des Dünndarms und des Dickdarms leicht unter UV-Licht aus. Das Maus-Modell war ein Grundstein, um einen Gewebeblock, die ein enteroendokrinen Zelle erkennen und verarbeiten es für SBEM. Das Korrelationsverfahren wird hier als cocem3D bezogen und auf der zuvor veröffentlichten Protokollen 10,11 aufgebaut.
Optimierung der Abmessungen der Gewebeblocks ist kritisch für die Korrelation. In Vorversuchenes wurde festgestellt, dass in einem Gewebeblock 300 & mgr; m lang x 50 um dick die Anzahl der SBEM Bilder wird auf ein Minimum reduziert, während die den gesamten Körper der Zelle von Interesse. 1A zeigt einen Overlook der Dissektion mit einem Schwingklinge microtrome zu erhalten die Gewebeblock mit den richtigen Abmessungen. Mindestens 100 Blöcke werden vor dem Sortieren durch sie an Menschen mit intakten Zellen von Interesse holen erhalten. Ausgewählten Blöcke durch konfokale Mikroskopie und Bild z-Stapel abgebildet werden optisch beabstandet jeden 1 & mgr; m (1B und C). 1D zeigt eine volumenwiederAnsicht einer enteroendokrine Zelle im Ileum der Maus. Seine herausragende neuropod sich unterhalb Epithelzellen für ~ 60 & mgr; m.
Die Zelle von Interesse wird durch Korrelation der konfokalen z-Stacks mit einer SBEM Bild des gesamten Blockfläche gefunden. Ein Beispiel ist hier aus einem Block aus dem distalen Kolon eines PyyGFP vorgestelltMaus. Nach Erhalten konfokalen Z-Stapel (2A), wird der Block in einer dünnen Schicht von Agarose mit niedrigem Schmelz (2B) eingebettet. Dies ist für die nachfolgende Bearbeitung des Blocks erforderlich. Es ist wichtig, die Ausrichtung des Blocks so flach wie möglich, um die optischen Scheiben mit den Scheiben SBEM übereinstimmen aufrechtzuerhalten. In 2C wird ein repräsentatives Bild von der gesamten Fläche der Gewebeblock gezeigt. Teile dieser Figur wurden zuvor in Bohorquez et al., 2014 8. Dieses wurde anschließend mit dem konfokalen Bild unter Berücksichtigung Teilpunkte und Maße korreliert. 2D zeigt eine Überlagerung der konfokalen und SBEM Bild des Blocks und enthüllt die genaue Lage unserer Zelle von Interesse.
Sobald die Zelle identifiziert wurde, wird SBEM Bildgebung bei einer Auflösung hoch genug, um sekretorischen Vesikeln und anderen Zellorganellen zu identifizieren getan. Der Datensatz präsentierte erre wurde bei 7 nm pro Pixel abgebildet wird, wie Bilder des Blocks wurden alle 70 nm gemacht. Der Datensatz enthält 643 Bilder, 45 um der Gewebetiefe erstrecken. Wenige Mikrometer sind während der anfänglichen Bilderzeugung der gesamten Blockfläche bei geringer Vergrößerung rasiert. Die rohe SBEM Datensatz wurde in .dm3 Format erworben und TIFF für nachfolgende Handhabung umgewandelt. Die SBEM Stapel wurde mit der Fidschi Software-Plugin "crop (3D)" Block, nur den Bereich von Interesse enthalten, abgeschnitten. Dies reduziert die Menge an RAM-Speicher für Volumenberechnungen notwendig. Die Zelle wurde durch seine Position und sekretorischen Vesikeln identifiziert und segmentiert Verwendung Imaris Software, Fig. 3 enthält eine Wiedergabe in 3D des enteroendokrine Zelle.
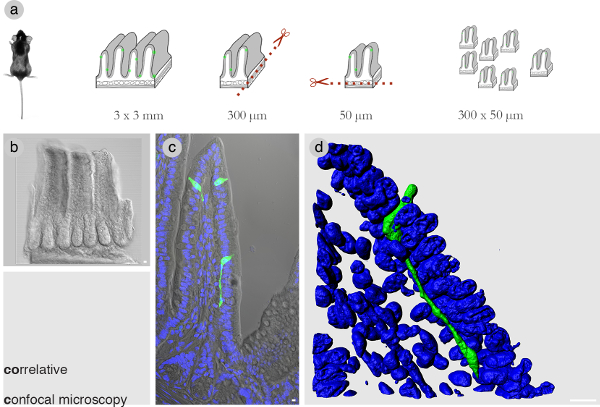
Abb. 1: Correlative Konfokalmikroskopie (A) Arbeitsablauf, um ein Stück zu sezierenDarmgewebe in ein Gewebe Block 300 x 50 & mgr; m dick. (B) Eine repräsentative Gewebeblock der richtigen Dimensionen. Man beachte, dass ein Gewebeblock dieser Dimensionen aus der Maus Dünndarm erstreckt sich über etwa 3 Zotten oder im Falle des Kolons etwa 8 Krypten. (C) Eine Zotten des Dünndarms, die eine Zelle von Interesse. (D) Ein konfokales Z-Stapel in 3D mit Imaris Software gerendert zeigt eine enteroendokrine Zelle mit einem prominenten neuropod unterhalb des Darmepithels läuft. Blau = DAPI Kernfärbung. Bars = 10 & mgr; m. Bitte klicken Sie hier, um eine größere Version dieser Figur zu sehen.
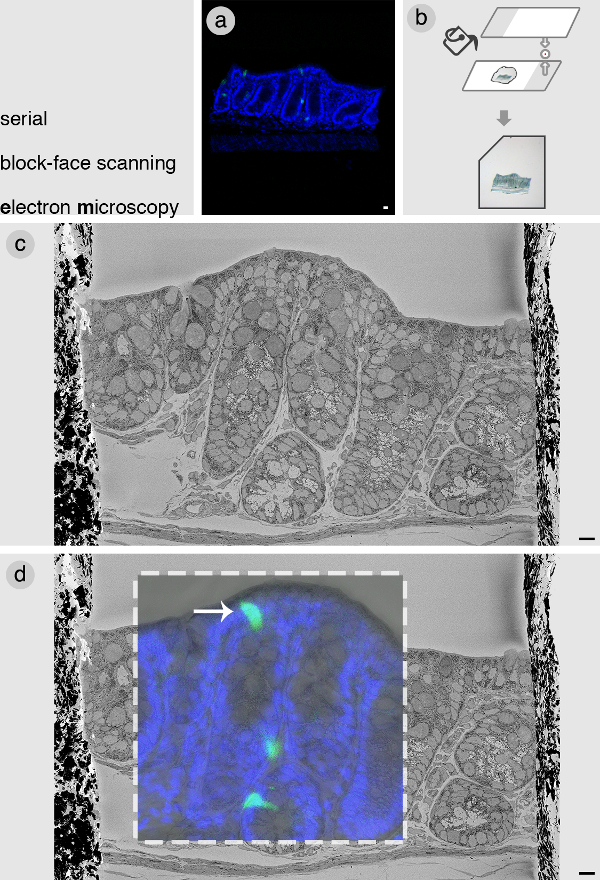
Figur 2: Serienblockfläche Rasterelektronenmikroskopie (A) konfokale Bild Fischspeziali.cted Gewebeblock aus dem Dickdarm PyyGFP enteroendokrinen Zellen (grün) von Interesse enthält. (B) Wenn der Block durch konfokale Mikroskopie abgebildet werden, es wird dann flach in niedrig schmelzenden Agarose unter Verwendung von Glasplättchen eingebettet. Einbetten der Gewebeblock in Agarose erleichtert anschließende Manipulation und die Kerbe in der linken Ecke hilft, den Block innerhalb des SBEM in der richtigen Orientierung zu montieren. (C) SBEM Bild des Gesichts des Blocks. (D) Korrelation der Daten mit konfokalen SBEM zu Zelle von Interesse (weißer Pfeil) zu identifizieren. Bilder c und d der Figur sind aus Bohorquez et al. Modifiziert 2014 8. Blau = DAPI Kernfärbung. Bars = 10 & mgr; m. Bitte klicken Sie hier, um eine größere Version dieser Figur zu sehen.
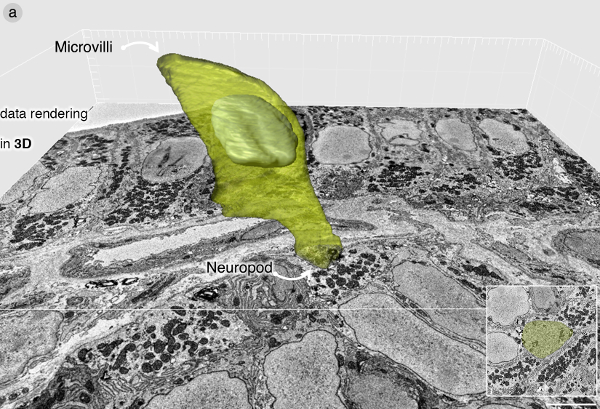
Abb. 3: Die Daten in 3D-Rendering (A) Volume-Rendering enteroendokrinen Zelle mit Imaris Software. Die Zelle enthält eine neuropod mit sekretorischen Vesikeln verpackt. Für eine vollständige Beschreibung der Ultrastruktur einer Zelle enteroendokrinen Sie bitte Bohórquez et al., 2014 8 beziehen. Bitte klicken Sie hier, um eine größere Version dieser Figur zu sehen.
Diskussion
Magen-Darm-Chemosensorik wird als ein aufregendes neues Gebiet in der biomedizinischen Forschung entstehen. Dies ist zum großen Teil aufgrund der Entdeckung der funktionellen Geschmacksrezeptoren in enteroendokrinen Zellen 18. Nachfolgende Studien haben gezeigt, dass enteroendokrinen Zellen exprimieren Rezeptoren für bestimmte Nährstoffe, einschließlich Kohlenhydraten, Lipiden und Aminosäuren 5,6,19,20. Die katalytische Faktor für diesen Entdeckungen wurde die Entwicklung von Reporter Mäusen, bei denen enteroendokrinen Zellen fluoreszierend markiert 20. Wir entwickelten eine dieser Mäuse, die PyyGFP Maus, um enteroendokrinen Zellen des distalen Dünndarm und Dickdarm 17,21 studieren. Diese Zellen sind von Interesse, weil sie PYY und Glucagon-like Peptid 1, wobei beide Induktoren Sättigungs 22,23 absondern. Zu der Zeit, eine komplette Ultrastruktur Konto dieser Zellen fehlte, die wir glaubten, war notwendig, um die Signalmechanismen zu verstehen.
NHALT "> Hier haben wir beschrieben, eine optisch ein Verfahren zur konfokalen Mikroskopie mit SBEM überbrücken. Die Methode wird als Cocem3D, die uns um die komplette Ultrastruktur von Zellen enteroendokrinen dokumentieren. Wir haben berichtet, dass diese Zellen eine neuropod, die drei- enthält zugelassen bezeichnet Viertel aller sekretorischen Vesikeln 8. Im neuropod gibt Neurofilamente und ähnlich wie neuronale Axone werden neuropods durch Darm Gliazellen 8 gefördert. Noch wichtiger ist es, obwohl diese neuropods das Zellen enteroendokrinen physische Verbindungen mit Neuronen innerviert den Darm und Doppelpunkt 9.Eine der Stärken der cocem3D ist seine Einfachheit. Verringern der Gewebeprobe in einen Block, der durch konfokale abgebildet werden kann und SBEM erleichtert die Identifizierung einer bestimmten Zelle in einem Gewebe. Sekretorischen Vesikeln und anderen Organellen mit Leichtigkeit bei einer Auflösung von 7 nm / Pixel identifiziert werden. Da die Abschnitte in SBEM werden verworfen, weitere processingen von Geweben, um spezifische Proteine zu identifizieren ist keine Option an dieser Stelle. Jedoch ist die Entwicklung von Methoden wie ATUM 24, wobei Abschnitte von dem Gewebe-Block erhalten werden, geeignet sind, die Identifizierung von spezifischen Proteinen in der Zelle zu ermöglichen. Bestimmung der spezifischen Lage der chemosensorische Rezeptoren auf enteroendokrinen Zellen wesentliche Informationen für die Entwicklung von Arzneimitteltherapien für Übergewicht, weil enteroendokrinen Zellen sind eine sensorische Schnittstelle zwischen Nahrung im Darm und das Sättigungsgefühl im Gehirn.
Offenlegungen
Dr. Satish Medicetty ist Mitarbeiter von und erhält 100% Gehalt von Renovo Neural Inc; Allerdings bedeutet dies nicht des Autors Einhaltung JoVE Politik ändern auf den Austausch von Daten und Materialien. Kein Interessenkonflikt ist für Rest Autoren erklärt.
Danksagungen
Our sincere appreciation is expressed to the following people: Drs. Sam Johnson and Benjamin Carlson of the Duke Light Microscopy Core Facility for their assistance with data visualization software, and Ms. Valerie Lapham and Dr. John M. Mackenzie, Jr. of the Center for Electron Microscopy at North Carolina State University for their advice on electron microscopy. We thank Dr. Elaine B. Bohórquez for her editorial assistance. Authors contributed in the following manner: DVB, SM, and RAL designed experiments and analyzed data. DVB performed experiments and FH performed manual rendering of data. SM is director of Renovo Neural, where SBEM data was acquired. DVB wrote the manuscript and all authors reviewed and edited the final manuscript. This work was supported by NIH grants R01DK091946 and Veterans Affairs grant I01BX002230 to RAL, and F32DK094704, to DVB.
Materialien
| Name | Company | Catalog Number | Comments |
| Phosphate buffered saline | Life technologies | 10010023 | |
| Heparin sodium salt | Sigma | H4784 | |
| Paraformaldehyde | Sigma | 158127 | 4%, freshly made in PBS, final pH 7.4 |
| Glutaraldehyde | Sigma | G5882 | |
| Dental wax | Electron Microscopy Sciences | 72660 | |
| Low-melting agarose | Life technologies | 16520-100 | 5%, freshly made in PBS |
| Standard Tissue-Tek Cryomold | Electron Microscopy Sciences | 62534-25 | |
| DAPI nuclear stain | Life technologies | D1306 | |
| Postively charged glass slides and coverslips | |||
| Fine art paintbrush #1 | |||
| Cacodylate buffer | Electron Microscopy Sciences | 11650 | |
| Tannic acid | Electron Microscopy Sciences | 21700 | |
| EMbed 812 kit | Electron Microscopy Sciences | 14120 | |
| Liquid releasing agent | Electron Microscopy Sciences | 70880 | |
| Liquid silver colloidal | Electron Microscopy Sciences | 12630 | |
| CircuitWorks conductive epoxy | ITW Chemtronics | CW2400 | |
| Variable flow peristaltic pump | VWR | 70730-064 | |
| VT1200S Vibrating blade microtome | Leica | ||
| Zeiss 780i confocal microscope | Carl Zeiss | ||
| Sigma VP Scanning Electron Microscope | Carl Zeiss | ||
| 3view system | Gatan | ||
| Renovo Neural Inc (Cleveland, OH) | http://www.renovoneural.com | Renovo provides 3d EM services | |
| Fiji software | Open access software | ||
| Computer station with 16 GB of RAM or more | |||
| Data visualization software Imaris 7.5 | Bitplane |
Referenzen
- White, J. G., Southgate, E., Thomson, J. N., Brenner, S. The structure of the nervous system of the nematode Caenorhabditis elegans. Philosophical transactions of the Royal Society of London. Series B, Biological. 314, 1-340 (1986).
- Smith, C. L., et al. Novel cell types, neurosecretory cells, and body plan of the early-diverging metazoan Trichoplax adhaerens. Current biology : CB. 24, 1565-1572 (2014).
- Denk, W., Horstmann, H. Serial block-face scanning electron microscopy to reconstruct three-dimensional tissue nanostructure. PLoS biology. 2, e329 (2004).
- Engelstoft, M. S., et al. Seven transmembrane G protein-coupled receptor repertoire of gastric ghrelin cells. Molecular metabolism. 2, 376-392 (2013).
- Chandra, R., et al. Immunoglobulin-like domain containing receptor 1 mediates fat-stimulated cholecystokinin secretion. The Journal of clinical investigation. 123, 3343-3352 (2013).
- Wang, Y., et al. Amino acids stimulate cholecystokinin release through the Ca2+-sensing receptor. Am J Physiol Gastrointest Liver Physiol. 300, G528-G537 (2011).
- Rogers, G. J., et al. Electrical activity-triggered glucagon-like peptide-1 secretion from primary murine L-cells. The Journal of physiology. 589, 1081-1093 (2011).
- Bohórquez, D. V., et al. An enteroendocrine cell-enteric glia connection revealed by 3D electron microscopy. PloS one. 9, e89881 (2014).
- Bohórquez, D. V., et al. Neuroepithelial circuit formed by innervation of sensory enteroendocrine cells. The Journal of clinical investigation. , (2015).
- Knott, G. W., Holtmaat, A., Trachtenberg, J. T., Svoboda, K., Welker, E. A protocol for preparing GFP-labeled neurons previously imaged in vivo and in slice preparations for light and electron microscopic analysis. Nature protocols. 4, 1145-1156 (2009).
- Deerinck, T., Bushong, E., Thor, A., Ellisman, M. . NCMIR methods for 3D EM: a new protocol for preparation of biological specimens for serial block face scanning electron microscopy. , (2010).
- Bohórquez, D. V., Chandra, R., Samsa, L. A., Vigna, S. R., Liddle, R. A. Characterization of basal pseudopod-like processes in ileal and colonic PYY cells. Journal of molecular histology. 42, 3-13 (2011).
- Nilsson, O., et al. Distribution and immunocytochemical colocalization of peptide YY and enteroglucagon in endocrine cells of the rabbit colon. Endocrinology. 129, 139-148 (1991).
- Mizuhira, V., Futaesaku, Y. New fixation method for biological membranes using tannic acids. Acta Histochem Cytochem. 5, 233-236 (1972).
- Reymond, O. L., Pickett-Heaps, J. D. A routine flat embedding method for electron microscopy of microorganisms allowing selection and precisely orientated sectioning of single cells by light microscopy. Journal of microscopy. 130, 79-84 (1983).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nature. 9, 676-682 (2012).
- Bohórquez, D., Chandra, R., Samsa, L., Vigna, S., Liddle, R. Characterization of basal pseudopod-like processes in ileal and colonic PYY cells. J Mol Histol. 42, 3-13 (2011).
- Jang, H. J., et al. Gut-expressed gustducin and taste receptors regulate secretion of glucagon-like peptide-1. Proceedings of the National Academy of Sciences of the United States of America. 104, 15069-15074 (2007).
- Liou, A. P., et al. The G-protein-coupled receptor GPR40 directly mediates long-chain fatty acid-induced secretion of cholecystokinin. Gastroenterology. 140, 903-912 (2011).
- Reimann, F., et al. Glucose sensing in L cells: a primary cell study. Cell metabolism. 8, 532-539 (2008).
- Bohorquez, D. V., Liddle, R. A. Axon-like basal processes in enteroendocrine cells: characteristics and potential targets. Clinical and translational science. 4, 387-391 (2011).
- Batterham, R. L., et al. Gut hormone PYY(3-36) physiologically inhibits food intake. Nature. 418, 650-654 (2002).
- Flint, A., Raben, A., Astrup, A., Holst, J. J. Glucagon-like peptide 1 promotes satiety and suppresses energy intake in humans. The Journal of clinical investigation. 101, 515-520 (1998).
- Hayworth, K. J., et al. Imaging ATUM ultrathin section libraries with WaferMapper: a multi-scale approach to EM reconstruction of neural circuits. Frontiers in neural circuits. 8, 68 (2014).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten