É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Um ELISA baseada Encadernação e Método de competição de determinar rapidamente interacções ligando-receptor
Neste Artigo
Resumo
The presented protocols describe two enzyme-linked immunosorbent assay (ELISA) based techniques for the rapid investigation of ligand-receptor interactions: The first assay allows the determination of dissociation constant between ligand and receptor. The second assay enables a rapid screening of blocking peptides for ligand-receptor interactions.
Resumo
A comprehensive understanding of signaling pathways requires detailed knowledge regarding ligand-receptor interaction. This article describes two fast and reliable point-by-point protocols of enzyme-linked immunosorbent assays (ELISAs) for the investigation of ligand-receptor interactions: the direct ligand-receptor interaction assay (LRA) and the competition LRA. As a case study, the ELISA based analysis of the interaction between different lambda interferons (IFNLs) and the alpha subunit of their receptor (IL28RA) is presented: the direct LRA is used for the determination of dissociation constants (KD values) between receptor and IFN ligands, and the competition LRA for the determination of the inhibitory capacity of an oligopeptide, which was designed to compete with the IFNLs at their receptor binding site. Analytical steps to estimate KD and half maximal inhibitory concentration (IC50) values are described. Finally, the discussion highlights advantages and disadvantages of the presented method and how the results enable a better molecular understanding of ligand-receptor interactions.
Introdução
Uma compreensão abrangente de vias de sinalização requer conhecimento detalhado sobre a interação ligando-receptor. A maioria dos métodos para avaliar a interacção de um ligando particular com o seu receptor específico são caros, consomem tempo, trabalho intensivo e exige equipamentos e conhecimentos específicos 1.
Este artigo descreve dois protocolos rápidos e fiáveis ponto-a-ponto para investigar a interacção ligando-receptor baseado em um ensaio de imunoabsorção enzimática (ELISA): o ensaio de interacção ligando-receptor directa (ERS) e o LRA competição. ELISA é uma técnica altamente sensível, específico e facilmente disponível, rotineiramente utilizados em quase todos os laboratórios. ELISA pode ser realizada e adaptada de diversas formas. Os protocolos apresentados são otimizados para a investigação da interação entre diferentes interferons lambda (INFLs) e seu receptor.
O LRA direta permite uma quantificatina de ligação ligando-receptor em relação à concentração do ligando e, portanto, produz uma curva de ligação. Usando um modelo apropriado para a interacção ligando-receptor, os dados podem ainda ser analisados para se avaliar a constante de dissociação (K D).
No protocolo apresentado, a equação de Hill vulgarmente utilizado é aplicada para modelar a ligação do ligando ao receptor. Embora outros métodos, tais como a tecnologia de ressonância de plasmon de superfície 2,3 permitir a determinação das afinidades de ligação entre as duas proteínas, esta tecnologia é muitas vezes um trabalho intensivo e caro, e requer equipamento laboratorial especial.
O LRA competição permite o rastreio de péptidos inibidores: A ligação ligando-receptor é quantificada em relação à concentração de péptido. Obteve-se uma curva de dose-resposta que descreve o efeito inibidor do péptido. Os dados podem ainda ser analisados para se estimar a concentração inibitória máxima metade (IC50 ) do péptido de bloqueio.
Ambos os protocolos de ELISA são fáceis de utilizar e podem ser adaptadas a uma ampla variedade de questões de pesquisa. As proteínas recombinantes, de qualquer tipo pode ser utilizada para determinar de forma fiável e rápida das peças de interacção. Além disso, o LRA competição pode ser usado para determinar sítios de interacção fundamentais de ligandos e receptores, utilizando os péptidos de bloqueio, que são concebidos para imitar ou o ligando ou o receptor. Se o péptido de bloqueio apresenta uma inibição eficiente e específico, o péptido ocupa um local crítico interacção do ligando (se o péptido imita o receptor) ou do ligando (se o péptido imita o ligando).
O primeiro protocolo descreve a determinação de K D valor de diferentes INFLs e a subunidade alfa do seu receptor, isto é, o receptor de interleucina-28 (IL28RA) usando o LRA directa. Em seguida, o segundo protocolo mostra como para determinar a capacidade de um péptido de comprimento de 20 aminoácidos deinibir as interacções INFL-IL28RA. O péptido é concebido para competir com IFNLs no seu local de ligação ao receptor e, portanto, permite uma compreensão molecular da interacção. Além disso, este péptido pode ser utilizado para bloquear IL28RA em experiências in vitro para determinar o impacto sobre os efeitos de sinalização a jusante 4.
Protocolo
1. Preparação de Reagentes
- Para preparar tampão de revestimento de carbonato, dissolve-se 0,36 g de Na 2 CO 3 e 0,84 g de NaHCO3 em 100 ml de água destilada; filtro estéril o buffer usando um vácuo impulsionado 0,22 polietersulfona (PES) filtro de membrana e armazenar a temperatura ambiente, até o uso.
- Preparar a solução de lavagem através da adição de 0,05% v / v de Tween 20 em tampão fosfato salino (PBS).
- Prepara-se uma 5% Albumina de Soro Bovino (BSA) (solução de bloqueio) em solução de PBS por dissolução de 5 g de BSA (≥98%) em 100 ml de PBS e armazenar a 4 ° C.
- Recombinante do receptor, os ligandos e os péptidos de bloqueio
- Reconstitutethe subunidade recombinante humana alfa do receptor da interleucina (IL28RA) e recombinantes ligantes His-tag de IFN humana (IFNL1-3) de acordo com instruções e armazenamento do fabricante a -80 ° C. Sintetizar péptidos de bloqueio e utilizados tal como previamente descrito 4. Use PBS para preparar a diferentes concentrações de ligandos e péptidos para utilização nos ensaios.
- Para preparar o anticorpo primário, diluir 6X His anticorpo monoclonal de ratinho em PBS com 0,1% de BSA a 1: 1000 de diluição. Para preparar o anticorpo secundário, dilui-se a peroxidase de rábano (HRP) conjugado bode anti-rato IgG (H + L) em PBS com 0,1% de BSA a diluição de 1: 10.000.
- Preparar a solução de TMB por mistura dos reagentes A e B de acordo com as instruções do fabricante.
- Preparar a solução de paragem através da adição de ácido sulfúrico 5 N (H 2 SO 4), em água destilada e armazenar a RT.
2. imunoabsorvente ligado a enzima (ELISA Assays)
NOTA: A interacção directa ligando-receptor de ELISA (LRA directa, a Figura 1) pode ser utilizado para medir a dissociação do receptor-ligando constante (K D), como uma medida da afinidade de ligação ao receptor-ligando. A interacção ligando-receptor ELISA de competição (LRA competição, Figura 2) tudoOWS rastreio de péptidos (e outros compostos de bloqueio), que actuam de modo a interferir com a interacção entre ligando e receptor. O protocolo básico que foi publicado anteriormente 5 foi ainda mais aperfeiçoado.
NOTA: Em ambos os métodos ELISA usar pipeta multicanal para a adição de soluções para os poços da placa de 96 poços em cada passo. Em solução decantar ou etapas de lavagem, jogue fora as soluções diretamente na pia.
- Directo Ligando-Receptor Interaction Assay (LRA directa)
NOTA: Para obter uma ilustração do fluxo de trabalho (ver Figura 1).- Placa de revestimento com o receptor recombinante
- Dilui-se o receptor recombinante em tampão de carbonato a uma concentração final de 100 ng / ul. revestimento de poços de 96 poços de placas de microtitulação com uma concentração fixa de receptores (100 ng / ul) por pipetagem de 100 ul a cada poço utilizando uma pipeta de canais múltiplos. Excluir paredes exteriores da placa para evitar bem artefato borda. Cobrir a placa com uma tampa e incubar aem placas a 4 ° CO / N.
- Bloqueio e adição de ligantes
- No dia seguinte, remover a solução de revestimento através da inclinação da placa contra a pia e lavar a placa 3 vezes com solução de lavagem (PBS + 0,05% v / v de Tween 20).
- Bloquear os sítios de ligação de receptores livres na placa revestida com 200 ul de solução de BSA a 5% a cada poço utilizando uma pipeta de canais múltiplos e incubar a placa durante 2 horas à temperatura ambiente.
- Descartar a solução de bloqueio (ver passo 2.1.2.1.) E lavar a placa 3 vezes com solução de lavagem.
- Prepara-se o recombinante marcada com His ligandos em diferentes concentrações (por exemplo, 8 ug / mL, 4 jig / mL, 2 ug / mL, 1 ug / mL, 0,5 ug / mL, 0,25 ug / mL, 0,125 ug / mL, 0,063 ug / mL, 0,031 ug / mL, 0,0 ug / ml) em PBS. Adicione somente PBS nos poços branco.
- Adiciona-se 100 ul de cada concentração de ligando-se aos poços em duplicado e incubar a placa durante 2 horas à temperatura ambiente permitindo receptor-ligandointeração.
- A incubação com os anticorpos
- A seguir à incubação com os ligandos, lavar a placa 3 vezes com solução de lavagem.
- Pipetar 100 l de Seu anti-solução principal do rato anticorpo monoclonal (1: 1000) a cada poço.
- Incubar a placa a temperatura ambiente durante 2 horas; Após a incubação, rejeitar a solução de anticorpo (ver o passo 2.1.2.1.) e lavar a placa 3 vezes com solução de lavagem.
- Adicionar 100 ul de HRP de cabra acoplado solução de anticorpo secundário IgG anti-ratinho (1: 10000) a cada poço. Incubar a placa durante 45 minutos à TA.
- Descartar a solução do anticorpo (ver o passo 2.1.2.1.) E lavar a placa 3 vezes com solução de lavagem.
- Adição de substrato e Desenvolvimento
- Traga as soluções de substrato TMB para a TA e preparar a solução de substrato TMB A e B na proporção 1: 1. Adicionar 100 ul de substrato preparadas de fresco a cada poço e manter a placa à temperatura ambiente durante 15-30 min. Após col suficiente ou desenvolvimento adicionar solução de parada de 50 mL.
- Lendo a Análise Placa e Dados
NOTA: O protocolo descrito baseia-se no pressuposto de que o sinal medido sobe de ligação específica. Pode ser necessário para estimar a contribuição de ligação não especifica para o sinal, mas esta está fora do âmbito deste protocolo.- Leia a absorvância (densidade óptica, OD) a 450 nm directamente.
- Subtrair o sinal de fundo a partir dos valores medidos e OD normalizar-los. Transformar todos os valores da concentração do ligando a escala logarítmica (base 10, log 10).
- Traça-se a normalizada e de fundo corrigido valores de densidade óptica (Y-eixo, corresponde à fracção de locais de ligação do receptor ocupada) em função do logaritmo da concentração do ligando (eixo-X, escala log10).
- Para estimar o valor K D, ajustar os dados para a seguinte forma da equação Hill:
tp_upload / 53575 / 53575eq1.jpg "/>
NOTA: Aqui Y denota a fração de sítios receptores ocupados ligação e Y max a ligação máxima; [L] indica a concentração de ligando livre e o coeficiente de Hill. Se houver apenas um local de ligação para o ligando, o coeficiente de Hill é n = 1. Para sistemas com local de ligação mais do que um ligando, a exposições de ligação cooperatividade positiva se n> 1, cooperatividade negativa se n <1 e não se n cooperatividade = 1. a constante de dissociação é denominado microscópica e corresponde a metade do máximo de concentração eficaz CE50 6. A constante de dissociação aparente é K d = (K D) n. No caso mais simples, em que n = 1, a constante de dissociação corresponde à concentração do ligando na qual metade dos sítios de ligação do receptor são ocupados e K d = k D. Este modelo parte do princípio de acção de massa de ligação sob condições de equilíbrio, assim como a que apenas uma pequena fracção deo ligando é adicionado ligado ao receptor, isto é, [L] >> [RL].
- Placa de revestimento com o receptor recombinante
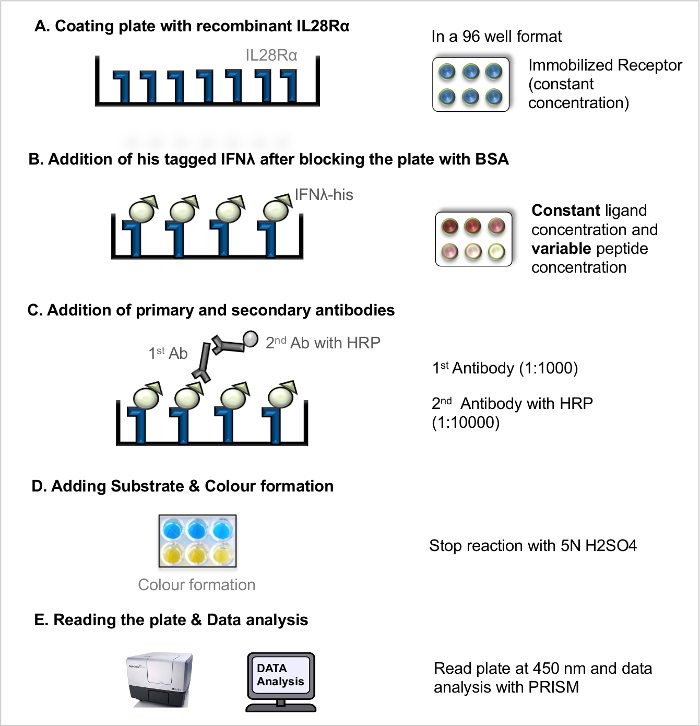
Figura 1. direto ensaio ligante receptor-interação (LRA direta). Protocolo passo-a-passo para a LRA direto. Por favor clique aqui para ver uma versão maior desta figura.
- Competição Ligand-Receptor-Interaction Assay (competição LRA)
NOTA:. Para uma ilustração do fluxo de trabalho veja a Figura 2 O processo de concurso LRA segue os mesmos passos que o LRA direta (revestimento da placa, incubação do anticorpo, desenvolvimento placa), exceto para mudanças importantes no ligante e peptídeos passo além. controles negativos adequados são essenciais para este ensaio. Em um estudo de triagem anterior 4, o peptídeo bloqueando mexidos não mostraram efeitos antagónicos.- Bloqueio - A adição de ligantes e Peptídeos de bloqueio
- No dia seguinte, remover a solução de revestimento e lavar a placa (ver 2.1.2.1).
- Bloquear a placa revestida através da adição de 200 ul de solução de BSA a 5% a cada poço e incubar a placa durante 2 horas à temperatura ambiente.
- Prepare os ligandos recombinantes His-tag (IFNL1-3) a uma concentração fixa (2x-20 ng / ml) em PBS.
- Prepara-se o péptido de bloqueio (cf. Tabela 3) com diferentes concentrações variando desde 10 nM a 100 pM em PBS, para garantir uma curva de dose-resposta.
NOTA: Este permite a determinação subsequente do valor de IC 50 para o péptido de bloqueio. Em cavidades de controlo, adicionar ligando única concentração fixa sem péptido para derivar o máximo (100%) de ligação. O espaço em branco, adicione apenas PBS sem ligando ou peptídeo. - Adicionar 50 ul de ligandos (IFNL1-3) e 50 ul de cada peconcentração ptide aos poços em duplicado.
- Incubar a placa durante 2 horas à TA.
- Lendo a Análise Placa e dados
NOTA: O protocolo descrito baseia-se no pressuposto de que o sinal medido sobe de ligação específica. Pode ser necessário para estimar a contribuição de ligação não especifica para o sinal, mas esta está fora do âmbito deste protocolo.- Leia a absorvância (densidade óptica, OD) a 450 nm directamente.
- Subtrair o sinal de fundo a partir dos valores medidos e OD normalizar-los. Transformar todos os valores da concentração de peptídeo a escala logarítmica (base 10, log 10).
- Traça-se a normalizada e de fundo corrigido valores de densidade óptica (Y-eixo, corresponde à fracção de locais de ligação do receptor ocupada) em função do logaritmo da concentração do ligando (eixo-X, escala log10).
- Para estimar o valor de IC 50, ajustar os dados com a equação seguinte:
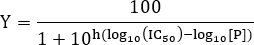
NOTA: Aqui [P] é a concentração de péptido e o declive Hill. A encosta descreve o declive da curva de dose-resposta. O IC50 corresponde à concentração de inibidor a que se observa% de inibição da ligação entre o ligando e o receptor 50.
- Bloqueio - A adição de ligantes e Peptídeos de bloqueio

Figura 2. Concorrência ensaio ligante receptor-interação (LRA concorrência). Protocolo passo-a-passo para a LRA concorrência. Por favor clique aqui para ver uma versão maior desta figura.
Resultados
As constantes de dissociação entre INFL1-3 e sua subunidade alfa do receptor de IL28RA foram determinados utilizando o LRA directa. Os resultados são apresentados na Figura 3: A fracção de sítios de ligação ocupados é representada graficamente contra o logaritmo da concentração de IFN respectivo. O gráfico de Scatchard dos dados é mostrado no canto inferior direito. Os resultados mostram que o LRA directa produz uma curva de ligação, o qual pode ser ainda ...
Discussão
ELISA é um método padrão e bem estabelecido para muitos laboratórios. Temos ainda mais modificado e melhorado a 5,7 método previamente publicado. O protocolo passo-a-passo demonstrou mostra como ela pode ser usada de uma maneira simples para determinar os valores de KD de interacções ligando-receptor. Além disso, pode ser determinado o IC50 de um péptido de bloqueio que interfere com a interacção ligando-receptor.
As principais vantagens são a rápida instal...
Divulgações
A.E. was supported by a research grants from the "SNSF Ambizione Score" program (PZ00P3_154709), "Forschungsfond, Förderung strategischer Projekte" University of Basel, Stiftungsinfektionskrankheiten Basel, and Bangeter Rhyner Stiftung. J.L. acknowledges support by an iPhD fellowship of the SystemsX.ch initiative in systems biology program (9th call).
Agradecimentos
We thank Prof. J. Stelling (Department of Biosystems Science and Engineering, ETH Zurich and Swiss Institute for Bioinformatics, Basel, Switzerland) for his critical review of the manuscript.
Materiais
| Name | Company | Catalog Number | Comments |
| Nunc-Immunoplate (F96 Maxi sorp) | Thermo Scientific | 442404 | ELISA plate |
| Sodium carbonate (Na2CO3) | Merck | 497-19-8 | For ELISA plate coating buffer |
| Sodium hydrogen carbomnate(NaHCO3) | Merck | 144-55-8 | For ELISA plate coating buffer |
| Bovine Serum Albumin (BSA) | Sigma | A7030-100G | 5% BSA in PBS for Blocking |
| rhIL-28Rα/IFNλR1 | R&D systems | 5260-MR | Recombinant human interlukin-28 Receptor alpha |
| rhIL-29/IFNλ1 | R&D systems | 1598-IL/CF | Recombinant human interlukin-29/Carrier free/C-terminal 10-His tag |
| rhIL-28A/IFNλ2 | R&D systems | 1587-IL/CF | Recombinant human interlukin-28A/Carrier free/C-terminal 6-His tag |
| rhIL-28B/IFNλ3 | R&D systems | 5259-IL/CF | Recombinant human interlukin-28B/Carrier free/C-terminal 6-His tag |
| 6x His Monoclonal antibody (Mouse) | Clontech | 631212 | Primary antiboy to capture His tagged Ligands |
| Goat anti-Mouse igG (H+L) | Jackson Immuno Research | 115-035-166 | Horseradish Peroxidase conjucated secondary antibody |
| BDoptEIA TMB reagent set | BD Biosciences | 555214 | ELISA - TMB substrate solution |
| Sulfuric acid (H2SO4) | Fulka | 84720 | 5 N H2SO4 (Enzyme reaction stop solution) |
| Synergy/H1 - Microplate reader | BioTeK | ELISA plate reader |
Referências
- Schneider, P., Willen, L., Smulski, C. R. Tools and techniques to study ligand-receptor interactions and receptor activation by TNF superfamily members. Methods in enzymology. 545, 103-125 (2014).
- Rossi, G., et al. Biosensor analysis of anti-citrullinated protein/peptide antibody affinity. Analytical biochemistry. 465, 96-101 (2014).
- van der Merwe, P. A., Barclay, A. N. Analysis of cell-adhesion molecule interactions using surface plasmon resonance. Curr Opin Immunol. 8, 257-261 (1996).
- Egli, A., et al. IL-28B is a key regulator of B- and T-cell vaccine responses against influenza. PLoS Pathog. 10, e1004556 (2014).
- Rosenbluh, J., et al. Positively charged peptides can interact with each other, as revealed by solid phase binding assays. Analytical biochemistry. 352, 157-168 (2006).
- Goutelle, S., et al. The Hill equation: a review of its capabilities in pharmacological modelling. Fundamental & clinical pharmacology. 22, 633-648 (2008).
- Levin, A., et al. Peptides derived from HIV-1 integrase that bind Rev stimulate viral genome integration. PLoS One. 4, e4155 (2009).
- Egli, A., Santer, M. D., O'Shea, D., Tyrrell, D. L., Houghton, M. The impact of the interferon-lambda family on the innate and adaptive immune response to viral infections. Emerging infectious diseases. , e51 (2014).
- Gad, H. H., Hamming, O. J., Hartmann, R. The structure of human interferon lambda and what it has taught us. J Interferon Cytokine Res. 30, 565-571 (2010).
- Folch, B., Rooman, M., Dehouck, Y. Thermostability of salt bridges versus hydrophobic interactions in proteins probed by statistical potentials. Journal of chemical information and modeling. 48, 119-127 (2008).
- Yuzlenko, O., Lazaridis, T. Interactions between ionizable amino acid side chains at a lipid bilayer-water interface. The journal of physical chemistry. B. 115, 13674-13684 (2011).
- Tissot, A. C., Vuilleumier, S., Fersht, A. R. Importance of two buried salt bridges in the stability and folding pathway of barnase. Biochemistry. 35, 6786-6794 (1996).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados