Method Article
Avaliação simultânea de Cardiomyocyte DNA Síntese e Ploidia: Um Método de Assistência Quantificação dos cardiomiócitos Regeneração e volume de negócios
Neste Artigo
Resumo
Quantification of cardiomyocyte turnover is challenging. The protocol described here makes an important contribution to this challenge by enabling accurate and sensitive quantification of neo-cardiomyocyte nuclei generation and nuclei ploidy.
Resumo
Embora se aceite que o coração tem um limitado potencial para regenerar cardiomiócitos após a lesão e que os baixos níveis de volume de negócios de cardiomiócitos ocorrem durante o envelhecimento normal, quantificação desses eventos permanece desafiador. Isto é em parte devido à raridade do processo e o fato de que várias fontes celulares contribuem para a manutenção do miocárdio. Além disso, a duplicação do ADN dentro de cardiomiócitos muitas vezes leva a uma cardiomiócitos poliplóide e apenas raramente conduz a novos cardiomiócitos por divisão celular. A fim de quantificar com precisão o volume de negócios cardiomiócitos discriminação entre estes processos é essencial. O protocolo aqui descrito emprega rotulagem nucleósido a longo prazo de forma a marcar todos os núcleos que surgiram como resultado de núcleos de replicação de ADN e de cardiomiócitos identificados através da utilização de núcleos e subsequente isolamento de imunomarcação PCM1. Em conjunto, isto permite a identificação precisa e sensível da rotulagem do nucleósido Cardiomyocyte população de núcleos. Além disso, 4 ', rotulagem e análise de ploidia núcleos 6-diamidino-2-fenilindole, permite a discriminação de núcleos neo-cardiomiócitos de núcleos que incorporaram nucleósido durante poliploidização. Embora este método não pode controlar para binucleação cardiomiócitos, permite uma quantificação rápida e robusta de núcleos neo-cardiomiócitos, enquanto respondendo por poliploidização. Este método tem uma série de aplicações a jusante, incluindo a avaliação das terapias potenciais para melhorar cardiomiócitos regeneração ou a investigar os efeitos da doença cardíaca no volume de negócios de cardiomiócitos e ploidia. Esta técnica é também compatível com técnicas de imuno-histológicos adicionais a jusante, permitindo a quantificação de incorporação nucleósido em todos os tipos de células cardíacas.
Introdução
Nos últimos anos tem havido um acúmulo de evidências desafiando a suposição de que o coração está, a 1,2 órgão pós-mitótico terminalmente diferenciadas. No entanto, a quantificação do volume de negócios dos cardiomiócitos e regeneração continua sendo um desafio.
As dificuldades em identificar com precisão a geração de cardiomiócitos rara utilizando técnicas de imuno-histoquímica padrão são bem relatado 3. Além disso, a fonte celular de geração de cardiomiócitos permanece incerto com evidência para as contribuições pela proliferação de cardiomiócitos, bem como por diferenciação de células estaminais 4-6. Portanto, a utilização dos modelos de rastreio de linhagem que requerem o conhecimento do fenótipo de cardiomiócitos progenitora é impossível e quantificação da proliferação de uma população única, incluindo cardiomiócitos, é inadequado. Além disso, um dos cardiomiócitos tem o potencial para endoreplication sem cariocinese (resultando em um carro poliplóidediomyocyte) ou cariocinese na ausência de citocinese (resultando numa cardiomiócitos binucleadas) 7,8. A quantificação exacta do volume de negócios cardiomiócitos depende da capacidade de distinguir entre estes eventos e verdadeiro geração neo-cardiomiócitos. Isto cria complicações únicas porque a replicação do ADN e a expressão de quinases dependentes de ciclina em cardiomiócitos não demonstram exclusivamente divisão celular verdadeiro 9,10.
Para auxiliar na quantificação da produção de neo-cardiomiócito, que se combinaram uma técnica de isolamento de núcleos estabelecida, e rotulagem imunológica de material pericentriolar 1 (PCM1) para identificação núcleos dos cardiomiócitos como descrito por Bergmann et ai. 7,11 com novos métodos de longo prazo de marcação de ADN e análise de ploidia. PCM-1 é uma proteína que se acumula centrossoma na superfície nuclear de miócitos diferenciados, sem o ciclo. Estudos anteriores demonstraram que os anticorpos contraPCM-1 rotular especificamente núcleos dos cardiomiócitos 7,11 e como tal PCM1 tem sido usado por um certo número de grupos independentes para identificar cardiomiócitos 1,12,13. Além disso, nós demonstramos que a expressão PCM1 mapeia para os núcleos dos cardiomiócitos geneticamente marcados no modelo de TNT-cre rato transgénico 14 (Recurso Figura 1).
O protocolo aqui descrito permite a identificação precisa e sensível de geração de cardiomiócitos núcleos neo no coração de rato, independentemente das origens celulares (Figura 1A e B), enquanto, simultaneamente, excluindo rotulagem nucleósido devido a poliploidização a partir da análise (Figura 1C e D). Embora este método não pode controlar para binucleação cardiomiócitos, permite uma quantificação rápida e robusta de núcleos neo-cardiomiócitos que é necessário para a quantificação exacta do volume de negócios dos cardiomiócitos. Além disso,ele fornece uma ferramenta de triagem rápida de avaliar potenciais mudanças na dinâmica de geração de cardiomiócitos.
Enquanto marcação de ADN geralmente envolve 5-bromo-2'-desoxiuridina (BrdU) como o análogo da timidina, o protocolo descrito aqui utiliza um ensaio de 5-etinil-2'-desoxiuridina (EdU) com base, uma vez que requer menos passos de processamento para um mais rápido através colocar-e não requer a desnaturação do DNA para detecção de imuno-, que o torna compatível com outros protocolos de imunocoloração e aumentando assim os potenciais aplicações a jusante do método.
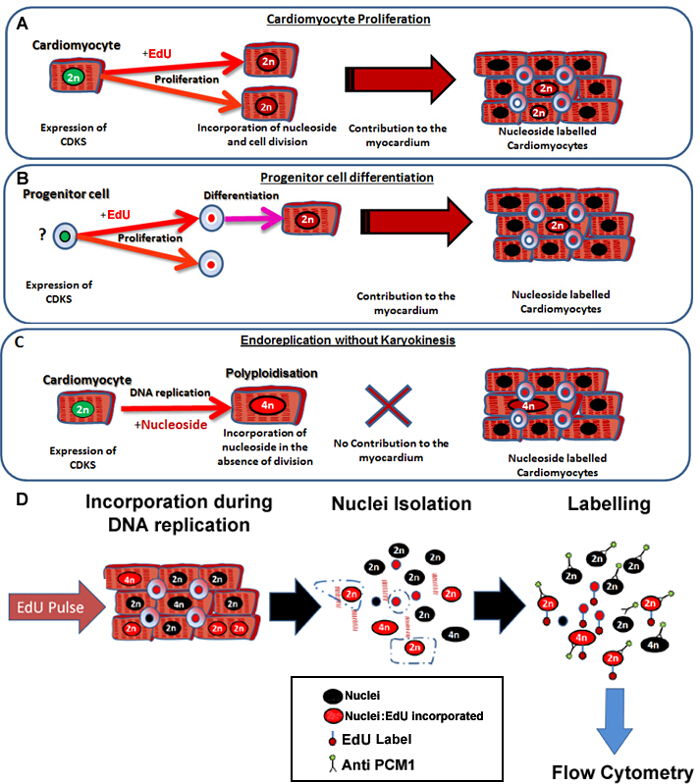
Figura 1: pulsação contínua com EdU etiquetas neo-cardiomiócitos, independentemente de seus progenitores. (A) EdU está incorporado no ADN de cardiomiócitos durante a divisão celular. Proliferação na população de cardiomiócitos vai Result em um aumento no, ou a substituição de cardiomiócitos e é, por conseguinte, a síntese de ADN produtiva (contribui para a manutenção e reparação de tecidos). (B) EdU está incorporado no ADN de células progenitoras cardíacas durante a divisão celular. Isto irá ser retido na célula durante a diferenciação para a linhagem de cardiomiócitos. Esta diferenciação de células estaminais também vai resultar num aumento do número de cardiomiócitos e, por conseguinte, contribui para a manutenção e a reparação dos tecidos. (C) Os cardiomiócitos têm o potencial para sofrer replicação de ADN "não produtiva" resultando em aumento da ploidia cardiomiócito, que está associado com hipertrofia de cardiomiócitos e remodelação do miocárdio, mas não substitui cardiomiócitos perdidos. O processo de poliploidização difere binucleação, que resulta em um dos cardiomiócitos com um único núcleo, que contém quatro ou mais conjuntos de dois cromossomas homólogos (> 2 N). (D) Na sequência de um núcleo contínuo pulse, este protocolo descreve o isolamento de núcleos e identificação dos núcleos dos cardiomiócitos por expressão PCM1 para permitir a quantificação de ambos ploidy cardiomiócitos e Edu incorporação. Expressão PCM1 e incorporação EdU detectada por citometria de fluxo. Por favor clique aqui para ver uma versão maior desta figura.
Protocolo
trabalho animal foi autorizado e aprovado pelo conselho de revisão Newcastle Ética da Universidade. Todos os procedimentos com animais foram realizados em conformidade com as diretrizes da Directiva 2010/63 / UE do Parlamento Europeu sobre a protecção dos animais utilizados para fins científicos.
1. Administração EdU
- Dissolver EdU em solução salina estéril (0,9% w / v) a uma concentração final de 12,5 mg / ml.
- Aquece-se a solução para 40 ° C e agitar com vortex para dissolver completamente. Armazenar o suficiente EdU / solução salina a 4 ° C para injectar todos os ratinhos no estudo permitindo 6 injecções diárias.
Nota: Tipicamente mais de 6 ratinhos são necessários para cada grupo experimental. No entanto, cálculos de potência deve ser realizada para estudos independentes.
- Aquece-se a solução para 40 ° C e agitar com vortex para dissolver completamente. Armazenar o suficiente EdU / solução salina a 4 ° C para injectar todos os ratinhos no estudo permitindo 6 injecções diárias.
- Pesar ratinhos individuais. Calcular o volume requerido para administrar 100 ug / g de solução / solução salina EdU para cada ratinho (8 ul de solução de reserva por EdU g).
- Desenhe o volum apropriadae de Edu / salina nos seringa de insulina com uma agulha 25 G.
- Execute intraperitoneal (ip) injeções durante o manuseio de ratos utilizando técnicas Home Office aprovados.
- Coloque um rato na tampa gaiola. Gentilmente puxar para trás na base da cauda e segure o mouse pela nuca.
- Expor o abdómen para injecção, segurando o mouse com o dedo indicador eo polegar perto da base da cabeça e inclinando a cabeça do animal para trás em direção ao chão.
- Limpe o abdômen com solução de álcool 70%. Localize linha média do animal e quadrante inferior direito. Injectar a solução EdU / Saline no animal para o quadrante inferior direito.
- Injetar ratos com uma solução / solução salina EdU e registrar a hora do dia.
- Repita os passos de 1,2 a 1,4 ao mesmo tempo do dia, durante 6 dias consecutivos.
Nota: Como um controle EdU negativo é necessário para definir o gating para análise de citometria de fluxo, injetar uma idade adicional, sexo e experimentalmente corresponderrato ed com um volume equivalente de solução salina sem EdU. Isto irá fornecer o controle necessário (sem controlo da UDE). Este controlo deve ser incluída para cada estudo para permitir variações em lotes de reagentes e citómetro de configurar.
2. Corações Colhendo
- Às 24 h após a injeção EdU 6º, sacrificar ratos usando deslocamento cervical (ou de um esquema alternativo 1 Home Office aprovado método).
- Coloque o animal sacrificado em decúbito dorsal e fazer incisão na pele a partir de meados do abdômen para o diafragma com uma tesoura cirúrgica.
- Cortar o diafragma, e segurando o esterno afastado da cavidade do corpo, corta bilateralmente para expor o coração.
- Levante o coração um pouco e cortar os vasos sanguíneos principais da via de saída para dissecar o coração fora da cavidade torácica.
- Colocar imediatamente cada coração para um tubo de centrífuga de 15 ml individuais contendo 10 ml de PBS arrefecido a 4 ° C.
- Transferir corações para separate 10 cm placas de Petri, utilizando um bisturi cortar cada coração em dois numa orientação sagital e lavar o coração apertando suavemente com uma pinça. Em seguida substituir o PBS para remover o máximo de sangue possível. Repita esta etapa duas vezes.
- Coloque as duas partes de cada coração para o mesmo indivíduo 1,5 ml tubo de microcentrífuga.
- Continue até a etapa 3 ou alternativamente espécimes armazenar -80 ° C, conforme detalhado nas etapas 2.9 e 2.10.
- Usando uma agulha G 25, faça um pequeno orifício na tampa do tubo de microcentrífuga para impedir que a tampa da abertura, devido à expansão dos gases quando o coração é processada.
- Rotular cada tubo de microcentrífuga e colocar em um recipiente de nitrogênio líquido para congelar.
3. Núcleos dissociação e Cardiomyocyte Núcleos Labeling
Nota: Este núcleos rotulagem de dissociação e cardiomiócitos núcleos é uma adaptação do que de Bergmann et al 11 Execute este procedimento em todas as amostras injetadas.com o Edu e soro fisiológico injetado (sem controlo da UDE) controle negativo. Ver Tabela 1 para a receita de soluções necessárias para executar estes passos.
- Para cada coração a ser analisado, colocar 36 ml de BSA a 1% / solução de PBS para um tubo de ultracentrífuga, incubar durante 30 minutos, rejeitar a solução e em seguida deixar secar ao ar.
Nota: Este tubo é usado no passo 3.6. - Coloque corações congelados individuais em um prato de 10 mm no gelo e tritura utilizando um bisturi permitindo que os corações para descongelar durante o processo de trituração.
- Coloque corações picadas em 15 ml de tampão de lise celular num tubo de centrífuga de 50 ml e homogeneizar com um homogeneizador de sonda durante 15 segundos a 25.000 rpm, à temperatura ambiente.
- Adicionar mais 15 ml de Tampão de Lise Celular para o tubo de centrífuga e transferir para um Dounce de 40 ml com uma grande folga pilão. Execute 10 cursos com o pilão e filtrar através de 100 mm, em seguida, 40 coador mm de células em um tubo de centrífuga de 50 ml.
- Centrifugar durante 10 minutos, umat 700 xg, a 4 ° C e remover o sobrenadante.
- Ressuspender o sedimento nuclear em 25 ml de sacarose Solução de gradiente e a camada de núcleos de células que contêm a solução no topo de 10 ml de solução de gradiente de sacarose fresca no tubo de ultracentrífuga preparado em 3.1.
- Centrifuga-se a 13000 xg durante 1 h a 4 ° C utilizando um rotor de balanço do tubo para fora.
- Após a centrifugação, desprezar o sobrenadante e ressuspender o sedimento de núcleos em 1300 ul de tampão de armazenagem os núcleos num tubo de microcentrífuga de 1,5 ml e rótulo PCM1.
- Retirar 300 ul da suspensão para um novo tubo de microcentrifugação e adicionar aos 700 ul de tampão de armazenamento núcleos fresco. Rotular este tubo como controle ISO.
- Adicionar anti-PCM1 a uma concentração final de 8 ug / ml para o tubo de microcentrífuga rotulado PCM1 e adicionar anticorpo de controlo de isotipo IgG de coelho a uma concentração de 8 ug / ml para o tubo de microcentrífuga rotulado controlo ISO.
- Incubar durante a noite a 4 ° C. Seguindo incubção, centrifugar a 700 xg durante 10 min, descartar o sobrenadante, substitua com 1 ml de tampão de armazenamento núcleos fresco, ressuspender e centrifugar de novo durante 10 min a 700 x g. Descartar o sobrenadante e substituir com 1 ml de tampão de armazenamento núcleos fresco.
- Adicionar 1 ml de F (ab ') 2 de cabra anti-coelho IgG (H + L) anticorpo (FITC ou corante verde-fluorescente equivalente) para obter uma concentração final de 2 ug / ml.
Nota: O uso de um F (ab ') 2 um fragmento de anticorpo diminui o potencial para a marcação não-específica das células imunitárias, incluindo macrófagos e células B. - tubos Envoltório em folha de alumínio para proteger da luz e incubar durante 1 hora a 4 ° C.
4. Detecção de Edu Incorporação
- Diluído 10x concentrada EdU tampão de reacção de 1:10 com água deionizada.
- Centrifugar o PCM-1 e ISO controlo núcleos marcados suspensões a 700 xg durante 10 min, descartar o sobrenadante e ressuspender o sedimento de núcleos em 1 ml deBSA a 1% / solução de PBS. Lava-se a pelete duas vezes núcleos.
- Após a lavagem final, descartar o sobrenadante e substituir com 100 ul de EdU fixador.
- Enrole tubos em folha de alumínio para proteger da luz e incubar à temperatura ambiente durante 15 min.
- Lavam-se as amostras duas vezes com 1 ml de BSA a 1% / solução de PBS, centrifugação a 700 xg e incubar com uma solução de permeabilização à base de saponina 1x à temperatura ambiente durante 15 min.
- Prepare 1x cocktail rotulagem EdU (Tabela 2). Um mínimo de 2 reacções serão obrigados a incluir o controlo não EdU.
- Adicionar 500 ul de 1x cocktail EdU rotulagem directamente a cada amostra já contendo núcleos em 100 ul de solução de permeabilização à base de saponina 1x e incubar à temperatura ambiente protegida da luz durante 30 min.
- Centrifuga-se a 700 x g e ressuspender em 1 ml de 1% de BSA / PBS e repetir este passo mais duas vezes.
- Centrifugar a 700 xg, elimine o sobrenadante e substituir por 400 ml de solução de coloração de ADN (ver Tabela de Materiais).
| Nome do reagente |
| 1. Tampão de Lise Celular |
| 0,32 M de sacarose |
| mM de Tris-HCl a 10 (pH = 8) |
| 5 mM de CaCl2 |
| acetato de magnésio 5 mM |
| EDTA 2,0 mM |
| 0,5 mM de EGTA |
| 1 mM de DTT |
| 2.Sucrose Solution Gradiente |
| 2.1 M de sacarose |
| mM de Tris-HCl a 10 (pH = 8) |
| acetato de magnésio 5 mM |
| 1 mM de DTT |
| 3. O tampão de armazenamento Núcleos (NSB) |
| 0,44 M de sacarose |
| mM de Tris-HCl a 10 (pH = 7,2) |
| KCl 70 mM |
| 10 mM de MgCl2 |
| espermina 1,5 mM |
Tabela 1: Núcleos ingredientes tampão isolamento Receita para todos os buffers usados na seção de protocolo 3 (Núcleos dissociação e núcleos de cardiomiócitos rotulagem)..
5. análise citométrica de fluxo
Nota: Executar A citometria de fluxo em núcleos isolados como descrito anteriormente 7,11,15.
- Em primeiro lugar, criar um gráfico descrevendo Forward Scatter (FSC) e Dispersão Lateral (SSC) para permitir a discriminação de núcleos de detritos (Figura 2A).
- Criar um plano para discriminar núcleos individuais de agregados (Figura 2B) traçando 4 ', 6-diamidino-2-phenylindole) DAPI) área ((3-405 / 450/50-A) versus altura (33-405 / 450 / 50-H). Certifique-that da-A 50 de sinal 3-405 / 450 / é recolhido na escala linear para permitir a correta determinação do teor de DNA.
- Crie um gráfico descrevendo 488/535/30-A (PCM-1) vs SSC e exibir apenas os eventos a partir do portão singlet criado em 5.2, a fim de avaliar PCM-1 expressão na população singlet.
- Executar a amostra de controlo de isotipo, de modo a estabelecer um portão positivo PCM-1 (Figura 2C). Executar uma pequena quantidade de PCM1 marcado amostra para verificar o PCM1 expressando a posição da população e gating.
- Crie um gráfico descrevendo SSC-A vs 405/450/50-A (para detectar DAPI). Neste enredo, exibição somente singlet PCM1 núcleos expressando, usando as portas criadas em 5.3. Utilize este enredo para criar um portão hierárquica adicional que contém apenas 2N núcleos (Figura 2D).
- Criar uma trama que descreve 488/535 / 30-A vs 1-633 / 660/20-A para permitir a identificação de rotulagem EdU do PCM1 expressar população de cardiomiócitos. Exibir apenas núcleos que são singlet, P CM1 + 2N e usando o gating acima.
- Amostra coração executado a partir de qualquer controlo EdU para definir o portão positivo EdU (Figura 2E). Criar o portão apropriado para células Edu +.
- Record e quantificar singlet,, núcleos 2N PCM-1 expressando que incorporaram EdU.
- Calcule esta população como uma percentagem do total de camisola, PCM1 + núcleos. Isto irá fornecer a taxa de geração de núcleos neo-cardiomiócito, durante o período de impulso EdU 7 dias, como uma percentagem do total dos núcleos dos cardiomiócitos.
Observação: Como DAPI ligação ao ADN é estequiométrica a intensidade de fluorescência é proporcional à quantidade de DNA. Determinar ploidia com base na intensidade do / 450/50 Um sinal 405, como descrito anteriormente 7. - Para avaliar a ploidia dentro da população total de cardiomiócitos utilizar a trama estabelecida em 5,5 e uma estratégia de propagação com base na concentração de ADN 7.
/files/ftp_upload/53979/53979fig2.jpg "/>
Figura 2:. Gating estratégia para quantificar cardiomiócitos EdU incorporação e ploidia (A) Núcleos são discriminados de detritos com base na dispersão para a frente (FSC) e dispersão lateral (SSC). (B) Um portão linear é criada e a população de núcleos singuleto é identificado com base na rotulagem DAPI e a altura 405/450/50 vs sinal área. (C) gating fluorescente permite a separação de núcleos de cardiomiócitos núcleos (PCM-1-positivo) e não-cardiomiócitos (PCM-1-negativas) a partir de tecido do coração. (D) A intensidade de sinal de DAPI é usado para determinar a concentração de ADN e, assim, os núcleos de ploidia da população de cardiomiócitos PCM1 +. No rato> 80% dos núcleos dos cardiomiócitos são diplóides (2n). (E) gating fluorescente permite a separação de 2N, os núcleos dos cardiomiócitos que incorporaram EdU (PCM + / + EDU = 0,9%) a partir de 2N, cardiomiócitos que não foram incorporados EdU (PCM + educação). Todos os passos são detalhados no protocolo 5.0. Exemplo mostrado de MDX -. / Mice y no fundo C57 / BL10 Por favor clique aqui para ver uma versão maior desta figura.
Resultados
This method allows the quantification of increased EdU incorporation due to cardiomyocyte nuclei division while controlling for increased polyploidization. Using a BrdU based assay, we previously demonstrated that the mammalian heart responds to chronic cardiomyocyte loss, in the mdx mouse model of Duchenne muscular dystrophy, with the regeneration of new cardiomyocytes. We have used the methods described here to further validate our published findings and to demonstrate the utility of the protocol. As per the protocol described; adult (12 week old) mdx and control bl10 mice were pulsed with one daily injection of EdU for 7 days, reported previously to allow statically analysis between experimental groups5,13. Immunohistological analysis of pulsed hearts demonstrates the presence of PCM1 expressing cardiomyocytes which have incorporated EdU (Figure 3A). However, the data obtained using immunohistological methods can be misinterpreted, as other cell types which have incorporated EdU can be misidentified as cardiomyocytes. An example of this is demonstrated in Supplementary Figure 1A and B. Furthermore immunohistological methods do not distinguish polyploidization from nuclear division. The flow cytometry analysis detailed in this protocol enables the rapid quantification of cardiomyocyte nuclear division in mdx and control mice, while excluding cells which had incorporated EdU due to polyploidization. Similar to previously published data13, analysis revealed an increase in the rates of neo-cardiomyocyte nuclei generation in the mdx mouse hearts compared to age matched controls (1.2% versus 0.17%; Figure 3B and C).
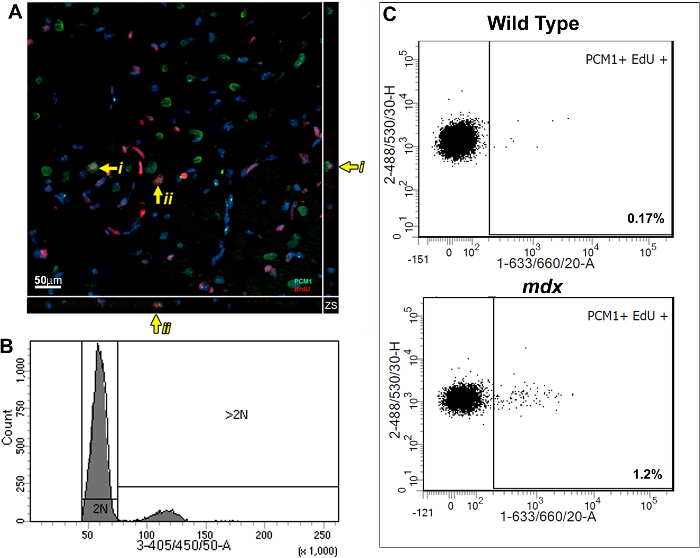
Figure 3: Quantification of EdU labeled cardiomyocytes in wildtype and mdx hearts. (A) Image of Z-stack projections taken from 40 µm thick sections of mdx hearts. PCM-1 expressing cardiomyocyte nuclei (green) which has incorporated EdU (red). Yellow arrow indicates EdU labeled cardiomyocytes. i and ii indicates individual EdU labeled cardiomyocytes shown in the Z-plane. Nuclei labeled with DAPI (blue) (B and C) Flow cytometry of isolated nuclei showing representative plots from 12-13 week old wild type (C57/BL10) and mdx mice (mdx-/y on the C57/BL10 background). (B) Histogram showing DAPI intensity and discrimination between 2N and >2N nuclei. (C) Plots showing EdU incorporation in the 2N cardiomyocyte nuclei population when analyzed as detailed in this protocol. For all mice Edu was administered for 1 week from 12 weeks of age. Please click here to view a larger version of this figure.
Supplementary Figure 1: PCM-1 is a specific marker for cardiomyocyte nuclei. Using the protocol described nuclei were isolated from the double transgenic mouse produced by crossing TNT-cre mice14 with the ROSAnT/nGcre sensitive reporter line16. In the resulting mouse cre activation under the control of Troponin T (TNT) promoter leads to irreversible expression of GFP with is localized to the nuclei. Nuclei labeled with Isotype control (Iso) demonstrates the ability to identify the GFP expressing cardiomyocyte nuclei population (488/530/30-A). Labeling of nuclei with anti-PCM1 (secondary antibody detected by 633-660/20-A) demonstrates the correlation of PCM1 expression with the GFP expressing cardiomyocyte nuclei population. In this example 98.8% of cardiomyocyte nuclei as identified by TNT promoter activity, are labeled by PCM1 antibody. Please click here to download Supplementary Figure 1.
Supplementary Figure 2: Challenges for quantifying cardiomyocyte renewal by immunohistological techniques. (A) 2D Image shows two PCM-1 expressing cardiomyocyte nuclei (green) which appear to be also labeled for EdU (red). Yellow arrows indicate what appears to be cardiomyocytes which have incorporated EdU. This is due to the apparent co-localization of PCM1 expression and EdU labeling when visualized using 2-dimentional imaging. (B) A 3-dimensional projection of this image identifies the EdU labeled cells are not cardiomyocytes but are non-cardiomyocyte cells overlying PCM1 expressing cardiomyocyte nuclei. C57/BL10 mice at 12 weeks of age used for experiments. Please click here to download Supplementary Figure 2A. Please click here to download Supplementary Figure 2B.
| Reaction Components | Number of reactions | |
| 2 | 5 | |
| PBS | 875 µl | 2.19 ml |
| Copper protectant | 20 µl | 50 µl |
| Fluorescent dye picolyl azide | 5 µl | 12.5 µl |
| Diluted reaction buffer prepared in 4.1 | 100 µl | 250 µl |
| Total reaction volume | 1 ml | 2.5 ml |
Table 2: EdU cocktail ingredients. Recipe for EdU labeling cocktail required in protocol section 4 (Detection of EdU incorporation).
Discussão
To accurately quantify cardiomyocyte turnover and regeneration assays must distinguish between true cardiomyocyte generation and nonproductive DNA division. Many studies continue to simply ignore these nonproductive events, quantifying cardiomyocyte proliferation solely via the expression of cyclin kinesis and cell cycle markers. To date a single method that allows the accurate quantification of cardiomyocyte turnover while controlling for these nonproductive events remains allusive. In particular it remains difficult to account for cardiomyocyte polyploidization which contributes up to ~65% of cardiomyocyte DNA replication13. Therefore to assist in the accurate quantification of cardiomyocyte generation we have developed a protocol that allows the robust quantification of the rates of neo cardiomyocyte nuclei while excluding DNA replication that results in increased ploidy. Although this protocol cannot discrimination between neo-cardiomyocyte generation and cardiomyocyte bi-nucleation, it can be used rapidly and accurately calculate the upper limit (accounting for ploidy) of cardiomyocyte generation. This protocol therefore provides a screening tool to assess potential changes in the rates of cardiomyocyte generation and polyploidization in disease models or to evaluate the potential efficiency of therapeutics. Once changes in the rate of neo-cardiomyocyte nuclei generation are identified using this protocol subsequent studies can be used to ascertain if this due to changes in cardiomyocyte generation of cardiomyocyte nucleation number, as described previously2,13,17,18. These include the use of histological quantification cardiomyocyte nucleation dynamics during the pulse period or analyses of tissue sections obtained from EdU pulsed animals in to compare EdU incorporation in the mononucleated and multinucleated cardiomyocyte populations.
Due to the low levels of cardiomyocyte turnover this protocol uses multiple injections of EdU over a 7 day period. This also allows the "chasing" of all potential cellular sources of cardiomyocyte generation and permits quantification of cumulative cardiomyocyte nuclei generation over this time period. Depending on the study, this timeframe may be adjusted to suit the predicted levels of cardiomyocyte generation. For the accurate quantification of EdU incorporation in cardiomyocyte nuclei, it is imperative that there is no non-specific labeling of the nuclei with the secondary antibody used to detect PCM-1 reactivity. It would therefore be prudent to undertake additional secondary antibody titration experiments in order to optimize this aspect of the protocol, particularly if a secondary antibody other than that suggested in this protocol is to be used. The protocol described here uses PCM-1 expression to identify cardiomyocyte nuclei. While this is an established cardiomyocyte marker, alternative markers can be used to validate data; these include antibodies specific for cardiac Troponin T which has been identified as partly localized in cardiomyocyte nuclei1. Similarly, alternative nuclear localized proteins may be used to identify and quantify EdU incorporation in nuclei populations other than that of the cardiomyocytes. It is important that all cardiomyocyte nuclei that are actively undergoing mitosis are excluded from the analysis, as the fate of this DNA synthesis is unknown and may result in either cell division or increased ploidy. PCM1 is disassembled during the M phase of the cell cycle therefore cardiomyocytes undergoing mitosis will not be identified by PCM1 expression. In addition, all nuclei in the s phase of the cell cycle should be excluded from subsequent analysis. This can be achieved by gating out all nuclei with a DAPI intensity above the 2N population including those with a DAPI intensity between the 2N and 4N populations.
Although it is increasingly accepted that the heart has the capacity to replace cardiomyocytes during normal aging and following acute injury, the source and degree of this potential remains controversial. In addition, disparate rates of cardiomyocyte turnover have been reported1,7,20-22. This may be in part due to the difficulties in accurately identifying and quantifying neo-cardiomyocyte generation19. To date the majority of studies have relied only on the use of histological analysis and the identification of cardiomyocytes via the expression of cytoplasmic proteins, including proteins of the sarcomere, for the quantification of cardiomyocyte turnover and renewal2,4,23,24. The use of these methods to detect the expression of proliferation markers, or as demonstrated here, the incorporation of thymidine analogues can easily result in the misidentification of other cardiac cell types as cardiomyocytes. While the use of 3D confocal imaging can help to alleviate these problems these methods are expensive and time consuming. Interestingly, the protocol described here demonstrates neo-cardiomyocyte nuclei generation occurs at a rate of 0.17% per week. This is consistent with other flow cytometry based studies demonstrating weekly turnover rates of up to 0.13%5. Although it is tempting to extrapolate annual turnover rates based on this data, as in previous studies2,5,25,26, this is inappropriate as the rates of turnover are dynamic during life time of an animal13.
This method has a number of potential applications including assessing the potential therapeutics to enhance cardiomyocyte regeneration or investigating the effects of cardiac disease on cardiomyocyte turnover and the rates of cardiomyocyte polyploidization.
Divulgações
The authors have nothing to disclose.
Agradecimentos
This work was supported by the British Heart Foundation, project grant PG/13/69/30454.
Materiais
| Name | Company | Catalog Number | Comments |
| 0.32 M sucrose | Sigma | 84100 | |
| 10 mM Tris-HCl (pH = 8) | Sigma | T3253 | |
| 5 mM CaCl2 | Sigma | c5086 | |
| 5 mM magnesium acetate | sigma | M-5661 | |
| 2.0 mM EDTA | Sigma | E5134 | |
| 0.5 mM EGTA | Sigma | 63779 | |
| 1 mM DTT | Sigma | D0632 | |
| 70 mM KCl | Sigma | P9541 | |
| 10 mM MgCl2 | Sigma | M8266 | |
| 1.5 mM spermine | Sigma | 85590 | |
| Isotype rabbit IgG- ChIP Grade | abcam | abc7415 | |
| Rabbit anti-PCM-1 antibody | Sigma | HPA023374 | |
| Alexa Fluor 488 F(ab')2 Fragment of Goat Anti-Rabbit IgG (H+L) Antibody | Life technologies | A-11070 | |
| cell strainers 70 μm and 100 μm | Fisher scientific | 11597522, 11517532 | |
| Glass dounce (40 ml) and pestle large clearance | Sigma | D9188-1SET | |
| EdU (5-ethynyl-2’-deoxyuridine) | Life technologies | A10044 | |
| Click-iT Plus EdU Alexa Fluor 647 Flow Cytometry Assay Kit | Life technologies | C10634 | This kit inlcudes reagents required for section, EdU reaction buffer, EdU fixative, saponin-based permeabilization solution and the reagents required for the EdU labelling cocktail. |
| CyStain DNA 2 step kit, | Sysmex Partec | 05 5005 | This kit inlcudes reagents required for DAPI labelling (DNA staining solution) |
| Probe homogeniser, e.g., TissueRuptor | Qiagen | 9001273 | |
| TissueRuptor Disposable Probes | Qiagen | 990890 | |
| ultracentrifuge | Sorvall | ||
| Facscanto II | BD Biosciences | ||
| Ultracentrifuge Tube, Thinwall, Polypropylene. 38.5 ml, 25 x 89 mm | Beckman Coulter | 326823 | |
| Bovine serum albumin | Sigma | A2153 |
Referências
- Bergmann, O., et al. Evidence for cardiomyocyte renewal in humans. Science. 324, 98-102 (2009).
- Senyo, S. E., et al. Mammalian heart renewal by pre-existing cardiomyocytes. Nature. 493, 433-436 (2013).
- Soonpaa, M. H., Rubart, M., Field, L. J. Challenges measuring cardiomyocyte renewal. Biochim Biophys Acta. 1833, 799-803 (2013).
- Loffredo, F. S., Steinhauser, M. L., Gannon, J., Lee, R. T. Bone marrow-derived cell therapy stimulates endogenous cardiomyocyte progenitors and promotes cardiac repair. Cell Stem Cell. 8, 389-398 (2011).
- Malliaras, K., et al. Cardiomyocyte proliferation and progenitor cell recruitment underlie therapeutic regeneration after myocardial infarction in the adult mouse heart. EMBO Mol Med. 5, 191-209 (2013).
- Hsieh, P. C., et al. Evidence from a genetic fate-mapping study that stem cells refresh adult mammalian cardiomyocytes after injury. Nat Med. 13, 970-974 (2007).
- Bergmann, O., et al. Identification of cardiomyocyte nuclei and assessment of ploidy for the analysis of cell turnover. Exp Cell Res. 317, 188-194 (2011).
- Herget, G. W., Neuburger, M., Plagwitz, R., Adler, C. P. DNA content, ploidy level and number of nuclei in the human heart after myocardial infarction. Cardiovasc Res. 36, 45-51 (1997).
- Scholzen, T., Gerdes, J. The Ki-67 protein: from the known and the unknown. J Cell Physiol. 182, 311-322 (2000).
- Carmena, M., Earnshaw, W. C. The cellular geography of aurora kinases. Nat Rev Mol Cell Biol. 4, 842-854 (2003).
- Bergmann, O., Jovinge, S. Isolation of Cardiomyocyte Nuclei from Post-mortem Tissue. J. Vis. Exp. , e4205 (2012).
- Gilsbach, R., et al. Dynamic DNA methylation orchestrates cardiomyocyte development, maturation and disease. Nat Commun. 5, 5288 (2014).
- Richardson, G., Laval, S., Owens, W. A. Cardiomyocyte regeneration in the mdx mouse model of non-ischemic cardiomyopathy. Stem Cells Dev. , (2015).
- Jiao, K., et al. An essential role of Bmp4 in the atrioventricular septation of the mouse heart. Genes Dev. 17, 2362-2367 (2003).
- Bergmann, O., et al. Cardiomyocyte renewal in humans. Circ Res. 110, e17-e18 (2012).
- Prigge, J. R., et al. Nuclear double-fluorescent reporter for in vivo and ex vivo analyses of biological transitions in mouse nuclei. Mamm Genome. , (2013).
- Naqvi, N., et al. A proliferative burst during preadolescence establishes the final cardiomyocyte number. Cell. 157, 795-807 (2014).
- Liu, Z., Yue, S., Chen, X., Kubin, T., Braun, T. Regulation of cardiomyocyte polyploidy and multinucleation by CyclinG1. Circ Res. 106, 1498-1506 (2010).
- Ang, K. L., et al. Limitations of conventional approaches to identify myocyte nuclei in histologic sections of the heart. Am J Physiol Cell Physiol. 298, C1603-C1609 (2010).
- Kajstura, J., et al. Myocyte turnover in the aging human heart. Circ Res. 107, 1374-1386 (2010).
- Kajstura, J., et al. Cardiomyogenesis in the adult human heart. Circ Res. 107, 305-315 (2010).
- Walsh, S., Ponten, A., Fleischmann, B. K., Jovinge, S. Cardiomyocyte cell cycle control and growth estimation in vivo--an analysis based on cardiomyocyte nuclei. Cardiovasc Res. 86, 365-373 (2010).
- Anversa, P., Leri, A., Kajstura, J. Cardiac regeneration. J Am Coll Cardiol. 47, 1769-1776 (2006).
- Gonzalez-Valdes, I., et al. Bmi1 limits dilated cardiomyopathy and heart failure by inhibiting cardiac senescence. Nat Commun. 6, 6473 (2015).
- Kimura, W., et al. Hypoxia fate mapping identifies cycling cardiomyocytes in the adult heart. Nature. 523, 226-230 (2015).
- Soonpaa, M. H., Field, L. J. Assessment of cardiomyocyte DNA synthesis in normal and injured adult mouse hearts. Am J Physiol. 272, H220-H226 (1997).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados