É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Identificação de ácidos graxos no
Neste Artigo
Resumo
We propose a protocol to identify fatty acids without the need to purify them. It combines information on the retention times with the mass spectra of three types of fatty acid derivatives: fatty acid methyl esters (FAMEs), 4,4-dimethyl oxazoline derivatives (DMOX), and 3-pyridylcarbinyl esters (picolinyl).
Resumo
As espécies de Bacillus contêm cadeia ramificada e ácidos graxos insaturados (FAS), com diversas posições do ramo de metilo (ISO ou anteiso) e da ligação dupla. Alterações na composição da FA desempenha um papel crucial na adaptação de bactérias ao seu ambiente. Estas modificações implicar uma mudança na proporção de iso contra anteiso ramificada CC, e na proporção de CC insaturado em relação ao FAS saturado, com ligações duplas em posições específicas criadas. Identificação precisa do perfil FA é necessário compreender os mecanismos de adaptação de espécies de Bacillus.
Muitas das FAs de Bacillus não estão comercialmente disponíveis. A estratégia aqui proposto identifica CC através da combinação de informação sobre o tempo de retenção (por cálculo do comprimento de cadeia equivalente (ECL)) com o espectro de massa de três tipos de derivados FA: ésteres metílicos de ácido gordo (FAMEs), 4,4-dimetil-oxazolina derivados (DMOX), eéster de 3-pyridylcarbinyl (picolinilo). Este método pode identificar os CC sem a necessidade de purificar o CC desconhecido.
Comparando os perfis cromatográficos de FAME preparado a partir de Bacillus cereus com uma mistura comercial de padrões permite a identificação de cadeia linear-CC, o cálculo da ECL, e saturada hipóteses sobre a identidade da outra FAs. FAMEs saturado ramificado de CC, iso ou anteiso, exibir uma mudança negativa constante no ECL, em comparação ao FAS lineares saturados com o mesmo número de carbonos. FAMEs do FAS insaturado pode ser detectada pela massa dos seus iões moleculares, e conduzir a uma mudança positiva na LCE em relação ao FAS saturados correspondentes.
A posição de ramificação de AF e a posição da ligação dupla CC insaturado pode ser identificado por espectros de massa de ionização de electrões de derivados de picolinilo e DMOX, respectivamente. Esta abordagem identifica todo o ramo saturado desconhecidoed FAs, insaturados de cadeia linear FAs e AF ramificados insaturados a partir do extrato de B. cereus.
Introdução
cromatografia de ácido gordo metil éster (FAME) gasosa (GC) é um método essencial para a caracterização de lípidos. Ele separa rapidamente e quantifica os vários ácidos gordos (FAS) de uma amostra após um curto passo de extracção. Os derivados de ésteres de metilo são altamente voláteis, estável e inerte no sentido da coluna cromatográfica, evitando desse modo os picos de decantação. A sua identificação é bastante simples quando a amostra é constituída por FAs bem conhecidas porque os perfis cromatográficos são publicados nem em relação aos padrões. Além disso, a injecção repetida de padrões de calibração para a quantificação de diferentes FAs não é necessário, devido à sua resposta quase constante para a detecção de ionização de chama (FID) 1.
Além FID, espectrometria de massa de detecção (MS) fornece um conjunto complementar de informações para confirmar FAMEs. No entanto, quando EMAGs são cobrados através de ionização de elétrons (EI), os espectros resultantes nem sempre permitem the identificação de estrutura fina FA. Por exemplo, a posição de ramificação (isto é, um grupo metilo ramificado) é difícil prever porque os iões de diagnóstico são difíceis de detectar uma característica e a alteração na abundância de iões alvo é dependente da máquina, evitando a utilização de espectros de massa bibliotecas 2. Outro desafio está em identificar a posição ligação dupla porque EI provoca a migração ligação dupla. Assim, isómeros FA com diferentes posições de ligação dupla não pode ser diferenciados pelo seu espectro de massa. Felizmente, outras ferramentas têm sido desenvolvidos para a identificação FA. Por exemplo, a presença e a posição de ramificação ou de ligações duplas em FAs pode ser conjecturado calculando o comprimento de cadeia equivalente (ECL) 3.
Outros métodos de derivatização resultar em diferentes espectros de massa, dependente da localização de uma ligação dupla ou um grupo metilo ramificado. 4,4-dimetil derivados oxazolina (DMOX) 4 permitem easY identificação da posição das ligações duplas de ácidos gordos monoinsaturados. 3-pyridylcarbinyl éster (éster picolinil) derivados de permitir uma identificação inequívoca da localização de metilo ramificado CC 5. Combinando retenção cromatográfica (ECL) e espectro de massa (DMOX e picolinil) informação permite a identificação da maioria dos CC sem a necessidade de utilizar métodos de complexos de purificação, tal como exigido para a ressonância magnética nuclear (RMN) do espectro, o método incontestável para a caracterização estrutural 1 .
As bactérias do gênero Bacillus, que incluem alguns patógenos humanos e animais, são capazes de colonizar muito diversos nichos e são, portanto, amplamente distribuído no ambiente 6. Entre o género Bacillus, a composição de FA é influenciada pela nicho ecológico das espécies com modulações nos padrões de FA para se adaptar a uma vasta gama de mudanças ambientais (por exemplo, meio de crescimento, temperatura,pH, etc.) 7-9. Devido à relativa homogeneidade do padrão de FA em várias espécies do género Bacillus durante o crescimento em condições padronizadas, a determinação da composição de FA é um dos critérios essenciais utilizados para definir as espécies de Bacillus. Um atributo exclusivo do gênero Bacillus é a abundância de FAs de cadeia ramificada contendo 12-17 carbonos 10-12 com a relação entre a ISO e Anteiso isômeros sendo um factor determinante da adaptação às condições ambientais. As espécies de Bacillus também adaptar-se a variações ambientais, alterando a proporção de ácidos gordos insaturados. Em algumas espécies, tais como Bacillus cereus, duas dessaturases ácidos graxos criar ligações duplas em diferentes posições da cadeia de alquilo 13 com diferentes papéis na adaptação 9. O exemplo do género Bacillus ilustra a importância de identificar precisamente a posição da ligação dupla e FA ramificação. coletarvamente, a identificação de padrões de Bacillus FA tem várias aplicações úteis. Aqui, nós propomos uma nova abordagem GC-MS para Bacillus FA identificação padrão que supera as limitações inerentes de uma análise de GC-MS clássica.
Esta abordagem inovadora pode ser utilizado directamente no material biológico em bruto, e é composto de uma combinação de técnicas existentes: informações sobre os tempos de retenção (ECL) e espectros de massa de derivados de diferentes FAs (FAME, DMOX e éster-picolinil).
Nós usamos a seguinte FA nomenclatura. I, A, e n indicam iso, Anteiso metil ramificada, e ácido gordo de cadeia linear, respectivamente. CC insaturadas foram nomeados por C: d em que C é o número de átomos de carbono no ácido gordo e d é o número de ligações duplas. Δ x indica a posição da ligação dupla, em que a ligação dupla está localizada na ligação carbono-carbono Xth, contando a partir da extremidade de ácido carboxílico.
Protocolo
1. As culturas bacterianas
- Prepara-se uma grama de bactérias (Bacillus cereus ATCC 14579) estirpe espalhando 100 jil de uma cultura de uma noite da estirpe incubada a 30 ° C em meio LB (meio de Luria-Bertani), sobre a superfície de uma placa de meio LB com agar. Incubar a placa durante a noite a 30 ° C.
2. ECL: Comprimento Cadeia Equivalent
- Calcular ECL como se segue:
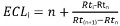 com:
com:
i, o soluto de interesse;
n, o número de carbono do éster de metilo de ácido gordo de cadeia linear saturado eluindo antes soluto I;
N + 1, o número de carbono do éster de metilo de ácido gordo saturado de cadeia linear eluindo depois de soluto I;
Rti, Rtn, Rt (n + 1) os tempos de retenção dos picos de FAME descritos acima.
NOTA: obter os tempos de retenção dos ésteres de ácidos gordos saturados de cadeia reta metilo por injecção de uma mistura de padrões (BAME).
3. FAME Preparação e Análise
- De forma a obter ácidos gordos de lípidos, células bacterianas colheita por raspagem das colónias das placas de agar e transferir 40 mg (peso fresco, equivalente a 10 células viáveis 9) de bactérias dentro de um tubo de vidro de 10 ml com tampas de rosca e vedantes de PTFE.
- Execute transesterificação via o éster método de ligação 14,15 detalhado abaixo.
- Adicionar 5 ml de 0,2 M de KOH em metanol às células bacterianas frescas e incubar a 37 ° C durante 1 h. Esta reacção consiste em metanólise alcalina, quebrar a ligação éster no lípido e a produção de ésteres metílicos de ácidos gordos.
- Adicionar 1 ml de ácido acético a 1 M para baixar o pH para 7,0. Verificar o pH com tiras de teste de pH.
- Adicionar 3 ml de hexano para extrair FAMEs.
- Transferir o sobrenadante (fase orgânica) para tubos limpos e concentra-se por evaporação à temperatura ambiente, sob um fluxo contínuo de azoto para se obter cerca de 200 ulde extrato. Transferir a amostra para um frasco de GC com inserção.
- Injetar extratos em uma espectrometria de cromatografia de massa de gás do sistema (GC-MS).
4. GC / Condições MS
- Injectar as amostras FAME para um instrumento de GC-MS equipado com uma coluna capilar ZB-WAX (comprimento, 30 m; diâmetro, 0,25 mm, espessura do filme, 0,25 um).
- Definir a porta de injecção (no modo de splitless) temperatura a 250 ° C. Use hélio como um gás de transporte, com uma velocidade linear de 37 cm / seg. Mantenha a temperatura do forno a 50 ° C durante 1 min, aumentar para 190 ° C a uma velocidade de 20 ° C / min, e aumentar ainda mais a uma temperatura final de 230 ° C a uma velocidade de 2 ° C / min.
- Para a MS, gravar os espectros de massa por ionização de electrão (El) a 70 eV, e definir a aquisição da corrente iónica total entre 50 e 400 unidades de massa atómica (amu) (2 leituras / seg).
- Quando necessário, injetar derivados DMOX e picolinil sob a mesma condição except o programa de temperatura do forno como se segue:
DMOX: 50 ° C (1 min), 20 ° C / min até 210 ° C e 2 ° C / min até 240 ° C (5 min);
Picolinilo: 6 ° C (1 min), 20 ° C / min até 220 ° C e 2 ° C / min até 250 ° C (20 min).
5. Preparação de éster picolinilo 16 FAME
- Evapora-se o extracto de FAME da secção 3 com um fluxo de azoto (pelo menos 10 mg de material seco) e dissolver em 1 ml de diclorometano seco.
- Prepara-se uma solução 1,0 M de ter-butóxido de potássio t em tetra-hidrofurano.
- Adicione o extrato de FAME e 0,2 ml de 3 piridinametanol a 0,1 ml de solução efectuadas no passo 5.2.
- Aquece-se a solução a 40 ° C durante 30 minutos num frasco fechado.
- Após arrefecimento até à temperatura ambiente, adicionar água purificada desionizada (2 ml, ver Tabela Materiais) e hexano (4 ml). Misturar com um vórtice, permitir fase para separar e recolher a fase orgânica.
- Dry-lo por adição de sulfato de sódio anidro, até a fase orgânica é perfeitamente claro. Transferi-lo para um tubo limpo. Em seguida, evapora-se a 200 ul. Transferir a amostra para um frasco de GC com inserção.
6. Preparação de FAME DMOX 17
- Evapora-se o extracto de FAME da secção 3 com um fluxo de azoto (material seco de pelo menos 10 mg).
- Ao extracto seco FAME, adicionar 250 mg de 2-amino-2-metil-1-propanol. Lavar o reactor com azoto, adicionar uma rolha, e colocá-lo em um bloco de aquecimento durante a noite a 190 ° C.
- Após arrefecimento até à temperatura ambiente, adicionar 3 ml de diclorometano para o tubo, e 5 ml de água purificada desionizada (Ver Tabela Materiais).
- Agitar por separação de fases e em seguida, remover a fase aquosa.
- Lava-se a fase orgânica com 5 ml de água. Agitar por separação de fases e em seguida, remover a fase aquosa.
- Seco por adição de sulfato de sódio anidro, até a fase orgânica é perfeitamente clara etransferi-lo para um tubo limpo. Evapora-se sob uma corrente de azoto até atingir um volume de 200 ul. Transferir a amostra para um frasco de GC com inserção.
Resultados
A estratégia de identificação FA a partir de células bacterianas é apresentado na Figura 1. Cada etapa fornece informações espectrais complementar ou informações sobre retenção cromatográfica. Passo 1 consiste de identificação preliminar FA usando uma solução padrão. Passo 2 permite a interpretação de espectros EI FAME e sua ECL, de forma a identificar os produtos tentativamente. Passo 3 identifica a localização exata de ramificação em cadeia ramif...
Discussão
Os perfis de cromatogramas FAs mostrados na Tabela 1 correspondem a B. cereus ATCC 14579 cresceram em uma superfície da placa de agar. Perfis semelhantes foram obtidos quando a bactéria foi cultivada em meio líquido gaseificado à mesma temperatura 8. No caso de bactérias cultivadas em meio líquido, a biomassa bacteriana é recolhido por centrifugação do meio de crescimento e podem ser lavados de acordo com protocolos anteriormente descritos, dependendo das condições de cres...
Divulgações
The authors declare that they have no competing financial interests.
Agradecimentos
Os autores são gratos a Thomas Mison por seu apoio técnico, e Rachel Kopec para a revisão do manuscrito.
Materiais
| Name | Company | Catalog Number | Comments |
| GC/MS | Shimadzu | QP2010 | |
| capillary column ZB WAX | Phenomenex | 7HG-G007-11 | 30 m x 0.25 mm x 0.25 µm |
| Methanol Lichrosolv | VWR | 1.06018.2500 | |
| potassium hydroxide | Aldrich | P1767 | |
| THF | Hipersolv Chromanorm | 28559.320 | |
| Dichloromethane | Hipersolv Chromanorm | 23373.320 | |
| Hexane | Hipersolv Chromanorm | 24575.320 | |
| 3-pyridinemethanol | Aldrich | P6-680-7 | |
| potassium tertiobutoxide | Aldrich | 156671 | |
| 2-amino-2-methyl-1-propanol | A-9879 | ||
| MilliQ Academic | Millipore | ZMQS50001 | |
| Bacterial Acid Methyl Ester (BAME) Mix | Sigma-Aldrich | 47080-U Supelco |
Referências
- Christie, W. W., Han, X. . Lipid Analysis 4th Edition. , (2010).
- HÜbschmann, H. -. J. . Handbook of GC-MS: fundamental and application. Third edition. , (2015).
- Sasser, M. Identification of Bacteria by Gas Chromatography of Cellular Fatty Acids. MIDI Technical note. 101, 1-6 (1990).
- Spitzer, V. Structure analysis of fatty acids by gas chromatography - Low resolution electron impact mass spectrometry of their 4,4-dimethyloxazoline derivatives - A review. Prog Lipid Res. 35 (4), 387-408 (1996).
- Harvey, D. J., Christie, W. W. . Advances in lipid methodology. Volume 1. , 19-80 (1992).
- Diomande, S. E., Nguyen-The, C., Guinebretière, M. -. H., Broussolle, V., Brillard, J. Role of fatty acids in Bacillus environmental adaptation. Front Microbiol. 6, (2015).
- Brillard, J., et al. Identification of Bacillus cereus Genes Specifically Expressed during Growth at Low Temperatures. Appl Environ Microbiol. 76 (8), 2562-2573 (2010).
- de Sarrau, B., et al. Influence of Anaerobiosis and Low Temperature on Bacillus cereus Growth, Metabolism, and Membrane Properties. Appl Environ Microbiol. 78 (6), 1715-1723 (2012).
- Diomandé, S. E., et al. Involvement of the CasK/R two-component system in optimal unsaturation of the Bacillus cereus fatty acids during low-temperature growth. Int J Food Microbiol. 213, 110-117 (2015).
- Berkeley, R. C. W., Heyndrickx, M., Logan, N., De Vos, P., Berkeley, R. C. W. . Applications and Systematics of Bacillus and Relatives. , 1-7 (2002).
- Kämpfer, P. Limits and Possibilities of Total Fatty Acid Analysis for Classification and Identification of Bacillus Species. System. Appl. Microbiol. 17 (1), 86-98 (1994).
- Kaneda, T. Fatty-acids of genus bacillus - example of branched-chain preference. Bacteriol Rev. 41 (2), 391-418 (1977).
- Chazarreta Cifre, L., Alemany, M., de Mendoza, D., Altabe, S. Exploring the Biosynthesis of Unsaturated Fatty Acids in Bacillus cereus ATCC 14579 and Functional Characterization of Novel Acyl-Lipid Desaturases. Appl Environ Microbiol. 79 (20), 6271-6279 (2013).
- Sasser, M., et al. Identification of Bacillus anthracis from culture using gas chromatographic analysis of fatty acid methyl esters. J AOAC Int. 88 (1), 178-181 (2005).
- Schutter, M. E., Dick, R. P. Comparison of fatty acid methyl ester (FAME) methods for characterizing microbial communities. Soil Sci Soc Am J. 64 (5), 1659-1668 (2000).
- Destaillats, F., Angers, P. One-step methodology for the synthesis of FA picolinyl esters from intact lipids. J Am Oil Chem Soc. 79 (3), 253-256 (2002).
- Fay, L., Richli, U. Location of double-bonds in polyunsaturated fatty-acids by gas-chromatography mass-spectrometry after 4,4-dimethyloxazoline derivatization. J Chromatogr. 541 (1-2), 89-98 (1991).
- Zhang, J. Y., Yu, Q. T., Liu, B. N., Huang, Z. H. Chemical modification in mass spectrometry IV-2-alkenyl-4,4-dimethyloxazolines as derivatives for the double bond location of long-chain olefinic acids. Biol Mass Spect. 15 (1), 33-44 (1988).
- de Sarrau, B., et al. Unsaturated fatty acids from food and in the growth medium improve growth of Bacillus cereus under cold and anaerobic conditions. Food Microbiol. 36 (2), 113-122 (2013).
- Miwa, T. K., Mikolajczak, K. L., Earle, F. R., Wolff, I. A. Gas chromatographic characterization of fatty acids.Identification constants for mono- and dicarboxylic methyl esters. Anal Chem. 32 (13), 1739-1742 (1960).
- van Den Dool, H., Kratz, P. A generalization of the retention index system including linear temperature programmed gas-liquid partition chromatography. J Chromatogr A. 11, 463-471 (1963).
- Stransky, K., Jursik, T., Vitek, A. Standard equivalent chain length values of monoenic and polyenic (methylene interrupted) fatty acids. J High Res Chromatogr. 20 (3), 143-158 (1997).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados