Method Article
De alta resolução Volume Imaging de neurônios pela exclusão da utilização da fluorescência método e dedicado Microfluidic dispositivos
Neste Artigo
Resumo
Volume é um parâmetro importante sobre células características fisiológicas e patológicas. Nós descrevemos um método de exclusão fluorescente permitindo a medição de campo integral em vitro neuronal volume com sub micrométrica resolução axial, necessária para a análise de neuritos e estruturas dinâmicas implicadas em crescimento neuronal.
Resumo
Volume é um parâmetro importante referentes às características fisiológicas e patológicas dos neurônios em escalas de tempo diferentes. Os neurônios são células bastante singular sobre suas morfologias ramificadas estendidas e consequentemente aumentar vários desafios metodológicos para a medição do volume. No caso particular de em vitro crescimento neuronal, a metodologia escolhida deve incluir a resolução axial sub micrométrica, combinada com a observação de campo integral em escalas de tempo de minutos a horas ou dias. Ao contrário de outros métodos, como a reconstrução de forma celular usando a imagem latente confocal, eletricamente com base em medições ou microscopia de força atômica, o método de exclusão de fluorescência recentemente desenvolvido (FXm) tem o potencial para atender a esses desafios. No entanto, apesar de ser simples em seu princípio, implementação de um FXm de alta resolução para os neurônios requer vários ajustes e uma metodologia dedicada. Apresentamos aqui um método baseado na combinação de exclusão de fluorescência, dispositivos microfluídicos de múltiplos compartimentos de baixa rugosidade e finalmente micropatterning para alcançar em vitro medições de volume neuronal local. A alta resolução fornecida pelo dispositivo nos permitiu medir o volume local de processos neuronais (neuritos) e o volume de algumas estruturas específicas envolvidas no crescimento neuronal, tais como cones de crescimento (GCs).
Introdução
O conhecimento preciso do volume celular tem atraído maior atenção nos últimos anos, impulsionada pelo problema da homeostase de tamanho de célula em microorganismos unicelulares 1 e, mais geralmente, em células mitóticas 2. No entanto, a questão do volume das células é pertinente também para células pós mitóticas, para o qual os neurônios constituem um exemplo paradigmático.
Volume é de fato uma importante assinatura de eventos fisiológicos e patológicos em diferentes escalas e pontos de tempo na vida neuronal, da deformação de axonal transitória associada à atividade elétrica (escala de milissegundos) 3 para o irreversível neuronal inchaço ocorrendo durante a fase assintomática da Neurodegenerativas Doenças (ao longo de anos em humanos) 4. No entanto, a maior alteração de volume ocorre em uma escala de tempo intermediário de dias ou semanas (dependendo do organismo considerado) durante o crescimento neuronal. A prolongada e complexa morfologia de neurônios levanta questões de múltiplos, entre os quais a regulação do tamanho da célula. Diâmetro e comprimento axonal são na verdade firmemente regulado na vivo, com valores específicos para cada tipo neuronal 5,6.
Estas questões, complexos de abordar na vivo, também podem ser abordadas de uma forma simplificada in vitro. Com esse objetivo, um método dedicado à medição de volume rápido suficiente para acompanhar a dinâmica de crescimento (ou seja, em uma escala de tempo de minutos) e compatível com a observação ao longo de horas ou dias é necessária. Vários métodos têm sido desenvolvidos ao longo dos anos para proporcionar um acesso directo ou indirecto ao volume celular in vitro. Célula de reconstrução de imagem latente confocal é um deles, mas este método implica a rotulagem e repetidas exposições à luz ao mesmo tempo mostrando uma resolução axial limitada de cerca de 500 nm 7. Note que estes dois últimos inconvenientes são parcialmente superados por um método mais sofisticado e mais recente chamado retículo microscopia de luz-folha 8. Microscopia de força atômica tem sido usado 9 , mas este método de varredura é por essência lenta e tediosa. Além disso, o contato físico com a célula requer pode interferir com a medição Considerando a extrema suavidade dos neurônios 10. Método indireto usando impedância ou ressonância têm sido empregadas para 11tipos de célula diferente, mas são insuficientes para estendido adesivas células como os neurônios.
Um dos métodos mais promissores baseia-se na medida do volume excluído das células em uma câmara de perto com um corante fluorescente. O método de exclusão de fluorescência (FXm) é simples no seu princípio, pois requer sem rotulagem e é apropriado para a imagem latente ótica rápido, a longo prazo, de populações de células com uma resolução axial potencialmente sub óptica. Mais precisamente, a resolução em z depende da intensidade de fluorescência máxima na cultura câmara (ou seja, na região desprovida de células) dividida pela faixa dinâmica da câmera, embora várias fontes de ruído limitam esta resolução final. Este método tem sido muito poderoso a seguir o volume da migração de células aderentes 12 ou para estudar a mudança de volume durante a mitose de células de mamíferos, como exaustivamente descrito em 13. No entanto, os neurônios constituem um desafio metodológico para FXm considerando sua extensa ramificação em processos sub micrométricas.
Apresentamos aqui um método que conduz para a fabricação das câmaras de FXm suaves acesso com alta precisão o volume e altura de ramificações neuronais e estruturas dinâmicas envolvidas em crescimento neuronal, como cones de crescimento.
Câmaras devem ter alturas semelhantes que o objeto para medir a fim de otimizar a resolução axial. Portanto, nós projetamos dispositivos diferentes do FXm caracterizados por câmaras de medição central de três alturas diferentes. O mais fino (3 µm de altura) dedica-se à medição do axônio: esta baixa altura exclui a soma, que permanecem na próximo câmara intermediária alta 15 µm. Câmaras de centrais mais espessas (10 a 12 µm) são suficientemente altas para acompanhar o crescimento de células inteiras. O dispositivo também inclui dois reservatórios localizados em ambos os lados da câmara central. Quatro furos de injeção (IH), portanto, são implementados e são designados como segue: a entrada e saída servem para introduzir as suspensões celulares no chip, Considerando que os dois outros alimentam os reservatórios.
Temos a primeira calibração fabricados lamelas para medições de altura, usando estruturas de fotorresiste da geometria conhecida. Nós então ter fotografado livre neurônios crescentes, mas também morfologicamente restrito neurônios em micropatterns de adesão.
Protocolo
O estudo foi realizado em conformidade com as diretrizes da Comunidade Europeia sobre o cuidado e o uso de animais de laboratório: 86/609/CEE. A finalidade da pesquisa e o protocolo são descritos no anexo ético de ERCadg projeto violoncelo, que foi aprovado e é regularmente revisado pelo ERCEA. Institut Curie facilidade animal recebeu licença #C75-05-18, 24/04/2012, reportando ao Comitê d'Ethique en matière d'expérimentation animale Paris Centre et Sud (número de registo nacional: #59).
1. a fabricação do molde
Nota: O molde inclui câmaras centrais e intermediárias, conectadas a uma entrada e uma saída, além de dois reservatórios (e enseadas) localizado em ambos os lados da câmara central.
- Use uma pinça plana para manipular wafers de silício 51 milímetros de diâmetro e transferi-los de um lugar para outro.
- Molde de silicone de câmara central
- Encha uma pipeta plástica de 2 mL com fotorresiste positivo. Posicionar a pipeta no centro de um wafer de silício de 51 mm de diâmetro e carregue seu reservatório até cobrindo cerca de 75% da superfície da bolacha com o fotorresiste. Rotação-casaco a 3000 rpm por 30 s.
- Transferir a bolacha da rotação-aplicador para uma placa de aquecimento, com uma temperatura de 100 ° C, durante 50 s.
- Transferi a bolacha da placa de aquecimento ao titular do substrato (chuck) do alinhador de máscara. Expor através da "máscara DRIE" (ver arquivos suplementares "masks_neuron_volume_chips.tiff" e "masks_neuron_volume_chips.dxf").
- Retire a bolacha o chuck depois mergulharia em um copo de 100 mL, cristalizando o prato que contém o desenvolvedor (diluição 1:4 em água desionizada). Liberar a bolacha e mantê-lo por 1 min, agitando continuamente e delicadamente o prato de cristalização.
Nota: Para salvar os reagentes, encha o prato de cristalização de 1 cm de altura no máximo. - Levar de volta a bolacha do desenvolvedor e mergulhe-o em um prato de cristalização 100 mL enchido com água desionizada para cerca de 10 s.Then lugar a bolacha em um papel absorvente e secá-lo com nitrogênio pressurizado usando uma arma de sopro de ar.
- Coloque a bolacha para 50 s sobre uma placa de aquecimento com uma temperatura de superfície de 115 ° C.
- Realizar profundo-Reactive íon Etching (DRIE) com os seguintes parâmetros (mais informações sobre a técnica DRIE podem ser encontradas nas referências 14 e 15); Passo de passivação: 50 sccm de C4F8, ele apoio fluxo 10 sccm, CP 10 W, indutivamente acoplado Plasma (ICP) 1500 W, total pressão 24 mbar, temperatura de 18 ° C; Passo de gravura: 100 sccm de SF6, ele apoio fluxo 10 sccm, CP 11 W, ICP 1500 W, total pressão 24 mbar, temperatura de 18 ° C; Tempo de passivação: 4 s; Tempo de gravação: 7 s; Processo de duração total: tipicamente 5 min para cerca de 10 µm gravado profundidade.
- Dissolva o fotorresiste por mergulhar o substrato um cristalização prato cheio de acetona.
- Mergulhe a bolacha em uma solução de piranha (2/3 de H2O2 (30%) e 1/3 de H2SO4 (95 %)) por 5 min.
Cuidado: Sempre adicione H2O2 primeiro e em seguida H24 e enxaguar pelo menos 3 vezes em água desionizada após a limpeza.
- Fabrica os moldes correspondentes às secções intermédias executando etapas 1.2.1 para 1.2.11 seguindo os parâmetros listados nas tabelas 1-3.
Nota: Cada tabela corresponde a um determinado dispositivo, caracterizado por uma altura específica da câmara de observação central.
Nota: Execute todo o processo usando crescente photoresists alturas (máscaras de 1 a 3 para cada dispositivo, consulte arquivos suplementares "masks_neuron_volume_chips.tiff" e "masks_neuron_volume_chips.dxf").- Coloque a bolacha de silício gravado sobre o titular de substrato de rotação-aplicador.
- Verificar que o spin-revestidor está funcionando corretamente confirmando que a aspiração do substrato é eficaz (o substrato deve girar e ficar no lugar durante a velocidade de rotação nominal).
- A viscosidade do SU-8 aumenta com a altura da faixa de espessura do alvo. Sempre use garrafas de 20-30 mL para armazenar SU-8 photoresists e derramá-lo sobre a bolacha antes de girar-revestimento (SU-8 pode ser demasiado viscosas para ser manipulado com uma pipeta de plástico).
- Introduzir o negativo do tipo epóxi fotorresiste SU-8 no centro do substrato até cobrindo cerca de 75% do wafer de silício e, em seguida, girar-revestimento usando os parâmetros indicados na linha "Spincoating" dos quadros.
- Lugar a bolacha revestida em uma placa de aquecimento para a duração e a temperatura indicada em Asse linha"suave".
- Monte a bolacha gravada e a máscara adequada"SU-8" o alinhador de máscara.
- Alinhar a bolacha gravada com a máscara usando as cruzes de alinhamento dedicado (tamanho típico de cruzes: 50 µm × 150 µm) projetado em cada máscara.
Nota: Duas cruzes em cada lado do dispositivo são suficiente (um no canto inferior esquerdo) e outro no canto superior direito. - Expor utilizando eu linha de alinhador a máscara (comprimento de onda de 365 nm) com a dose adequada de UV, como indicado na linha "Energia de exposição".
Nota: O tempo de exposição é calculada dividindo-se o específico de energia E de exposição para cada fotorresiste pelo poder eficaz da lâmpada UV, modulada pela absorção da máscara: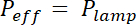 x (1 - absorçãomáscara). A absorção é cerca de 20% para máscaras flexíveis e insignificante para máscara de cromo duro.
x (1 - absorçãomáscara). A absorção é cerca de 20% para máscaras flexíveis e insignificante para máscara de cromo duro. - Coloque a bolacha revestida em uma placa de aquecimento para a duração e a temperatura indicada na linha "Post coze".
- Prepare duas 100 mL vidro-cristalização pratos, um contendo o desenvolvedor, o outro vazio. Mergulhe a bolacha no desenvolvedor durante o período indicado na linha "Desenvolvimento". Agite suavemente o prato cristalização ao longo do desenvolvimento
- Polvilhe a bolacha com isopropanol acima o prato vazio e cristalização por cerca de 5 s. Finalmente, coloque a bolacha em um papel absorvente e secá-lo com nitrogênio pressurizado usando uma arma de sopro de ar.
- Lugar a bolacha revestida em uma placa de aquecimento para a duração e a temperatura indicada em Asse linha"dura" (opcional).
Nota: Este passo é útil para evitar rachaduras no fotorresiste e fornecem uma superfície plana homogênea para os próximos passos. - Repita as etapas 1.3.1 - 1.3.10 para completar os processos listados na tabela 1-3.
- Após a última etapa de fotolitografia, silanize o mestre final do molde que consiste de 3 camadas de photoresists negativo por despachar dois 100 µ l as gotas de silano Trichloro(1H,1H,2H,2H-perfluoro-octyl) em cada lado do mestre em um prato de Petri-100mm. Selo do disco de Petri com uma película de plástico da parafina e incubar 20 min à temperatura ambiente (RT).
Nota: O molde mestre está pronto e pode ser utilizado várias vezes.
- Coloque a bolacha de silício gravado sobre o titular de substrato de rotação-aplicador.
2. fabricação do chip PDMS
- Despeje a 90 g de polímero orgânico à base de silicone (polidimetilsiloxano: PDMS) em um copo de plástico de 100 mL. Adicione 10 g de seu agente de cura (01:10 em peso). Agite a mistura usando uma pipeta plástica para 2-3 min.
Nota: Misture 90g de PDMS com 10 g de agente para a fabricação de 6 chips de cura. - Coloque a mistura dentro de um vácuo exsicador e bomba para cerca de 30 min para remover as bolhas de ar para o PDMS.
- Coloque o molde mestre em um prato de Petri P100 e despeje 15 mL da mistura sobre o molde usando uma seringa.
Nota: 15ml leva a uma altura total de microplaqueta de 1,5 mm. - Coloque a estrutura de PDMS-molde dentro de um vácuo exsicador e bomba até há não mais bolhas de ar explodindo na superfície do PDMS.
Nota: Este passo leva cerca de 30 min. - Empurre a válvula na parte inferior da caixa de Petri com uma ponta de cone para evitar volume morto de PDMS abaixo a bolacha. Coloque o dispositivo de PDMS-molde no forno conjunto a 70 ° C, durante pelo menos 2 h.
- Demold o bloco PDMS sob uma capa de química utilizando uma espátula plana de aço inoxidável e pelo isopropanol derramando no chip. Coloque-o sobre a bancada de aço inoxidável do exaustor.
- Corte em torno da pastilha de silício e demold réplica de PDMS utilizando um bisturi.
- Cortou o chip usando um bisturi ou uma lâmina de barbear (margem de deixar um 2-mm).
- Soco entradas premindo o perfurador de 1,5 mm de diâmetro com firmeza e accionar para cortar e esculpir o buraco da entrada. Faça o mesmo para as quatro zonas dedicadas do chip onde líquido será injetado.
- Limpe o chip por degola e descascando a fita adesiva no lado microstructured. Polvilhe o isopropanol em ambos os lados. Depois seque o chip com nitrogênio pressurizado usando uma arma de sopro de ar.
3. fabricação de lamelas padronizadas (24 × 24 mm2)
Nota: Manipule as lamelas com pinça curvada.
- Padrões de poli-ornitina (PLO)
- Aplica um O2 limpeza plasma sobre as lamelas de vidro. Parâmetros do plasma: bombeamento de pressão: 0.25 mbar; Ó2 alimentação duração: 3 min; fluxo de gás: 10 sccm; desvio máximo: ± 5 sccm; duração de plasma: 3 min; definir pressão: 0,36 mbar; desvio máximo: ±0.10 mbar; definir energia: 50 W; desvio máximo: 5%; duração de ventilação: 45 s.
- 484 Mix µ l de ácido acético e 56 µ l de 3-methacryloxypropyl-trimethoxysilane, completa com etanol absoluto para obter um volume total de 15 mL.
- Usando um cone de ponta de 1 mL, coloque uma gota de 500 µ l desta solução em cada lamela, esperar 2-3 min. seque usando um limpador de microfibra de salas limpas.
Nota: As lamelas de vidro silanizadas podem ser armazenadas 1 mês em temperatura ambiente dentro de caixas de plástico selado com um filme plástico de parafina. - Coloque cada lamela em um aplicador de rotação localizado em um ambiente de sala limpa. Coloque uma gota de um positivo fotorresiste cobrindo cerca de 75% da lamela (cerca de 500 µ l) e rotação-casaco a 4000 rpm por 30 s para chegar a uma espessura final de 0,45 µm.
- Lugar de lamelas para 1 min em uma placa, com uma temperatura de superfície de 115 ° C.
- Usando um alinhador de máscara, expor cada lamela no comprimento de onda de 435 nm (linha G) através da máscara dedicada de acordo com os parâmetros de fabricant (dose de UV sobre 50-60 mJ.cm–1)
- Prepare 2 pratos de cristalização de vidro, um contendo o desenvolvedor (sem diluição), o outro contendo água desionizada.
- Mergulhe um por um cada lamela no desenvolvedor por 1 min, agitando continuamente e delicadamente o prato de cristalização. Em seguida, mergulhe a bolacha na água desionizada para cerca de 5 s. lugar a bolacha em um papel absorvente e secá-lo com nitrogênio pressurizado usando uma arma de sopro de ar.
- Aplica um plasma de2 ativação O com os mesmos parâmetros como em 3.1.1.
- Sob o capô, depor quatro 170 µ l gotas de uma solução de 100 µ g/mL PLO por P100 placa de Petri. Coloque o rosto modelado das lamelas em cada um destas gotas. Selo da placa de Petri com uma película de parafina de plástico para evitar a secagem. Incubar durante a noite a RT
Nota: A solução de PLO deve permanecer anexada para as lamelas por capilaridade. - Preparar quatro destinatários (tipicamente P60 placas de Petri), três deles encher com PBS e o quarto com água desionizada. Prepare dois pratos de cristalização de vidro de etanol puro.
- Tirar cada lamela desde os pratos de Petri, mergulhe-o no primeiro banho de PBS por 10-15 s, evacuar o líquido sobre um tecido absorvente, colocando a lamela verticalmente no lado, inseri-lo com , por exemplo, o rosto modelado acima dentro do banho de álcool etílico.
Nota: Uma vez que a dissolução do fotorresiste é concluída, torna-se difícil localizar o lado padrão da lamela. Portanto, é importante nesta fase para rastrear sua localização. - Coloque o etanol cristalizar prato dentro de um sonicador de banho ultra-sônico (120 W / 35 kHz) e deixe o fotorresiste ser dissolvido por 3 min.
Nota: Mude o banho de etanol cada 4 lamelas para limitar a diluição pela PBS que pode afectar a dissolução do fotorresiste. - Pegue a lamela do banho de etanol e, em seguida, mergulhar é várias vezes o segundo banho de PBS. Verificar o aspecto de superfície repetidamente até a superfície gordurosa, como que os resultados do filme líquido remanescente de etanol desaparece.
- Mergulhe por 5-10 s a lamela o terceiro banho de PBS. Então imediatamente transferi-lo para o banho de água desionizada. Colocar a lamela sobre um papel absorvente e secá-lo com nitrogênio pressurizado usando uma arma de sopro de ar.
Nota: A última lavagem em água desionizada é usada para evitar a formação de cristais de PBS durante a etapa de secagem.
- Estruturas de fotorresiste para calibração de altura
- Execute apenas etapas 3.1.4. a 3.1.8. usando a máscara dedicada (máscara "Fotorresiste listras", veja o arquivo complementar "Mask_Photoresist-stripes.dxf").
4. implementação final e montagem da microplaqueta
- Chip de montagem em pratos de vidro inferior
- Colocar o chip PDMS e o prato de vidro no qual irá ser lig na câmara de plasma para a ativação de superfície. Parâmetros: bombeamento para baixo pressão: 0.25 mbar; Ó2 alimentação duração: 3 min; fluxo de gás: 10 sccm; desvio máximo: ± 5 sccm; duração de plasma: 30 s; definir pressão: 0.40 mbar; desvio máximo: ±0.10 mbar; definir energia: 50 W; desvio máximo: 5%; duração de ventilação: 45 s.
- Gentilmente colocou o chip PDMS ativado em contato com a lamela de vidro e delicadamente, aplique pressão nas bordas do chip a ligação do chip para a lamela. Para aumentar a força de ligação, coloque o dispositivo no forno a 70 ° C durante 5 a 10 min.
Nota: Não pressione as peças contendo pilares, eles podem entrar em colapso sob muita pressão. - Sob o capô (ou seja, em RT) e dentro de 30 minutos após a ligação, coloque um cone de ponta de 10 µ l preenchido com uma solução de 100 µ g/mL PLO no IHs e, em seguida, injetar o líquido. Ajuste o volume, a fim de formar uma gota na parte superior de cada IHs. Em seguida, usando um cone de ponta de 1 mL, adicione PBS na prato de Petri todo o chip.
- Deixe o chip no RT com um mínimo de tempo de incubação de 2 h. Para incubação durante a noite, sele a placa de Petri com um filme plástico de parafina para evitar a secagem.
Nota: O líquido não deve vazar fora senão o chip, que o chip deve ser Descartado. - Pressione um cone de ponta de 10 µ l levemente em cada IH e absorver o excesso de líquido. Então fique completamente o cone de ponta dentro da tomada e elaborar o líquido restante.
- Substitua o PLO laminina seguindo as instruções dadas nas etapas 4.1.5 (esvaziamento) e 4.1.3 (enchimento). Incube a RT por 1h.
- Substitua a laminina pelo meio de cultura seguinte as instruções dadas nas etapas 4.1.5 (esvaziamento) e 4.1.3 (enchimento). Composição do meio de cultura: MEM 81,8%; Piruvato de sódio 1% de 100 mM; Glutamax 200mm 1%; Soro do cavalo 5%; B27 suplemento de 2%, N2 suplemento 1%, gentamicina 0,2%; Filtre a solução através de um filtro de 220 nm. Usando um cone de ponta de 1 mL, substitua também a PBS em torno do chip por este meio.
- Colocar o chip em incubadora regulada a 37 ° C e 5% CO2 pelo menos 5 h (ou durante a noite) antes de neurônio semeadura.
- Chip de montagem usando estampadas lamelas
Nota: Se as lamelas modeladas incluem objetos de referência positiva fotorresiste, execute etapas 4.2.1 para 4.2.9. Caso contrário, execute somente etapa 4.2.3, vara, o dispositivo PDMS na lamela PLO modelado conforme indicado no ponto 4.1.2, pôr meio de cultura dentro e ao redor do chip vá para a etapa 4.2.10.- Depositar uma gota de água em um microscópio grosso vidro retangular e enfiar a lamela sobre a lâmina de vidro por capilaridade (não-padronizada lado virado para a lâmina de vidro). Sob um microscópio, marca a localização das listras fotorresiste na parte traseira da lâmina de vidro, usando uma caneta de feltro.
- Coloca a lamela de vidro modelado no suporte da máscara de alinhador a máscara. Contam com a marca feita com a caneta de feltro para centralizar os objetos de referência de fotorresiste.
- Execute o passo de plasma conforme descrito em 4.1.1 do chip de PDMS.
- Coloque o chip PDMS no suporte da carcaça móvel (chuck) do alinhador de máscara.
Nota: Para aumentar o contraste óptica, coloque uma bolacha de silicone sob o chip PDMS. O wafer de silício deve permanecer firmemente anexado o Chuck durante o processo de alinhamento (uso uma fita transparente para furá-lo para o chuck). - Levante o chuck no limite de contato mecânico para alinhar o chip com a matriz de listras fotorresiste localizado na lamela.
- Obter contato mecânico entre o chip e a lamela, terminando de levantamento do mandril até a superfície do PDMS pilares toque a lamela de vidro.
- Baixe o chuck. Remova a lamela agora ligada à microplaqueta do suporte da máscara. Em seguida, coloque o dispositivo dentro de um prato de Petri 35mm e transferir tudo para o forno (temperatura: 70 ° C) por 5 a 10 min aumentar a força de ligação.
- Execute como na etapa 4.1.3.
Nota: se usar PLO modelado lamelas, ir diretamente da etapa 4.2.7 para passo 4.2.9. - Substitua o PLO seguindo o chapeamento médio que os procedimentos descritos nas etapas 4.1.5 (esvaziamento) e 4.1.3 (enchimento). Usando um cone de ponta de 1 mL, substitua também a PBS em torno do chip por este meio.
- Colocar o chip em incubadora regulada a 37 ° C e 5% de CO2 até neurônio semeadura, com um mínimo de tempo de incubação de 5 h.
5. cultura de neurônio
- Prepare 100 mL de meio de dissecação (meio HH) misturando-se 10 mL de HBSS 10 x, 2 mL de Hepes 1 M e 88 mL de água esterilizada num frasco de plástico de 200 mL.
Nota: O meio HH pode ser preparado no dia anterior a cultura. - Disse o hipocampo de embriões de ratos E18 extraído de uma mãe sacrificada por deslocamento cervical (camundongos C57BL/6J de Charles Rivers). São , por exemplo, detalhados em 16etapas de dissecação.
- Coloque o hipocampo em um tubo de plástico contendo tripsina (0,3 mL de tripsina 2,5%, w/o EDTA em 2,7 mL de meio HH) por 10 min a 37 ° C para iniciar a dissociação química.
- Descartar a quase todo o líquido e substitua-o por 5 a 10 mL de HH usando pipetas plásticas descartáveis. Fazê-lo 3 vezes. Para o último preenchimento, utilize 1 mL de chapeamento médio em vez de HH.
- Mecanicamente, dissocia tecidos usando um cone de ponta 1ml por aspirantes e ejetar todo o volume várias vezes, evitando fazer bolhas e usando não mais que 15-20 passagens.
- Num recipiente separado de 500 µ l, prepare uma solução usando 5 µ l de suspensão de células diluído em 45 µ l de PBS. Tomar 1 µ l desta solução usando uma pipeta de 10 µ l e inserir a suspensão diluída em um contador de célula de Malassez. Use as indicações fornecidas em 17 para estimar o número de células.
Nota: Um único hipocampo geralmente fornece aproximadamente 0,5 milhões de neurônios. - Centrifugar 100 x g por 6 min em RT.
- Desprezar o sobrenadante e substituí-lo pelo volume de chapeamento médio necessário para atingir uma concentração de 10 milhões de células/mL. Os eritrócitos por sucessivamente aspiram e ejetar a suspensão de células com um cone de ponta de 1 mL.
- Elaborar o meio de chapeamento presente no chip (consulte a etapa 4.1.5). Coletar 2-3 µ l da solução recém ressuspensa usando uma pipeta de 10 µ l e injetá-lo na entrada (consulte o procedimento de injeção descrito na etapa 4.1.3). Repita imediatamente a mesma operação na saída.
- Injetar aproximadamente o mesmo volume de meio de chapeamento em cada reservatório (consulte a etapa 4.1.3).
Nota: Observe rapidamente o chip ao microscópio para verificar a densidade de células. A ordem de grandeza da densidade celular ideal corresponde a aproximadamente 5-10 pilhas dentro da superfície quadrada delimitado por 4 pilares (cerca de 0,3 mm2, ou seja, sobre células de 15 a 20 por mm2). - Eventualmente, repita etapa 5.10 usando, em vez disso, 0,5-1 µ l de suspensão de células para alcançar a densidade de células alvo.
- Coloque o chip semeado em uma incubadora regulada a 37 ° C 5% CO2.
6. observação de exclusão fluorescência
- Substituição do meio de cultura por meio de imagens.
- Prepare o suporte de imagens como em 4.1.7 mas use MEM desprovido de vermelho de fenol e adicionar dextrano fluorescente. Com esse objetivo, diluir o dextran (peso molecular 10.000 g/mol, solução concentrada a 10 mg/mL em PBS) para atingir uma concentração final de 0,5-1 mg/mL em meio de geração de imagens.
Nota: O uso Dextran conjuga com maxima de absorção/emissão 496/524 ou 650/668. Prefiro o primeiro a imagem 0,45 µm fotorresiste positiva alta estruturas (para se livrar de sua fluorescência automático em largura de banda da vermelha) e o segundo para os neurônios de imagem (menos tóxico). - Esvazie todas as entradas usando uma pipeta de 10 µ l e re-preenchê-los completamente com o meio de imagem (consulte os passos 4.1.3 e 4.1.5 da metodologia precisa de substituição média).
- Prepare o suporte de imagens como em 4.1.7 mas use MEM desprovido de vermelho de fenol e adicionar dextrano fluorescente. Com esse objetivo, diluir o dextran (peso molecular 10.000 g/mol, solução concentrada a 10 mg/mL em PBS) para atingir uma concentração final de 0,5-1 mg/mL em meio de geração de imagens.
- Imagem latente
- Coloque o chip num microscópio de epifluorescência equipado com uma câmara ambiental regulamentada a 37 ° C e 5% de CO2. Uso um 40 X, abertura numérica (NA) 0.8 seca objetiva, 30% da potência máxima (potência máxima: 3 W) e 30 ms de tempo de exposição. Adquira imagens de células em foco (de único para várias imagens sucessivas no caso de experimentos de lapso de tempo).
- Análise de imagem
- Normalize a imagens usando a rotina de redução de fundo implementada no software dedicado para obter um fundo homogêneo. Veja 13 para os detalhes do passos incluídos neste software de processamento de imagem. A imagem de saída tem um. Formato de esteira.
- Converta. MAT arquivo em. Imagens de 8 bits TIFF usando a rotina de colocar no material complementar (conversion_mattotiff.m, que chama para importfilevol.m).
- Executar importação > sequência de imagem no ImageJ para construir um vídeo da. Imagens TIFF.
- Calcule a média intensidade P de uma área de quadrados, localizada no centro de um pilar (objeto de referência) e a intensidade média B de uma área quadrada da câmara desprovido de células (plano de fundo, ou seja, altura zero). Ver 18 para obter um exemplo de metodologia detalhada de processamento de imagem.
Nota: A dimensão lateral das áreas quadradas utilizadas como referências de intensidade devem ser cerca de metade do diâmetro do pilar para obter um número suficiente de pixels, evitando a poluição luminosa entre as bordas do pilar. - Use os valores de P e B para estabelecer a lei de conversão linear de intensidade a altura h:
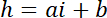
com hc a altura conhecida da câmara,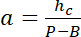 e
e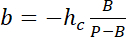
Nota: PDMS exibe não detectável autofluorescência. - Selecionar uma área em torno da zona de interesse, integrar a intensidade usando o ImageJ (ver 19 para mais detalhes) e aplicar a lei de conversão obtida em 6.3.5 para medir o volume de um compartimento de pilha.
Nota: A zona de interesse pode ser selecionada com base em fluorescência do elementos sub celulares como cones de actin, por exemplo, para a seleção de crescimento em neurônios de GFP-LifeAct, selecionados a zona de interesse no canal de emissão da GFP, salvar o contorno do presente zona usando uma ferramenta ROI manager e, em seguida, medida que o volume de célula fechado dentro da mesma zona no canal de emissão do dextran (em vermelho).
Resultados
O resultado do processo de fabricação descrito nas seções 1 e 2 é ilustrado pelas imagens da figura 1A-1B e a curva da Figura 1. A tabela da Figura 1 exibe os valores de rugosidade de duas diferentes áreas representativas de PDMS chip, ou seja, na região central e a câmara intermediária alta próxima de 20 µm. Obteve-se uma diminuição da rugosidade por um fator de cerca de 7 usando gravado da bolacha de Si em vez de fotorresiste SU-8. Em seguida, FXm primeiro foi aplicado em uma faixa de fotorresiste da geometria conhecida (Figura 2A) dentro de uma câmara de alta de 10 µm. Após processamento de imagem e intensidade para conversão de altura (ver gráfico da Figura 2B), perfis de FXm realizadas em cortes transversais ao longo desta faixa (Figura 2) fornecem os perfis de altura desejada (Figura 2D). Figura 2D mostra a comparação entre perfis obtidos usando profilometry mecânico e métodos FXm. Estes perfis, incluindo o valor de borda e planalto, são muito semelhantes, validando o método. Observe que a dispersão dos dados FXm não é representativa da resolução final do método, como novo avaliada na Figura 3 e Figura 4, mas resultados da baixa intensidade empregadas para evitar um possível efeito de muito fraco autofluorescência do fotorresiste no canal da GFP.
Em seguida, observamos neuritos em 3 µm e 10 µm alta câmaras (Figura 3). O desvio padrão de ruído de fundo é de cerca de 18 nm após a intensidade para correção de conversão e fundo de altura. Este valor é ligeiramente maior do que o física rugosidade das superfícies PDMS fundidas em superfícies de silício (12 nm, consulte a tabela da Figura 1) mas muito mais baixo do que a rugosidade medida em PDMS obtidos de moldes de SU-8. Esses resultados destacam o valor acrescentado da perfuração de poços em wafers de silício, em vez de abrir buracos em SU-8 fotorresiste para lançar pilares. Um valor tão baixo permite que um sinal elevado à relação de ruído e imagens muito claras em volume como o exibido na Figura 3A. Como um exemplo dos dados que podem ser obtidos de tais imagens, nós calculado o volume de 1,6 µm (ou seja, 10 pixels) de axônio grande fatia (ver o gráfico da Figura 3B). Usando em uma primeira aproximação um ajuste linear destes dados dá um valor de altura média axônio de cerca de 400 nm, para ser comparado com , por exemplo, o 500 nm diâmetro axonal encontrado em 10 dias velhos filhotes dentro do corpo caloso 5. Também combinamos FXm com micropatterns de adesão consiste em série confinou 2 µm e 6 µm de listras largas de 30 µm de comprimento. Nosso objetivo foi estudar a influência da largura do axônio na sua forma 3D. A Figura 3 mostra uma representação 3D em cor falsa de uma neurônio toda imagem obtida em uma câmara de alta de 10 µm. Neuritos estão se espalhando em 2 µm e 6 µm de listras largas, Considerando que a soma está localizado na extremidade da faixa maior. Perfis de altura foram divididos em três seções diferentes de cruz. Em coerência com o gráfico apresentado na Figura 3A, a superfície integrado sobre o aumento de seções transversais com a largura do axônio (Figura 3D).
Enfocamos também estruturas 3D de crescimento cone (GC). Figura 4 A-B exibe dois diferentes GC perfis obtidos em uma câmara de alta 3 µm, que destacam sua estrutura ramificada de sub. Além disso, foram realizadas experiências de lapso de tempo para acompanhar a dinâmica do volume de GCs em uma câmara de alta 12 µm. A Figura 4 mostra um ciclo de encolhimento e reativação de um GC determinado dentro de uma escala de tempo de algumas dezenas de minutos. Graças à utilização de GFP-lifeact ratos, cones de crescimento foram localizados no comprimento de onda de emissão de GFP (510 nm) de sua concentração elevada de actina. A superfície identificada no comprimento de onda foi usada para integrar-se ao longo do comprimento de onda de emissão de dextran em 647 nm para calcular o volume de GC. Figura 4 mostra finalmente a distribuição do volume de GC em pontos diferentes de tempo e local em três diferentes neurônios, centrados em um valor de cerca de 6 µm3.
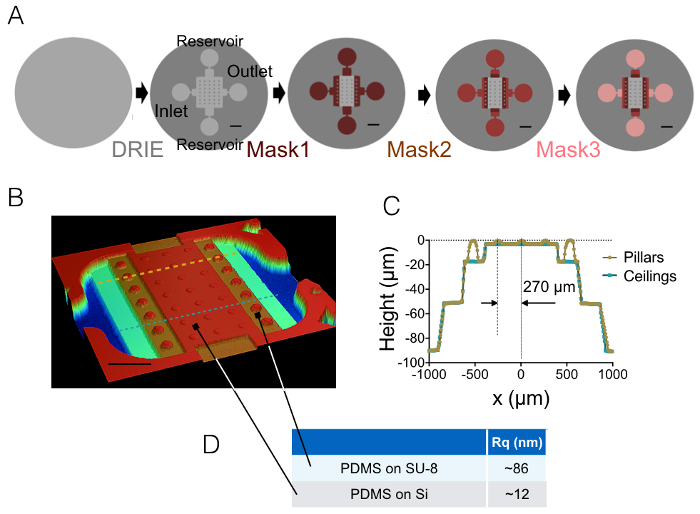
Figura 1: câmaras FXm PDMS. (A) esquemas das quatro etapas principais diferentes de microfabrication levando ao molde final. A localização da entrada, saída e reservatórios são indicados. Escala de bares: 1 mm. (B) imagem da câmara de PDMS FXm obtida usando um perfilômetro óptico. Esta imagem mostra a câmara central contendo 3 fileiras de 10 µm altos pilares e as câmaras intermédias de 20, 50 e 90 µm de altura. Barra de escala: 500 µm. (C) vista transversal do chip ao longo de duas linhas tracejadas desenhado em (B). Amarelo/ouro: seção transversal ao longo de pilares, azuis: secção transversal entre pilares. (D) significa valores de rugosidade do PDMS medido em 50 × 50 µm2 áreas moldadas em silicone e sobre a câmara intermediária alta de 20 µm SU-8 (veja as setas para a localização dessas áreas). Valores médios foram obtidos de medições de três áreas diferentes. Clique aqui para ver uma versão maior desta figura.
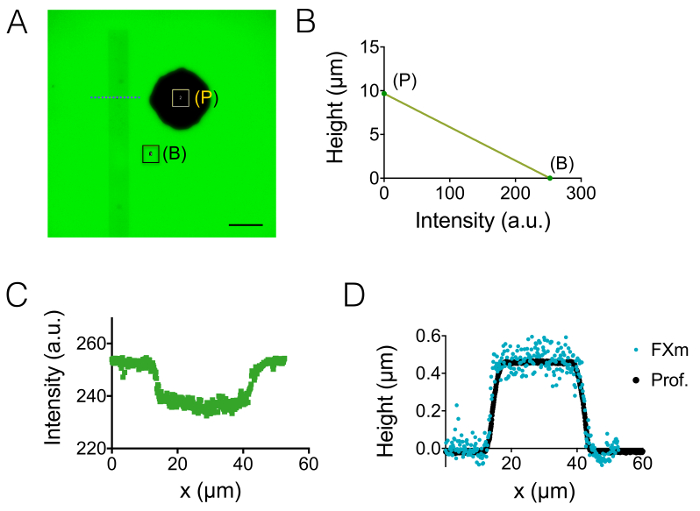
Figura 2: calibração de FXm método usando uma faixa de fotorresiste como o objeto de interesse. (A) imagem de GFP-fluorescência de uma câmara de alta de 10 µm tirada repleta de 10.000 dextrano MW absorvente em 488 nm a 1 mg/mL. (B: plano de fundo, p: pilar). Observação com um objectivo de ND X 0.8 40 seco. Barra de escala: lei 50 µm. (B) calibração Linear obtida a intensidade média dos dois retângulos coloridos mostrado no perfil de intensidade da fluorescência (C) obtido ao nível da linha tracejada azul exibido em (A), cruzando o fotorresiste listra (0,45 µm alta positiva fotorresiste). (D) comparação dos perfis obtidos de mecânico perfilômetro (pontos pretos) e FXm depois de intensidade para conversão da altura dos dados de (B) (pontos azuis). Clique aqui para ver uma versão maior desta figura.
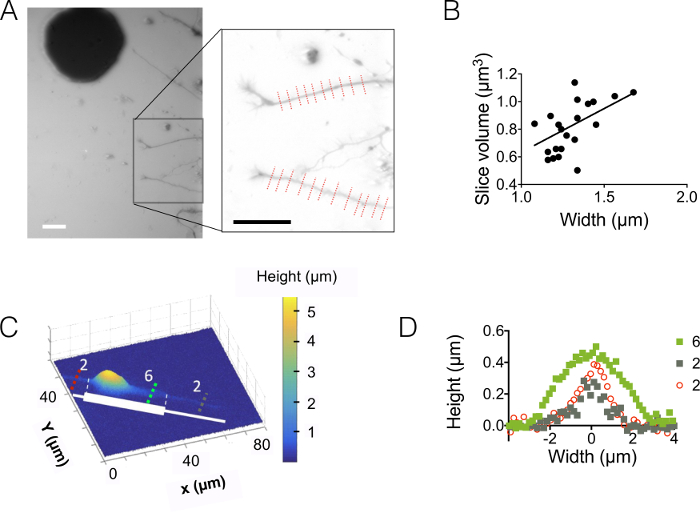
Figura 3: imagem de volume de axônio. (A) axônio estendendo-se para a câmara alta central de 3 µm de soma, localizado na câmara intermédia em seguida 15 µm. Imagem executada usando 10.000 dextrano MW absorvente em 488 nm e uma 40 X, at 0.8 secar o objectivo. O baixo-relevo obtidos após o uso de rotina de redução de fundo destaca os dois neuritos e escolhido para plotar o gráfico à direita. Escala de barras:de 30 µm. (B) axônio fatia de volume em função da largura do axônio obtidos a partir do 22 perfis (média de 10 pixels, ou seja, em um 1,6 µm "fatia do axônio") mostrados em (A). A linha sólida representa um ajuste linear de inclinação 0,4 µm passa a origem. (C) imagem em falsa cor de um neurônio estampado em uma faixa adesiva feita de sucessivas de 2 µm e 6 µm de largura tocos (representados em branco). As medições foram feitas em uma câmara alta de 10 µm, cheia de 10.000 dextrano MW absorvente em 647 nm e usando um at 40 x 0.8 secar o objectivo. (D) perfis de altura correspondente para as linhas tracejadas coloridas mostradas em (C), mantendo o mesmo código de cor. Clique aqui para ver uma versão maior desta figura.
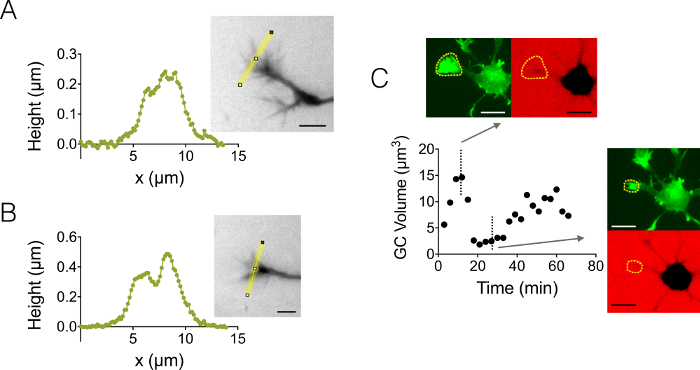
Figura 4: imagem de cone de crescimento dinâmico e estático. (A-B) Perfis de altura de cone de crescimento obtidas em uma câmara de alta 3 µm após intensidade para conversão de altura ao longo das linhas amarelas exibido em imagens associadas. Observação realizada utilizando um preenchimento com 10.000 dextrano MW absorvente em 488 nm e uma 40 X, at 0.8 secar o objectivo. (C) imagem de neurônio toda em uma câmara de alta 12 µm repleto de 10.000 dextrano MW absorvente em 647 nm. As observações foram feitas em dois canais fluorescentes: GFP para localização de cone de crescimento (linhas tracejadas para amarelas) e CY5 para calcular o volume de GC da exclusão de fluorescência. A superfície incluída por linhas tracejadas amarelas foi usada para calcular o volume de GC. O gráfico mostra a variação do volume de GC com o tempo e morfologias associadas em ambos GFP e CY5 canais em dois pontos representativos de tempo diferentes. Todos os dados foram adquiridos usando um at 40 x 0.8 secar objectivo todos os bares de escala de 3 min.: 10 µm. clique aqui para ver uma versão maior desta figura.
| Passo | 1:8 µm camada de máscara | Máscara de 02:30 µm camada | Máscara 03:40 µm camada |
| Tipo de SU-8 | 2007 | 2025 | 2050 |
| Spincoating | 30 s @ 2000RPM | 30 s @ 3050 rpm | 30 s @ 3250 rpm |
| Bolos mole | 3 min @ 95 ° C | 2 min @ 65 ° C + 6 min @ 95 ° C | 3 min @ 65 ° C + 7 min @ 95 ° C |
| Energia de exposição | 110 mJ/cm2 | 155 mJ/cm2 | 170 mJ/cm2 |
| Bolos de pós-exposição | 4 min @ 95 ° C | 1 min @ 65 ° C + 6 min @ 95 ° C | 2 min @ 65 ° C + 7 min @ 95 ° C |
| Desenvolvimento | 2 min 30 s | 5 min | 6 min |
| Difícil Asse (opcional) | 3-5 min @ 200 ° C | 3-5 min @ 200 ° C | 3-5 min @ 200 ° C |
Tabela 1: fotolitos etapas executadas para construir um dispositivo que contém uma câmara central de 12 μm de altura. Alturas das câmaras intermediárias: 20, 50 e 90 µm.
| Passo | Máscara 01:10 µm de camada | Máscara de 02:30 µm camada | Máscara 03:40 µm camada |
| Tipo de SU-8 | 2007 | 2025 | 2050 |
| Spincoating | 30 s @ 1500rpm | 30 s @ 3050 rpm | 30 s @ 3250 rpm |
| Bolos mole | 3 min @ 95 ° C | 2 min @ 65 ° C + 6 min @ 95 ° C | 3 min @ 65 ° C + 7 min @ 95 ° C |
| Energia de exposição | 125 mJ/cm2 | 155 mJ/cm2 | 170 mJ/cm2 |
| Bolos de pós-exposição | 4 min @ 95 ° C | 1 min @ 65 ° C + 6 min @ 95 ° C | 2 min @ 65 ° C + 7 min @ 95 ° C |
| Desenvolvimento | 2 min 30 s | 5 min | 6 min |
| Difícil Asse (opcional) | 3-5 min @ 200 ° C | 3-5 min @ 200 ° C | 3-5 min @ 200 ° C |
Tabela 2: fotolitos etapas executadas para construir um dispositivo que contém uma câmara central de 10 μm de altura. Alturas das câmaras intermediárias: 20, 50 e 90 µm.
| Passo | Máscara 01:12 µm de camada | Máscara 02:32 µm camada | Máscara 03:40 µm camada |
| Tipo de SU-8 | 2015 | 2025 | 2050 |
| Spincoating | 30 s @ 3250 rpm | 30 s @ 2500rpm | 30 s @ 3250 rpm |
| Bolos mole | 3 min @ 95 ° C | 2 min @ 65 ° C + 5 min @ 95 ° C | 3 min @ 65 ° C + 7 min @ 95 ° C |
| Tempo de exposição | 140 mJ/cm2 | 157 mJ/cm2 | 170 mJ/cm2 |
| Bolos de pós-exposição | 4 min @ 95 ° C | 1 min @ 65 ° C + 5 min @ 95 ° C | 2 min @ 65 ° C + 7 min @ 95 ° C |
| Desenvolvimento | 3 min | 5 min | 6 min |
| Difícil Asse (opcional) | 3-5 min @ 200 ° C | 3-5 min @ 200 ° C | 3-5 min @ 200 ° C |
Tabela 3: fotolitos etapas executadas para construir um dispositivo que contém uma câmara central de 3 μm de altura. Alturas das câmaras intermediárias: 18, 50 e 90 µm.
Dados complementares 1: masks_neuron_volume_chips.tiff. Visão esquemática das máscaras usadas para fabricar o dispositivo PDMS (máscara DRIE e máscaras 1-3). Clique aqui para baixar este arquivo.
Dados adicionais 2: arquivo "masks_neuron_volume_chips.dxf". Arquivos eletrônicos que permite fabricar a máscara DRIE e máscaras 1-3. Clique aqui para baixar este arquivo.
Dados suplementares 3: "Mask_Photoresist-stripes.dxf". Arquivos eletrônicos que permite fabricar a máscara utilizada para os fotolitos de fotorresiste listras. Clique aqui para baixar este arquivo.
Dados adicionais 4: arquivo conversion_mattotiff.m clique aqui para baixar este arquivo.
Dados suplementares 5: arquivo importfilevol.m clique aqui para baixar este arquivo.
Discussão
Imagem de volume de neurônios constitui um desafio para a técnica de FXm devido as longas e finas extensões dessas células. Este protocolo descreve as variantes do mesmo tipo de dispositivo microfluidic dedicado a imagem latente do neurônio.
Ao lado os aspectos do design microfluídicos, a escolha do objetivo é fundamental para a imagem latente de exclusão de fluorescência e implica um trade-off entre resolução lateral e a nitidez da imagem. Tem sido demonstrado em 13 que um at alta, levando a uma profundidade de foco menor do que a altura da câmara não foi prejudicial para a precisão de medição de volume se a imagem foi executada em foco e se é deixada uma margem suficiente entre o contorno do objeto de nterest e os limites da superfície de integração. No entanto, o uso de uma câmara muito maior do que a profundidade de foco prejudica a clareza de imagem devido à difusão de fótons, que suaviza as bordas dos objetos de interesse. A fabricação de uma câmara alta 3 µm reduzida a essa indefinição lateral e forneceu imagens de exclusão fluorescente excepcionalmente bem definidas mesmo usando alta at 40 (0,8) X objetivos Visualizar neuronais ramos com alta resolução lateral.
Chip de montagem é uma etapa crítica, em particular no caso de 3 µm câmaras altas, mas manipulação cuidadosa conforme descrito no ponto 4.1.2 evita o colapso do telhado. A superfície elevada à relação do volume associada a estas câmaras finas também levantou a questão da estabilidade da concentração de dextrano ao longo do tempo. Temos verificado que a superfície absorção do Dextran após uma noite de incubação foi insignificante: depois de substituir o Dextran pela PBS, a diferença de intensidade entre o pilar e o plano de fundo foi cerca de 1 por 1000 do contraste entre a intensidade inicial Estas duas regiões na presença de dextrano. Note-se que os neurônios podem aderir na lamela inferior e o telhado de PDMS. Este efeito desaparece quando usando estampadas lamelas (ou seja, quando nós não incubar adesivas moléculas dentro da câmara PDMS), como o revestimento, portanto, é estritamente localizado na parte inferior da câmara.
Além de sua morfologia desafiador, os neurônios são bastante adequados para FXm devido ao fato de que dentre a grande limitação do método, ou seja, endocitose de dextrano, é muito limitado nestas células. Nós escolhemos um 10 kDa formulação para suprimir a longo alcance (horas) qualquer fenômeno visível endocitose.
Em conclusão, a simplicidade conceitual do FXm é equilibrada por um conjunto de questões experimentais que foram resolvidos pelo presente protocolo, como nanométrica aspereza PDMS e altura de câmara micrométrica ou correcção de fundo para corrigir o desnível de o teto de PDMS entre pilares. No entanto, o uso de uma câmara estreita microfluidic confinar o meio fluorescente produz alguns condicionalismos específicos, como a necessidade de pilares de apoio, que reduz a superfície eficaz disponível para adesão celular, ou a necessidade de excluir a soma da central Câmara de observar neuronais extensões com a maior clareza, o que restringe as regiões da célula acessível à observação de alta resolução. Uma possível evolução desse método seria para se livrar deste confinamento físico, a ser substituído por um óptico. O novo desenvolvimento da microscopia de luz folha pode ser combinado vantajosamente com FXm no futuro.
Divulgações
Os autores declaram não haverá conflito de interesses.
Agradecimentos
Os autores querem reconhecer ChiLab, materiais e laboratório Microsystems - Politecnico di Torino - DISAT, na pessoa do Prof C F Pirri, Dr. M Cocuzza e Dr. S L Marasso, pelo seu precioso apoio no processo de desenvolvimento e fabricação de dispositivo. Agradecemos Victor Racine de Quantacell para discussão e suporte no processamento de imagem. Estamos gratos a Isabelle Grandjean e Manon Chartier da instalação de Animal do Institut Curie para sua sustentação para os ratos e Pablo Vargas e Ana-Maria Lennon (Institut Curie) para nos fornecer com os ratos de GFP LifeAct. Estamos gratos à Olivier Thouvenin do Institut Langevin e Clotilde Cadart, Larisa Venkova e Matthieu Piel do Institut Curie - UMR 144, por sua ajuda na compreensão da exclusão da fluorescência método. Finalmente, agradecemos a plataforma tecnológica do Institut Pierre-Gilles de Gennes (CNRS UMS 3750) pelo seu apoio em microfabrication. Este trabalho foi financiado em parte pelo Europeu pesquisa Conselho Advanced Grant no. 321107 "Violoncelo", Université de PSL (projeto SwithNeuroTrails), d'Avenir ANR Investissement e o Labex IPGG e Equipex.
Materiais
| Name | Company | Catalog Number | Comments |
| Equipments | |||
| Plasmalab System 100 | Oxford Instruments | To perform DRIE | |
| MJB4 mask aligner | SUSS MicroTec | SU-8 photolithography | |
| NXQ 4006 Mask aligner | Neutronix Quintel | Photolithography associated to DRIE | |
| Plasma cleaner | Diener Electronic | Pico PCCE | |
| Cell culture hood | ADS Laminaire | Optimale 12 | |
| Centrifuge | Thermo Fisher Scientific | Heraeus Multifuge X1R | |
| Incubator 37 °C 5% CO2 | Panasonic | MCO-170AICUVH-PE IncuSafe CO2 Incubator | |
| Epifluorescence microscope | Leica | DMi8 | |
| PDMS Oven between 65 and 80 °C | Memmert | ||
| Mechanical profilometer | Veeco | Dektak 6M Stylus Profiler | |
| Optical profilometer | Veeco | Wyko NT9100 | |
| Vacuum desiccator | Verrerie Villeurbanaise (Kartel Labware) | 230KAR | Diameter 200 mm |
| Ultrasonic bath sonicator | Labo Moderne | SHE1000 | Volume of the bath: 0.8 liter |
| Hotplate | Stuart SD162 | SD162 | |
| Masks | |||
| DRIE | Supplementary data | ||
| Su8 Mask 1 | Supplementary data | ||
| Su8 Mask 2 | Supplementary data | ||
| Su8 Mask 3 | Supplementary data | ||
| S1805 calibration stripes | Supplementary data | ||
| Small laboratory equipment | |||
| 1.5 mm hole puncher | Sigma-Aldrich | 29002519 (US reference) | |
| Scalpel or razor blade | |||
| 9" Stainless Steel Flat Spatula with Spoon | VWR International | 82027-532 | To demold PDMS |
| Top Lip Wafer Handling | VWR International | 63042-096 | |
| Curved tweezer | FST | Dumont #7 Forceps - Standard / Dumoxel | To manipulate glass coverslips |
| Substrates | |||
| Silicon wafer | Prolog Semicor Ltd | ||
| 24x24 mm glass coverslips | VWR | 631-0127 | |
| Photoresists and developpers | |||
| AZ 1518 positive photoresist | Microchemicals GmbH | Before DRIE process, thicknes 1.8 µm | |
| AZ 351B developer | Microchemicals GmbH | To develop positive AZ 1518 photoresist | |
| SU-8 2007 | MicroChem | ||
| SU-8 2025 | MicroChem | ||
| SU-8 2050 | MicroChem | ||
| PGMA developer | Technic | To develop SU-8 negative photoresist | |
| Microposit S1805 resist | Chimie Tech Services | Positive photoresist used to obtain 0.5µm high structures | |
| MF 26A developer | Chimie Tech Services | To develop positive S1805 photoresist | |
| Laboratory consumables | |||
| Disposable plastic pipette 3 mL | LifeTechnologies - ThermoFisher | ||
| P100 Petri dishes | TPP | 93100 | |
| 20 mL syringe | Terumo | SS+20ES1 | |
| Transparent scotch tape | |||
| Square wipes | VWR | 115-2148 | |
| Parafilm | DUTSCHER | 90260 | Plastic paraffin film |
| Chemicals | |||
| (3-Methacryloxypropyl)trichlorosilane | abcr | AB 109004 | |
| PDMS and curing agent Sylgard 184 | Sigma-Aldrich | 761036 | |
| isopropanol | W292907 | ||
| poly-ornithine | Sigma-Aldrich | P4957 - 50 mL | |
| Ethanol absolute | Sigma-aldrich | 02865 | 99.8% |
| 3-methacryloxypropyl-trimethoxysilane | Sigma-aldrich | M6514-25ML | (C4H5O2)-(CH2)3- Si(OCH3)3 |
| acetic acid | Sigma-aldrich | 71251-5ML-F | |
| Dextran 10kW conjugated with Alexa488 | LifeTechnologies - ThermoFisher | D22910 | Absoprtion at 488 nm |
| Dextran 10kW conjugated with Alexa647 | LifeTechnologies - ThermoFisher | D22914 | Absoprtion at 647 nm |
| Culture medium | |||
| MEM | LifeTechnologies - ThermoFisher | 21090-022 | |
| Horse Serum | LifeTechnologies - ThermoFisher | 26050088 | |
| B27 | LifeTechnologies - ThermoFisher | 12587-010 | |
| Glutamax 200 mM | LifeTechnologies - ThermoFisher | 35050-061 | |
| Sodium Pyruvate GIBCO 100 mM | LifeTechnologies - ThermoFisher | 11360-070 | |
| Gentamicin | LifeTechnologies - ThermoFisher | 15710-049 | |
| PBS | Sigma-Aldrich | D8537-500ML | |
| HBSS 10x | LifeTechnologies - ThermoFisher | 14180-046 | |
| Hepes 1M | LifeTechnologies - ThermoFisher | 15630-056 | |
| trypsin-EDTA | Sigma-Aldrich | 59418C-100ML | |
| Neurobasal | LifeTechnologies - ThermoFisher | 21103-049 | |
| Neurobasal without phenol red | LifeTechnologies - ThermoFisher | 12348-017 | |
| Softwares | |||
| Routine in Matlab for background normalization | Quantacell | Contact Victor Racine: victor.racine@quantacell.com | |
| ImageJ | To select specific ROI for image analysis | ||
| Routine | ImageJ | Supplementary data | |
| Routine | Matlab | Supplementary data |
Referências
- Jun, S., Taheri-Araghi, S. Cell-size maintenance: universal strategy revealed. Trends Microbiol. 23 (1), 4-6 (2015).
- Zlotek-Zlotkiewicz, E., Monnier, S., Cappello, G., Le Berre, M., Piel, M. Optical volume and mass measurements show that mammalian cells swell during mitosis. J. Cell Biol. 211 (4), 765-774 (2015).
- Kim, G. H., Kosterin, P., Obaid, A. L., Salzberg, B. M. A mechanical spike accompanies the action potential in Mammalian nerve terminals. Biophys J. 92 (9), 3122-3129 (2007).
- Iacono, D., et al. Neuronal Hypertrophy in Asymptomatic Alzheimer Disease. J Neuropathol Exp Neurol. 67 (6), 578-589 (2008).
- Cheli, V. T., et al. Conditional Deletion of the L-Type Calcium Channel Cav1. 2 in Oligodendrocyte Progenitor Cells Affects Postnatal Myelination in Mice. J Neurosci. 36 (42), 10853-10869 (2016).
- Kneynsberg, A., Collier, T. J., Manfredsson, F. P., Kanaan, N. M. Quantitative and semi-quantitative measurements of axonal degeneration in tissue and primary neuron cultures. J Neurosci Methods. 266, 32-41 (2016).
- Fouquet, C., et al. Improving axial resolution in confocal microscopy with new high refractive index mounting media. PloS one. 10 (3), (2015).
- Aguet, F., et al. Membrane dynamics of dividing cells imaged by lattice light-sheet microscopy. Mol Biol Cell. 27 (22), 3418-3435 (2016).
- Roland, A. B., et al. Cannabinoid-induced actomyosin contractility shapes neuronal morphology and growth. eLife. 3, (2014).
- Lu, Y. B., et al. Viscoelastic properties of individual glial cells and neurons in the CNS. Proc Natl Acad Sci USA. 103 (47), 17759-17764 (2006).
- Chen, J., Xue, C., Zhao, Y., Chen, D., Wu, M. H., Wang, J. Microfluidic impedance flow cytometry enabling high-throughput single-cell electrical property characterization. Int J Mol Sci. 16 (5), 9804-9830 (2015).
- Bottier, C., et al. Dynamic measurement of the height and volume of migrating cells by a novel fluorescence microscopy technique. Lab Chip. 11, 3855-3863 (2011).
- Cadart, C., et al. Fluorescence eXclusion Measurement of volume in live cells. Methods Cell Biol. 139, 103-120 (2017).
- Marasso, S. L., et al. A novel graphene based nanocomposite for application in 3D flexible micro-supercapacitors. Materials Research Express. 3 (6), 065001(2016).
- Welch, C. C., Goodyear, A. L., Wahlbrink, T., Lemme, M. C., Mollenhauer, T. Silicon etch process options for micro-and nanotechnology using inductively coupled plasmas. Microelectronic Engineering. 83 (4), 1170-1173 (2006).
- Fath, T., Ke, Y. D., Gunning, P., Götz, J., Ittner, L. M. Primary support cultures of hippocampal and substantia nigra neurons. Nat. Protoc. 4 (1), 78-85 (2008).
- abcam. Counting cells using a hemocytometer. , Available from: http://www.abcam.com/protocols/counting-cells-using-a-haemocytometer (2017).
- ImageJ. Getting intensity values from single ROI. Image Intensity Processing. , Available from: https://imagej.net/Image_Intensity_Processing (2017).
- ImageJ. 19. Tools. ImageJ User Guide. , Available from: https://imagej.nih.gov/ij/docs/guide/146-19.html (2017).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados