Method Article
Observação em tempo real da reação de troca de fita de DNA mediada por Rad51
Neste Artigo
Resumo
Fluorescência ressonância energética baseada em transferência em tempo real observação sistemas do DNA vertente reação de troca, mediada por Rad51 foram desenvolvidos. Usando os protocolos aqui apresentados, somos capazes de detectar a formação de intermediários de reação e sua conversão em produtos, também na análise da cinética enzimática da reação.
Resumo
A reação de troca de vertente de DNA mediada por Rad51 é uma etapa crítica de recombinação homóloga. Nesta reação, Rad51 constitui um filamento de cadeia de DNA de cadeia simples (ssDNA) e captura de DNA de cadeia dupla (dsDNA) não especìfica para interrogá-lo para uma sequência homóloga. Depois de encontrar a homologia, Rad51 catalisa troca de strand DNA para mediar o emparelhamento do ssDNA com a cadeia complementar do dsDNA. Esta reação é altamente regulada por numerosas proteínas accessary na vivo. Embora ensaios bioquímicos convencionais têm sido empregados com sucesso para examinar o papel de tais proteínas acessórias em vitro, análise cinética de formação intermediária e sua progressão em um produto final provou desafiadora devido à natureza transitória e instável dos intermediários de reação. Ao observar estas etapas de reação diretamente na solução, transferência de energia de ressonância de fluorescência (FRET)-com base em observação em tempo real, estabeleceram-se sistemas desta reação. Análise cinética de observações em tempo real mostra que a reação de troca de vertente de DNA mediada por Rad51 obedece a um modelo de reação de três etapas que envolvem a formação de um DNA de três filamentos intermédio, maturação deste intermediário e o lançamento do ssDNA de o intermediário maduro. O complexo de Swi5-Sfr1, uma proteína accessary conservado em eucariontes, aumenta fortemente as segunda e terceiros etapas desta reação. Os ensaios baseados em FRET aqui apresentados nos permitem descobrir os mecanismos moleculares através de recombinação que proteínas accessary estimulam a atividade de troca de vertente de DNA de Rad51. O objetivo principal do presente protocolo é para aumentar o repertório de técnicas disponíveis para pesquisadores da área de recombinação homóloga, particularmente aqueles que trabalham com proteínas de espécies diferentes de Schizosaccharomyces pombe, para que o conservação evolutiva dos achados aqui apresentados pode ser determinada.
Introdução
Recombinação homóloga (HR) facilita o embaralhamento de informação genética entre duas diferentes moléculas de DNA. HR é essencial para dois fenômenos biológicos fundamentais: a geração de diversidade genética durante a gametogénese1 e a reparação do DNA dobro-Costa (DSBs)2 se rompe durante a mitose. DSBs são a forma mais grave de dano do ADN e constituem uma quebra no cromossomo. Reparação incorreta de DSBs pode causar extensivos rearranjos cromossômicos e instabilidade genômica, que são as duas características de câncer3.
A reação de troca de vertente de DNA é a fase central de RH. A proteína Rad51, que é um membro da família de RecA-tipo altamente conservada de recombinases, é a proteína-chave que catalisa esta reação em eucariontes4,5. Nesta reação, Rad51 vincula a single-stranded DNA (ssDNA) gerado pelo processamento de antípodas da extremidade ORL e formulários uma cadeia helicoidal complexa denominado filamento pré-sináptica. Este filamento intacto DNA de cadeia dupla (dsDNA) nonspecifically para procurar uma sequência homóloga de capturas. Quando o filamento encontra uma sequência homóloga, uma reação intermediária contendo três ADN é formada e o filamento Rad51 Medeia vertente troca dentro desta estrutura6,7,8. Para realizar esta reação eficiente, Rad51 requer vários tipos de proteínas acessórias, tais como BRCA1 e BRCA2, produtos de mama câncer susceptibilidade genes9,10.
Compreensão como acessórios fatores regulam Rad51 é um passo integral para descobrir as causas da instabilidade genômica durante a tumorigênese. Embora muita pesquisa está preocupada com os efeitos desses fatores na formação do filamento pré-sináptica e estabilidade11,12,13,14,15,16, o contribuição desses fatores para a formação de intermediário a três fios e sua transformação em produto final ainda não está clara. Observando estas etapas de reação através de experimentos bioquímicos convencionais é muito difícil porque o intermediário de três fios é instável e propenso a entrar em colapso por manipulações experimentais comuns tais como desproteinização de amostras ou eletroforese.
Para superar este problema, adaptamos dois sistemas anteriormente desenvolvidos observação em tempo real da reação de troca de vertente de DNA utilizando a transferência de energia de ressonância de fluorescência (FRET): o emparelhamento de vertente de DNA e DNA vertente deslocamento ensaios17, 18 (Figura 1). A cadeia de DNA emparelhar ensaio, Rad51 formas um filamento pré-sináptica com fluoresceína amidite (FAM) - rotulado ssDNA e então homóloga carboxy-x-rodamina (ROX) - rotulado dsDNA é adicionado para iniciar a reação de troca do fio. Quando o filamento pega o ROX-rotulado dsDNA e forma o intermediário de três fios, os dois fluorophores entrar em proximidade e emissão de fluorescência da FAM é extinto por ROX (figura 1A). No ensaio de deslocamento da vertente de DNA, um filamento pré-sináptica formado-se em ssDNA sem rótulo é incubado com FAM e ROX dsDNA duplo-rotulados. Quando troca de vertente é concluída e o FAM rotulado ssDNA é liberado a partir de três fios intermediários, a emissão de FAM aumenta porque FAM não está em estreita proximidade com ROX (figura 1B). Estes ensaios permitem-nos observar a formação de três filamentos intermediários e sua transformação em produtos finais em tempo real sem qualquer perturbação para a reação.
Usando este sistema de observação em tempo real, nós encontramos que a reação de troca de vertente de DNA mediada por Rad51 procede em três etapas-incluindo a formação da primeira reação intermediária (C1), do primeiro intermediário, transita para um segundo intermediário (C2) e lançamento do ssDNA de C219. Também encontramos esse fermento de fissão (S. pombe) Swi5-Sfr1, que é um evolutivamente conservada Rad51 acessório proteína complexa13,16,20,21,22, estimula a transição de C1-C2 e lançamento do ssDNA de C2, de forma que é dependente da hidrólise de ATP por Rad5119.
Se esses achados são conservados evolutivamente permanece desconhecido. Este protocolo é fornecido com a esperança de que os pesquisadores da área de RH, especialmente aqueles que trabalham com proteínas de organismos que não sejam S. pombe, podem aplicar estas técnicas para determinar a medida em que o mecanismo molecular de orientado a Rad51 troca de vertente é conservada. Além disso, estas técnicas têm sido altamente bem sucedidas em determinar o papel do Swi5 de S. pombe -Sfr1. Assim, é uma previsão racional que estas técnicas será inestimáveis para descobrir as funções precisas dos outros fatores acessórios HR.
Protocolo
1. preparação de proteínas e DNA substratos
- Purifica as proteínas Rad51 S. pombe e Swi5-Sfr1 a homogeneidade (julgados pela coloração Coomassie), como relatado anteriormente13,21.
- Prepare substratos de DNA do oligonucleotide listados na tabela 118.
Nota: Os oligonucleotides foram comprados (consulte Tabela de materiais) e sintetizado no grau HPLC. Para a cadeia de DNA emparelhar reação, oligonucleotides 16FA(-), 16A _40bp (-) e 16AR (+) _40bp são necessários. Para o ensaio de deslocamento de DNA, oligonucleotides 16A(-), 16FA _40bp (-) e 16AR (+) _40bp são necessários (Figura 1 e tabela 1). Todas as concentrações de DNA neste protocolo se refere a concentrações em oposição a concentrações de nucleótidos do fragmento. - Para formar o doador dsDNA, Misture quantidades equimolar de costas complementares em um tubo de parede fina de PCR com tampão de recozimento (10 mM Tris-HCl pH 7,5, 100 mM de NaCl, 10 mM MgCl2), garantindo um volume total superior a 20 µ l. executar esta mistura em um rack de metal prechilled na gelo (entre 2 ° C e 4 ° C).
Nota: A combinação de oligonucleotídeos para o ensaio de emparelhamento são 16A _40bp (-) e 16AR (+) _40bp. Para o ensaio de deslocamento, recoze oligonucleotides 16FA _40bp (-) e 16AR (+) _40bp. - Aqueça a mistura de recozimento a 90 ° C por 5 min e refrigerar para baixo mais de 3h para 30 ° C, utilizando uma máquina PCR. Armazenar o DNA recozido a-20 ° C.
2. DNA Strand emparelhamento e ensaios de deslocamento
- Execute uma cadeia de DNA emparelhar o ensaio.
- Preparar 1,6 mL de tampão de reação A (pH 30mm HEPES-KOH 7.5, 1mm ditiotreitol [TDT], 15 mM MgCl2, 0,25 mM ATP, albumina de soro bovino de 0,1 mg/mL [BSA] e 0,0075% polyoxyethylenesorbitan monolaurato) contendo 36 nM 16FA(-) em uma microcentrífuga de 2,0 mL tubo de plástico (polipropileno) e pre-lo incubar a 37 ° C por 5 min.
- Para formar filamentos Rad51-ssDNA, adicionar proteína Rad51 para uma concentração final de 1,5 µM no buffer pré-incubado reação e ele incubar a 37 ° C por 5 min.
- Adicionar a proteína Swi5-Sfr1 para a mistura para uma concentração final de 0,15 µM e incubar a 37 ° C por um mais 5 min.
- 1,5 ml da mistura e transferi-lo para uma cubeta de quartzo de 1.0 x 1.0 cm contendo um agitador magnético e definir a cubeta em um spectrofluorometer. Configurar o controlador de temperatura peltier do espectrofotómetro a 37 ° C e definir o agitador magnético para 450 rpm para garantir rápida mistura da amostra injetada.
- Começar a medir a emissão de fluorescência da FAM em 525 nm (largura de banda: 20 nm) sobre excitação em 493 nm (largura de banda: 1 nm). Colete dados a cada segundo.
- Depois de iniciar a medição por 100 s, injetar dsDNA ROX-rotulado doador para uma concentração final de 36 nM a mistura usando uma seringa e medida a alteração na emissão em intervalos de 1 s para um mais 30 min.
- Realize o ensaio de deslocamento de vertente de DNA.
- Prepare-se 1,6 mL de tampão de reação A contendo 36 nM 16A(-) em um plástico de microcentrífuga de 2,0 mL tubo e pre-lo incubar a 37 ° C por 5 min.
- Formar filamentos Rad51-ssDNA na presença de Swi5-Sfr1 a 37 ° C, conforme descrito nas etapas 2.1.2. e 2.1.3.
- 1,5 ml da mistura e transferi-lo para a cubeta de quartzo, contendo um agitador magnético e definir a cubeta em espectrofotómetro, conforme descrito na etapa 2.1.4.
- Começar a medir a emissão de fluorescência e após 100 s, injetar dsDNA FAM - e ROX-etiquetadas do doador, conforme descrito na etapa 2.1.6. Medir a mudança na emissão de fluorescência em intervalos de 1 s para um mais 30 min.
3. análise de dados experimentais de emparelhamento e ensaios de deslocamento
- Estimar a eficiência máxima do traste.
- Preparar 16FA(-) recozido com 16AR (+) _40bp e 16FA _40bp (-) recozido com 16AR (+) _40bp usando o mesmo procedimento descrito nos passos 1.3 e 1.4.
- Preparar 130 μL de tampão de reação A contendo 36 nM de qualquer 16FA(-), 16FA(-) recozido com 16AR (+) _40bp, 16FA _40bp (-) recozido com 16AR (+) _40bp ou 16FA _40bp (-) em uma cubeta de quartzo de 1,0 x 0,2 cm.
- Definir a cubeta para o spectrofluorometer e incubar a 37 ° C por 5 min.
- Medir os espectros de fluorescência de 500 a 600 nm após excitação em 493 nm.
- Para testar o efeito de Rad51 sobre a emissão de FAM e têmpera de FAM por ROX, adicionar Rad51 para uma concentração final de 1,5 μM para a mistura e incubar a 37 ° C por 5 min.
- Medir os espectros de fluorescência de 500 a 600 nm após excitação em 493 nm.
- Calcule a máxima eficiência FRET (Emáxima) usando a equação descrita abaixo:
Emáximo = (intensidade de fluorescência em 525 nm de dsDNA rotulado com FAM e ROX) / (intensidade de fluorescência em 525 nm de FAM rotulado ssDNA)
- Analise os dados experimentais de ensaio de deslocamento.
- Para converter a mudança na fluorescência observada no ensaio de deslocamento para a mudança na quantidade de produto final, normalizar dados experimentais brutos obtidos neste ensaio utilizando a equação descrita abaixo, onde Fcru é a intensidade da fluorescência de dados brutos e Fnormalizada é a mudança na fluorescência calculada pela seguinte equação.
Fnormalizado = ([Fcru ao tempo x]-[Fcru ao tempo 0]) / (([Fcru ao tempo 0] / Emáxima)-[Fcru ao tempo 0])
Fcru ao tempo 0 é a média fluorescência monitorizada para as primeiras 5 s após o tempo morto (ou seja, o tempo necessário para a mistura, depois de um injectant é introduzida a cubeta). - Para excluir os efeitos de fotobranqueamento e deslocamento espontâneo, subtrai Fnormalizado sem proteína de Fnormalizado da amostra para obter FD, que é a mudança na quantidade de produto final neste ensaio.
FD = [Fnormalizado da amostra] - [Fnormalizado sem proteína]
- Para converter a mudança na fluorescência observada no ensaio de deslocamento para a mudança na quantidade de produto final, normalizar dados experimentais brutos obtidos neste ensaio utilizando a equação descrita abaixo, onde Fcru é a intensidade da fluorescência de dados brutos e Fnormalizada é a mudança na fluorescência calculada pela seguinte equação.
- Analise os dados experimentais de ensaio de emparelhamento.
- Normalizar os dados experimentais obtidos de ensaio de emparelhamento usando a equação descrita abaixo, onde Fcru é a intensidade da fluorescência de dados brutos e Fnormalizada é a mudança na fluorescência calculada pela seguinte equação.
Normalizado F = (Fcru ao tempo x) / (Fcru no tempo 0)
Fcru ao tempo 0 é a média fluorescência monitorizada para os últimos 20 s antes de iniciar a reação injetando o substrato dsDNA. - Para converter a mudança na fluorescência em mudança na quantidade de substrato e excluir os efeitos de fotobranqueamento e espontânea de emparelhamento, normalizar Fnormalizado da amostra usando a equação descrita abaixo, onde FP é a mudança na quantidade de substrato neste ensaio.
FP = 1 - (([Fnormalizado sem proteína] - [Fnormalizado da amostra]) / [1-Emáxima]) - Para examinar a cinética da reação de troca de DNA strand, realizar a análise de regressão não-linear do último-Praça da reação de emparelhamento usando o programa de análise23 (consulte Tabela de materiais).
- Prepare um arquivo no formato. txt, que contém dados de curso tempo de FP.
- Inicie o programa e cole o script no Arquivo de código suplementar em uma janela do programa:
- Começa a análise de regressão não-linear do último quadrado. Os resultados desta análise serão exibidos na mesma janela.
- Normalizar os dados experimentais obtidos de ensaio de emparelhamento usando a equação descrita abaixo, onde Fcru é a intensidade da fluorescência de dados brutos e Fnormalizada é a mudança na fluorescência calculada pela seguinte equação.
Resultados
A fim de efetivamente analisar os dados experimentais de ensaios o emparelhamento e deslocamento, é necessário definir como uma mudança na emissão de fluorescência da FAM corresponde a uma conversão de substratos de DNA em produtos. Para conseguir isso, deve ser determinada a gama relevante da intensidade de fluorescência. Para o ensaio de emparelhamento, a emissão de fluorescência de 16FA(-), que corresponde ao substrato ssDNA, é comparada com a emissão de 16FA(-) recozido com 16AR (+) _40bp, que corresponde aos produtos finais desta reação (Figura 2A). Isso equivale a máxima eficiência de FRET e, portanto, a máxima redução na intensidade da fluorescência que seria esperado se todo ssDNA substrato foi convertido em produto dsDNA. Para o ensaio de deslocamento, a emissão de 16FA (-) _40bp recozido com 16AR (+) _40bp, que corresponde ao substrato, é comparada com a emissão de 16FA _40bp (-), que corresponde ao produto final (Figura 2B). Neste caso, o aumento máximo na intensidade da fluorescência das FAM transmite um cenário em que todos do substrato dsDNA é convertido em produto ssDNA. S. pombe Rad51 não afetou de emissão de fluorescência da FAM ou têmpera eficiência de FAM por ROX em ambos os ensaios (Figura 2). A máxima eficiência de traste deve ser re-measured com cada nova preparação dos oligonucleotides como é dependente da eficiência rotulagem dos oligonucleotides.
Dados representativos de reações de emparelhamento e deslocamento de vertente de DNA são mostrados na Figura 3. Os efeitos das reações espontâneas entre DNAs de substrato e fotobranqueamento eram pequenos em ambos os ensaios, como revelado pelas mudanças insignificantes vistas na emissão de FAM sem Rad51 em comparação com as mudanças substanciais, visto com Rad51 (Figura 3A e Figura 3B). Baseia-se os dados mostrados na Figura 3A e Figura 3B, a mudança de fluorescência foi convertida na mudança na quantidade de substrato (FP) ou o produto final (F.D), respectivamente, utilizando as equações descritas em etapas 3.2.2 ou 3.3.2 ( Figura 3 e Figura 3D).
A reação de emparelhamento foi simulada usando um modelo sequencial de três etapas de reação, que consiste da formação do primeiro intermediário de três fios (C1), do primeiro intermediário, transita para o segundo intermediário (C1 para C2) e o lançamento do ssDNA desde o segundo intermediário para formar os dois produtos (D + E) (Figura 3E). Para testar se a simulação usando um ajuste do modelo de reação de três etapas sequenciais os dados experimentais, resíduos entre dados experimentais da cadeia do DNA emparelhamento de ensaio e uma curva teórica obtidos por simulação foram calculados (Figura 3F). Além disso, resíduos entre a reação de emparelhamento e uma curva teórica gerada usando um modelo de reação em duas fases sequenciais também foram calculados (Figura 3). Os resíduos para a reação de emparelhamento e o modelo de duas etapas mostram um desvio sistemático na fase inicial, Considerando que os resíduos para a reação de emparelhamento e o modelo de três passos não mostram tal um desvio. Isso indica que o modelo de três passos é um ajuste melhor do que o modelo de duas etapas para simular a reação de emparelhamento.
Para testar se o modelo de três passos é consistente com a reação de deslocamento que detecta a etapa final de troca de DNA strand, geramos uma curva teórica da reação de deslocamento, usando os parâmetros cinéticos obtidos de simulação do emparelhamento reação mostrada na Figura 3 e comparou-o com os dados experimentais da reação de deslocamento, mostrada na Figura 3D (Figura 3-H). A curva teórica ajustar os dados experimentais de ensaio de deslocamento. Nesses resultados, podemos concluir que a simulação usando o modelo de três passos é capaz de razoavelmente avaliar a reação de troca de vertente de DNA mediada por Rad51.
Dados representativos da cadeia do DNA emparelhamento reação contendo o complexo de Swi5-Sfr1, uma proteína accessary de Rad51 e Rad51 são mostrados na Figura 4A. O complexo de Swi5-Sfr1 estimulou fortemente a atividade emparelhamento de Rad51. Como foi visto na ausência de Swi5-Sfr1, a reação de emparelhamento é melhor ajustar o modelo de três passos que o modelo de duas etapas na presença de Swi5-Sfr1 (Figura 4B). Através da simulação da reação usando o modelo de três passos, constantes de equilíbrio de reação de cada etapa de reação, com ou sem Swi5-Sfr1 foram calculados. As constantes de equilíbrio de reação indicaram que o complexo de Swi5-Sfr1 não estimula a primeira etapa da reação (Figura 4, painel de um), no qual um intermediário de três fios é formado, mas estimula fortemente C1-C2 em transição ( Figura 4, painel b) e o lançamento do ssDNA de intermediário a C2 (Figura 4, painel c).
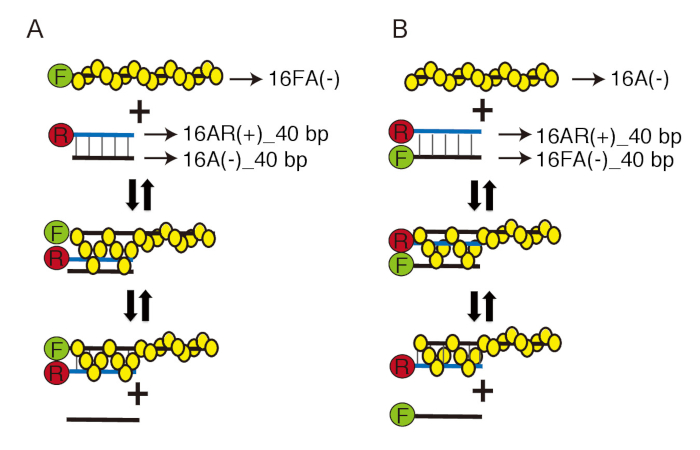
Figura 1: desenho Experimental de DNA strand ensaios emparelhamento e deslocamento. Esquemas de DNA vertente emparelhamento (A) e ensaios de deslocamento (B). Círculos amarelos representam Rad51 monômeros. Verde círculos contendo "F" e círculos vermelhos que contêm "R" representam fluoresceína amidite (FAM) e carboxi-X-rodamina (ROX), respectivamente. Preto de DNA é idênticos em sequência e complementar ao azul de DNA. Linhas pretas finas com pontas de seta apontam para o nome de cada do oligonucleotide, como descrito na tabela 1. Esta figura foi adaptada da Ito et al . 19 e modificados. Clique aqui para ver uma versão maior desta figura.
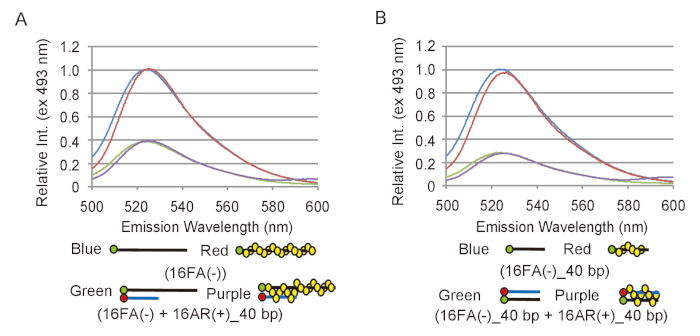
Figura 2: medições da eficiência máxima dos ensaios de emparelhamento e deslocamento FRET. (A) comparação dos espectros de fluorescência entre o substrato ssDNA, 16FA(-) e o produto de dsDNA, 16FA(-) emparelhado com 16AR (+) _40bp, do ensaio do emparelhamento. Linhas azuis e vermelhas representam os espectros de fluorescência do substrato sem e com Rad51, respectivamente. Linhas verdes e roxas mostram os espectros de fluorescência do produto final sem e com Rad51, respectivamente. (B) comparação dos espectros de fluorescência entre o substrato dsDNA, 16FA _40bp (-) emparelhado com 16AR (+) _40bp e o produto do ssDNA, 16FA _40bp (-), do ensaio do deslocamento. Linhas azuis e vermelhas representam os espectros de fluorescência do produto final sem e com Rad51, respectivamente. Linhas verdes e roxas mostram os espectros de fluorescência do substrato sem e com Rad51, respectivamente. Esta figura é uma adaptação de Ito et al . 19 e modificados. Clique aqui para ver uma versão maior desta figura.
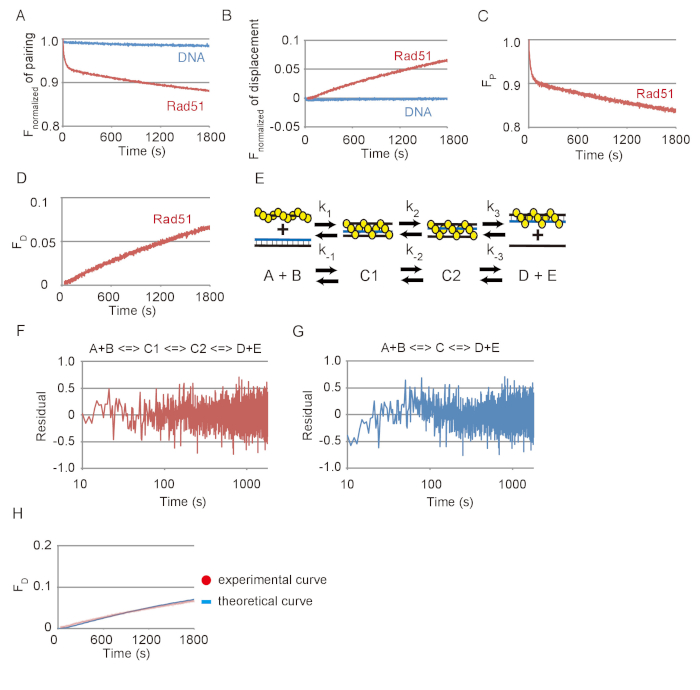
Figura 3: reações de emparelhamento e deslocamento de vertente de DNA mediada por Rad51. (A) curso de tempo da fluorescência normalizado da reação de emparelhamento com ou sem Rad51. (B) tempo de curso da fluorescência normalizado da reação de deslocamento, com ou sem Rad51. (C) Curso de tempo da alteração na quantidade de substrato na reação de emparelhamento com a Rad51. (D) curso de tempo da alteração na quantidade de substrato na reação de deslocamento com Rad51. (E) A diagrama esquemático do modelo sequencial de três etapas de reação. A e B correspondem a pré-sináptica dsDNA filamento e doador. C1 corresponde o primeiro intermediário de três fios (imaturo). C2 corresponde o segundo intermediário de três fios (maduro). D e E correspondem a um doheteroduplex e ssDNA libertado da C2. (F e G) resíduos entre dados experimentais da cadeia do DNA emparelhamento de ensaio e uma curva teórica obtidos por simulação usando os três passos (F) ou modelo em duas fases (G). (H) pontos vermelhos indicam os dados experimentais da reação de deslocamento com Rad51 mostradas no painel D. azul linha indica a curva teórica dos produtos finais. A curva teórica foi gerada pela simulação usando as constantes de velocidade de reação obtidos de ensaio de emparelhamento mostrado no painel C. Esta figura é uma adaptação de Ito et al . 19 e modificados. Clique aqui para ver uma versão maior desta figura.
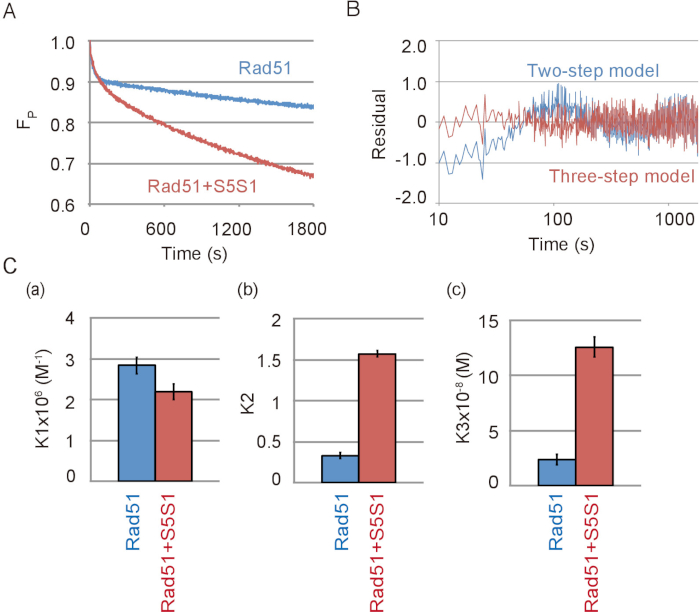
Figura 4: Swi5-Sfr1 estimula a segunda e terceiros etapas do DNA vertente reação troca. (A) curso do tempo da alteração na quantidade de substrato na reação de emparelhamento com ou sem Swi5-Sfr1 (S5S1). (B) resíduos entre dados experimentais do DNA strand emparelhamento do ensaio com Swi5-Sfr1 e uma curva teórica obtidos por simulação utilizando o modelo de três etapas (linha vermelha) ou em duas fases (linha azul). (C) o emparelhamento reação mostrada na Figura 4A foi simulada pelo modelo de três passos usando o programa de análise23 (consulte Tabela de materiais). As constantes de equilíbrio de reação de cada etapa de reação, K1 (um), (b) de K2 e K3 (c), foram obtidos a partir da simulação. Esta figura é uma adaptação de Ito et al . 19 e modificados. Clique aqui para ver uma versão maior desta figura.
| Oligonucleotídeos para uma cadeia de DNA emparelhar ensaio | ||||
| 16FA(-) | 5'-[FAM] - AAATGAACATAAAGTAAATAAGTATAAGGATAATACA AAA AAATAAGTAAATGAATAAACATAGAAAATAAAGTAAAGGATAT -3 ' | |||
| 16A _40bp (-) | 5'-AAATGAACATAAAGTAAATAAGTATAAGGATAATACAAAA-3' | |||
| 16AR (+) _40bp | 5 '- TTTTGTATTATCCTTATACTTATTTACTTTATGTTCATTT-[ROX] -3' | |||
| Oligonucleotídeos para ensaio de deslocamento do DNA strand | ||||
| 16A(-) | 5'-AAATGAACATAAAGTAAATAAGTATAAGGATAATACAAAATA AAA AGTAAATGAATAAACATAGAAAATAAAGTAAAGGATAT -3 ' | |||
| 16FA _40bp (-) | AAATGAACATAAAGTAAATAAGTATAAGGATAATACAAAA-[FAM] - 5'-3' | |||
| 16AR (+) _40bp | 5 '- TTTTGTATTATCCTTATACTTATTTACTTTATGTTCATTT-[ROX] -3' | |||
Tabela 1: uma lista de oligonucleotídeos utilizados os ensaios de emparelhamento e deslocamento de strand DNA em. Se for caso disso, as posições de fluorophores (fluoresceína amidite, FAM; carboxy-x-rodamina, ROX) são indicadas em quadrado parênteses.
Discussão
Aqui, descrevemos um protocolo detalhado que utiliza traste para medir a troca de vertente orientada a Rad51 DNA em tempo real. Importante, estas medidas permitem a determinação da cinética de reação. Enquanto as descrições acima apresentadas são suficientes para reproduzir nossos resultados publicados, existem vários pontos críticos que serão descritos nesta seção. Além disso, as vantagens e desvantagens das metodologias baseadas em FRET para estudar a troca do fio de DNA serão discutidas, juntamente com a aplicação de tais técnicas para estudar outros aspectos do metabolismo de DNA.
Tal como acontece com todas as reconstituições bioquímicas, garantindo que todos os substratos da reação são de alta pureza é essencial. É negligente para assumir a ausência de contaminantes atividades baseadas unicamente na pureza aparente de uma preparação de proteína julgada pela coloração Coomassie. Em particular, a presença de vestígios de nucleases ou helicases drasticamente pode afetar os resultados dos ensaios de emparelhamento e deslocamento. Assim, recomendamos que teste para tais atividades, cada vez que um novo lote de proteína é purificado. Além disso, é prudente verificar a pureza de substratos de DNA sintetizados por eletroforese em gel de poliacrilamida nativo. Apesar de muitas empresas, garantindo a pureza dos oligonucleotides, muitas vezes encontramos com o nosso próprio teste que a pureza do DNA sintetizado pode variar entre lotes.
É importante considerar os seguintes dois pontos quando conduzindo experimentos com cubetas de quartzo. Em primeiro lugar, algumas proteínas são propensas a vincular cubetas de quartzo nonspecifically. Para combater isso, monolaurato de BSA e polyoxyethylenesorbitan estão incluídos nos buffers de reação. Em segundo lugar, a temperatura tem um efeito drástico sobre a intensidade de velocidade e fluorescência de reação. Para minimizar este efeito, a cubeta de quartzo deve ser previamente incubada a 37 ° C antes de usar.
Apesar de ensaios bioquímicos convencionais foram extremamente úteis no estudo troca de strand DNA, eles têm várias desvantagens. Em um experimento de tempo-curso típico, uma reação é incubada a uma determinada temperatura e alíquotas são retiradas em momentos desejados e desproteinizadas com tratamento com detergente e protease para terminar a reação. Após a conclusão do curso de tempo, amostras são então submetidas a electroforese para separar substratos de DNA de produtos. A vantagem principal do método descrito aqui é que ela permite a observação em tempo real da reação sem qualquer perturbação. Qualquer Commit durante a reação pode ser inspecionada sem interrupções para a reação em si e não há nenhuma necessidade de deproteinize amostras ou sujeitá-los às forças potencialmente perturbador da electroforese. Isto é especialmente relevante quando monitoramento lábil estruturas de DNA.
Apesar dessas forças sobre ensaios convencionais, o método descrito aqui tem algumas desvantagens. Enquanto o uso de substratos de DNA do oligonucleotide para troca de vertente simplifica a interpretação dos resultados, é importante lembrar que tais substratos não se assemelham os substratos de DNA envolvida em RH na célula. Alguns ensaios convencionais utilizam substratos tamanho do plasmídeo de DNA, que são mais propensos a refletir o número de pares de bases que são trocados em vivo. Além disso, a utilização de substratos de dsDNA circular topologicamente restrita em um subconjunto dos ensaios convencionais, pelo menos parcialmente pode recriar as tensões no DNA fisiológica.
A aplicação do método descrito aqui começou a desvendar os mecanismos moleculares do exchange de strand DNA orientado a Rad51. No entanto, existem muitas questões interessantes que continuam a ser respondida. Não há evidências claras que HR durante a meiose requer Rad51 e Dmc1, a recombinase RecA-tipo de meiose específicos em eucariontes24. No entanto, a falta de grandes diferenças bioquímicas entre estes dois recombinases confundiu os pesquisadores no campo por anos. Além disso, os papéis dos grupos distintos numerosos factores acessório de recombinação tem sido um tema focal da pesquisa no campo de RH. Além de elucidar as diferenças bioquímicas entre Rad51 e Dmc1, pretendemos investigar e comparar os efeitos de fatores acessórios diferentes de recombinação na cinética de troca de strand DNA no futuro imediato. Finalmente, é importante salientar que a metodologia baseada em FRET descrita aqui não é limitada ao estudo da troca de vertente de DNA. Com modificações relativamente menores, vislumbramos muitos tipos de aplicações para esta técnica na investigação funcionalmente diversas proteínas envolvidas no metabolismo DNA25,26,27,.28. Esperamos que os desenvolvimentos descritos aqui irão fornecer mais opções de pesquisadores pertencentes a muitas disciplinas diferentes.
Divulgações
Os autores não têm nada para divulgar.
Agradecimentos
Este trabalho foi financiado pelo Grants-in-Aid para investigação científica (A) (18H 03985) e em áreas inovadoras (15H 05974) à HI, para jovens cientistas (B) (17K 15061) para BA e para Scientific Research (B) (18H 02371) para HT da sociedade para a promoção da ciência (Japão JSPS).
Materiais
| Name | Company | Catalog Number | Comments |
| 0.2 x 1.0 cm quartz cuvette | Hellma Analytics | 105-250-15-40 | |
| 1.0 x 1.0 cm quartz cuvette | Hellma Analytics | 101-10-40 | |
| adenosine triphosphate (ATP) | Sigma | A2383 | |
| DynaFit | BioKin, Ldt. | DynaFit is a program to analyze kinetics of biochemical reactions. | |
| Fluorescent labeled and non-labeled oligonucleotides | Eurofins Genomics | The sequences of oligos are listed in Table. 1. | |
| Magnetic stirrer | Aisis (Japan) | CM1609 | |
| PCR machine | TAKARA (Japan) | TP600 | TAKARA PCR Thermal Cycler Dice |
| Spectrofluorometer | JASCO | FP8300 | Contains a peltier temperature controller and magnetic stirrer system |
| Syringe | HAMILTON | 1702RN 25ul SYR (22s/2"/2) |
Referências
- Camerini-Otero, R. D., Hsieh, P. Homologous recombination proteins in prokaryotes and eukaryotes. Annual Review of Genetics. 29 (1), 509-552 (1995).
- Cromie, G. A., Connelly, J. C., Leach, D. R. Recombination at double-strand breaks and DNA ends: conserved mechanisms from phage to humans. Molecular Cell. 8 (6), 1163-1174 (2001).
- Pierce, A. J., et al. Double-strand breaks and tumorigenesis. Trends in cell biology. 11 (11), S52-S59 (2001).
- Haber, J. E. Genome Stability. , Garland Science. (2013).
- Kowalczykowski, S. C. An Overview of the Molecular Mechanisms of Recombinational DNA Repair. Cold Spring Harbor perspectives in biology. 7 (11), a016410(2015).
- Bianco, P. R., Tracy, R. B., Kowalczykowski, S. C. DNA strand exchange proteins: a biochemical and physical comparison. Frontiers in bioscience: a journal and virtual library. 3, D570-D603 (1998).
- Renkawitz, J., Lademann, C. A., Jentsch, S. Mechanisms and principles of homology search during recombination. Nature Reviews Molecular Cell Biology. 15 (6), 369-383 (2014).
- Greene, E. C. DNA Sequence Alignment during Homologous Recombination. The Journal of biological chemistry. 291 (22), 11572-11580 (2016).
- Sung, P., Krejci, L., Van Komen, S., Sehorn, M. G. Rad51 recombinase and recombination mediators. Journal of Biological Chemistry. 278 (44), 42729-42732 (2003).
- Prakash, R., Zhang, Y., Feng, W., Jasin, M. Homologous recombination and human health: the roles of BRCA1, BRCA2, and associated proteins. Cold Spring Harbor perspectives in biology. 7 (4), a016600(2015).
- Sung, P. Function of yeast Rad52 protein as a mediator between replication protein A and the Rad51 recombinase. Journal of Biological Chemistry. 272 (45), 28194-28197 (1997).
- Sung, P. Yeast Rad55 and Rad57 proteins form a heterodimer that functions with replication protein A to promote DNA strand exchange by Rad51 recombinase. Genes & Development. 11 (9), 1111-1121 (1997).
- Kurokawa, Y., Murayama, Y., Haruta-Takahashi, N., Urabe, I., Iwasaki, H. Reconstitution of DNA strand exchange mediated by Rhp51 recombinase and two mediators. PLoS biology. 6 (4), e88(2008).
- Jensen, R. B., Carreira, A., Kowalczykowski, S. C. Purified human BRCA2 stimulates RAD51-mediated recombination. Nature. 467 (7316), 678-683 (2010).
- Liu, J., et al. Rad51 paralogues Rad55-Rad57 balance the antirecombinase Srs2 in Rad51 filament formation. Nature. 479 (7372), 245-248 (2011).
- Lu, C. -H., et al. Swi5-Sfr1 stimulates Rad51 recombinase filament assembly by modulating Rad51 dissociation. Proceedings of the National Academy of Sciences of the United States of America. , (2018).
- Bazemore, L. R., Takahashi, M., Radding, C. M. Kinetic analysis of pairing and strand exchange catalyzed by RecA. Detection by fluorescence energy transfer. Journal of Biological Chemistry. 272 (23), 14672-14682 (1997).
- Gupta, R. C., Bazemore, L. R., Golub, E. I., Radding, C. M. Activities of human recombination protein Rad51. Proceedings of the National Academy of Sciences. 94 (2), 463-468 (1997).
- Ito, K., Murayama, Y., Takahashi, M., Iwasaki, H. Two three-strand intermediates are processed during Rad51-driven DNA strand exchange. Nature Structural & Molecular Biology. 25 (1), 29-36 (2018).
- Akamatsu, Y., Dziadkowiec, D., Ikeguchi, M., Shinagawa, H., Iwasaki, H. Two different Swi5-containing protein complexes are involved in mating-type switching and recombination repair in fission yeast. Proceedings of the National Academy of Sciences. 100 (26), 15770-15775 (2003).
- Haruta, N., et al. The Swi5-Sfr1 complex stimulates Rhp51/Rad51- and Dmc1-mediated DNA strand exchange in vitro. Nature Structural & Molecular Biology. 13 (9), 823-830 (2006).
- Argunhan, B., Murayama, Y., Iwasaki, H. The differentiated and conserved roles of Swi5-Sfr1 in homologous recombination. FEBS Letters. 591 (14), 2035-2047 (2017).
- Kuzmic, P. Program DYNAFIT for the analysis of enzyme kinetic data: application to HIV proteinase. Analytical biochemistry. 237 (2), 260-273 (1996).
- Brown, M. S., Bishop, D. K. DNA strand exchange and RecA homologs in meiosis. Cold Spring Harbor perspectives in biology. 7 (1), a016659(2014).
- Rudert, W. A., et al. Double-labeled fluorescent probes for 5' nuclease assays: purification and performance evaluation. BioTechniques. 22 (6), 1140-1145 (1997).
- Xiao, J., Singleton, S. F. Elucidating a key intermediate in homologous DNA strand exchange: structural characterization of the RecA-triple-stranded DNA complex using fluorescence resonance energy transfer. Journal of Molecular Biology. 320 (3), 529-558 (2002).
- Grimme, J. M., et al. Human Rad52 binds and wraps single-stranded DNA and mediates annealing via two hRad52-ssDNA complexes. Nucleic Acids Research. 38 (9), 2917-2930 (2010).
- Algasaier, S. I., et al. DNA and Protein Requirements for Substrate Conformational Changes Necessary for Human Flap Endonuclease-1-catalyzed Reaction. The Journal of biological chemistry. 291 (15), 8258-8268 (2016).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados