Method Article
Usando Nanoplasmon-Enhanced espalhamento e microscópio de baixa ampliação de imagem para quantificar Exosomas derivados do tumor
Neste Artigo
Resumo
A tradução clínica de biomarcadores derivados de exosome para células doentes e malignas é dificultada pela falta de métodos de quantificação rápidos e precisos. Este relatório descreve o uso de imagens do microscópio do escuro-campo da baixo-ampliação para quantificar subtipos específicos do exossomo em amostras pequenas do soro ou do plasma do volume.
Resumo
As células infectadas ou malignas freqüentemente secretam mais exosomas, levando a níveis elevados de exosomas associados à doença na circulação. Estes exosomas têm o potencial para serir como biomarcadores para o diagnóstico da doença e para monitorar a progressão da doença e a resposta do tratamento. Entretanto, a maioria de procedimentos da análise do exossomo exigem etapas do isolamento e da purificação do exossomo, que são geralmente demorado e trabalhoso, e assim da utilidade limitada em ajustes clínicos. Este relatório descreve um procedimento rápido para analisar biomarcadores específicos na membrana exterior dos exossomos sem exigir etapas separadas da isolação e da purificação. Neste método, os exosomas são capturados na superfície de uma corrediça por anticorpos exosome-específicos e então hibridizados com as pontas de prova Nanoparticle-conjugated do anticorpo específicas a uma doença. Após a hibridação, a abundância da população do exossomo do alvo é determinada analisando imagens do microscópio do escuro-campo da baixo-ampliação (lmdfm) das nanopartículas encadernadas. Esta abordagem pode ser facilmente adotada para pesquisa e uso clínico para analisar biomarcadores exossomo associados à membrana associada à doença.
Introdução
Os exosomas são liberados da maioria de tipos da pilha e jogam um papel chave em comunicações Cell-to-Cell, incluindo os processos fisiopatológicos associados com as várias doenças, desde que podem ser home aos tecidos ou aos tipos específicos da pilha, e contêm uma variedade de ácidos nucleico , proteínas e lipídios que refletem sua célula de origem e podem exercer efeitos regulatórios em suas células-receptor1,2,3,4. Os exosomas são secretados frequentemente em níveis elevados em Estados da doença, podem interagir com as pilhas adjacentes e distantes, e são encontrados na concentração relativamente elevada na circulação assim como a maioria outros de líquidos de corpo, incluindo a saliva, a urina, o pancreatic e a bilis suco, e fluido de lavagem broncoalveolar5,6,7,8,9,10,11. Esta abundância e estabilidade de exosomas em fluidos corporais humanos, juntamente com a sua natureza rica em informações, torna-os biomarcadores ideais para o diagnóstico da doença e monitorização do tratamento.
Isto inclui os exossomos tumor-derivados (TDES), que contêm os fatores tumor-específicos ou seletivos, que podem serir como Biomarkers da doença, incluindo os alelos tumor-associados do mutante. Os TDEs podem participar na remodelação do microambiente do tumor para facilitar o desenvolvimento e a metástase do tumor, e regulam respostas antitumorais12. O secretion aumentado de TDE é um phenotype comum de a maioria de cancros e de diversas características do microambiente do tumor, incluindo a hipóxia, o pH ácido, e a inflamação, são sabidos para promover a secreção exossomo. Surpreendentemente, dado o número de células que secretam exosomas, um aumento no nível total de exossomo pode, em si, funcionar como um biomarcador de câncer. Por exemplo, um estudo recente constatou que a concentração total de EV no suco de bile discrimina os pacientes malignos e não malignos em estenose do ducto biliar comum com 100% de acurácia7. Resultados semelhantes foram encontrados com estudos que utilizam outros fluidos corporais, incluindo o plasma. No entanto, devido ao potencial para a variação do assunto, e outros fatores de confusão, a maioria dos estudos que investigam os exosomas como biomarcadores de doença têm se concentrado na detecção de biomarcador que estão seletivamente associados com TDES em vez de exossomo total Números.
Traduzir biomarcadores exossomo na prática clínica, entretanto, permanece desafiante desde que a maioria de aproximações relatadas do ensaio do exossomo exigem procedimentos demorados e labor-intensive da isolação 13. Os métodos de isolamento exossomo atualmente populares incluem ultracentlee, gradientes de densidade, tamanho-exclusão, coprecipitação, captura de afinidade e abordagens de isolamento microfluídico. Ultracentlee é o método "padrão-ouro", e é mais comumente usado para isolamentos exossomo, mas este procedimento é demorado e resulta em danos exossomo e clustering de membrana exossomo, e produz amostras exossomo que estão contaminados com proteínas, lipoproteínas e outros fatores que podem influenciar as análises subsequentes14. A maioria dos métodos de isolamento de exosomas, incluindo a ultracentramentação, não pode separar os exossomos (30 – 150 nm) de Microvesículas (100 – 1.000 nm) e de corpos apoptóticos (100 – 5000 nm), que surgem através de diferentes mecanismos e têm diferentes funções, devido ao tamanho sobreposição entre esses grupos, e a diversidade de populações de exossomo15. Novas abordagens são necessárias para melhorar a sensibilidade e a reprodutibilidade dos ensaios de exossomo, melhorando a recuperação de exossomo, reduzindo o dano e a contaminação exossomo, embora quaisquer ensaios baseados em tais métodos também precisem ser otimizados para torná-los apropriado para a tradução às aplicações em ajustes clínicos.
Vários estudos recentes têm proposto empregar plataformas integradas para capturar e analisar os exosomas diretamente dos fluidos corporais. Estes métodos empregam microfluídico, eletrocinética, captura de afinidade, e vários outros métodos para isolamento exossomo, e eletroquímica, ressonância Plasmon superfície, e outros métodos para detectar exosomas capturados. Não está claro como viável muitas dessas abordagens serão em configurações clínicas, devido à sua complexidade, despesa, baixa taxa de transferência ou outros problemas.
Nós desenvolvemos um ensaio rápido e barato que possa ser usado para a quantificação sensível e específica de exossomos totais e de subtypes específicos do exossomo, incluindo os exossomos doença-associados, tais como TDES, que exige somente uma pequena quantidade de amostra e que emprega um fluxo de trabalho simplificado adequado para ambientes clínicos. Neste ensaio, uma corrediça é revestida com os anticorpos que ligam um marcador exossomo-específico ou doença-específico expressado na superfície do exossomo a fim capturar diretamente os exossomos do alvo atuais no plasma do volume pequeno ou nas amostras do soro aplicadas aos poços no Slide. Os exosomas capturados são então hibridizados com uma nanopartícula anticorpo-conjugada que reconheça o biomarcador do interesse nestes exossomos, que pode ser um marcador exossomo geral ou um fator específico para um SubType do exossomo do interesse. As imagens desses poços amostrais são então capturadas por meio de um microscópio de campo escuro (DFM) e analisadas para mensurar a luz dispersa de nanopartículas vinculadas a exosomas de juros capturados em cada amostra bem6,16,17. Notavelmente, a imagem latente uma amostra inteira bem pela baixo-ampliação DFM (LMDFM) evita uma polarização da seleção encontrada com análises elevadas da ampliação DFM quando os usuários devem diretamente escolher que campos a capturar para a análise de imagem subseqüente. A análise de imagem LMDFM está sujeita a artefatos de espalhamento de luz de irregularidades superficiais, incluindo arranhões e detritos amostrais, mas este fundo pode ser reduzido usando um algoritmo simples de redução de ruído que desenvolvemos para rodar no programa de análise de imagem NIH, ImageJ (https://imagej.nih.gov/ij/). Este algoritmo primeiro aplica um limite de contorno de entrada que é usado para detectar os limites da amostra bem para definir a região da imagem para análise subseqüente. A região definida por esta região de contorno é então dividida para separar o sinal presente nos canais vermelho, azul e verde da imagem e o canal azul é subtraído do canal vermelho para remover o sinal decorrente de artefatos de superfície e iluminação desigual de diamante Sinal.
Este artigo descreve como usar este ensaio para quantificar rapidamente níveis de exossomo totais ou específicos em amostras de plasma ou soro.
Protocolo
1. preparação de sondas de nanopartículas
Nota: Este ensaio utiliza Nanorods de ouro funcionalizada (aunrs; 25 nm de diâmetro x 71 nm de comprimento) que são covalentemente conjugados com polímeros de neutravidina (AV) e têm um pico de ressonância Plasmon superfície que produz um vermelho (641 nm pico) espalhamento sinal sobre DFM Iluminação.
- Lave 40 μL de AuNR-AV (2,56 x 1011 partículas) três vezes com 200 ΜL de PBS (pH 7,0) por centrifugação e aspiração (8.500 x g a 4 ° c por 10 minutos), seguido de uma centrifugação final e passo de aspiração após o qual o pellet aunr-AV está suspenso em 40 μL de PBS.
- Misture esta suspensão aunr-AV com 10 μL de anticorpo biotinilado (0,5 mg/ml) específico para um antígeno na superfície do subtipo exossomo de interesse e 150 μL de PBS e, em seguida, misture a 4 ° c por 2 h usando um misturador para permitir a ligação da neutravidina-biotina para alcançar a conclusão.
- Lave as AuNRs conjugadas com anticorpos resultantes (AuNR-IgG) três vezes por centrifugação e aspiração (6.500 x g a 4 ° c por 10 minutos) e, em seguida, suspenda-as em 200 ΜL de PBS e guarde-as a 4 ° c até o uso.
Nota: a técnica estéril e os tempos de armazenagem curtos devem ser utilizados para evitar a contaminação e a degradação da AuNR-IgG. É melhor usar as AuNRs conjugadas com anticorpos dentro de 24 horas de sua conjugação.
2. preparação de slides de captura EV
- Diluir os anticorpos de captura exossomo selecionados para 0, 25 mg/ml em PBS e adicionar 1 μl/poço desta diluição em um slide de proteína a/g multipoços e, em seguida, incubar esta corrediça a 37 ° c por 1 h em uma câmara humidificada para permitir a ligação de anticorpos de captura à proteína a/g imobilizada em o slide.
- Aspirar poços para remover anticorpos não acoplados, e lavar os poços três vezes pela adição e aspiração de 1 μL/poço de PBS, em seguida, carregar cada poço com 1 μL de tampão de bloqueio (ver tabela de materiais) e incubar o slide para 2 h a 37 ° c em uma câmara umidificada para b bloquear quaisquer locais de ligação de proteínas restantes.
- Aspirar poços para remover o tampão de bloqueio, lave os poços três vezes pela adição e aspiração de 1 μl/poço de PBS, e use imediatamente os slides bloqueados para captura e análise de exossomo.
3. preparação padrão da curva
- Para quantificar com precisão a abundância absoluta ou relativa de um subtipo específico exossomo, o usuário deve gerar uma curva padrão com uma população pura exossomo que expressa uniformemente o biomarcador de superfície exossomo de interesse. Este estudo analisa a abundância de exosomas expressando uma proteína de membrana associada à metástase, o receptor de ephrin a2, que tem uma relação relatada com o estágio e o prognóstico do câncer pancreático6,18.
Nota: a linha de células cancerosas pancreáticas humanas Panc-1 e seus exosomas são conhecidas por expressar esta proteína e exosomas isolados desta linha celular foram utilizados para gerar uma curva padrão para quantificar o número de exosomas que expressam esta proteína em exossomo complexo Amostras. - Células de cultura por 48 horas a 37 ° c em meios de cultura livres de soro para permitir acúmulo de exossomo na mídia, em seguida, isolar sobrenadantes de cultura celular por centrifugação de culturas de suspensão ou aspiração direta de meios de cultura de culturas de células aderentes.
- Centrifugue a mídia coletada em 2000 x g por 30 min para remover detritos e recuperar o sobrenadante.
- Filtre o sobrenadante de cultura clarificada através de uma unidade de filtro de ligação de baixa proteína de 0,45 μm de capacidade adequada (por exemplo, uma unidade de filtração a vácuo de polietersulfona 250 ml).
- Concentrar o filtrado resultante por centrifugação a 3200 x g utilizando um sistema de filtro de limite de peso molecular 100.000 nominal para um volume final de 250 μl. Colete o volume retido deste filtro, em seguida, lave o filtro com 200 μL de PBS, e combine este volume de lavagem com o volume de amostra exossomo coletado.
- Centrifugue esta amostra em 21.000 x g por 45 minutos e recupere com cuidado o sobrenadante, tomando o cuidado para não coletar nenhum material precipitado.
- Centrifugue o sobrenadante recuperado em 100.000 x g por 3 horas para precipir os exosomes. Aspirar afastado o sobrenadante e recolher o pellet exossomo em 100 μl de PBS.
- Armazene as suspensões exossomo resultantes em 4 ° c se usado dentro de 24 horas ou em-80 ° c para o armazenamento a longo prazo.
Nota: não sujeitar amostras de exossomo a repetir os ciclos de congelamento-degelo. - Quantificar uma alíquota da suspensão exossomo após a mistura por medição direta de números exossomo (por exemplo, por análise de rastreamento de nanopartículas ou sensoriamento de pulso resistivo sintonável ou medindo a concentração de proteínas de lisados exossomo por micro-bicinchoninic ensaio ácido, ou um método equivalente, como um meio para aproximar a quantidade de exossomo)16,17.
- Gere um conjunto de diluições seriais da suspensão exossomo para permitir a comparação do sinal de nanopartículas para introduzir o número de exossomo ou o teor de proteínas.
- Transfira 1 μL de cada padrão de exossomo para cada um dos seus poços replicantes na placa de ensaio.
Nota: as curvas padrão podem ser usadas para calcular a inclinação da linha de correlação entre o sinal de nanopartícula e a concentração de exossomo para (1) avaliar o desempenho do ensaio e (2) determinar a concentração relativa de exosomas-alvo em amostras experimentais.
4. processamento de amostras de plasma ou soro humano
- Colete amostras de plasma ou soro por métodos padrão e armazene a-80 ° c até que seja necessário para a análise de exossomo. Descongelar rapidamente amostras em um banho de água da temperatura ambiente. Misture repetidamente as amostras descongelados por inversão para promover a suspensão homogênea.
Nota: os resultados das amostras de soro e plasma podem não ser equivalentes, uma vez que existe uma libertação significativa de exosomas durante a reação de coagulação. - Centrifugue amostras do plasma ou do soro em 500 x g por 15 minutos para precipir agregados da proteína e outros restos. Transfira uma alíquota da amostra do plasma ou do soro a um tubo fresco e adicione PBS para gerar uma diluição 1:1. Misture a amostra diluída por vortexing suave ou inversão, conforme apropriado. Transfira 1 μL de cada suspensão plasmática ou sérica para cada um dos seus poços replicantes na placa de ensaio.
5. exosome captura e detecção
- Carregue poços de uma corrediça bloqueada da captação de EV com 1 μl/poço da amostra do exossomo, usando 8 repetições por a amostra, e incubar a corrediça durante a noite em 4 ° c em uma câmara humidificada. Aspire todos os poços da amostra e, em seguida, adicione 1 μl/poço de PBS para lavar poços e remover exosomas não acoplados e outros contaminantes da amostra de exossomo carregada.
- Carregar poços amostrais com 1 μL/poço de uma suspensão AuNR-IgG previamente preparada (ver secção 1 supra) e incubar a lâmina durante 2 h a 37 ° c numa câmara humidificada. Aspirar a solução de nanopartículas e lavar o slide em PBS suplementado com 0, 1% Tween-20 (PBST) por 10 min usando um misturador, em seguida, aspirar e lavar todos os poços de amostra com água deionizada por 10 min usando um misturador rotativo, e ar-seco para imagens subsequentes LMDFM.
Nota: os coeficientes de variação entre ensaios (CVs) são avaliados a partir de oito repetições da mesma amostra. Amostras que exibem CVs > 20% são consideradas não informativas e devem ser repetidas, se houver amostra suficiente.
6. captura de imagem DFM
- Capture imagens para quantificação de exossomo iluminação consistente usando uma câmera digital conectada a um microscópio equipado com um condensador de campo escuro (1,2 < na < 1,4) e um objetivo de 4x empregando um tempo de exposição de 1/220 s.
- Abra o software de captura de imagem.
Nota: utilizamos o software de imagem de microscópio NIS-Elements (ver tabela de materiais) para o protocolo descrito abaixo, mas é possível utilizar outro software que possa corresponder aos seus parâmetros de captura de imagem. NIS-Elements Viewer Imaging software é um programa autônomo gratuito para visualizar arquivos de imagem e conjuntos de dados que contém ferramentas de análise, visualização e arquivamento. Os parâmetros abaixo também são para um microscópio com autofoco e um estágio automatizado que permite que várias imagens sejam automaticamente capturadas e costuradas em uma única imagem. - Coloque a corrediça de cabeça para baixo no estágio do microscópio, ajuste a posição da corrediça e aplique uma gota pequena do óleo da imersão na parte traseira da corrediça, onde a lente do condensador contata a corrediça.
- Clique no botão ao vivo na interface do software, e ajustar o tempo de exposição contra um padrão de alta concentração bem para garantir que a imagem não está saturada.
- Abra a janela digitalizar imagem grande da guia adquirir e defina os parâmetros da interface do software da seguinte forma: imagem macro Optical conf = atual; Objetivo: 2:10x, digitalização de conf óptico = atual, objetivo: 2:10x; Sobreposição de costura = 20%; Costura via = caminho ideal.
- Escolha criar imagem grande, feche o obturador ativo durante o movimento do palco, aguarde antes de cada captura: 20 ms, concentre-se manualmente no inícioe use o foco passo a passo a cada 20 campos. Estas definições serão guardadas com as imagens digitalizadas.
- Mova o estágio do microscópio para definir os limites inferiores direito superior esquerdo do campo de verificação de destino. Ajuste o foco para conseguir uma imagem desobstruída no monitor e ajuste as configurações do condensador e a iluminação ambiental como necessário para minimizar todas as irregularidades da iluminação na imagem focalizada.
- Nomeie o arquivo de saída de imagem no software. Clique no botão digitalizar e permitir que o microscópio para digitalizar e criar e salvar uma imagem costurada de todo o slide.
- Abra a imagem salva com o software de captura de imagem que você está usando e salvá-lo em 1/8 escala para análise subsequente no plugin DSM no ImageJ.
7. análise de imagem DFM
- Faça o download do programa ImageJ (https://imagej.nih.gov/ij/). Instale o plugin do algoritmo DSM no ImageJ usando as instruções listadas em https://imagej.net/Plugins#Installing_plugins_manually.
- Abra o software ImageJ e, em seguida, defina os seguintes parâmetros de entrada dentro do algoritmo DSM: limite de contorno (CT) = 253, 20, tipo = vermelho, escala de centro (S) = 0,8, limite de quantificação baixo (lt)/alto (HT) = 0/62.
- Abra a imagem guardada a partir da secção 6,8 com o ImageJ. Escolha o botão DSM Scan na guia plugins e, em seguida, defina o número de colunas e linhas de acordo com a imagem aberta. O programa pode reconhecer as áreas de detecção e análise da intensidade de dispersão de nanopartícula em áreas de acordo automaticamente. Defina os seguintes parâmetros de entrada dentro da janela do DSM Scan : redimensionar porcentagem = 25, diâmetro do ponto (em pixels) = 190 – 200, faixa de diâmetro = 32, diâmetro de incremento (em pixels) = 8, configuração do DSM-baixa limite = 0, limite alto = 62, distância adjacente = 100, subtrair Bias = 0.
Nota: os resultados da intensidade de dispersão da nanopartícula refletem a quantidade de exosomas acoplados no slide.
Resultados
As lâminas revestidas de proteína A/G multipoços (Figura 1a) foram funcionalizadas com anticorpo EphA2 e, em seguida, usadas para capturar especificamente Exosomas EphA2-positivas de amostras de soro de pacientes com e sem câncer pancreático (1 μl/poço) e incubadas com Nanorods dourados conjugados com um anticorpo CD9 (Figura 1b). As imagens de DFM da luz dispersadas destas nanopartículas foram analisadas usando o encaixe de DSM no software de ImageJ para quantificar os exossomos positivos Bound de EPHA-2 em cada poço. O algoritmo DSM define automaticamente o limite de uma amostra bem, filtra o ruído de artefatos, calcula o sinal disperso de cada poço e gera essas informações (Figura 2). O algoritmo DSM atenua fortemente artefatos de espalhamento de luz de arranhões ou detritos presentes na amostra e melhora a sensibilidade e a reprodutibilidade da detecção de nanopartículas e pode processar automaticamente um lote de imagens de slides para alta taxa de transferência Usar. Esse algoritmo usa comandos ImageJ e entrada de parâmetros pelo usuário para subtrair o plano de fundo da imagem, calcular o sinal de dispersão de cada poço e gerar um arquivo de dados e imagem (Figura 3). As regiões de interesse são definidas pelos limites de poço de alta intensidade na imagem de captura usando a função de limite de contorno do programa macro ImageJ. As análises de imagem usam um limiar de contorno predefinido e parâmetros de imagem para calcular a intensidade da dispersão de nanopartículas para cada poço.
Conforme relatado em nosso trabalho anterior (informações complementares de Liang et al.6), exosomas isolados de culturas de células Panc-1 caracterizadas por microscopia eletrônica de transmissão e Western blot exibiram a faixa de tamanho, morfologia e marcador de proteínas expressão consistente com uma amostra exossomo de alta pureza. Os exosomas Panc-1, preparados com o mesmo procedimento que nosso trabalho anterior, foram utilizados aqui para validar nosso ensaio de NPEs para quantificação de exossomo. Este ensaio usou um anticorpo EphA2 para capturar uma grande população dos exossomos da população total do exossomo e de um anticorpo de encontro à proteína geral do exossomo CD9 para detectar exossomos capturados. Os resultados obtidos por meio de amostras de Panc-1 exossomo, com concentrações proteicas variando de 0,24 a 1,2 μg/μl, mostraram boa reprodutibilidade em poços replicantes (figura 4a) e uma forte correlação linear entre a resposta de dispersão e concentração de proteínas exossomo (Figura 4B).
Para demonstrar a aplicação potencial deste método, as amostras do soro dos pacientes com e sem cancro pancreatic foram analisadas para detectar a abundância de exossomos do soro que expressam o biomarcador cancro-associado EphA2, usando um anticorpo EphA2 a Capture diretamente os exossomos do alvo do soro e das nanopartículas conjugados com um anticorpo CD9 para detectar exossomos encadernados. Esta análise revelou que as amostras de soro dos pacientes com câncer pancreático apresentaram níveis significativamente mais elevados de EphA2 + exosomas (Figura 5) do que seus controles.
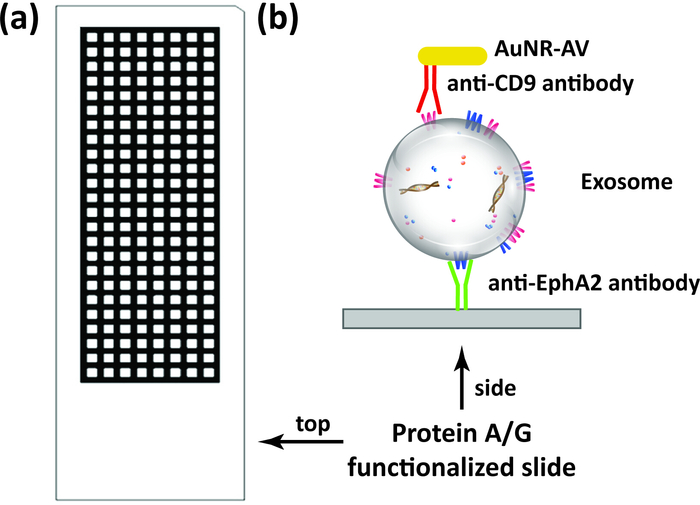
Figura 1: esquema de quantificação de Exosome. (A) esquema de lâminas de proteína A/G multipoços (192 poços) utilizados neste ensaio. (B) os exosomas alvo são diretamente capturados a partir de amostras, incluindo soro e plasma, por imobilização superficial no anticorpo de captura (por exemplo, anticorpo EphA2) ligado à corrediça e, em seguida, incubados com Nanorods dourados conjugados com uma detecção anticorpo (por exemplo, anticorpo CD9) antes da análise por análise de imagem DFM. Por favor clique aqui para ver uma versão maior desta figura.
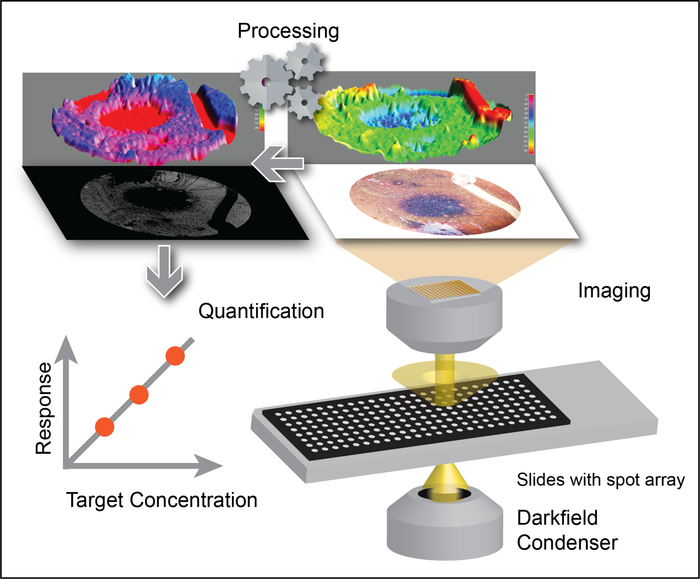
Figura 2: quantificação de Exosome aplicando o algoritmo DSM a imagens LMDFM. Imagens de baixa ampliação são processadas com o algoritmo DSM para eliminar o sinal de fundo e artefatos de sinal que podem surgir de arranhões, misturando vazios, detritos e iluminação desigual da amostra para permitir a detecção robusta de sinal de diamante de ouro, que correlaciona-se com a concentração do exossomo. Este número foi adaptado com a permissão de Sun, D. et al. método de redução de ruído para quantificar a dispersão de luz de nanopartículas em imagens de campo distante de microscópio de campo escuro de baixa ampliação. Química analítica. 88 (24), 12001-12005 (2016). Copyright (2016) sociedade química americana. Por favor clique aqui para ver uma versão maior desta figura.
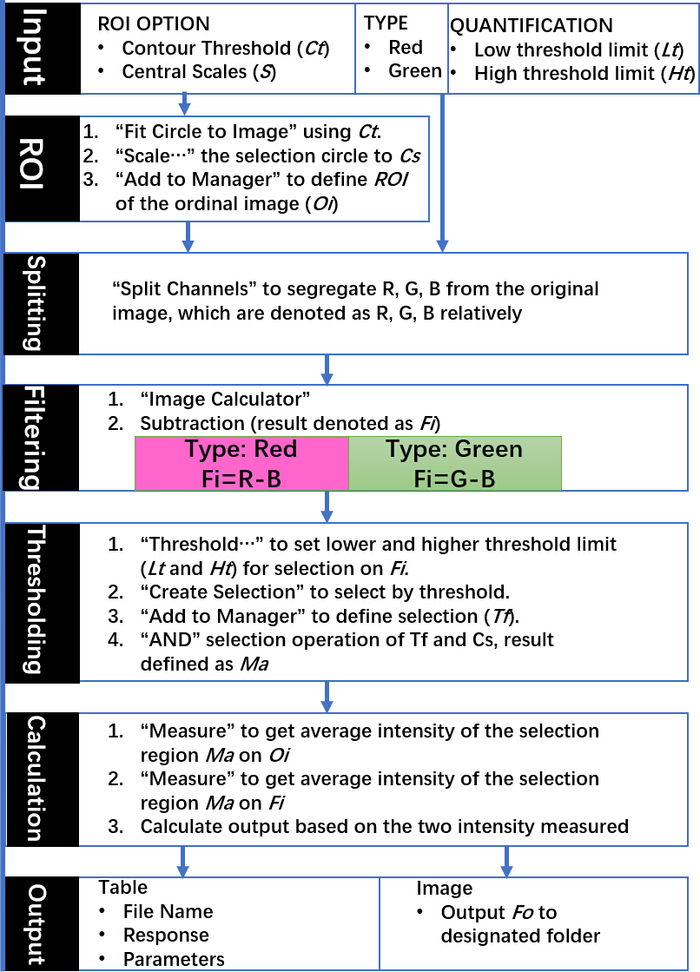
Figura 3: esquema dos comandos e saídas do algoritmo DSM. As etapas indicadas usam comandos nativos do ImageJ e todos os parâmetros de entrada são selecionados de acordo com os requisitos de experimentos através da interface gráfica do usuário. Este número foi adaptado com a permissão de Sun, D. et al. método de redução de ruído para quantificar a dispersão de luz de nanopartículas em imagens de campo distante de microscópio de campo escuro de baixa ampliação. Química analítica. 88 (24), 12001-12005 (2016). Copyright (2016) sociedade química americana. Por favor clique aqui para ver uma versão maior desta figura.
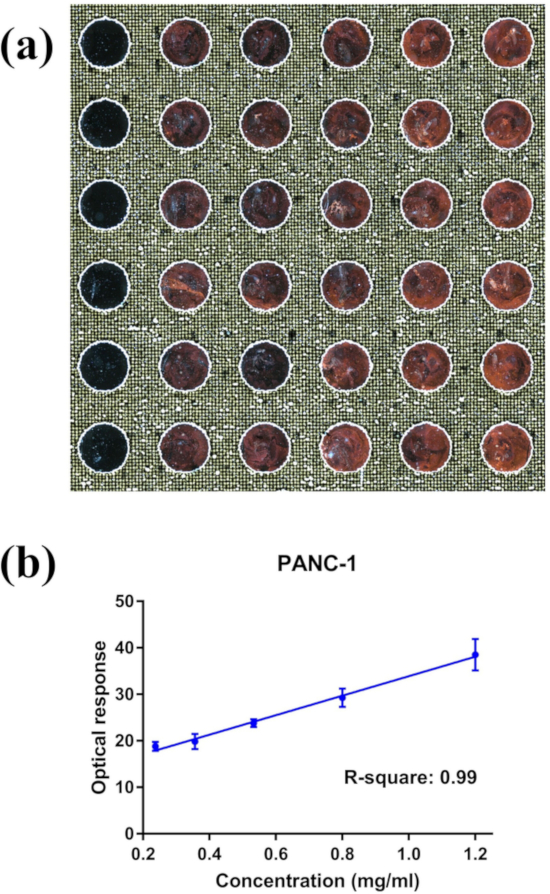
Figura 4: imagens representativas do nPES LMDFM e dados de saída do DSM. (A) imagem de lmdfm de um ensaio de NPEs analisando um gradiente de concentração de exosomas Panc-1 e (B) a correlação linear do sinal óptico e a concentração de exossomo deste slide (da esquerda para a direita: 0,24, 0,356, 0,53, 0,80, 1,20 μg/μl respectivamente para cada coluna). Os dados são apresentados como média ± SE, n = 6, com coeficiente de correlação de Pearson R2 = 0,99 e coeficiente de variação para as repetições de cada concentração < 0,2. Por favor clique aqui para ver uma versão maior desta figura.
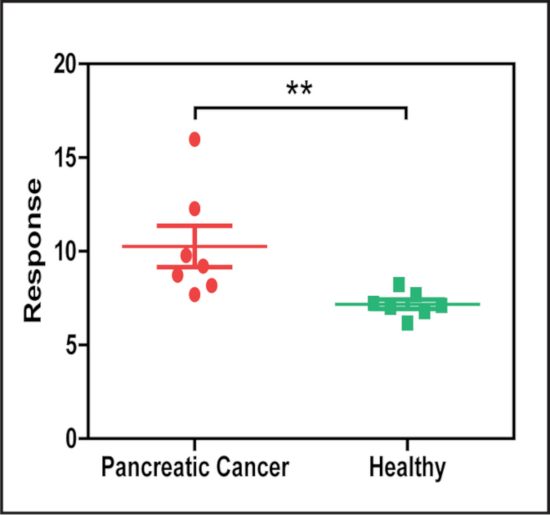
Figura 5: o sinal de lmdfm de EphA2+ exossomos difere no soro dos pacientes com e sem cancro pancreatic. Amostras de soro analisadas por NPEs usando um anticorpo de captura EphA2 (associada ao câncer) e um anticorpo de detecção CD9 (marcador geral de exossomo) exibiram uma diferença significativa na concentração de EphA2 + exosomas em amostras de soro de pacientes com e sem cancro pancreático (N = 7/grupo). Os resultados são apresentados como média ± SE. * *p = 0, 2 pelo teste U de Mann-Whitney (dois lados). Este valor foi modificado a partir de [Sun, D. et al. método de redução de ruído para quantificar a dispersão de luz de nanopartículas em imagens de campo distante do microscópio de campo escuro de baixa ampliação. Química analítica. 88 (24), 12001-12005 (2016)]. Por favor clique aqui para ver uma versão maior desta figura.
Discussão
Os exosomes levantam-se das invaginações reguladas da membrana endossomo exterior que produzem corpos multivesiculares, um subconjunto especializado dos endossomos que contêm um grande número vesículas intraluminal que se submetem à fusão com a membrana de plasma para liberar maduro exosomas no espaço extracelular. Devido a esta via de biogênese, os exosomas podem transportar fatores ligados à membrana associados a frações de membrana que compõem ou se fundem com a membrana endossomo, bem como vários tipos diferentes de componentes citosónicos e, assim, contêm cargas de proteínas, DNA e vários subtipos de RNA (mRNAs, microRNAs, RNAs de longa não codificação) que podem refletir o fenótipo de sua célula de origem20. Uma vez que os exosomas são secretados pela maioria, se não todos os tipos de células, podem apresentar aumento da secreção de células doentes ou malignas, e se acumulam na maioria dos fluidos corporais, os exosomas são objeto de investigação abrangente e sistemática como uma promissora minimamente meios invasivos para detectar condições específicas da doença e monitorar suas respostas ao tratamento21.
A isolação exossomo, que é exigida para a maioria de análises exossomo atuais, é um procedimento demorado e labor-intensivo, restringindo a tradução clínica de biomarcadores exossomo-associados com relevância médica potencial. Muitos métodos comuns de isolamento (ultracentlee, tamanho-exclusão, precipitação, etc.) muitas vezes não distinguem suficientemente os exosomas (30 – 100 nm) de Microvesículas (100 – 1.000 nm) e de corpos apoptóticos (100 – 5000 nm) devido a sobreposições em suas faixas de tamanho ou Propriedades físicas ou pode danificar a integridade exossomo15. Novas abordagens estão em desenvolvimento que podem permitir análises exossomo mais rápidas, mas não é claro como viável pode ser implementar muitas dessas plataformas em configurações clínicas devido.
Neste relatório, nós apresentamos uma aproximação nova que permita a quantificação do exossomo da elevado-taxa de transferência nanopartícula-baseada usando baixas imagens escuras do microscópio do campo da ampliação. Este método não exige a purificação exossomo, o equipamento dedicado caro, ou a perícia técnica nova e deve assim ser passíveis à tradução rápida em a maioria de pesquisa e de ajustes clínicos. Nosso ensaio pode ser aplicado para quantificar precisamente a concentração de uma população alvo exossomo que carrega um biomarcador específico quando os resultados do ensaio são comparados a uma curva padrão, uma vez que nossos resultados exibem uma forte correlação linear (R2 = 0,99) entre a resposta óptica e a concentração de exossomo. Para demonstrar o potencial do mundo real dessa abordagem, fornecemos dados onde empregamos esse método para quantificar a concentração de um biomarcador exossomo associado ao câncer pancreático em amostras de soro obtidas de pacientes com e sem cancro pancreático.
Em LMDFM, o poço inteiro da amostra é imaged para evitar o viés de seleção encontrado em análises de DFM da elevado-ampliação, onde os usuários devem diretamente escolher os campos da amostra a capturar para a análise subseqüente da imagem, mas são sujeitos aos artefatos de espalhamento da luz da superfície irregularidades, incluindo arranhões e detritos amostrais. Este fundo pode ser reduzido para detectar o sinal derivado de exosome alvo usando nosso algoritmo de redução de ruído DSM que é executado no programa de análise de imagem NIH, ImageJ, mas ainda deve ser tomado para evitar a introdução de tais artefatos que podem reduzir a gama dinâmica de o ensaio.
Materiais utilizados neste ensaio:
As corrediças multi-well de SuperProtein A/G que prendem 1 μL/poço foram compradas de Arrayit Corporaçõ (AGMSM192BC). Nanopartículas foram obtidas de Nanopartz (C12-25 -650-TN-DIH-50-1, 6,4 x 1012/ml). Imagens DFM foram capturadas com uma câmera digital Nikon DiR2 anexado a um microscópio Nikon ti-eclipse, com iluminação consistente e um tempo de exposição de 1/220 s. A linha de células PANC-1 utilizada neste estudo foi adquirida na coleção de cultura de tipo americano.
Divulgações
Os autores declaram que não têm interesses financeiros concorrentes.
Agradecimentos
O trabalho foi apoiado primeiramente pelo financiamento da pesquisa fornecido por NIH (U01CA214254, R01HD090927, R01AI122932, R01AI113725 e R21Al126361-01), concessão nova do investigador da Comissão de pesquisa biomédica de Arizona (ABRC).
Materiais
| Name | Company | Catalog Number | Comments |
| Eppendorf Repeater stream | Fisher Scientific | 05-401-040 | |
| Eppendorf Research plus | Eppendorf | 3120000011 | 0.1 – 2.5 µL, dark gray |
| Functionalized Gold Nanorods | Nanopartz | C12-25-650-TN-DIH-50-1 | In vitro neutravidin polymer functionalization |
| HulaMixer Sample Mixer | Thermo Fisher Scientific | 15920D | |
| Incu-shaker 10L | Benchmark Scientific | H1010 | |
| Inverted Research Microscope | Nikon | Ti-DH | With Dark field condenser, DS-Ri2 camera, and Ti-SH-U universal holder, and motorized stage |
| NIS-Elements | Nikon | Microscope imaging software | |
| Phosphate Buffered Saline (1X) | GE Healthcare Life Sciences | SH30256.02 | HyClone |
| Protein A/G Treated Glass Substrate Slides | Arrayit Corp. | AGMSM192BC | Premium microarray substrate |
| Q500 Sonicator | Qsonica, LLC | Q500-110 | With standard probe (#4220) |
| Superblock blocking buffer | Thermo Scientific | ||
| TWEEN 20 | Sigma Life Sciences | 9005-64-5 |
Referências
- Andaloussi S, E. L., Mager, I., Breakefield, X. O., Wood, M. J. Extracellular vesicles: biology and emerging therapeutic opportunities. Nature Reviews Drug Discovery. 12 (5), 347-357 (2013).
- Choi, D. S., Kim, D. K., Kim, Y. K., Gho, Y. S. Proteomics, transcriptomics and lipidomics of exosomes and ectosomes. Proteomics. 13 (10-11), 1554-1571 (2013).
- Schorey, J. S., Cheng, Y., Singh, P. P., Smith, V. L. Exosomes and other extracellular vesicles in host-pathogen interactions. EMBO Reports. 16 (1), 24-43 (2015).
- Hoshino, A., et al. Tumour exosome integrins determine organotropic metastasis. Nature. 527 (7578), 329-335 (2015).
- Zaborowski, M. P., Balaj, L., Breakefield, X. O., Lai, C. P. Extracellular Vesicles: Composition, Biological Relevance, and Methods of Study. Bioscience. 65 (8), 783-797 (2015).
- Liang, K., et al. Nanoplasmonic Quantification of Tumor-derived Extracellular Vesicles in Plasma Microsamples for Diagnosis and Treatment Monitoring. Nature Biomedical Engineering. 1, (2017).
- Severino, V., et al. Extracellular Vesicles in Bile as Markers of Malignant Biliary Stenoses. Gastroenterology. 153 (2), e498 495-504 (2017).
- Osteikoetxea, X., et al. Detection and proteomic characterization of extracellular vesicles in human pancreatic juice. Biochemical and Biophysical Research Communications. 499 (1), 37-43 (2018).
- Bulacio, R. P., Nosetto, E. C., Brandoni, A., Torres, A. M. Novel finding of caveolin-2 in apical membranes of proximal tubule and first detection of caveolin-2 in urine: A promising biomarker of renal disease. Journal of Cellular Biochemistry. , (2018).
- Nair, S., Tang, K. D., Kenny, L., Punyadeera, C. Salivary exosomes as potential biomarkers in cancer. Oral Oncology. 84, 31-40 (2018).
- Kim, J. E., et al. Diagnostic value of microRNAs derived from exosomes in bronchoalveolar lavage fluid of early-stage lung adenocarcinoma: A pilot study. Thoracic Cancer. 9 (8), 911-915 (2018).
- Boussadia, Z., et al. Acidic microenvironment plays a key role in human melanoma progression through a sustained exosome mediated transfer of clinically relevant metastatic molecules. Journal of Experimental & Clinical Cancer Research. 37 (1), 245(2018).
- An, M., Wu, J., Zhu, J., Lubman, D. M. Comparison of an Optimized Ultracentrifugation Method versus Size-Exclusion Chromatography for Isolation of Exosomes from Human Serum. Journal of Proteome Research. , (2018).
- Brenner, A. W., Su, G. H., Momen-Heravi, F. Isolation of Extracellular Vesicles for Cancer Diagnosis and Functional Studies. Methods in Molecular Biology. 1882, 229-237 (2019).
- Li, P., Kaslan, M., Lee, S. H., Yao, J., Gao, Z. Progress in Exosome Isolation Techniques. Theranostics. 7 (3), 789-804 (2017).
- Sun, D., et al. Noise Reduction Method for Quantifying Nanoparticle Light Scattering in Low Magnification Dark-Field Microscope Far-Field Images. Analytical Chemistry. 88 (24), 12001-12005 (2016).
- Sun, D., Hu, T. Y. A low cost mobile phone dark-field microscope for nanoparticle-based quantitative studies. Biosensors and Bioelectronics. 99, 513-518 (2018).
- Koshikawa, N., Minegishi, T., Kiyokawa, H., Seiki, M. Specific detection of soluble EphA2 fragments in blood as a new biomarker for pancreatic cancer. Cell Death & Disease. 8 (10), e3134(2017).
- Clayton, A., Turkes, A., Navabi, H., Mason, M. D., Tabi, Z. Induction of heat shock proteins in B-cell exosomes. Journal of Cell Science. 118 (Pt 16), 3631-3638 (2005).
- Henne, W. M., Buchkovich, N. J., Emr, S. D. The ESCRT pathway. Developmental Cell. 21 (1), 77-91 (2011).
- Panagiotara, A., Markou, A., Lianidou, E. S., Patrinos, G. P., Katsila, T. Exosomes: a cancer theranostics road map. Public Health Genomics. 20 (2), 116-125 (2017).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados