Method Article
Colonoid humana monocamadas para estudar interações entre epitélio Intestinal do hospedeiro, comensais e patógenos
Neste Artigo
Resumo
Aqui, apresentamos um protocolo para a cultura humana enteroid ou monocamadas de colonoid que têm a função de barreira intacta para estudar interações de epitélio-microbiota de acolhimento a nível celular e bioquímico.
Resumo
3-dimensional (3D) enteroid ou colonoid as culturas humanas derivadas de células-tronco base cripta são atualmente o modelo mais avançado ex vivo do epitélio intestinal. Devido a suas estruturas fechadas e significativa apoio da matriz extracelular, culturas 3D não são ideais para estudos de patógeno-hospedeiro. Enteroids ou colonoids podem ser crescidas como epiteliais monocamadas na cultura de tecidos permeáveis membranas para permitir a manipulação de ambos luminal e superfícies de celular basolateral e acompanhamento de fluidos. Essa acessibilidade aprimorada de superfície luminal facilita a modelagem de interações epiteliais bacteriano-host, tais como a capacidade de muco-degradante de enterohemorrhagic Escherichia coli (EHEC) no epitélio do cólon. Um método para a fragmentação da cultura 3D, monocamada semeadura e medições de resistência elétrica (TER) transepitelial para monitorar o progresso em direção a confluência e diferenciação são descritos. Diferenciação de monocamada Colonoid produz muco secretado que pode ser estudado através das técnicas de imunofluorescência ou immunoblotting. Mais geralmente, monocamadas de enteroid ou colonoid permitem uma plataforma fisiologicamente relevantes avaliar populações de células específicas que podem ser direcionadas pela microbiota patogênica ou comensal.
Introdução
Colonoids, enteroids e organoids intestinal conduziram a muitos avanços em compreender o comportamento de células-tronco, desenvolvimento intestinal, função de barreira/transporte e diferenciação celular. 1 no entanto, cultura 3D limita o estudo da interação de epiteliais patógeno hospedeiro porque o lúmen não é diretamente acessível às bactérias ou factores de virulência, a menos que as estruturas fechadas são submetidas a microinjeção. 2 , 3 além disso, secretada materiais tais como pequenas moléculas, proteínas, ou muco não pode ser facilmente amostrado de cultura 3D para a análise a jusante. O impacto de agentes patogénicos na barreira epitelial função4 e íon transporte5 foi avaliado em cultura 3D usando corantes fluorescentes e microscopia de lapso de tempo, mas monocamadas cultivadas em cultura de tecidos permeáveis suporta são passíveis de técnicas adicionais como TER medição e gravação de braçadeira Ussing câmara/tensão. 6 , 7
Inúmeras publicações têm descrito protocolos para 2D ou monocamada de cultura de enteroids/colonoids. Materiais encontrados para promover a fixação da célula epitelial incluem colágeno hidrogel,8,9 0.1% gelatina,10 camada fina murino sarcoma de derivados da membrana basal matrix (BMM),11,12, 13 e colágeno humano IV. 6 , 7 , 14 , 15 , 16 , 17 a semeadura de abordagens incluem fragmentação mecânica pipetando6,14,15 e/ou dissociação de fatores de aderência de célula usando a tripsina,10,11 Dispase,13 ou EDTA. 9 alguns protocolos usam cultura de tecidos não-porosa plasticware para propagação, mas isso restringe acesso basolateral, para a maioria dos aplicativos dependem de inserções de cultura de tecido permeável. Documentação de formação estável monocamada confluente e manutenção varia amplamente entre as publicações. Além disso, crescimento e diferenciação composições de mídia para as culturas humanas diferem entre os vários grupos e continuam a evoluir à medida que mais pesquisadores adotam e ajustar a metodologia para se adequar a sua aplicação e dos recursos disponíveis.
Para lidar com as limitações da cultura de epitélio intestinal 3D em estudos de interação patógeno-hospedeiro, apresentamos um protocolo modificado para a conversão 3D enteroids humana ou colonoids para uma monocamada. Depois de alcançar a confluência em um estado imaturo de cripta, retirada dos fatores de crescimento WNT3A, RSPO1 e os inibidores da-83-01 e SB 202190 leva à diferenciação representativa de pequenas vilosidades intestinais ou superfície epitélio do cólon. Descrevemos o matrix ideal, humana de colágeno tipo IV, para revestir as inserções e obter fragmentos uniformes de enteroid ou colonoid para o chapeamento. Demonstramos que o presente protocolo produz uma monocamada confluente com uma alta de monocamadas de ter Colonoid secretam uma camada de muco espesso apical, permitindo estudos ex vivo de interação patógeno-muco via immunoblotting ou immunostaining. Um procedimento de fixação modificados para preservar a camada de muco do colo para immunostaining também é descrito. Este método visa proporcionar um modelo tractable para estudar as primeiras interações hospedeiro-patógeno intestinal após infecção.
Protocolo
Este protocolo é baseado em estudos publicados anteriormente pelos autores. 6 , 7 , 14 , 15 , 16 as etapas a seguir devem ser efectuadas em um gabinete de biossegurança estéril usando técnicas assépticas apropriadas. Todos os métodos envolvendo espécimes humanos foram aprovados pelo institucional Review Board da Johns Hopkins University School of Medicine (IRB NA_00038329).
1. casaco inserções de cultura de células com matriz extracelular
- Prepare-se 5 mL da solução estoque colagénio IV (1mg/mL) em 100 mM de ácido acético. Deixe descansar a 4 ° C por aproximadamente 4h para totalmente hidrato/dissolver.
- Alíquota do colagénio IV solução e armazenar a 4 ° C (≤ 1 mês) ou a-20 ° C (> 1 mês).
- Imediatamente antes do seu revestimento, dilua a solução-mãe colagénio IV em água de grau de cultura de tecido estéril para uma concentração final de 34 μg/mL. Para placa de 24 insere cultura celular, casaco cada um insert com 100 μL de solução que corresponde a 10 μg/cm2.
- Incubar a placa em uma padrão CO2 tecido cultura incubadora a 37 ° C para ≥ 2 h, ou por conveniência, selar as bordas da placa com a película de parafina e incubar a 4 ° C durante a noite ou até 1 semana.
2. isolar enteroids/colonoids da cultura 3D
Nota: Enteroids ou colonoids são estabelecidas de biópsias de doador e mantidas em cultura 3D de acordo com protocolos padrão descrita anteriormente. 14 , 18 brevemente, criptas são colhidas biópsias intestinais ou ressecções via quelação e agitação mecânica. As criptas são lavadas, colhidas e banhadas em BMM. Mídia de expansão (descrita na etapa 4.2) é adicionada à cultura e substituída a cada 2 dias. Formação de cultura 3D é visível dentro de horas após o chapeamento.
- Aspire o meio de cultura da placa de 24 poços e substituir com 1 mL de solução de colheita gelada (ver Tabela de materiais) por poço.
- Use um raspador de celular mini para desalojar e terminar a pelota de matriz de membrana basal, dando especial atenção a qualquer material perto das bordas bem.
- Agite a placa em um agitador orbital em aproximadamente 200 rpm, 4 ° C por 30-45 min.
3. dissociar 3D enteroids/colonoids
- Use uma pipeta de canal único P200 ou uma pipeta multicanal equipado com pontas de filtro estéril para triture a suspensão de eritrócitos.
- Piscina da célula suspension(s) em um frasco cônico de 15 mL. Use vários frascos se suspensão total volume é > 6 mL.
- Adicionar um volume igual de meio DMEM/F12 avançado contendo 10 mM HEPES, dipeptídeo de L-alanil-L-glutamina (1x) e penicilina-estreptomicina (1x) (média de lavagem). Inverter o tubo 3 - 4 vezes para misturar. Centrifugação a 300 x g, 10 min, 4 ° C.
- Opcionalmente, Aspire o meio de lavagem e substituir com 0,5 mL/poço tripsina (ver Tabela de materiais). Resuspenda colonoids com uma pipeta P200, tampa do tubo e colocar em banho maria a 37 ° C por aproximadamente 2 min. imediatamente remover o tubo, adicionar meio de lavagem até um volume final de 10 mL e repetir a centrifugação como passo 3.3.
Nota: Este passo pode ser preferível se trituração não está resultando em fragmentos de tamanhos uniforme.
- Opcionalmente, Aspire o meio de lavagem e substituir com 0,5 mL/poço tripsina (ver Tabela de materiais). Resuspenda colonoids com uma pipeta P200, tampa do tubo e colocar em banho maria a 37 ° C por aproximadamente 2 min. imediatamente remover o tubo, adicionar meio de lavagem até um volume final de 10 mL e repetir a centrifugação como passo 3.3.
4. chapeamento da suspensão de enteroid/colonoid
- Se revestido inserções foram armazenadas a 4 ° C, se equilibrar em uma incubadora de cultura de tecidos de2 °C/5% CO 37 pelo menos 30 min antes de chapeamento de célula.
- Para cada inserção ser chapeada, prepare-se 1 mL de meio de expansão quente (entre 25 e 37 ° C) (EM) e adiciona 10 μM Y-27632 e 10 μM CHIR 99021.
Nota: EM é composto de Advanced DMEM/F12 contendo B27 (1x), 50% WNT3A condicionado médio, médio do RSPO1 condicionado de 15%, 10% Noggin condicionado médio, 50 ng/mL EGF humano, 500 nM A-83-01, 10 μM SB 202190, antibiótico/antimicótico cocktail (1x), 10 mM HEPES, L-alanil-L-glutamina dipeptídeo (1x) e penicilina-estreptomicina (1x) (ver Tabela de materiais). - Aspirar o meio de lavagem do tubo contendo o centrifugado e ressuspender em um volume de los suficientes para produzir pelo menos 100 μL/inserir.
- Aspirar a solução IV de colágeno de cada inserção e duas lavagens com 150 µ l de meio de lavagem por inserção.
- Pipeta 600 μL de travessão para o espaço por baixo de cada inserção.
- Pipetar 100 μL da suspensão de células em cada inserção.
- Devolve a chapa para a incubadora de cultura de tecidos e sair sem ser perturbado pelo menos 12 h.
Nota: Evite tremer ou agudamente inclinando a placa para evitar a distribuição desigual de colonoid fragmentos da membrana de inserção. - Monitorar o acessório de célula e espalhar-se para formar uma monocamada confluente, colocando a placa em um microscópio de contraste de fase sob um 2.5 x-10 x lente objetiva.
- Após 1-2 dias, a maioria dos fragmentos devem aderir para a matriz de colagénio. Refrescar o meio de cultura e interromper o tratamento com Y-27632 e CHIR 99021. Continue a atualizar o médio a cada 2-3 dias até confluente. Confluência é geralmente alcançada em 7 a 10 dias.
5. medida transepitelial resistência elétrica
- Anexe o eletrodo pistas e poder sobre o voltohmmeter epitelial (EVOM). Verifique se a função de medição é definida a ohms.
- Mergulhe as pontas de eletrodo brevemente em etanol a 70% e seque com um tecido de laboratório. Equilibrar o eléctrodo em 5 mL de meio de lavagem por cerca de 5 min.
- Mergulhe a ponta do eletrodo menor no meio de inserir e orientar a ponta do eletrodo mais a placa bem menor. Manter o eletrodo em uma orientação vertical completo até a leitura EVOM é relativamente estável, ou por não mais de 60 s.
Nota: Se o assembly de eletrodo é inclinado longe vertical, ou a altura da ponta é ajustada incorretamente, a ponta mais curta pode entrar em contacto com e perturbar a monocamada de células. - Compare as medições EVOM para o valor obtido a partir de uma inserção sem célula submergida em meio de cultura para avaliar o progresso em direção a confluência ou diferenciação.
6. diferenciação de monocamadas confluentes do enteroid/colonoid
- Troca EM por meio de diferenciação (DM) e continuar a atualizar a mídia a cada dois dias até o dia 5. DM é EM que carece de WNT3A, RSPO1, A-83-01 e SB 202190.
- Continue a monitorar a TER para verificar que a diferenciação está correndo como o esperado. TER continuará a aumentar durante dias 1-5 de diferenciação. Monocamadas podem continuar a TER de aumentar e manter a viabilidade além do dia 5-6 mas apresentam resultados mais reprodutíveis e máximas quando usado em dia 5-6.
7. bacteriana infecção com comensais ou patogênicas de Escherichia coli
- Um dia antes da realização da infecção, lavar e alimentar monocamadas com antibiótico-livre EM ou DM.
- Use um loop de microbiologia estéril suavemente raspar a superfície de um estoque de glicerol bacteriana congelados e inocular 2 mL de caldo LB. Transferi o tubo para um padrão tremendo incubadora a 37 ° C por 12-16 h.
- Diluir 50 μL da cultura starter no caldo LB fresco 5 mL (1: 100) e continuar a incubação com agitação por 90 min. Isto irá produzir uma cultura de fase de registo com uma densidade de 105-106 formadoras unidades (cfu) / mL.
- Opcionalmente, confirmar a concentração bacteriana por medição da densidade óptica em 600 nm (OD600) em um espectrofotômetro.
- Spin para baixo a cultura bacteriana a 12.000 x g por 10 min, retire o sobrenadante e ressuspender as bactérias em sem antibióticos EM ou DM para uma concentração final de 107 UFC/mL.
- Adicione 10 μL da suspensão bacteriana para a inserção (concentração final 106 UFC/mL). Pipetar lentamente para misturar e evitar perturbar a camada de muco extracelular.
- Devolve a chapa para uma incubadora de cultura de tecido para o período desejado de infecção.
8. fixação para preservar e delgados muco
- Elevador da cultura de pilha insere da placa de 24 poços, inverter cuidadosamente em um laboratório de lenços de papel para remover o meio apical e limpar afastado líquido externo apegar-se a inserção da.
- Em uma nova placa 24, mergulhe a inserção em uma solução de 1:3 do ácido acético em etanol absoluto (solução de Clarke) por 10 min à temperatura ambiente.
- Inverta a inserção para remover o fixador e Re-hidratar as células em 1X PBS durante 10 min. Proceda com protocolos de imunocoloração padrão.
- Use uma lâmina de barbear para cortar ao redor do perímetro da membrana inserir. Transferir a membrana para um vidro de microscópio usando fórceps e apor uma lamela com meio de montagem.
9. preparação de lisados celulares para immunoblotting
Nota: Execute as seguintes etapas no gelo ou em uma sala fria.
- Aspire insert médio e lave uma vez com PBS.
- Adicionar 150 μL do lysis buffer contendo inibidores de protease (ver tabela de materiais) para a inserção e deixe repousar 5 a 10 min. do lysis contém pH de Tris 50mm 8, 150 mM de NaCl, 1% IGEPAL CA-630, 0,5% de sódio Deoxycholate do, 0,1% SDS, inibidor de protease de 1: 100 cocktail.
- Uso um celular mini raspador para remover as células da inserção e, em seguida, usar uma pipeta P200 para brevemente triture a suspensão e a transferência para um tubo de microcentrifugadora.
- Proceda à sonicação a suspensão com 5 x 1 pulsos de s em amplitude de 20% usando um microtip sonda (ver Tabela de materiais).
- Adicione o amortecedor do carregamento SDS-PAGE se imediatamente executar eletroforese ou alíquota e armazenar lysates a-20 ° C.
Resultados
Culturas humanas, enteroid e colonoid são crescidas como estruturas 3D, em seguida, dissociadas e fragmentadas para o chapeamento em inserções de cultura de célula IV-revestido de colágeno humano. O progresso da formação monocamada facilmente é monitorado diariamente através de microscopia de campo claro, mancha (Figura 1) e por um aumento na resistência elétrica transepitelial (TER) (Figura 2), que reflete a permeabilidade das junções apertadas para íons e correlaciona-se com a confluência de monocamada. A TER de uma inserção de 24-poço vazia é aproximadamente 50-100 Ω·cm2e ao atingir a confluência aumenta para aproximadamente 400-500 Ω·cm2. Todas as células epiteliais em monocamadas confluentes são ligadas por complexos juncionais detectados pelo anel F-Actina no perímetro da célula (Figura 1). Monocamadas na mídia de fator de crescimento completo representam o epitélio cripta-como proliferativo, constituído principalmente por ativamente dividindo pilhas que incorporam o nucleosídeo EdU analógico (Figura 2). Retirada dos fatores de crescimento, WNT3A e RSPO1 promove diferenciação, levando a culturas como vilosidades que faltam pilhas proliferating. Monocamadas diferenciadas contêm enterócitos (enteroids) ou colonocytes (colonoids) e desenvolvem células epiteliais intestinais especializadas, como ficou demonstrado em culturas 3D,18 , incluindo células de cálice e enteroendócrina. 15 monocamadas diferenciadas também demonstram um aumento significativo em TER (> 1000 Ω·cm2) (Figura 2), indicando maduras junções apertadas.
Na fisiologia humana normal, a camada epitelial intestinal separa nutrientes e espaço luminal de micróbio-enriquecido o ambiente estéril serosas. O epitélio firmemente regula o cross-talk entre estes dois compartimentos. Monocamadas de Enteroid e colonoid preservar esta propriedade importante compartimentalização como mostrado pela análise proteômica em tabela 1. Existem diferenças substanciais entre a composição de proteína apical e basolateral condicionado mídia coletadas de monocamadas enteroid diferenciada. Acesso a ambos os lados apicais e basolateral permite a recolha de ambos os sobrenadantes em uma maneira dependente do tempo para medição de secreção diferencial de outras moléculas tais como citocinas e quimiocinas. 15 , 17
Monocamadas são modelos altamente reprodutíveis e convenientes para detectar as alterações na expressão de proteínas usando imunocoloração e immunoblotting. Assim, muda em mucina 2 (MUC2) expressão por nocaute shRNA (KD) pode ser detectada usando ambas as técnicas (Figura 3). A transdução de shRNA foi executada em 3D colonoids e mantida em meios de seleção de antibiótico. Depois de verificar o KD, colonoids pode ser continuamente mantida como culturas 3D e/ou chapeado como monocamadas para fins experimentais. Além disso, MUC2 KD não afeta a eficiência de formação monocamada em comparação com a linha de colonoid parental do tipo selvagem. Coleção de monocamadas para immunoblotting é simples e semelhante aos protocolos descritos por linhas humano célula epitelial derivado de adenocarcinoma. Normalmente, cerca de 50 µ g ou mais de proteína total podem ser extraído de uma monocamada de inserção-crescido2 cm 0,33 único. Como mostrado na Figura 3, MUC2 é mal detectável em monocamadas de colonoid (UD) WT indiferenciadas, mas é altamente expresso em monocamadas diferenciadas do WT (DF). MUC2 está abaixo do nível de detecção em ambos UD e DF colonoid monocamadas transfectadas com MUC2 shRNA.
Importante, monocamadas são um modelo adequado para estudar interações hospedeiro-microbiana na superfície apical dos epitélios. Monocamadas Colonoid formam uma espessa camada de muco positivo MUC2 anexada em cima de diferenciação que não é facilmente permeada por commensal bactéria Escherichia coli HS (Figura 4), semelhante ao que foi sugerido no cólon humano normal. 19 no entanto, enterohemorrhagic Escherichia coli (EHEC), um patógeno humano do cólon, foi mostrado para ter a capacidade de destruir a camada de muco anexado MUC2 enriquecido para chegar à superfície apical do epitélio (Figura 4). 14 , 20 restantes MUC2 está presente apenas no interior das células do cálice.
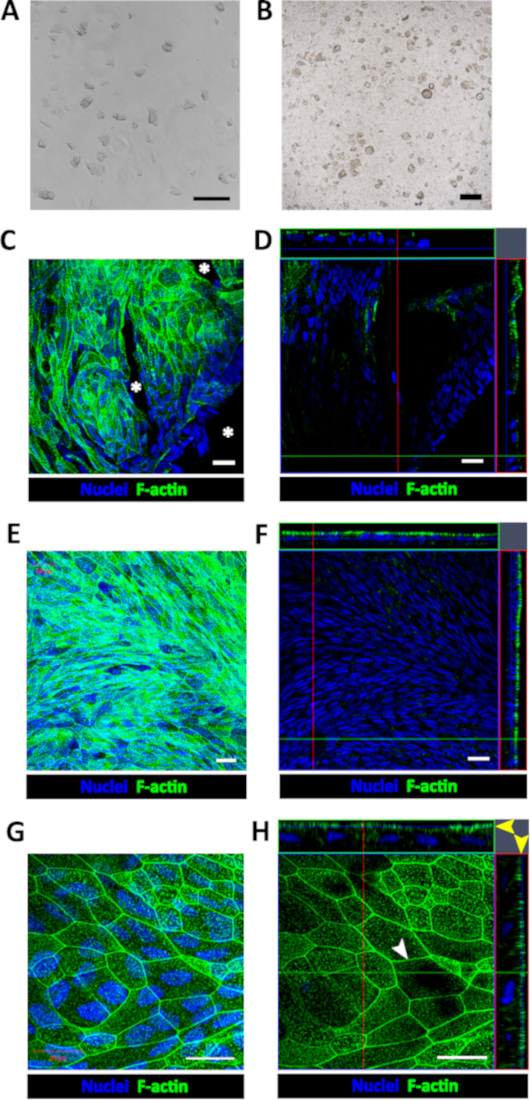
Figura 1: Criação de humanas monocamadas de enteroid/colonoid. (A) exemplo de colonoid fragmentos após a dissolução da BMM e trituração. (B) exemplo de inserção imediatamente após colonoid fragmentos de chapeamento. Escala da barra (A-B) = 200 μm. Representativa projeção de intensidade máxima (C) e (D) confocal Z-secção óptica com as projecções ortogonais correspondentes mostram que colonoid fragmentos semearam para formulário de filtros IV-revestido de colágeno humano várias ilhas monocamada 2-4 dias após a semeadura. As áreas livres de célula (asterisco) são identificáveis pela ausência de ambos nuclear (Hoechst 33342, azul) e F-Actina apical (faloidina, verde) coloração em C e D. representativa projeção de intensidade máxima (E) e (F) confocal optical seção com as projecções ortogonais correspondentes mostram uma monocamada confluente colonoid com superfície apical contínua detectado pelo F-Actina immunostaining aproximadamente 1 semana pós-semeadura. (G) alta magnificação de uma projeção de intensidade máxima representativa e secção de óptica confocal (H) com as correspondentes projecções ortogonais mostram que células em monocamadas colonoid confluentes formam o perijunctional F-Actina anéis (seta branca) e uma fronteira de escova apical imaturo (cabeças de seta amarela). Escala da barra (C-H) = 20 μm. Clique aqui para ver uma versão maior desta figura.
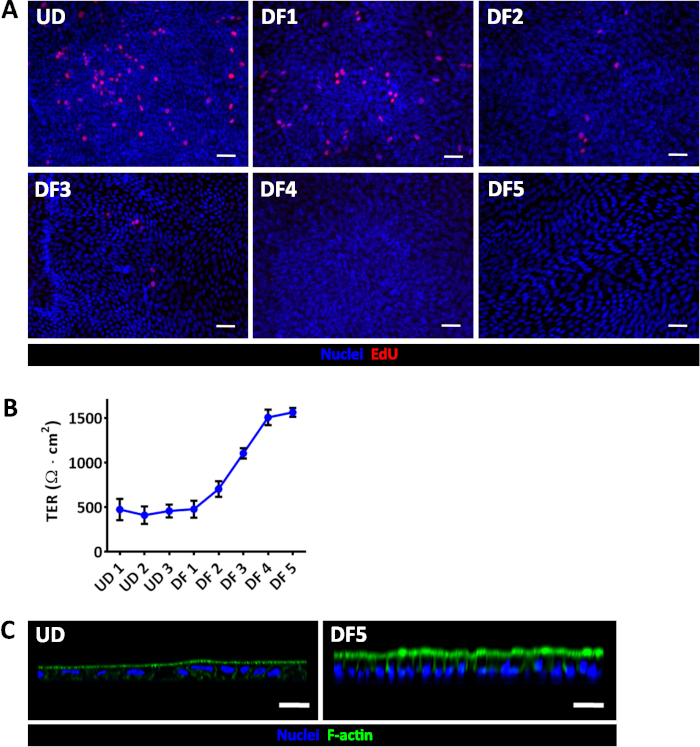
Figura 2: avaliação da diferenciação monocamada enteroid/colonoid. (A) incorporação de EdU (vermelho) demonstra uma perda progressiva da proliferação durante a diferenciação de monocamada jejunal. (B) TER média medições em monocamadas jejunal confluentes no meio de expansão ou diferenciação. Barras de erro representam SEM. UD, indiferenciado; DF, diferenciada. Os números correspondem aos dias sob a condição especificada; UD1 foi o primeiro dia de confluência, cerca de 1 semana após a semeadura. (C) monocamadas de jejunal UD têm células amplas, mais curtas e uma borda menos-maduras apical escova de actina-baseado de monocamadas de jejunal DF dia 5. Escala da barra (A, C) = 50 μm. Monocamadas todos foram descritos pelo menos 1 semana pós-semeadura e foram confluente antes de começar a diferenciação. Clique aqui para ver uma versão maior desta figura.
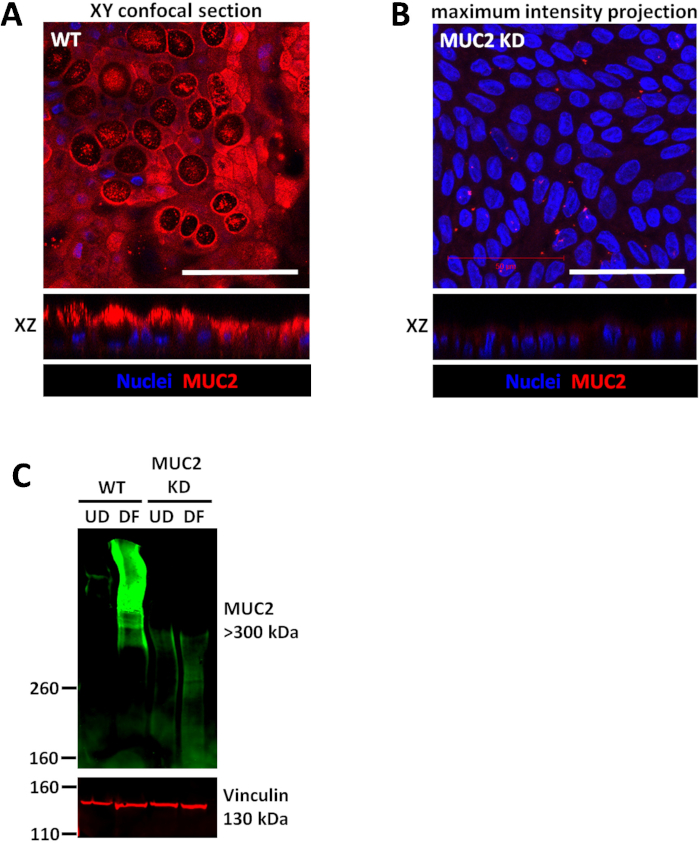
Figura 3: Colonoids tipo selvagem e shRNA transfectadas nocaute (KD) colonoids cada monocamadas de forma confluente. (A) imagens representativas do tipo selvagem (WT) humana colonoid confluente monocamada diferenciadas por 5 dias e (B) da mesma forma, crescida monocamadas derivado de culturas de colonoid MUC2 KD. Escala da barra (A-B) = 50 μm (C) representante immunoblot das culturas colonoid transfectadas com ovos mexido shRNA ou MUC2 shRNA demonstra que as mudanças na expressão da proteína devido a KD podem ser quantificadas por immunoblot. Clique aqui para ver uma versão maior desta figura.
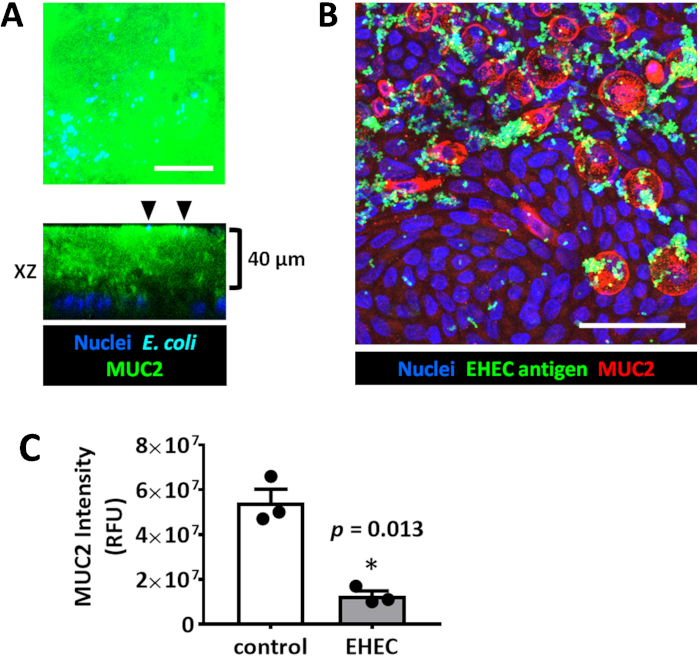
Figura 4: Enteroid/colonoid monocamadas são modelos adequados para estudar interações micróbio-hospedeiro luminal. Projeção de intensidade máxima representante do (A) e seção ótica ortogonal de uma monocamada colonoid mostra a apical MUC2 positivo muco camada grossa que não é permeável a Escherichia coli HS (setas pretas) sentado no muco apical superfície. A monocamada estava infectada por 6 horas com 106 UFC/mL HS. (B) projeção de intensidade máxima representante da colonoid humana monocamada apicalmente infectada com EHEC (106 UFC/mL, 6 horas). Escala da barra (A-B) = 50 μm. (C) medições de intensidade de imunofluorescência MUC2 mostram uma diminuição significativa no MUC2 em monocamadas de EHEC infectados colonoid comparadas aos controles não infectados. Barras de erro representam SEM. clique aqui para ver uma versão maior desta figura.
| Proteína | Número de adesão | Abundância de peptídeo |
| Basolateral Media | ||
| proteína ligadora de ácidos graxos, fígada [Homo sapiens] | NP_001434.1 | 1299 |
| criação-1 [Homo sapiens] | NP_005013.1 | 797 |
| apolipoproteína A-IV precursor [Homo sapiens] | NP_000473.2 | 744 |
| ecto-ADP-ribosyltransferase 4 precursor [Homo sapiens] | NP_066549.2 | 633 |
| gliceraldeído-3-fosfato desidrogenase isoform 1 [Homo sapiens] | NP_002037.2 (+ 1) | 616 |
| serotransferrin precursor [Homo sapiens] | NP_001054.1 (+ 1) | 572 |
| precursor de isoform 3 proteína ligadora de vitamina D [Homo sapiens] | NP_001191236.1 + (2) | 567 |
| catalase [Homo sapiens] | NP_001743.1 | 427 |
| Agrin isoform 1 precursor [Homo sapiens] | NP_940978.2 (+ 1) | 377 |
| lactotransferrin isoform 1 precursor [Homo sapiens] | NP_002334.2 (+ 1) | 262. |
| Mídia apical | ||
| precursor de fator 3 trifólio [Homo sapiens] | NP_003217.3 | 326 |
| precursor de fator 1 trevo [Homo sapiens] | NP_003216.1 | 304 |
| específicas de membrana basal heparan sulfato proteoglycan núcleo proteínas isoform um precursor [Homo sapiens] | NP_001278789.1 + (3) | 303 |
| FBLIM1-B isoform 1 [Homo sapiens] | NP_001157789.1 + (2) | 240 |
| precursor de fator 2 trifólio [Homo sapiens] | NP_005414.1 | 182 |
| excluído no precursor de b de isoforma para 1 proteína de tumores cerebrais malignos [Homo sapiens] | NP_015568.2 + (3) | 169 |
| queratina, tipo II do citoesqueleto 1 [Homo sapiens] | NP_006112.3 | 157 |
| miosina-9 [Homo sapiens] | NP_002464.1 | 150 |
| aminopeptidase N precursor [Homo sapiens] | NP_001141.2 (+ 1) | 145 |
| Agrin precursor [Homo sapiens] | NP_940978.2 (+ 1) | 112 |
Tabela 1. Uma lista parcial de peptídeos identificados por espectrometria de massa de cromatografia/tandem líquida apical e basolateral fluidos amostrados de diferenciadas jejunal monocamadas.
Discussão
Parâmetros críticos para a formação bem sucedida de monocamadas de enteroid/colonoid incluem 1) saudável, proliferando 3D culturas como matéria-prima; 2) revestimento de superfície de inserção de cultura celular com colágeno humano IV antes da semeadura de monocamadas; 3) fragmentação do 3D culturas mecanicamente ou enzimaticamente, mas não para o nível de célula única.
Durante o isolamento de enteroids/colonoids 3D, a velocidade de agitação ideal pode variar dependendo do diâmetro rotacional de um modelo determinado abanador. A intenção é não só agitar a placa para uma mistura eficiente, mas também evitar salpicos a suspensão de eritrócitos na tampa do prato ou em poços adjacentes. Períodos de incubação de 30 min de < podem render BMM residual, agarrando-se às células, que podem impedir o apego e a formação de monocamadas de células uniforme quando banhado em inserções. Incubação extensiva (≥ 1 h) produzirá a viabilidade celular significativamente diminuída.
A extensão da trituração necessária para dissociar 3D enteroids/colonoids pode variar com a destreza de rigidez e usuário do pistão. Recomenda-se exames periódicos do conteúdo bem em um microscópio de contraste de fase para determinar a uniformidade de fragmento durante a trituração, com um gol de aproximadamente 30 células por fragmento. Em vez de, ou além de trituração, a suspensão pode ser digerida brevemente com tripsina obter fragmentos menores de uniformes. Resumo de tripsina pode ser desejável se monocamadas contêm uma grande proporção de estruturas 3D entre uma caso contrário única camada epitelial. No entanto, a dissociação de células únicas não é desejável como isto diminui significativamente a viabilidade celular. Um poço de enteroids/colonoids cultivadas em uma gota BMM 35 μL geralmente será suficiente para preencher as inserções de 2-3, mas este fator pode variar dependendo do número e tamanho médio da enteroids/colonoids.
Colonoid monocamadas podem absorver um volume substancial de médio porte apical como eles diferenciar. Isto pode ser contornado através da aplicação de DM adicional para a câmara de inserção superior (150-200 μL), apesar de secagem parcial da câmara alta não parece afetar negativamente a viabilidade de monocamada ou função em ensaios a jusante. Depois de aproximadamente 4-5 dias de diferenciação, monocamadas colonoid desenvolvem muco extracelular, que pode ser visível como material grosso/gelatinoso na superfície das células após aspiração cuidadosa de DM.
Para a interação de EHEC com a camada de muco exterior, usamos rotineiramente o período de incubação e título bacteriano especificado no protocolo. No entanto, cepas bacterianas exclusivas inicialmente devem ser analisadas em várias concentrações e períodos de incubação para determinar os parâmetros adequados para o efeito desejado.
Durante a fixação de muco, aspirando a apical médio provavelmente removerá a maioria da camada de muco desanexado extracelular. Portanto, é importante que com cuidado, despeje o meio. Se o meio é retido na inserção pela tensão de superfície, use o canto de um tecido dobrado laboratório para quebrar a tensão superficial e pavio afastado a maioria de médio porte. Após fixação e coloração, a camada de muco pode ser involuntariamente desalojada ou achatada durante a montagem de uma lamela sobre o filtro de inserção. Para preservar a altura de muco, colocar o filtro em uma gota de montagem mídia e coloque cuidadosamente a lamela sobre o filtro. Não toque ou pressione para baixo sobre a lamela isto significativamente pode achatar a camada de muco.
Para formar uma monocamada polarizada de células em cultura 3D, é necessário reproduzir a interação entre integrinas de membrana basolateral das células epiteliais intestinais e proteínas da matriz extracelular (ECM). Uma matriz de proteoglicanos, colágeno IV, fibronectina e laminina constituem o ECM epitelial intestinal. 21 , 22 comparamos humana derivado de células laminina, fibronectina, colágeno IV e BMM murino-derivado, como revestimentos de inserção. Somente o colagénio IV apoiou a formação de estável, a longo prazo (até 4 semanas) confluentes monocamadas de enteroid/colonoid (figuras 1-2). Pequenas manchas de monocamada, como crescimento foram obtidas em cada uma das outras matrizes testadas, mas nestas regiões não conseguiram progredir a confluência. Uso de colágeno humano IV como o substituto de ECM tem uma série de vantagens. BMM derivados de Engelbreth-Holm-enxame (EHS) células de sarcoma murino não são de origem humana, considerando humano colágeno IV é comercialmente disponível e geralmente mais econômica. Além disso, o BMM é uma mistura complexa de proteínas com variabilidade inerente e pode conter fatores de crescimento secretados pelo sarcoma que afetam a expressão genética. 23
Interações entre as células epiteliais intestinais e o ECM subjacente em restituição epitelial intestinal é compreendido ainda incompleta. A importância do colágeno IV, mas não a laminina, como um ligante de adesão para o colonocytes humano24 e potenciador da cripta intestinal célula epitelial restituição25 sugeriu usando anticorpos contra colágeno que impedia o anexo IV . Epitelial-expresso β1-integrina parece ser importante para a interação de ECM, como um anticorpo específico para β1-integrina significativamente bloqueado a adesão de colonocytes de colagénio IV tipo. 24 enquanto colágeno IV fornece uma confiável ECM para enteroid/colonoid monocamada formação em inserções, epiteliais remodelação do ECM ao longo do tempo não ainda foi avaliada neste modelo, nem tem a influência de misturas de ECM mais complexas e definidas de colágeno IV, laminina e fibronectina.
Enteroids/colonoids humanas em formato de monocamada (figuras 1-4) permitem manipulações e amostragem que seria complicado ou impossível de alcançar usando 3D culturas matriz-incorporado. Enteroids/colonoids 3D variam em tamanho, complexidade estrutural e volume luminal, e, portanto, microinjeção de micróbios ou pequenas moléculas são difíceis de quantificar com precisão. Além disso, em contraste com as monocamadas (tabela 1), culturas 3D impedem acesso direto para as superfícies luminal e basolateral para medir íons, nutrientes, citocinas ou fatores secretados associados aos processos fisiológicos ou fisiopatológicas . Confluência (figuras 1-2) é uma das principais propriedades da monocamada de enteroid/colonoid necessária para estabelecer não só os gradientes fisiológicos de nutrientes, íons e outras macromoléculas,16 , mas também criando a barreira adequada entre a microbiota luminal e mesenquimais/imune celular-preenchido serosas ambientes estéreis. Essas facetas são importantes a considerar para futuros estudos que incorporam monocamadas de enteroid/colonoid com tipos de células mesenquimais, imune ou neuronal para construir modelos fisiológicos mais complexos.
Observou as limitações do modelo de inserção de cultura celular descrito aqui incluem falta de forças físicas como fluido tensão de cisalhamento e estiramento/compressão mecânica (peristaltismo) e a ausência de um ambiente anaeróbio apical normalmente experimentada por intestinal epitélios in vivo. Esses elementos têm o potencial de ser abordadas com mais sofisticadas plataformas microphysiological,26 , mas eles também exigem a despesa adicional, equipamento e experiência para implementar. Culturas tanto 3D e monocamada também estão sem interações com ou contribuições da microbiome intestinal, populações de células do estroma e o sistema imunológico, a menos que esses componentes são adicionados propositadamente.
Futuras aplicações da monocamada enteroid/colonoid em inserções de cultura de célula IV-revestido de colágeno podem incluir outros estudos de interação microbiana patogénica ou comensal, droga ou nutriente absorção, toxicidade, metabolismo, função de barreira e funcional realce catalisada por co-cultura com tipos adicionais de células intestinais. Avaliações podem ser feitas apenas entre os epitélios dos doadores saudáveis, mas também de indivíduos com mutações genéticas ou distúrbios intestinais, desde que os fenótipos in vivo são estabelecidos para ser preservado em ex vivo enteroid/colonoid cultura.
Divulgações
Os autores não têm nada para divulgar.
Agradecimentos
Este trabalho foi apoiado por subsídios NIH P01 AI125181, K01 DK106323 (JGI) e K01 DK113043 (JFA). Agradecemos a James Kaper (Universidade de Maryland, Baltimore, MD, EUA) para a prestação de e. coli estirpe HS e EHEC. Também reconhecemos a fisiologia integrada e núcleos de imagem do Conte digestivo doença básica de Hopkins e Translational Research Center do núcleo (P30 DK089502) e espectrometria de massa do Johns Hopkins e Proteomics Core.
Materiais
| Name | Company | Catalog Number | Comments |
| 2-Amino-2-(hydroxymethyl)-1,3-propanediol | Sigma | T4661 | Tris base, Component of lysis buffer |
| A-83-01 | Tocris | 2939 | ALK4/5/7 inhibitor |
| Acetic acid, glacial | Fisher Scientific | A38 | |
| Advanced DMEM/F12 | Life Technologies | 12634-010 | Component of growth medium |
| Alexa Fluor 488 phalloidin | Life Technologies | A12379 | Fluorescent probe for F-actin |
| Antibiotic/antimycotic cocktail | Invivogen | ant-pm-2 | Primocin (100x) |
| B27, 50x | Life Technologies | 17504-044 | Component of growth medium |
| Cell culture inserts | Corning | 3470 | Transwell, PET membrane, 0.4 μm pore, 24-well plate |
| CHIR 99021 | Tocris | 4423 | GSK3β inhibitor |
| Click-iT Plus EdU Alexa Fluor 594 Imaging Kit | Life Technologies | C10639 | |
| Collagen IV, from human placenta | Sigma | C5533 | |
| Cultrex Organoid Harvesting Solution | Trevigen | 3700-100-01 | Depolymerizes basement membrane matrix |
| Enterohemorrhagic Escherichia coli (EHEC) | Kaper lab, University of Maryland | ||
| Epithelial voltohmmeter | World Precision Instruments | EVOM2 | |
| Epithelial voltohmmeter electrode | World Precision Instruments | STX3 | |
| Escherichia coli strain HS | Kaper lab, University of Maryland | ||
| Ethanol, absolute | Pharmco | 111000200 | |
| FluorSave mounting medium | Millipore | 345789 | For mounting insert membrane on microscope slide |
| GlutaMAX | Life Technologies | 35050-061 | L-alanyl-L-glutamine dipeptide, 200 mM |
| HEK293T/Noggin-Fc cell line | van den Brink lab, Tytgat Institute for Liver and Intestinal Research | For production of Noggin conditioned medium | |
| HEK293T/RSPO1-Fc-HA cell line | Trevigen | 3710-001-K | For production of Rspondin-1 conditioned medium |
| HEPES, 1 M | Life Technologies | 15630-080 | Component of growth medium |
| Heraeus Multifuge X1R Centrifuge | Thermo Fisher Scientific | 75004250 | |
| Hoechst 33342 | Life Technologies | H3570 | Fluorescent nuclear dye |
| Human epidermal growth factor (EGF) | R&D Systems | 236-EG-01M | Component of growth medium |
| IGEPAL CA-630 | Sigma | I8896 | Component of lysis buffer |
| Inverted cell culture light microscope | Olympus | CKX51 | |
| LB broth | EMD Millipore | 1.10285.0500 | |
| L-WNT3A cell line | ATCC | CRL-2647 | For production of Wnt3a conditioned medium |
| Matrigel, growth factor reduced | Corning | 356231 | Basement membrane matrix for 3D culture |
| Mini cell scraper | United BioSystems | MCS-200 | |
| MUC2 antibody, mouse monoclonal | Abcam | ab11197 | Use at 1:100 for immunostaining, 1:500 for immunoblotting |
| MUC2 shRNA lentiviral particles | GE Dharmacon | RHS4531 | GIPZ lentiviral shRNA, ID: 4583 |
| Orbital shaker | Grant Instruments | PSU-10i | |
| Penicillin-streptomycin, 100x | Life Technologies | 15140-122 | Component of growth medium |
| Phosphate buffered saline | Corning | 21-031 | |
| Probe sonicator with microtip | Branson Ultrasonics | 450 | |
| Protease inhibitor cocktail for mammalian cells | Sigma | P8340 | Component of lysis buffer |
| SB 202190 | Tocris | 1264 | p38 MAPK inhibitor |
| Sodium chloride | Sigma | S3014 | Component of lysis buffer |
| Sodium dexoycholate | Sigma | D6750 | Component of lysis buffer |
| Sodium dodecyl sulfate | Sigma | L3771 | Component of lysis buffer |
| TrypLE Express, 1x | Life Technologies | 12605-010 | Trypsin, for digesting enteroid/colonoid fragments |
| Vinculin antibody, rabbit monoclonal | Abcam | ab129002 | Use at 1:1000 for immunoblotting |
| Water, tissue culture grade, sterile filtered | Corning | 25-055 | |
| Y-27632 | Tocris | 1254 | RhoA/ROCK inhibitor |
Referências
- In, J. G., et al. Human mini-guts: new insights into intestinal physiology and host-pathogen interactions. Nature Reviews Gastroenterology & Hepatology. 13, 633-642 (2016).
- Hill, D. R., et al. Bacterial colonization stimulates a complex physiological response in the immature human intestinal epithelium. eLife. 6, e29132 (2017).
- Williamson, I. A., et al. A High-Throughput Organoid Microinjection Platform to Study Gastrointestinal Microbiota and Luminal Physiology. Cellular and Molecular Gastroenterology and Hepatology. 6, 301-319 (2018).
- Leslie, J. L., et al. Persistence and Toxin Production by Clostridium difficile within Human Intestinal Organoids Result in Disruption of Epithelial Paracellular Barrier Function. Infection and Immunity. 83, 138 (2015).
- Foulke-Abel, J., et al. Human Enteroids as a Model of Upper Small Intestinal Ion Transport Physiology and Pathophysiology. Gastroenterology. 150, (2016).
- Yin, J., et al. Molecular Basis and Differentiation-Associated Alterations of Anion Secretion in Human Duodenal Enteroid Monolayers. Cellular and Molecular Gastroenterology and Hepatology. 5, 591-609 (2018).
- Tse, C., et al. Enterohemorrhagic E. coli (EHEC)—Secreted Serine Protease EspP Stimulates Electrogenic Ion Transport in Human Colonoid Monolayers. Toxins. 10, 351 (2018).
- Jabaji, Z., et al. Use of collagen gel as an alternative extracellular matrix for the in vitro and in vivo growth of murine small intestinal epithelium. Tissue engineering. Part C, Methods. 19, 961-969 (2013).
- Wang, Y., et al. Self-renewing Monolayer of Primary Colonic or Rectal Epithelial Cells. Cellular and Molecular Gastroenterology and Hepatology. 4, 165-182 (2017).
- Moon, C., VanDussen, K. L., Miyoshi, H., Stappenbeck, T. S. Development of a primary mouse intestinal epithelial cell monolayer culture system to evaluate factors that modulate IgA transcytosis. Mucosal Immunology. 7, 818-828 (2014).
- VanDussen, K. L., et al. Development of an enhanced human gastrointestinal epithelial culture system to facilitate patient-based assays. Gut. 64, 911 (2015).
- Ettayebi, K., et al. Replication of human noroviruses in stem cell–derived human enteroids. Science. 353, 1387 (2016).
- Kozuka, K., et al. Development and Characterization of a Human and Mouse Intestinal Epithelial Cell Monolayer Platform. Stem Cell Reports. 9, 1976-1990 (2017).
- In, J., et al. Enterohemorrhagic Escherichia coli Reduces Mucus and Intermicrovillar Bridges in Human Stem Cell-Derived Colonoids. Cellular and Molecular Gastroenterology and Hepatology. 2, 48-62 (2016).
- Noel, G., et al. A primary human macrophage-enteroid co-culture model to investigate mucosal gut physiology and host-pathogen interactions. Scientific Reports. 7, 45270 (2017).
- Vernetti, L., et al. Functional Coupling of Human Microphysiology Systems: Intestine, Liver, Kidney Proximal Tubule, Blood-Brain Barrier and Skeletal Muscle. Scientific Reports. 7, 42296 (2017).
- Noel, G., et al. Enterotoxigenic Escherichia coli is phagocytosed by macrophages underlying villus-like intestinal epithelial cells: modeling ex vivo innate immune defenses of the human gut. Gut Microbes. 9, 382-389 (2018).
- Sato, T., et al. Long-term Expansion of Epithelial Organoids From Human Colon, Adenoma, Adenocarcinoma, and Barrett's Epithelium. Gastroenterology. 141, 1762-1772 (2011).
- Johansson, M. E. V., Sjövall, H., Hansson, G. C. The gastrointestinal mucus system in health and disease. Nature Reviews Gastroenterology & Hepatology. 10, 352-361 (2013).
- Hews, C. L., et al. The StcE metalloprotease of enterohaemorrhagic Escherichia coli reduces the inner mucus layer and promotes adherence to human colonic epithelium ex vivo. Cellular Microbiology. 19, e12717 (2017).
- Laurie, G. W., Leblond, C. P., Martin, G. R. Localization of type IV collagen, laminin, heparan sulfate proteoglycan, and fibronectin to the basal lamina of basement membranes. The Journal of Cell Biology. 95, 340 (1982).
- Timpl, R. Macromolecular organization of basement membranes. Current Opinion in Cell Biology. 8, 618-624 (1996).
- Hughes, C. S., Postovit, L. M., Lajoie, G. A. Matrigel: A complex protein mixture required for optimal growth of cell culture. Proteomics. 10, 1886-1890 (2010).
- Ishii, S., et al. Normal colonic epithelium adheres to carcinoembryonic antigen and type IV collagen. Gastroenterology. 106, 1242-1250 (1994).
- Moore, R., Madri, J., Carlson, S., Madara, J. L. Collagens facilitate epithelial migration in restitution of native guinea pig intestinal epithelium. Gastroenterology. 102, 119-130 (1992).
- Bein, A., et al. Microfluidic Organ-on-a-Chip Models of Human Intestine. Cellular and Molecular Gastroenterology and Hepatology. 5, 659-668 (2018).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados