Для просмотра этого контента требуется подписка на Jove Войдите в систему или начните бесплатную пробную версию.
Method Article
Мониторинг Равновесие Изменения в структуре РНК "Peroxidative» и «Окислительный" гидроксильных радикалов Footprinting
В этой статье
Резюме
Этот протокол описывает, как количественного Mg (II)-зависимой РНК формирование третичной структуры двумя методами гидроксильных радикалов footprinting.
Аннотация
Молекулы РНК играют важную роль в биологии. В дополнение к передаче генетической информации, РНК может спасовать в уникальной третичной структуры выполняют особую роль в качестве регулятора биологических, связующее или катализатор. Информация о третичном образовании контакта необходимо понимать функции молекул РНК. Гидроксильных радикалов (OH •) представляют собой уникальные зонды структуры нуклеиновых кислот из-за их высокой реакционной способностью и небольшим размером. 1 При использовании в качестве footprinting зонд, гидроксильные радикалы карте растворителя доступной поверхности фосфодиэфирных основа ДНК и РНК 1 2 с тонкая, как одиночных нуклеотидных разрешения. Гидроксильных радикалов footprinting может быть использована для идентификации нуклеотидов в межмолекулярных контактной поверхности, например, в ДНК-белковых 1 и РНК-белковых комплексов. Равновесие 3 и 4 кинетические переходы могут быть определены путем проведения гидроксильных радикалов footprinting в зависимости от solutiна переменную или времени, соответственно. Ключевой особенностью является то, что footprinting ограниченным воздействием зонда (например, "одним хитом кинетика ') приводит к равномерной выборке каждый нуклеотид полимера 5.
В этом видео статье мы используем P4-P6 области Tetrahymena рибозим, чтобы проиллюстрировать РНК подготовки проб и определения Mg (II)-опосредованной складной изотерм. Мы описываем использование известных гидроксильных радикалов протокол, который требует footprinting H 2 O 2 (мы называем это "peroxidative" протокол) и ценную, но не очень широко известно, альтернатива, которая использует естественно растворенного O 2 (мы называем это ' окислительный "протокол). Обзор обработки данных, трансформация и анализ процедур представлена.
протокол
1. Подготовка Footprinting Реагенты
- Подготовка 10х буфера реакции, содержащего 100 мМ натрий какодилатном, 1 мМ ЭДТА и 1 М KCl. Отрегулируйте рН до 7,4. Фильтры буфера с помощью 0,2 мкМ ацетата фильтрующее устройство (Nalgene). Примечание: не пипеткой РНК прямо в 10х буфера.
- Подготовьте смесь титрования реакции для каждой реакции, как это указано в таблице 1. Объем титрования смеси (1x буфера и Mg (II) в нужной концентрации) должно быть 90 мкл, перед добавлением 10 мкл РНК в буфере 1x.
- Подготовка пищеварения РНКазы Т1 буфер, содержащий 6.63M мочевины, 20 мМ цитрат натрия, 1 мМ ЭДТА, 0,25 мкг / мкл тРНК, 0,025% ксилола cyanol, и 0,025% бромфенол синий. Этот буфер может храниться при температуре 4 ° С до 6 месяцев.
- Реакция Фентона требует свежеприготовленных водных растворов 250 мМ Fe (NH 4) 2 (SO 4) 2, 250 мМ ЭДТА и 500 мМ натрий-L-аскорбиновой кислоты. Концентраций отмечалось в главе 10,5 являются оптимальными для равновесия эксперименты в буфер, такие как натрий какодилатном, не убирать мусор радикалов и разнообразный двухвалентных концентрации ионов.
- Реагенты для peroxidative (1.5.1) и окислительной (1.5.2) footprinting реакции.
- Peroxidative реакции Фентона требует свежеприготовленные водные растворы 100 мМ Fe-ЭДТА решение, и 50 ммоль натрия L-аскорбиновая кислота и 1,5% H 2 O 2. Fe-ЭДТА раствор готовят путем смешивания Fe (NH 4) 2 (SO 4) 2 с ЭДТА иметь 1,1 раза избытка ЭДТА над железом.
- Незадолго до начала реакции окислительного расщепления (3.4.2), создайте смесь Фентон реакции путем комбинирования 2 мкл Fe (NH 4) 2 (SO 4) 2 (250 мм), 2,2 мкл ЭДТА (250 мм), 62,5 мкл натрия L-аскорбиновая кислота (500 мм), 22,5 мкл 10х буфера реакции и 135,5 мкл H 2 O добраться до конечной концентрации в смеси footprinting реакции (3.4.2) 0.10 мМ, 0,11 мМ и 6,61 мМ соответственно.
2. РНК Подготовка к Footprinting эксперимент
- РНК могут быть получены стандартными в пробирке транскрипции ДНК шаблонов 6 или, если короче, чем 50 нуклеотидов, быть приобретены (например, при Комплексная ДНК технологий, www.idtdna.com). Следуйте рекомендуемым процедурам лаборатории чистоту поддерживать целостность образца РНК во время footprinting экспериментов 7.
- Дефосфорилирования 8 в пробирке транскрипции РНК необходимо до 32 P маркировки 5 'конца РНК. 10 пмоль РНК конечных помеченных kinasing 9. Решение должно быть облачно после успешного инактивации фосфатазы.
- Purify радиоактивно меченых РНК с использованием денатурирующих гель-электрофореза. РНК выделяли два последующих гель извлечения использованием 0,3 М ацетата натрия (рН 5,5). РНК осаждали путем смешивания 0,5 мл водного раствора с 1 мл этанола и последующееинкубации в течение 1 минуты на сухом льду. Спиновые образца при 4 ° С, 12500 оборотов в минуту в течение 0,5 час. Удалите супернатант и мыть РНК гранул с 70% этанола. Удалить супернатант после центрифугирования и дополнительных сухих гранул в вакууме.
- Растворите 32 P-меченой РНК образцов в 330 мкл 1x Buffer.Pipet 30 мкл раствора РНК в реакционную трубку, осадок с этанолом и сухой РНК в вакууме, чтобы эта труба для генерации ссылки лестницы (шаг 3.5). Денатурировать буфер РНК решение путем нагревания при 95 ° С в течение 2 мин. Охладите образцы до комнатной температуры в течение 15 минут и дать быстрые вращения, чтобы сбить конденсата на крышках из трубок обратно в раствор.
3. Footprinting эксперимент
- Электрофорез гребень определяет максимальное количество точек данных в аналитических геля. Мы используем 30 и расческу. Ссылка лестнице занимает 2 скважины и uncleaved один элемент управления. Подготовка 27 труб реакции. Окончательный Mg (II) concentratiДополнения должны быть равномерно распределены по логарифмической шкале охватывающей порядка вокруг титрования середине.
- Отдельно, инкубировать РНК и Mg (II) растворов при 50 ° С в течение 5 мин. Смешайте 10 мкл раствора РНК с 90 мкл соответствующего количества Mg (II) в 1x буфера для достижения конечного объема 100 мкл. Инкубируйте решения в течение 30 мин при 50 ° C.
- Пусть решения равновесие при 25 ° С в течение 1 ч в то время как складные происходит.
- Peroxidative (3.4.1) или окислительного (3.4.2) гидроксильных радикалов реакции footprinting.
- Начало peroxidative реакции путем размещения капли 2 мкл Fe-ЭДТА (100 мм), 2 мкл натрия L-аскорбиновая кислота (50 мМ) и 2 мкл Н 2 О 2 (1,5%) отделены друг от друга в верхней внутренней реакции пробирку, содержащую раствор РНК и начать footprinting реакцию интенсивного перемешивания. Остановить реакцию через 15 секунд, добавив 300 мкл холодного абсолютного этанола. Включите трубы в 3-5 раз. Осадок, мытье исухих гранул, как в пункте 2.4.
- Начало окислительного гидроксильных радикалов footprintingreaction добавлением 5 мкл свежеприготовленного Фентон реакционной смеси (1,6). Инкубируйте в течение 30 мин при 25 ° C. Добавить 300 мкл холодного абсолютного этанола, чтобы утолить реакции и смешивают поворотом трубы в 3-5 раз. Осадок, вымойте и высушите осадок, как указано в пункте 2.4.
- Создание РНКазы Т1 переварить эталонного образца в соответствии со стандартными процедурами 10, используя раствор, приготовленный в шаге 1.3.
- Растворите гранулы РНК в 8 мкл геля загрузки красителя II (Ambion) и подтвердите РНК ресуспендировали с помощью счетчика Гейгера.
- Подготовка денатурации, 8% полиакриламидном геле последовательности в соответствии со стандартными протоколами. 11 образцов нагрузки в том числе две ссылки и контроля. Отдельные фрагменты РНК при 60 Вт - 75 Вт в течение 2,5 час. Expose высушенный гель для экрана хранения фосфора в одночасье. Сканирование люминофор экрана с изображениями системы беспленочной авторадиографии, например,. Буря 865 (GE Healthcare) или Typhoon (GE Healthcare). Передача файлов гель изображение на компьютер.
4. Анализ данных
- Гидроксильных радикалов реакционной способности каждого нуклеотида определяется количественно интенсивность каждой полосы по аналитической гель. Скачайте, установите и откройте SAFA, простой в использовании открытого программного обеспечения для установки одной группы и количественного определения. 12, 13, следуйте инструкциям руководства пользователя. Короче говоря, нагрузка последовательности РНК. Текстовый файл затем гель изображение как. Гель файл. Настройка интенсивности полос, определять полосы движения, выбирать якорь переулок, а также выполнять гель выравнивания. Назначение полос нуклеотидов происходит по отношению к РНКазы лестнице пищеварения T1. Количественная группа интегралы и использовать нормализацию / colorplot функция для нормализации и назначить потенциально инвариантных остатков. Сохранить в файл как. Текстовый файл.
- SAFA выходы таблицы, содержащей столбцы представляют полос на геле и строки, представляющие винтегрированная группа плотности отдельных полос, соответствующих фрагментов РНК. Во-первых, определить защиту сайтов, которые показывают заметные изменения в растворителе доступности, сравнивая защиту профиля происходит от не Mg (II) образца с профилем конечная точка Mg (II) концентрации (50 мМ в нашем примере). Чем меньше значение, тем более защищены нуклеотид против атаки гидроксильные радикалы, и наоборот.
- Создать складной изотерм от нормированных интегралов группы отдельных или группы нуклеотидов по сравнению с Mg (II) концентрации. Изотерм индивидуально масштабируется до дробной насыщения р = L + (U - L) •
 где F обозначает интегральная плотность полосы (ы) анализируются, L и U представляет нижняя и верхняя границы для перехода и
где F обозначает интегральная плотность полосы (ы) анализируются, L и U представляет нижняя и верхняя границы для перехода и 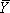 является дробным насыщения. 14 данные пригодны использованием нелинейных лвосток квадратных программа анализа (Мы используем либо происхождения (OriginLab) или GraphPad (GraphPad Software, Inc.)) для уравнения Хилла (1),
является дробным насыщения. 14 данные пригодны использованием нелинейных лвосток квадратных программа анализа (Мы используем либо происхождения (OriginLab) или GraphPad (GraphPad Software, Inc.)) для уравнения Хилла (1), 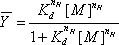
где К г равновесная константа диссоциации, [M] соответствует концентрации переменной, которая является посредником складной реакции, Mg (II) в этом примере, и п Н коэффициент Хилла. Эта процедура масштабах переход на дробное насыщения, определяет его середину и обеспечивает феноменологической испытание ли переход сигмоидального. Изотермы основе секвенирования гелей (рис. 3А), которые были проанализированы SAFA (рис. 3В) и установите, как описано выше, показаны на рисунке 3C. Дробных изотерм насыщения показано на рисунке 3 были получены за счет масштабирования в таблице значения, сгенерированные SAFA против наилучшим образом соответствует верхним и нижним пределами (U и L) с помощью уравнения фракция = значение / (U - L) - L / (U - L).
5. Представитель Результаты:
На рисунке 3 показан представителю результаты P4-P6 РНК • экспериментов ОН footprinting. Гель изображение (слева) показывает, что) фоне расщепления минимальна (правая полоса), б) одного нуклеотида назначение стало возможным благодаря четко определены полосы в полосу T1 (T1 РНКаза расщепляет на каждом G) и в) гидроксильных радикалов индуцированной фрагментации РНК значительно выше фона. Переход от низких до высоких Mg (II) является филиалом с уменьшением плотности интегрированной группы индивидуальных и групп полос указывает образование РНК третичной структуры. Одно-или групп смежных нуклеотидных которого расщепления изменения одновременно, называются "защиты". • ОН защиты характеристика Mg (II) опосредовано складывания P4-P6 РНК тесно связаны с растворителем труднодоступных районов молекулы наблюдаются в его кристаллической структуры. 15
Сложенный молекулы РНК с имеют очень разной степени (то есть, насколько близко к фоновым снижение группа плотности) высшего защиты контактов, которые не были связаны с ясным структурным или динамическим явлением. Таким образом, некоторые молекулы РНК покажет четко определены структурные переходы, как показано на рис 3, а некоторые не будут. Интенсивности полос количественно и нормированный SAFA 12 анализа (рис. 3В). Выход "тепловой" сюжет визуализации относительная степень защиты от • ОН. Цветовой переход описывает доступность меняется при Mg (II) дополнение. Белый с красным или синим цветом показаны более доступной и более защищены нуклеотидов, соответственно. Каждая степень затенения связан с числовым значением, которые могут быть нанесены в качестве защиты кривой (рис. 3C) и проанализирована модель привязки, такие как уравнения Хилла (1). Равновесии константа диссоциации связан с Защита 153-155 примерно вдвое больше, чем соответствующее значение Защита 163-164.
ontent ">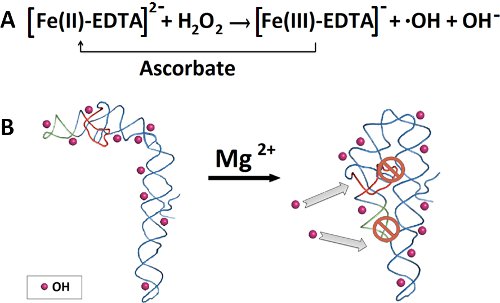
Рисунок 1. Гидроксильных радикалов и P4-P6 РНК складывания. А) Fe (II) катализирует образование гидроксильных радикалов из перекиси водорода. Аскорбат снижает Fe (III) обратно к черным железом. Б) В отсутствие Mg (II), не P4-P6 третичная структура формируется, позволяя гидроксильные радикалы проникать и прилепится всех доступных позиций позвоночника. Добавление Mg (II) инициирует складывания P4-P6, позволяя только растворителем доступны позвоночник будет расщепляться гидроксильных радикалов.
Рисунок 2. Наброски на гидроксильных радикалов эксперимент footprinting. ) Dephophorylation и 32 Р 5'-конце маркировки РНК. Б) Очистка 32 P-меченой РНК на денатурирующих полиакриламидном геле. C) Иссечение группы РНК, РНК последующего извлечения и осаждением этанолом. D) и Prefoldingсворачивания РНК. Е) Добавление свежеприготовленные смеси реакции Фентона для генерации гидроксильных радикалов. F) фрагмент РНК разделения денатурирующих электрофореза в полиакриламидном геле. G) Количественный анализ фрагментов РНК программного обеспечения SAFA.
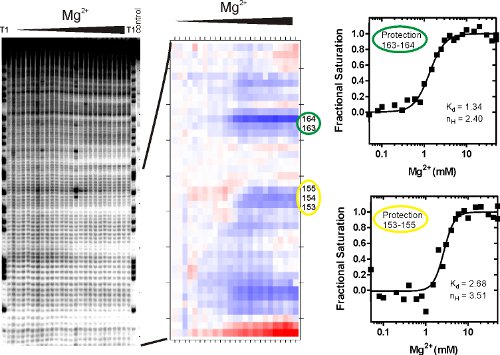
Рисунок 3. Анализ фрагментов РНК после peroxidative реакции footprinting Фентона. (А) РНК подвергаются гидроксильных радикалов и расщепления продуктов были решены использованием денатурирующих электрофореза в полиакриламидном геле (PAGE). На снимке peroxidative продуктов расщепления с ростом концентрации Mg (II). Ведения и контроля полосы помечены. Этот файл является входным для программы SAFA которые quantitates объем каждой группы. (B) "тепловой" сюжет порожденных SAFA. (C) таблицы значений плотности, касающихся интегрированной группы до нуклеотида номер выхода из SAFA анализируется модель привязки, таких как Hill equatiна (1). Регионы защиты были выбраны в соответствии с увеличением в группе интегральной плотности в зависимости от Mg (II) концентрации. К г является Mg (II), концентрация, при которой половина РНК складывается на месте проводится мониторинг. Коэффициент Хилла, п H, является мерой наклона кривой, которая дает информацию об обязательных кооперативности и обеспечивает более низкую оценку числа ионов магния участвует в складывания данного сайта.
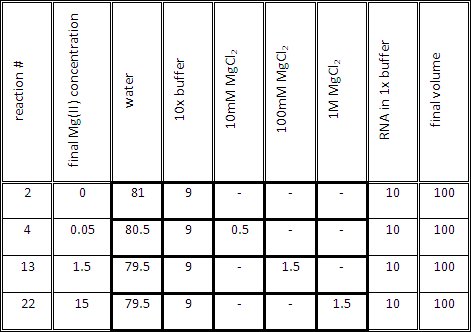
Таблица 1. Представителю объемы для создания образцов, содержащих РНК различных Mg (II) концентрации.
Обсуждение
Гидроксильных радикалов footprinting является ценным инструментом для оценки растворителя доступной площади поверхности нуклеиновых кислот. Качественный и количественный формирование третичной структуры 14 можно проследить как функцию параметров, таких как ион типа и концентрации,...
Раскрытие информации
Нет конфликта интересов объявлены.
Благодарности
Эта работа была поддержана грантами от Национального института здоровья RO1-GM085130 и Национальный научный фонд MCB0929394. Мы благодарим доктора Марион Шмидт за гостеприимство и за предоставленную нам возможность фильма в своей лаборатории.
Материалы
| Name | Company | Catalog Number | Comments |
| Name | Company | Cat# | |
| Sodium Cacodylate(Caution! Toxic) | Sigma-Aldrich | C4945-25g | |
| EDTA (0.5 M) | Ambion | AM9260G | |
| DEPC treated water | Ambion | AM9915G | |
| Sodium Acetate (3 M) | Ambion | AM9740 | |
| MgCl2 (1 M) | Ambion | AM9530G | |
| Urea | Ambion | AM9902 | |
| Sodium Citrate | Sigma-Aldrich | W302600 | |
| tRNA | Sigma-Aldrich | R-7876 | |
| Sodium-L-ascorbate | Sigma-Aldrich | A7631-25g | |
| Fe(NH4)2(SO4)2 . 6 H2O | Sigma-Aldrich | F1543-500g | |
| RNase T1 | Fermentas | EN0541 | |
| Hydrogen Peroxide (30%) | Sigma-Aldrich | 349887 |
Ссылки
- Tullius, T. D., Dombroski, B. A. Hydroxyl radical "footprinting": high-resolution information about DNA-protein contacts and application to lambda repressor and Cro protein. Proc. Natl. Acad. Sci. U.S.A. 83, 5469-5473 (1986).
- Celander, D. W., Cech, T. R. Iron(II)-ethylenediaminetetraacetic acid catalyzed cleavage of RNA and DNA oligonucleotides: similar reactivity toward single- and double-stranded forms. Biochemistry. 29, 1355-1361 (1990).
- Celander, D. W., Cech, T. R. Visualizing the higher order folding of a catalytic RNA molecule. Science. 251, 401-407 (1991).
- Sclavi, B., Sullivan, M., Chance, M. R., Brenowitz, M., Woodson, S. A. RNA folding at millisecond intervals by synchrotron hydroxyl radical footprinting. Science. 279, 1940-1943 (1998).
- Tullius, T. D., Dombroski, B. A., Churchill, M., Kam, L. Hydroxyl radical footprinting: a high-resolution method for mapping protein-DNA contacts. Methods. Enzym. 155, 537-558 (1987).
- Milligan, J. F., Groebe, D. R., Witherell, G. W., Uhlenbeck, O. C. Oligoribonucleotide synthesis using T7 RNA polymerase and synthetic DNA templates. Nucleic. Acids. Res. 15, 8783-8798 (1987).
- Schlatterer, J. C., Brenowitz, M. Complementing global measures of RNA folding with local reports of backbone solvent accessibility by time resolved hydroxyl radical footprinting. Methods. 49, 142-147 (2009).
- Sambrook, J., Russell, D. W. . Molecular Cloning: A Laboratory Manual. , (2001).
- Zaug, A. J., Grosshans, C. A., Cech, T. R. Sequence-specific endoribonuclease activity of the Tetrahymena ribozyme: enhanced cleavage of certain oligonucleotide substrates that form mismatched ribozyme-substrate complexes. Biochemistry. 27, 8924-8931 (1988).
- Knapp, G. Enzymatic approaches to probing of RNA secondary and tertiary structure. Methods. Enzym. 180, 192-212 (1989).
- Slatko, B. E., Albright, L. M. Denaturing gel electrophoresis for sequencing. Curr. Protoc. Mol. Biol. Chapter 7, Unit 7.6-Unit 7.6 (2001).
- Das, R., Laederach, A., Pearlman, S. M., Herschlag, D., Altman, R. B. SAFA: semi-automated footprinting analysis software for high-throughput quantification of nucleic acid footprinting experiments. RNA. 11, 344-354 (2005).
- Simmons, K., Martin, J. S., Shcherbakova, I., Laederach, A. Rapid quantification and analysis of kinetic *OH radical footprinting data using SAFA. Methods. Enzym. 468, 47-66 (2009).
- Uchida, T., He, Q., Ralston, C. Y., Brenowitz, M., Chance, M. R. Linkage of monovalent and divalent ion binding in the folding of the P4-P6 domain of the Tetrahymena ribozyme. Biochemistry. 41, 5799-5806 (2002).
- Cate, J. H., Gooding, A. R., Podell, E., Zhou, K., Golden, B. L., Kundrot, C. E., Cech, T. R., Doudna, J. A. Crystal structure of a group I ribozyme domain: principles of RNA packing. Science. 273, 1678-1685 (1996).
- Petri, V., Brenowitz, M. Quantitative nucleic acids footprinting: thermodynamic and kinetic approaches. Curr. Opin. Biotechnol. 8, 36-44 (1997).
- Takamoto, K., Das, R., He, Q., Doniach, S., Brenowitz, M., Herschlag, D., Chance, M. R. Principles of RNA compaction: insights from the equilibrium folding pathway of the P4-P6 RNA domain in monovalent cations. J. Mol. Biol. 343, 1195-1206 (2004).
- Brenowitz, M., Senear, D. F., Shea, M. A., Ackers, G. K. Quantitative DNase footprint titration: a method for studying protein-DNA interactions. Methods. Enzym. 130, 132-181 (1986).
- Shcherbakova, I., Mitra, S. Hydroxyl-radical footprinting to probe equilibrium changes in RNA tertiary structure. Methods. Enzym. 468, 31-46 (2009).
- Howlett, G. J., Minton, A. P., Rivas, G. Analytical ultracentrifugation for the study of protein association and assembly. Curr. Opinion. Chem. Biol. 10, 430-436 (2006).
- McGinnis, J. L., Duncan, C. D., Weeks, K. M. High-throughput SHAPE and hydroxyl radical analysis of RNA structure and ribonucleoprotein assembly. Methods. Enzym. 468, 67-89 (2009).
- Jonikas, M. A., Radmer, R. J., Laederach, A., Das, R., Pearlman, S., Herschlag, D., Altman, R. B. Coarse-grained modeling of large RNA molecules with knowledge-based potentials and structural filters. RNA. 15, 189-199 (2009).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеСмотреть дополнительные статьи
This article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены