Method Article
Generazione rapida ed efficace delle ricombinante cellule staminali umane pluripotenti da ricombinasi mediata scambio Cassette in
* Questi autori hanno contribuito in egual misura
In questo articolo
Riepilogo
Qui riportiamo un metodo di modifica genetica rapida ed efficiente in base RMCE nel locus AAVS1 di umani cellule staminali pluripotenti (hPSCs) che migliora i sistemi precedentemente descritti. Usando questa tecnica, linee isogeniche possono essere rapidamente e affidabile generati per adeguati studi comparativi, facilitando la ricerca transgenesi mediata con hPSCs.
Abstract
Anche con la rivoluzione delle tecnologie gene targeting guidati da CRISPR-Cas9, la modificazione genetica delle cellule staminali umane pluripotenti (hPSCs) è ancora in tempo. Studi comparativi che utilizzano linee ricombinanti con transgeni integrati in loci porto sicuro potrebbero beneficiare di approcci che utilizzano ricombinasi mirati site-specific, come Cre o FLPE, che sono più rapidi e meno incline agli effetti esce a lato. Sono state descritte Tali metodi, pur non significativamente gene outperform targeting in molti aspetti. Utilizzando nucleasi zinc-finger, abbiamo precedentemente creato una linea cellulare maestro nel locus AAVS1 di hPSCs che contiene una GFP-Hygromycin-tk esprimere cassetta, affiancato da sequenze FRT eterotipica. Qui, si descrivono le procedure per eseguire FLPE scambio cassetta recombinase-mediata (RMCE) utilizzando questa linea. Il Master Clinea ell è trasfettate con un vettore RMCE donatore, che contiene un promoterless resistenza puromicina, e con FLPE ricombinasi. Applicazione di entrambi programma di selezione positiva (puromicina) e negativo (FIAU) porta alla selezione di RMCE senza integrazioni casuali. RMCE genera pienamente caratterizzato pluripotenti linee transgeniche policlonali in 15 d con un'efficienza del 100%. Nonostante le limitazioni del locus AAVS1 recentemente descritto, la facilità del sistema apre la strada per HPSC transgenesi nelle impostazioni isogenici, è necessario per gli studi comparativi, e consente schermi genetici semi-high-throughput per guadagno / perdita di analisi funzione che sarebbe altrimenti essere molto tempo.
Introduzione
Mirata ricombinazione del genoma ha facilitato rapidi progressi in molti settori della ricerca. Nei topi, la sinergia tra la modifica del genoma e ricerca sulle cellule staminali ha permesso una maggiore comprensione del complesso meccanismo della funzione del gene e la regolazione genica. Tale progresso è previsto in cellule umane pluripotenti staminali (hPSCs) e, anche se per molti anni da quando il primo isolamento di cellule staminali embrionali umane (hESC), e successivamente indotto umano cellule staminali pluripotenti (hiPSCs), la modifica genetica ha costituito un ostacolo tecnico . I recenti progressi nel campo dell'ingegneria del genoma utilizzando nucleasi-zinco mirati Finger nucleasi (ZFNs), trascrizione attivatore come nucleasi effettrici (Talen), o cluster regolarmente intercalare breve ripete palindromiche / CRISPR-proteina associata 9 (CRISPR / Cas9) ci hanno permesso di superare queste difficoltà, facendo mirati ricombinazione un processo efficiente 1-3.
loci specifica nel mOuse genoma che permettono l'espressione del transgene stabile, affidabile, e onnipresente in assenza di effetti negativi, come Rosa26, Hprt1 o COL1A1, sono diventati strumenti essenziali nella realizzazione di studi genetici. loci di porto sicuro consentono l'analisi confrontabili tra le diverse linee di topi in contesti isogenici. Ciò non può essere ottenuto utilizzando metodi di integrazione casuale standard, che sono associati con limitazioni ben note come mutagenesi inserzionale, effetti dose-dipendente, o l'espressione del transgene variegata. Gene editing facilità e flessibilità nella loci porto sicuro è aumentata attraverso l'uso di ricombinasi mirati site-specific come Cre o flippase (FLPE), che riconoscono specificamente sequenze bersaglio (loxP o FRT, rispettivamente) e catalizzano efficiente ricombinazione tra obiettivi identici. Grazie a queste caratteristiche, la modifica genetica recombinase mediata utilizzando loxP o FRT sequenze in preintegrate loci Safe Harbor è uno strumento comunemente usato nel topo transgenesi. Oltre all'inserimento cassetto o escissione mediata tra sequenze bersaglio identici, l'uso di siti loxP o FRT incompatibili consente uno scambio cassette ricombinasi-mediata (RMCE) 4.
Nell'uomo, i tentativi di identificare loci approdo sicuro sono state effettuate. Il HPRT orthologous del mouse, nonostante la sua associazione con la sindrome della perdita-di-funzione di Lesch-Nyhan, e ROSA26 sono stato preso di mira in hPSCs. HPRT è stato segnalato per mettere a tacere rapidamente l'espressione del transgene nel ESC e, come ROSA26, la sua capacità di sostenere l'espressione del transgene nel terminale cellule differenziate non è stato studiato 5-7. Perché un null mutazione omozigote del gene CCR5 sembra essere ben tollerato nell'uomo, il suo valore come porto sicuro è stata valutata. CCR5 stato preso di mira e segnalato per sostenere l'espressione del transgene stabile in diverse linee cellulari umane, tra cui CES 8,9. Però,quest'ultimo non è stato dimostrato a livello clonale durante la coltura a lungo termine, e l'espressione ubiquitaria transgene non è stata dimostrata in progenie differenziata dei tre strati germinali. AAVS1, il sito di integrazione naturale del adeno associati tipo Virus 2 (AAV), è stato testato in hESC così, a causa di resistenza riferito a transgene tacere 10. Successivamente, molti gruppi usati locus AAVS1 e descritti espressione del transgene stabile in hPSCs indifferenziate, nonché nella loro progenie differenziata di tutti tre strati germinali, sia in vitro che in vivo 2,8,11,12. I recenti risultati comunque nuance questi risultati, come il locus AAVS1 è stato trovato a esercitare l'inibizione transgene variabile in vitro in hESC indifferenziato e in epatociti progenie 13.
Ulteriori studi di screening che utilizzano approcci di integrazione casuali e metodi per determinare l'integrazione copia singola mirato per trovare genositi di integrazione mic resistenti al transgene silenziamento durante l'espansione e la differenziazione hPSCs 14,15. Nel complesso, fino ad ora, nessun sito genomico è stato completamente convalidato come un porto sicuro in hPSCs e la loro progenie; identificazione di un sito appropriato per ubiquitous espressione del transgene stabile, non solo in hPSCs ma anche nelle loro progenie differenziate in vitro e in vivo, rimane da risolvere. Tra tutti i loci studiati e nonostante i suoi limiti, AAVS1 rimane il più caratterizzata e più utilizzato nel campo della ricerca sulle cellule staminali.
RMCE è stato eseguito con successo in hPSCs in alcuni di questi loci 6,7,14,16, utilizzando per lo più Cre ricombinasi, anche se ci sono indicazioni che FLPE è più efficiente di Cre 17. In tutti questi casi, una cassetta resistenza ai farmaci positivo è stato utilizzato per la selezione delle colonie ricombinanti. Anche se di successo, queste procedure non costituiscono un progresso tecnico sopra gene- seriele procedure di modifica utilizzando ZFNs, Talens o CRISPR / Cas9, come una singola procedura di selezione antibiotica non esclude integrazioni casuali e richiede lo screening colonia per identificare cloni correttamente mirati.
In questa procedura, si descrivono metodi per eseguire RMCE in hPSCs nel locus AAVS1 utilizzando una combinazione di positivo (all'interno della cassetta in entrata) e negativo (all'interno della cassetta preintegrate) selezioni che consentono la generazione di linee transgeniche policlonali di ± 15 giorni con 100% di efficienza e privo di eventi di integrazione casuali. Pertanto, questo metodo rappresenta un progresso al di là delle tecnologie RMCE attualmente descritte in hPSCs.
Protocollo
1. Preparazione del FRT contenenti HPSC master Cell Line per Transfection
- Cultura linee cellulari maestro hESC / IPSC sotto procedure standard o disattivare inattivati fibroblasti embrionali di topo (Imef) utilizzando hESC o medio IPSC senza alimentatore, rispettivamente (Tabella 1). Preparare 2 (su alimentatori) o 1 pozzetti (senza alimentatore) di hPSCs in un 6-pozzetti per ogni condizione sperimentale.
- Osservare le culture, e quando le cellule raggiungono 60 - 70% di confluenza, piastra resistente ai farmaci IMEF (iDR4). Brevemente, rivestire i pozzetti necessarie di un 12-pozzetti con 0,5 mL di soluzione di gelatina 0,1% e incubare per 5 minuti a temperatura ambiente. Piastra 125.000 iDR4s per pozzetto e incubare O / N con il mezzo Imef (Tabella 1).
2. HPSC Trasfezione da Nucleofection
- Il giorno seguente, pre-incubare hESC / culture IPSC con mezzi freschi, tra cui 10 micron proteina chinasi Rho-associata (ROCK) inibitore (Y-27632), per 1 ora a 37 ° C. Next, prendere soluzione trasfezione hESC dal frigorifero di pre-caldo a temperatura ambiente.
- Preparare 1 ml di nucleofection placcatura media (Tabella 1) per ogni condizione di nucleofection. Prendere medio IMEF fuori i pozzi, lavare con 1 ml di PBS temperatura ambiente, e aggiungere 500 ml di nucleofection placcatura media. Trasferire gli altri 500 ml di sterili tubi da 1,5 ml e conservarli a 37 ° C fino a quando la placcatura. NOTA: Un tipico esperimento RMCE contiene tre condizioni diverse con rapporti molecolari di RMCE donatore: vettori pFLPe di 2: 0, 2: 1, e 0: 1.
- Staccare hESC / IPSC in una sospensione singola cella.
- Togliere i media, lavarlo con 2 ml di RT PBS, e aggiungere 1 ml di tripsina 0,05% o accutase su alimentatore o senza alimentatore HPSC culture, rispettivamente. Incubare per 7 (tripsina) o e 2-5 (accutase) min a 37 ° C. Per il trattamento Accutase - celle devono rimanere sciolto, ma in allegato.
- Rimuovere con cautela la piastra dall'incubatore e senzallowing le cellule a staccare, rimuovere l'agente di dissociazione per aspirazione. Aggiungere 1 ml di hESC supporti e dissociare le cellule sciolte da vampate di calore delicatamente i media sulla superficie della piastra, evitando la formazione di schiuma. Assicurarsi che le cellule sono in una sospensione singola cella.
- Raccogliere le cellule in un 15 o 50 provetta pulita e sterile mL. Lavare i pozzetti con 1 ml di mezzi hESC e raccogliere le cellule nello stesso 15 o tubo da 50 ml. Prendete una aliquota di 50 microlitri per il conteggio (durante il passaggio successivo) utilizzando un dispositivo di conteggio delle cellule. Annotare il volume della raccolta e calcolare il numero di cellule totale. Centrifugare le cellule a 300 xg per 5 minuti a temperatura ambiente.
- Risospendere il pellet di cellule ad una sospensione di 10 6 cellule / ml con PBS utilizzando una pipetta da 5 mL sierologica e delicato pipettaggio. Evitare la formazione di schiuma. Trasferire 2 ml per condizione sperimentale (circa 2 x 10 6 cellule) per pulire, sterili 15 mL.
- Mentre centrifugazione, prepare il RMCE donor- plasmidi pFLPe mescola in un volume massimo di 10 ml.
- Trasferire 2,5 mg di pFLPe (6.5 Kb) ad una sterile 1,5 mL provetta pulita. Aggiungere un rapporto 2: 1 molecolare del vettore RMCE donatore 13 al vettore pFLPe. Ad esempio, circa 10 mg di una Kb RMCE vettore 12 donatori.
- Procedere con nucleofection, evitando la formazione di schiuma in tutti i passaggi.
NOTA: Alcuni protocolli di trasfezione HPSC applicano una fase di esaurimento Imef ulteriore prima del punto 2.4. Tuttavia, in pratica, Imef è una frazione cellulare minore singola sospensione che non alteri significativamente l'efficienza di trasfezione, né si crea contaminanti cellule ricombinanti poiché sono non proliferativa. Inoltre, è essenziale procedere rapidamente durante il processo di transfezione per aumentare la vitalità. Per queste ragioni, l'esaurimento Imef non viene applicata.- Rimuovere la piastra iDR4 ei tubi da 1,5 ml con il mezzo placcaturadal termostato. Un tubo alla volta, pipettare largo della PBS accuratamente senza disturbare il pellet. Rimuovere il più possibile. Risospendere il pellet con 100 ml di soluzione di trasfezione pipettando delicatamente. Trasferire la sospensione cellulare alla provetta ml 1,5 contenente le miscele di plasmidi e mescolare delicatamente.
- Trasferire la miscela di cellule-DNA alla provetta transfector, facendo attenzione a non introdurre bolle. Introdurre la provetta nel dispositivo Nucleofector e applicare il programma A13 (cellule coltivate su alimentatori) o F16 (-alimentatore gratuito).
NOTA: Altri metodi di trasfezione o Nucleofector dispositivi possono richiedere l'ottimizzazione di questi programmi o condizioni di trasfezione. - Riprendere la cuvetta e, utilizzando le pipette monouso disponibili nel kit nucleofection, raccogliere il contenuto applicando 0,5 ml del mezzo di placcatura ed aspirando tutto il volume. Condurre questa fase rapidamente, ma con dolcezza e in un unico movimento.
- Piastra goccia a goccia in 12-wPiastra ell contenente la iDR4 e 500 ml di placcatura media. Ripetere i punti 2.6.1 - 2.6.4 per il prossimo condizione sperimentale.
- Posizionare il piatto di coltura su uno scaffale in incubatrice e spostarlo lentamente avanti e indietro e un lato all'altro per distribuire uniformemente la sospensione cellulare. Incubare per 24 ore prima del successivo cambio media per consentire alle cellule di recuperare in presenza di 10 inibitore ROCK micron (Y-27632).
3. Selezione positivi e negativi di cellule in fase RMCE
- mezzi cambiamento culturale al giorno (1 ml / pozzetto), e 2 - 3 d dopo la trasfezione, iniziano la selezione con 100 ng / ml di puromicina.
NOTA: concentrazioni ottimali di reagenti di selezione positive e negative devono essere determinati sperimentalmente per ciascuna linea cellulare maestro HPSC appena generato. Effettuare una curva di uccidere 13, utilizzando la linea cellulare master per determinare la dose bassa (concentrazione alla quale la tossicità visiva minima è evidente dopo 7d di selezione, ma la cultura non è invaso), dose ottimale (la concentrazione più bassa alla quale tutte le cellule sono morti dopo 7 d di selezione), e ad alte dosi (concentrazione che ha causato la tossicità evidente, uccidendo tutte le cellule dopo 2 - 3 d) sia per puromicina e Fialuridine (FIAU).- Aspirare il mezzo di hESC. Lavare con 1 ml di PBS. Aggiungere mezzo hESC con 100 ng / ml di puromicina.
- Osservare la crescita cellulare in modo che la morte e la crescita sono bilanciati. Cambiare media con puromicina quotidiano, seguendo passo 3.1.1. Quando il tasso di crescita supera morte cellulare, aumentare la concentrazione puromicina in blocchi di 25 - 50 ng / mL fino ad un massimo di 250 ng / mL. Continuare selezione puromicina per 5 - 7 d.
- 3 - 4 d dopo aver iniziato la selezione puromicina, intorno al giorno 6 dopo la trasfezione, iniziare la selezione con 0,5 micron FIAU. Modificare i media giornaliera e mantenere FIAU per non più di 7 giorni consecutivi.
4. Ampliamento e caratterizzazione di linee RMCE
- Dopo il completamento della selezione, intorno D14 - 15, colonie RMCE resistenti sono presenti e costituiscono la nuova linea RMCE. Split alla rinfusa in un rapporto 1: 2 seguendo le procedure standard (passaggio 1, P1).
- Piastra 2 pozzetti di una 12-pozzetti (o disattivare alimentatori, a seconda delle condizioni di coltura regolari della linea cellulare master originale). Utilizzare un pozzetto per un'ulteriore espansione, stoccaggio o set-up sperimentale (senza ulteriore descrizione di queste procedure è fatto in questo protocollo), e l'altra per la caratterizzazione.
- Quando le cellule da P1 sono pronte per dividere, dissociare il bene per la caratterizzazione in una sospensione di cellule singole, come descritto in 2.4 e raccogliere le cellule in una provetta da 15 ml. NOTA: utilizzare le cellule dal HPSC WT (senza modificazione genetica) e linea di cellule master come controlli negativi e positivi, rispettivamente, per la caratterizzazione.
- Centrifugare a 300 xg per 5 minuti a temperatura ambiente. Risospendere in 1 ml di PBS e diviso il campione in due per citometria a flussoe l'analisi del DNA 13.
- Citometria a flusso di analisi 13 (300 ml):
- Aggiungere siero fetale di vitello alle cellule in PBS fino ad una concentrazione finale del 5%. Trasferire le cellule in una provetta FACS pulita con un colino cella e tenerli in ghiaccio.
NOTA: La vitalità cellulare è alto in queste condizioni, quindi è possibile, ma non necessario, utilizzare un colorante fluorescente per le cellule non vitali (ad esempio, 7-AAD o PI). - Procedere immediatamente ad analisi in un citofluorimetro analizzatore cellulare e registrare 20.000 - 30.000 eventi. Per l'analisi, impostare le porte di cui sopra GFP o TDT sfondo fluorescenza utilizzando il campione di controllo negativo WT.
- Aggiungere siero fetale di vitello alle cellule in PBS fino ad una concentrazione finale del 5%. Trasferire le cellule in una provetta FACS pulita con un colino cella e tenerli in ghiaccio.
- Analisi del DNA 13 (700 ml):
- Trasferire le cellule in una provetta pulita da 1,5 ml. Spin down a 300 xg per 5 minuti a temperatura ambiente e aspirare il surnatante. Procedere alla estrazione del DNA utilizzando un kit commerciale e seguire le istruzioni del produttore. Conservare il pellet secco a -20 ° C per latanalisi ER.
- Misurare la concentrazione di DNA.
- Preparare reazioni PCR standard secondo le condizioni della figura 3 e nella tabella 2 13 campioni Run di PCR su un gel di agarosio al 2% contenente 1x macchia gel DNA a 150 V per 15 -. 25 min. Analizzare la corsa in un analizzatore di gel.
Risultati
Utilizzando AAVS1 ZNFs specifico d', linee cellulari maestro pienamente caratterizzato hESC / IPSC contenenti eterotipica sequenze bersaglio FRT sono stati generati e descritti 13. Le linee cellulari di master FRT contenenti mantenuti pluripotency e l'integrità del genoma dopo il trattamento ZFN e stabilmente espressi GFP in vitro e in vivo. RMCE è eseguita da cotrasfezione delle linee cellulari matrimoniale con FLPE ricombinasi e vettori donatori RMCE (Figura 1A). Vettori RMCE contengono identiche sequenze FRT a quelli della linea cellulare maestro AAVS1 che fiancheggiano il transgene Figura 1B controlla RMCE utilizzando il esprimono costitutivamente TDT pZ:.. F3-P CAGGS tdTPH-F dopo la transfezione, HPSC formare piccoli gruppi di cellule uniformemente distribuiti su lo strato iDR4 MEF, e transfettate HPSC esprimono GFP sia così come transitoria TDT (Figura 1B, D 2). Le cellule possono recuperare per 2 - 3d post-trasfezione prima di iniziare la selezione. selezione positiva viene prima iniziato a utilizzare una bassa dose di puromicina al fine di favorire l'inserimento dei donatori RMCE. Puromicina è applicato delicatamente all'inizio, perché la morte delle cellule iniziale massiccia potrebbe portare a singole cellule in fase di ricombinazione, che li rende in grado di sopravvivere al processo. Nei giorni successivi, concentrazione puromicina deve essere sollevato gradualmente fino alla dose ottimale per consentire alle cellule ricombinanti di crescere in piccole colonie mentre le cellule muoiono nonrecombined gradualmente.
3 - 4 d dopo l'inizio della selezione positiva (circa D 6 dopo la trasfezione), selezione negativa è iniziata per selezionare solo per eventi di ricombinazione FRT-mediate. RMCE provoca la perdita della timidina chinasi (TK) gene suicida e quindi la sensibilità Fialuridine (FIAU). Selezione negativa applicata a questo punto, in cui piccoli gruppi di di 2 - 4 GFP - / + TDT (RMCE) cellule sonoin grado di resistere a concentrazioni ottimali di FIAU (Figura 1B, D 6), impedisce l'integrazione casuale, perché in tali eventi, il gene Tk nel locus AAVS1 rimane inalterato. Il verificarsi di simultanea FRT-mediata e l'integrazione casuale è altamente improbabile, come ha dimostrato l'assenza di eventi di integrazione casuali più tardi, durante la caratterizzazione (Figura 2B).
Il RMCE GFP mista - colonie / TDT + continuano a crescere, diventando più omogenea, in modo che entro D 9 - 10 dopo la trasfezione, alcune colonie RMCE misti non pienamente selezionati possono essere trovati (Figura 1B). GFP - colonie / TDT + RMCE sono presenti solo quando entrambi RMCE donatore e FLPE (2: 1) vengono utilizzati, mentre RMCE non si verifica in assenza di FLPE (2: 0). Selezione completa con FIAU fino al giorno 13 - 15 post-trasfezione dà luogo ad una GFP omogenea - / + TDT culture. RMCE produce una media di 12,8 ± 6,8 (n = 6) colonie Puro R / R FIAU 15 giorni 13. Citometria a flusso caratterizzazione della linea RMCE appena generato (colonie RMCE Puro R / FIAU R combinate in una popolazione di cellule nonclonal) conferma che il 100% delle cellule presentano il RMCE GFP - / + TDT fenotipo (Figura 2A). Quando si utilizzano i donatori RMCE senza espressione costitutiva di un reporter fluorescente, la conseguente linea di Puro R / R FIAU RMCE HPSC è omogeneo GFP -.
PCR caratterizzazione della linea RMCE nonclonal dimostra lo scambio cassetto pieno (tracce della cassetta della linea cellulare master può essere rilevato) e che il programma di selezione utilizzato genera linee privo di integrazione casuale (sia del donatore RMCE e FLPE esprimenti vector) (Figura 2B). Questi risultati sono stati ulteriormente comprovati da southern macchia e, in aggiunta, abbiamo dimostrato che le linee RMCE rimangono pluripotenti 13. Ciò rende più necessario effettuare sia la valutazione genoma a livello di integrazione casuale o la prova pluripotenza durante gli esperimenti RMCE di routine. In sintesi, utilizzando questo protocollo, HPSC linee cellulari transgeniche nel AAVS1 locus libera di integrazione casuale può essere facilmente generati in 15 d con un'efficienza del 100% e senza la necessità di un'ampia caratterizzazione.

Figura 1: Schema di RMCE (A) linea temporale del programma di selezione RMCE.. A sinistra: la linea cellulare master (MCL), l'ospitare una cassetta FRT-fiancheggiato (triangoli) che esprimono GFP e Hygromycin-tk (collegata da peptidi auto-scissione 2A), è trasfettate da nucleofection (NF) con il vettore FLPE-esprimere e il vettore RMCE donatore, which contiene un gene promoterless puromicina resistenza (splicing accettore - SA- Puro R) e una cassetta sperimentale variabile (X). A destra: la nuova linea RMCE (RMCEL) ottenuto dopo il completamento della selezione con puromicina (triangolo rosso) e FIAU (linea blu). (B) RMCE utilizzando un costitutiva TDT-esprimono donatore e differenti rapporti di DNA donatore e FLPE DNA (0: 1, 2: 1, 2: 0) monitorato mediante microscopia a fluorescenza (GFP - fluorescenza verde, TDT - fluorescenza rossa) a diversi punti di tempo. D 2 e 6: scala barre rappresentano 100 micron. D 10: barre di scala rappresentano 200 micron. Clicca qui per vedere una versione più grande di questa figura.
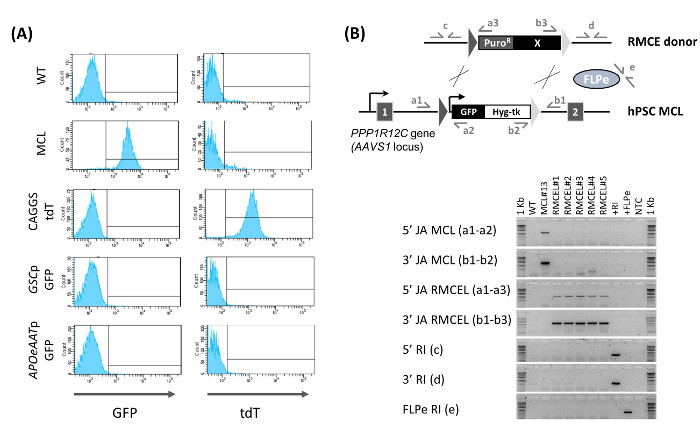
Figura 2:. Caratterizzazione di RMCE linee (A) Determinazione del RMCE bycitometria a flusso. GFP e di espressione TdT è indicata per la linea WT, linea cellulare master (MCL), e appena generato linea RMCE a D 15 post-nucleofection utilizzando un vettore costitutiva TDT-espressione (CAGGS TdT), o donatori con GFP guidata da un promotore GOOSECOID (GSCP GFP, marcatore endoderma definitivo) o di un promotore AAT (APOeAATp GFP, marcatore epatociti). (B) La conferma della RMCE (5 '/ 3' JA RMCEL), assenza di (MCL) cassette della linea cellulare master (5 '/ 3' JA MCL), e la mancanza di integrazione casuale (5 '/ 3' / FLPE RI ) mediante PCR utilizzando le coppie di primer raffigurati. DNA da WT, MCL, linee RMCE (1 - 5), donatori RMCE vettore (+ RI), FLPE vettore che esprime, e nessun controllo template (NTC) sono stati utilizzati. Figura modificato da (Ordovas et al., 2015) 13. Si prega di cliccare qui per vedere una versione più grande di questa figura.
entro-page = "1"> 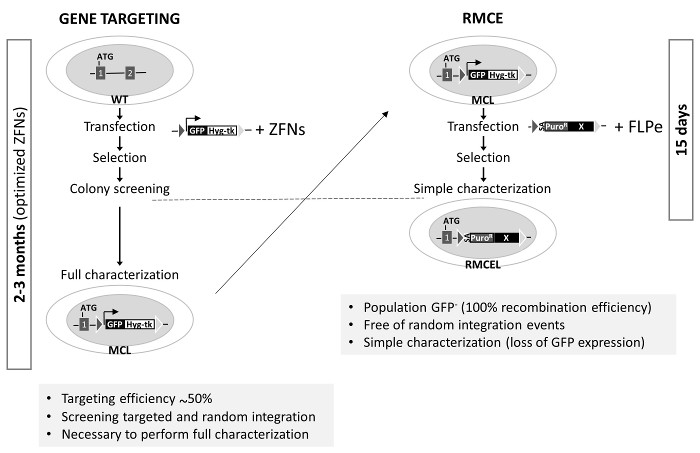
Figura 3. Generazione del RMCE-adatto master linea cellulare e Confronto di gene targeting con RMCE in AAVS1 sinistra:. Gene targeting utilizza cellule non modificati WT che sono cotransfected con un donatore (cassette descritto nella figura 1A per la generazione della linea cellulare maestro , MCL) e ZFNs ottimizzate specifiche. Il processo per completare la caratterizzazione completa richiede fino a 3 mesi. A destra: RMCE viene eseguita cotrasfezione del donatore con vettori FLPE, e una linea completamente caratterizzati viene generata in 15 d. Gene targeting efficienza in AAVS1 è da studi precedenti 1,2. Si prega di cliccare qui per vedere una versione più grande di questa figura.
Tabella 1. Composizione Media.
| saggio | Inoltrare | Inverso | amplicon | ciclo di PCR | ||
| 5'JA MCL | CACTTTGAGCTCTACTGGCTTC | CGTTACTATGGGAACATACGTCA | 1.1 Kb | 95 ° C, 5 '- [95 ° C, 30' '- 68 ° C (-0,5 ° C / ciclo), 1' 30 ''] X15 - [95 ° C, 30 '' - 58 ° C, 30 '' - 72 ° C, 1 '30' '] X25 - 72 ° C, 5' | ||
| 3'JA MCL | TAACTGAAACACGGAAGGAG | AAGGCAGCCTGGTAGACA | 1.4 Kb | 95 ° C, 5 '- [95 ° C, 30' '- 68 ° C (-0,5 ° C / ciclo), 1' 30 ''] X15 - [95 ° C, 30 '' - 58 ° C, 30 '' - 72 ° C, 1 '30' '] X25 - 72 ° C, 5' | ||
| 5'JA RMCEL | CACTTTGAGCTCTACTGGCTTC | CATGTTAGAAGACTTCCTCTGC | 1.1 Kb | 95 ° C, 5 '- [95 ° C, 30' '- 68 ° C (-0,5 ° C / ciclo), 1' 30 ''] X15 - [95 ° C, 30 '' - 58 ° C, 30 '' -72 ° C, 1 '30' '] X25 - 72 ºC, 5' | ||
| 3'JA RMCEL | TTCACTGCATTCTAGTTGTGG | AAGGCAGCCTGGTAGACA | 1.5 Kb | 95 ° C, 5 '- [95 ° C, 30' '- 68 ° C (-0,5 ° C / ciclo), 1' 30 ''] X15 - [95 ° C, 30 '' - 58 ° C, 30 '' - 72 ° C, 1 '30' '] X25 - 72 ° C, 5' | ||
| 5'RI DONATORE | GTACTTTGGGGTTGTCCAG | TTGTAAAACGACGGCCAG | 0.5 Kb | 95 ° C, 5 '- [95 ° C, 30' '- 60 ° C, 30' '- 72 ° C, 30' '] X25 - 72 ° C, 5' | ||
| 3'RI DONATORE | CCTGAGTTCTAACTTTGGCTC | ACACAGGAAACAGCTATGAC | 0.5 Kb | 95 ° C, 5 '- [95 ° C, 30' '- 60 ° C, 30' '- 72 ° C, 30' '] X25 - 72 ° C, 5' | ||
| RI FLPE | CCTAGCTACTTTCATCAATTGTG | GTATGCTTCCTTCAGCACTAC | 0.65 Kb | 95 ° C, 5 '- [95 ° C, 30' '- 60 ° C, 30' '- 72 ° C, 30' '] X25 - 72 ° C, 5' | ||
Tabella 2. set di primer utilizzati per la PCR genotipizzazione. Modificato da Ordovas et al. 2015 13.
Discussione
metodi di modifica gene in loci di porto sicuro rimangono uno strumento essenziale per lo sviluppo transgenesi in hPSCs. Anche se il carattere porto sicuro di AAVS1 è recentemente stata messa in discussione per alcune applicazioni 13,18, questo locus rimane attualmente la migliore caratterizzato nelle linee di cellule di derivazione umana. La consapevolezza dei suoi limiti in hPSCs può contribuire ad ottenere dati affidabili. Pertanto, AAVS1 ancora si pensa che essere un sito utile, ad esempio, per gli studi GAIN- e la perdita-di-funzione o espressione inducibile / costitutiva di fattori che richiedono un contesto isogenico all'interno e tra determinati background genetico.
Il locus AAVS1 è stato preso di mira da molti gruppi che utilizzano ZFNs, TALEN o CRISPR / Cas9 1,2,19. Queste nucleasi aumentano significativamente l'efficienza di ricombinazione omologa in un determinato locus. Tuttavia, il processo di schermo e caratterizzare completamente cloni correttamente mirati, gratuitamente integ casualerazione e di mantenere la pluripotenza e genomica integrità può richiedere fino a 3 mesi (utilizzando strumenti di editing gene ottimizzati) (Figura 3). Gli ultimi due possono essere causati da eventuali mutazioni generati dall'attività nucleasi centra la porta. Il sistema RMCE utilizzato qui, tuttavia, offre un metodo rapido, efficiente ed elegante per raggiungere questo obiettivo in sole due settimane, una volta specifici ricombinasi sequenze bersaglio sono preintegrate nel locus AAVS1. Uso di selezione positiva / negativa e un programma di selezione adeguata sono i principali fattori che contribuiscono alla semplificazione del montaggio gene nel locus AAVS1 da RMCE.
caratterizzazione genotipica delle nuove linee transgeniche è notevolmente ridotta (non screening clonale è necessario), e la caratterizzazione associata a centrare la porta nucleasica è reso superfluo a causa della specificità della FLPE per FUz. La caratterizzazione può anche essere ridotto in esperimenti di routine RMCE dimostrando l'completa perdita di espressione GFP dalla linea cellulare master (Figura 2A), in quanto lo scambio cassetta piena e mancanza di integrazione casuale sono già sufficientemente dimostrato mediante PCR (Figura 2B) e Southern blot 13. L'uso della selezione positiva / negativa costituisce anche la principale differenza con le relazioni precedenti. Diversi gruppi che in precedenza descritti RMCE in hPSCs, sia nel AAVS1 o di altri loci, impiegato solo un singolo passo selezione positiva 7,14,16. Ciò non costituisce un importante vantaggio tecnico su editing gene eseguita con nucleasi, perché lo screening colonia deve essere ugualmente eseguita per dimostrare corretta integrazione e assenza di integrazione casuale a livello clonale.
L'uso di questo sistema RMCE in molteplici linee hESC / IPSC richiede preintegrazione del descritto cassette FRT contenenti nel locus AAVS1 di ciascuna linea indipendente. Tuttavia, una volta generate,la sua rapidità e semplicità rende possibile lo sviluppo di semi-high throughput schermi genetiche in impostazioni isogenici definite per le applicazioni di cui sopra, che altrimenti sarebbe tecnicamente molto tempo. Inoltre, tutti i vettori RMCE sono costruiti all'interno PZ AAVS1 vettore gene-targeting utilizzato per indirizzare il locus con ZFNs, ma Talens o CRISPR / Cas9 sono stati segnalati anche. dualità del vettore RMCE è molto utile per generare più righe per un transgene determinato hPSCs con o senza FRT. Ad esempio, un colpo ha dimostrato con successo in uno schermo genetica decisa svolta da RMCE idealmente bisogno di essere testato in più righe HPSC per confermare i risultati. In assenza di diverse linee cellulari maestro RMCE-adatto, gene targeting diretto del colpo con nucleasi potrebbe essere eseguita. L'utilità del duplice carattere dei vettori RMCE è stato dimostrato dal nostro gruppo 20. In questo studio, la correzione genica è stata effettuata in paziente derivateDemenza frontotemporale (FTD) iPSCs che hanno una mutazione che causa progranulin (PGRN) carenza di espressione. Gene complementazione mediante inserimento ZFNs mediata nel locus AAVS1 di una cassetta che ripristinato livelli PGRN, correggere il fenotipo difettoso in corticogenesi associata alla presenza della mutazione. Inoltre, un analogo è stato generato da RMCE in hESC WT per essere usato come controllo per l'espressione PGRN esogena.
In assenza di colonie ricombinanti, controlla l'efficienza di trasfezione del vettore RMCE donatore. efficienze di trasfezione superiori al 30% danno i risultati sopra indicati. Bassa efficienza di trasfezione diminuiscono l'efficienza di ricombinazione. Infine, il sistema RMCE è stato generato nel locus AAVS1, ma è applicabile a qualsiasi altro locus.
Divulgazioni
The authors have nothing to disclose.
Riconoscimenti
L.O. was funded by IWT/OZM/090838, IACS BPAMER3/08/04, and the Government of Aragon FMI048/08; RB by IWT fellowship SB-121396; M.P. by FWO 1288714N; and R.S. by the Dutch Diabetes Foundation. N.H., J.V., K.C., and Q.C. were supported by IWT fellowships SB-121396, SB-101230, SB-91228, and SB-093228, respectively. Funding to C.M.V. was from FWO G.0667.07, G.0975.11, and KU Leuven (EIW-B4855-EF/05/11, ETH-C1900-PF, EME-C2161-GOA/11/012), IWT-HEPSTEM, BELSPO-IUAP-DEVREPAIR, FP7-HEMIBIO (266777).
Materiali
| Name | Company | Catalog Number | Comments |
| DR4 Mouse Embryonic Fibroblasts (MEF) | AMS bio | GSC-6204G | hPSC culture on feeers |
| CF-1 MEF, mitomycin-C treated | AMS Bio | GSC-6001M | |
| EmbryoMax 0.1% Gelatin Solution | Millipore | ES-006-B | iMEF plating |
| DMEM/F-12, HEPES | Gibco | 31330-038 | |
| KnockOut Serum Replacement | Gibco | 10828-028 | |
| L-Glutamine | Sigma Aldrich | G8540 | For hESC medium |
| L-Glutamine 200 mM | Gibco | 25030-081 | For iMEF medium |
| 2-Mercaptoethanol 50 mM | Gibco | 31350-010 | |
| MEM Non-Essential Amino Acids Solution (100X) | Gibco | 11140-035 | |
| Penicillin-Streptomycin (10,000 U/mL) | Gibco | 15140122 | |
| Human basic FGF | Peprotech | 100-18C | |
| Y-27632 in Solution | VWR | 688001-500 | |
| DMEM High Glucose | Gibco | 41965-039 | |
| Fetal Bovine Serum | Sigma Aldrich | F7524 | |
| Trypsin-EDTA (0.05%), phenol red | Gibco | 25300-054 | hPSC culture on feeders |
| Matrigel hESC qualified | VWR | 734-1440 | |
| mTeSR1 complete medium kit | Stem cell technologies | 05850 | For Feeder free culture |
| StemPro Accutase Cell Dissociation Reagent | Gibco | A1110501 | For feeder free culture |
| Y-27632 in solution | VWR | 56726 | |
| Human Stem Cell Nucleofector Kit 2 | Lonza | VVPH-5022 | |
| Puromycine Dihydrochloride | Gibco | A11138-03 | |
| FIAU (Fialuridine) | ABX | 2910 | |
| Hygromycin B | Sigma Aldrich | H3274 | |
| pFLPe | Modified to from pCAGGS-FLPe (MES4488, Open Biosystems) to remove puromycin | ||
| RMCE donors | - | - | Modified from pZ donor AAVS1 puromycin vector (PZD0020-1KT, Sigma Aldrich)13 |
| 5mL Round Bottom Polystyrene Test Tube, with Cell Strainer | Falcon | 352235 | |
| PureLink Genomic DNA kit | Invitrogen | K182001 | |
| GoTaq Flexi DNA Polymerase | Promega | M8298 | |
| Agarose | Sigma | A9539 | |
| SYBR Safe DNA Gel Stain | Invitrogen | S33102 | |
| Nucleofector 2b Device | Lonza (Amaxa) | AAB-1001 | |
| NucleoCounter NC-100 | Chemometec | NC-100 | |
| BD FACS Canto | BD Biosciences | 337175 | |
| NucleoCassette | Chemometec | 941-0002 |
Riferimenti
- Hockemeyer, D., et al. Genetic engineering of human pluripotent cells using TALE nucleases. Nat. Biotechnol. 29 (8), 731-734 (2011).
- Hockemeyer, D., et al. Efficient targeting of expressed and silent genes in human ESCs and iPSCs using zinc-finger nucleases. Nat. Biotechnol. 27 (9), 851-857 (2009).
- Mali, P., et al. RNA-guided human genome engineering via Cas9. Science. 339 (6121), 823-826 (2013).
- Baer, A., Bode, J. Coping with kinetic and thermodynamic barriers: RMCE, an efficient strategy for the targeted integration of transgenes. Curr. Opin. Biotechnol. 12 (5), 473-480 (2001).
- Sakurai, K., et al. Efficient integration of transgenes into a defined locus in human embryonic stem cells. Nucleic Acids Res. 38 (7), e96 (2010).
- Irion, S., et al. Identification and targeting of the ROSA26 locus in human embryonic stem cells. Nat. Biotechnol. 25 (12), 1477-1482 (2007).
- Di Domenico, A. I., Christodoulou, I., Pells, S. C., McWhir, J., Thomson, A. J. Sequential genetic modification of the hprt locus in human ESCs combining gene targeting and recombinase-mediated cassette exchange. Cloning Stem Cells. 10 (2), 217-230 (2008).
- Lombardo, A., et al. Site-specific integration and tailoring of cassette design for sustainable gene transfer. Nat. Methods. 8 (10), 861-869 (2011).
- Lombardo, A., et al. Gene editing in human stem cells using zinc finger nucleases and integrase-defective lentiviral vector delivery. Nat. Biotechnol. 25 (11), 1298-1306 (2007).
- Simth, J. R., et al. Robust , Persistent Transgene Expression in Human Embryonic Stem Cells Is Achieved with AAVS1-Targeted Integration. Stem Cells. 26, 496-504 (2008).
- DeKelver, R. C., et al. Functional genomics, proteomics, and regulatory DNA analysis in isogenic settings using zinc finger nuclease-driven transgenesis into a safe harbor locus in the human genome. Genome Res. 20 (8), 1133-1142 (2010).
- Qian, K., et al. A simple and efficient system for regulating gene expression in human pluripotent stem cells and derivatives. Stem Cells. 32 (5), 1230-1238 (2014).
- Ordovás, L., et al. Efficient Recombinase-Mediated Cassette Exchange in hPSCs to Study the Hepatocyte Lineage Reveals AAVS1 Locus-Mediated Transgene Inhibition. Stem Cell Reports. 5 (5), 918-931 (2015).
- Du, Z. -. W., Hu, B. -. Y., Ayala, M., Sauer, B., Zhang, S. -. C. Cre recombination-mediated cassette exchange for building versatile transgenic human Embryonic Stem Cells lines. Stem Cells. 27, 1032-1041 (2009).
- Papapetrou, E. P., et al. Genomic safe harbors permit high β-globin transgene expression in thalassemia induced pluripotent stem cells. Nat. Biotechnol. 29 (1), 73-78 (2011).
- Ramachandra, C. J., et al. Efficient recombinase-mediated cassette exchange at the AAVS1 locus in human embryonic stem cells using baculoviral vectors. Nucleic Acids Res. 39 (16), e107 (2011).
- Takata, Y., Kondo, S., Goda, N., Kanegae, Y., Saito, I. Comparison of efficiency between FLPe and Cre for recombinase-mediated cassette exchange in vitro and in adenovirus vector production. Genes to Cells. 16 (7), 765-777 (2011).
- Mizutani, T., Li, R., Haga, H., Kawabata, K. Transgene integration into the human AAVS1 locus enhances myosin II-dependent contractile force by reducing expression of myosin binding subunit 85. Biochem. Biophys. Res. Commun. 465 (2), 270-274 (2015).
- Mali, P., et al. RNA-guided human genome engineering via Cas9. Science. 339 (6121), 823-826 (2013).
- Raitano, S., et al. Restoration of progranulin expression rescues cortical neuron generation in an induced pluripotent stem cell model of frontotemporal dementia. Stem Cell Reports. 4 (1), 16-24 (2015).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon