Method Article
Imaging multimodale gerarchica delle sezioni di serie per l'individuazione di specifici obiettivi cellulari all'interno di grandi volumi
In questo articolo
Riepilogo
Questo protocollo mira alle cellule specifiche nel tessuto per immagini a risoluzione nanometrica usando un microscopio elettronico a scansione (SEM). Un numero elevato di sezioni di serie da materiale biologico resina-incastonato prima è imaged in un microscopio chiaro per identificare la destinazione e quindi in modo gerarchico in SEM.
Abstract
Targeting per celle specifiche ultrastrutturali risoluzione all'interno di una popolazione mista delle cellule o un tessuto può essere ottenuto gerarchica imaging utilizzando una combinazione di luce e microscopia elettronica. Campioni incorporati in resina sono sezionati in matrici costituito da nastri di centinaia di sezioni ultrasottili e depositati su pezzi di wafer di silicio o vetrini coprioggetti conduttivamente rivestiti. Le matrici sono imaging a bassa risoluzione utilizzando un consumatore digitale come smartphone fotocamera microscopio ottico (LM) per una panoramica rapida grande area o un microscopio a fluorescenza campo largo (luce microscopia di fluorescenza (FLM)) dopo l'etichettatura con fluorofori. Dopo post-colorazione con metalli pesanti, le matrici sono imaging in un microscopio elettronico a scansione (SEM). Selezione del target è possibile da ricostruzioni 3D generati da FLM o da ricostruzioni 3D fatti dagli stack di immagine di SEM a risoluzione intermedia se non marcatori fluorescenti sono disponibili. Per l'analisi ultrastrutturale, target selezionati sono infine registrato nel SEM presso ad alta risoluzione (pixel immagine di pochi nanometri). È dimostrato uno strumento di gestione nastro che possa essere adattato a qualsiasi ultramicrotomo. Aiuta con la produzione di matrice e rimozione del substrato dalla barca coltello taglio. Una piattaforma software che permette l'imaging automatici di matrici nel SEM è discussa. Rispetto ad altri metodi di generazione di dati di grande volume EM, come seriale blocco-viso SEM (SBF-SEM) o fascio ionico focalizzato (FIB-SEM), SEM questo approccio ha due vantaggi principali: (1) il campione di resina-incastonato è conservato, sia pure in una versione tagliata. Può essere tinto in modi diversi e ripreso con differenti risoluzioni. (2) come le sezioni possono essere post-macchiate, non è necessario utilizzare campioni fortemente macchiato di blocco con metalli pesanti per introdurre di contrasto per imaging di SEM o blocchi di rendering del tessuto conduttivo. Ciò rende il metodo applicabile a una vasta gamma di materiali e domande biologiche. Materiali particolarmente con prefisso ad es., da banche di biopsia e laboratori di patologia, può direttamente essere incorporato e ricostruito in 3D.
Introduzione
Per la ricostruzione di grandi volumi di tessuto alla risoluzione ultrastrutturale un numero di differenti approcci di imaging basata su SEM è stati usati1: recensioni complete sono disponibili ad es., per SBF-SEM2, FIB-SEM3e Array Tomografia (a)4. Mentre per il secondo metodo il materiale campione è conservato come una matrice di sezioni di serie su un substrato, SBF-SEM e FIB-SEM sono metodi distruttivi, lavorando sul blocco campione e consumarlo durante la formazione immagine. A causa della ricarica della resina nel SEM, dipendono anche fortemente metallizzato campione blocchi5.
D'altra parte, identificando determinate cellule o strutture di interesse all'interno di un campione di tessuto possa trarre profitto in particolare da correlativo luce e microscopia elettronica (CLEM)6,7,8. Utilizzando FLM per il targeting osti all'applicazione di grandi quantità di metalli pesanti, poiché ciò sarebbe spegnere il segnale di fluorescenza9. Per tali campioni solo leggermente metallizzati, AT è il metodo di scelta dato che le matrici possono facilmente essere post-macchiate con metallo pesante dopo formazione immagine LM. Inoltre, quasi ogni tipo di campione può essere utilizzato per AT, anche routine campiona il patologo tesoro petto10.
Un altro grande vantaggio di AT è il potenziale per gerarchica11 o multi-resolution imaging12: non è necessario tutto in alta risoluzione di immagine, come gli obiettivi possono essere selezionati in una modalità diversa (ad es., FLM) o in immagini a bassa risoluzione del SEM. Solo le regioni interessanti di una popolazione dei tessuti o cellule a ad alta risoluzione di imaging consente di risparmiare spazio di archiviazione di dati digitali e produce più piccoli insiemi di dati di immagine, che sono più facili da gestire. Qui, il flusso di lavoro AT è dimostrato utilizzando un campione piuttosto debolmente metallizzato: alta pressione congelato le radici delle piante (Arabidopsis thaliana) incorporato in resina idrofila.
Come le matrici sono preparate, macchiate e fotografate in entrambi FLM e SEM, e come la serie di immagini sono registrati sono spiegati. Inoltre, come la ricostruzione 3D del volume FLM può essere utilizzata per selezionare celle specifiche per formazione immagine nel SEM a risoluzione nanometrica è illustrata.
Protocollo
Nota: Blocchi campione dovrebbero essere polimerizzati e contengono alcuni metalli pesanti. Fissazione e l'incorporamento di protocolli per i due campioni, mostrati in Figura 1A-B sono stati descritti altrove11. In breve, l'esempio mostrato in Figura 1A è stato fissato chimicamente, macchiato del blocco dell'en prima con OsO 1%4, quindi con 1% di acetato di uranile e incorporati in resina di Spurr. L'esempio mostrato in Figura 1B è stato congelato ad alta pressione, congelare sostituito con acetato di uranile 0,4% in acetone e incorporati in resina Lowicryl HM20. Utilizzare guanti senza polvere per le operazioni di preparazione successiva.
1. creazione di Array
- Taglio del blocco campione
Nota: Serrare sempre bene durante l'inserimento di parti.- Inserire il blocco porta-campione il portacampioni dell'ultramicrotomo, posizionare il supporto nel blocco di taglio e farlo slittare il livello più basso dell'ultramicrotomo.
- Tagliare via resina intorno al tessuto incorporato con una lama di rasoio lasciando solo un piccolo bordo di resina intorno al campione. Tagliare dalla parte superiore fino a raggiungere il target.
Nota: La forma del blocco-viso può essere rettangolare o trapezoidale (abbiamo utilizzato con successo entrambe le forme). È importante utilizzare i lati più lunghi del rettangolo come il leader e il bordo finale per assicurare che una vasta area è coperta da miscela colla stabilizzando i nastri. - Inserire il supporto del campione nel braccio dell'ultramicrotomo e sostituire il blocco di taglio nella fase inferiore del portalama. Inserire un coltello di taglio di diamante (normalmente 45°), il portalama. Allineare il blocco-faccia esattamente parallela al bordo di taglio coltello.
Nota: Per produrre esattamente parallelo iniziali (in basso) e finali i margini (superiore), ruotare o spostare solo il coltello per tagliare i lati opposti. Per grandi volumi (più di parecchie centinaia sezioni), un coltello di taglio di 90° è vantaggioso. - Lisciare tutti e quattro i lati, quindi ruotare il supporto del campione in modo che i leader e il bordo d'uscita sono ora in posizione orizzontale.
- Cappotto con attenzione i lati iniziali e finali del blocco con miscela adesiva. Utilizzare un pennello piccolo, formato da pochi peli fissati a un stuzzicadenti13. Eseguire questo passaggio rapidamente perché il solvente di questa miscela evapora in pochi secondi. Non contaminare il blocco-viso con questa miscela. Asciugare il blocco campione rivestito per 5-10 min.
Nota: per un più grande numero di sezioni (> 200), potrebbe essere meglio rivestire il bordo solo, perché con il tempo, un rigonfiamento di colla potrebbe costruire sul bordo d'uscita e potenzialmente tirare indietro sezioni sopra il filo del rasoio.
- Preparazione del substrato
- Tagliare pezzi di wafer di silicio a una dimensione che si adatta in barca coltello (circa 2 x 2,5 cm2 per il coltello Jumbo). Se necessario, contrassegnare i pezzi di wafer (numeri o lettere) con uno scriber diamante prima di pulire o con pennarello indelebile dopo la pulizia. Pulire la lastra di silicio manualmente con isopropanolo e salvietta.
Nota: Per formazione immagine correlativa utilizzare ossido di indio-stagno (ITO)-rivestito vetrini coprioggetti. Essi devono essere maneggiati con cura e pulizia supplementare non è necessaria. - Difficoltà il substrato ad una estremità della placca utilizzando un adesivo rimovibile.
Nota: In alternativa, i bordi del substrato possono essere appuntati paralleli il filo del rasoio utilizzando due strisce di nastro adesivo verso il basso. - Al plasma attivare (Scarica a bagliore) il substrato con aria per ottenere una superficie idrofila. Questo dovrebbe essere fatto in modo che una goccia d'acqua inserito i substrato si diffonde in un film molto sottile (basso angolo di contatto, Figura 2, D). I parametri idrofilizzazione dipendono dal dispositivo al plasma utilizzato; per i parametri utilizzati qui, Vedi Tabella materiali.
Nota: L'attivazione del Plasma è molto volatile, quindi eseguire questo immediatamente prima dell'uso di substrato. - Inserire il supporto nel morsetto di un titolare di substrato, con il substrato montato più vicino per il filo del rasoio per raggiungere ottimale bagnabilità del substrato sulla barca di coltello.
Nota: Una descrizione dettagliata del proprietario del substrato, compresi i disegni di progettazione assistita da elaboratore (CAD), è data in Wacker et al. 11
- Tagliare pezzi di wafer di silicio a una dimensione che si adatta in barca coltello (circa 2 x 2,5 cm2 per il coltello Jumbo). Se necessario, contrassegnare i pezzi di wafer (numeri o lettere) con uno scriber diamante prima di pulire o con pennarello indelebile dopo la pulizia. Pulire la lastra di silicio manualmente con isopropanolo e salvietta.
- Preparazione di sezionamento
- Inserire una lama di diamante Jumbo del portalama, impostare l'angolo di spoglia (0° per coltello Jumbo) e riempire la barca di coltello con acqua distillata. Il coltello di approccio ad una distanza di 1 – 2 mm al campione.
- Abbassare il substrato in acqua tramite viti 1 – 3 (Figura 2A) del proprietario del substrato. Controllare che la linea di galleggiamento si trova nel terzo superiore del substrato.
Nota: Controllare che la colla tra il substrato e portante la piastra è molto curata da spingendo il substrato con una pinza pulita. Non deve muoversi. - Perché è difficile vedere l'altezza da terra quando si utilizza un wafer di silicio, abbassare il substrato fino a che si toccano il pavimento. Ora alza il substrato una piccola quantità. Assicurarsi che il substrato né il vettore tocca la barca di lama durante il taglio.
- Utilizzare una siringa o una pipetta per regolare il livello dell'acqua nella barca. Mentre si guarda attraverso il binocolo, aggiungere o rimuovere l'acqua fino a quando l'intera area della superficie dell'acqua presenta una riflessione omogenea della luce illuminazione superiore dell'ultramicrotomo.
- Accendere la luce di fondo dell'ultramicrotomo. Assicurarsi che il braccio è in posizione centrale e non in ritrazione utilizzando la rotella di mano dell'ultramicrotomo. Approccio il coltello al campione fino a quando il riflesso del del coltello è visibile sulla faccia del blocco.
- Utilizzare le opzioni di regolazione dell'ultramicrotomo per allineare il campione al coltello. Prima di ruotare il coltello, poi ruotare e inclinare il campione.
Nota: È allineato correttamente se la striscia di luce, che può essere visto nel divario tra il campione e il coltello, Mostra bordi paralleli (linee parallele dritto e non a forma di Cuneo). - Controllare l'inclinazione del campione: assicurarsi che la striscia di luce non diventa più spessa o più sottili, mentre il campione in movimento su e giù. Se necessario, è possibile utilizzare la vite di regolazione ad arco per risolvere il problema. Spostare il coltello più vicino al campione fino a quando è appena sopra la faccia di blocco (ma non lo tocchi).
- Impostare lo spessore di sezione (feed), taglio di velocità e la finestra di taglio presso l'unità di controllo.
- Avviare il sezionamento. Se necessario, attendere fino a quando la prima sezione completa viene tagliata. Tagliare alcune sezioni per essere sicuri che si attaccano insieme ai nastri di forma (altrimenti la colla deve essere applicato nuovamente). Iniziare con un alto valore di alimentazione (Max, 200 nm per il coltello Ultra) fino a quando la prima sezione completa viene tagliata. Quindi, impostare il valore desiderato di mangimi. Per stabilità adeguata nastro, uno spessore della sezione di 100 nm e una velocità di taglio di 1 mm/s è un buon punto di partenza. Lo spessore di sezione realizzabile più basso è di circa 60 nm, a seconda della qualità del campione.
- Smettere di sezionamento. Rimuovere tutte le sezioni (parzialmente tagliate) non necessari dal filo del rasoio e barca usando i capelli di un gatto/ciglia. Se c'è un sacco di piccoli detriti galleggianti intorno, rimuovere l'acqua completamente con una pipetta e riempire la barca con acqua fresca. Ora il processo è pronto per il primo fiocco produttivo.
- Sezionamento
- Avviare il sezionamento. Una volta che è stato tagliato un numero di sezioni (il numero effettivo dipende dalle dimensioni delle sezioni e substrato), interrompere il procedimento di taglio e rilasciare il nastro dal filo del rasoio accarezzando delicatamente sopra il filo del rasoio con un ciglio14 o meglio ancora, con un molto capelli morbidi da pelliccia di un gatto.
- Manipolare (push/pull) il nastro con le ciglia verso il substrato e fissare la prima sezione al substrato.
Nota: È necessario spingere delicatamente il nastro finché non si attacca alla parte secca del substrato. - Continuare sezionamento ed allegando i nastri al substrato. Incominciare da un lato e spostare gradualmente oltre a altro con ogni nuova barra multifunzione.
Nota: Evitare movimenti massiccia dell'acqua per evitare l'allentamento i nastri già allegati. Lo stesso vale per correnti d'aria. Usare lo scudo di respiro consegnato con l'ultramicrotomo. In condizioni ambientali sfavorevoli, un recinto per l'ultramicrotomo è consigliato15. - Quando il substrato è completamente coperto con i nastri (4 – 5 nastri sono solitamente fattibile), il substrato delicatamente alza-fuori dalla barca coltello usando le viti micromanipolatore del proprietario del substrato.
Nota: I movimenti adatti sono: sollevare verticalmente (vite 1) e di rotazione/inclinazione (vite 3), o una combinazione di entrambi. - Lasciate la matrice di nastro asciugare prima di riporlo in un ambiente privo di polvere. Dopo l'essiccazione, rimuovere l'adesivo substrato montato appena possibile da parte del vettore (sullo stesso giorno, altrimenti il substrato potrebbe essere troppo difficile da rimuovere o potrebbe persino rompere durante lo smontaggio).
2. colorazione per l'Imaging di LM
Nota: Diversi metodi di colorazione/etichettatura sono possibili, inclusi i protocolli di immunofluorescenza. Qui un diretto, piuttosto aspecifiche macchia viene scelto per delineare le pareti cellulari.
-
Ioduro di propidio
- Coprire il fondo di un bicchiere grande di Petri (diametro 30 cm) con parafilm e linea il bordo del piatto con tessuto bagnato per costruire una camera umida.
- Usare circa 300-500 µ l di soluzione al vetrino coprioggetti. Depositare una goccia per ogni vetrino coprioggetti sul parafilm e mettere sottosopra il vetro sulla goccia, in modo che le sezioni sono a contatto con il fluido di colorazione. Incoperchiare il recipiente e avvolgerlo con un foglio di alluminio per proteggere i campioni dalla luce. Incubare i campioni per 16 h a 4 ° C.
- Rimuovere il vetrino coprioggetto con le pinzette e lavarlo con un movimento su e giù in un becher da 100 mL riempito con 80 mL di acqua distillata. Ripetere questo passaggio in un altro becher con acqua fresca. Asciugare il vetrino coprioggetto accuratamente con aria compressa.
3. registrare la serie di immagini nella FLM
- Posto il coprioggetto sul palco di un comune FLM grandangolari.
- Scegliere il filtro appropriato set (Tabella materiali) per la fluorescenza devono essere rispettate.
- Con un obiettivo adatto, scattare una foto dell'oggetto in ogni sezione: cercare di riempire il campo di vista e mantenere costante l'orientamento. Per la punta della radice, è stato usato un obiettivo 40x aria.
Nota: Se i nastri non sono perfettamente allineati, un palco rotante può aiutare a riorientare le sezioni durante l'assunzione di immagini. Se possibile, centrare l'immagine su una caratteristica specifica o mantenere una funzionalità come ad esempio il bordo della sezione ad una distanza uguale al bordo dell'immagine registrata. - Se possibile, utilizzare 16 bit per limitare pixel saturi e mantenere costante il tempo di esposizione.
4. registrazione dello Stack immagine FLM
- Importare la serie di immagini in Fiji16 come uno stack virtuale.
- Aprire un nuovo TrakEM17 (vuota) dal menu file.
- Fare clic con il pulsante destro nel campo immagine e importare lo stack in TrakEM come "Una fetta per strato".
- Allineare i livelli (click destro nel campo image), impostare l'intervallo (prima immagine all'ultimo) e scegliere nessuno come riferimento. Per tutte le impostazioni, utilizzare i valori predefiniti e scegliere rigida come la trasformazione desiderata.
- Quando la registrazione è finito e soddisfacente, Risparmia il dataset allineato da tasto destro del mouse e scegliere Esporta. Rendere l'immagine piatta, impostare l'intervallo dalla prima all'ultima immagine e lasciare che il software Visualizza lo stack risultante. Salvare lo stack in formato tif.
5. colorazione e montaggio per l'Imaging di SEM
Nota: Per la preparazione delle soluzioni di colorazione Vedi Tabella materiali. Soluzioni possono essere conservati a 4 ° C fino a 12 mesi, protette dalla luce e aria.
Attenzione: Acetato di piombo citrato e uranile contengono metalli pesanti tossici. Indossare guanti e smaltire i rifiuti secondo le istruzioni delle autorità locali.
- Coprire il fondo di un bicchiere grande di Petri (diametro 30 cm) con parafilm e linea il bordo del piatto con tessuto bagnato per costruire una camera umida.
Nota: È importante che diversi pellet di NaOH sono posizionati all'interno di Petri vicino le gocce di colorazione per evitare eccessiva precipitazione di citrato di piombo. -
La macchiatura di acetato di uranile
- Centrifugare la soluzione di acetato di uranile a 2.680 x g per pochi secondi per piccole particelle di sedimento.
- Usare circa 300 – 500 µ l di soluzione al vetrino coprioggetti. Depositare una goccia per ogni vetrino coprioggetti sul parafilm e mettere sottosopra il vetro sulla goccia, in modo che le sezioni sono a contatto con il fluido di colorazione.
- Incubare per 10 minuti a temperatura ambiente e coprire il piatto durante la colorazione.
- Rimuovi il coprioggetto con forcipe e lavare spostando su e giù in un bicchiere riempito con acqua distillata (Vedi punto 2.1.3).
-
Piombo citrato macchiatura
- Durante l'incubazione di acetato di uranile, preparare la soluzione di citrato di piombo.
Nota: Piombo citrato dovrebbe sempre essere filtrata immediatamente prima dell'uso per rimuovere eventuali precipitati. Anche Centrifugare la soluzione di citrato di piombo a 2.680 x g per pochi secondi come descritto al punto 5.2.1. - Usare circa 300 – 500 µ l di soluzione al vetrino coprioggetti. Depositare una goccia per ogni vetrino coprioggetti sul parafilm immediatamente prima di lavare i vetrini coprioggetti dopo la macchiatura di acetato di uranile e mettere sottosopra il vetro sulla goccia, in modo che le sezioni sono a contatto con il fluido di colorazione. Posizionare le gocce (300 – 500 µ l) sul parafilm immediatamente prima di lavare i vetrini coprioggetti dopo la macchiatura di acetato di uranile.
Nota: Per evitare la formazione di precipitati, non respirare sulle goccioline di citrato di piombo. - Porre il coprivetrino lavato upside-down sul menu (non c'è nessun bisogno di asciugare).
- Incubare per 5 min a temperatura ambiente e coprire il piatto durante la colorazione.
- Rimuovere il vetrino coprioggetto con le pinzette e lavarlo come descritto in precedenza in un becher con acqua fresca (punto 5.2.4).
- Durante l'incubazione di acetato di uranile, preparare la soluzione di citrato di piombo.
- Asciugare il vetrino coprioggetto accuratamente con aria compressa.
-
Esempi di montaggio per l'imaging di SEM
- Montare i wafer di silicio su stub di alluminio con un pad di carbonio appiccicoso.
Nota: Le lamelle rivestite con ITO possono essere montate utilizzando vernice argento e Cu-nastro — assicurarsi che la superficie conduttiva è collegata allo stub — o con pastiglie di carbonio come sopra. In tal caso, un collegamento conduttivo da ITO-superficie allo stub può essere fatto con una goccia di vernice argento.
- Montare i wafer di silicio su stub di alluminio con un pad di carbonio appiccicoso.
6. gerarchico di formazione immagine nel SEM
Nota: In un'emissione di campo SEM, scegliere un basso consumo energetico primario (3 kV o inferiore), un fascio di corrente in un intervallo da 50 a 800 pA per evitare di caricare e una distanza di lavoro adatto per efficiente raccolta di elettroni secondari e/o retro-sparsi. Selezione della larghezza corrente dipende dalle proprietà del campione (ad es., l'incorporamento di resina); la dose di elettrone sarà anche un compromesso tra una piccola corrente (meno dannosa per il campione) e a corrente forte, che è benefico per l'imaging di velocità e di conseguenza abbassa il tempo di acquisizione immagine totale. Dedicato rivelatori per elettroni sparsi schiena forniscano buon contrasto, sono meno sensibili alla carica del campione e mostrano meno di manufatti di superficie del campione (pieghe, segni di coltello). Contrasto e luminosità deve essere regolate in modo tale che l'istogramma è centrato.
-
Formazione immagine di SEM
- Prima di definire i quattro angoli della matrice afferrando un'immagine di ogni angolo a basso ingrandimento, circa 100. Creare un'area di interesse (ROI) che racchiude l'intera matrice. Assegnare un protocollo di imaging con i seguenti parametri: utilizzare un rivelatore elettronico secondario (SE), che consente per immagini ad alta velocità a una dimensione di pixel di grande immagine (per esempio 1.000 nm) e un tempo di sosta breve (ad es., 0,2 µs).
Nota: Per superare limiti ottici elettrone alle grandi scansione campi di vista (FOV), che potrebbe causare distorsioni alla periferia delle immagini, utilizzare modalità di ingrandimento basso dedicato (forniti dalla maggior parte dei produttori di SEM) o usare mezzo, campi 1 e 2 k scansione per singole immagini. - Generare una sezione impostata creando un ROI che delinea solo il tessuto nella prima sezione. Clonarlo per tutte le sezioni successive utilizzando lo strumento timbro. Ruotare il ROIs quando necessario per ospitare nastri piegati.
- Registrare la serie di immagine utilizzando una dimensione di pixel intermedi (circa 50 nm) e una pausa di tempo abbastanza lungo per identificare e riconoscere la struttura di destinazione. Utilizzare un FOV per singole immagini in un intervallo di 6-10 k pixel.
Nota: Il software Atlas 5 può raccogliere automaticamente mosaici composti da immagini adiacenti per coprire vaste aree ROI/sezione attraverso le sezioni seriali. - Creare un sito situato all'interno di questo set di sezione, che contiene la struttura di destinazione per l'imaging ad alta risoluzione SEM superiore. Rendere il ROI abbastanza grande per tenere conto di precisione di fase. Controllare e regolare le posizioni dei siti.
Nota: È importante posizionare il ROIs in modo tale che il centro, dove verrà eseguite la messa a fuoco automatica e autostigmation, non si siede sul materiale "vuoto" con nessun dettaglio strutturale, ad es., vacuoli. - Definire le impostazioni di messa a fuoco automatica e controllare le prestazioni almeno la lunghezza di un nastro (cioè, la distanza più lunga che la fase deve viaggiare) su un piccolo ROI nei pressi del sito che verrà essere imaged.
- Definire un protocollo di imaging per l'acquisizione di SEM ad alta risoluzione. Per vedere i compartimenti di membrana, scegliere dimensione pixel immagine di 3 – 5 nm. Selezionare un tempo di sosta a seconda il rivelatore in modo che l'immagine non è troppo rumoroso.
- Prima di iniziare l'acquisizione, è possibile definire i valori di messa a fuoco su almeno la prima sezione di ogni nastro utilizzando l'opzione del protocollo di controllo.
- Ricominciare l'imaging automatici SEM tutta la serie di destinazione ROIs.
- Esportare i dati acquisiti come serie di immagini, preferibilmente in formato tif.
- Prima di definire i quattro angoli della matrice afferrando un'immagine di ogni angolo a basso ingrandimento, circa 100. Creare un'area di interesse (ROI) che racchiude l'intera matrice. Assegnare un protocollo di imaging con i seguenti parametri: utilizzare un rivelatore elettronico secondario (SE), che consente per immagini ad alta velocità a una dimensione di pixel di grande immagine (per esempio 1.000 nm) e un tempo di sosta breve (ad es., 0,2 µs).
7. registrazione dello Stack immagine SEM
- Importazione di serie di immagini in Fiji come uno stack virtuale.
Nota: Questi saranno i file di dati di grandi dimensioni nel range di pochi GB a seconda del numero di sezioni e la dimensione del ROI. - Ritaglia l'immagine di SEM dello stack per l'ulteriore elaborazione in un'area più vicino la struttura di interesse possibile e regolare la luminosità e il contrasto.
- Aprire un nuovo TrakEM17 (vuota) dal menu file.
- Fare clic con il pulsante destro nel campo immagine e importare lo stack in TrakEM come "Una fetta per strato".
- Allineare gli strati (click destro nel campo image), scegliere minimi quadrati come modalità, impostare l'intervallo (prima immagine all'ultimo) e scegliere nessuno come riferimento. Per le impostazioni, utilizzare i valori predefiniti e scegliere rigida come la trasformazione desiderata.
- Quando la registrazione è completata e soddisfacente, Risparmia il dataset allineato da tasto destro del mouse e scegliere Esporta. Rendere un'immagine piatta, impostare l'intervallo dalla prima all'ultima immagine e lasciare che il software Visualizza lo stack risultante. Salvare lo stack in formato tif.
Risultati
Il flusso di lavoro descritto qui (Figura 1) inizia con un campione incorporato in un blocco di resina. Durante la preparazione del campione, alcuni metalli pesanti dovrebbe essere introdotto nel tessuto, ma non è necessario l'utilizzo di protocolli ottimizzati per metallizzazione piuttosto forte. Figura 1A Mostra una radice di pianta (crescione) blocco tinto convenzionalmente con 1% OsO4 e 1% acetato di uranile, mentre la radice di Arabidopsis in Figura 1B è solo debolmente metallizzato con acetato di uranile 0,5%. Il tipo di campione di quest'ultimo è più adatto per correlativi approcci come alcuni metalli pesanti tendono a placare la fluorescenza. Con un supporto dedicato di substrato (Figura 2), le matrici di parecchie centinaia di sezioni possono essere prodotte (Figura 1). Dopo l'etichettatura fluorescente, tali matrici sono fotografate in un FLM grandangolari standard (Figura 1), poi tinto con soluzioni di metalli pesanti e fotografate in un SEM a diverse risoluzioni (Figura 1E–G).
Importanti strumenti per la generazione riproducibile di matrici, particolarmente quando immissione diversi nastri da barca lama del microtomo su un substrato, sono titolare del substrato (Figura 2A, progettata in laboratorio degli autori) e un diamante di Jumbo coltello con una barca abbastanza grande da ospitare vetrini da microscopio (Figura 2B). Un menisco piatto, permettendo la buona osservazione dei nastri, è necessario e può essere realizzato dalla pulizia al plasma del substrato: una piccola goccia di acqua distillata non deve formare una struttura lente sul substrato come in Figura 2 (substrato non trattato), ma un film sottile (Figura 2D, substrato del plasma attivato). In queste condizioni, nastri attaccate alla parte secca di un vetrino coprioggetti rivestite con ITO sono facilmente visibili (Figura 2E) e può essere osservato e controllato durante l'alza-fuori del substrato dall'acqua.
Ad esempio, le matrici colorate con ioduro di propidio per etichettare le pareti cellulari della pianta erano imaged con un ampio campo standard FLM (Figura 3A). Poiché le sezioni sono solo 100 nm di spessore, anche over-macchiatura come illustrato di seguito introduce little sfocatura. Dopo la registrazione, sono state selezionate le due celle completamente racchiuse nel volume ricostruito dallo stack di immagine (Figura 3B) per imaging ad alta risoluzione in 3D (veda inoltre Supplementare film S1). A seguito di ulteriori macchiatura con acetato e piombo citrato di uranile, le matrici erano imaged in SEM. Figura 3 C Mostra una panoramica, registrata con 60 pixel di immagine nm; il quadrato scuro al centro dell'immagine indica la posizione dove sono state eseguite le funzioni di messa a fuoco automatica, e la dose aggiuntiva ha portato alla contaminazione leggera. ROIs in quelle sezioni seriali (fette 51-248 di 435 fette in totale) del caso contenente le celle di due destinazione selezionate nello stack di FLM sono state poi registrate con una dimensione di pixel di immagine nm 5 (Figura 3D; veda inoltre Supplementare film S2).
Imaging automatici gerarchica delle matrici nel SEM descritto qui è stato fatto con la soluzione di piattaforma hardware/software ZEISS Atlas 5. In primo luogo, una panoramica di tutto l'array è stata creata utilizzando il rivelatore di SE, con molto grande (1.000 nm) immagine pixel e tempo di permanenza molto basso (Figura 4A). Un ROI che delinea solo il tessuto viene posizionato sulla prima sezione e propagato a tutte le altre sezioni della matrice. Questo set di sezione è stato poi registrato con 60 pixel di immagine nm utilizzando un più lungo tempo di sosta (Figura 4B). Infine, un insieme di sito, che contiene le cellule due bersaglio più uno "strato" di che circonda le cellule per tenere conto per l'inesattezza di fase, è stato istituito con i seguenti parametri: rilevatore di ESB (energia Backscatter selettiva), immagine di nm 5 pixel, molto lungo (40 µs) abitare ( del tempo Figura 4). Zoomando a tale immagine Mostra dettaglio subcellulare (Figura 4) quali vacuoli (V), mitocondri (M), nucleo (N) e del reticolo endoplasmatico (frecce). Veda inoltre il Supplemental film S3 per lo zoom in da una panoramica di tutto l'array al dettaglio subcellulare di una cella di destinazione.
La matrice mostrata qui (200 sezioni) più un ulteriore uno dei 250 sezioni ha preso circa 8 h per produrre, una notte a macchiare per LM e un giorno registrare (manualmente) presso il FLM. Post-colorazione dura circa 1-2 h in totale, a seconda del numero di singole matrici. Per la registrazione di SEM, poche ore sono necessarie per impostare l'esecuzione di Atlas e registrazione automatizzata era 3 – 4 h per il set di sezione (dimensione dei pixel 60 nm) intermedio ad alta risoluzione (200 sezioni, 450 x 200 µm2) e circa 5 giorni per l'alta risoluzione (5 nm dimensioni in pixel) ROI contenente le celle di due destinazione (200 sezioni, 55 x 30 µm2). Si noti che a causa del basso contenuto di metallo dell'esempio mostrato qui, una velocità di scansione molto lenta dovuto essere utilizzato per raggiungere una buona rilevazione di segnale-rumore, che implicita (per il rilevatore attualmente disponibile) un tempo di sosta di 40 µs per il ROI ad alta risoluzione.
Ci sono diversi passaggi del flusso di lavoro intera incline ai trabocchetti: idealmente nastri dovrebbero essere più o meno dritto e collocato nel giusto ordine (Figura 5A). Tuttavia, sono spesso prodotti piegato (figura 5B), curve (Figura 5) o nastri anche rotti. Ciò può provocare a causa di errato taglio (leader e bordi d'uscita non esattamente paralleli) o adesivo non uniformemente applicato, ma anche da un campione asimmetrico o irregolarmente infiltrato. Particolarmente problematici sono i campioni che contengono componenti sia morbidi e molto difficile. I componenti di quest'ultimi possono essere difficili per infiltrarsi come la parete cellulare delle radici della pianta mostrato qui (Figura 5). In tal caso, pieghe (frecce) facilmente possono essere causati da compressione variabile e rilassamento durante il sezionamento. Per automatizzato di imaging nel SEM, curvi nastri non sono un grande problema, poiché il ROIs può essere ruotato per ospitare la curvatura della barra multifunzione.
Un altro passo fondamentale nel protocollo sta macchiando: lavaggio inadeguato può portare a residui sulla sezione (Figura 5E, F) e nel peggiore dei casi, coprire la zona più interessante (cerchio su una delle celle di due destinazione in Figura 5F). Inoltre, polvere (Figura 5, particelle fortemente light scattering) introdotto in barca il coltello, per esempio, con un vettore di substrato sporco, può causare seri problemi: nel FLM, la polvere può essere altamente fluorescente (cfr alcune fette in film supplementare S1) a tal punto che alcuni algoritmi di registrazione non funzionano. Tuttavia, la funzione di "allineare" in TrakEM17 in grado di gestire tali pile come dimostrato nel Supplementare film S1.
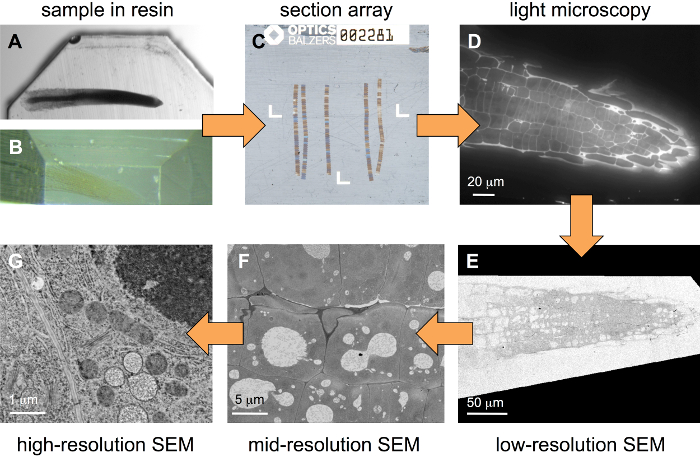
Figura 1: flusso di lavoro per l'imaging gerarchica correlativo. A partire da un campione incorporato in un blocco di resina (A, fortemente metallizzato campione), il campione è il primo profilato (B, campione debolmente metallizzato) e poi matrici composto da diversi nastri delle sezioni di serie (C), qui collocato su un Rivestite con ITO vetrino coprioggetti, sono prodotte con un ultramicrotomo. Dopo colorazione con un colorante fluorescente, pile di immagini vengono registrate in un grande campo FLM (D). Dopo ulteriore colorazione giri con metalli pesanti sali, gli stack sono imaging in un SEM (E–G) a diverse risoluzioni (dimensioni di pixel di immagine). Clicca qui per visualizzare una versione più grande di questa figura.

Figura 2: strumenti per la preparazione di matrici. Titolare del substrato assemblati da micromanipolatori con sette assi di movimento collegato a un ultramicrotomo standard (A): le viti, evidenziate con i cerchi, sono per il movimento orizzontale (2) e verticale (1) e per l'inclinazione (3) del vettore substrato . Il coltello di diamante jumbo con una barca oversize per ospitare grandi substrati (freccia), qui con un pezzo di wafer di silicio attivato al plasma montato su un supporto di alluminio dimensioni diapositiva (B). 20 gocce di µ l di acqua distillata collocato su un substrato di wafer di silicio non trattati (C) o su un substrato di plasma-attivato (D). Quattro nastri galleggianti sulla barca di coltello, collegato a un vetrino coprioggetti ITO-rivestito dalle loro estremità inferiore. Clicca qui per visualizzare una versione più grande di questa figura.
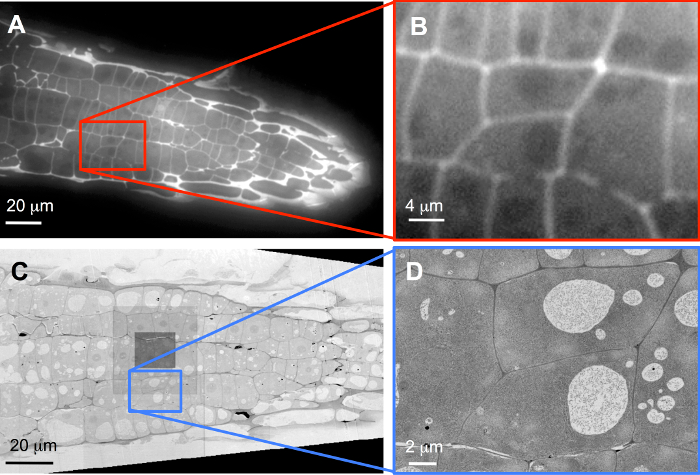
Figura 3: correlazione dei dati LM con i dati SEM. Panoramiche (A, C) e cellule bersaglio (B, D) registrarono con FLM (A, B) e SEM (C, D). (B) è uno zoom di software, e i dati originali sono stati registrati con una lente dell'obiettivo X 40 su un chip di 1.388 x 1.040 pixel fotocamera, mentre (C) viene registrato con 60 nm immagine dimensioni in pixel e (D) con 5 dimensioni di immagine pixel nm, che illustrano il vero aumento della risoluzione nel SEM. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 4: gerarchico di formazione immagine nel SEM utilizzando ZEISS Atlas 5 at Panoramica di una matrice registrata con 1.000 pixel di immagine nm utilizzando il rivelatore di SE (A). Sezione imposta con il ROI sul tessuto in ogni sezione e registrato con i pixel di immagine 60 nm (B). Sito imposta con un ROI seriale posizionato sulle cellule bersaglio e registrata con 5 pixel di immagine nm (C). Quando lo zoom in tali immagini ad alta risoluzione (D), compartimenti intracellulari membrana quali vacuoli (V), nucleo (N), mitocondri (M) e del reticolo endoplasmatico (frecce) diventano visibili. Clicca qui per visualizzare una versione più grande di questa figura.
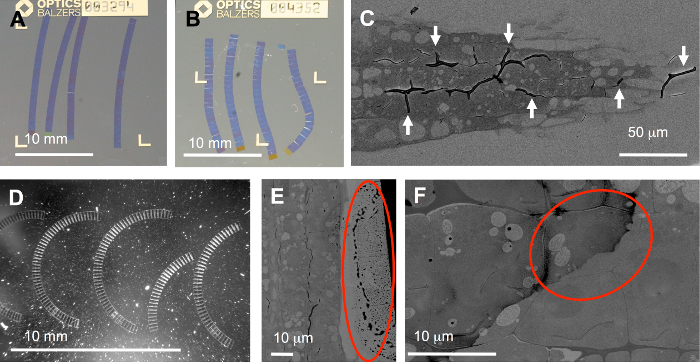
Figura 5: problemi tipici. 1. derivanti dal processo di sezionamento: nastri immessi sul lamelle rivestite con ITO sono idealmente dritto (A), ma irregolare compressione durante il sezionamento possono causare piegato (B) o nastri curvi (D) o anche pieghe (C). 2. causati da manipolazione substrato e nastri in acqua, per esempio, durante il sezionamento e la macchiatura: Light scattering di particelle sul substrato (D), cerchio di goccioline sulla sezione (cerchio in E), o sporcizia spalmato fuori del tessuto a causa di inadeguata lavaggio dopo la colorazione (F). Clicca qui per visualizzare una versione più grande di questa figura.
Supplementare film S1: serie di immagini FLM. 435 immagini allineate in Fiji16 utilizzando TrakEM17 e salvato come file di filmato (. avi). Per favore clicca qui per scaricare questo file.
Supplementare film S2: serie di immagini di SEM. 210 immagini allineate in Fiji16 utilizzando TrakEM17. La pila originale (300 immagini) di questo set di dati è stato 15 GB. Per ridurre le dimensioni dello stack da 3.3 GB (dopo l'allineamento e il ritaglio per solo due cellule bersaglio), che è stata ridimensionata in x e y di un fattore pari a 0.2 utilizzando Fiji e poi salvato come. avi film. Per favore clicca qui per scaricare questo file.
Supplementare film S3: zoom con livelli di risoluzione diversa nel SEM. Film creato ed esportato dal software Atlas 5 in formato. MP4. Per favore clicca qui per scaricare questo file.
Discussione
Un flusso di lavoro per il targeting celle specifiche all'interno di un tessuto da multi-modale gerarchici AT è stata dimostrata: un campione di resina-incastonato è tagliata a fette in matrici di sezioni di serie, che sono collocate su un substrato conduttivo utilizzando un supporto di substrato personalizzati. Dopo etichettatura con un fluoroforo e imaging nella FLM, il volume ricostruito viene utilizzato per la selezione delle cellule bersaglio. Dopo ulteriori macchiatura turni con metalli pesanti per introdurre il contrasto, questi obiettivi sono immaginati durante cento diverse sezioni a risoluzione nanometrica in un SEM utilizzando una piattaforma software automatizzato.
Per la produzione di matrici densamente imballate con parecchi lunghi nastri, titolare di un substrato simile a quello qui descritto è necessario. Una persona abile e paziente può essere in grado di collegare diversi nastri ad un substrato di silicio, semi immerso nella barca coltello e recuperare la matrice abbassando gradualmente il livello dell'acqua fino a quando i nastri sono seduti sul substrato. Tuttavia, nella nostra esperienza, c'è una tendenza a formazione in frantumi quando il substrato è toccare qualsiasi parte della barca coltello (cfr nota in 1.3.2 nel protocollo). Inoltre, questa procedura è molto più difficile con substrati rivestiti ITO: (1) grazie alla trasparenza del vetro di ITO, è difficile vedere il bordo dell'acqua dove le estremità dei nastri devono essere allegati; e (2) perché la superficie rivestita con ITO è molto più ruvida rispetto il wafer di silicio altamente lucidato, i nastri tendono a rompersi durante il sollevamento e frammenti più piccoli, composto da alcune sezioni possono galleggiare, distruggendo così l'ordine delle sezioni.
L'intero flusso di lavoro è anche fattibile senza correlazione ai dati FLM. In questo caso, raccolta di dati nel SEM potrebbe essere necessario essere eseguita in diverse sessioni. Una ricostruzione 3D iniziale o almeno valutazione dei dati di bassa o media risoluzione può essere necessario identificare bersagli. Inoltre, macchie istologiche convenzionali per campo chiaro LM (non richiedendo FLM) possono essere applicate. Naturalmente, altre opzioni6,7,8 sono anticorpo etichettatura su matrici, come già dimostrato nel libro iniziale a18, o geneticamente codificato proteine fluorescenti (XFPs) o pre-embedding Etichettatura con conservazione della fluorescenza durante la preparazione del campione.
Una limitazione generale del metodo discusso è l'uso di sezioni di un certo spessore e il campionamento discreto risultante del volume 3D: risoluzione in Z può essere solo buono come lo spessore delle sezioni poiché il SEM raccoglie solo i dati dalla superficie sezione (d epending sull'energia primaria energia/atterraggio selezionato). Ciò significa che il volume 3D risultante ha anisotropo voxel, ad es., 5 x 5 x 100 nm3 se 100 nm sezioni e una dimensione di pixel di immagine di 5 nm sono utilizzati. Per entità molto piccole in un intervallo di grandezza inferiore a 1 µm, questo potrebbe non essere sufficiente per una vera Descrizione ultrastrutturale. Una limitazione più tecnica è l'accuratezza della fase utilizzata nel SEM per automatizzato di imaging. Per questo motivo, è necessario scegliere un ROI maggiore le specifiche di accuratezza fase per garantire che l'area di destinazione completo è ripreso.
Rispetto al SBF-SEM e FIB-SEM come metodi di formazione immagine del blocco-viso, correlativo AT ha lo svantaggio definitivo di voxel anisotropo, come descritto sopra. Con FIB-SEM, voxel isotropo di 5 x 5 x 5 nm3 può essere ottenuto quando una correzione adeguata drift è a posto.
Lacune nel volume ricostruito a causa della perdita delle sezioni durante la preparazione di matrici potrebbero anche essere una preoccupazione che non si è verificata con SBF-SEM o FIB-SEM. Con la stabilizzazione di buon nastro di colla, questo di solito è solo un problema per l'ultima sezione di un nastro: potrebbe essere danneggiato quando liberandola dal filo del rasoio usando le ciglia. Tuttavia, nella nostra esperienza, la perdita di una sezione in ogni 20 – 50 sezioni non influenza la registratura delle immagini.
D'altra parte, la possibilità di post-macchia matrici conferisce buon segnale e contrasto per imaging di SEM, anche su campioni debolmente metallizzati come l'alta pressione congelato punte delle radici mostrati qui. Di conseguenza, non è necessario di compromettere la conservazione ottimale ultrastrutturale da numerosi fissazione chimica e passaggi di metallizzazione. Inoltre, routine campioni da laboratorio di patologia con gradi intermedi di metallizzazione consegnare dati eccellente10. Tale aumento di contrasto una Post-embedding non è in generale possibile per SBF-SEM e FIB-SEM. Inoltre, poiché questi metodi sono distruttivi, vale a dire, consumando il campione durante imaging, gerarchica imaging presso siti e diverse risoluzioni o formazione immagine ripetuta nei punti successivi nel tempo è impossibile. In linea di principio, volumi illimitati, composto da grande FOVs (ad es., fino a parecchi millimetri per cervelli di topo tutta in connectomics) creato da impunture mosaici, e un numero enorme di sezioni può essere acquistato da AT, mentre in FIB-SEM, FOVs oltre 100 µm x 100 µm sono difficili da raggiungere con strumenti di routine.
Ulteriore automazione del flusso di lavoro-AT descritto sarebbe un indubbio vantaggio, poiché i metodi suddetti SBF-SEM e FIB-SEM l'esecuzione sia di sezionamento e di imaging all'interno dello strumento stesso in modo completamente automatizzato. Esiste un tipo di automazione di sezionamento: The ATUMtome12 in grado di generare e raccogliere migliaia di sezioni, ma l'uso di nastro Kapton come un substrato rende tali matrici difficili all'immagine in un FLM. Il lamelle rivestite con ITO utilizzate qui, imaging anche super-risoluzione dovrebbe essere possibile. Un obiettivo ulteriore, molto desiderabile per automazione sarebbe la registrazione degli stack dei dati FLM. D'altra parte, automazione può essere costoso e fatta eccezione per il titolare di substrato, il flusso di lavoro qui presentato si basa (in termini di hardware) solo su strumentazione generalmente disponibile in una routine EM laboratorio o core struttura, rendendo il basso livello di accesso.
Divulgazioni
KIT ha ricevuto rimborsi da Boeckeler strumenti per la fornitura di un modello funzionale del proprietario del substrato. Marlene Thaler è dipendente della ZEISS microscopia GmbH, produttore di sistemi di microscopia citati in questo articolo. Inoltre, ZEISS offre alcune soluzioni come pacchetti di soluzione ZEISS Atlas per una vasta gamma di applicazioni a grande superficie, 3D imaging per strumenti SEM e FIB-SEM. Tutti gli altri autori non hanno nulla a rivelare.
Riconoscimenti
Questo lavoro è stato supportato da grant FKZ 13GW0044 dal Ministero federale per formazione e ricerca, progetto MorphiQuant-3D. Ringraziamo Carolin Bartels per il supporto tecnico.
Materiali
| Name | Company | Catalog Number | Comments |
| Instrumentation | |||
| Ultramicrotome | RMC | PT-PC | Alternative: Leica UC7 |
| Substrate holder | RMC | ASH-100 | Alternative: home built |
| Plasma cleaner | Diener | Zepto 40kHz | Alternatives: Ted Pella Pelco or other benchtop plasma cleaner Example Parameters for Diener Zepto with 40kHz generator (0-100W); 0.5 mbar, 5 sccm (Air), 10% performance |
| Widefield fluorescence light microscope | Zeiss | Axio Observer.Z1 | Alternatives: Leica, Nikon, Olympus |
| Fluorescence filter set | Zeiss | 43 HE (Cy3/DsRed) | |
| Objective lens | Zeiss | Zeiss Neofluar 40x | 0.75 NA |
| Decent workstation able to handle GB-sized image data | |||
| FESEM | Zeiss | Ultra 55 | Alternatives: FEI, Jeol, Hitachi, TESCAN |
| Name | Company | Catalog Number | Comments |
| Sectioning | |||
| Razor blades | Plano | T585-V | |
| Diamond knife for trimming 45° | Diatome | DTB45 | |
| Diamond knife for trimming 90° | Diatome | DTB90 | |
| Jumbo diamond knife for sectioning | Diatome | DUJ3530 | |
| Silicon wafer (pieces) | Si-Mat | Custom Made | Doping: P/Bor, orientation: <100>, thickness: 525 ± 25 µm, resistivity: 1-30 Ω-cm http://si-mat.com/silicon-wafers.html |
| ITO-coated coverslips | Balzers | Type Z | 22 × 22 × 0.17 mm https://www.opticsbalzers.com/de/produkte/deckglas-fenster/corrslide.html |
| Aluminium carrier | Custom Made | 76 × 26 mm | |
| Wafer forceps | Ideal-tek | 34A.SA | |
| Stubs forceps | Dumont | 0103-2E1/2-PO-1 | Dumoxel-H 2E 1/2 |
| Diamond scriber | Plano | T5448 | |
| Eyelash/very soft cat's hair | Selfmade | Alternative: Plano | |
| Brush | Selfmade | ||
| Pattex contact adhesive | Pattex | PCL3C | Kraftkleber Classic (the yellowish one) |
| Fixogum | Marabu | 290110001 | for fixing substrate to carrier |
| Adhesive tape | 3M | 851 | for fixing substrate to carrier |
| Isopropanol | Bernd Kraft | 07029.4000 | |
| Xylene | Carl Roth | 4436 | thinner for glue mixture |
| Rotihistol | Carl Roth | 6640 | alternative, limonene based thinner |
| Name | Company | Catalog Number | Comments |
| Software | |||
| Image processing | Open source | Fiji (http://fiji.sc/#download) | |
| Image acquisition | Zeiss | Atlas 5 AT (module for Zeiss SEM) | Alternative for automated image acquisition: WaferMapper: https://software.rc.fas.harvard.edu/lichtman/LGN/WaferMapper.html |
| Name | Company | Catalog Number | Comments |
| Staining | |||
| Propidiumiodide | Sigma-Aldrich | P4170 | Stock solution: 1.5 mM in 0.1 % sodium azide |
| Uranylacetate | Science Services | E22400 | |
| Lead(II) Nitrate | Merck | 107398 | |
| Tri Sodium Citrate Dihydrate | Merck | 106448 | |
| NaOH pellets | Merck | 106469 | |
| 1M NaOH solution | Bernd Kraft | 01030.3000 | |
| Glass petri dish | Duran | 23 755 56 | |
| Name | Company | Catalog Number | Comments |
| Mounting | |||
| Stubs | Plano | G301F | |
| Carbon pads | Plano | G3347 | |
| Copper tape | Plano | G3397 | double-sided adhesive, conductive |
| Silver paint | Plano | G3692 | Acheson Elektrodag 1415M |
| Name | Company | Catalog Number | Comments |
| Solutions/mixtures | |||
| Adhesive mixture for coating blocks | Pattex contact adhesive /xylene as thinner, ratio 1:3. (Alternative for xylene: Rotihistol) | ||
| Reynolds lead citrate | 50 mL: Dissolve 1.33 g of lead(II) nitrate in 10 mL of dH2O. Dissolve 1.76 g of tri-sodium citrate dihydrate in 10 ml dH2O. Mix both and add 1 M sodium hydroxide until the solution is clear. Fill up with dH2O to 50 mL. | ||
| Propidium iodide staining solution | Prepare 1:1500 dilution from stock in dH2O. Vortex for adequate mixing. | ||
| Aqueous uranyl acetate | Dissolve 3 % uranyl acetate in dH2O (mix thoroughly). |
Riferimenti
- Peddie, C. J., Collinson, L. M. Exploring the third dimension: Volume electron microscopy comes of age. Micron. 61, 9-19 (2014).
- Wanner, A. A., Kirschmann, M. A., Genoud, C. Challenges of microtome-based serial block-face scanning electron microscopy in neuroscience. J Microsc. 259 (2), 137-142 (2015).
- Kizilyaprak, C., Daraspe, J., Humbel, B. Focused ion beam scanning electron microscopy in biology. J Microsc. 254 (3), 109-114 (2014).
- Wacker, I., Schröder, R. R. Array tomography. J Microsc. 252 (2), 93-99 (2013).
- Tapia, J. C., Kasthuri, N., Hayworth, K. J., Schalek, R., Lichtman, J. W., Smith, S. J., Buchanan, J. High-contrast en bloc staining of neuronal tissue for field emission scanning electron microscopy. Nat protoc. 7 (2), 193-206 (2012).
- Lucas, M. S., Günthert, M., Gasser, P., Lucas, F., Wepf, R. Bridging Microscopes: 3D Correlative Light and Scanning Electron Microscopy of Complex Biological Structures. Methods Cell Biol. 111, 325-356 (2012).
- De Boer, P., Hoogenboom, J. P., Giepmans, B. N. Correlated light and electron microscopy: ultrasructure lights up!. Nat Methods. 12 (6), 503-513 (2015).
- Verkade, P., Müller-Reichert, T. . Methods in Cell Biology Correlative Light and Electron Microscopy III. 140, 1-352 (2017).
- Gibson, K. H., Vorkel, D., Meissner, J., Verbavatz, J. -. M. Fluorescing the Electron: Strategies in Correlative Experimental Design. Methods Cell Biol. 124, 23-54 (2014).
- Wacker, I., Schröder, R. R., Schroeder, J. A. Pathology goes 3D: Exploring the potential of array tomography versus FIB nanotomography for a CADASIL sample. Ultrastruct Pathol. 41 (1), 114-115 (2017).
- Wacker, I., Spomer, W., Hofmann, A., Thaler, M., Hillmer, S., Gengenbach, U., Schröder, R. R. Hierarchical imaging: a new concept for targeted imaging of large volumes from cells to tissues. BMC Cell Biol. 17 (1), 38 (2016).
- Hayworth, K. J., Morgan, J. L., Schalek, R., Berger, D. R., Hildebrand, D. G. C., Lichtman, J. W. Imaging ATUM ultrathin section libraries with WaferMapper: a multi-scale approach to EM reconstruction of neural circuits. Front Neural Circuits. 8, (2014).
- Micheva, K. D., O'Rourke, N., Busse, B., Smith, S. J. Array Tomography: Production of Arrays. Cold Spring Harb Protoc. 11, 1267-1269 (2010).
- Fahrenbach, W. H. Continuous serial thin sectioning for electron microscopy. J. Elec. Microsc. Tech. 1 (4), 387-398 (1984).
- Harris, K. M., et al. Uniform serial sectioning for transmission electron microscopy. J Neurosci. 26 (47), 12101-12103 (2006).
- Schindelin, J., Arganda-Carreras, I., Frise, E., Kaynig, V., Longair, M., Pietzsch, T., Preibisch, S., Rueden, C., Saalfeld, S., Schmid, B., Tinevez, J. -. Y., White, D. J., Hartenstein, V., Eliceiri, K., Tomancak, P., Cardona, A. Fiji: an open-source platform for biological-image analysis. Nat Methods. 9 (7), 676-682 (2012).
- Cardona, A., Saalfeld, S., Schindelin, J., Arganda-Carreras, I., Preibisch, S., Longair, M., Tomancak, P., Hartenstein, V., Douglas, R. J. TrakEM2 software for neural circuit reconstruction. PLoS ONE. 7 (6), e38011 (2012).
- Micheva, K. D., Smith, S. J. Array Tomography: A new tool for imaging the molecular architecture and ultrastructure of neural circuits. Neuron. 55 (1), 25-36 (2007).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon