Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Espectroscopía de neutrones de alta resolución para estudiar la dinámica de picosegundos-nanosegundos de proteínas y agua de hidratación
En este artículo
Resumen
La espectroscopia de retrodispersión de neutrones ofrece un acceso no destructivo y sin etiquetas a la dinámica ps-ns de las proteínas y su agua de hidratación. El flujo de trabajo se presenta con dos estudios sobre proteínas amiloides: sobre la dinámica resuelta en el tiempo de la lisozima durante la agregación y sobre la dinámica del agua de hidratación de tau en la formación de fibra.
Resumen
La dispersión de neutrones ofrece la posibilidad de sondear la dinámica dentro de las muestras para una amplia gama de energías de una manera no destructiva y sin etiquetar más que el deuterio. En particular, la espectroscopia de retrodispersión de neutrones registra las señales de dispersión en múltiples ángulos de dispersión simultáneamente y es muy adecuada para estudiar la dinámica de los sistemas biológicos en la escala de tiempo ps-ns. Al emplear D2O, y posiblemente componentes tampón deuterados, el método permite monitorear tanto la difusión del centro de masa como los movimientos de la columna vertebral y la cadena lateral (dinámica interna) de las proteínas en estado líquido.
Además, la dinámica del agua de hidratación se puede estudiar empleando polvos de proteínas perdeuteradas hidratadas conH2O. Este artículo presenta el flujo de trabajo empleado en el instrumento IN16B en el Institut Laue-Langevin (ILL) para investigar la dinámica del agua de proteínas e hidratación. Se explica la preparación de muestras de solución y muestras de proteína hidratada en polvo mediante intercambio de vapor. El procedimiento de análisis de datos para la dinámica del agua de proteínas e hidratación se describe para diferentes tipos de conjuntos de datos (espectros cuasielásticos o escaneos de ventana fija) que se pueden obtener en un espectrómetro de retrodispersión de neutrones.
El método se ilustra con dos estudios que involucran proteínas amiloides. Se muestra que la agregación de lisozima en agregados esféricos de tamaño μm -partículas denotadas- ocurre en un proceso de un solo paso en el rango de espacio y tiempo sondeado en IN16B, mientras que la dinámica interna permanece sin cambios. Además, se estudió la dinámica del agua de hidratación de tau en polvos hidratados de proteína perdeuterada. Se muestra que los movimientos de traslación del agua se activan tras la formación de fibras amiloides. Finalmente, se discuten los pasos críticos en el protocolo sobre cómo se posiciona la dispersión de neutrones con respecto al estudio de la dinámica con respecto a otros métodos biofísicos experimentales.
Introducción
El neutrón es una partícula masiva y sin carga que se ha utilizado con éxito a lo largo de los años para sondear muestras en diversos campos, desde la física fundamental hasta la biología1. Para aplicaciones biológicas, la dispersión de neutrones de ángulo pequeño, la dispersión inelástica de neutrones y la cristalografía y reflectometría de neutrones se utilizan ampliamente 2,3,4. La dispersión inelástica de neutrones proporciona una medición promediada por conjuntos de la dinámica sin requerir un etiquetado específico per se, y una calidad de señal que no depende del tamaño o la proteína5. La medición se puede hacer utilizando un ambiente altamente complejo para la proteína en estudio que imita el medio intracelular, como un lisado bacteriano deuterado o incluso in vivo 3,6,7. Se pueden usar diferentes configuraciones experimentales para estudiar la dinámica, a saber, i) acceso de tiempo de vuelo a la dinámica sub-ps-ps, ii) acceso de retrodispersión a la dinámica ps-ns, y iii) acceso de spin-echo a la dinámica de ns a cientos de ns. La retrodispersión de neutrones hace uso de la ley de Bragg 2d sinθ = nλ, donde d es la distancia entre planos en un cristal, θ el ángulo de dispersión, n el orden de dispersión y λ la longitud de onda. El uso de cristales para la retrodispersión hacia los detectores permite lograr una alta resolución en energía, típicamente ~ 0.8 μeV. Para medir el intercambio de energía, se utiliza una unidad Doppler que lleva un cristal en retrodispersión para definir y ajustar la longitud de onda de neutrones entrantes 8,9,10 (Figura 1), o se puede usar una configuración de tiempo de vuelo a costa de una disminución en la resolución de energía 11.
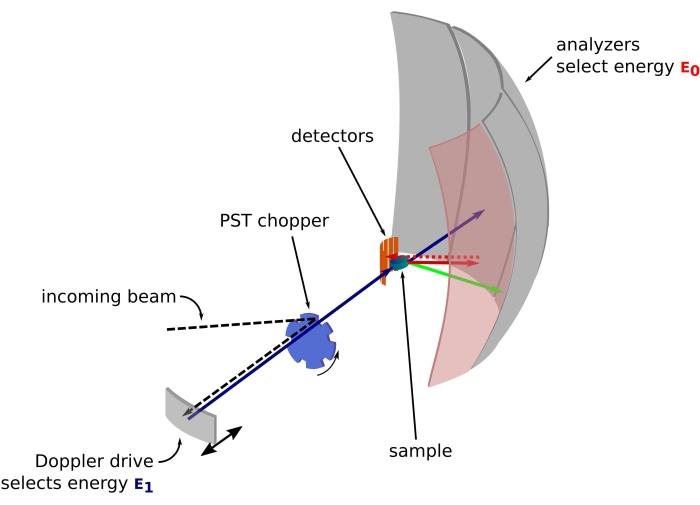
Figura 1: Croquis de un espectrómetro de retrodispersión de neutrones con una unidad Doppler. El haz entrante golpea el helicóptero de transformación del espacio de fase (PST)42, lo que aumenta el flujo en la posición de la muestra. Luego se retrodispersa hacia la muestra mediante la unidad Doppler, que selecciona una energía E1 (flecha cian). Los neutrones son dispersados por la muestra (con diferentes energías representadas por el color de las flechas) y los analizadores, hechos de cristales de Si 111, solo dispersarán neutrones con una energía específica E0 (flechas de color rojo aquí). Por lo tanto, la transferencia de momento q se obtiene de la posición detectada del neutrón en la matriz del detector, y la transferencia de energía se obtiene de la diferencia E1- E0. El tiempo de vuelo esperado para el pulso de neutrones producido por el PST se utiliza para descartar la señal de los neutrones dispersos directamente hacia los tubos detectores. Abreviatura: PST = transformación del espacio de fase. Haga clic aquí para ver una versión más grande de esta figura.
Para la espectroscopia de retrodispersión, la principal contribución a la señal de muestras ricas en protones de hidrógeno, como las proteínas, proviene de la dispersión incoherente, para la cual la intensidad de dispersión Sinc(q, ω) se muestra mediante Eq (1)12
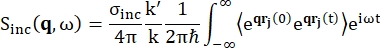 (1)
(1)
Donde σinc es la sección transversal incoherente del elemento considerado, k' es la norma del vector de onda disperso, k la norma del vector de onda entrante, q (= k - k') la transferencia de momento, r j(t) el vector de posición del átomo j en el tiempo t, y ω la frecuencia correspondiente a la transferencia de energía entre el neutrón entrante y el sistema. Los corchetes angulares denotan el promedio del conjunto. Por lo tanto, la dispersión incoherente sondea la autocorrelación de partículas individuales promediada por conjuntos de las posiciones de los átomos con el tiempo y da la autodinámica promediada sobre todos los átomos en el sistema y diferentes orígenes de tiempo (promedio de conjunto). La función de dispersión es la transformada de Fourier en el tiempo de la función de dispersión intermedia I(q, t), que puede ser vista como la transformada de Fourier en el espacio de la función de correlación de van Hove mostrada por Eq (2):
 (2)
(2)
Donde ρ(r,t) es la densidad de probabilidad de encontrar un átomo en la posición r y el tiempo t 13.
Para un proceso de difusión fickiano, la función de autodifusión resulta (ver Eq (3)) después de una doble transformada de Fourier en una función de dispersión que consiste en un lorentziano de ancho de línea dado por γ = Dq2.
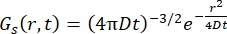 (3)
(3)
Se desarrollaron modelos más sofisticados que se encontraron útiles, como el modelo de difusión de salto de Singwi y Sjölander para la dinámica interna de proteínas ps-ns14 o el modelo de rotación de Sears para el agua de hidratación15,16,17.
En el instrumento de retrodispersión de neutrones (NBS) IN16B8,9 en el ILL, Grenoble, Francia (Figura suplementaria S1), una configuración comúnmente utilizada con proteínas consiste en cristales de Si 111 para los analizadores con un accionamiento Doppler para ajustar la longitud de onda entrante (Figura suplementaria S2A), dando así acceso al rango de transferencia de momento ~0.2 Å-1 < q < ~2 Å-1 y rango de transferencia de energía de -30 μeV < 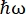 < 30 μeV- correspondiente a escalas de tiempo que van desde unos pocos ps a unos pocos ns y distancias de unos pocos Å. Además, IN16B ofrece la posibilidad de realizar escaneos elásticos e inelásticos de ventana fija (E/IFWS)10, que incluyen la adquisición de datos en una transferencia de energía fija. Como el flujo es limitado cuando se trabaja con neutrones, E/IFWS permite maximizar el flujo para una transferencia de energía, reduciendo así el tiempo de adquisición necesario para obtener una relación señal-ruido satisfactoria. Una opción más reciente es el modo11 del espectrómetro de retrodispersión y tiempo de vuelo (BATS), que permite la medición de una amplia gama de transferencias de energía (por ejemplo, -150 μeV < <
< 30 μeV- correspondiente a escalas de tiempo que van desde unos pocos ps a unos pocos ns y distancias de unos pocos Å. Además, IN16B ofrece la posibilidad de realizar escaneos elásticos e inelásticos de ventana fija (E/IFWS)10, que incluyen la adquisición de datos en una transferencia de energía fija. Como el flujo es limitado cuando se trabaja con neutrones, E/IFWS permite maximizar el flujo para una transferencia de energía, reduciendo así el tiempo de adquisición necesario para obtener una relación señal-ruido satisfactoria. Una opción más reciente es el modo11 del espectrómetro de retrodispersión y tiempo de vuelo (BATS), que permite la medición de una amplia gama de transferencias de energía (por ejemplo, -150 μeV < < 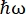 150 μeV), con un flujo más alto que con el accionamiento Doppler, pero a costa de una resolución de energía más baja (Figura suplementaria S2B).
150 μeV), con un flujo más alto que con el accionamiento Doppler, pero a costa de una resolución de energía más baja (Figura suplementaria S2B).
Una propiedad importante de la dispersión de neutrones es que la sección transversal incoherente σinc tiene un valor 40 veces mayor para el hidrógeno que para el deuterio y es insignificante para otros elementos que se encuentran comúnmente en muestras biológicas. Por lo tanto, la dinámica de las proteínas en un ambiente líquido se puede estudiar mediante el uso de un tampón deuterado, y el estado del polvo permite el estudio de la dinámica interna de la proteína con proteína en polvo hidrogenada hidratada con D2O, o el estudio del agua de hidratación para la proteína perdeuterada en polvo hidratada conH2O. En estado líquido, la retrodispersión de neutrones permite típicamente el acceso simultáneo a la autodifusión del centro de masa de las proteínas (difusión de tipo fickiano) y su dinámica interna. Estos últimos son movimientos de columna vertebral y de cadena lateral generalmente descritos por el llamado modelo de difusión de salto u otros 3,18. En los polvos de proteína hidrogenada, la difusión de proteínas está ausente y solo se necesita modelar la dinámica interna. Para el agua de hidratación, las contribuciones de los movimientos de traslación y rotación de las moléculas de agua presentan una dependencia diferente de la transferencia de momento q, lo que permite su distinción en el proceso de análisis de datos17.
Este artículo ilustra el método de retrodispersión de neutrones con el estudio de proteínas que se encontraron capaces de desplegarse, agregarse en una forma canónica que consiste en pilas de β hebras -el llamado patrón de β cruzado19,20- y formar fibras alargadas. Se trata de la llamada agregación amiloide, ampliamente estudiada debido a su papel central en trastornos neurodegenerativos como el Alzheimer o el Parkinson21,22. El estudio de las proteínas amiloides también está motivado por el papel funcional que pueden desempeñar 23,24 o su alto potencial para el desarrollo de nuevos biomateriales25. Los determinantes fisicoquímicos de la agregación amiloide siguen sin estar claros, y no se dispone de una teoría general de la agregación amiloide, a pesar del tremendo progreso durante los últimos años21,26.
La agregación amiloide implica cambios en la estructura y estabilidad de las proteínas con el tiempo, cuyo estudio implica naturalmente dinámica, vinculada a la estabilidad de la conformación de proteínas, la función de las proteínas y el paisaje de energía de las proteínas27. La dinámica está directamente relacionada con la estabilidad de un estado específico a través de la contribución entrópica para los movimientos más rápidos28, y la función de la proteína puede ser sostenida por movimientos en varias escalas de tiempo desde sub-ps para proteínas sensibles a la luz29 hasta ms para movimientos de dominio, que pueden ser facilitados por la dinámica de picosegundos-nanosegundos30.
Se presentarán dos ejemplos de uso de la espectroscopia de retrodispersión de neutrones para estudiar proteínas amiloides, uno en estado líquido para estudiar la dinámica de proteínas y otro en estado de polvo hidratado para estudiar la dinámica del agua de hidratación. El primer ejemplo se refiere a la agregación de lisozima en esferas de tamaño μm (llamadas partículas) seguidas en tiempo real5, y el segundo a una comparación de la dinámica del agua en estados nativos y agregados de la proteína humana tau31.
La lisozima es una enzima implicada en la defensa inmune y está compuesta por 129 residuos de aminoácidos. La lisozima puede formar partículas en tampón deuterado a pD de 10,5 y a una temperatura de 90 °C. Con la dispersión de neutrones, demostramos que la evolución temporal del coeficiente de difusión del centro de masa de la lisozima sigue la cinética exponencial única de la fluorescencia de tioflavina T (una sonda fluorescente utilizada para monitorear la formación de patrones de β cruzada amiloide32), lo que indica que la formación de superestructuras particuladas y patrones de β cruzada ocurren en un solo paso con la misma velocidad. Además, la dinámica interna se mantuvo constante durante todo el proceso de agregación, lo que puede explicarse por un cambio conformacional rápido que no se puede observar en los instrumentos NBS, o por la ausencia de un cambio significativo en la energía interna de las proteínas tras la agregación.
La proteína humana tau es una proteína intrínsecamente desordenada (IDP) que consta de 441 aminoácidos para la llamada isoforma 2N4R, que está notablemente involucrada en la enfermedad de Alzheimer33. Usando retrodispersión de neutrones en polvos de proteína tau perdeuterada, demostramos que la dinámica del agua de hidratación aumenta en el estado de fibra, con una mayor población de moléculas de agua sometidas a movimientos de traslación. El resultado sugiere que un aumento en la entropía del agua de hidratación podría conducir a la fibrilación amiloide de tau.
Protocolo
1. Preparar el tampón deuterado para proteínas en estado líquido
- Disolver todos los componentes del tampón enD2Opuro.
- Si el electrodo de pH se calibró enH2O, ajuste el pD de acuerdo con la fórmula pD = pH + 0.4 usando NaOD o DCl34.
NOTA: El uso de D2O en lugar deH2Opodría afectar la solubilidad de las proteínas y las condiciones tampón podrían necesitar ser adaptadas (por ejemplo, por un ligero cambio en la concentración de sal).
2. Preparar los polvos hidratados H2O-hidratados de proteína perdeuterada
- Prepare el portamuestras.
- Limpie a fondo un portamuestras plano de aluminio con su sello de alambre de indio y tornillos con agua y etanol y déjelo secar.
NOTA: Se utiliza un portamuestras plano de tal manera que el polvo se pueda distribuir homogéneamente sobre la superficie. La cantidad de polvo debe ser suficiente para que pueda mantenerse entre las paredes y no caiga cuando el portamuestras se coloca verticalmente. - Pese las diferentes partes del soporte de la muestra (fondo, tapa y alambre de indio) por separado en una balanza de precisión.
- Coloque el sello de alambre de indio de 1 mm en la ranura de la parte inferior del portamuestras, dejando una pequeña superposición donde se unen los dos extremos (Figura 2A).
- Coloque una cantidad adecuada de proteína liofilizada (típicamente ~ 100 mg de proteína) de modo que llene la superficie interna de la parte inferior del portamuestras.
- Limpie a fondo un portamuestras plano de aluminio con su sello de alambre de indio y tornillos con agua y etanol y déjelo secar.
- Hidrata la proteína en polvo.
- Coloque el portamuestras en un desecador con una placa de Petri que contenga polvo deP2O5durante 24 h para secar completamente la proteína en polvo35 (Figura 2B). Pesar la parte inferior seca del portamuestras que contiene el sello de indio y el polvo seco para obtener mseco.
PRECAUCIÓN: El polvo de P2O5 es muy corrosivo. - Retire elP2O5del desecador y coloque una placa de Petri conD2Odentro. Controle la masa del polvo regularmente para verificar el nivel de hidratación h = m hyd / m seco donde mhyd y m seco son la masa del polvo hidratado y el polvo seco, respectivamente.
NOTA: Para proteínas altamente hidrófobas como la insulina, puede ser necesario aumentar la temperatura dentro del desecador para obtener una presión de vapor más alta y alcanzar el nivel de hidratación h deseado. - Repita los pasos 2.2.1 y 2.2.2 al menos tres veces para convertir correctamente todos los hidrógenos intercambiables en deuterón.
NOTA: Alternativamente, los ciclos de liofilización y disolución en D2O puro podrían usarse para un mejor intercambio de H / D siempre que la proteína no se vea afectada por ella. - Hidrate el polvo ligeramente por encima del nivel deseado, deje que la parte inferior del portamuestras con el alambre de indio y el polvo hidratado permanezca en la balanza de precisión, y espere a que la masa disminuya lentamente al valor deseado para obtener el objetivo h (típicamente 0.2-0.4 si una proteína globular de tamaño mediano debe cubrirse con una capa de hidratación completa).
- Coloque rápidamente la tapa en la parte inferior y cierre primero el portamuestras con cuatro tornillos para detener el intercambio de vapor (Figura suplementaria S3A).
- Coloque y apriete todos los tornillos restantes hasta que no se vea ningún espacio entre la parte inferior y la tapa (Figura suplementaria S3B).
- Pese el portamuestras sellado para comprobar si hay una posible pérdida de hidratación por fugas después del experimento con neutrones.
- Coloque el portamuestras en un desecador con una placa de Petri que contenga polvo deP2O5durante 24 h para secar completamente la proteína en polvo35 (Figura 2B). Pesar la parte inferior seca del portamuestras que contiene el sello de indio y el polvo seco para obtener mseco.
3. Realizar el experimento de dispersión de neutrones incoherente
- Discuta y verifique la configuración del instrumento necesario para el experimento con el contacto local algunas semanas antes del tiempo de haz asignado.
- Prepare la muestra en estado líquido.
- Disuelva la proteína en el tampón deuterado.
- Determinar el volumen apropiado de líquido que se colocará en el portamuestras con agua (asegúrese de que no haya desbordamiento cuando el portamuestras esté cerrado; Figura 2C).
NOTA: Los pasos siguientes (3.3 y 3.4) describen un experimento realizado en el espectrómetro NBS IN16B en el ILL8,9, utilizando un criohorno como entorno de muestra. El sistema de control de instrumentos cambiará de un instrumento a otro, pero los principios de funcionamiento siguen siendo los mismos.
- Inserte la muestra.
- Seque bien la varilla de muestra (Figura 2D) y retire la muestra anterior, si la hubiera, después de comprobar que la dosis de radiación ionizante es inferior a 100 μSv/h antes de manipular cualquier material (en el ILL).
- Coloque la muestra, verifique el centrado adecuado en relación con el centro del haz (Figura suplementaria S4) e inserte la barra de muestra en el criohorno (Figura 2D). Encienda la bomba de vacío para alcanzar menos de 10-3 bar y elimine el aire dentro del criohorno repitiendo las siguientes tres veces: llene el criohorno con gas helio hasta que se alcance la presión atmosférica y retire el gas nuevamente usando la bomba de vacío.
NOTA: En el caso de un portamuestras plano, el portamuestras debe estar orientado en un ángulo de 45° con respecto al haz entrante. El rango de transferencia de momento útil podría reducirse debido a la absorción y dispersión por parte de la célula. Se puede usar un absorbedor de neutrones fuerte como el cadmio para enmascarar ciertas partes del soporte de la muestra (por ejemplo, tornillos, partes gruesas). - Introducir un poco de gas helio en el criohorno de tal manera que la presión sea ~0,05 bar.
- Adquirir datos (por ejemplo, usando NOMAD en IN16B en el ILL, se supone que el usuario prefiere una temperatura de 200 K antes de adquirir un espectro de neutrones cuasielástico (QENS), luego E/IFWS durante una rampa de temperatura a 310 K a 0.5 K por minuto y finalmente un QENS a 310 K).
- Con NOMAD, en la pestaña de ejecución, arrastre y suelte un controlador FurnaceCryostat en el Launch Pad. Ajuste la temperatura a 200 K. Utilice el modo rápido y un tiempo de espera de 30 minutos para que la temperatura tenga tiempo de estabilizarse. Haga clic en el icono de flechas giratorias para ejecutarlo en segundo plano, de modo que los datos se puedan adquirir durante la disminución de la temperatura.
- Arrastre y suelte el controlador IN16DopplerSettings , establezca el perfil de velocidad en Velocidad precisa establecida por Max ΔE, un valor de 0.00 μeV y 128 canales para obtener una configuración EFWS.
- Arrastre y suelte un controlador de recuento , rellene el campo Subtítulo con un nombre que permita una fácil identificación de los datos y establezca 60 repeticiones de escaneos de 30 s (Figura suplementaria S5A).
- Arrastre y suelte un controlador IN16DopplerSettings , establezca el perfil de velocidad en Sine establecido por Speed con un valor de 4.5 m / s y 2,048 canales para obtener una configuración QENS.
- Arrastre y suelte un controlador de conteo con 4 repeticiones de escaneos de 30 minutos (Figura suplementaria S5B).
- Para la rampa de temperatura, arrastre y suelte un controlador FurnaceCryosta , ajuste la temperatura a 310 K, establezca Rampa en SetPoint con Δ = 0.05 K y 6 s. Use un tiempo de 220 minutos (Figura suplementaria S6A).
- Usa un bucle for con 65 repeticiones. En el interior, inserte un controlador IN16DopplerSettings como en el paso 3.4.2, seguido de un solo recuento de 30 s. Posteriormente, inserte IN16DopplerSettings, como se describió anteriormente, pero utilizando un desplazamiento de energía de 1,5 μeV y 1.024 canales seguido de un recuento único de 3 min (Figura suplementaria S6B).
- Para adquirir el último QENS a 310 K, arrastre y suelte los controladores IN16DopplerSettings y Count configurados como se describe en los pasos 3.4.4 y 3.4.5, respectivamente.
- Presione el botón de inicio (triángulo derecho en la parte inferior de la ventana) para ejecutar el script.
NOTA: Cada experimento requerirá la adquisición de datos de calibración; es decir, la celda vacía para las correcciones de resta o absorción, el tampón solo a las diferentes temperaturas utilizadas para modelar el fondo, y una medición de vanadio (o equivalentemente, la muestra a una temperatura de 10 K o inferior) para obtener la función de resolución del instrumento.
4. Análisis de datos - QENS
- Importe el conjunto de datos utilizando el método 'IN16B_QENS.process()' en el software Python nPDyn v3.x36
>>>> desde nPDyn.dataParsers importar IN16B_QENS
>>> muestra = IN16B_QENS(
...
... [detGroup=... formato>]
... ). proceso()
>>> muestra = sample.get_q_range(0.3, 1.8) - Realice correcciones de datos (opcional) con los siguientes comandos (consulte la documentación de nPDyn para obtener más información, figura 3):
#it se supone que los datos de celda vacía, vanadio y tampón
# ya se importaron en un conjunto de datos llamado 'empty_cell', 'vanadio',
# y 'buffer', respectivamente.
# para restar celdas vacías con un factor de escala
# (los errores se propagan automáticamente)
>>> muestra = muestra - 0,95 * empty_cell
# para corrección usando el coeficiente Paalman-Ping
# (mutuamente excluyente con el ejemplo anterior)
>>> sample = sample.absorptionCorrection(empty_cell)
# para normalización
>>> muestra = muestra.normalizar(vanadio)
# para agrupar a lo largo del eje observable
# observable es el tiempo de agregación aquí
>>> muestra = muestra.bin(3, eje=0) - Ajuste los datos de calibración. El conjunto de datos (muestras, celda vacía, tampón deuterado (si es necesario) y vanadio) se puede ajustar utilizando modelos integrados o un modelo definido por el usuario (consulte la documentación de nPDyn):
>>> desde nPDyn.models.builtins import (
... modeloPVoigt,
... modelWater,
... modeloCalibratedD2O,
... )
# Los modelos incorporados utilizan un vector columna del momento
# transferir valores q
>>> q = vanadio.q[:, Ninguno]
# el vanadio se ajusta utilizando un perfil pseudo-Voigt
>>> vanadium.fit(modeloPVoigt(q)) - Utilice el modelo incorporado para el agua de hidratación llamado 'modelWater'. Este modelo se lee como se muestra en Eq (4)17
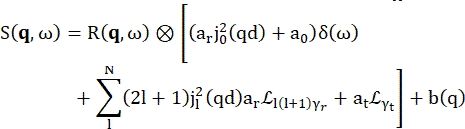 (4)
(4)
Donde un0, unar y unat son escalares que representan la contribución relativa de la señal elástica, los movimientos de rotación y los movimientos de traslación, respectivamente; j1(qd) es la función de Bessel esféricade orden l , siendo q la transferencia de momento; d la distancia O-H en la molécula de agua; δ(ω) es el delta de Dirac, que se multiplica por la EISF aquí; N es el orden más alto de la función de Bessel esférica utilizada (típicamente ~5);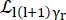 y
y 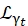 son los movimientos rotacionales y traslacionales lorentzianos, respectivamente; b(q) es un término de fondo plano. Las funciones esféricas de Bessel dan la contribución relativa de cada estado de momento angular de las moléculas de agua, y el número N se determina en función del rango q de transferencia de momento. En el caso de un espectrómetro NBS típico, los términos hasta N = 4 explican casi por completo la señal (Figura suplementaria S7).
son los movimientos rotacionales y traslacionales lorentzianos, respectivamente; b(q) es un término de fondo plano. Las funciones esféricas de Bessel dan la contribución relativa de cada estado de momento angular de las moléculas de agua, y el número N se determina en función del rango q de transferencia de momento. En el caso de un espectrómetro NBS típico, los términos hasta N = 4 explican casi por completo la señal (Figura suplementaria S7).
# Aquí, la ecuación 2 se usa para el agua de hidratación
# Convolución con función de resolución y adición de
# El fondo D2O se realiza automáticamente con el botón
# Argumentos proporcionados
>>> sample.fit(modelWater(q),
... res=vanadio,
... bkgd=buffer,
... volume_fraction_bkgd=0,95
... )
NOTA: Las contribuciones de los movimientos rotacionales y de traslación deben ser complicadas para ser perfectamente rigurosas. El éxito de un modelo aditivo debe atribuirse a la presencia de distintas poblaciones de agua en la superficie de la proteína y al limitado rango de energía accesible. - Utilice lo siguiente para trazar los datos (figura 4):
>>> de nPDyn.plot import plot
>>> parcela(muestra)
5. Análisis de datos: rampa de temperatura, escaneos elásticos de ventana fija (EFWS)
- Utilice un procedimiento similar a la sección 4 para normalizar los datos de rampa de temperatura mediante la señal a la temperatura más baja (normalmente 10 K):
>>> desde nPDyn.dataParsers importar IN16B_FWS
>>> muestra = IN16B_FWS(
... ,
... detGroup=[detGroup=]
... ). proceso()
# normalización con los 5 primeros puntos en el observable
# eje, que corresponde a la temperatura
>>> muestra /= muestra[:5].mean(0)
# El rango Q de transferencia de momento utilizado aquí es más pequeño
# ya que el modelo utilizado es válido solo para q bajo
>>> muestra = sample.get_q_range(0.2, 0.8) - Utilice un modelo gaussiano simple para comenzar, cuyo ancho está dado por el llamado desplazamiento cuadrático medio (MSD). Compile y ajuste el modelo mediante los siguientes comandos:
>>> importar numpy como np
>>> desde nPDyn.models importar parámetros, modelo, componente
# a es un factor de escala
>>> params = Parámetros(
... a={'value': 1, 'bounds': (0, np.inf)},
... msd={'value': 1, 'bounds': (0, np.inf)}
... )
>>> model = Modelo(parámetros)
>>> model.addComponent(Component(
... «gaussiano»,
... lambda x, a, msd: a * np.exp(-x ** 2 * msd / 6)
... ))
>>> sample.fit(model, x=sample.q[:, None])
>>> parcela(muestra)
NOTA: La aproximación gaussiana siempre es válida para q2MSD << 1, pero se puede usar un rango de transferencia de momento más amplio para la comparación relativa entre muestras. Se han desarrollado modelos más sofisticados, que van más allá de la aproximación gaussiana37,38,39.
6. Análisis de datos: escaneos de ventana fija elásticos e inelásticos (E/IFWS)
- Similar al paso 4, importe el conjunto de datos pero usando la clase 'IN16B_FWS':
>>> desde nPDyn.dataParsers importar IN16B_FWS
>>> muestra = IN16B_FWS(
...
... [detGroup=]
... ). proceso()
>>> muestra = sample.get_q_range(0.3, 1.8) - Ajuste los datos de calibración y los datos de muestra.
- Analice los datos E/IFWS utilizando un MSD40 generalizado o considerándolos como espectros QENS gruesos (que tienen solo unos pocos puntos de datos en el eje de energía). Cuando E/IFWS se considera QENS grueso, los modelos utilizados para QENS se utilizan para ajustar todo el conjunto de datos E/IFWS a la vez (ajuste global de transferencias de energía y transferencias de impulso).
NOTA: La última solución, que utiliza modelos para QENS en datos E/IFWS, se utiliza aquí donde se impone la dependencia de transferencia de momento de la difusión del centro de masa y la dinámica interna de las proteínas. - Modele la dinámica de proteínas en líquidos utilizando el siguiente Eq (5) ('modelProteinJumpDiff' en nPDyn):
 (5)
(5)
Donde R(q,ω) es la función de resolución; β un escalar independiente para cada transferencia de momento q; a0 es el factor de estructura incoherente elástica (EISF);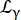 un lorentziano que explica la difusión del centro de masa con una anchura dada por Eq (6);
un lorentziano que explica la difusión del centro de masa con una anchura dada por Eq (6); 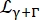 es un lorentziano que incluye la difusión del centro de masa y una contribución siguiendo el modelo de difusión de salto14 que tiene en cuenta la dinámica interna (Eq (7);
es un lorentziano que incluye la difusión del centro de masa y una contribución siguiendo el modelo de difusión de salto14 que tiene en cuenta la dinámica interna (Eq (7);  siendo la señal ajustada de D2O reescalada por su fracción de volumen en la muestra.
siendo la señal ajustada de D2O reescalada por su fracción de volumen en la muestra.
γ = Dsq2 (6)
Ds es el coeficiente de autodifusión.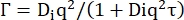 (7)
(7)
Di es el coeficiente de difusión aparente para la dinámica interna y τ un tiempo de relajación para los movimientos difusivos.
>>> desde nPDyn.models.builtins import (
... modeloPVoigt,
... modelProteinJumpDiff,
... modeloCalibratedD2O,
... )
# Los modelos incorporados utilizan un vector columna del momento
# transferir valores q
>>> q = vanadio.q[:, Ninguno]
# el vanadio se ajusta utilizando un perfil pseudo-Voigt
>>> vanadium.fit(modeloPVoigt(q))
# para D2O puro, un modelo con ancho de línea calibrado
# para diferentes temperaturas está incluido en nPDyn
>>> buffer.fit(modelCalibratedD2O(q, temp=363))
# Aquí, la ecuación 3 se usa para muestras líquidas
# Convolución con función de resolución y adición de
# El fondo D2O se realiza automáticamente con los # argumentos proporcionados
>>> sample.fit(modelProteinJumpDiff(q),
... res=vanadio,
... bkgd=buffer,
... volume_fraction_bkgd=0,95
... )
- Analice los datos E/IFWS utilizando un MSD40 generalizado o considerándolos como espectros QENS gruesos (que tienen solo unos pocos puntos de datos en el eje de energía). Cuando E/IFWS se considera QENS grueso, los modelos utilizados para QENS se utilizan para ajustar todo el conjunto de datos E/IFWS a la vez (ajuste global de transferencias de energía y transferencias de impulso).
- Trazar los datos ajustados utilizando:
>>> de nPDyn.plot import plot
>>> parcela(muestra)
Resultados
La agregación de lisozima en partículas se realizó a 90 °C con una concentración de proteína de 50 mg/mL en un tampón deuterado (0,1 M NaCl a pD 10,5). La formación de partículas se desencadena por el aumento de la temperatura a 90 °C y se produce dentro de las 6 h (Figura suplementaria S8). La adquisición de datos se realizó en IN16B, como se describe en el protocolo anterior (los datos son seleccionados permanentemente por el ILL y accesibles en http://dx.doi.org/10.5291/ILL-DATA.8-04-811)....
Discusión
La espectroscopia de neutrones es el único método que permite sondear la dinámica ps-ns promediada por conjuntos de muestras de proteínas, independientemente del tamaño de la proteína o de la complejidad de la solución cuando se utiliza la deuteración6. Específicamente, al sondear la autodifusión de ensamblajes de proteínas en solución, el tamaño hidrodinámico de dichos ensamblajes puede determinarse inequívocamente. No obstante, el método está comúnmente limitado por el bajo flu...
Divulgaciones
Los autores no tienen conflictos de intereses que revelar.
Agradecimientos
Los autores agradecen a Michaela Zamponi en el Centro Jülich para la Ciencia de Neutrones en el Heinz Maier-Leibnitz Zentrum, Garching, Alemania, por parte de los experimentos de dispersión de neutrones realizados en el instrumento SPHERES. Este trabajo se ha beneficiado de las actividades del consorcio Deuteration Laboratory (DLAB) financiado por la Unión Europea bajo los Contratos HPRI-2001-50065 y RII3-CT-2003-505925, y de la actividad financiada por el Consejo de Investigación de Ingeniería y Ciencias Físicas del Reino Unido (EPSRC) dentro del Institut Laue Langevin EMBL DLAB bajo las subvenciones GR / R99393/01 y EP / C015452 / 1. Se reconoce el apoyo de la Comisión Europea en el marco del 7º Programa Marco a través de la Acción Clave: Fortalecimiento del Espacio Europeo de Investigación, Infraestructuras de Investigación [Contrato 226507 (NMI3)]. Kevin Pounot y Christian Beck agradecen al Ministerio Federal de Educación e Investigación (BMBF, número de subvención 05K19VTB) por la financiación de sus becas postdoctorales.
Materiales
| Name | Company | Catalog Number | Comments |
| Aluminum sample holder | Not commercially available. Either the local contact on the instrument can provide them or they can be manufactured based on a technical drawing that can be provided by the local contact. | ||
| Deuterium chloride, 35 wt. % in D2O, ≥99 atom % D | Sigma-Aldrich | 543047 | |
| Deuterium oxide (D, 99.9%) | Eurisotop | DLM-4DR-PK | |
| Dow Corning high-vacuum silicone grease | Sigma-Aldrich | Z273554-1EA | |
| Ethanol 96%, EMSURE Reag. Ph Eur | Sigma-Aldrich | 1.5901 | |
| Glass dessicator | VWR | 75871-660 | |
| Glass dessicator plate, 140 mm | VWR | 89038-068 | |
| Indium wire, 1.0 mm (0.04 in) dia, Puratronic, 99.999% | Alfa Aesar | 00470.G1 | |
| Lysozyme from chicken egg white dialyzed, lyophilized, powder, ~100,000 U/mg | Sigma-Aldrich | 62970 | |
| nPDyn | v3.x | see github.com/kpounot/nPDyn, model functions fot fitting also included in the software | |
| OHAUS AX324 Adventurer balance, internal calibration | Dutscher | 92641 | |
| Phosphorus pentoxide, ReagentPlus, 99% | Sigma-Aldrich | 214701 | |
| Pipette ErgoOne 0.5-10 μL | Starlab | S7100-0510 | |
| Pipette ErgoOne 100-1,000 μL | Starlab | S7100-1000 | |
| Pipette ErgoOne 20-200 μL | Starlab | S7100-2200 | |
| Pipette tip TipOne 1,000 μL | Starlab | S1111-6001 | |
| Pipette tip TipOne 10 μL | Starlab | S1111-3200 | |
| Pipette tip TipOne 200 μL | Starlab | S1111-0206 | |
| Sodium deuteroxide solution, 40 wt. % in D2O, 99.5 atom % D | Sigma-Aldrich | 372072 |
Referencias
- Jacrot, B. Des neutrons pour la science: Histoire de l'Institut Laue-Langevin. Des neutrons pour la science. EDP Sciences. , (2021).
- Mahieu, E., Gabel, F. Biological small-angle neutron scattering: recent results and development. Acta Crystallographica Section D. 74 (8), 715-726 (2018).
- Grimaldo, M., Roosen-Runge, F., Zhang, F., Schreiber, F., Seydel, T. Dynamics of proteins in solution. Quarterly Reviews of Biophysics. 52, 7 (2019).
- Martel, A., et al. Membrane permeation versus amyloidogenicity: A multitechnique study of islet amyloid polypeptide interaction with model membranes. Journal of the American Chemical Society. 139 (1), 137-148 (2017).
- Pounot, K., et al. Tracking internal and global diffusive dynamics during protein aggregation by high-resolution neutron spectroscopy. The Journal of Physical Chemistry Letters. 11 (15), 6299-6304 (2020).
- Grimaldo, M., et al. Protein short-time diffusion in a naturally crowded environment. The Journal of Physical Chemistry Letters. 10 (8), 1709-1715 (2019).
- Jasnin, M., Stadler, A., Tehei, M., Zaccai, G. Specific cellular water dynamics observed in vivo by neutron scattering and NMR. Physical Chemistry Chemical Physics. 12 (35), 10154-10160 (2010).
- Frick, B. The neutron backscattering spectrometer IN16 at ILL-high energy resolution with high intensity and excellent signal-to-noise ratio. Neutron News. 13 (2), 15-22 (2002).
- Frick, B., Mamontov, E., van Eijck, L., Seydel, T. Recent backscattering instrument developments at the ILL and SNS. Zeitschrift für Physikalische Chemie. 224 (1-2), 33-60 (2010).
- Frick, B., Combet, J., van Eijck, L. New possibilities with inelastic fixed window scans and linear motor Doppler drives on high resolution neutron backscattering spectrometers. Nuclear Instruments and Methods in Physics Research Section A: Accelerators, Spectrometers, Detectors and Associated Equipment. 669, 7-13 (2012).
- Appel, M., Frick, B., Magerl, A. A flexible high speed pulse chopper system for an inverted neutron time-of-flight option on backscattering spectrometers. Scientific Reports. 8 (1), 13580 (2018).
- Squires, G. L. . Introduction to the theory of thermal neutron scattering. , (1996).
- Singwi, K. S., Sjölander, A. Diffusive motions in water and cold neutron scattering. Physical Review. 119 (3), 863-871 (1960).
- Sears, V. F. Theory of cold neutron scattering by homonuclear diatomic liquids: i. free rotation. Canadian Journal of Physics. 44 (6), 1279-1297 (1966).
- Sears, V. F. Theory of cold neutron scattering by homonuclear liquid: ii. hindered rotation. Canadian Journal of Physics. 44 (6), 1299-1311 (1966).
- Schirò, G., et al. Translational diffusion of hydration water correlates with functional motions in folded and intrinsically disordered proteins. Nature Communications. 6, 6490 (2015).
- Grimaldo, M., et al. Hierarchical molecular dynamics of bovine serum albumin in concentrated aqueous solution below and above thermal denaturation. Physical Chemistry Chemical Physics. 17 (6), 4645-4655 (2015).
- Eanes, E. D., Glenner, G. G. X-ray diffraction studies on amyloid filaments. Journal of Histochemistry & Cytochemistry. 16 (11), 673-677 (1968).
- Bonar, L., Cohen, A. S., Skinner, M. M. Characterization of the Amyloid Fibril as a Cross-β Protein. Proceedings of the Society for Experimental Biology and Medicine. 131 (4), 1373-1375 (1969).
- Chiti, F., Dobson, C. M. Protein Misfolding, Amyloid Formation, and Human Disease: A Summary of Progress Over the Last Decade. Annual Review of Biochemistry. 86 (1), 27-68 (2017).
- Knowles, T. P. J., Vendruscolo, M., Dobson, C. M. The amyloid state and its association with protein misfolding diseases. Nature Reviews Molecular Cell Biology. 15 (6), 384-396 (2014).
- Maji, S. K., et al. Functional amyloids as natural storage of peptide hormones in pituitary secretory granules. Science. 325 (5938), 328-332 (2009).
- Li, J., et al. The RIP1/RIP3 necrosome forms a functional amyloid signaling complex required for programmed necrosis. Cell. 150 (2), 339-350 (2012).
- Knowles, T. P. J., Mezzenga, R. Amyloid fibrils as building blocks for natural and artificial functional materials. Advanced Materials. 28 (31), 6546-6561 (2016).
- Stephens, A. D., Kaminski Schierle, G. S. The role of water in amyloid aggregation kinetics. Current Opinion in Structural Biology. 58, 115-123 (2019).
- Adamcik, J., Mezzenga, R. Amyloid polymorphism in the protein folding and aggregation energy landscape. Angewandte Chemie International Edition. 57 (28), 8370-8382 (2018).
- Liu, Z., et al. Entropic contribution to enhanced thermal stability in the thermostable P450 CYP119. Proceedings of the National Academy of Sciences of the United States of America. 115 (43), 10049-10058 (2018).
- Coquelle, N., et al. Chromophore twisting in the excited state of a photoswitchable fluorescent protein captured by time-resolved serial femtosecond crystallography. Nature Chemistry. 10 (1), 31-37 (2018).
- Henzler-Wildman, K. A., et al. A hierarchy of timescales in protein dynamics is linked to enzyme catalysis. Nature. 450 (7171), 913-916 (2007).
- Fichou, Y., et al. Hydration water mobility is enhanced around tau amyloid fibers. Proceedings of the National Academy of Sciences of the United States of America. 112 (20), 6365-6370 (2015).
- Burns, J., Pennock, C. A., Stoward, P. J. The specificity of the staining of amyloid deposits with thioflavine T. The Journal of Pathology and Bacteriology. 94 (2), 337-344 (1967).
- Iqbal, K., Liu, F., Gong, C. -. X., Grundke-Iqbal, I. Tau in Alzheimer disease and related tauopathies. Current Alzheimer Research. 7 (8), 656-664 (2010).
- Krȩżel, A., Bal, W. A formula for correlating pKa values determined in D2O and H2O. Journal of Inorganic Biochemistry. 98 (1), 161-166 (2004).
- Dolman, M., Halling, P. J., Moore, B. D., Waldron, S. How dry are anhydrous enzymes? Measurement of residual and buried 18O-labeled water molecules using mass spectrometry. Biopolymers. 41 (3), 313-321 (1997).
- Pounot, K. kpounotnPDyn: v3.0.0. Zenodo. , (2021).
- Yi, Z., Miao, Y., Baudry, J., Jain, N., Smith, J. C. Derivation of mean-square displacements for protein dynamics from elastic incoherent neutron scattering. Journal of Physical Chemistry B. 116 (16), 5028-5036 (2012).
- Peters, J., Kneller, G. R. Motional heterogeneity in human acetylcholinesterase revealed by a non-Gaussian model for elastic incoherent neutron scattering. The Journal of Chemical Physics. 139 (16), 165102 (2013).
- Zeller, D., Telling, M. T. F., Zamponi, M., García Sakai, V., Peters, J. Analysis of elastic incoherent neutron scattering data beyond the Gaussian approximation. The Journal of Chemical Physics. 149 (23), 234908 (2018).
- Roosen-Runge, F., Seydel, T. A generalized mean-squared displacement from inelastic fixed window scans of incoherent neutron scattering as a model-free indicator of anomalous diffusion confinement. EPJ Web of Conferences. 83, 02015 (2015).
- Ortega, A., Amorós, D., García de la Torre, J. Prediction of hydrodynamic and other solution properties of rigid proteins from atomic- and residue-level models. Biophysical Journal. 101 (4), 892-898 (2011).
- Hennig, M., Frick, B., Seydel, T. IUCr Optimum velocity of a phase-space transformer for cold-neutron backscattering spectroscopy. Journal of Applied Crystallography. 44 (3), 467-472 (2011).
- Paalman, H. H., Pings, C. J. Numerical evaluation of X-ray absorption factors for cylindrical samples and annular sample cells. Journal of Applied Physics. 33 (8), 2635-2639 (1962).
- Ow, S. -. Y., Dunstan, D. E. The effect of concentration, temperature and stirring on hen egg white lysozyme amyloid formation. Soft Matter. 9 (40), 9692-9701 (2013).
- Tominaga, T., Sahara, M., Kawakita, Y., Nakagawa, H., Yamada, T. Evaluation of sample cell materials for aqueous solutions used in quasi-elastic neutron scattering measurements. Journal of Applied Crystallography. 54 (6), 1631-1640 (2021).
- Beck, C., et al. Following protein dynamics in real time during crystallization. Crystal Growth & Design. 19 (12), 7036-7045 (2019).
- Smith, A. A., Testori, E., Cadalbert, R., Meier, B. H., Ernst, M. Characterization of fibril dynamics on three timescales by solid-state NMR. Journal of Biomolecular NMR. 65 (3-4), 171-191 (2016).
- Wang, T., Jo, H., DeGrado, W. F., Hong, M. Water distribution, dynamics, and interactions with Alzheimer's β-amyloid fibrils investigated by solid-state NMR. Journal of the American Chemical Society. 139 (17), 6242-6252 (2017).
- Rezaei-Ghaleh, N., Giller, K., Becker, S., Zweckstetter, M. Effect of zinc dinding on β-amyloid structure and dynamics: Implications for Aβ aggregation. Biophysical Journal. 101 (5), 1202-1211 (2011).
- Vugmeyster, L., et al. Fast motions of key methyl groups in amyloid-β fibrils. Biophysical Journal. 111 (10), 2135-2148 (2016).
- Yang, X., Wang, B., Hoop, C. L., Williams, J. K., Baum, J. NMR unveils an N-terminal interaction interface on acetylated-α-synuclein monomers for recruitment to fibrils. Proceedings of the National Academy of Sciences of the United States of America. 118 (18), (2021).
- Tuttle, M. D., et al. Solid-state NMR structure of a pathogenic fibril of full-length human α-synuclein. Nature Structural & Molecular Biology. 23 (5), 409-415 (2016).
- Karamanos, T. K., Kalverda, A. P., Thompson, G. S., Radford, S. E. Mechanisms of amyloid formation revealed by solution NMR. Progress in Nuclear Magnetic Resonance Spectroscopy. 88-89, 86-104 (2015).
- Lai, Y. -. C., Kuo, Y. -. H., Chiang, Y. -. W. Identifying protein conformational dynamics using spin-label ESR. Chemistry - An Asian Journal. 14 (22), 3981-3991 (2019).
- Franck, J. M., Han, S. Overhauser dynamic nuclear polarization for the study of hydration dynamics, explained. Methods in Enzymology. 615, 131-175 (2019).
- Pavlova, A., et al. Protein structural and surface water rearrangement constitute major events in the earliest aggregation stages of tau. Proceedings of the National Academy of Sciences of the United States of America. 113 (2), 127-136 (2016).
- Lin, Y., et al. Liquid-liquid phase separation of tau driven by hydrophobic interaction facilitates fibrillization of tau. bioRxiv. , (2020).
- Decatur, S. M. Elucidation of residue-level structure and dynamics of polypeptides via isotope-edited infrared spectroscopy. Accounts of Chemical Research. 39 (3), 169-175 (2006).
- Chatani, E., Tsuchisaka, Y., Masuda, Y., Water Tsenkova, R. molecular system dynamics associated with amyloidogenic nucleation as revealed by real time near infrared spectroscopy and aquaphotomics. PLoS One. 9 (7), 101997 (2014).
- Goret, G., Aoun, B., Pellegrini, E. MDANSE: An interactive analysis environment for molecular dynamics simulations. Journal of Chemical Information and Modeling. 57 (1), 1-5 (2017).
- Fujiwara, S., et al. Internal dynamics of a protein that forms the amyloid fibrils observed by neutron scattering. Journal of the Physical Society of Japan. 82, (2013).
- Schiró, G., et al. Neutron scattering reveals enhanced protein dynamics in concanavalin a amyloid fibrils. Journal of Physical Chemistry Letters. 3 (8), 992-996 (2012).
- Pounot, K., et al. Zinc determines dynamical properties and aggregation kinetics of human insulin. Biophysical Journal. 120 (5), 886-898 (2021).
- Fujiwara, S., et al. Dynamic properties of human α-synuclein related to propensity to amyloid fibril formation. Journal of Molecular Biology. 431 (17), 3229-3245 (2019).
- Sanz, A., et al. High-pressure cell for simultaneous dielectric and neutron spectroscopy. Review of Scientific Instruments. 89 (2), 023904 (2018).
- Adams, M. A., et al. Simultaneous neutron scattering and Raman scattering. Applied Spectroscopy. 63 (7), 727-732 (2009).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados