Method Article
Drosophila'da Tek Moleküllü Ölçeklerde Kas Transkripsiyon Dinamiğinin İncelenmesi
Bu Makalede
Özet
Drosophila, miyogenezi düzenleyen anahtar molekülleri incelemek için iyi kurulmuş bir modeldir. Bununla birlikte, mevcut yöntemler, mRNA transkripsiyonel dinamiklerini ve sinsitya içindeki uzamsal dağılımı belirlemek için yetersizdir. Bu sınırlamayı ele almak için, mRNA'ların tek molekül ölçeğinde saptanmasına ve miktarının belirlenmesine izin veren bir RNA floresan in situ hibridizasyon yöntemini optimize ettik.
Özet
İskelet kasları, kuvvet üreten ve vücut hareketini sağlayan birçok demetlenmiş miyofiberden oluşan büyük sinsitialardır. Drosophila , kas biyolojisini incelemek için klasik bir modeldir. Hem Drosophila genetiğinin hem de gelişmiş omik yaklaşımların kombinasyonu, kas morfogenezini ve rejenerasyonunu düzenleyen anahtar korunmuş moleküllerin tanımlanmasına yol açtı. Bununla birlikte, bu moleküllerin transkripsiyonel dinamikleri ve haberci RNA'larının sinsitya içindeki uzamsal dağılımı geleneksel yöntemlerle değerlendirilemez. Burada, yetişkin uçuş kasları ve kas kök hücreleri içindeki bireysel mRNA moleküllerinin saptanmasını ve ölçülmesini sağlamak için mevcut bir tek moleküllü RNA floresan in situ hibridizasyon (smFISH) yöntemini optimize ettik. Kavramın bir kanıtı olarak, iki evrimsel korunmuş transkripsiyon faktörünün, Mef2 ve Zfh1/Zeb'nin mRNA ekspresyonunu ve dağılımını analiz ettik. Bu yöntemin, kas öncü hücrelerindeki, yetişkin kaslarındaki ve kas kök hücrelerindeki her iki transkript için tek mRNA moleküllerini verimli bir şekilde tespit edebildiğini ve ölçebildiğini gösteriyoruz.
Giriş
Yetişkin iskelet kasları, kasılma özellikleri hareketler üreten farklılaşmış çok çekirdekli miyoliflerden yapılır. Kas büyümesi, bakımı ve rejenerasyonu, embriyonik gelişim sırasında belirtilen kas progenitörlerine ve kas kök hücrelerine (MuSC'ler) dayanır1. Miyojenik program, bir dizi çekirdek miyojenik transkripsiyon faktörü (TF'ler) tarafından hassas bir şekilde kontrol edilir (örneğin, Pax3 / 7, MYOD, Mef2 ve ZEB)2,3,4. Kas biyolojisini düzenleyen moleküler mekanizmaların deşifre edilmesi, miyoloji ile ilgili temel soruların aydınlatılması ve kas dejeneratif hastalıkların tedavisinde olası terapötik kullanım için önemlidir.
Drosophila, miyogenez5'i incelemek için genetik bir model olarak uzun bir geçmişe sahiptir. Son zamanlarda kas rejenerasyonunu araştırmak için yeni bir model olarak ortaya çıkmıştır 6,7,8. Kas yapısı ve çekirdek miyojenik programlar, sinekler ve memeliler arasında yüksek oranda korunur. Örneğin, TF'ler Mef2 ve Zfh1 / ZEB, kas gelişimini ve rejenerasyonunu düzenlemede korunmuş bir işleve sahiptir 3,9,10,11. Dolaylı Uçuş Kasları (IFM'ler) gibi yetişkin Drosophila kasları, Yetişkin Kas Progenitörleri (AMP'ler) olarak bilinen belirli bir MuSC popülasyonundan oluşur12. Bu AMP'ler embriyogenez sırasında belirlenir ve kanat ve bacak diskleri gibi epitel yapılarıyla ilişkilidir. Embriyonik ve larva aşamaları boyunca, AMP'ler, IFM'leri oluşturmak için farklılaşma ve füzyona girdiklerinde metamorfoza kadar farklılaşmadan kalırlar13,14. TF Spalt majör (spalt, salm) uçuş kas gelişimi sırasında eksprese edilir ve yapısal kimliklerini belirlemek için gereklidir15. Mef2, yetişkin kas oluşumu için gerekli olan bir diğer önemli miyojenik TF'dir 16,17. AMP'lerde ifade edilir ve yetişkin IFM'lerde10,18 korunur. AMP'lerin çoğunluğu fonksiyonel kaslara farklılaşırken, bir alt küme farklılaşmadan kaçar ve yetişkin MuSC'lerioluşturur 11. Omurgalılara benzer şekilde, yetişkin MuSC'lerin erken farklılaşmasını önlemek ve gövdelerini korumak için TF Zfh1/ZEB gereklidir 9,10.
Gen ekspresyon dinamiklerinin çeşitli kas biyolojik süreçlerini düzenlediği kanıtlanmıştır 19,20. Yüksek verimli tek hücreli ve tek çekirdekli RNA dizileme tekniklerinin ortaya çıkışı, bu transkripsiyonel dinamiklerin kapsamlı bir şekilde araştırılmasını sağlamıştır21,22. Bu yaklaşımların dikkate değer bir sınırlaması, çok çekirdekli kas liflerinde mRNA moleküllerinin uzamsal dağılımını sağlayamamalarıdır. Bu özellikler, iki tip mRNA gövdesini ortaya çıkaran tek moleküllü floresan in situ hibridizasyon (smFISH) ile araştırılabilir: 1. Sitoplazmada yayılmış ve olgun RNA'yı temsil eden bireysel mRNA molekülleri ve 2. En fazla iki parlak nükleer odak, yeni ortaya çıkan transkripte karşılık gelir ve transkripsiyonel olarak aktif alelleriortaya çıkarır 23. Bu nedenle, smFISH, bireysel mRNA molekülü miktar tayini için tercih edilen bir yöntemdir, uzamsal dağılımlarını araştırır ve gen transkripsiyonel dinamiklerinin anlık görüntülerini sağlar.
smFISH yöntemi, hedef transkripti tamamlayıcı olacak şekilde özel olarak tasarlanmış bir dizi kısa floresan oligonükleotid probuna dayanır. Tavlama üzerine, konfokal mikroskopi24 kullanılarak mRNA'nın tek molekül seviyesinde saptanmasına izin veren yüksek yoğunluklu nokta kaynakları üretir. Bu yöntem, memeli kas dokuları da dahil olmak üzere çok çeşitli hücre tipleri için giderek daha fazla uygulanmaktadır19,20. Bununla birlikte, diğer hayvan modellerinde olduğu gibi Drosophila'da, yetişkin kas gen ekspresyonu hakkındaki bilgilerin çoğu, mRNA moleküllerinin uzamsal konumu hakkında nicel bilgiden yoksun olan toplu moleküler deneylerden türetilmiştir. Burada, kas öncü hücreleri ve yetişkin Drosophila kasları 23,25 üzerinde smFISH gerçekleştirmek için bir yöntemi optimize ettik. Bu protokol, çekirdek segmentasyonu ve mRNA sayımı ve lokalizasyonu için tam otomatik bir analiz hattı içerir.
Protokol
1. Kanat diski ve erişkin kas diseksiyonu ve hazırlanması
NOT: Diseksiyon tezgahını ve tüm diseksiyon aletlerini RNAse inhibitör solüsyonu ile temizleyin (Malzeme Tablosu). Tüm deney prosedürü boyunca laboratuvar eldivenleri giyilmelidir.
- Kanat diski
- L3 aşamalı larvaları soğuk 1x fosfat tamponlu salin (PBS) içine yerleştirin (Şekil 1A).
- Larvaları 3x 1x PBS ile durulayın ve 5 dakika boyunca bu solüsyonda tutun.
- İki çift keskin forseps kullanarak, larvaları ön kısımdan tutun ve vücut dokularının geri kalanını (yaklaşık 2/3) çekin ve atın.
- Larvaların ilk 1 / 3'ünü bir çift forseps ile ve ağız kancalarını diğer çiftle tutun. Larva içindeki ağız kancalarını tamamen ters çevrilene kadar geri çekin.
NOT: Analiz için yeterli numune büyüklüğünü sağlamak için deney başına yaklaşık 30 larvayı inceleyin. - Kanat disklerinin, larvaların her iki tarafı boyunca uzanan trakeanın iki ana dalı ile yakından ilişkili olduğunu gözlemleyin.
- Trakeaya bağlı bir çift kanat diski ile temiz bir ters çevrilmiş larva karkası elde etmek için diğer tüm larva bileşenlerini (bacak diskleri ve beyin) çıkarın (Şekil 1B).
- Numuneleri 1 mL'lik bir santrifüj tüpüne yerleştirin ve çalkalama altında oda sıcaklığında 45 dakika boyunca 1x PBS'de seyreltilmiş %4 paraformaldehit (PFA) içinde sabitleyin.
NOT: Numune çalkalama salınımlı bir karıştırıcı ile sağlanır (Malzeme Tablosu). - Numuneleri 3 x 5 dakika %70 etanol (RNAse'sız suda seyreltilmiş EtOH) ile yıkayın ve aynı yıkama solüsyonunda 4 °C'de en az 2 gün ve 1 haftaya kadar saklayın.
- Dolaylı uçuş kasları
- Yetişkin sinekleri bir CO2 sinek pedi üzerinde uyuşturun ve kafaları, karınları, bacakları ve kanatları çıkarın.
- Diseke edilmiş toraksları soğuk 1x PBS'ye yerleştirin ve tüm numuneler diseke edilene kadar devam edin (Şekil 1D).
- Toraksları ajitasyon altında oda sıcaklığında 20 dakika boyunca% 4 PFA, (% 1 Triton) ile önceden sabitleyin.
- Numuneleri 1x PBT yıkama solüsyonu (1x PBS'de %1 Triton) ile 3 x 5 dakika durulayın.
- Toraksları bir cam slayt üzerindeki çift taraflı bir bant (Malzeme Tablosu) üzerine yerleştirin ve iki hemi-toraks oluşturmak için keskin bir mikrotom bıçağıyla ikiye bölün (Şekil 1E,F).
- Numuneleri çalkalama altında oda sıcaklığında 45 dakika boyunca %4 PFA (%1 Triton) içinde sabitleyin.
- 2 x 20 dakika 1x PBT yıkama solüsyonu (%1 Triton, 1x PBS) ile oda sıcaklığında çalkalama altında yıkayın.
- Hemi-toraksları soğuk 1x PBS'ye yerleştirin ve IFM'leri hemi-thorastan nazikçe izole etmek için forseps kullanın (Şekil 1G).
NOT: Bunu başarmak için, kütikülü mümkün olduğunca iyice çıkarmak önemlidir. - Numuneleri geçirgenleştirmek için, izole edilmiş IFM'leri %70 EtOH çözeltisinde en az 2 gün ve 4 °C'de 1 haftaya kadar saklayın.
NOT: Analiz için yeterli numune boyutunu sağlamak için IFM'leri deney başına yaklaşık 10 torakstan izole edin.
2. Hibridizasyon, immün boyama ve montaj
NOT: Hem kanat diskleri hem de IFM numuneleri için benzer hibridizasyon prosedürü uygulanır. Hibridizasyona başlamadan önce kanat disklerini larva karkaslarından izole etmek ve 200 μL Tampon A'ya koymak çok önemlidir.
- 12.5 μM'lik bir nihai stok çözeltisi için probları 400 μL TE tamponunda (10 mM Tris-HCl, 1 mM EDTA, pH 8.0) yeniden süspanse edin.
- Numuneleri Tampon A (Malzeme Tablosu) içeren 0,5 mL'lik bir santrifüj tüpüne aktarın.
NOT: Kabın hacmini azaltarak, numuneleri görselleştirmek ve yıkama işlemi sırasında kaybolmalarını önlemek daha kolay hale gelir. - Numuneleri Tampon A ile 2 x 20 dakika yıkayın.
- Tampon A'yı aspire edin, 400 μL hibridizasyon tamponu (Malzeme Tablosu) ile değiştirin ve numuneleri bir termal karıştırıcıda (Malzeme Tablosu) 37 °C'de 30 dakika önceden hibridize edin.
- Probları, hibridizasyon tamponunda kanat diskleri için 125 nM ve yetişkin kasları için 200 nM'lik bir nihai konsantrasyona seyreltin ve birincil antikoru ekleyin.
NOT: Antikor seyreltmesi, kullanılan spesifik antikora bağlı olarak değişir (Malzeme Tablosu). Antikor eklemek isteğe bağlıdır. Kullanıcılar protein boyamadan vazgeçmeyi tercih edebilir ve bunun yerine antikor ilavesini içermeyen benzer bir smFISH protokolü kullanabilir. - Hibridizasyon tamponunu aspire edin ve probu ve antikoru içeren 100 μL hibridizasyon tamponu ile değiştirin.
- Numuneleri karanlıkta 37 °C'de bir termal karıştırıcıda 300 rpm çalkalama altında en az 16 saat inkübe edin.
- Tampon A'nın bir kısmını 37 °C'de ısıtın.
- Numuneleri termal karıştırıcıda 37 °C'de 300 rpm çalkalama altında 3 x 10 dakika yıkayın.
- Son yıkama sırasında, ikincil antikoru (1/200) ve DAPI'yi (1/10.000) Tampon A'da seyreltin ve numuneleri bu çözelti içinde 37 °C'de en az 1 saat inkübe edin.
- Numuneleri Tampon B (Malzeme Tablosu) ile 3 x 20 dakika yıkayın.
- Numuneleri dikkatlice bir mikroskop lamı üzerine aktarın, kalan Tampon B'yi silin ve 30 μL montaj ortamı ekleyin ve 18 x 18 mm'lik bir lamel ile örtün. Preparasyonu kapatmak için bir oje kullanın.
- Numuneleri 4 °C'de 1 haftaya kadar saklayın.
NOT: Ancak, prob sinyalinin bozulmasını önlemek için görüntülemenin mümkün olan en kısa sürede yapılması önerilir.
3. Görüntüleme
- 40x ve 63x yağa daldırma hedefleriyle donatılmış bir konfokal mikroskop kullanarak xyz toplama moduyla görüntüler elde edin (Malzeme Tablosu). smFISH sinyali, HyD dedektörü tarafından bir foton sayma modu aracılığıyla algılanır.
- DAPI sinyalini ve UV lambasını kullanarak numuneleri bulun.
- Kanat diski ile ilişkili kas progenitörlerini ve IFM'leri (Şekil 2 ve Şekil 3) görüntülemek için, lazer çizgileri ve emisyon filtreleri için aşağıdaki kurulumu uygulayın: DAPI'yi seçin (uyarma (örn.) 405 nm, emisyon (em.) 450 nm), Alexa Fluor 488 (ör. 496 nm, em. 519 nm) ve smFISH sondaları için Quasar 670 (örn. 647 nm, em. 670 nm).
- Yetişkin MuSC'leri görüntülemek için (Şekil 2D), lazer çizgileri ve emisyon filtreleri için aşağıdaki kurulumu uygulayın: DAPI'yi seçin (örn. 405 nm, em. 450 nm), Alexa Fluor 488 (ör. 496 nm, em. 519 nm), Alexa Fluor 555 (ör. 555 nm, em. 565 nm) ve smFISH sondaları için Quasar 670 (örn. 647 nm, em. 670 nm).
NOT: Görüntüleme için dalga boyları, ikincil antikorlar ve problarla ilişkili boyalara göre uyarlanabilir. - Konfokal görüntüleri olarak kaydedin. TIFF dosyaları.
4. Görüntüleme sonrası analiz
- Başlamak için ImageJ'i başlatın ve Eklentiler menüsüne gidin. Oradan, Makrolar | Makro kaynak kodunu açmak için Düzenle'yi seçin.
NOT: Bu, parametrelerin gerektiği gibi ayarlanmasını sağlar ve gerekirse, makronun kaynak kodundaki her kanal için klasör dizinini değiştirir. - Bu protokolü takip etmek ve larvalardaki Mef2 smFISH lekelerini ölçmek için aşağıdaki ayarları kullanın: Log_radius = 3.0 ve Log_quality = 20.0. Bulanık Mef2-pozitif çekirdekler = 2; nucleus_scale_parameter = 30; nucleus_threshold = -8.0; nucleus_size = 300.
- Kas liflerinde aşağıdaki parametreleri kullanın: Log_radius = 2.5; Log_quality = 60.0; bulanıklık = 2; nucleus_scale_parameter = 100; nucleus_threshold = 0.0; nucleus_size = 300 olur.
- Makroyu başlatmak için, çalıştır komutunu çalıştırın ve makronun belirlenen klasördeki tüm görüntüleri otomatik olarak yüklemesini ve bunları sıralı bir şekilde ölçmesini bekleyin.
NOT: Bu işlemin süresinin, analiz edilen veri hacmine bağlı olarak birkaç saniye ile birkaç saat arasında değişebileceğini lütfen unutmayın. - İki yeni .csv dosyasında görüntülenen sonuçları arayın: file_FISH_results ve file_nuclei_results. Birincisi, transkripsiyonel noktaların sayısını ve bunların ortalama ve maksimum yoğunluklarını gösterir. İkincisi, hem toplam çekirdek sayısını hem de her çekirdeğin içindeki nokta sayısını verir.
Sonuçlar
Bu protokolde, Mef2 ve zfh1 mRNA'yı hedefleyen ticari olarak üretilmiş problar kullandık. Probları, üreticinin FISH prob tasarımcısı (Malzeme Tablosu) ile tasarladık. Mef2 seti, tüm Mef2 RNA izoformlarında (ekzon 3'ten ekzon 10'a) ortak olan ekzonları hedefler. zfh1 seti, hem zfh1-RB hem de zfh1-RE izoformları10 için ortak olan üçüncü ekzonu hedefler. Her iki prob da Quasar 670 floresan boyasına konjuge edilir.
Ham .tif verilerini otomatik olarak analiz etmek için ImageJ yazılımıyla uyumlu bir makroyu şirket içinde oluşturduk. Makro, Fiji eklentisi BIOP ile Cellpose26 ile çekirdeklerin 3B segmentasyonuna ve Trackmate eklentisi27'de uygulandığı gibi Gauss'un bir Laplacian tarafından transkripsiyonel nokta ölçümüne izin verir. MorpholibJ eklentisi28 , son işlem için kullanılır (Ek Dosya 1).
İlk adım olarak, Mef2 ve zfh1'in AMP'lerde ifade edildiği bilinen kanat görüntüleme disklerinde smFISH gerçekleştirdik. Bu veriler, her iki transkriptin de Mef2 veya Zfh1 antikorları ile birlikte boyanmış AMP popülasyonunda eşit olarak tespit edildiğini göstermektedir (Şekil 2A, B). Aktif transkripsiyon bölgeleri (TS), bireysel sitoplazmik transkriptlerden veya olgun nükleer transkriptlerden daha büyük olma eğiliminde oldukları ve daha yoğun sinyallere sahip oldukları için smFISH tarafından olgun mRNA'dan ortaya çıkarılabilir ve ayırt edilebilir. Tutarlı bir şekilde, AMP'lerin daha yüksek büyütmeleri, TS odakları ile sitoplazmaya dağılmış olgun mRNA arasında ayrım yapar (Şekil 2A',B'), bu smFISH protokolünün duyarlılığını doğrular. Bununla birlikte, hem zfh1 hem de Mef2 için çekirdek başına bir aktif TS gözlemlediğimiz belirtilmelidir (Şekil 2A',B'). Bu veriler, AMP'lerdeki Mef2 ve zfh1 transkripsiyonel paternleri, ilgili proteinlerinin tespiti ile birlikte lokalize olduğundan, prob tasarımının doğruluğunu daha da doğrular (Şekil 2).
İkinci adımda, farklılaşmış yetişkin IFM'lerde ve ilişkili kök hücrelerde Mef2 ve zfh1 mRNA'larının transkripsiyon yeri ve dağılımını inceledik (Şekil 2C,D). Bu veriler, sinsityal kas çekirdeklerindeki Mef2 TS'leri ve sitoplazma boyunca Mef2 mRNA dağılımını açıkça göstermektedir (Şekil 2C,C'). Zfh1 özellikle MuSC'lerin nüfusunu10,11 olarak işaretler. Bu smFISH protokolünü kullanarak, GFP ekspresyonu > Zfh1-Gal4 ile işaretlenmiş MuSC'lerde zfh1 transkripsiyonunu başarıyla tespit ettik. Daha yüksek büyütme, hem zfh1 TS hem de sitoplazmik tek mRNA'ların saptanmasını gösterir (Şekil 2D').
Son olarak, Mef2 transkripsiyonel dinamiklerini ve uzamsal dağılımı nicel olarak analiz etmek için şirket içi yerleşik ImageJ makromuzu kullandık. Hesaplama boru hattı, Mef2 noktalarını ve kas çekirdeklerini verimli bir şekilde tespit edebilir ve segmentlere ayırabilir (Şekil 3A-C). Ancak, sinyal-gürültü oranı sorunları nedeniyle, bazı durumlarda bazı noktalar algılanamayabilir (Şekil 3A',B'). Mef2 mRNA bolluğunun kas öncü hücreleri (AMP'ler) ve farklılaşmış yetişkin IFM'ler arasında değişip değişmediğini araştırmak için bu otomatik yöntemi kullandık. Analizimiz, yetişkin IFM'lerde ve AMP'lerde çekirdek başına Mef2 mRNA sayısının önemli ölçüde farklı olmadığını göstermektedir (Şekil 3D).
Yetişkin IFM kaslarındaki Mef2 transkriptlerinin uzamsal dağılımı hakkında fikir edinmek için, sitoplazmik ve nükleer Mef2 mRNA noktalarının fraksiyonunu ölçtük (Şekil 3E, F). Programımızı kullanarak hem toplam Mef2 mRNA nokta sayısını hem de çekirdek boyama (Mef2) ile örtüşen Mef2 mRNA nokta sayısını saydık (Mef2). Çekirdekle ilişkili mRNA'nın toplam mRNA sayısından çıkarılması, sitoplazmik mRNA noktalarının sayısını sağlar. Bulgularımız, Şekil 3E,F'de gösterildiği gibi, Mef2 mRNA'nın yaklaşık %92'sinin sitoplazmada bulunduğunu, kalan %8'inin ise kas çekirdekleri ile ilişkili olduğunu ortaya koymaktadır.
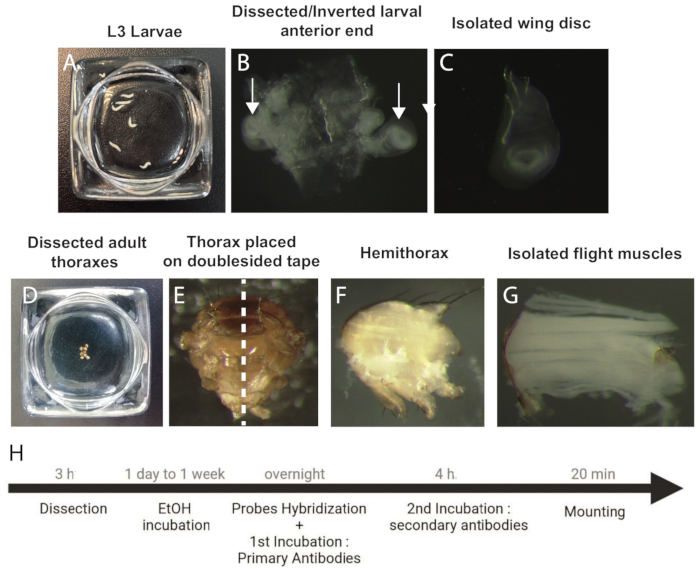
Şekil 1: smFISH için kanat disklerini ve IFM örneklerini inceleme ve hazırlama prosedürü. (A) L3 aşamalı larvalar. (B) Larvaların disseke edilmiş ve ters çevrilmiş ön ucu. Oklar kanat disklerini gösterir. (C) İzole kanat diski. (D) Diseke yetişkin torakslar. (E) Bisection öncesi çift taraflı bir bant üzerine yönlendirilmiş yetişkin toraks (noktalı çizgi). (F) Yetişkin hemitoraks. (g) İzole IFM'ler. (H) smFISH protokolünün iş akışı. Kısaltmalar: IFM'ler = dolaylı uçuş kasları; smFISH = tek moleküllü RNA floresansı in situ hibridizasyon; EtOH = etanol. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
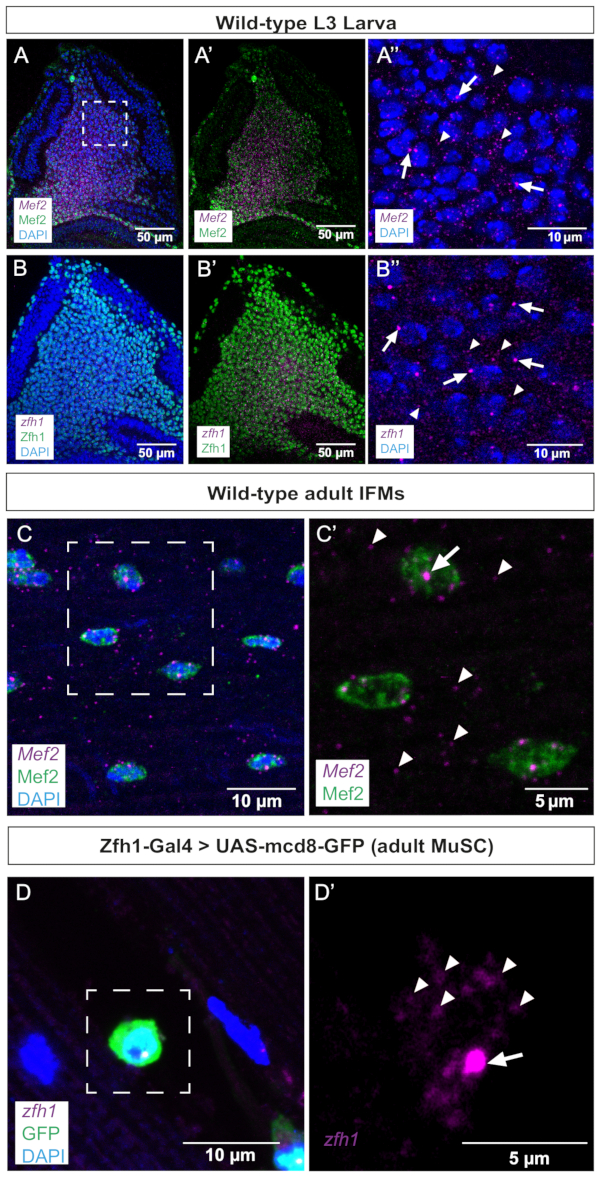
Şekil 2: Kanat disklerinde ve yetişkin IFM'lerde Mef2 ve zfh1 smFISH'in temsili görüntüleri. (A,B) Mef2 ve zfh1 mRNA'ları (mor) AMP'lerde homojen olarak tespit edilir ve sırasıyla (A,A') Mef2 ve (B,B') Zfh1 proteinleri (yeşil) ile birlikte lokalize olur. (A'',B'') AMP'lerin daha yüksek büyütme oranı. (C) Yetişkin IFM'lerde Mef2 mRNA'ları ve Mef2 proteini tespit edilir. (C') C'de kutulu bölgenin daha yüksek büyütmesi. (D) Zfh1-Gal4 >UAS-mCD8GFP ekspresyonu (yeşil) yetişkin MuSC'leri etiketler. ZFH1 RNA, yetişkin MuSC'lerde tespit edilir. (D') D'de kutulu bölgenin daha yüksek büyütmesi. Zfh1 transkripsiyon başlangıç bölgeleri ve tek mRNA'lar sırasıyla oklar ve ok uçları ile gösterilir. DAPI boyama (mavi). Ölçek çubukları = 50 μm (A,B,A',B'), 10 μm (A",B",C,D), 5 μm (C',D'). Kısaltmalar: IFM'ler = dolaylı uçuş kasları; smFISH = tek moleküllü RNA floresansı in situ hibridizasyon; AMP'ler = yetişkin kas progenitörleri; MuSC'ler = kas kök hücreleri; DAPI = 4',6-diamidino-2-fenilindol; GFP = yeşil floresan proteini. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
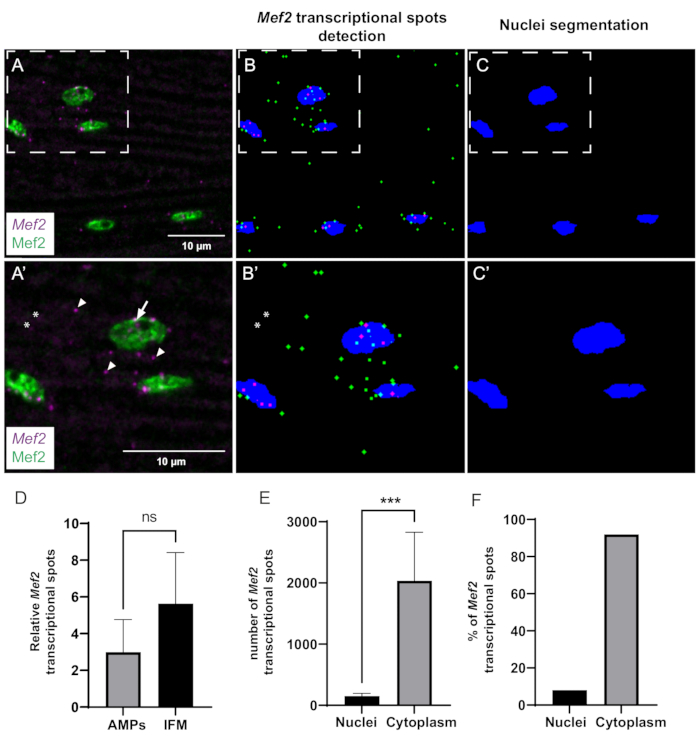
Şekil 3: Mef2 transkripsiyonunun ve sitoplazmik mRNA birikiminin smFISH ile miktarının belirlenmesi. (A) Yabani tip yetişkin IFM'lerde Mef2 transkriptlerinin (mor) ve Mef2 proteininin (yeşil) dağılımı. (Bir') A'da kutulu bölgenin daha yüksek büyütmesi. Transkripsiyon başlangıç bölgeleri ve tek mRNA'lar sırasıyla oklar ve ok uçları ile gösterilir. (B,C) Otomatik Mef2 nokta bulma ve nükleer segmentasyon. Sitoplazmik ve nükleer Mef2 mRNA'lar sırasıyla yeşil ve macenta ile gösterilir. (B',C') B ve C'de kutulu bölgelerin daha yüksek büyütme oranları. Yıldız işaretleri, makro tarafından sayılmayan noktaları gösterir. (D) Toplam çekirdek sayısına göre AMP'lerde ve yetişkin IFM'lerde Mef2 nokta ölçümü örneği. (p=0,0785, Student t-testi, kanat diskleri (n=4), IFM'ler (n=11)). (E) Yetişkin IFM'lerde Mef2 spot dağılımının nicelleştirilmesi örneği. (*** p< 0.0007, Öğrenci t-testi, n = 5). (F) Çekirdeğin içinde ve dışında tespit edilen Mef2 mRNA noktalarının yüzdesi (n = 5). Ölçek çubukları = 10 μm (A,A'). Kısaltmalar: IFM'ler = dolaylı uçuş kasları; smFISH = tek moleküllü RNA floresansı in situ hibridizasyon; AMP'ler = yetişkin kas progenitörleri. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
Ek Dosya 1: Son işlem için kullanılan makro. Bu Dosyayı indirmek için lütfen buraya tıklayın.
Tartışmalar
smFISH yönteminin uygulanması, çeşitli hücre tiplerine ve model organizmalara kadar uzanan kullanımı ile son zamanlarda popülerlik kazanmıştır. Bununla birlikte, Drosophila durumunda, yetişkin kas gen ekspresyonu hakkındaki bilgilerin çoğu, mRNA moleküllerinin kesin uzamsal konumu hakkında nicel bilgi sağlayamayan toplu moleküler deneylerden türetilir. Bu boşluğu gidermek için, kas öncü hücreleri ve yetişkin Drosophila kasları üzerinde smFISH gerçekleştirmek için bir yöntemi optimize ettik. Bu yaklaşım daha önce yayınlanmış bir protokol23'ten uyarlanmış ve Drosophila kas dokuları için optimize edilmiştir.
Yüksek kaliteli smFISH görüntüleri elde etmenin önündeki en büyük engel, probların optimum penetrasyonunu engelleyen yetişkin kaslarının kalınlığıdır. Bu nedenle, yetişkin kaslarını hayvanın diğer dokularından ayırmak ve hibridizasyon işlemini hafif ajitasyon ile gerçekleştirmek çok önemlidir. Bu özel adım, dokuların etkili bir şekilde geçirgenleşmesini sağlar.
Bir diğer kritik husus, sinyal-gürültü oranının, hesaplama boru hattının belirli mRNA noktalarını tespit etmede başarısız olmasına neden olabilmesidir. Sinyal-gürültü oranının arttırılmasının, probların optimal konsantrasyonunu bularak elde edilebileceğini bulduk. Optimum seyreltme, konjuge boya ve oligonükleotid bileşimi dahil olmak üzere her bir prob setinin bileşimine bağlı olarak farklılık gösterebilir. Farklı seyreltmeler denemenizi öneririz; 200 nM'lik son bir seyreltme, bu deneylerde yetişkin dokular için en iyi sinyal-gürültü oranını verdi.
smFISH yöntemi ile TS odaklarında yeni sentezlenen RNA'ların sayısını ölçmek mümkündür. Bir gen aktif olarak kopyalandığında, TS'de aynı anda birden fazla yeni RNA üretilir. Sonuç olarak, TS'nin yoğunluğu olgun sitoplazmik RNA'nınkini aşacaktır ve bu özellik, nükleer lokalizasyonu ile birleştiğinde, TS'yi bireysel sitoplazmik RNA'lardan ayırt edebilir. Kas biyolojisi bağlamında, TS tespiti ve miktar tayini, aynı sinsityum içindeki kas çekirdekleri arasında belirli bir genin transkripsiyonunun senkronizasyonunu belirlemek için özellikle önemlidir. Bununla birlikte, bu hesaplama boru hattı, sitoplazmik mRNA ve TS sinyalleri arasında ayrım yapmak için tasarlanmamıştır. Alternatif olarak, bu smFISH yöntemini BayFISH veya FISH-quant29,30 gibi diğer köklü smFISH analiz araçlarıyla birleştirmenizi öneririz. Bu araçların, RNA agregalarının otomatik segmentasyon ve floresan yoğunluğu hesaplamalarını dikkate değer bir hassasiyetle kolaylaştırdığı kanıtlanmıştır.
Son olarak, smFISH, yüksek uzamsal çözünürlüğe sahip mRNA moleküllerini tespit ederken, aynı anda az sayıda mRNA'yı analiz etmekle sınırlıdır. merFISH (çoklanmış hata-sağlam floresan in situ hibridizasyon) gibi çok ölçekli yöntemler, çok sayıda farklı mRNA31'in aynı anda analiz edilmesini sağlar. Oligonükleotid probları ve hata düzeltme kodlarını birleştiren bu yöntem, tek hücrelerde yüzlerce RNA türünün tespitini kolaylaştırır.
Açıklamalar
Yazarların ifşa edecek herhangi bir çıkar çatışması yoktur.
Teşekkürler
Alain Vincent'a el yazmasını eleştirel okuması için teşekkür ederiz. Zfh1 antikorunu sağladığı için Ruth Lehmann'a teşekkür ederiz. MRic Görüntüleme Platformu'nda konfokal mikroskopi için Stephanie Dutertre ve Xavier Pinson'ın yardımlarını kabul ediyoruz. EL, ministère de l'enseignement supérieur et de la recherche scientifique'den doktora bursu ile desteklenmektedir. NA, AFM-Telethon Ph.D. burs 23846. TP, Chan Zuckerberg Girişimi DAF'ın T.P.'ye (2019-198009) verdiği hibe ile finanse edilmektedir. HB, CNRS, Atip Avenir programı ve AFM-Telethon trambolin hibesi 23108 tarafından desteklenmektedir.
Malzemeler
| Name | Company | Catalog Number | Comments |
| Confocal microscope SP8 | Leica | SP8 DMI 6000 | |
| DAPI | ThermoFisher | 62248 | |
| Double-sided tape | Tesa | 57910-00000 | |
| Dumont #55 Forceps | Fine Science Tools | 11255-20 | |
| Formaldehyde 16% | Euromedex | EM-15710 | |
| Mef2 antibody | DHSB | NA | |
| PBS 10x | Euromedex | ET330 | |
| RNaseZAP | Sigma-Aldrich | R2020 | |
| Stellaris probes | LGC | NA | |
| Stellaris RNA FISH Hybridization Buffer | LGC | SMF-HB1-10 | |
| Stellaris RNA FISH Wash Buffer A | LGC | SMF-WA1-60 | |
| Stellaris RNA FISH Wash Buffer B | LGC | SMF-WB1-20 | |
| Swann-Morton Blades | Fisher scientific | 0210 | |
| ThermoMixer | Eppendorf | 5382000015 | |
| Triton X-100 | Euromedex | 2000 | |
| UAS-mCD8::GFP line | Bloomington | RRID:BDSC_5137 | |
| Ultrapure water | Sigma-Aldrich | 95284 | |
| Watch Glass, Square, 1 5/8 in | Carolina | 742300 | |
| Zfh1 antibody | Gifted by Ruth Leahman | ||
| Zfh1-Gal4 | Bloomington | BDSC: 49924, FLYB: FBtp0059625 |
Referanslar
- Relaix, F., et al. Perspectives on skeletal muscle stem cells. Nature Communications. 12 (1), 692 (2021).
- Chal, J., Pourquie, O. Making muscle: skeletal myogenesis in vivo and in vitro. Development. 144 (12), 2104-2122 (2017).
- Taylor, M. V., Hughes, S. M. Mef2 and the skeletal muscle differentiation program. Seminars in Cell and Developmental Biology. 72, 33-44 (2017).
- Siles, L., et al. ZEB1 imposes a temporary stage-dependent inhibition of muscle gene expression and differentiation via CtBP-mediated transcriptional repression. Molecular and Cellular Biology. 33 (7), 1368-1382 (2013).
- Weitkunat, M., Schnorrer, F. A guide to study Drosophila muscle biology. Methods. 68 (1), 2-14 (2014).
- Boukhatmi, H. Drosophila, an integrative model to study the features of musclestem cells in development and regeneration. Cells. 10 (8), 2112 (2021).
- Bothe, I., Baylies, M. K. Drosophila myogenesis. Current Biology. 26 (17), R786-R791 (2016).
- Catalani, E., et al. RACK1 is evolutionary conserved in satellite stem cell activation and adult skeletal muscle regeneration. Cell Death Discovery. 8 (1), 459 (2022).
- Siles, L., Ninfali, C., Cortes, M., Darling, D. S., Postigo, A. ZEB1 protects skeletal muscle from damage and is required for its regeneration. Nature Communications. 10 (1), 1364 (2019).
- Boukhatmi, H., Bray, S. A population of adult satellite-like cells in Drosophila is maintained through a switch in RNA-isoforms. Elife. 7, e35954 (2018).
- Chaturvedi, D., Reichert, H., Gunage, R. D., VijayRaghavan, K. Identification and functional characterization of muscle satellite cells in Drosophila. Elife. 6, e30107 (2017).
- Gunage, R. D., Reichert, H., VijayRaghavan, K. Identification of a new stem cell population that generates Drosophila flight muscles. Elife. 3, e03126 (2014).
- Figeac, N., Daczewska, M., Marcelle, C., Jagla, K. Muscle stem cells and model systems for their investigation. Developmental Dynamics. 236 (12), 3332-3342 (2007).
- Aradhya, R., Zmojdzian, M., Da Ponte, J. P., Jagla, K. Muscle niche-driven insulin-Notch-Myc cascade reactivates dormant adult muscle precursors in Drosophila. Elife. 4, e08497 (2015).
- Schonbauer, C., et al. Spalt mediates an evolutionarily conserved switch to fibrillar muscle fate in insects. Nature. 479 (7373), 406-409 (2011).
- Bryantsev, A. L., Baker, P. W., Lovato, T. L., Jaramillo, M. S., Cripps, R. M. Differential requirements for Myocyte Enhancer Factor-2 during adult myogenesis in Drosophila. Developmental Biology. 361 (2), 191-207 (2012).
- Soler, C., Han, J., Taylor, M. V. The conserved transcription factor Mef2 has multiple roles in adult Drosophila musculature formation. Development. 139 (7), 1270-1275 (2012).
- Soler, C., Taylor, M. V. The Him gene inhibits the development of Drosophila flight muscles during metamorphosis. Mechanisms of Development. 126 (7), 595-603 (2009).
- Pinheiro, H., et al. mRNA distribution in skeletal muscle is associated with mRNA size. Journal of Cell Science. 134 (14), jcs256388 (2021).
- Denes, L. T., Kelley, C. P., Wang, E. T. Microtubule-based transport is essential to distribute RNA and nascent protein in skeletal muscle. Nature Communications. 12 (1), 6079 (2021).
- Li, H., et al. Fly Cell Atlas: A single-nucleus transcriptomic atlas of the adult fruit fly. Science. 375 (6584), eabk2432 (2022).
- Santos, M. D., et al. Extraction and sequencing of single nuclei from murine skeletal muscles. STAR Protocols. 2 (3), 100694 (2021).
- Raj, A., van den Bogaard, P., Rifkin, S. A., van Oudenaarden, A., Tyagi, S. Imaging individual mRNA molecules using multiple singly labeled probes. Nature Methods. 5 (10), 877-879 (2008).
- Femino, A. M., Fay, F. S., Fogarty, K., Singer, R. H. Visualization of single RNA transcripts in situ. Science. 280 (5363), 585-590 (1998).
- Raj, A., Tyagi, S. Detection of individual endogenous RNA transcripts in situ using multiple singly labeled probes. Methods in Enzymology. 472, 365-386 (2010).
- Stringer, C., Wang, T., Michaelos, M., Pachitariu, M. Cellpose: a generalist algorithm for cellular segmentation. Nature Methods. 18 (1), 100-106 (2021).
- Tinevez, J. Y., et al. TrackMate: An open and extensible platform for single-particle tracking. Methods. 115, 80-90 (2017).
- Legland, D., Arganda-Carreras, I., Andrey, P. MorphoLibJ: integrated library and plugins for mathematical morphology with ImageJ. Bioinformatics. 32 (22), 3532-3534 (2016).
- Mueller, F., et al. FISH-quant: automatic counting of transcripts in 3D FISH images. Nature Methods. 10 (4), 277-278 (2013).
- Gomez-Schiavon, M., Chen, L. F., West, A. E., Buchler, N. E. BayFish: Bayesian inference of transcription dynamics from population snapshots of single-molecule RNA FISH in single cells. Genome Biology. 18 (1), 164 (2017).
- Chen, K. H., Boettiger, A. N., Moffitt, J. R., Wang, S., Zhuang, X. RNA imaging. Spatially resolved, highly multiplexed RNA profiling in single cells. Science. 348 (6233), aaa6090 (2015).
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiDaha Fazla Makale Keşfet
This article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır